このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ミリ秒時間スケールでのタンパク質複合体のダイナミクスを調べるため体外蛍光共鳴エネルギー移動の使用
要約
蛋白質蛋白質の相互作用、生物学的システムにとって重要と拘束運動の研究は、ダイナミクスとタンパク質複合体の機能に対する洞察を提供します。蛋白質の蛍光共鳴エネルギー移動ストップト フロー法を適用した複合体の速度論的パラメーターを定量化する手法について述べる。
要約
タンパク質は生物学的システムの主な演算子と通常他のマクロ - または彼らの生物学的機能を遂行する小分子とやり取りします。このような相互作用は、非常にダイナミックな相互作用の亜単位は常に関連付けられているし、特定のレートで解離を意味することができます。高速方法に洞察力を提供定量的プルダウン バインディング機構研究、相互作用の強さを明らかにするようなテクニックを使用する結合親和性を測定しながら相互作用が発生し、それぞれの複合体が存在することができますどのくらい。さらに、相互作用の重要な知識を提供する他の因子によって調節されているメカニズムを明らかにするのに役立ちます蛋白質交換因子、薬物など、その他の要因の存在下での相互作用の動力学を測定、医学生物学の進歩。ここでは、高い組み込み協会率を持ち、別の蛋白質によってすぐに解離することができます蛋白質の複合体の結合動態を測定するためのプロトコルについて述べる。メソッドでは、蛍光共鳴エネルギー移動を使用して、in vitro タンパク質複合体の形成を報告でき高速協会・ ストップト フロー蛍光上でリアルタイムに複合体の解離を監視できます。この試金を使用して、複雑なタンパク質の協会と解離速度定数は定量化しました。
概要
生物活性最終的に最もうちで適切な生物学的機能の他のユーザーと対話する蛋白質によって実行されます。計算論的アプローチを使用して、650,000 〜1、人間蛋白質蛋白質の相互作用の総量を推定し、病2につながる多くの場合これらの相互作用の中断。酵母 2 ハイブリッド、分子蛍光補完、分割ルシフェラーゼなどのタンパク質間の相互作用を研究する細胞と個体のプロセスを制御するに重要な役割のための多数の方法を開発されています。補完、および co 免疫沈降分析3。これらのメソッドが検出および蛋白質蛋白質の相互作用を確認するのに優れている、彼らは通常定量的なしたがって、相互作用タンパク質の親和性について限られた情報を提供します。定量的プルダウン ・ リストを使用して、結合親和性 (例えば、解離定数Kd) を測定できますが、バインディングのキネティクスはからずも、それKdが不十分のため非常に低い場合に適用されます。信号対雑音比の4。表面プラズモン共鳴 (SPR) 法による拘束運動を定量化が比表面積、表面、反応5のバインドのプロパティを変えることができる潜在的に 1 つの反応の固定化が必要です。さらに、高速協会と解離率5を測定するための SPR の困難だし、SPR を使用して蛋白質の複合体の蛋白質の亜単位の交換イベントを特徴付ける適切ではないです。ここでは、蛋白質複雑なアセンブリおよびミリ秒のタイム スケールでの分解の率を測定できるようにする方法をについて説明します。このメソッドは、F 箱蛋白質交換要因6,7としてCullin-ssociated -Nedd8 -dissociated 蛋白質1 (Cand1)の役割を決定するために不可欠だった。
Cand1 は、Cullin リング ユビキチンリ ガーゼの大家族に属している Skp1•Cul1•F ボックス蛋白質 (SCF) E3 リガーゼのダイナミクスを調整します。SCFs は、リング ドメイン蛋白質 Rbx1 を結合する Cul1、F ボックス蛋白質が交換可能な基板を募集し、アダプター蛋白質 Skp18を介して Cul1 をバインドのカリンで構成されます。E3 リガーゼとして SCF は、その基板にユビキチンの共役を触媒して基板が F 箱蛋白質によって採用されていると Cul1 がユビキチン様タンパク質 Nedd89によって変更されたときそれがアクティブになります。Cand1 はそのまま Cul1 をバインドし、結合、それが両方の Skp1•F ボックス蛋白質 Cul1 の協会、Cul110、11,12,13Nedd8 活用妨害します。その結果、Cand1 は in vitro での SCF 活動の阻害剤のように見えたが、生物における Cand1 欠乏 vivo14,15,16の SCF の活動を調節する Cand1 の肯定的な役割を示唆している欠陥が発生,17. このパラドックスは最終的に Cul1、Cand1、Skp1•F ボックス蛋白質間の動的相互作用を明らかにする量的な研究によって説明されました。SCF と Cul1•Cand1 複合体の形成を検出蛍光共鳴エネルギー移動 (FRET) 試金を使用して、協会と解離速度定数 (k上とオフk、それぞれ) あった個別に測定します。測定は、Cand1 と Skp1•F ボックス蛋白質フォーム非常にタイトな複雑な Cul1、しかし、 kオフの SCF とは Cand1 で劇的に増加し、 kオフの Cul1•Cand1 が大幅に向上を明らかにしました。Skp1•F ボックス蛋白質6,7。これらの結果は、古い SCF 複合体から Cul1 のリサイクルを通じて新しい SCF 複合体の形成を触媒するタンパク質の交換要因として Cand1 の役割を定義する初期および重要なサポートを提供します。
ここでは、開発と Cul1•Cand1 複雑な7ダイナミクスを研究するフレットの試金を使用しての手順を提案して同じ原理は、様々 な生体分子のダイナミクスを研究に適用できます。フレットは、適切な波長で励起ドナーとドナーの発光スペクトルを重複する励起スペクトルを持つ受容体は 10-100 の範囲内で存在が発生します Å。励起状態は、ドナーの強度を減少させる受容体強度18を増加し、受容体に転送されます。フレット (E) の効率・ フォシュテル半径 (R0) およびドナーとアクセプター フルオロ (r) の間の距離によって異なります、によって定義されます: E = R06/(R06 + r6)。フェルスター半径 (R0) 配向角度を含むいくつかの要因に依存し、ドナー ・ アクセプターのペアとソリューションのスペクトルの重複使用19。リアルタイムでドナーの排出量の変化を監視し、高速でkとkオフの測定が可能、ストップト フロー蛍光にフレット アッセイを適用する効率的なフレットを確立する必要があることドナーの排出量の大幅な削減の結果。したがって、染料を付ける適切な蛍光染料とターゲット蛋白質のサイトのペアを選択することで効率的なフレットの設計は重要であり、このプロトコルで説明。
プロトコル
1. フレット アッセイをデザインします。
- 蛋白質構造データバンク (1U6G ファイル) から複雑な Cul1•Cand1 の構造ファイルをダウンロードします。
- ソフトフェアの複雑な Cul1•Cand1 の構造を表示します。
- Cand1 の最初のアミノ酸と Cul1 の最後のアミノ酸との間の距離を推定するソフトフェアのウィザードメニューの下で測定機能を使用 (図 1)。
- オンライン スペクトル ビューアーを読み込む (材料表参照) 励起と 7-アミノ-4-methylcoumarin (AMC) とフラッシュの発光スペクトルを同時に表示して (図 2)。AMC はフレット ドナー フラッシュは FRET アクセプターに注意してください。
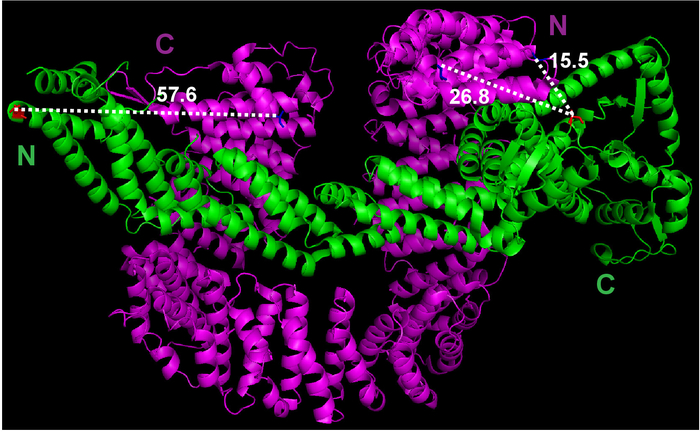
図 1: 結晶構造 Cul1•Cand1 と潜在的なサイトのラベル間の距離の測定。結晶構造ファイルだったから Protein Data Bank (ファイル 1U6G) をダウンロードし、ソフトフェアで表示。選択した原子間の測定は、ソフトフェアによって行われました。この図の拡大版を表示するのにはここをクリックしてください。
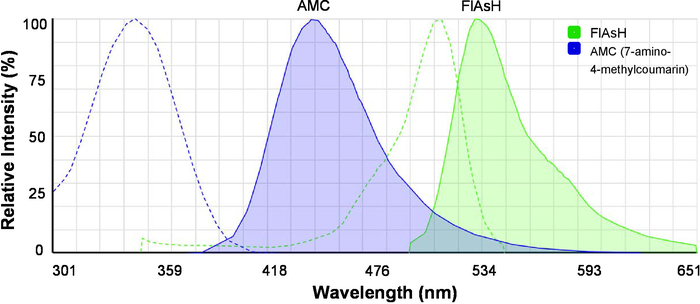
図 2: 励起とフレット用蛍光色素の発光スペクトル。AMC (7-アミノ-4-methylcoumarin) とフラッシュのスペクトルが表示されます。点線は励起スペクトルと発光スペクトルを示す実線。画像は蛍光 SpectraViewer によって生成されたよりわかりやすくするために変更されました。この図の拡大版を表示するのにはここをクリックしてください。
2. Cul1 の準備AMC•Rbx1、フレット供与体蛋白質
- エシェリヒア属大腸菌細胞の人間の Cul1sortase•Rbx1 を表現するためのプラスミドを構築します。エシェリヒア属大腸菌細胞の人間 Cul1•Rbx1 の共発現 2 種のプラスミドが前レポート20でくわしく述べることに注意してください。
- 標準 PCR, クローニング方法21,22を通じて Cul1 コード配列の 3' 末端に"LPETGGHHHHHH"(sortase 彼の6札) をコーディング DNA シーケンスを追加します。
- 遺伝子挿入が正確なことを確認する新しいプラスミッドをシーケンスします。
- エシェリヒア属大腸菌細胞における共同エクスプレス Cul1sortase•Rbx1。メソッドは、以前のレポート20から派生されます。
- ミックス 100 ng BL21 の 2 種のプラスミドの熱を使用して共同の変形のため化学的に有能なセルを (DE3) 衝撃法23。一晩 100 μ g/mL アンピシリンと 37 ° C で 34 μ G/ml クロラムフェニ コールを含む LB 寒天培地プレート上のセルを成長します。
- 新しく変換されたコロニーと LB 文化の 50 mL を接種して 250 rpm の揺れで 37 ° C で一晩成長します。これはスターター文化を与えます。
- 接種する 6 のフラスコ、それぞれの LB 培地 1 L に 5 mL スターター文化そして揺れ外径 φ600まで 〜 1.0 250 rpm と 37 ° C で育ちます。16 ° C に文化を冷却し、イソプロピル-β-D-thiogalactoside (IPTG) を 0.4 mM に追加します。一晩 250 rpm の揺れで 16 ° C で文化を保ちます。
- 15 分間 5,000 × gで遠心分離による大腸菌の細胞を収穫し、50 mL の円錐管の細胞ペレットを収集します。
注: 細胞ペレットは蛋白質の浄化のために処理することができます。 または蛋白質精製の手順に進む前に-80 ° C で凍結します。
- 複雑な Cul1 のsortaseの •Rbx1 の浄化。このメソッドは、以前のレポート20から派生されます。
- Cul1sortase•Rbx1 を発現する大腸菌細胞のペレットに 50 mL の換散バッファー (30 mM トリス-HCl、200 mM の NaCl、5 mM DTT、10% グリセロール 1 タブレットのプロテアーゼ阻害剤カクテル、pH 7.6) を追加します。
- 氷 50% 振幅で超音波で細胞を溶解させます。1 秒オン/1 秒オフの間を交互し、3 分間実行します。
- 2.3.2 のステップ 2 〜 3 倍を繰り返します。
- 50 mL 遠心チューブに細胞ライセートを転送し、45 分 25,000 × gで遠心分離によって細胞の残骸を削除します。
- 明確な細胞の 5 ml の 2 h の 4 ° C、グルタチオン ビーズのライセートを孵化させなさい。
- 4 ° C で 2 分間 1,500 × gでビーズ ライセートの混合物を遠心分離します。上澄みを除去します。
- 5 ml の溶解バッファー (ないプロテアーゼ阻害剤) とのビーズを洗浄し、4 ° C で 2 分間 1,500 × gで遠心分離後、上清を除去
- 2.3.7 手順を繰り返します 2 倍。
- 洗浄のビーズに 3 mL の溶解バッファーを追加し、空の列にビーズ スラリーを転送します。
- 列には、5 mL の溶出バッファー (50 mM トリス-HCl、200 mM の NaCl、10 mM 減少グルタチオン、pH 8.0) を追加します。10 分間インキュベートし、溶出液を収集します。
- 3-4 x 2.3.10 のステップを繰り返します。
- 5 mg/mL トロンビンを 200 μ l 添加 (材料の表を参照してください) グルタチオン ビーズから溶出液を 4 ° C で一晩インキュベートし、
注: プロトコルはここで一時停止することができます。 - バッファー A の蛋白質のサンプル (25 mM HEPES、1 mM DTT、5% のグリセロール、pH 6.5) を 3 倍希釈します。
- 陽イオン交換クロマトグラフィー コラムを平衡させ (材料表参照) バッファー A. FPLC システムに
- 0.5 mL/分の流量で釣合いの陽イオン交換クロマトグラフィー カラムに蛋白質のサンプルをロードします。
- 40 mL に塩化ナトリウムのこう配をもつタンパク質を溶出バッファー A と 1 mL/分の流量で 0 ~ 50% バッファー B (25 mM 1 M NaCl、1 mM DTT、5% のグリセロール、HEPES pH 6.5) を混合することによって。
- SDS ページ24使用の異なる画分の溶出蛋白質を確認します。
注: プロトコルはここで一時停止することができます。 - Cul1sortase•Rbx1 を含有する溶出画分をプールします。
- 限外ろ過膜 (30 kDa カットオフ) バッファーを渡すことによって 2.5 mL にプールされた Cul1sortase•Rbx1 サンプルを集中します。
- Sortase を介した transpeptidation21,22を通じて Cul1 の C 末端に AMC を追加します。
- Sortase バッファー (50 mM トリス-HCl、150 mM の NaCl、10 mM CaCl2、pH 7.5) に Cul1sortase•Rbx1 サンプルではバッファーを変更脱塩カラムを使用して (材料の表を参照してください)。
- 25 ml sortase バッファーの脱塩カラムを平衡します。
- Cul1sortase•Rbx1 サンプルの 2.5 mL を列に読み込みます。流れを破棄します。
- Sortase バッファーの 3.5 ml サンプルを溶出します。流れを収集します。
- Sortase バッファー内 Cul1sortase•Rbx1 ソリューションの 900 μ L、600 μ M 精製 sortase ソリューションの 100 μ L と 25 mM GGGGAMCペプチドの 10 μ L を追加します。暗い一晩で 30 ° C で反応混合物を孵化させなさい。メモこの手順が Cul1 を生成するAMC•Rbx1。
注意: 蛍光染料は光、だから可能な限り蛋白質およびサンプル準備の間に周囲の光にさらすことを避けるために敏感です。
注: プロトコルはここで一時停止することができます。 - Ni NTA アガロース ビーズの 50 μ L を反応混合物に追加し、30 分間室温でインキュベートします。
- 2 分間 5,000 × gで遠心分離によって Ni NTA アガロース ビーズをペレットし、上澄みを収集します。
- サイズ排除クロマトグラフィー コラムを平衡させ (材料の表を参照してください) FPLC システム上のバッファー (トリス-HCl、100 mM の NaCl、1 mM DTT、10% グリセロール 30 mM) を持つ。
- すべての Cul1 の読み込みサイズ排除クロマトグラフィー コラムのAMC•Rbx1 サンプル。1.5 x 列の量バッファーで溶出します。
- SDS ページ24によって溶出画分を確認してください。
- Cul1 を含有する溶出画分をプールAMC•Rbx1。
- 280 その吸光度を用いたタンパク質濃度を測定分光光度計の nm。
- タンパク質溶液の約数と-80 ° C でストア
注: プロトコルはここで一時停止することができます。
- Sortase バッファー (50 mM トリス-HCl、150 mM の NaCl、10 mM CaCl2、pH 7.5) に Cul1sortase•Rbx1 サンプルではバッファーを変更脱塩カラムを使用して (材料の表を参照してください)。
3.フラッシュCand1、 FRET アクセプター蛋白質の作製
メモ: この部分の手順のほとんどは、ステップ 2 と同じです。異なる条件は、以下で詳しく説明します。
- エシェリヒア属大腸菌細胞の人間のTetraCysCand1 を表現するためのプラスミドを構築します。
- 通常の PCR25 Cand1 の 15番目のアミノ酸の中の前に"CCPGCCGSG"(tetracysteine/TetraCys タグ) をコーディング DNA シーケンスを追加 (プライマー シーケンス: TGCTGTCCGGGCTGCTGCGGCAGCGGCATGACATCCAGCGACAAGGACTTTAG;CTAACTAGTGTCCATTGATTCCAAG)。
- PGEX 4 t 2 ベクトルに PCR の製品を挿入します。遺伝子挿入が正確でフレームを確認するプラスミドをシーケンスします。
- プラスミッドはロゼッタの有能なセルに変換ことを除いては、2.2 の手順と同じ方法でTetraCysCand1inエシェリヒア属大腸菌のセルを表現します。
- 大腸菌の細胞からTetraCysCand1 の浄化。
- エシェリヒア属大腸菌細胞ペレットを溶解し、 TetraCysCand1using グルタチオン ビーズを抽出します。これらの手順は、手順 2.3.1–2.3.12 と同じです。
- 3 つのフォールドによってバッファー C (50 mM トリス-HCl、1 mM DTT、5% のグリセロール、pH 7.5)、グルタチオン ビーズからタンパク質の溶出液を希釈します。陰イオン交換クロマトグラフィー カラムの平衡 (材料表参照) FPLC システム バッファー C と負荷 0.5 mL/分の流量で希薄蛋白質のサンプルの。
- 40 mL に塩化ナトリウムのこう配をもつタンパク質を溶出バッファー C と 1 mL/分の流量で 0 ~ 50 %d バッファー (50 mM 1 M NaCl、1 mM DTT、5% のグリセロール、トリス塩酸 pH 7.5) を混合することによって。SDS ページ24、使用の異なる画分の溶出蛋白質を確認し、 TetraCysCand1 を含む画分をプールします。TetraCysCand1 が無料 GST より大量保有を持っていることに注意してください。
- 限外ろ過膜 (30 kDa カットオフ) バッファーを渡すことによってプールTetraCysCand1 サンプルを集中します。
- ラベル バッファー (20 mM トリス-HCl、100 mM の NaCl、2 mM TCEP、1 mM EDTA、5% のグリセロール) とサイズ排除クロマトグラフィー コラムを平衡させ、FPLC システムに。TetraCysCand1 サンプル (各時間 500 μ L) をロードし、SDS ページ24溶出画分をチェックします。
- TetraCysCand1 を含むすべての画分をプール、限外濾過膜 (30 kDa カットオフ) バッファーを渡すことによって 〜 40 μ M に蛋白質を集中します。280 その吸光度を用いたタンパク質濃度を推定 nm。-80 ° C で 50 μ L の因数としてタンパク質を格納します。
注: プロトコルはここで一時停止することができます。
- Cand1 のフラッシュの準備。
- フラッシュ溶液 1 μ L を追加 (材料の表を参照) を 50 μ L TetraCysCand1 ソリューション。
- よく混ぜるし、Cand1 のフラッシュを取得する 1-2 h、暗所に室温で混合物を孵化させなさい。
注: プロトコルはここで一時停止することができます。
4. Cand1、フレット チェイス蛋白質の作製
注: 蛋白質の準備のプロトコルは、次の変更でステップ 3 に似ています。
- PGEX 4 t 2 ベクトルにフルレングスの Cand1 のコーディングのシーケンスを挿入します。
- 3.3.5 30 mM トリス-HCl、100 mM の NaCl、1 mM DTT、10% グリセロールを格納するバッファーへのステップで使用するバッファーを変更します。
- 3.3.7 と 3.3.8 の手順を排除します。
5. テストし、フレット アッセイを確認
- 30 mM 100 mM の NaCl、0.5 mM DTT、1 mg/mL オヴァルブミンの pH 7.6、トリス塩酸を含むフレット バッファーを準備し、常温で使用します。
- Cul1 間のフレットをテストAMC•Rbx1 と、蛍光のフラッシュCand1。
- フレット バッファーの 300 μ L は、追加 Cul1AMC•Rbx1 (フレット ドナー) 70 の最終的な集中に nM。キュベットにソリューションを転送します。
- キュヴェットを蛍光のサンプル ホルダーに。350 nm の励起光を試料し、400 から発光信号をスキャン 600 nm nm 1 nm 刻みで。
- 5.2.1 手順を繰り返しますが、Cand1 のフラッシュに変更 Cul1AMC•Rbx1 (FRET アクセプター)。5.2.2 の手順と同じ方法を使用してフラッシュCand1 サンプルをスキャンします。
- (省略可能)510 nm 励起光とフラッシュCand1 を興奮し、500 から発光信号をスキャン 650 nm nm。
- 300 μ L フレット バッファーに追加両方 Cul1AMC•Rbx1 と 70 の最終的な集中にフラッシュCand1 nM。5.2.2 の手順と同じ方法で、サンプルを分析します。
- Cul1 間のフレットを確認AMC•Rbx1 ・ チェイス (ラベル Cand1) 蛋白質 (図 3) を追加することによって Cand1 をフラッシュします。
- フレット バッファー 300 μ、70 を追加 nM Cul1AMC•Rbx1 と 700 nM Cand1。5.2.2 の手順とサンプル放出をスキャンします。
- フレット バッファー 300 μ、70 を追加 nMフラッシュCand1、700 nM Cand1。5.2.2 の手順とサンプル放出をスキャンします。
- フレット バッファー 300 μ、70 を追加 nM Cul1AMC•Rbx1 と 70 nMフラッシュCand1、5 分間室温でサンプルをインキュベートし、。追加 700 nM Cand1、添加後すぐにスキャン ステップ 5.2.2 のようにサンプル放出)。この手順のステップ 5.2.5 に似ていることに注意してください。
- 300 μ L フレット バッファーで順次追加 70 nM Cul1AMC•Rbx1、700 nM、Cand1、70 nMフラッシュCand1。5 分間室温でサンプルをインキュベートし、ステップ 5.2.2 のようにサンプル放出をスキャンします。これはチェイスの例 (図 3の緑線) に注意してください。
6. Cul1•Cand1 の (kで) 協会速度定数を測定します。
注: 以前のレポート26ストップト フロー蛍光を動作の詳細が記載されています。
- ストップト フロー蛍光を測定に備えます。
- 製造元の指示に従ってストップト フロー蛍光を入れます。
- 350 nm と 450 nm の発光を可能にするバンドパス フィルターを通過する光し、500-650 nm 放射光をブロック使用に励起された光を設定します。
- サンプル バルブを埋める位置に維持し、水で満たされた 3 mL シリンジを接続します。上下に数回サンプル シリンジ ドライブを移動することによって水で 2 つ (A と B) サンプル注射器を洗います。この手順で使用されるすべての水を捨てます。
- サンプル バルブを埋める位置に維持し、フレット バッファーでいっぱい 3 mL シリンジを接続します。上下にいくつかの時間サンプル シリンジ ドライブを移動することによってフレット バッファーと 2 つのサンプルの注射器を洗います。この手順で使用されるすべてのフレット バッファーを破棄します。
- コントロール測定 (図 4) を取る。
- 3 mL 注射器を接続し、フレット バッファーで 100 nM Cul1AMC•Rbx1 を注射器 A を読み込みます。ドライブ位置にサンプル バルブを回します。
- 3 mL シリンジを接続し、フレット バッファーとシリンジ B をロードします。ドライブ位置にサンプル バルブを回します。
- ソフトウェアの取得の下のコントロール パネルを使用する (シリンジ A とシリンジ B からサンプルの等量をミックス) 5 つのショット ストップト フロー蛍光に結果を記録も。
- ソフトウェアで、取得の下のコントロール パネルを開き、プログラムの Cul1 の排出量を記録するAMC以上 60 s。1 つのショットを取る。
- 6.2.4 手順を繰り返します 2 倍。
- サンプル バルブを埋める位置に。空の注射器 B とフレット バッファーで洗浄します。
- メジャーの Cul1•Cand1 の協会速度定数 (kobs) を観察 (図 4 b)。
- 注射器 A にサンプルをおいて 6.2.1 の手順と同じです。
- 3 mL 注射器を接続し、100 で注射器 B フレット バッファーのフラッシュCand1 nM。ドライブ位置にサンプル バルブを回します。
- ソフトウェアの取得の下でコントロール パネルを使用して、結果を記録することがなく 5 つのショットを取る。
- ソフトウェアで、取得の下のコントロール パネルを開き、プログラムの Cul1 の排出量を記録するAMC以上 60 s。1 つのショットを取る。
- 6.3.4 手順を繰り返します 2 倍。
- 空の注射器 B とフレット バッファーで洗浄します。
- 数回フレット バッファーのフラッシュCand1 の濃度の増加と手順 6.3.1–6.3.6 を繰り返します。
- 単一指数曲線に各ショットから時間をかけて測定した蛍光信号の変化 (△) に適応します。これは各測定におけるkobsを与えるだろうし、単位は s-1。この算定の基礎は、以前レポート27でよく議論されているに注意してください。
- 平均値と各フラッシュCand1 濃度使用kobsの標準偏差を計算します。Cand1 濃度 (図 4) に対する平均kobsをプロットし、 kのCul1•Cand1 の M-1の-1単位で、直線の傾きを表します。
7. Skp1•F ボックス蛋白質の存在下での Cul1•Cand1 (オフk) 解離反応の速度定数を測定します。
注: この手順は、次の変更手順 6 と同様です。
- 入力位置の下で、注射器で負荷 100 nM Cul1AMC•Rbx1 のソリューションと 100 nMフラッシュCand1 フレット バッファー。「ドライブ」の位置にサンプル バルブを回します。
- 注射器 B の記入位置の下 Skp1•Skp2 (次の以前のレポート20準備) のソリューションを読み込みます。「ドライブ」の位置にサンプル バルブを回します。
- ソフトウェアで、取得の下のコントロール パネルを開き、プログラムの Cul1 の排出量を記録するAMC以上 30 s。1 つのショットを取る。蛍光信号はシリンジ A とシリンジ B (図 5) からソリューションを混合した後時間の経過と共に増加します。
結果
Cul1 間のフレットをテストするAMCとフラッシュCand1、我々 はまず 70 の発光強度を決定 nM Cul1AMC (ドナー) と 70 nMフラッシュCand1 (受容体)、それぞれ (図 3 a-c、青いライン)。各分析だけで 1 つの発光ピークは現状と Cand1 のフラッシュの発光 (受容体) は低かった。とき 70 nM の各 Cul1 のAMCと...
ディスカッション
フレットは、勉強し、生物学的システム19を理解に非常に役立つは物理現象です。ここでは、テストのフレットを使用して 2 つの相互作用の蛋白質の結合の動力学を調査するためのプロトコルを提案する.フレットをデザインするとき、我々 は 3 つの主要な要因を考慮: ドナーの排出量と受容体の励起、2 つの同時と fluorophores28の双極子の方向間の距離間ス?...
開示事項
著者が明らかに何もありません。
謝辞
フレットの試金の開発に洞察力に富んだ議論ありがとう集 Ou シャン (カリフォルニア工科大学)。X.L.、Y.Z.、m. g. Y.Z. にパデュー大学からスタートアップ資金によって資金が供給された、X.L.This 作業はパデュー大学センターからのシード補助金によって植物生物学の部分で支えられました。
資料
| Name | Company | Catalog Number | Comments |
| Anion exchange chromatography column | GE Healthcare | 17505301 | HiTrap Q FF anion exchange chromatography column |
| Benchtop refrigerated centrifuge | Eppendorf | 2231000511 | |
| BL21 (DE3) Competent Cells | ThermoFisher Scientific | C600003 | |
| Calcium Chloride | Fisher Scientific | C78-500 | |
| Cation exchange chromatography column | GE Healthcare | 17505401 | HiTrap SP Sepharose FF |
| Desalting Column | GE Healthcare | 17085101 | |
| Floor model centrifuge (high speed) | Beckman Coulter | J2-MC | |
| Floor model centrifuge (low speed) | Beckman Coulter | J6-MI | |
| Fluorescence SpectraViewer | ThermoFisher Scientific | https://www.thermofisher.com/us/en/home/life-science/cell-analysis/labeling-chemistry/fluorescence-spectraviewer.html | |
| FluoroMax fluorimeter | HORIBA | FluoroMax-3 | |
| FPLC | GE Healthcare | 29018224 | |
| GGGGAMC peptide | New England Peptide | custom synthesis | |
| Glutathione beads | GE Healthcare | 17075605 | |
| Glycerol | Fisher Scientific | G33-500 | |
| HEPES | Fisher Scientific | BP310-100 | |
| Isopropyl-β-D-thiogalactoside (IPTG) | Fisher Scientific | 15-529-019 | |
| LB Broth | Fisher Scientific | BP1426-500 | |
| Ni-NTA agarose | Qiagen | 30210 | |
| Ovalbumin | MilliporeSigma | A2512 | |
| pGEX-4T-2 vector | GE Healthcare | 28954550 | |
| Protease inhibitor cocktail | MilliporeSigma | 4693132001 | |
| Reduced glutathione | Fisher Scientific | BP25211 | |
| Refrigerated shaker | Eppendorf | M1282-0004 | |
| Rosetta Competent Cells | MilliporeSigma | 70953-3 | |
| Size exclusion chromatography column | GE Healthcare | 28990944 | Superdex 200 10/300 GL column |
| Sodium Chloride (NaCl) | Fisher Scientific | S271-500 | |
| Stopped-flow fluorimeter | Hi-Tech Scientific | SF-61 DX2 | |
| TCEP·HCl | Fisher Scientific | PI20490 | |
| Thrombin | MilliporeSigma | T4648 | |
| Tris Base | Fisher Scientific | BP152-500 | |
| Ultrafiltration membrane | MilliporeSigma | UFC903008 | Amicon Ultra-15 Centrifugal Filter Units, Ultra-15, 30,000 NMWL |
参考文献
- Stumpf, M. P. H., et al. Estimating the size of the human interactome. Proceedings of the National Academy of Sciences of the United States of America. 105 (19), 6959-6964 (2008).
- Kuzmanov, U., Emili, A. Protein-protein interaction networks: probing disease mechanisms using model systems. Genome Medicine. 5 (4), 37 (2013).
- Titeca, K., Lemmens, I., Tavernier, J., Eyckerman, S. Discovering cellular protein-protein interactions: Technological strategies and opportunities. Mass Spectrometry Reviews. , 1-33 (2018).
- Lapetina, S., Gil-Henn, H. A guide to simple, direct, and quantitative in vitro binding assays. Journal of Biological Methods. 4 (1), 62 (2017).
- Zheng, X., Bi, C., Li, Z., Podariu, M., Hage, D. S. Analytical methods for kinetic studies of biological interactions: A review. Journal of Pharmaceutical and Biomedical Analysis. 113, 163-180 (2015).
- Pierce, N. W., et al. Cand1 promotes assembly of new SCF complexes through dynamic exchange of F box proteins. Cell. 153 (1), 206-215 (2013).
- Liu, X., Reitsma, J. M., Mamrosh, J. L., Zhang, Y., Straube, R., Deshaies, R. J. Cand1-Mediated Adaptive Exchange Mechanism Enables Variation in F-Box Protein Expression. Molecular Cell. 69 (5), 773-786 (2018).
- Zheng, N., et al. Structure of the Cul1-Rbx1-Skp1-F boxSkp2 SCF ubiquitin ligase complex. Nature. 416 (6882), 703-709 (2002).
- Kleiger, G., Deshaies, R. Tag Team Ubiquitin Ligases. Cell. 166 (5), 1080-1081 (2016).
- Zheng, J., et al. CAND1 binds to unneddylated CUL1 and regulates the formation of SCF ubiquitin E3 ligase complex. Molecular Cell. 10 (6), 1519-1526 (2002).
- Hwang, J. W., Min, K. W., Tamura, T. A., Yoon, J. B. TIP120A associates with unneddylated cullin 1 and regulates its neddylation. FEBS Letters. 541 (1-3), 102-108 (2003).
- Min, K. W., Hwang, J. W., Lee, J. S., Park, Y., Tamura, T., Yoon, J. B. TIP120A associates with cullins and modulates ubiquitin ligase activity. Journal of Biological Chemistry. 278 (18), 15905-15910 (2003).
- Goldenberg, S. J., et al. Structure of the Cand1-Cul1-Roc1 complex reveals regulatory mechanisms for the assembly of the multisubunit cullin-dependent ubiquitin ligases. Cell. 119 (4), 517-528 (2004).
- Chuang, H. W., Zhang, W., Gray, W. M. Arabidopsis ETA2, an apparent ortholog of the human cullin-interacting protein CAND1, is required for auxin responses mediated by the SCF(TIR1) ubiquitin ligase. Plant Cell. 16 (7), 1883-1897 (2004).
- Feng, S., et al. Arabidopsis CAND1, an Unmodified CUL1-Interacting Protein, Is Involved in Multiple Developmental Pathways Controlled by Ubiquitin/Proteasome-Mediated Protein Degradation. the Plant Cell Online. 16 (7), 1870-1882 (2004).
- Cheng, Y., Dai, X., Zhao, Y. AtCAND1, a HEAT-repeat protein that participates in auxin signaling in Arabidopsis. Plant Physiology. 135 (June), 1020-1026 (2004).
- Lo, S. -. C., Hannink, M. CAND1-Mediated Substrate Adaptor Recycling Is Required for Efficient Repression of Nrf2 by Keap1. Molecular and Cellular Biology. 26 (4), 1235-1244 (2006).
- Okamoto, K., Sako, Y. Recent advances in FRET for the study of protein interactions and dynamics. Current Opinion in Structural Biology. 46, 16-23 (2017).
- Hussain, S. A. An introduction to fluorescence resonance energy transfer (FRET). arXiv preprint. , (2009).
- Li, T., Pavletich, N. P., Schulman, B. A., Zheng, N. High-level expression and purification of recombinant SCF ubiquitin ligases. Methods in Enzymology. 398 (1996), 125-142 (2005).
- Popp, M. W., Antos, J. M., Grotenbreg, G. M., Spooner, E., Ploegh, H. L. Sortagging: A versatile method for protein labeling. Nature Chemical Biology. 3 (11), 707-708 (2007).
- Antos, J. M., Ingram, J., Fang, T., Pishesha, N., Truttmann, M. C., Ploegh, H. L. Site-Specific Protein Labeling via Sortase-Mediated Transpeptidation. Current Protocols in Protein Science. 89, (2017).
- Froger, A., Hall, J. E. Transformation of Plasmid DNA into E. coli Using the Heat Shock Method. Journal of Visualized Experiments. , (2007).
- Simpson, R. J. SDS-PAGE of Proteins. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Kleiger, G., Saha, A., Lewis, S., Kuhlman, B., Deshaies, R. J. Rapid E2-E3 Assembly and Disassembly Enable Processive Ubiquitylation of Cullin-RING Ubiquitin Ligase Substrates. Cell. 139 (5), 957-968 (2009).
- Biro, F. N., Zhai, J., Doucette, C. W., Hingorani, M. M. Application of Stopped-flow Kinetics Methods to Investigate the Mechanism of Action of a DNA Repair Protein. Journal of Visualized Experiments. (37), 2-8 (2010).
- Patel, J. T., Belsham, H. R., Rathbone, A. J., Friel, C. T. Use of Stopped-Flow Fluorescence and Labeled Nucleotides to Analyze the ATP Turnover Cycle of Kinesins. Journal of Visualized Experiments. (92), 1-6 (2014).
- Bajar, B. T., Wang, E. S., Zhang, S., Lin, M. Z., Chu, J. A guide to fluorescent protein FRET pairs. Sensors (Switzerland). 16 (9), 1-24 (2016).
- Chen, A. K., Cheng, Z., Behlke, M. A., Tsourkas, A. Assessing the sensitivity of commercially available fluorophores to the intracellular environment. Analytical Chemistry. 80 (19), 7437-7444 (2008).
- Lin, C. T., Rorabacher, D. B. Mathematical approach for stopped-flow kinetics of fast second-order reactions involving inhomogeneity in the reaction cell. Journal of Physical Chemistry. 78 (3), 305-308 (1974).
- Toseland, C. P., Geeves, M. A. Rapid Reaction Kinetic Techniques. Fluorescent Methods for Molecular Motors. , 49-65 (2014).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: Synthesis and biological applications. Journal of the American Chemical Society. 124 (21), 6063-6076 (2002).
- Lin, C. W., Ting, A. Y. Transglutaminase-catalyzed site-specific conjugation of small-molecule probes to proteins in vitro and on the surface of living cells. Journal of the American Chemical Society. 128 (14), 4542-4543 (2006).
- Yin, J., et al. Genetically encoded short peptide tag for versatile protein labeling by Sfp phosphopantetheinyl transferase. Proceedings of the National Academy of Sciences of the United States of America. 102 (44), 15815-15820 (2005).
- Diaspro, A., Chirico, G., Usai, C., Ramoino, P., Dobrucki, J. Photobleaching. Handbook Of Biological Confocal Microscopy. , 690-702 (2006).
- Aoki, K., Kamioka, Y., Matsuda, M. Fluorescence resonance energy transfer imaging of cell signaling from in vitro to in vivo: Basis of biosensor construction, live imaging, and image processing. Development, Growth & Differentiation. 55 (4), 515-522 (2013).
- Kilic, S., et al. Single-molecule FRET reveals multiscale chromatin dynamics modulated by HP1α. Nature Communications. 9 (1), (2018).
- Shen, K., Arslan, S., Akopian, D., Ha, T., Shan, S. O. Activated GTPase movement on an RNA scaffold drives co-translational protein targeting. Nature. 492 (7428), 271-275 (2012).
- Bajar, B. T., et al. Improving brightness and photostability of green and red fluorescent proteins for live cell imaging and FRET reporting. Scientific Reports. 6 (February), 1-12 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved