Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Использование в Vitro флуоресценции резонанс передачи энергии для изучения динамики белковых комплексов в масштабе времени миллисекунду
В этой статье
Резюме
Белок белковых взаимодействий являются критическими для биологических систем, и исследования кинетики привязки обеспечивают понимание динамики и функции белковых комплексов. Мы описываем метод, который дает количественную оценку кинетических параметров белкового комплекса, с помощью передачи энергии резонансной флюоресценции и остановить поток техники.
Аннотация
Белки являются основным операторам биологических систем, и они обычно взаимодействуют с другими макро - или малых молекул выполнять свои биологические функции. Такое взаимодействие может быть высокодинамичный, означает взаимодействующих подразделений постоянно связаны и отделить некоторые ставкам. В то время как измерения сродство с использованием методов, таких как количественные выпадающем показывает силу взаимодействия, изучение кинетики привязки предоставляет информацию о том, как быстро происходит взаимодействие и как долго каждый комплекс может существовать. Кроме того, измерения кинетика взаимодействия присутствии дополнительным фактором, как фактор обмена белков или наркотиков, помогает раскрыть механизм, по которому взаимодействие регулируется другим фактором, предоставляя важные знания для улучшения биологических и медицинских исследований. Здесь мы описываем протокол для измерения кинетики привязки комплекс белков, который имеет высокое внутренне присущей ей связи скорость и может быть отделен быстро по другим белком. Этот метод использует передачи энергии резонансная флюоресценция сообщить формирования комплекса белков в пробирке, и позволяет мониторинга быстро ассоциации и диссоциация комплекса в режиме реального времени на fluorimeter остановить поток. Используя этот assay, количественно ассоциации и диссоциации константы скорости комплекс белков.
Введение
В конечном счете биологическая деятельность осуществляется белков, Последнее из которых взаимодействовать с другими для надлежащего биологических функций. Используя вычислительный подход, общее количество белок белковых взаимодействий в человека оценивается в1~ 650 000, и нарушения этих взаимодействий часто приводит к заболеваниям2. Из-за их существенно важную роль в контроле процессов клеточной и научные многочисленные методы были разработаны для изучения взаимодействия протеин протеина, такие как дрожжи 2 гибрида, bimolecular флуоресценции комплементарности, Сплит Люцифераза дополнения и co иммунопреципитация пробирного3. Хотя эти методы являются хорошими на открытие и подтверждение белок белковых взаимодействий, они обычно неколичественного и таким образом предоставляют ограниченную информацию о сходство между взаимодействия партнеров белка. Количественные тянуть Даунс может использоваться для измерения сродство (например, Константа диссоциации Kd), но он не измерить кинетика привязки не могут быть применены, когда Kd очень низка из-за неадекватной Соотношение сигнал шум4. Спектроскопия резонанса (СРП) поверхностного плазмон количественно кинетики привязки, но она требует удельной поверхности и иммобилизация одной реагент на поверхности, которые потенциально могут изменить свойство привязки реагент5. Кроме того это трудно для СРП для измерения быстро ассоциации и диссоциации ставки5, и это не целесообразно использовать СРП для характеризуют событие обмена субблоков протеина в комплекс белков. Здесь мы описываем метод, который позволяет измерения количества белка сложные сборки и разборки в масштабе времени миллисекунду. Этот метод имеет важное значение для определения роли Cullin -ssociated -Nedd8 -дissociated белок 1 (Cand1) как F-box белка обмен фактор6,7.
Cand1 регулирует динамика Skp1•Cul1•F-бокс белка (SCF) E3 ligases, которые принадлежат к большой семье Каллин-кольцо убиквитин ligases. Заявок состоят из Каллин Cul1, который связывает кольцо домен белка Rbx1, и взаимозаменяемыми F-box белка, который вербует субстратов и связывает Cul1 через адаптер белок СКП18. Лигаза E3 SCF катализирует спряжение убиквитин к его основанию, а он активируется, когда субстрат завербован F-box белка, и когда Cul1 изменяется убиквитин подобных белков Nedd89. Cand1 связывает неизмененном Cul1, и после привязки, она разрушает как ассоциации белка Skp1•F-бокс с Cul1 и спряжение Nedd8 Cul110,11,12,13. В результате Cand1, по-видимому, ингибитор SCF активности в пробирке, но Cand1 дефицита в организмах причиной дефектов, которые предполагает позитивную роль Cand1 в регулировании деятельности компаний СКФ в vivo14,,1516 , 17. Этот парадокс объясняется наконец количественного исследования, которые показали динамического взаимодействия между Cul1, Cand1 и Skp1•F-бокс белка. С помощью флуоресценции резонанс энергии передачи (ЛАДА) анализов, которые обнаруживают формирования СКФ и Cul1•Cand1 комплексов, ассоциации и диссоциации оценить константы (kна и kот, соответственно) были измеряется в отдельности. Измерения показали, что Cand1 и Skp1•F-бокс белка форме крайне сжатые комплекс с Cul1, но kот SCF резко увеличивается на Cand1 и kот Cul1•Cand1 резко увеличилось Skp1•F-бокс белок6,7. Эти результаты обеспечивают поддержку первоначальных и критических для определения роли Cand1 как фактор обмена белков, который катализирует образование комплексов новых SCF путем переработки Cul1 от старых СКФ комплексами.
Здесь мы представляем процедура разработки и использования ладу assay для изучения динамики Cul1•Cand1 комплекс7, и тот же принцип может применяться для изучения динамики различных биомолекул. ЛАДУ происходит, когда донор возбужденных с соответствующей длины волны, и акцептора с перекрывающихся доноров спектр излучения спектра возбуждения присутствует на расстоянии 10-100 Å. Возбужденное состояние передается акцептор, тем самым уменьшается интенсивность доноров и увеличения интенсивности акцептора18. Эффективность ладу (E) зависит от радиуса Фёрстер (R0) и расстояние между доноров и акцепторов флуорофоров (r) и определяется: E = R06/ (R0 6 + r6). Радиус Фёрстер (R0) зависит от нескольких факторов, включая угловые ориентацию диполя, спектральные совпадения донорно акцепторной пара и решение используется19. Применять assay ладу на остановился потока fluorimeter, который отслеживает изменения выбросов доноров в режиме реального времени и обеспечивает измерение быстро kна и kот, необходимо создать эффективный ладу, приводит к значительному сокращению выбросов доноров. Таким образом проектирование эффективных ладу, выбрав соответствующие пары флуоресцентных красителей и сайтов на целевых белков прикрепить красители имеет важное значение и будут обсуждаться в настоящем Протоколе.
протокол
1. дизайн assay ладу.
- Скачайте файл структуры Cul1•Cand1 комплекс из банка данных белков (файл 1U6G).
- Просмотр структуры комплекса в PyMOL Cul1•Cand1.
- Используйте функцию измерения под меню Мастер PyMOL оценить расстояние между первой amino acid Cand1 и последней амино кислоты Cul1 (рис. 1).
- Загрузить средство просмотра онлайн спектры (см. Таблицу материалы) и одновременно просматривать возбуждения и выбросов спектры 7-амино-4-methylcoumarin (AMC) и FlAsH (рис. 2). Обратите внимание, что АМС является донором ладу и FlAsH акцептор ладу.
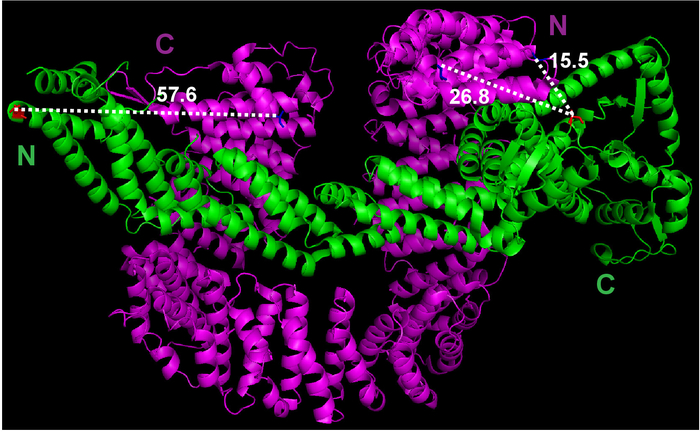
Рисунок 1: Кристаллическая структура измерения расстояния между потенциал маркировки сайтов и Cul1•Cand1. Файл структуры Кристалл был загружен из банка данных белков (файл 1U6G) и в PyMOL. Измерения между отдельных атомов были сделаны PyMOL. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
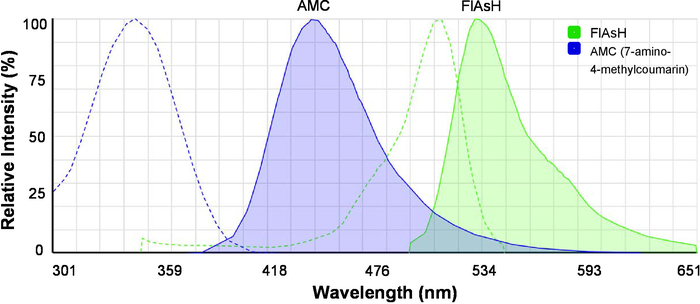
Рисунок 2: возбуждение и выбросов спектры флуоресцентных красителей для ладу. Приведены спектры AMC (7-4-амино methylcoumarin) и FlAsH. Пунктирные линии показывают возбуждения спектров, и сплошной линии указывают выбросов спектров. Изображение первоначально порожденных флуоресценции SpectraViewer и был изменен для большей ясности. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
2. Подготовка Cul1 •Rbx1КУА, белок доноров ладу
- Постройте плазмиды для выражения человека Cul1sortase•Rbx1 в клетках E. coli . Обратите внимание, что два плазмид, совместно выражая человека Cul1•Rbx1 в клетках E. coli подробно описаны в предыдущем докладе20.
- Добавьте последовательность ДНК кодирования «LPETGGHHHHHH» (sortase его6 тег) 3' конца Cul1 кодирования последовательности через стандартное PCR и клонирования методы21,22.
- Последовательности новых плазмиду для подтверждения вставка гена является точной.
- Совместное Экспресс Cul1sortase•Rbx1 в клетках E. coli . Этот метод является производным от предыдущего доклада20.
- Смесь 100 нг двух плазмид с BL21 (DE3) химически сведущие клетки для совместного преобразования с использованием тепла шок метод23. Рост клеток на табличке агар LB, содержащие 100 мкг/мл Ампициллин и хлорамфеникол 34 мкг/мл при 37 ° C на ночь.
- Прививок 50 мл LB культуры с свежезаваренным преобразованные колоний и расти на ночь при 37 ° C встряхивании 250 об/мин. Это дает закваски.
- Прививок 6 колб, каждый с 1 Л LB средних, с 5 мл закваски и растут при 37 ° C с 250 об/мин, пожимая до од600 ~ 1.0. Cool культуры до 16 ° C и добавить изопропил β-D-thiogalactoside (IPTG) до 0,4 мм. Держите культуры на 16 ° C ночь встряхивании 250 об/мин.
- Урожай клеток E. coli путем центрифугирования в 5000 x g 15 мин и собирать клеток окатышей в 50 мл конические трубы.
Примечание: Гранулы клетки могут быть обработаны для очистки белков или заморожены на-80 ° C, прежде чем продолжить шаги очистки белков.
- Очистка Cul1sortase•Rbx1 комплекс. Этот метод является производным от предыдущего доклада20.
- Добавьте 50 мл буфера lysis (30 мм трис-HCl, 200 NaCl, 5 мм DTT, 10% глицерина, 1 таблетка ингибитора протеазы коктейль, рН 7,6) Пелле клеток кишечной палочки , выражая Cul1sortase•Rbx1.
- Лизируйте клетки на льду с sonication на 50% амплитуды. Чередовать 1-второй и 1-секундный выключения и запустить на 3 мин.
- Повторите шаг 2.3.2 x 2-3.
- Передача lysate клетки в 50-мл центрифугирования трубку и удалить мусор клетки центрифугированием в 25000 х g 45 мин.
- Инкубируйте Светлоклеточная lysate с 5 мл глутатион бусы на 4 ° C на 2 ч.
- Центрифуга бусы lysate смеси на 1500 x g на 2 мин при 4 ° C. Удалите супернатант.
- Бусины с 5 мл буфера lysis (не ингибиторами протеазы) вымыть и удалить супернатант после центрифугирования в 1500 x g на 2 мин при 4 ° C.
- Повторите шаг 2.3.7 2 x.
- Добавить 3 мл буфера lysis промывают бусины и передача шлам бусины в пустой столбец.
- Добавьте 5 мл буфера (50 Tris-HCl, 200 мм мм NaCl, 10 мм уменьшена глутатиона, pH 8.0) в столбце. Проинкубируйте втечение 10 мин и собирать элюата.
- Повторите шаг 2.3.10 3-4 x.
- 200 мкл тромбина 5 мг/мл (см. Таблицу материалы) до элюата из бисера глутатиона и инкубировать на ночь при 4 ° C.
Примечание: Протокол может быть приостановлена здесь. - Разбавьте образец протеина с буфером A (25 мм HEPES, 1 мм DTT, 5% глицерина, рН 6,5) три раза.
- Сбалансировать столбец хроматографии катионного обмена (см. Таблицу материалы) на ПСОК систему с буфером а.
- Загрузите образец протеина в уравновешенной катионообмен хроматографии столбец со скоростью потока 0,5 мл/мин.
- Элюировать белка с градиентом NaCl в 40 мл, смешивая буфер A и 0 до 50% буфера B (25 мм HEPES, 1 M NaCl, 1 мм DTT, 5% глицерина, рН 6,5) при скорости потока 1 мл/мин.
- Проверьте eluted белка в различных фракций с использованием SDS-PAGE24.
Примечание: Протокол может быть приостановлена здесь. - Бильярд элюата фракций, содержащих Cul1sortase•Rbx1.
- Концентрат пула Cul1sortase•Rbx1 для образца 2,5 мл, передав в буфер через ультрафильтрационные мембраны (отсечки 30 кДа).
- Добавьте AMC C-конечная Cul1 через sortase опосредованной transpeptidation21,22.
- Изменить содержимое буфера в образце •Rbx1sortaseCul1 sortase буфер (50 мм трис-HCl, 150 мм NaCl, 10 мм2CaCl, рН 7,5) с помощью столбца опреснительные (см. Таблицу материалы).
- Сбалансировать опреснительной столбец с 25 мл буфера sortase.
- В столбце Загрузите 2,5 мл Cul1sortase•Rbx1 образца. Отказаться от потока через.
- Элюировать образца с 3,5 мл буфера sortase. Соберите потока через.
- В 900 мкл раствора •Rbx1sortaseCul1 в sortase буфера добавьте 100 мкл 600 мкм очищенный sortase A решения и 10 мкл пептид GGGGAMC 25 мм. Инкубируйте реакционной смеси при 30 ° C в темной ночи. Обратите внимание, что этот шаг будет генерировать Cul1КУА•Rbx1.
Предупреждение: Флуоресцентными красителями чувствительны к свет, так что избежать, подвергая их воздействию окружающего света во время белковых и образец подготовки как можно больше.
Примечание: Протокол может быть приостановлена здесь. - Добавьте 50 мкл Ni-НТА агарозы бусины реакционной смеси и инкубации при комнатной температуре за 30 мин.
- Пелле бусины агарозы Ni-НТА центрифугированием в 5000 x g на 2 мин и собирать супернатант.
- Сбалансировать размер столбца исключение хроматографии (см. Таблицу материалы) с буфером (30 мм трис-HCl, 100 мм NaCl, 1 мм DTT, 10% глицерина) на ПСОК систему.
- Загрузить все Cul1КУА•Rbx1 образца на столбце хроматографии исключения размер. Элюировать объемом 1.5 x столбца буфера.
- Проверьте элюата фракций, SDS-PAGE24.
- Бассейн элюата фракций, содержащих Cul1КУА•Rbx1.
- Измерить концентрацию белка, используя его поглощения в 280 Нм с спектрофотометром.
- Алиготе раствор белка и хранить при температуре-80 ° C.
Примечание: Протокол может быть приостановлена здесь.
- Изменить содержимое буфера в образце •Rbx1sortaseCul1 sortase буфер (50 мм трис-HCl, 150 мм NaCl, 10 мм2CaCl, рН 7,5) с помощью столбца опреснительные (см. Таблицу материалы).
3. Подготовка FlAsHCand1, белок акцептора ладу
Примечание: Большинство из шагов в этой части являются так же, как шаг 2. Ниже подробно описаны условия, которые отличаются.
- Постройте плазмиду для выражения человека TetraCysCand1 в клетках E. coli .
- Добавление последовательности ДНК кодирования «CCPGCCGSG» (tetracysteine/TetraCys тег) до 15й аминокислоты из Cand1 через регулярные ПЦР25 (грунтовка последовательности: TGCTGTCCGGGCTGCTGCGGCAGCGGCATGACATCCAGCGACAAGGACTTTAG; CTAACTAGTGTCCATTGATTCCAAG).
- Вставьте продукт PCR в pGEX 4Т-2 вектора. Последовательность плазмиду для подтверждения вставка гена является точной и в кадре.
- Экспресс TetraCysCand1in E. coli клетки в так же, как шаг 2.2, за исключением того, что плазмида преобразуется в Rosetta сведущие клетки.
- Очистка TetraCysCand1 из клеток кишечной палочки .
- Лизировать гранулы клеток кишечной палочки и экстракт TetraCysCand1using глутатиона бусины. Эти шаги являются так же, как 2.3.1–2.3.12 шаги.
- Разбавьте элюата белка из бисера глутатиона с буфера C (50 мм трис-HCl, 1 мм DTT, 5% глицерина, pH 7.5), три складки. Сбалансировать столбец хроматографии обмену анионов (см. Таблицу материалы) на ПСОК систему с буфера C и нагрузки образец разбавленный протеина при скорости потока 0,5 мл/мин.
- Элюировать белка с градиентом NaCl в 40 мл, смешивая буфера C и 0 до 50% D буфера (50 мм трис-HCl, 1 M NaCl, 1 мм DTT, 5% глицерина, рН 7,5) при скорости потока 1 мл/мин. Проверьте eluted белка в различных фракций с использованием SDS-PAGE24и бассейн фракций, содержащих TetraCysCand1. Обратите внимание, что TetraCysCand1 имеет больший объем хранения чем бесплатно GST.
- Концентрат пула TetraCysCand1 образца, передав в буфер через ультрафильтрационные мембраны (30 кДа отсечки).
- Сбалансировать размер исключения хроматографии столбец с маркировки буфера (20 мм трис-HCl, 100 мм NaCl, 2 TCEP, 1 мм ЭДТА, 5% глицерина) на ПСОК систему. Загрузить образец TetraCysCand1 (500 мкл каждый раз) и проверьте элюата фракций SDS-PAGE24.
- Объединить всех фракций, содержащих TetraCysCand1 и концентрата белка до ~ 40 мкм, передав в буфер через ультрафильтрационные мембраны (30 кДа отсечки). Оценить концентрацию белка, используя его поглощения в 280 Нм. Хранить белка как 50 мкл аликвоты-80 ° c.
Примечание: Протокол может быть приостановлена здесь.
- Подготовка флэш-Cand1.
- 1 мкл флэш-решения (см. Таблицу материалы) до 50 мкл TetraCysCand1 решения.
- Хорошо перемешайте и инкубировать смесь при комнатной температуре в темноте для 1-2 h получить FlAsHCand1.
Примечание: Протокол может быть приостановлена здесь.
4. Подготовка Cand1, Чейз белка ладу
Примечание: Белка подготовка протокол похож на шаг 3, со следующими изменениями.
- Вставьте кодирующая последовательность полнометражные Cand1 в pGEX 4Т-2 вектора.
- Измените буфер, используемый на шаге 3.3.5 буфер, содержащий 30 мм трис-HCl, 100 мм NaCl, 1 мм DTT, 10% глицерина.
- Ликвидировать шаги 3.3.7 и 3.3.8.
5. проверить и подтвердить пробирного ладу
- Подготовить ладу буфер, содержащий 30 мм трис-HCl, 100 мм NaCl, 0.5 мм DTT, овальбумина 1 мг/мл, pH 7,6 и использовать при комнатной температуре.
- Испытания Лада между Cul1 •Rbx1КУАи флэш-Cand1 на fluorimeter.
- Добавьте Cul1 в 300 мкл буфера ладу,AMC•Rbx1 (ладу доноров) до конечной концентрации 70 нм. Передача решения в кювет.
- Поместите кювету в держателе образца fluorimeter. Возбуждают образца с 350 Нм возбуждения светом и проверки выбросов сигналы от 400 Нм до 600 Нм при 1 нм с шагом.
- Повторите шаг 5.2.1, но изменения Cul1КУА•Rbx1 для FlAsHCand1 (ладу акцептор). Сканировать в образце Cand1 FlAsH, используя тот же метод, что и в шаге 5.2.2.
- (Необязательно) Возбуждают FlAsHCand1 возбуждения светом 510 нм и проверки выбросов сигнал от 500 Нм до 650 Нм.
- Добавьте в 300 мкл буфера ладу, оба Cul1КУА•Rbx1 и FlAsHCand1 до конечной концентрации 70 нм. Анализ образца таким же образом, как в шаге 5.2.2.
- Подтвердить Лада между Cul1 •Rbx1КУАи FlAsHCand1 путем добавления белка Чейз (немеченого Cand1) (рис. 3 c).
- Добавьте в 300 мкл буфера ладу, 70 нм Cul1КУА•Rbx1 и 700 Нм Cand1. Проверка образца выбросов как шаг 5.2.2.
- Добавьте в 300 мкл буфера ладу, 70 нм FlAsHCand1 и 700 Нм Cand1. Проверка образца выбросов как шаг 5.2.2.
- Добавьте в 300 мкл буфера ладу, 70 нм Cul1КУА•Rbx1 и 70 нм FlAsHCand1 и Проинкубируйте образцы при комнатной температуре в течение 5 мин. Затем добавить 700 Нм Cand1 и сразу же после того, сканирования образца выбросов как шаг 5.2.2). Обратите внимание, что этот шаг похож на шаг 5.2.5.
- В 300 мкл буфера ладу, последовательно добавить 70 нм Cul1КУА•Rbx1, 700 Нм Cand1 и 70 нм FlAsHCand1. Инкубировать образца при комнатной температуре в течение 5 мин и проверки образцов выбросов как шаг 5.2.2. Обратите внимание, что это пример Чейз (зеленая линия на рис. 3 c).
6. Измерьте константа скорости ассоциации (kна) по Cul1•Cand1
Примечание: Детали работы остановлены потока fluorimeter были описаны в предыдущем докладе26.
- Подготовьте остановить поток fluorimeter для измерения.
- Включите fluorimeter остановить поток согласно инструкции производителя.
- Установите возбуждения света на 350 Нм и использование полосовой фильтр, который позволяет 450 Нм выбросов свет пройти и блокирует 500-650 Нм выбросов света.
- Держите образца клапанов на заполнить позиции и подключить 3 мл шприц с водой. Путем перемещения образца шприц диск вверх и вниз несколько раз промойте водой два образца шприцы (A и B). Отменить все воды, используемой в этом шаге.
- Держите образца клапанов на заполнить позиции и подключить 3 мл шприц, заполнены с буфером ладу. Мыть два образца шприцы с буфером ладу, перемещая образца шприц диск вверх и вниз несколько раз. Отменить все буфера ладу, используемые на этом шаге.
- Возьмите измерения управления (рис. 4 c).
- Подключите 3 мл шприц и загрузить шприц А с 100 Нм Cul1КУА•Rbx1 в буфере ладу. Поверните клапан образца в положение диска .
- Подключите 3 мл шприц и загрузить B шприц с буфером ладу. Поверните клапан образца в положение диска .
- Используйте Панель управления при приобретении программного обеспечения принять пять выстрелов (смешать равные количества образцов из шприца A и B шприц) на fluorimeter остановить поток без записи результатов.
- Откройте Панель управления при приобретении программного обеспечения и программа для записи выбросов Cul1КУА более 60 s. Затем принять ни одного выстрела.
- Повторите шаг 6.2.4 2 x.
- Поверните клапан образца для заполнения позиции. Пустой шприц B и мыть с буфером ладу.
- Мера, наблюдается ассоциации константы скорости (kobs) Cul1•Cand1 (рис. 4В).
- Держите образец в шприц А так же, как и шаг 6.2.1.
- Подключите 3 мл шприц и загрузить B шприц с 100 Нм FlAsHCand1 в буфере ладу. Поверните клапан образца в положение диска .
- Используйте Панель управления при приобретении программного обеспечения принять пять выстрелов без записи результатов.
- Откройте Панель управления при приобретении программного обеспечения и программа для записи выбросов Cul1КУА более 60 s. Затем принять ни одного выстрела.
- Повторите шаг 6.3.4 2 x.
- Пустой шприц B и мыть с буфером ладу.
- Повторите шаги 6.3.1–6.3.6 несколько раз с увеличением концентрации FlAsHCand1 в буфере ладу.
- Изменение (уменьшение) помещается в флуоресцентные сигналов измеряется временем от каждого выстрела для экспоненциальной кривой. Это даст kobsв каждом измерении, и подразделение-s-1. Обратите внимание, что основой такого расчета хорошо обсуждался в предыдущем докладе27.
- Вычислить среднее и стандартное отклонение kobsдля каждой концентрации Cand1 FlAsH. Участок средняя kobsпротив концентрации Cand1 (рис. 4 d), и наклон линии представляет kна Cul1•Cand1, с блоком M-1 s-1.
7. Измерьте скорость Константа диссоциации (kвыкл) из Cul1•Cand1 в присутствии белка Skp1•F-бокс.
Примечание: Этот шаг похож на шаге 6, со следующими изменениями.
- В шприц А, под заполнить позиции, загрузите решение 100 Нм Cul1КУА•Rbx1 и 100 Нм FlAsHCand1 в буфере ладу. Поверните клапан образца в положение «Драйв».
- В шприц B под заполнить позиции, загрузите решение Skp1•Skp2 (подготовлен после предыдущего доклада20). Поверните клапан образца в положение «Драйв».
- Откройте Панель управления при приобретении программного обеспечения и программа для записи выбросов Cul1КУА более 30 s. Затем принять ни одного выстрела. Флуоресцентный сигналы увеличить со временем после смешивания решения из шприца A и B шприца (рис. 5).
Результаты
Для тестирования в ладу между Cul1КУА и FlAsHCand1, мы сначала определили интенсивности выбросов 70 нм Cul1AMC (донора) и 70 нм FlAsHCand1 (акцептор), соответственно (Рисунок 3А-C, синие линии). В каждом анализе только один пик выбросов был п?...
Обсуждение
Лада-это физическое явление, которое представляет большой интерес для изучения и понимания биологических систем19. Здесь мы представляем собой протокол для тестирования и использования лад для изучения кинетики привязки двух взаимодействующих белков. При проектировании ...
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Мы благодарим Шу-Ou Шань (Калифорнийский технологический институт) за глубокий дискуссии о развитии assay ладу. М.г., ВМФ и X.L. финансировались запуска средств из Университета Пердью в ВМФ и X.L.This работы была частично поддержана семян грант от центра Университета Purdue для биологии растений.
Материалы
| Name | Company | Catalog Number | Comments |
| Anion exchange chromatography column | GE Healthcare | 17505301 | HiTrap Q FF anion exchange chromatography column |
| Benchtop refrigerated centrifuge | Eppendorf | 2231000511 | |
| BL21 (DE3) Competent Cells | ThermoFisher Scientific | C600003 | |
| Calcium Chloride | Fisher Scientific | C78-500 | |
| Cation exchange chromatography column | GE Healthcare | 17505401 | HiTrap SP Sepharose FF |
| Desalting Column | GE Healthcare | 17085101 | |
| Floor model centrifuge (high speed) | Beckman Coulter | J2-MC | |
| Floor model centrifuge (low speed) | Beckman Coulter | J6-MI | |
| Fluorescence SpectraViewer | ThermoFisher Scientific | https://www.thermofisher.com/us/en/home/life-science/cell-analysis/labeling-chemistry/fluorescence-spectraviewer.html | |
| FluoroMax fluorimeter | HORIBA | FluoroMax-3 | |
| FPLC | GE Healthcare | 29018224 | |
| GGGGAMC peptide | New England Peptide | custom synthesis | |
| Glutathione beads | GE Healthcare | 17075605 | |
| Glycerol | Fisher Scientific | G33-500 | |
| HEPES | Fisher Scientific | BP310-100 | |
| Isopropyl-β-D-thiogalactoside (IPTG) | Fisher Scientific | 15-529-019 | |
| LB Broth | Fisher Scientific | BP1426-500 | |
| Ni-NTA agarose | Qiagen | 30210 | |
| Ovalbumin | MilliporeSigma | A2512 | |
| pGEX-4T-2 vector | GE Healthcare | 28954550 | |
| Protease inhibitor cocktail | MilliporeSigma | 4693132001 | |
| Reduced glutathione | Fisher Scientific | BP25211 | |
| Refrigerated shaker | Eppendorf | M1282-0004 | |
| Rosetta Competent Cells | MilliporeSigma | 70953-3 | |
| Size exclusion chromatography column | GE Healthcare | 28990944 | Superdex 200 10/300 GL column |
| Sodium Chloride (NaCl) | Fisher Scientific | S271-500 | |
| Stopped-flow fluorimeter | Hi-Tech Scientific | SF-61 DX2 | |
| TCEP·HCl | Fisher Scientific | PI20490 | |
| Thrombin | MilliporeSigma | T4648 | |
| Tris Base | Fisher Scientific | BP152-500 | |
| Ultrafiltration membrane | MilliporeSigma | UFC903008 | Amicon Ultra-15 Centrifugal Filter Units, Ultra-15, 30,000 NMWL |
Ссылки
- Stumpf, M. P. H., et al. Estimating the size of the human interactome. Proceedings of the National Academy of Sciences of the United States of America. 105 (19), 6959-6964 (2008).
- Kuzmanov, U., Emili, A. Protein-protein interaction networks: probing disease mechanisms using model systems. Genome Medicine. 5 (4), 37 (2013).
- Titeca, K., Lemmens, I., Tavernier, J., Eyckerman, S. Discovering cellular protein-protein interactions: Technological strategies and opportunities. Mass Spectrometry Reviews. , 1-33 (2018).
- Lapetina, S., Gil-Henn, H. A guide to simple, direct, and quantitative in vitro binding assays. Journal of Biological Methods. 4 (1), 62 (2017).
- Zheng, X., Bi, C., Li, Z., Podariu, M., Hage, D. S. Analytical methods for kinetic studies of biological interactions: A review. Journal of Pharmaceutical and Biomedical Analysis. 113, 163-180 (2015).
- Pierce, N. W., et al. Cand1 promotes assembly of new SCF complexes through dynamic exchange of F box proteins. Cell. 153 (1), 206-215 (2013).
- Liu, X., Reitsma, J. M., Mamrosh, J. L., Zhang, Y., Straube, R., Deshaies, R. J. Cand1-Mediated Adaptive Exchange Mechanism Enables Variation in F-Box Protein Expression. Molecular Cell. 69 (5), 773-786 (2018).
- Zheng, N., et al. Structure of the Cul1-Rbx1-Skp1-F boxSkp2 SCF ubiquitin ligase complex. Nature. 416 (6882), 703-709 (2002).
- Kleiger, G., Deshaies, R. Tag Team Ubiquitin Ligases. Cell. 166 (5), 1080-1081 (2016).
- Zheng, J., et al. CAND1 binds to unneddylated CUL1 and regulates the formation of SCF ubiquitin E3 ligase complex. Molecular Cell. 10 (6), 1519-1526 (2002).
- Hwang, J. W., Min, K. W., Tamura, T. A., Yoon, J. B. TIP120A associates with unneddylated cullin 1 and regulates its neddylation. FEBS Letters. 541 (1-3), 102-108 (2003).
- Min, K. W., Hwang, J. W., Lee, J. S., Park, Y., Tamura, T., Yoon, J. B. TIP120A associates with cullins and modulates ubiquitin ligase activity. Journal of Biological Chemistry. 278 (18), 15905-15910 (2003).
- Goldenberg, S. J., et al. Structure of the Cand1-Cul1-Roc1 complex reveals regulatory mechanisms for the assembly of the multisubunit cullin-dependent ubiquitin ligases. Cell. 119 (4), 517-528 (2004).
- Chuang, H. W., Zhang, W., Gray, W. M. Arabidopsis ETA2, an apparent ortholog of the human cullin-interacting protein CAND1, is required for auxin responses mediated by the SCF(TIR1) ubiquitin ligase. Plant Cell. 16 (7), 1883-1897 (2004).
- Feng, S., et al. Arabidopsis CAND1, an Unmodified CUL1-Interacting Protein, Is Involved in Multiple Developmental Pathways Controlled by Ubiquitin/Proteasome-Mediated Protein Degradation. the Plant Cell Online. 16 (7), 1870-1882 (2004).
- Cheng, Y., Dai, X., Zhao, Y. AtCAND1, a HEAT-repeat protein that participates in auxin signaling in Arabidopsis. Plant Physiology. 135 (June), 1020-1026 (2004).
- Lo, S. -. C., Hannink, M. CAND1-Mediated Substrate Adaptor Recycling Is Required for Efficient Repression of Nrf2 by Keap1. Molecular and Cellular Biology. 26 (4), 1235-1244 (2006).
- Okamoto, K., Sako, Y. Recent advances in FRET for the study of protein interactions and dynamics. Current Opinion in Structural Biology. 46, 16-23 (2017).
- Hussain, S. A. An introduction to fluorescence resonance energy transfer (FRET). arXiv preprint. , (2009).
- Li, T., Pavletich, N. P., Schulman, B. A., Zheng, N. High-level expression and purification of recombinant SCF ubiquitin ligases. Methods in Enzymology. 398 (1996), 125-142 (2005).
- Popp, M. W., Antos, J. M., Grotenbreg, G. M., Spooner, E., Ploegh, H. L. Sortagging: A versatile method for protein labeling. Nature Chemical Biology. 3 (11), 707-708 (2007).
- Antos, J. M., Ingram, J., Fang, T., Pishesha, N., Truttmann, M. C., Ploegh, H. L. Site-Specific Protein Labeling via Sortase-Mediated Transpeptidation. Current Protocols in Protein Science. 89, (2017).
- Froger, A., Hall, J. E. Transformation of Plasmid DNA into E. coli Using the Heat Shock Method. Journal of Visualized Experiments. , (2007).
- Simpson, R. J. SDS-PAGE of Proteins. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Kleiger, G., Saha, A., Lewis, S., Kuhlman, B., Deshaies, R. J. Rapid E2-E3 Assembly and Disassembly Enable Processive Ubiquitylation of Cullin-RING Ubiquitin Ligase Substrates. Cell. 139 (5), 957-968 (2009).
- Biro, F. N., Zhai, J., Doucette, C. W., Hingorani, M. M. Application of Stopped-flow Kinetics Methods to Investigate the Mechanism of Action of a DNA Repair Protein. Journal of Visualized Experiments. (37), 2-8 (2010).
- Patel, J. T., Belsham, H. R., Rathbone, A. J., Friel, C. T. Use of Stopped-Flow Fluorescence and Labeled Nucleotides to Analyze the ATP Turnover Cycle of Kinesins. Journal of Visualized Experiments. (92), 1-6 (2014).
- Bajar, B. T., Wang, E. S., Zhang, S., Lin, M. Z., Chu, J. A guide to fluorescent protein FRET pairs. Sensors (Switzerland). 16 (9), 1-24 (2016).
- Chen, A. K., Cheng, Z., Behlke, M. A., Tsourkas, A. Assessing the sensitivity of commercially available fluorophores to the intracellular environment. Analytical Chemistry. 80 (19), 7437-7444 (2008).
- Lin, C. T., Rorabacher, D. B. Mathematical approach for stopped-flow kinetics of fast second-order reactions involving inhomogeneity in the reaction cell. Journal of Physical Chemistry. 78 (3), 305-308 (1974).
- Toseland, C. P., Geeves, M. A. Rapid Reaction Kinetic Techniques. Fluorescent Methods for Molecular Motors. , 49-65 (2014).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: Synthesis and biological applications. Journal of the American Chemical Society. 124 (21), 6063-6076 (2002).
- Lin, C. W., Ting, A. Y. Transglutaminase-catalyzed site-specific conjugation of small-molecule probes to proteins in vitro and on the surface of living cells. Journal of the American Chemical Society. 128 (14), 4542-4543 (2006).
- Yin, J., et al. Genetically encoded short peptide tag for versatile protein labeling by Sfp phosphopantetheinyl transferase. Proceedings of the National Academy of Sciences of the United States of America. 102 (44), 15815-15820 (2005).
- Diaspro, A., Chirico, G., Usai, C., Ramoino, P., Dobrucki, J. Photobleaching. Handbook Of Biological Confocal Microscopy. , 690-702 (2006).
- Aoki, K., Kamioka, Y., Matsuda, M. Fluorescence resonance energy transfer imaging of cell signaling from in vitro to in vivo: Basis of biosensor construction, live imaging, and image processing. Development, Growth & Differentiation. 55 (4), 515-522 (2013).
- Kilic, S., et al. Single-molecule FRET reveals multiscale chromatin dynamics modulated by HP1α. Nature Communications. 9 (1), (2018).
- Shen, K., Arslan, S., Akopian, D., Ha, T., Shan, S. O. Activated GTPase movement on an RNA scaffold drives co-translational protein targeting. Nature. 492 (7428), 271-275 (2012).
- Bajar, B. T., et al. Improving brightness and photostability of green and red fluorescent proteins for live cell imaging and FRET reporting. Scientific Reports. 6 (February), 1-12 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены