A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
שימוש בהעברת אנרגיה פלורסצנטיות תהודה חוץ גופית ללמוד את הדינמיקה של חלבון מתחמי בקנה מידה הזמן אלפיות השנייה
In This Article
Summary
אינטראקציות חלבון-חלבון קריטי עבור מערכות ביולוגיות, מחקרים של קינטיקה איגוד לספק תובנות הדינמיקה והתפקוד של מתחמי חלבון. אנו מתארים שיטה מכמת את הפרמטרים קינטי של חלבון מורכב באמצעות קרינה פלואורסצנטית והעברת אנרגיה תהודה הטכניקה זרימה עצר.
Abstract
חלבונים הן חברות הסלולר העיקרי של מערכות ביולוגיות, הם בדרך כלל לקיים אינטראקציה עם אחרים מאקרו או מולקולות קטנות כדי לבצע תפקודים ביולוגיים שלהם. אינטראקציות כזה יכול להיות מאוד דינמיים, כלומר subunits שמעצבת כל הזמן משויך, חלופה מועדפת בשיעורים מסוימים. בעוד מדידה של זיקה מחייבת שימוש בטכניקות כגון הנפתח כמותיים חושף את עוצמת האינטראקציה, לומד את קינטיקה איגוד מספק תובנות על איך מהר האינטראקציה מתרחשת, כמה זמן כל מתחם יכולה להתקיים. יתר על כן, מדידה של קינטיקה של אינטראקציה בנוכחות גורם נוסף, כגון פקטור המרת חלבון או סם, מסייע לחשוף את המנגנון שבאמצעותו האינטראקציה מוסדר על ידי הגורם השני, מתן ידע חשוב עבור קידום מחקר ביולוגי ורפואי. כאן, אנו מתארים את פרוטוקול למדידת קינטיקה של איגוד של קומפלקס החלבונים יש שיעור גבוה האגודה מהותי, יכול להיות חלופה מועדפת במהירות על ידי חלבון אחר. השיטה משתמשת העברת אנרגיה של קרינה פלואורסצנטית תהודה לדווח היווצרות של המתחם חלבון במבחנה, והוא מאפשר ניטור של דיסוציאציה של המתחם בזמן אמת על fluorimeter זרימה הפסיק וארגון מהר. שימוש assay הזה, הם לכמת את קבועי קצב דיסוציאציה וארגון של החלבונים.
Introduction
פעילויות ביולוגיות בסופו של דבר תבוצענה על ידי חלבונים, ביותר אשר אינטראקציה עם אחרים עבור תפקודים ביולוגיים נאותה. שימוש בגישה חישובית, הסכום הכולל של אינטראקציות חלבון-אנוש מוערך ~ 650,0001, שיבוש אינטראקציות אלה לעיתים קרובות מוביל מחלות2. עקב תפקידיהם חיוני בשליטה הסלולר תהליכים organismal, פותחו שיטות רבות ללמוד אינטראקציות חלבון-חלבון, כגון שמרים-שני-היברידית, קומפלמנטציה פלורסצנטיות ריאקציה דו-מולקולרית, פיצול-לוציפראז קומפלמנטציה ו- co-immunoprecipitation assay3. בעוד ששיטות אלה טובים גילוי ולאשר אינטראקציות חלבון-חלבון, הם בדרך כלל שאינם כמותיים, ובכך לספק מידע מוגבל לגבי הזיקה בין בני הזוג חלבון אינטראקציה. השפעה כמותית-דאונס יכול לשמש כדי למדוד את זיקה מחייבת (למשל, קבוע דיסוציאציה Kd), אבל זה לא למדוד את קינטיקה של האיגוד, ולא ניתן להחיל אותה כאשר Kd נמוכה מאוד עקב לקוי יחס אות לרעש4. פלזמון משטח תהודה (SPR) ספקטרוסקופיה מכמת את קינטיקה מחייב, אך זה דורש על משטח ספציפי בטקטיקות של מגיבים אחד על פני השטח, אשר פוטנציאל תוכל לשנות את המאפיין איגוד של מגיב5. יתר על כן, קשה למדוד דיסוציאציה המחירים5וארגון מהר SPR, וזה לא ראוי לעשות שימוש SPR לאפיין את האירוע של החלפת החלבוניות בתוך קומפלקס החלבונים. כאן, אנו מתארים שיטה המאפשרת מדידת שיעור של חלבונים מורכבים הרכבה ופירוק בקנה מידה הזמן אלפיות השנייה. שיטה זו היתה חיונית לצורך קביעת התפקיד של Cullin - ssociated -Nedd8 -dissociated חלבון 1 (Cand1)כמו F-box חלבון exchange גורם6,7.
Cand1 מסדיר את הדינמיקה של Skp1•Cul1•F-תיבת חלבון (SCF) E3 ligases, אשר שייכת למשפחה גדולה של Cullin-טבעת אוביקוויטין ligases. SCFs מורכב cullin Cul1, אשר נקשר החלבון תחום טבעת Rbx1, ולא של חלבון F-box להחלפה, אשר מגייס סובסטרטים ומאגד Cul1 דרך החלבון מתאם Skp18. E3 ליגאז, SCF מזרז את ההטיה של אוביקוויטין את המצע שלה, וגם זה מופעל כאשר המצע הוא גויס על ידי החלבון F-box, וכאשר Cul1 משתנה על ידי החלבון אוביקוויטין דמוי Nedd89. Cand1 נקשר Cul1 שלא שונתה, על הכריכה, זה משבש גם איגוד חלבון Skp1•F-box עם Cul1 וגם את ההטיה של Nedd8 Cul110,11,12,13. כתוצאה מכך, Cand1 שנראה מעכב פעילות SCF במבחנה, אבל חוסר Cand1 אורגניזמים גרם פגמים שמרמז תפקיד חיובי של Cand1 בוויסות פעילות SCF vivo14,15,16 , 17. פרדוקס זה הוסבר בסופו של דבר על ידי מחקר כמותי שגילה את האינטראקציות דינמי בין חלבון Cul1, Cand1 ו Skp1•F-box. באמצעות קרינה פלואורסצנטית תהודה אנרגיה העברה (סריג) מבחני המאתרות את היווצרות מתחמי SCF ו- Cul1•Cand1, שיוך של דיסוציאציה לדרג קבועים (k- ו kחופש, בהתאמה) היו נמדד בנפרד. המדידות חשפו כי הן Cand1 והן Skp1•F-תיבת חלבון טופס הדוק ביותר מתחם עם Cul1, אבל את kהנחה של SCF מתגברת באופן דרמטי על ידי Cand1, kהנחה של Cul1•Cand1 הוא גדלו דרמטית. על ידי Skp1•F-תיבת חלבון6,7. תוצאות אלה מספקים את התמיכה הראשונית וקריטיים להגדרת תפקידו של Cand1 כגורם exchange חלבון, אשר מזרז היווצרות של מתחמי SCF חדש באמצעות מיחזור Cul1 מ מתחמי SCF הישן.
כאן, אנו מציגים את ההליך של פיתוח ושימוש וזמינותו סריג ללמוד את הדינמיקה של מורכבות Cul1•Cand17, ניתן להחיל את אותו עיקרון ללמוד את הדינמיקה של מולקולות שונות. סריג מתרחשת כאשר תורם הוא מתרגש עם הגל המתאים, מקבל עם ספקטרום עירור חפיפה ספקטרום הפליטה תורם קיים למרחק של 10-100 Å. המדינה נרגשת מועבר מקבל, ובכך להקטין את עוצמת התורם, הגדלת עוצמת מקבל18. היעילות של קשת/קשתית (E) תלוי רדיוס פורסטר (R0) והן את המרחק בין התורם ואת מקבל fluorophores (r), מוגדר על-ידי: E = R06/ (R0 6 + r6). רדיוס פורסטר (R0) תלויה במספר גורמים, כולל כיוון זוויתי דיפול, החפיפה ספקטרלי של הזוג מקבל התורם, והפתרון היה19. למרוח וזמינותו סריג fluorimeter עצר-flow, אשר מפקח על השינוי של הפליטה תורם בזמן אמת ומאפשר מדידות של מהיר k- ו kאת, זה הכרחי ליצור סריג יעיל זה תוצאות לירידה משמעותית של פליטת התורם. לכן, עיצוב סריג יעיל על-ידי בחירת הזוג המתאים של צבעי פלורסנט, ואתרים על החלבונים היעד כדי לצרף את צבע חשוב, יידונו ב פרוטוקול זה.
Protocol
1. עיצוב וזמינותו סריג.
- הורד את הקובץ מבנה של Cul1•Cand1 מורכבות מ בנק מידע החלבונים (קובץ 1U6G).
- הציגו את המבנה של Cul1•Cand1 מתחם PyMOL.
- השתמש בפונקציה מדידה תחת התפריט אשף של PyMOL כדי להעריך את המרחק בין החומצה אמינית הראשון של Cand1 חומצת אמינו האחרונה ביותר של Cul1 (איור 1).
- לטעון את הצופה ספקטרה באינטרנט (ראה טבלה של חומרים) ולהציג את עירור ועל ספקטרום הפליטה של 7-אמינו-4-methylcoumarin (AMC) ו- FlAsH בו זמנית (איור 2). שימו לב כי AMC התורם סריג, פלאש הוא מקבל סריג.
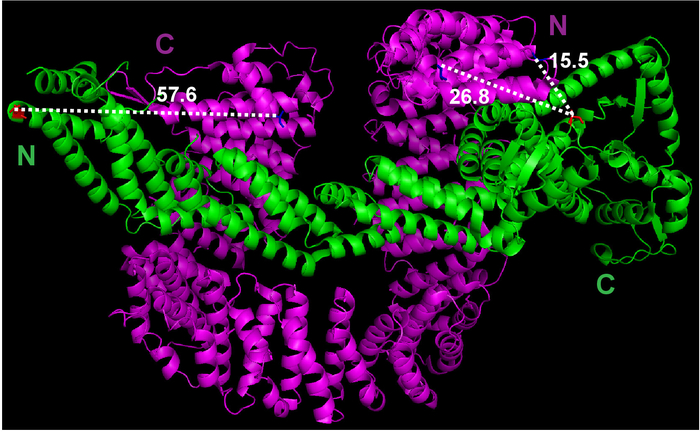
איור 1: מבנה הגביש Cul1•Cand1 מדידה של המרחק בין הפוטנציאל תיוג אתרים- הקובץ מבנה קריסטל הייתה שהורדו מ בנק מידע החלבונים (קובץ 1U6G), צפו ב PyMOL. מידות בין אטומים שנבחר נעשו על-ידי PyMOL. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
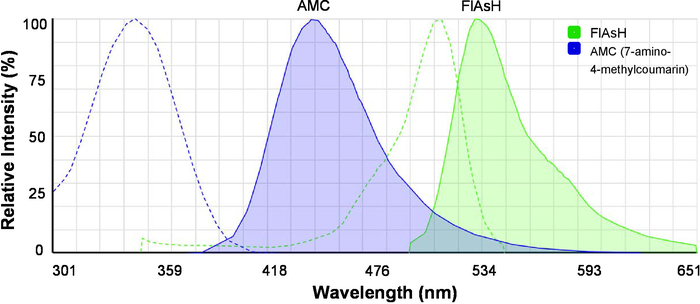
איור 2: עירור של ספקטרום הפליטה של צבעי פלורסנט עבור קשת/קשתית. ספקטרום של AMC (7-אמינו-4-methylcoumarin) ו- FlAsH מוצגים. קווים מקווקווים מציינים את עירור ספקטרה, קווים אחידים מציינים ספקטרום הפליטה. התמונה נוצר במקור על ידי SpectraViewer-ידי קרינה פלואורסצנטית ולשנות עבור בהירות יותר. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
2. הכנת Cul1AMC•Rbx1, החלבון התורם סריג
- לבנות פלסמידים לביטוי האנושי •Rbx1sortaseCul1 בתאים e. coli . שימו לב כי פלסמידים 2 שיתוף לבטא Cul1•Rbx1 האנושי בתאים e. coli מתוארים בפירוט הדוח הקודם20.
- להוסיף רצף ה-DNA קידוד "LPETGGHHHHHH" (sortase-שלו6 תג) 3' סוף Cul1 רצף קידוד באמצעות תקן ה-PCR ושיטות השיבוט21,22.
- רצף של פלסמיד חדש כדי לאשר תותב ג'ין הוא מדויק.
- שיתוף אקספרס Cul1 •Rbx1sortaseבתאים e. coli . השיטה נגזרת של הדוח הקודם20.
- Ng לערבב 100 כל של פלסמידים 2 עם BL21 (DE3) תאים כשיר מבחינה כימית לשינוי שיתוף באמצעות החום לחשמל שיטת23. לגדל תאים בצלחת אגר LB המכיל אמפיצילין µg/mL 100 ו µg 34/mL כלורמפניקול ב 37 מעלות צלזיוס למשך הלילה.
- לחסן 50 מ של תרבות ליברות עם מושבות טרי טרנספורמציה ולגדול בן לילה ב 37 ° C עם סל"ד 250 רועדת. זה נותן תרבות starter.
- לחסן 6 מבחנות, כל אחד עם 1 ליטר של מדיום ליברות, עם 5 מ"ל starter תרבות כל ולגדול ב 37 ° C עם סל"ד 250 לרעוד עד ה OD600 ~ 1.0. מגניב התרבות עד 16 ° C ולהוסיף איזופרופיל-β-D-thiogalactoside (IPTG) 0.4 מ מ. שמור את התרבות ב 16 ° C בלילה עם סל"ד 250 רועדת.
- הקציר התאים e. coli באמצעות צנטריפוגה ב x 5,000 g למשך 15 דקות ולאסוף תא כדורי צינורות חרוט 50 מ.
הערה: כדורי תא יעובד לטיהור חלבון או להיות קפוא ב-80 מעלות צלזיוס לפני שממשיכים לשלבים טיהור חלבון.
- טיהור של •Rbx1sortaseCul1 מורכבים. שיטה זו נגזרת של הדוח הקודם20.
- להוסיף 50 מ של פירוק מאגר (30 מ מ טריס-HCl, 200 מ"מ NaCl, 5 מ מ DTT, 10% גליצרול, 1 טבליה של מעכב פרוטאז קוקטייל, pH 7.6) בגדר של e. coli תאים לבטא Cul1sortase•Rbx1.
- Lyse התאים על הקרח עם sonication-משרעת 50%. לסירוגין שניה אחת ON ו- OFF שניה אחת ולרוץ 3 דקות.
- חזור על שלב 2.3.2 x 2-3.
- להעביר את התא lysate לתוך צינור 50-mL צנטריפוגה ולהסיר את שאריות תאים על ידי צנטריפוגה ב x 25,000 g למשך 45 דקות.
- דגירה התא ברורה lysate 5 מ של גלוטתיון חרוזים ב 4 מעלות צלזיוס במשך שעתיים.
- Centrifuge את התערובת חרוזים-lysate ב x 1,500 g למשך 2 דקות ב 4 º C. הסר את תגובת שיקוע.
- לשטוף את החרוזים עם 5 מ של פירוק מאגר (עם מעכבי פרוטאז לא) ולהסיר את תגובת שיקוע לאחר צנטריפוגה ב 1,500 x g למשך 2 דקות ב 4 º C.
- חזור על שלב 2.3.7 2 x.
- להוסיף 3 מ"ל של פירוק מאגר החרוזים שטף ולהעביר את slurry חרוז לתוך עמודה ריקה.
- הוסף 5 מ של מאגר • תנאי (50 מ"מ טריס-HCl, 200 מ"מ NaCl, 10 מ מ מופחת גלוטתיון, pH 8.0) לתוך העמודה. תקופת דגירה של 10 דקות ולאסוף את eluate.
- חזור על שלב 2.3.10 x 3-4.
- להוסיף 200 µL של תרומבין 5 מ"ג/מ"ל (ראה טבלה של חומרים) eluate של החרוזים גלוטתיון דגירה בין לילה ב 4 º C.
הערה: הפרוטוקול אפשר לעצור כאן. - לדלל את הדגימה חלבון עם מאגר A (25 מ מ HEPES, 1 מ"מ DTT, גליצרול 5%, pH 6.5) פי שלושה.
- Equilibrate עמודה כרומטוגרפיה קטיון (ראה טבלה של חומרים) על מערכת FPLC עם מאגר א
- לטעון את המדגם חלבון לעמודה כרומטוגרפיה equilibrated קטיון בספיקה 0.5 mL/min.
- Elute החלבון עם שיפוע של NaCl ב- 40 מ"ל על ידי ערבוב מאגר A ו- 0 עד 50% B מאגר (25 מ מ HEPES, 1 M NaCl, 1 מ"מ DTT, גליצרול 5%, pH 6.5) בספיקה 1 mL/min.
- בדוק את החלבון eluted בחלקים שונים באמצעות מרחביות-עמוד24.
הערה: הפרוטוקול אפשר לעצור כאן. - בריכת שברים eluate המכיל Cul1sortase•Rbx1.
- לרכז את Cul1sortase•Rbx1 הדגימה במאגר כדי 2.5 מ ל ע י העברת המאגר דרך קרום אולטראפילטרציה (הקיצוץ 30 kDa).
- להוסיף AMC קצה קרבוקסילי של Cul1 דרך transpeptidation בתיווך sortase21,22.
- לשנות את המאגר במדגם •Rbx1sortaseCul1 אל המאגר sortase (50 מ מ טריס-HCl, 150 מ מ NaCl, 10 מ מ CaCl2, pH 7.5) משתמש בעמודה desalting (ראה טבלה של חומרים).
- Equilibrate עמודה desalting 25 מ ל מאגר sortase.
- לטעון 2.5 מ ל Cul1sortase•Rbx1 מדגם לתוך העמודה. למחוק את הזרימה דרך.
- Elute הדגימה עם 3.5 מ של מאגר sortase. לאסוף את הזרימה דרך.
- ב- µL 900 של הפתרון •Rbx1sortaseCul1 במאגר sortase, להוסיף 100 µL של 600 מיקרומטר מטוהר sortase A פתרון ו 10 µL של 25 מ מ GGGGAMC פפטיד. דגירה תערובת התגובה ב 30 מעלות צלזיוס ב הלילה האפל. הערה שלב זה יפיק Cul1AMC•Rbx1.
התראה: צבעי פלורסנט רגישים אור, כדי להימנע לחשוף אותם תאורת במהלך הכנת חלבון ולדגום כמה שיותר.
הערה: הפרוטוקול אפשר לעצור כאן. - להוסיף תערובת התגובה µL 50 של ני-נ agarose חרוזים, דגירה בטמפרטורת החדר למשך 30 דקות.
- גלולה Ni-נ agarose חרוזים על ידי צנטריפוגה ב x 5,000 g למשך 2 דקות ולאסוף את תגובת שיקוע.
- Equilibrate גודל אי-הכללה של כרומטוגרפיה עמודה (ראה טבלה של חומרים) עם מאגר (30 מ מ טריס-HCl, 100 מ מ NaCl, 1 מ"מ DTT, 10% גליצרול) על מערכת FPLC.
- לטעון את כל Cul1 שלAMCמדגם •Rbx1 על העמודה כרומטוגרפיה של אי-הכללה של גודל. Elute עם 1.5 x בעמודה כמות המאגר.
- בדוק את השברים eluate על ידי מרחביות-עמוד24.
- בריכה שברים eluate המכיל Cul1AMC•Rbx1.
- למדוד את ריכוז חלבון באמצעות ספיגת שלה ב 280 ננומטר עם ספקטרופוטומטרים.
- Aliquot הפתרון חלבון וחנות ב-80 מעלות צלזיוס.
הערה: הפרוטוקול אפשר לעצור כאן.
- לשנות את המאגר במדגם •Rbx1sortaseCul1 אל המאגר sortase (50 מ מ טריס-HCl, 150 מ מ NaCl, 10 מ מ CaCl2, pH 7.5) משתמש בעמודה desalting (ראה טבלה של חומרים).
3. הכנת פלאשCand1, החלבון מקבל סריג
הערה: רוב השלבים בחלק זה הם אותו דבר כמו בשלב 2. תנאים שונים מתוארים בפירוט בהמשך.
- לבנות את פלסמיד לביטוי האנושי Cand1 TetraCysבתאים e. coli .
- הוסף את רצף ה-DNA קידוד "CCPGCCGSG" (tetracysteine/TetraCys תג) לפני 15th חומצת אמינו ביותר של Cand1 באמצעות PCR רגיל25 (פריימר רצפים: TGCTGTCCGGGCTGCTGCGGCAGCGGCATGACATCCAGCGACAAGGACTTTAG; CTAACTAGTGTCCATTGATTCCAAG).
- הכנס את המוצר PCR וקטור pGEX-4T-2. רצף של פלסמיד כדי לאשר תותב ג'ין הוא מדויק, מסגרת.
- אקספרס TetraCysCand1in e. coli תאים באותו אופן כשלב 2.2, חוץ מזה פלסמיד הוא הופך תאים המוסמכת רוזטה.
- טיהור של TetraCysCand1 את התאים e. coli .
- Lyse כדורי תא החיידק ולחלץ TetraCysCand1using גלוטתיון חרוזים. השלבים זהים שלבים 2.3.1–2.3.12.
- לדלל את eluate חלבון מ החרוזים גלוטתיון עם מאגר C (50 מ מ טריס-HCl, 1 מ"מ DTT, גליצרול 5%, pH 7.5) על ידי שלושה קיפולים. Equilibrate עמודת כרומטוגרפיה של exchange אניון (ראה טבלה של חומרים) על מערכת FPLC עם מאגר C וטעינה הדגימה חלבון מדולל בספיקה 0.5 mL/min.
- Elute החלבון עם שיפוע של NaCl ב- 40 מ"ל על ידי ערבוב מאגר C ו- 0 עד 50% D מאגר (50 מ מ טריס-HCl, 1 M NaCl, 1 מ"מ DTT, גליצרול 5%, pH 7.5) בספיקה 1 mL/min. בדוק את החלבון eluted בחלקים שונים באמצעות מרחביות-דף24ולאחר בריכה שברים המכיל TetraCysCand1. שימו לב כי TetraCysCand1 נפח שמירה גדול יותר מאשר GST חינם.
- לרכז את הדגימה Cand1 TetraCysבמאגר על-ידי העברת המאגר דרך קרום אולטראפילטרציה (הקיצוץ 30 kDa).
- Equilibrate את העמודה כרומטוגרפיה אי-הכללה של גודל עם מאגר תיוג (20 מ"מ טריס-HCl, 100 מ מ NaCl, 2 מ מ. TCEP, 1 מ"מ EDTA, 5% גליצרול) על מערכת FPLC. לטעון מדגם TetraCysCand1 (500 µL בכל פעם) וסמנו שברים eluate מרחביות-עמוד24.
- בריכה כל השברים המכיל TetraCysCand1 ולרכז החלבון מיקרומטר ~ 40 ע י העברת המאגר דרך קרום אולטראפילטרציה (הקיצוץ 30 kDa). מעריכים את ריכוז חלבון באמצעות ספיגת שלה ב 280 ננומטר. לאחסן את החלבון כמו 50 aliquots µL ב-80 מעלות צלזיוס.
הערה: הפרוטוקול אפשר לעצור כאן.
- הכנת Cand1 פלאש.
- להוסיף 1 µL של פתרון פלאש (ראה טבלה של חומרים) לפתרון Cand1 TetraCysµL 50.
- מערבבים היטב, דגירה התערובת בטמפרטורת החדר בחושך 1-2 h לקבל Cand1 פלאש.
הערה: הפרוטוקול אפשר לעצור כאן.
4. הכנת Cand1, החלבון צ'ייס סריג
הערה: פרוטוקול הכנה חלבון דומה בשלב 3, עם השינויים הבאים.
- הכנס את רצף קידוד של Cand1 באורך מלא וקטור pGEX-4T-2.
- לשנות את המאגר המשמש בשלב 3.3.5 למאגר המכיל 30 מ מ טריס-HCl, 100 מ מ NaCl, 1 מ"מ DTT, 10% גליצרול.
- לחסל את השלבים 3.3.7 ו 3.3.8.
5. לבדוק ולאשר וזמינותו סריג
- הכנת המאגר סריג המכיל 30 מ מ טריס-HCl, 100 מ מ NaCl, 0.5 מ מ DTT, אובלבומין 1 מ"ג/מ"ל, pH 7.6, ולהשתמש בטמפרטורת החדר.
- מבחן על סריג בין Cul1AMC•Rbx1, פלאשCand1 על fluorimeter.
- ב- µL 300 מאגר סריג, להוסיף Cul1AMC•Rbx1 (סריג התורם) כדי ריכוז סופי של 70 ננומטר. להעביר את הפתרון cuvette.
- מקם את cuvette בעל מדגם fluorimeter. לרגש את הדגימה עם 350 nm עירור אור ולסרוק את האותות פליטה מ- 400 nm 600 nm ב במרווחים 1 ננומטר.
- חזור על שלב 5.2.1 אבל שינוי Cul1 •Rbx1AMCכדי Cand1 פלאש(סריג מקבל). סרוק את הדגימה Cand1 FlAsHבאמצעות אותה שיטה כמו שלב 5.2.2.
- (אופציונלי) לרגש את Cand1 פלאשבאור עירור 510 ננומטר, וסרוק את האות פליטה בין 500 nm 650 ננומטר.
- במאגר 300 µL סריג, להוסיף שתי Cul1AMC•Rbx1 ו- Cand1 פלאשכדי ריכוז סופי של 70 ננומטר. לנתח את הדגימה באותו אופן כמו שלב 5.2.2.
- לאשר את סריג בין Cul1AMC•Rbx1, פלאשCand1 על-ידי הוספת החלבון צ'ייס (ללא תווית Cand1) (איור 3C).
- ב- µL 300 מאגר סריג, להוסיף 70 nM Cul1AMC•Rbx1 ו 700 nM Cand1. סרוק את הפליטה מדגם כמו שלב 5.2.2.
- ב- µL 300 מאגר סריג, להוסיף 70 nM Cand1 פלאשו 700 nM Cand1. סרוק את הפליטה מדגם כמו שלב 5.2.2.
- ב- µL 300 מאגר סריג, להוסיף 70 nM Cul1AMC•Rbx1 ו-70 ננומטר פלאשCand1, דגירה המדגם בטמפרטורת החדר במשך 5 דקות. לאחר מכן להוסיף 700 nM Cand1, מיד לאחר התוספת, סריקה הפליטה מדגם כמו שלב 5.2.2). שימו לב כי שלב זה דומה לשלב 5.2.5.
- במאגר 300 µL סריג, ברצף להוסיף 70 nM Cul1AMC•Rbx1, 700 nM Cand1, ו- 70 nM Cand1 פלאש. דגירה המדגם בטמפרטורת החדר במשך 5 דקות, ולאחר סרוק הפליטה מדגם כמו שלב 5.2.2. שים לב כי זוהי הדוגמה צ'ייס (הקו הירוק באיור 3C).
6. למדוד קבוע קצב האגודה (k-) Cul1•Cand1
הערה: פרטים של ההפעלה fluorimeter זרימה הפסיק תואר בשנת הדוח הקודם26.
- היכונו fluorimeter זרימה עצר את המדידה.
- הפעל את fluorimeter זרימה עצר לפי הוראות היצרן.
- להגדיר את עירור אור 350 nm, וכן שימוש מסנן הלהקה-pass המאפשר 450 nm פליטת האור לעבור וחוסם 500-650 nm פליטת אור.
- לשמור את השסתומים מדגם במיקום למלא וחבר מזרק 3 מ ל מים. לשטוף מדגם שני מזרקים (A ו- B) עם מים על-ידי הזזת את הכונן מזרק מדגם לאורך מספר פעמים. למחוק את כל המים בשימוש בשלב זה.
- לשמור את השסתומים מדגם במיקום למלא וחבר מזרק 3 מ"ל מלא מאגר סריג. לשטוף את מזרקים מדגם שני עם המאגר סריג על-ידי הזזת את הכונן מזרק מדגם לאורך כמה זמן. למחוק את כל המאגר סריג המשמש בשלב זה.
- קח שליטה מדידה (איור 4C).
- התחבר מזרק 3 מ"ל, לטעון את המזרק A עם 100 ננומטר Cul1AMC•Rbx1 במאגר סריג. להפוך את השסתום דוגמה למיקום כונן .
- התחבר מזרק 3 מ"ל, לטעון B מזרק עם המאגר סריג. להפוך את השסתום דוגמה למיקום כונן .
- השתמש בלוח הבקרה תחת ידרשו בתוכנה לקחת חמש יריות (לערבב נפח שווה של דגימות מן המזרק A ו- B מזרק) על fluorimeter זרימה עצר ללא הקלטה את התוצאות.
- פתח את לוח הבקרה תחת ידרשו בתוכנה, תוכנית להקליט את פליטת Cul1AMC יותר מ-60 s. אז לקחת ירייה אחת.
- חזור על שלב 6.2.4 2 x.
- להפוך את השסתום מדגם למיקום למלא . לרוקן את המזרק B, לשטוף עם המאגר סריג.
- מדד נצפתה האגודה קצב קבועים (kobs) של Cul1•Cand1 (איור 4B).
- לשמור את הדגימה במזרק A כמו שלב 6.2.1.
- להתחבר מזרק 3 מ"ל ולטעון B מזרק עם 100 ננומטר Cand1 פלאשבמאגר סריג. להפוך את השסתום דוגמה למיקום כונן .
- השתמש בלוח הבקרה תחת ידרשו בתוכנה כדי לקחת חמש יריות ללא הקלטה את התוצאות.
- פתח את לוח הבקרה תחת ידרשו בתוכנה, תוכנית להקליט את פליטת Cul1AMC יותר מ-60 s. אז לקחת ירייה אחת.
- חזור על שלב 6.3.4 2 x.
- לרוקן את המזרק B, לשטוף עם המאגר סריג.
- חזור על צעדים 6.3.1–6.3.6 מספר פעמים עם הגדלת ריכוזים של פלאשCand1 במאגר סריג.
- להתאים את השינוי (ירידה) אותות פלואורסצנט נמדד לאורך זמן של כל זריקה לעקומה מעריכית יחיד. זה ייתן kobsבמדידה כל, יחידת s-1. שימו לב כי הבסיס של חישוב זה טוב נדון הדוח הקודם27.
- לחשב את הממוצע, סטיית התקן של kobsבכל ריכוז Cand1 פלאשבשימוש. להתוות את הממוצע kobsכנגד ריכוז Cand1 (איור 4D) ומייצג השיפוע של הקו k- של Cul1•Cand1, עם יחידת ז-1 s-1.
7. למדוד את קצב קבוע דיסוציאציה (k.) של Cul1•Cand1 בנוכחות של חלבון Skp1•F-box.
הערה: השלב זה דומה לשלב 6, עם השינויים הבאים.
- במזרק A, תחת המיקום למלא , לטעון פתרון של 100 ננומטר Cul1AMC•Rbx1 ו- 100 ננומטר Cand1 פלאשבמאגר סריג. להפוך את השסתום מדגם למיקום "נסיעה".
- מזרק ב', תחת המיקום למלא , לטעון פתרון של Skp1•Skp2 (שהוכן בעקבות הדוח הקודם20). להפוך את השסתום מדגם למיקום "נסיעה".
- פתח את לוח הבקרה תחת ידרשו בתוכנה, תוכנית להקליט את פליטת Cul1AMC מעל גיל 30 s. אז לקחת ירייה אחת. אותות פלואורסצנט להגדיל לאורך זמן לאחר ערבוב פתרונות של מזרק A ו- B מזרק (איור 5).
תוצאות
כדי לבדוק את סריג בין Cul1AMC , פלאשCand1, קבענו לראשונה את עוצמת הפליטה של 70 nM Cul1AMC (התורם) ו- 70 nM Cand1 פלאש(מקבל), בהתאמה (איור 3 אקווים-C, כחול). כל ניתוח, רק פסגה אחת פליטה היה נוכח, ולא את פליטת Cand1 פלאש(מקבל) היה נמוך. כאשר 70 nM כל ?...
Discussion
סריג הוא תופעה פיזיקלית זה עניין רב בלימוד והבנה של מערכות ביולוגיות19. כאן, אנו מציגים עבור בדיקה ושימוש סריג ללמוד את איגוד קינטיקה של שני חלבונים שמעצבת פרוטוקול. בעת עיצוב סריג, שקלנו שלושה גורמים עיקריים: ספקטרלי החפיפה בין התורם פליטה, מקבל עירור, המרחק בין את fluorophores שני, ?...
Disclosures
המחברים אין לחשוף.
Acknowledgements
אנו מודים שן שו-Ou (המכון הטכנולוגי של קליפורניה) לדיון תובנה על התפתחות וזמינותו סריג. מ. ג, Y.Z. ו- X.L. במימון קרנות הפעלה מאוניברסיטת Purdue כדי Y.Z., X.L.This עבודה נתמך בחלקו על ידי מענק זרע מן המרכז של אוניברסיטת Purdue עבור ביולוגית הצמח.
Materials
| Name | Company | Catalog Number | Comments |
| Anion exchange chromatography column | GE Healthcare | 17505301 | HiTrap Q FF anion exchange chromatography column |
| Benchtop refrigerated centrifuge | Eppendorf | 2231000511 | |
| BL21 (DE3) Competent Cells | ThermoFisher Scientific | C600003 | |
| Calcium Chloride | Fisher Scientific | C78-500 | |
| Cation exchange chromatography column | GE Healthcare | 17505401 | HiTrap SP Sepharose FF |
| Desalting Column | GE Healthcare | 17085101 | |
| Floor model centrifuge (high speed) | Beckman Coulter | J2-MC | |
| Floor model centrifuge (low speed) | Beckman Coulter | J6-MI | |
| Fluorescence SpectraViewer | ThermoFisher Scientific | https://www.thermofisher.com/us/en/home/life-science/cell-analysis/labeling-chemistry/fluorescence-spectraviewer.html | |
| FluoroMax fluorimeter | HORIBA | FluoroMax-3 | |
| FPLC | GE Healthcare | 29018224 | |
| GGGGAMC peptide | New England Peptide | custom synthesis | |
| Glutathione beads | GE Healthcare | 17075605 | |
| Glycerol | Fisher Scientific | G33-500 | |
| HEPES | Fisher Scientific | BP310-100 | |
| Isopropyl-β-D-thiogalactoside (IPTG) | Fisher Scientific | 15-529-019 | |
| LB Broth | Fisher Scientific | BP1426-500 | |
| Ni-NTA agarose | Qiagen | 30210 | |
| Ovalbumin | MilliporeSigma | A2512 | |
| pGEX-4T-2 vector | GE Healthcare | 28954550 | |
| Protease inhibitor cocktail | MilliporeSigma | 4693132001 | |
| Reduced glutathione | Fisher Scientific | BP25211 | |
| Refrigerated shaker | Eppendorf | M1282-0004 | |
| Rosetta Competent Cells | MilliporeSigma | 70953-3 | |
| Size exclusion chromatography column | GE Healthcare | 28990944 | Superdex 200 10/300 GL column |
| Sodium Chloride (NaCl) | Fisher Scientific | S271-500 | |
| Stopped-flow fluorimeter | Hi-Tech Scientific | SF-61 DX2 | |
| TCEP·HCl | Fisher Scientific | PI20490 | |
| Thrombin | MilliporeSigma | T4648 | |
| Tris Base | Fisher Scientific | BP152-500 | |
| Ultrafiltration membrane | MilliporeSigma | UFC903008 | Amicon Ultra-15 Centrifugal Filter Units, Ultra-15, 30,000 NMWL |
References
- Stumpf, M. P. H., et al. Estimating the size of the human interactome. Proceedings of the National Academy of Sciences of the United States of America. 105 (19), 6959-6964 (2008).
- Kuzmanov, U., Emili, A. Protein-protein interaction networks: probing disease mechanisms using model systems. Genome Medicine. 5 (4), 37 (2013).
- Titeca, K., Lemmens, I., Tavernier, J., Eyckerman, S. Discovering cellular protein-protein interactions: Technological strategies and opportunities. Mass Spectrometry Reviews. , 1-33 (2018).
- Lapetina, S., Gil-Henn, H. A guide to simple, direct, and quantitative in vitro binding assays. Journal of Biological Methods. 4 (1), 62 (2017).
- Zheng, X., Bi, C., Li, Z., Podariu, M., Hage, D. S. Analytical methods for kinetic studies of biological interactions: A review. Journal of Pharmaceutical and Biomedical Analysis. 113, 163-180 (2015).
- Pierce, N. W., et al. Cand1 promotes assembly of new SCF complexes through dynamic exchange of F box proteins. Cell. 153 (1), 206-215 (2013).
- Liu, X., Reitsma, J. M., Mamrosh, J. L., Zhang, Y., Straube, R., Deshaies, R. J. Cand1-Mediated Adaptive Exchange Mechanism Enables Variation in F-Box Protein Expression. Molecular Cell. 69 (5), 773-786 (2018).
- Zheng, N., et al. Structure of the Cul1-Rbx1-Skp1-F boxSkp2 SCF ubiquitin ligase complex. Nature. 416 (6882), 703-709 (2002).
- Kleiger, G., Deshaies, R. Tag Team Ubiquitin Ligases. Cell. 166 (5), 1080-1081 (2016).
- Zheng, J., et al. CAND1 binds to unneddylated CUL1 and regulates the formation of SCF ubiquitin E3 ligase complex. Molecular Cell. 10 (6), 1519-1526 (2002).
- Hwang, J. W., Min, K. W., Tamura, T. A., Yoon, J. B. TIP120A associates with unneddylated cullin 1 and regulates its neddylation. FEBS Letters. 541 (1-3), 102-108 (2003).
- Min, K. W., Hwang, J. W., Lee, J. S., Park, Y., Tamura, T., Yoon, J. B. TIP120A associates with cullins and modulates ubiquitin ligase activity. Journal of Biological Chemistry. 278 (18), 15905-15910 (2003).
- Goldenberg, S. J., et al. Structure of the Cand1-Cul1-Roc1 complex reveals regulatory mechanisms for the assembly of the multisubunit cullin-dependent ubiquitin ligases. Cell. 119 (4), 517-528 (2004).
- Chuang, H. W., Zhang, W., Gray, W. M. Arabidopsis ETA2, an apparent ortholog of the human cullin-interacting protein CAND1, is required for auxin responses mediated by the SCF(TIR1) ubiquitin ligase. Plant Cell. 16 (7), 1883-1897 (2004).
- Feng, S., et al. Arabidopsis CAND1, an Unmodified CUL1-Interacting Protein, Is Involved in Multiple Developmental Pathways Controlled by Ubiquitin/Proteasome-Mediated Protein Degradation. the Plant Cell Online. 16 (7), 1870-1882 (2004).
- Cheng, Y., Dai, X., Zhao, Y. AtCAND1, a HEAT-repeat protein that participates in auxin signaling in Arabidopsis. Plant Physiology. 135 (June), 1020-1026 (2004).
- Lo, S. -. C., Hannink, M. CAND1-Mediated Substrate Adaptor Recycling Is Required for Efficient Repression of Nrf2 by Keap1. Molecular and Cellular Biology. 26 (4), 1235-1244 (2006).
- Okamoto, K., Sako, Y. Recent advances in FRET for the study of protein interactions and dynamics. Current Opinion in Structural Biology. 46, 16-23 (2017).
- Hussain, S. A. An introduction to fluorescence resonance energy transfer (FRET). arXiv preprint. , (2009).
- Li, T., Pavletich, N. P., Schulman, B. A., Zheng, N. High-level expression and purification of recombinant SCF ubiquitin ligases. Methods in Enzymology. 398 (1996), 125-142 (2005).
- Popp, M. W., Antos, J. M., Grotenbreg, G. M., Spooner, E., Ploegh, H. L. Sortagging: A versatile method for protein labeling. Nature Chemical Biology. 3 (11), 707-708 (2007).
- Antos, J. M., Ingram, J., Fang, T., Pishesha, N., Truttmann, M. C., Ploegh, H. L. Site-Specific Protein Labeling via Sortase-Mediated Transpeptidation. Current Protocols in Protein Science. 89, (2017).
- Froger, A., Hall, J. E. Transformation of Plasmid DNA into E. coli Using the Heat Shock Method. Journal of Visualized Experiments. , (2007).
- Simpson, R. J. SDS-PAGE of Proteins. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Kleiger, G., Saha, A., Lewis, S., Kuhlman, B., Deshaies, R. J. Rapid E2-E3 Assembly and Disassembly Enable Processive Ubiquitylation of Cullin-RING Ubiquitin Ligase Substrates. Cell. 139 (5), 957-968 (2009).
- Biro, F. N., Zhai, J., Doucette, C. W., Hingorani, M. M. Application of Stopped-flow Kinetics Methods to Investigate the Mechanism of Action of a DNA Repair Protein. Journal of Visualized Experiments. (37), 2-8 (2010).
- Patel, J. T., Belsham, H. R., Rathbone, A. J., Friel, C. T. Use of Stopped-Flow Fluorescence and Labeled Nucleotides to Analyze the ATP Turnover Cycle of Kinesins. Journal of Visualized Experiments. (92), 1-6 (2014).
- Bajar, B. T., Wang, E. S., Zhang, S., Lin, M. Z., Chu, J. A guide to fluorescent protein FRET pairs. Sensors (Switzerland). 16 (9), 1-24 (2016).
- Chen, A. K., Cheng, Z., Behlke, M. A., Tsourkas, A. Assessing the sensitivity of commercially available fluorophores to the intracellular environment. Analytical Chemistry. 80 (19), 7437-7444 (2008).
- Lin, C. T., Rorabacher, D. B. Mathematical approach for stopped-flow kinetics of fast second-order reactions involving inhomogeneity in the reaction cell. Journal of Physical Chemistry. 78 (3), 305-308 (1974).
- Toseland, C. P., Geeves, M. A. Rapid Reaction Kinetic Techniques. Fluorescent Methods for Molecular Motors. , 49-65 (2014).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: Synthesis and biological applications. Journal of the American Chemical Society. 124 (21), 6063-6076 (2002).
- Lin, C. W., Ting, A. Y. Transglutaminase-catalyzed site-specific conjugation of small-molecule probes to proteins in vitro and on the surface of living cells. Journal of the American Chemical Society. 128 (14), 4542-4543 (2006).
- Yin, J., et al. Genetically encoded short peptide tag for versatile protein labeling by Sfp phosphopantetheinyl transferase. Proceedings of the National Academy of Sciences of the United States of America. 102 (44), 15815-15820 (2005).
- Diaspro, A., Chirico, G., Usai, C., Ramoino, P., Dobrucki, J. Photobleaching. Handbook Of Biological Confocal Microscopy. , 690-702 (2006).
- Aoki, K., Kamioka, Y., Matsuda, M. Fluorescence resonance energy transfer imaging of cell signaling from in vitro to in vivo: Basis of biosensor construction, live imaging, and image processing. Development, Growth & Differentiation. 55 (4), 515-522 (2013).
- Kilic, S., et al. Single-molecule FRET reveals multiscale chromatin dynamics modulated by HP1α. Nature Communications. 9 (1), (2018).
- Shen, K., Arslan, S., Akopian, D., Ha, T., Shan, S. O. Activated GTPase movement on an RNA scaffold drives co-translational protein targeting. Nature. 492 (7428), 271-275 (2012).
- Bajar, B. T., et al. Improving brightness and photostability of green and red fluorescent proteins for live cell imaging and FRET reporting. Scientific Reports. 6 (February), 1-12 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved