É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Usando na transferência de energia de ressonância de fluorescência de Vitro para estudar a dinâmica dos complexos de proteínas em uma escala de tempo de milissegundo
Neste Artigo
Resumo
Interações da proteína-proteína são essenciais para sistemas biológicos, e estudos sobre a cinética de ligação fornecem insights sobre a dinâmica e a função dos complexos da proteína. Nós descrevemos um método que quantifica os parâmetros cinéticos de uma proteína complexa usando transferência de energia de ressonância de fluorescência e a técnica de fluxo parou.
Resumo
As proteínas são os operadores primários de sistemas biológicos, e geralmente interagem com outro macro - ou pequenas moléculas para realizar suas funções biológicas. Tais interações podem ser altamente dinâmicas, significando as subunidades interagindo constantemente são associadas e dissociadas a determinadas taxas. Enquanto a afinidade de ligação usando técnicas tais como suspenso quantitativo revela a força da interação, estudando a cinética de ligação de medição fornece insights sobre como rápido a interação ocorre e quanto tempo cada complexo pode existir. Além disso, medir a cinética da interação na presença de um fator adicional, como um fator de troca de proteína ou uma droga, ajuda a revelar o mecanismo pelo qual a interação é regulada por outro fator, fornecendo conhecimentos importantes para o avanço de pesquisas biológicas e médicas. Aqui, descrevemos um protocolo para avaliar a cinética de ligação de um complexo de proteínas que tem uma taxa de associação intrínseca elevada e pode ser rapidamente dissociado por outra proteína. O método utiliza a transferência de energia de ressonância de fluorescência para relatar a formação do complexo proteína in vitro, e permite monitorar a rápida associação e dissociação do complexo em tempo real em um fluorímetro de fluxo parou. Usando este ensaio, as constantes de taxa de associação e dissociação da proteína complexa são quantificadas.
Introdução
Atividades biológicas em última análise, são realizadas por proteínas, mais do que interagir com outras pessoas para funções biológicas. Usando uma abordagem computacional, a quantidade total de interações da proteína-proteína em humanos é estimada em 650.000 ~1, e interrupção dessas interações muitas vezes leva a doenças2. Devido a seus papéis essenciais no controle de processos celulares e organismos, inúmeros métodos foram desenvolvidos para estudar interações da proteína-proteína, tais como levedura-dois-híbrido, complementação de fluorescência bimolecular, split-luciferase complementação e co-imunoprecipitação ensaio3. Enquanto esses métodos são bons em descobrir e confirmando as interações da proteína-proteína, eles são geralmente não-quantitativos e, portanto, fornecem informações limitadas sobre a afinidade entre os parceiros de proteína interagindo. Pull-downs quantitativas podem ser usados para medir a afinidade de ligação (por exemplo, a constante de dissociação Kd), mas ele não mede a cinética da ligação, nem pode ser aplicada quando o Kd é muito baixa devido a uma insuficiente relação sinal-ruído4. Espectroscopia de ressonância (SPR) de plasmon de superfície quantifica a cinética de ligação, mas requer uma superfície específica e a imobilização de um reagente na superfície, o que potencialmente pode alterar a propriedade de ligação do reagente5. Além disso, é difícil para SPR medir Associação rápida e taxas de dissociação5, e não é apropriado usar SPR para caracterizar o evento de troca de subunidades de proteínas em um complexo proteico. Aqui, descrevemos um método que permite medir taxas de proteína complexa montagem e desmontagem em uma escala de tempo em milissegundos. Este método foi essencial para determinar a função de Cullin -umssociated -Nedd8 -dissociated proteína 1 (Cand1) como o F-caixa proteína troca fator6,7.
Cand1 regula a dinâmica da E3 ligases do proteína (SCF) Skp1•Cul1•F-caixa, que pertencem à grande família de ligases do ubiquitin Cullin-anel. SCFs consistem no neddylation Cul1, que se liga a proteína de domínio de anel Rbx1, e uma proteína F-caixa intercambiável, o que recruta substratos e liga Cul1 através da proteína do adaptador Skp18. Como um ligase E3, SCF catalisa a conjugação da ubiquitina de seu substrato, e é ativado quando o substrato é recrutado pela proteína F-box, e quando Cul1 é modificado pela proteína ubiquitina-como Nedd89. Cand1 liga Cul1 sem modificações, e após a ligação, ele perturba tanto a associação de proteína Skp1•F-caixa com Cul1 e a conjugação de Nedd8 de Cul110,11,12,13. Como resultado, Cand1 parecia ser um inibidor da atividade do SCF in vitro, mas Cand1 deficiência em organismos causou defeitos que sugere um papel positivo de Cand1 na regulação de atividades SCF em vivo14,15,16 , 17. este paradoxo foi finalmente explicado por um estudo quantitativo que revelou as interações dinâmicas entre Cul1, Cand1 e Skp1•F-caixa de proteína. Utilizando ensaios de transferência (FRET) de energia ressonância fluorescência que detectam a formação dos complexos SCF e Cul1•Cand1, a associação e dissociação taxa constantes (k,na e kfora, respectivamente) foram medido individualmente. As medições revelaram que tanto Cand1 e Skp1•F-caixa de forma extremamente apertado complexo proteico com Cul1, mas o kfora do SCF é dramaticamente aumentado pela Cand1 e o kfora de Cul1•Cand1 é dramaticamente aumentada por Skp1•F-caixa proteína6,7. Estes resultados fornecem o apoio inicial e crítico para definir o papel de Cand1 como um fator de troca de proteína, que catalisa a formação de novos complexos SCF através da reciclagem de Cul1 desde os antigos complexos SCF.
Aqui, apresentamos o processo de desenvolvimento e usando o ensaio FRET para estudar a dinâmica do complexo Cul1•Cand17, e o mesmo princípio pode ser aplicado para estudar a dinâmica de várias biomoléculas. TRASTE ocorre quando um doador está animado com o comprimento de onda adequado, e um aceitador com espectro de excitação sobrepondo o espectro de emissão do doador está presente dentro de uma distância de 10 a 100 Å. O estado excitado é transferido para o aceitador, desse modo diminuindo a intensidade do doador e aumentando a intensidade de aceitador18. A eficiência do traste (E) depende do raio de Förster (R0) e a distância entre o doador e aceptor fluorophores (r) e é definida por: E = R06/ (R0 6 + r6). O raio de Förster (R0) depende de alguns fatores, incluindo a orientação angular de dipolo, a sobreposição espectral do par doador-aceitador e a solução usada19. Para aplicar o traste do ensaio em um fluorímetro de fluxo parou, que monitora a mudança das emissões doador em tempo real e permite medições de rápido k,na e kfora, é necessário estabelecer FRET eficiente que resulta em uma redução significativa das emissões de doador. Portanto, projetar FRET eficiente, escolhendo o par apropriado de corantes fluorescentes e sites sobre as proteínas alvo para anexar as tinturas é importante e será discutido neste protocolo.
Protocolo
1. design o ensaio de traste.
- Baixe o arquivo de estrutura do complexo do Cul1•Cand1 do banco de dados da proteína (arquivo 1U6G).
- Exiba a estrutura de complexos em PyMOL o Cul1•Cand1.
- Use a função de medição no menu Assistente de PyMOL para estimar a distância entre o primeiro aminoácido da Cand1 e o último aminoácido de Cul1 (Figura 1).
- Carregar o Visualizador de espectros on-line (ver Tabela de materiais) e ver a excitação e espectros de emissão de 7-amino-4-metilcumarina (AMC) e FlAsH simultaneamente (Figura 2). Observe que a AMC é o doador FRET e FlAsH é o aceitador de traste.
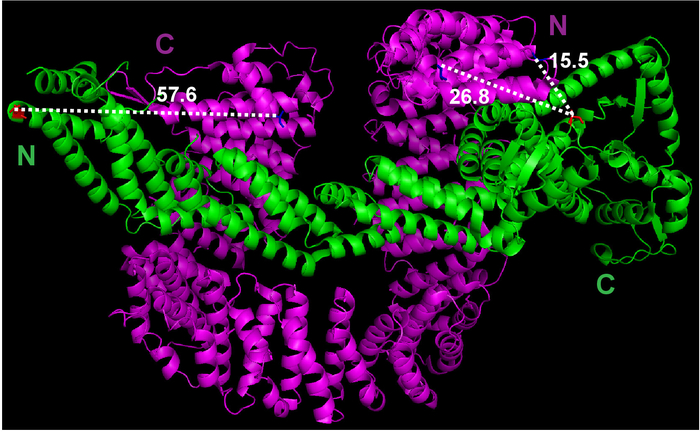
Figura 1: A estrutura de cristal de Cul1•Cand1 e medição da distância entre o potencial rotulagem sites. O arquivo de estrutura de cristal foi baixado do Protein Data Bank (arquivo 1U6G) e visualizado em PyMOL. Medições entre átomos selecionados foram feitas por PyMOL. Clique aqui para ver uma versão maior desta figura.
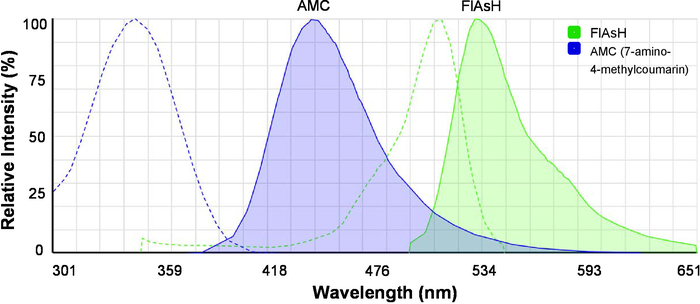
Figura 2: A excitação e espectros de emissão das tinturas fluorescentes para FRET. Espectros de AMC (7-amino-4-metilcumarina) e FlAsH são mostrados. Linhas tracejadas indicam espectros de excitação e linhas sólidas indicam espectros de emissão. A imagem foi originalmente gerada pela SpectraViewer de fluorescência e foi modificada para melhor clareza. Clique aqui para ver uma versão maior desta figura.
2. preparação de Cul1 •Rbx1 daAMC, a proteína de doador FRET
- Construa plasmídeos para expressar o humano Cul1sortase•Rbx1 em células de Escherichia coli . Observe que os dois plasmídeos expressando co humano Cul1•Rbx1 em células de Escherichia coli são descritos em detalhes no relatório anterior20.
- Adicione uma sequência de DNA codificação "LPETGGHHHHHH" (marca sortase-His6 ) à extremidade 3' do Cul1 a sequência de código através de PCR padrão e clonagem métodos21,22.
- Sequenciar o plasmídeo novo para confirmar a inserção do gene é exata.
- Express co Cul1sortase•Rbx1 em células de Escherichia coli . O método é derivado de um anterior relatório20.
- Mix 100 ng cada dos dois plasmídeos com BL21 (DE3) células quimicamente competentes para transformação co usando o calor choque método23. Desenvolvem-se células na placa de ágar LB contendo 100 µ g/mL ampicilina e cloranfenicol 34 µ g/mL a 37 ° C durante a noite.
- Inocular 50 mL de cultura LB com colônias recém transformadas e crescer durante a noite a 37 ° C, com agitação de 250 rpm. Isto dá uma cultura starter.
- Inocular 6 frascos, cada um com 1 L de meio LB, com cultura de acionador de partida 5 mL cada e crescer a 37 ° C com 250 rpm agitação até que o OD600 ~ 1.0. Arrefecer a cultura a 16 ° C e adicionar isopropil-β-D-thiogalactoside (IPTG) a 0,4 mM. Manter a cultura a 16 ° C durante a noite com a agitação de 250 rpm.
- Colheita de células de Escherichia coli através de centrifugação a 5.000 x g durante 15 min e coletar pelotas de célula em tubos de Erlenmeyer de 50 mL.
Nota: As pelotas de célula podem ser processadas para a purificação de proteínas ou ser congeladas a-80 ° C antes de prosseguir para as etapas de purificação de proteínas.
- Purificação da Cul1sortase•Rbx1 complexo. Este método é derivado de um anterior relatório20.
- Adicione 50 mL de tampão de lise (30 mM Tris-HCl, NaCl, 5 mM DTT, 10% de glicerol, 1 comprimido de 200 mM de inibidor da protease cocktail, pH 7,6) ao pellet de células de Escherichia coli expressando Cul1sortase•Rbx1.
- Lise as células no gelo com sonication em amplitude de 50%. Alterne entre 1 segundo e 1 segundo e concorrer a 3 min.
- Repita a etapa 2.3.2 x 2-3.
- Transfira o lisado celular em um tubo de centrifugação de 50 mL e remova os restos de células por centrifugação a 25.000 x g durante 45 min.
- Incube as células claras lisada com 5 mL de grânulos de glutationa em 4 ° C por 2 h.
- Centrifugue a mistura de grânulos-lisado a 1.500 x g por 2 min a 4 ° C. Remova o sobrenadante.
- Lave os grânulos com 5 mL de tampão de lise (com não inibidores de protease) e remover o sobrenadante após centrifugação a 1.500 x g por 2 min a 4 ° C.
- Repita a etapa 2.3.7 2x.
- Adicionar 3 mL de tampão de Lise para os grânulos lavados e transferir o chorume do grânulo para uma coluna vazia.
- Adicione 5 mL de tampão de eluição (50 mM Tris-HCl, 200 mM NaCl, glutationa reduzida de 10mm, pH 8.0) na coluna. Incubar durante 10 min e recolher o eluato.
- Repita a etapa 2.3.10 x 3-4.
- Adicionar 200 µ l de trombina de 5 mg/mL (ver Tabela de materiais) para o eluato de missanga a glutationa e incubar durante a noite a 4 ° C.
Nota: O protocolo pode ser pausado aqui. - Dilua a amostra de proteína com tampão A (25mm HEPES, 1 milímetro DTT, 5% de glicerol, pH 6.5) três vezes.
- Equilibrar uma coluna de cromatografia de permuta catiónica (ver Tabela de materiais) em um sistema FPLC com Buffer A.
- Carrega a amostra de proteína para a coluna de cromatografia de troca de cátion equilibrado em uma taxa de fluxo de 0,5 mL/min.
- Eluir a proteína com um gradiente de NaCl em 40 mL pela mistura de tampão A e 0 a 50% B Buffer (pH 6,5 de 25mm HEPES, 1 M NaCl, 1 milímetro DTT, 5% de glicerol,) em uma taxa de fluxo de 1 mL/min.
- Verifique a proteína eluted em diferentes frações utilizando SDS-PAGE24.
Nota: O protocolo pode ser pausado aqui. - As frações de eluato contendo Cul1sortase•Rbx1 da piscina.
- Concentre-se em pool Cul1sortase•Rbx1 amostra de 2,5 mL, passando a reserva através de uma membrana de ultrafiltração (corte de 30 kDa).
- Adicione AMC para o C-terminal da Cul1 através de mediada por sortase transpeptidation21,22.
- Alterar o buffer na amostra Cul1sortase•Rbx1 para o buffer de sortase (50 mM Tris-HCl, 150 mM NaCl, 10 mM CaCl2, pH 7,5) usando uma coluna do desalting (ver Tabela de materiais).
- Equilibrar uma coluna do desalting com 25 mL de tampão de sortase.
- Carrega 2,5 mL da amostra de •Rbx1 desortaseCul1 para a coluna. Descarte o escoamento.
- Eluir a amostra com 3,5 mL de tampão de sortase. Colete o escoamento.
- Em 900 µ l da solução de •Rbx1 de Cul1sortaseno buffer sortase, adicione 100 µ l de solução sortase 600 µM purificado e 10 µ l de peptídeo de GregorioAMC de 25 mM. Incube a mistura de reação a 30 ° C na noite escura. Nota que esta etapa irá gerar Cul1AMC•Rbx1.
Cuidado: Corantes fluorescentes são sensíveis à luz, então evite expô-los à luz ambiente durante a preparação da proteína e amostra tanto quanto possível.
Nota: O protocolo pode ser pausado aqui. - Adicionar 50 µ l de grânulos de agarose Ni-NTA a mistura de reacção e incubar a temperatura ambiente por 30 min.
- Os grânulos de agarose Ni-NTA de pelotas por centrifugação a 5.000 x g por 2 min e recolher o sobrenadante.
- Equilibrar uma coluna de cromatografia de exclusão de tamanho (ver Tabela de materiais) com buffer (30 mM Tris-HCl, 100 mM de NaCl, 1 milímetro DTT, 10% de glicerol) no sistema FPLC.
- Carregar todos os Cul1AMC•Rbx1 amostra na coluna de cromatografia de exclusão de tamanho. Eluir com volume de coluna x 1,5 de buffer.
- Verifique as frações de eluato por SDS-PAGE24.
- As frações de eluato contendo Cul1 da piscinaAMC•Rbx1.
- Medir a concentração de proteína usando sua absorbância em 280 nm com um espectrofotômetro.
- Alíquota da solução de proteína e loja a-80 ° C.
Nota: O protocolo pode ser pausado aqui.
- Alterar o buffer na amostra Cul1sortase•Rbx1 para o buffer de sortase (50 mM Tris-HCl, 150 mM NaCl, 10 mM CaCl2, pH 7,5) usando uma coluna do desalting (ver Tabela de materiais).
3. preparação de FlAsHCand1, a proteína do aceitador FRET
Nota: A maioria das etapas nesta parte é igual a etapa 2. Condições que diferem são descritas em detalhes abaixo.
- Construa o plasmídeo para expressar o humano Cand1 de TetraCysem células de Escherichia coli .
- Adicionar sequência de DNA codificação "CCPGCCGSG" (marca de tetracysteine/TetraCys) antes do 15th aminoácido de Cand1 através de PCR regular25 (sequências da primeira demão: TGCTGTCCGGGCTGCTGCGGCAGCGGCATGACATCCAGCGACAAGGACTTTAG; CTAACTAGTGTCCATTGATTCCAAG).
- Inserir o produto do PCR em um vetor do pGEX 4T-2. Sequenciar o plasmídeo para confirmar a inserção do gene é exata e no quadro.
- Express TetraCysCand1in Escherichia coli células da mesma forma como passo 2.2, exceto que o plasmídeo é transformado em células competentes de Rosetta.
- Purificação de Cand1 de TetraCyspartir de células de Escherichia coli .
- Lisar as pelotas de célula de e. coli e extrair TetraCysCand1using grânulos de glutationa. Estes passos são os mesmos que passos 2.3.1–2.3.12.
- Dilua o eluato de proteína dos grânulos de glutationa com Buffer C (50 mM Tris-HCl, 1 milímetro DTT, 5% de glicerol, pH 7,5) por três dobras. Equilibrar uma coluna de cromatografia de troca de ânion (ver Tabela de materiais) no sistema FPLC com Buffer C e carga a amostra diluída de proteína em uma taxa de fluxo de 0,5 mL/min.
- Eluir a proteína com um gradiente de NaCl em 40 mL misturando Buffer C e 0 a 50% D Buffer (pH 7.5 de 50 mM Tris-HCl, NaCl de 1 M, 1 mM DTT, 5% de glicerol,) em uma taxa de fluxo de 1 mL/min. Verifique a proteína eluted em diferentes frações utilizando SDS-PAGE24e piscina as frações contendo TetraCysCand1. Observe que o TetraCysCand1 tem um volume maior de retenção de GST livre.
- Concentre a amostra de Cand1 de TetraCysem pool, passando a reserva através de uma membrana de ultrafiltração (corte de 30 kDa).
- Equilibrar a coluna de cromatografia de exclusão de tamanho com buffer de rotulagem (20 mM Tris-HCl, 100 mM de NaCl, 2mm TCEP, 1 mM EDTA, 5% de glicerol) no sistema FPLC. Carregar TetraCysCand1 amostra (500 µ l cada vez) e verificar as frações de eluato por SDS-PAGE24.
- Reunir todas as frações contendo TetraCysCand1 e concentrar a proteína de ~ 40 µM, passando a reserva através de uma membrana de ultrafiltração (corte de 30 kDa). Estimar a concentração de proteína usando sua absorbância em 280 nm. Armazenar a proteína como 50 alíquotas µ l a-80 ° C.
Nota: O protocolo pode ser pausado aqui.
- Preparação de Cand1 FlAsH.
- Adicione 1 µ l de solução FlAsH (ver Tabela de materiais) para a solução de Cand1 50 µ l TetraCys.
- Misture bem e incubar a mistura à temperatura ambiente no escuro para 1-2 h obter Cand1 de FlAsH.
Nota: O protocolo pode ser pausado aqui.
4. preparação de Cand1, a proteína de perseguição FRET
Nota: O protocolo de preparação de proteína é semelhante para a etapa 3, com as seguintes modificações.
- Introduza a sequência de código de Cand1 completos do vetor do pGEX 4T-2.
- Altere o buffer usado na etapa 3.3.5 para um buffer que contém 30 mM Tris-HCl, 100 mM de NaCl, 1 milímetro DTT, 10% de glicerol.
- Elimine etapas 3.3.7 e 3.3.8.
5. testar e confirmar o ensaio FRET
- Preparar o traste buffer contendo 30 mM Tris-HCl, NaCl, 0.5 mM DTT, ovalbumina de 1 mg/mL, pH 7,6, de 100 mM e usar à temperatura ambiente.
- Testar o traste entre Cul1AMC•Rbx1 e Cand1 de FlAsHem um fluorímetro.
- Em 300 µ l de tampão FRET, adicionar Cul1 •Rbx1AMC(FRET doador) a uma concentração final de 70 nM. Transferi a solução para uma cubeta.
- Coloque a cubeta no suporte de amostra de um fluorímetro. Excitar a amostra com luz de excitação 350 nm e digitalizar os sinais de emissão de 400 nm a 600 nm em incrementos de 1 nm.
- Repita a etapa 5.2.1 mas mudança Cul1AMC•Rbx1 para FlAsHCand1 (FRET aceitador). Examinar a amostra de Cand1 FlAsHusando o mesmo método da etapa 5.2.2.
- (Opcional) Excitar o Cand1 FlAsHcom luz de excitação 510 nm e digitalizar o sinal de emissão de 500 nm a 650 nm.
- No buffer FRET 300 µ l, adicionar ambos Cul1AMC•Rbx1 e Cand1 de FlAsHpara uma concentração final de 70 nM. Analise a amostra da mesma forma como na etapa 5.2.2.
- Confirmar o traste entre Cul1 •Rbx1AMCe Cand1 FlAsHadicionando a proteína de perseguição (sem rótulo Cand1) (Figura 3).
- Em 300 µ l de tampão FRET, adicione 70 nM Cul1AMC•Rbx1 e 700 nM Cand1. Verificação das emissões de amostra como na etapa 5.2.2.
- Em 300 µ l de tampão FRET, adicione 70 nM Cand1 FlAsHe 700 nM Cand1. Verificação das emissões de amostra como na etapa 5.2.2.
- Em 300 µ l de tampão FRET, adicione 70 nM Cul1AMC•Rbx1 e 70 nM Cand1 FlAsHe incubar a amostra em temperatura ambiente por 5 min. Em seguida, adicione 700 nM Cand1 e imediatamente após a adição, digitalizar a emissão da amostra como na etapa 5.2.2). Note que este passo é semelhante ao passo 5.2.5.
- No buffer FRET 300 µ l, adicionar sequencialmente 70 nM Cul1AMC•Rbx1, 700 nM Cand1 e 70 nM Cand1 FlAsH. Incubar a amostra em temperatura ambiente por 5 min e digitalizar a emissão da amostra como na etapa 5.2.2. Note que esta é a amostra de perseguição (linha verde na Figura 3).
6. medir a constante de velocidade de associação (k,na) de Cul1•Cand1
Nota: Detalhes de um fluorímetro parou-fluxo de funcionamento tem sido descrito em um anterior relatório26.
- Prepare o fluorímetro fluxo parou para medição.
- Ligue o fluorímetro de fluxo interrompido de acordo com as instruções do fabricante.
- Defina a luz de excitação a 350 nm e uso um filtro passa-faixa que permite a emissão de 450 nm e luz para passar e bloqueia a emissão de 500-650 nm luz.
- Mantenha as válvulas de amostra na posição de preencher e conecte uma seringa de 3 mL com água. Lave as duas seringas de amostra (A e B) com água movendo-se a unidade de seringa amostra acima e para baixo várias vezes. Descarte toda a água utilizada nesta etapa.
- Mantenha as válvulas de amostra na posição de preencher e conecte uma seringa de 3 mL cheia de buffer de traste. Lave as duas seringas de amostra com o buffer FRET movendo o amostra seringa de carro subindo e descendo várias vez. Descarte todo o buffer FRET utilizado nesta etapa.
- Tome uma medida de controle (Figura 4).
- Conecte uma seringa de 3 mL e carregar a seringa A com 100 nM Cul1AMC•Rbx1 no buffer de traste. Gire a válvula de amostra para a posição de dirigir .
- Conecte uma seringa de 3 mL e carregar a seringa B com o buffer de traste. Gire a válvula de amostra para a posição de dirigir .
- Use o Painel de controle sob a adquirir no software para assumir o fluorímetro fluxo parou sem gravar os resultados cinco tiros (misturar igual volume de amostras da seringa A e B da seringa).
- Abra o Painel de controle sob a adquirir no software e programa para gravar a emissão de Cul1AMC mais de 60 s. Em seguida dar um único tiro.
- Repita a etapa 6.2.4 2x.
- Gire a válvula de amostra para a posição de preencher . Esvaziar a seringa B e lave com o buffer de traste.
- Medida observada constantes de velocidade de associação (kobs) de Cul1•Cand1 (Figura 4B).
- Manter a amostra em seringa à mesma etapa 6.2.1.
- Conecte uma seringa de 3 mL e carregar a seringa B com 100 nM Cand1 FlAsHno buffer de traste. Gire a válvula de amostra para a posição de dirigir .
- Use o Painel de controle sob a adquirir no software para levar cinco tiros sem gravar os resultados.
- Abra o Painel de controle sob a adquirir no software e programa para gravar a emissão de Cul1AMC mais de 60 s. Em seguida dar um único tiro.
- Repita a etapa 6.3.4 2x.
- Esvaziar a seringa B e lave com o buffer de traste.
- Repita os passos 6.3.1–6.3.6 várias vezes com concentrações crescentes de FlAsHCand1 no buffer de traste.
- Encaixa a alteração (diminuição) fluorescentes sinais medidos ao longo do tempo de cada tiro para uma única curva exponencial. Isto dará kobsem cada medição, e a unidade é s-1. Note que a base deste cálculo foi bem discutida em um anterior relatório27.
- Calcule a média e desvio padrão de kobspara cada concentração de Cand1 FlAsHutilizada. Plotar a média kobscontra a concentração de Cand1 (Figura 4), e o declive da linha representa o k de Cul1•Cand1, com uma unidade de M-1 s-1.
7. medir a taxa constante de dissociação (kfora) de Cul1•Cand1 na presença de proteína Skp1•F-caixa.
Nota: Este passo é semelhante ao passo 6, com as seguintes modificações.
- Na seringa A, sob a posição de encher , carregar uma solução de 100 nM Cul1AMC•Rbx1 e 100 nM Cand1 FlAsHno buffer de traste. Gire a válvula de amostra para a posição "DRIVE".
- Em seringa B, sob a posição de encher , carrega uma solução de Skp1•Skp2 (preparado na sequência de uma anterior relatório20). Gire a válvula de amostra para a posição "DRIVE".
- Abra o Painel de controle sob a adquirir no software e programa para gravar a emissão de Cul1AMC acima de 30 s. Em seguida dar um único tiro. Os sinais fluorescentes aumentam ao longo do tempo após a mistura de soluções de seringa A e B de seringa (Figura 5).
Resultados
Para testar o traste entre Cul1AMC e FlAsHCand1, primeiro determinamos a intensidade de emissão de 70 nM Cul1AMC (o doador) e 70 nM Cand1 FlAsH(o aceitador), respectivamente (Figura 3A-C, azul linhas). Em cada análise, somente um pico de emissão estava presente e a emissão de Cand1 FlAsH(o aceitador) foi baixa. Quando 70 nM cada de Cul1AMC e FlAsHCand1 foram mist...
Discussão
TRASTE é um fenômeno físico que é de grande interesse para o estudo e compreensão de sistemas biológicos19. Aqui, apresentamos um protocolo para teste e usando o FRET para estudar a cinética de ligação de duas proteínas de interação. Ao projetar o traste, consideramos três fatores principais: a sobreposição espectral entre o doador de emissão e aceitador da excitação, a distância entre os dois fluorophores e a orientação do dipolo do fluorophores28. Par...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Agradecemos perspicaz discussão sobre o desenvolvimento do ensaio FRET-Shu-UO Shan (California Institute of Technology). M.G., Y.Z. e X.L. foram financiados por fundos de inicialização da Universidade de Purdue para Y.Z. e X.L.This trabalho foi financiado em parte por uma concessão de semente da Purdue University Center para a biologia da planta.
Materiais
| Name | Company | Catalog Number | Comments |
| Anion exchange chromatography column | GE Healthcare | 17505301 | HiTrap Q FF anion exchange chromatography column |
| Benchtop refrigerated centrifuge | Eppendorf | 2231000511 | |
| BL21 (DE3) Competent Cells | ThermoFisher Scientific | C600003 | |
| Calcium Chloride | Fisher Scientific | C78-500 | |
| Cation exchange chromatography column | GE Healthcare | 17505401 | HiTrap SP Sepharose FF |
| Desalting Column | GE Healthcare | 17085101 | |
| Floor model centrifuge (high speed) | Beckman Coulter | J2-MC | |
| Floor model centrifuge (low speed) | Beckman Coulter | J6-MI | |
| Fluorescence SpectraViewer | ThermoFisher Scientific | https://www.thermofisher.com/us/en/home/life-science/cell-analysis/labeling-chemistry/fluorescence-spectraviewer.html | |
| FluoroMax fluorimeter | HORIBA | FluoroMax-3 | |
| FPLC | GE Healthcare | 29018224 | |
| GGGGAMC peptide | New England Peptide | custom synthesis | |
| Glutathione beads | GE Healthcare | 17075605 | |
| Glycerol | Fisher Scientific | G33-500 | |
| HEPES | Fisher Scientific | BP310-100 | |
| Isopropyl-β-D-thiogalactoside (IPTG) | Fisher Scientific | 15-529-019 | |
| LB Broth | Fisher Scientific | BP1426-500 | |
| Ni-NTA agarose | Qiagen | 30210 | |
| Ovalbumin | MilliporeSigma | A2512 | |
| pGEX-4T-2 vector | GE Healthcare | 28954550 | |
| Protease inhibitor cocktail | MilliporeSigma | 4693132001 | |
| Reduced glutathione | Fisher Scientific | BP25211 | |
| Refrigerated shaker | Eppendorf | M1282-0004 | |
| Rosetta Competent Cells | MilliporeSigma | 70953-3 | |
| Size exclusion chromatography column | GE Healthcare | 28990944 | Superdex 200 10/300 GL column |
| Sodium Chloride (NaCl) | Fisher Scientific | S271-500 | |
| Stopped-flow fluorimeter | Hi-Tech Scientific | SF-61 DX2 | |
| TCEP·HCl | Fisher Scientific | PI20490 | |
| Thrombin | MilliporeSigma | T4648 | |
| Tris Base | Fisher Scientific | BP152-500 | |
| Ultrafiltration membrane | MilliporeSigma | UFC903008 | Amicon Ultra-15 Centrifugal Filter Units, Ultra-15, 30,000 NMWL |
Referências
- Stumpf, M. P. H., et al. Estimating the size of the human interactome. Proceedings of the National Academy of Sciences of the United States of America. 105 (19), 6959-6964 (2008).
- Kuzmanov, U., Emili, A. Protein-protein interaction networks: probing disease mechanisms using model systems. Genome Medicine. 5 (4), 37 (2013).
- Titeca, K., Lemmens, I., Tavernier, J., Eyckerman, S. Discovering cellular protein-protein interactions: Technological strategies and opportunities. Mass Spectrometry Reviews. , 1-33 (2018).
- Lapetina, S., Gil-Henn, H. A guide to simple, direct, and quantitative in vitro binding assays. Journal of Biological Methods. 4 (1), 62 (2017).
- Zheng, X., Bi, C., Li, Z., Podariu, M., Hage, D. S. Analytical methods for kinetic studies of biological interactions: A review. Journal of Pharmaceutical and Biomedical Analysis. 113, 163-180 (2015).
- Pierce, N. W., et al. Cand1 promotes assembly of new SCF complexes through dynamic exchange of F box proteins. Cell. 153 (1), 206-215 (2013).
- Liu, X., Reitsma, J. M., Mamrosh, J. L., Zhang, Y., Straube, R., Deshaies, R. J. Cand1-Mediated Adaptive Exchange Mechanism Enables Variation in F-Box Protein Expression. Molecular Cell. 69 (5), 773-786 (2018).
- Zheng, N., et al. Structure of the Cul1-Rbx1-Skp1-F boxSkp2 SCF ubiquitin ligase complex. Nature. 416 (6882), 703-709 (2002).
- Kleiger, G., Deshaies, R. Tag Team Ubiquitin Ligases. Cell. 166 (5), 1080-1081 (2016).
- Zheng, J., et al. CAND1 binds to unneddylated CUL1 and regulates the formation of SCF ubiquitin E3 ligase complex. Molecular Cell. 10 (6), 1519-1526 (2002).
- Hwang, J. W., Min, K. W., Tamura, T. A., Yoon, J. B. TIP120A associates with unneddylated cullin 1 and regulates its neddylation. FEBS Letters. 541 (1-3), 102-108 (2003).
- Min, K. W., Hwang, J. W., Lee, J. S., Park, Y., Tamura, T., Yoon, J. B. TIP120A associates with cullins and modulates ubiquitin ligase activity. Journal of Biological Chemistry. 278 (18), 15905-15910 (2003).
- Goldenberg, S. J., et al. Structure of the Cand1-Cul1-Roc1 complex reveals regulatory mechanisms for the assembly of the multisubunit cullin-dependent ubiquitin ligases. Cell. 119 (4), 517-528 (2004).
- Chuang, H. W., Zhang, W., Gray, W. M. Arabidopsis ETA2, an apparent ortholog of the human cullin-interacting protein CAND1, is required for auxin responses mediated by the SCF(TIR1) ubiquitin ligase. Plant Cell. 16 (7), 1883-1897 (2004).
- Feng, S., et al. Arabidopsis CAND1, an Unmodified CUL1-Interacting Protein, Is Involved in Multiple Developmental Pathways Controlled by Ubiquitin/Proteasome-Mediated Protein Degradation. the Plant Cell Online. 16 (7), 1870-1882 (2004).
- Cheng, Y., Dai, X., Zhao, Y. AtCAND1, a HEAT-repeat protein that participates in auxin signaling in Arabidopsis. Plant Physiology. 135 (June), 1020-1026 (2004).
- Lo, S. -. C., Hannink, M. CAND1-Mediated Substrate Adaptor Recycling Is Required for Efficient Repression of Nrf2 by Keap1. Molecular and Cellular Biology. 26 (4), 1235-1244 (2006).
- Okamoto, K., Sako, Y. Recent advances in FRET for the study of protein interactions and dynamics. Current Opinion in Structural Biology. 46, 16-23 (2017).
- Hussain, S. A. An introduction to fluorescence resonance energy transfer (FRET). arXiv preprint. , (2009).
- Li, T., Pavletich, N. P., Schulman, B. A., Zheng, N. High-level expression and purification of recombinant SCF ubiquitin ligases. Methods in Enzymology. 398 (1996), 125-142 (2005).
- Popp, M. W., Antos, J. M., Grotenbreg, G. M., Spooner, E., Ploegh, H. L. Sortagging: A versatile method for protein labeling. Nature Chemical Biology. 3 (11), 707-708 (2007).
- Antos, J. M., Ingram, J., Fang, T., Pishesha, N., Truttmann, M. C., Ploegh, H. L. Site-Specific Protein Labeling via Sortase-Mediated Transpeptidation. Current Protocols in Protein Science. 89, (2017).
- Froger, A., Hall, J. E. Transformation of Plasmid DNA into E. coli Using the Heat Shock Method. Journal of Visualized Experiments. , (2007).
- Simpson, R. J. SDS-PAGE of Proteins. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Kleiger, G., Saha, A., Lewis, S., Kuhlman, B., Deshaies, R. J. Rapid E2-E3 Assembly and Disassembly Enable Processive Ubiquitylation of Cullin-RING Ubiquitin Ligase Substrates. Cell. 139 (5), 957-968 (2009).
- Biro, F. N., Zhai, J., Doucette, C. W., Hingorani, M. M. Application of Stopped-flow Kinetics Methods to Investigate the Mechanism of Action of a DNA Repair Protein. Journal of Visualized Experiments. (37), 2-8 (2010).
- Patel, J. T., Belsham, H. R., Rathbone, A. J., Friel, C. T. Use of Stopped-Flow Fluorescence and Labeled Nucleotides to Analyze the ATP Turnover Cycle of Kinesins. Journal of Visualized Experiments. (92), 1-6 (2014).
- Bajar, B. T., Wang, E. S., Zhang, S., Lin, M. Z., Chu, J. A guide to fluorescent protein FRET pairs. Sensors (Switzerland). 16 (9), 1-24 (2016).
- Chen, A. K., Cheng, Z., Behlke, M. A., Tsourkas, A. Assessing the sensitivity of commercially available fluorophores to the intracellular environment. Analytical Chemistry. 80 (19), 7437-7444 (2008).
- Lin, C. T., Rorabacher, D. B. Mathematical approach for stopped-flow kinetics of fast second-order reactions involving inhomogeneity in the reaction cell. Journal of Physical Chemistry. 78 (3), 305-308 (1974).
- Toseland, C. P., Geeves, M. A. Rapid Reaction Kinetic Techniques. Fluorescent Methods for Molecular Motors. , 49-65 (2014).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: Synthesis and biological applications. Journal of the American Chemical Society. 124 (21), 6063-6076 (2002).
- Lin, C. W., Ting, A. Y. Transglutaminase-catalyzed site-specific conjugation of small-molecule probes to proteins in vitro and on the surface of living cells. Journal of the American Chemical Society. 128 (14), 4542-4543 (2006).
- Yin, J., et al. Genetically encoded short peptide tag for versatile protein labeling by Sfp phosphopantetheinyl transferase. Proceedings of the National Academy of Sciences of the United States of America. 102 (44), 15815-15820 (2005).
- Diaspro, A., Chirico, G., Usai, C., Ramoino, P., Dobrucki, J. Photobleaching. Handbook Of Biological Confocal Microscopy. , 690-702 (2006).
- Aoki, K., Kamioka, Y., Matsuda, M. Fluorescence resonance energy transfer imaging of cell signaling from in vitro to in vivo: Basis of biosensor construction, live imaging, and image processing. Development, Growth & Differentiation. 55 (4), 515-522 (2013).
- Kilic, S., et al. Single-molecule FRET reveals multiscale chromatin dynamics modulated by HP1α. Nature Communications. 9 (1), (2018).
- Shen, K., Arslan, S., Akopian, D., Ha, T., Shan, S. O. Activated GTPase movement on an RNA scaffold drives co-translational protein targeting. Nature. 492 (7428), 271-275 (2012).
- Bajar, B. T., et al. Improving brightness and photostability of green and red fluorescent proteins for live cell imaging and FRET reporting. Scientific Reports. 6 (February), 1-12 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados