需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
通过乙基甲烷硫素突变在小谷物作物中靶向诱导局部病变(TILLING)种群的发展
摘要
描述是使用乙基甲酸酯 (EMS) 作为诱变剂在小谷物作物中开发靶向诱导局部病变(TILLING)人群的协议。还提供了使用Cel-1测定突变检测的协议。
摘要
靶向诱导局部病变在基因组(TILLING)是一个强大的反向遗传学工具,包括化学诱变和检测靶基因的序列变异。TILLING 是一种非常有价值的功能基因组学工具,用于基因验证,特别是在基于转化的方法存在严重局限性的小颗粒中。培养一个强大的诱变种群是确定基于TILLING的基因验证研究的效率的关键。总体突变频率较低的一个蒂林种群表明,必须筛选数量庞大的人群以找到所需的突变,而高诱变浓度会导致种群死亡率高,导致数量不足诱变个体的数量。一旦形成有效种群,有多种方法来检测感兴趣的基因突变,平台的选择取决于实验规模和资源的可用性。Cel-1测定法和基于甘蔗的凝胶基方法用于突变物鉴定,方便、可重复,且平台资源密集度较低。它具有优势,因为它简单,不需要计算知识,并且特别适用于使用基本实验室设备验证少量基因。本文介绍了良好的蒂林种群的发育方法,包括制备剂量曲线、突变种群的诱变和维持,以及使用基于PCR的Cel-1测定对突变种群进行筛查.
引言
基因组中的点突变可以为研究人员提供许多有用的用途。根据它们的性质和位置,这些突变可用于将功能分配给基因,甚至不同感兴趣的蛋白质域。另一方面,作为新基因变异的来源,可以使用表型屏筛选来选择有用的突变,并进一步用于作物改良。TILLING是一个强大的反向遗传学工具,包括化学诱变和检测靶基因中的序列变异。最早在阿拉伯拟南芥1和果蝇黑色素2中开发,TILLING种群已被开发和用于许多小谷物作物,如六倍体面包小麦(三叶草)3,大麦 (Ordeum 庸)4, 四倍体杜鲁姆小麦 (T. 三氯球菌杜鲁姆)5,双发化小麦 (T.单球体)6和"D" 基因组后代小麦Aegilops tauschii7.这些资源已被用来验证基因在调节非生物和生物应激性8,调节开花时间9,并开发营养优越的作物品种5的作用。
除使用烷基化诱变诱变剂(如乙酸甲酸酯(EMS)、阿齐德钠、N-甲基-N-硝酸钠(MNU)和甲基甲酸甲酸酯(MMS)外,其优越性优于其他反向遗传学工具,原因有若干原因。首先,诱变可以在几乎任何物种或植物10品种上进行,并且独立于转化瓶颈,在小谷物11的情况下尤其具有挑战性。其次,除了产生其他基因验证方法可以获得的敲除突变外,还可以诱导一系列误感和拼接突变,从而可以分辨感兴趣的蛋白质的单个域的功能。此外,TILLING生成整个基因组中不朽的突变集合;因此,单个种群可用于多个基因的功能验证。相反,其他反向遗传学工具只生成研究13下的基因特有的资源。通过TILLING识别的有用突变可以用于育种目的,不受调控,不像基因编辑,其非转基因分类在许多国家仍然不确定。这变得特别相关的小谷物,是国际贸易14。
TILLING是一种简单而有效的基因验证策略,需要开发诱变种群以研究感兴趣的基因。发展有效的诱变种群是确定基于TILLING的基因验证研究效率的关键。总体突变频率较低的一个蒂林种群表明,必须对不切实际的大种群进行所需的突变筛查,而高诱变浓度会导致种群死亡率高,且数量不足。诱变个体。一旦形成良好的种群,有多种方法来检测感兴趣的基因突变,平台的选择取决于实验规模和资源的可用性。全基因组测序和外兆体测序已被用来描述具有小基因组15、16的植物中蒂林种群的所有突变。两个TILLING种群的外泄序列测序已在面包和杜鲁姆小麦中进行,供公众识别理想的突变和订购突变的感兴趣线17。就理想突变的可用性而言,它是一个伟大的公共资源;然而,在基因验证研究中,野生型线应具有感兴趣的候选基因。不幸的是,对整个TILLING种群的外在体进行测序,以便在另一个背景下对几个候选基因进行反向遗传学验证,仍然是成本高昂的。Amplicon测序和基于Cel-1的检测已用于检测小麦靶向种群的突变,Cel-1测定更简单,无需计算知识,特别适用于验证少数具有基本基因的基因实验室设备6,18 。
在本文中,介绍了发展良好蒂林种群的方法,包括制备剂量曲线、突变种群的诱变和维持,以及使用基于PCR的Cel-1测定对突变种群进行筛查.该协议已经成功地实施,开发和利用三聚体、三聚体6、大麦、Aegilops tauchii和几个别人。其中包括这些方法的明确细节,以及有用的提示,这将有助于研究人员发展TILLING种群,使用EMS作为诱变任何小谷物植物的选择。
Access restricted. Please log in or start a trial to view this content.
研究方案
1. 有效诱变的剂量曲线的准备
- 将100粒具有基因型感兴趣的种子浸泡在6个250 mL玻璃瓶中(每个烧瓶100个),含有50 mL的蒸馏水。在室温 (RT) 下,在 100 rpm 下摇动 8 小时,以便种子浸渍。
- 在烟气罩中,通过在蒸馏水中分别溶解 0.167、0.249、0.331、0.415 和 0.498 mMS 溶液,制备 50 mL 的 0.4%、0.6%、0.8%、1.0% 和 1.2%(w/v)乙酸甲酸酯 (EMS) 溶液。
注: EMS 在 RT 上是液体,密度为 1.206 g/mL。
注意:在处理 EMS 时,请使用适当的个人防护设备 (PPE)。 - 将5个烧瓶中的水从5个烧瓶中挤出,在每个含有浸渍种子的烧瓶中加入50mL的EMS溶液,这样有6种不同的处理方法,包括0.0%、0.4%、0.6%、0.8%、1.0%和1.2%的EMS溶液。在 75 rpm 和 RT 下摇动烧瓶 16 小时。
- 使用奶酪布分别处理 EMS 溶液并收集处理过的种子。通过添加一卷 EMS 失活溶液(0.1M NaOH,20% w na2S2O 3)24 小时,使已使用的 EMS 解决方案失去激活。此外,使用 EMS 停用溶液处理受污染的烧瓶和移液器尖端 24 小时。
- 在自来水下清洗经EMS处理的种子2小时,将每个种子分别移植到含有灌注土壤的根培养器中。
- 在16小时光照条件下在20~25°C下种植植物。
- 记录移植15天后植物存活率的数据。使用以下等式计算每次治疗的存活率:
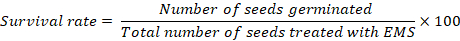
注:如果对照组发芽率低于100%,则在减去对照中未发芽的种子数量后,应计算所有治疗的准确存活率。有效诱变的存活率为40%-60%。可能需要根据经过处理的种子的存活率进行第二轮剂量优化,并根据经过处理的种子的存活率进行改性浓度优化,直到达到40%-60%的致期望致死率。
2. 突变种群的产生和维持
- 将最后一批至少3000粒种子平均(每粒600个种子)浸泡在5个1000mL烧瓶中,里面装有300mL蒸馏水。在 RT 下 100 rpm 下摇动 8 小时以进行浸渍。
注:理想种群的最终大小将取决于基因型的突变频率和多倍水平,但建议使用至少3,000个种子作为六倍体,4,000个种子用于四倍体,7,000多个种子用于二倍体。 - 在烟气罩中,在蒸馏水中制备 1,500 mL 的优化 EMS 浓度溶液。
- 将水从烧瓶中挤出,并在每个装有600个浸渍种子的烧瓶中加入300ml最佳浓度的EMS溶液。在 75 rpm 和 RT 下摇动烧瓶 16 小时。
- 在奶酪布中处理过的种子。如步骤 1.4 中所做的那样,使用 EMS 停用解决方案停用 EMS 解决方案和处理容器。
- 在自来水下清洗EMS处理的种子2小时。将每个EMS处理的M1种子分别移植到根培养器中。
- 在16小时光周期下在20~25°C下生长M1植物(来自M0种子)。
注:如果感兴趣的基因型具有冬季生长习惯,则可能需要在两叶阶段在4°C下对幼苗进行6周的花期。 - 允许M1植物自授,并为每个肥沃的M1植物分别收获M2种子。
注:为了避免潜在的交叉机会,在麻醉前用授粉袋盖住M1植物的尖峰。 - 从每个M1植物中种植一个M2种子,以避免遗传冗余。
- 在1.1 mL的96口井微管中,在双叶阶段收集M2植物的组织。从每个植物收集约80毫米的叶组织,并在组织收集计划中记录每个样本的ID。
- 使用冻干剂冷冻干燥叶组织,储存在-80°C。
- 在16小时光周期下,将M2工厂保持在20~25°C。
- 定期记录M2植物突变表型的数据。预期的表型为白化、氯、草芽、变异、部分肥沃、无菌等。
- 让M2植物自肥和成熟。单独收获和保存M2植物的M3种子(图1)。
注:M种子用于在反向遗传学研究中验证表型。因此,M 应在阴凉干燥条件下仔细编目和储存。或者,如果种子必须在田间增加,则可以为每个 M 植物种植头行,并且可以单独收获每行中的 M 种子并保存为 TILLING 人口资源。
3. 突变体遗传特征的Cel-1测定
- 根据制造商的建议,使用带有DNA纯化系统的植物DNA提取试剂盒从M2的叶组织中提取DNA(见材料表)。
- 使用分光光度计量化DNA,并使用96个井块中的无核酸酶水将DNA浓度标准化为25纳克/μL。
注:通过在凝胶上运行来检查DNA的质量很重要,因为低质量的DNA(涂污的DNA)可能导致池样品中的假阴性。 - 通过将来自四个 96 个井块的 DNA 组合到一个板中,同时保持每个样本的行和列标识,创建 4x DNA 池。将每个单独样品中的 50 μL DNA 添加到池板中,使每个池板井包含来自四个不同 96 个井块的 200 μL DNA。
- 以池板-排-列格式(例如,池 1 A1 + Box1A1 + Box2A1 + Box3A1 + Box4A1 + Box4A1) 格式对池子 DNA 的标识进行目录。
- 使用基因组特异性引源 (GSP) 页面 为感兴趣的基因设计基因组专用引源 [A1],并针对多倍体物种进行默认设置。对于二倍体,请使用引基 3 使用默认设置设计引体。如果需要,设计多个引物,以覆盖感兴趣的基因的整个编码区域。
注:最新的IWGSC组件可用于使用URGI BLAST工具获取小麦相关基因的序列 。最佳放大素长度在800~1,500bp的范围内。表1显示了六倍体小麦中蜡质基因的引物示例。 - 在集合DNA上运行基因特异性引源的PCR,如下所示:
- 加入5μLPCR缓冲液,2μL(每个)4μM正向和反向引物,0.1μL的DNA聚合酶(见材料表),5μL的池化DNA模板,然后增加体积到25μL使用无核酸酶水。
- 使用触控式曲线在热循环器上运行 PCR 反应,如下所示:95 °C 为 1 分钟,7 个循环为 95°C 1 分钟,67°C 至 60°C 为 1 分钟,每个循环温度降低 1 °C,2 分钟降低 72°C,随后是 30 个 95°C 周期,1 分钟,60°C 1 分钟,72°C 2 分钟,最后在 72°C 下延长 7 分钟。
注:PCR 上方的配置文件适用于使用引材 3 默认设置设计的大多数引材的小麦 DNA 模板。在非特异性扩增的情况下,在进行后续步骤之前,应严格PCR轮廓。
- 在不匹配的DNA之间生成异质双工,使用配置文件在热循环器中孵育PCR产品如下:95°C2分钟,5个周期95°C1秒,95°C至85°C5分钟,每个周期温度降低2°C,60周期85°C至25°C每个周期降低 1 °C。
- 在异质复用PCR产品中加入2.5 μL自制Cel-1内分母,并在45°C下孵育45分钟。通过加入 2.5 μL 的 0.5 M EDTA (pH 8.0) 来终止 Cel-1 反应。
注:Cel-1内糖酶可使用Till等人执行的协议从新鲜芹菜茎中提取。或市售的突变检测试剂盒。 - 在 100 V 的 3.0% 甘蔗凝胶上运行 Cel-1 处理产品,工作 2.5 小时。含有更小和独特的分片带的井,除了全长不洁带外,还将包含突变DNA样本。
- 登康突变池。
- 对于构成步骤 3.9 中确定的突变池的单个 M2 DNA 样本,请遵循步骤 3.6、3.7、3.8 和 3.9。
- 要确定突变体的酶度,为单个 M2 DNA运行两个 PCR(如步骤 3.6 所述),其中第一反应包含 2.5 μL M2 DNA 和 2.5 μL 的野生型 DNA,第二种反应仅包含 5 μL 的 M2 DNA。如果突变是杂音,则两种反应中都会存在一个额外的被锥带。另一方面,只有在突变体是同源性时,才会在第一反应中找到额外的被锥带。
- 为了识别突变的性质,请按照制造商的说明,使用Sanger测序平台对已确认突变体的PCR产品进行测序。
4. 突变频率的计算
注:一个类型突变频率是指该种群个体发生一次突变的平均物理距离。例如,在 TILLING 总体中,突变频率为 1/35 kb,这意味着该总体中平均每 35 kb 的个体拥有 1 个突变。
- 要确定一个TILLING种群的突变频率,计算筛选的碱基总数。
- 要计算筛选的基的总数,请将 PCR 产品大小乘以筛选的个体总数。
- 将筛选的碱基总数除以使用以下方程观察到的唯一突变数,这将产生在给定的 TILLING 总体中拥有 1 个突变的物理区域:
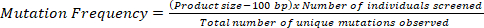
注:为了考虑基于基于角胶凝胶平台的两端50 bp的分辨率限制,在计算中从产品尺寸中减去100 bp。
Access restricted. Please log in or start a trial to view this content.
结果
图2显示了六倍体面包小麦品种Jagger、二倍体小麦三聚体单球体6的剂量曲线,以及小麦Aegilops tauschii7的基因组供体。期望50%生存率的EMS剂量分别为T.单孔、Ae.tauschii和T.aestivum的0.25%、0.6%和0.7%。 六倍体小麦的EMS容差较高,是由于其基因组缓冲能力。然而,尽管两者都是二倍体,EMS对T.单...
Access restricted. Please log in or start a trial to view this content.
讨论
TILLING是一种非常有价值的反向遗传学工具,用于基因验证,特别是对于基于转化的方法存在严重瓶颈的小颗粒。培养高突变频率的诱变种群是进行功能基因组学研究的关键步骤之一。发展强健的TILLING种群的最重要步骤是确定EMS的最佳浓度。M1中40%-60%的存活率是小麦和大麦4、6、18EMS诱变效果的良好指标。
Access restricted. Please log in or start a trial to view this content.
披露声明
作者声明没有相互竞争的经济利益。
致谢
这项工作得到了美国农业部国家粮食和农业研究所、Hatch项目1016879和马里兰州农业实验站通过MAES授权号2956952的支持。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| 96 well 1.1 ml microtubes in microracks | National Scientific | TN0946-08R | For collecting leaf tissues |
| Agarose I biotechnology grade | VWR | 0710-500G | |
| Biosprint 96 DNA Plant Kit | Qiagen | 941558 | Kit for DNA extraction |
| Cel-1 endonuclease | Extracted as described by Till et al 2006 | Single strand specific endonuclease | |
| Centrifuge 5430 R | Eppendorf | ||
| Ethyl methanesulfonate | Sigma Aldrich | M-0880-25G | EMS, Chemical mutagen |
| Freeze Dry/Shell freeze system | Labconco | For lyophilization of leaf tissue | |
| Kingfisher Flex purification system | Thermo fisher scientific | 5400610 | High throughput DNA extraction robot |
| My Taq DNA Polymerase | Bioline | BIO-21107 | |
| Nuclease free water | Sigma aldrich | W4502-1L | |
| NuGenius gel imaging system | Syngene | ||
| Orbit Environ-shaker | Lab-line | ||
| SPECTROstar Nano | BMG LABTECH | Nano drop for DNA quantification | |
| T100 Thermal cycler | BIO-RAD | 1861096 |
参考文献
- McCallum, C. M., Comai, L., Greene, E. A., Henikoff, S. Targeted screening for induced mutations. Nature Biotechnology. 18 (4), 455-457 (2000).
- Bentley, A., MacLennan, B., Calvo, J., Dearolf, C. R. Targeted Recovery of Mutations in Drosophila. Genetics. 156 (3), 1169-1173 (2000).
- Tsai, H., et al. Discovery of Rare Mutations in Populations: TILLING by Sequencing. Plant Physiology. 156 (3), 1257-1268 (2011).
- Caldwell, D. G., et al. A structured mutant population for forward and reverse genetics in Barley (Hordeum vulgare L.). The Plant Journal. 40 (1), 143-150 (2004).
- Hazard, B., et al. Induced Mutations in the Starch Branching Enzyme II ( SBEII ) Genes Increase Amylose and Resistant Starch Content in Durum Wheat. Crop Science. 52 (4), 1754-1766 (2012).
- Rawat, N., et al. A diploid wheat TILLING resource for wheat functional genomics. BMC Plant Biology. 12, 205(2012).
- Rawat, N., et al. TILL-D: An Aegilops tauschii TILLING Resource for Wheat Improvement. Frontiers in Plant Science. 9, (2018).
- Rawat, N., et al. Wheat Fhb1 encodes a chimeric lectin with agglutinin domains and a pore-forming toxin-like domain conferring resistance to Fusarium head blight. Nature Genetics. 48 (12), 1576-1580 (2016).
- Kippes, N., Chen, A., Zhang, X., Lukaszewski, A. J., Dubcovsky, J. Development and characterization of a spring hexaploid wheat line with no functional VRN2 genes. Theoretical and Applied Genetics. 129 (7), 1417-1428 (2016).
- Greene, E. A., et al. Spectrum of Chemically Induced Mutations From a Large-Scale Reverse-Genetic Screen in Arabidopsis. Genetics. 164 (2), 731-740 (2003).
- Harwood, W. A. Advances and remaining challenges in the transformation of barley and wheat. Journal of Experimental Botany. 63 (5), 1791-1798 (2012).
- Henikoff, S., Comai, L. Single-Nucleotide Mutations for Plant Functional Genomics. Annual Review of Plant Biology. 54 (1), 375-401 (2003).
- Uauy, C., et al. A modified TILLING approach to detect induced mutations in tetraploid and hexaploid wheat. BMC Plant Biology. 9 (1), 115(2009).
- Uauy, C., Wulff, B. B. H., Dubcovsky, J. Combining Traditional Mutagenesis with New High-Throughput Sequencing and Genome Editing to Reveal Hidden Variation in Polyploid Wheat. Annual Review of Genetics. 51 (1), 435-454 (2017).
- Li, G., et al. The Sequences of 1504 Mutants in the Model Rice Variety Kitaake Facilitate Rapid Functional Genomic Studies. The Plant Cell. 29 (6), 1218-1231 (2017).
- Jiao, Y., et al. A Sorghum Mutant Resource as an Efficient Platform for Gene Discovery in Grasses. The Plant Cell. 28 (7), 1551-1562 (2016).
- Krasileva, K. V., et al. Uncovering hidden variation in polyploid wheat. Proceedings of the National Academy of Sciences. , 201619268(2017).
- Dong, C., Dalton-Morgan, J., Vincent, K., Sharp, P. A Modified TILLING Method for Wheat Breeding. The Plant Genome. 2 (1), 39-47 (2009).
- Till, B. J., Zerr, T., Comai, L., Henikoff, S. A protocol for TILLING and Ecotilling in plants and animals. Nature Protocols. 1 (5), 2465-2477 (2006).
- Wu, J. -L., et al. Chemical- and Irradiation-induced Mutants of Indica Rice IR64 for Forward and Reverse Genetics. Plant Molecular Biology. 59 (1), 85-97 (2005).
- Feldman, M., Levy, A. A. Genome Evolution Due to Allopolyploidization in Wheat. Genetics. 192 (3), 763-774 (2012).
- Comai, L. The advantages and disadvantages of being polyploid. Nature Reviews Genetics. 6 (11), 836-846 (2005).
- Guo, H., et al. Development of a High-Efficient Mutation Resource with Phenotypic Variation in Hexaploid Winter Wheat and Identification of Novel Alleles in the TaAGP.L-B1 Gene. Frontiers in Plant Science. 8, (2017).
- Rakszegi, M., et al. Diversity of agronomic and morphological traits in a mutant population of bread wheat studied in the Healthgrain program. Euphytica. 174 (3), 409-421 (2010).
- Tsai, H., Ngo, K., Lieberman, M., Missirian, V., Comai, L. Tilling by Sequencing. Plant Functional Genomics: Methods and Protocols. , 359-380 (2015).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。