Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Desarrollo de lesiones locales inducidas dirigidas en las poblaciones de genomas (TILLING) en cultivos de granos pequeños por mutagénesis etnometametanada
En este artículo
Resumen
Descrito es un protocolo para el desarrollo de una población de lesiones locales inducidas dirigidas en genomas (TILLING) en cultivos de grano pequeño con el uso de metanoesulfonato etílico (EMS) como mutágeno. También se proporciona un protocolo para la detección de mutaciones utilizando el ensayo Cel-1.
Resumen
La orientación de lesiones locales inducidas en genomas (TILLING) es una poderosa herramienta genética inversa que incluye mutagénesis química y detección de variación de secuencia en genes diana. TILLING es una herramienta de genómica funcional muy valiosa para la validación de genes, especialmente en granos pequeños en los que los enfoques basados en la transformación tienen serias limitaciones. Desarrollar una población mutageneizada robusta es clave para determinar la eficiencia de un estudio de validación de genes basado en TILLING. Una población de TILLING con una frecuencia de mutación general baja indica que se debe examinar una población imprácticamente grande para encontrar las mutaciones deseadas, mientras que una alta concentración de mutágenos conduce a una alta mortalidad en la población, lo que conduce a una número de individuos mutagenizados. Una vez que se desarrolla una población eficaz, hay múltiples maneras de detectar mutaciones en un gen de interés, y la elección de la plataforma depende de la escala experimental y la disponibilidad de recursos. El ensayo Cel-1 y el enfoque basado en gel de agarosa para la identificación de mutantes es conveniente, reproducible y una plataforma menos intensiva en recursos. Es ventajoso porque es simple, no requiere conocimientos computacionales, y es especialmente adecuado para la validación de un pequeño número de genes con equipos básicos de laboratorio. En el presente artículo, se describen los métodos para el desarrollo de una buena población TILLING, incluyendo la preparación de la curva de dosificación, mutagénesis y mantenimiento de la población mutante, y la detección de la población mutante utilizando el ensayo Cel-1 basado en PCR .
Introducción
Las mutaciones puntuales en los genomas pueden servir para muchos propósitos útiles para los investigadores. Dependiendo de su naturaleza y ubicación, estas mutaciones se pueden utilizar para asignar funciones a genes o incluso dominios distintos de proteínas de interés. Por otro lado, como fuente de nueva variación genética, se pueden seleccionar mutaciones útiles para los rasgos deseados utilizando pantallas de fenotipado y se pueden seguir utilizando en la mejora de los cultivos. TILLING es una poderosa herramienta de genética inversa que incluye mutagénesis química y detección de variación de secuencia en el gen objetivo. Desarrollado por primera vez en Arabidopsis1 y Drosophilia melanogaster2, las poblaciones de TILLING se han desarrollado y utilizado en muchos cultivos de grano pequeño como el trigo de pan hexaploide (Triticum aestivum)3, cebada (Hordeum vulgare)4, tetraploide de trigo duro (T. dicoccoides durum)5, trigo diploide (T. monococcum)6 y el genitor genoma "D" de trigo Aegilops tauschii7 . Estos recursos se han utilizado para validar las funciones de los genes en la regulación de la tolerancia al estrés abiótico y biótico8, la regulación del tiempo de floración9,y el desarrollo de variedades de cultivos nutricionalmente superiores5.
TILLING, junto con el uso de agentes mutagénicos altiladores como el metanoesulfonato etílico (EMS), azida sódica, N-metil-N-nitrosourea (MNU) y metanoesulfonato de metilo (MMS), tiene ventajas sobre otras herramientas genéticas inversas por varias razones. En primer lugar, la mutagénesis se puede llevar a cabo en prácticamente cualquier especie o variedad de la planta10 y es independiente del cuello de botella de transformación, que es particularmente difícil en el caso de los granos pequeños11. En segundo lugar, además de generar mutaciones noqueantes que pueden obtenerse mediante otros enfoques de validación genética, se puede inducir una serie de mutaciones erróneas y de empalme, que pueden discernir las funciones de los dominios individuales de las proteínas de interés12. Además, TILLING genera una colección inmortal de mutaciones a lo largo del genoma; por lo tanto, una sola población se puede utilizar para la validación funcional de múltiples genes. Por el contrario, otras herramientas de genética inversa generan recursos específicos sólo del gen en estudio13. Las mutaciones útiles identificadas a través de TILLING pueden desplegarse con fines de reproducción y no están sujetas a regulación, a diferencia de la edición genética, cuya clasificación no transgénica sigue siendo incierta en muchos países. Esto se vuelve especialmente relevante para los granos pequeños que se comercializan internacionalmente14.
TILLING es una estrategia de validación de genes simple y eficiente y requiere que se desarrollen poblaciones mutagenéticas para investigar genes de interés. El desarrollo de una población mutageneizada eficaz es clave para determinar la eficiencia de un estudio de validación de genes basado en TILLING. Una población de TILLING con una frecuencia de mutación global baja indica que una población imprácticamente grande debe ser examinada para detectar las mutaciones deseadas, mientras que una alta concentración de mutágenos conduce a una alta mortalidad en la población y un número insuficiente de individuos mutagenizados. Una vez que se desarrolla una buena población, hay múltiples maneras de detectar mutaciones en los genes de interés, y la elección de la plataforma depende de la escala experimental y la disponibilidad de recursos. La secuenciación del genoma entero y la secuenciación del exónima se ha utilizado para caracterizar todas las mutaciones en las poblaciones de TILLING en plantas con pequeños genomas15,16. La secuenciación de exomas de dos poblaciones DE TILLING se ha realizado en pan y trigo duro y está a disposición del público para identificar mutaciones deseables y ordenar líneas mutantes de interés17. Es un gran recurso público en términos de disponibilidad de mutaciones deseables; sin embargo, en los estudios de validación de genes, la línea de tipo silvestre debe poseer el gen candidato de interés. Desafortunadamente, sigue siendo prohibitivo secuenciar el exóme de toda la población de TILLING para la validación inversa basada en genética de unos pocos genes candidatos en otro contexto. La secuenciación de Amplicon y los ensayos basados en Cel-1 se han utilizado para detectar mutaciones en poblaciones específicas en trigo, y los ensayos Cel-1 son más simples, no requieren conocimientos computacionales, y son especialmente adecuados para la validación de un pequeño número de genes con equipo de laboratorio6,18.
En el presente artículo, se describen métodos para el desarrollo de una buena población TILLING, incluyendo la preparación de la curva de dosificación, mutagénesis y mantenimiento de la población mutante, y la detección de la población mutante utilizando el ensayo Cel-1 basado en PCR . Este protocolo ya se ha implementado con éxito en el desarrollo y la utilización de poblaciones mutagenizadas de Triticum aestivum, Triticum monoccocum6, cebada, Aegilops tauchii7,y varios Otros. Se incluyen detalles explícitos de estos métodos junto con consejos útiles que ayudarán a los investigadores a desarrollar poblaciones de TILLING, utilizando EMS como mutágeno en cualquier planta de grano pequeño de elección.
Protocolo
1. Preparación de la curva dosis para la mutagénesis efectiva
- Remoje 100 semillas con el genotipo de interés en seis matraces de vidrio de 250 ml (100 en cada matraz) que contengan 50 ml de agua destilada. Agitar a 100 rpm durante 8 h a temperatura ambiente (RT) para la imbibición por las semillas.
- En una campana de humos, prepare 50 ml de 0,4%, 0,6%, 0,8%, 1,0% y 1,2% (p/v) solución de metanoesulfonato de etil (EMS) disolviendo 0,167, 0,249, 0,331, 0,415 y 0,498 ml de EMS en agua destilada, respectivamente.
NOTA: EMS es líquido en RT con una densidad de 1.206 g/ml.
ADVERTENCIA: Utilice el equipo de protección personal (PPE) adecuado mientras manipula EMS. - Decantar el agua de cinco matraces y añadir 50 ml de solución EMS en cada matraz que contenga semillas imbibed para que haya seis tratamientos diferentes con 0.0%, 0.4%, 0.6%, 0.8%, 1.0% y 1.2% solución EMS. Agitar los matraces durante 16 h a 75 rpm y RT.
- Decantar la solución de EMS y recoger las semillas tratadas por separado para cada tratamiento utilizando tela de queso. Inactivar la solución EMS usada añadiendo un volumen de solución de inactivación de EMS (0,1 M NaOH, 20% w/v Na2S2O3) durante 24 horas. Trate también los matraces y las puntas de pipeta contaminados con la solución inactivadora de EMS durante 24 horas.
- Lavar las semillas tratadas con EMS bajo agua corriente del grifo durante 2 h. Trasplante cada semilla individualmente en entrenadores de raíces que contengan tierra para macetas.
- Cultivar plantas a 20–25 oC bajo un período de luz de 16 h.
- Registrar datos sobre la supervivencia de las plantas después de 15 días de trasplante. Utilice la siguiente ecuación para calcular la tasa de supervivencia de cada tratamiento:
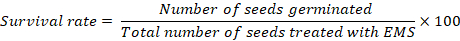
NOTA: Si la tasa de germinación es inferior al 100% en los controles, se deben calcular tasas de supervivencia precisas en todos los tratamientos después de restar el número de semillas que no germinaron en los controles. Una tasa de supervivencia de 40%–60% es deseable para la mutagénesis efectiva. Puede ser necesario realizar una segunda ronda de optimización de dosificación con una concentración modificada de acuerdo con la supervivencia de las semillas tratadas hasta lograr la tasa de letalidad deseable de 40%-60%.
2. Mutagénesis y mantenimiento de la población mutante
- Remoje el lote final de al menos 3.000 semillas dividiendo por igual (600 semillas cada una) en cinco matraces de 1.000 ml que contienen 300 ml de agua destilada. Agitar durante 8 h a 100 rpm bajo RT para imbibición.
NOTA: El tamaño final de la población deseable dependerá de la frecuencia de mutación y el nivel de ploidía del genotipo, pero es aconsejable utilizar al menos 3.000 semillas para hexaloides, 4.000 semillas para tetraploides y más de 7.000 semillas para diploides. - En una campana de humos, prepare 1.500 ml de la solución de concentración de EMS optimizada en agua destilada.
- Decantar el agua de los matraces y añadir 300 ml de la concentración óptima de emS solución en cada matraz que contiene 600 semillas imbilabidos. Agitar los matraces durante 16 h a 75 rpm y RT.
- Decantar el EMS y recoger las semillas tratadas en tela de queso. Inactivar la solución EMS y los recipientes de tratamiento con la solución de inactivación de EMS como se hace en el paso 1.4.
- Lavar las semillas tratadas con EMS bajo agua corriente del grifo durante 2 h. Trasplante cada semilla M1 tratada con EMS individualmente en entrenadores de raíces.
- Cultivar plantas M1 (derivadas de semillas M0) a 20–25 oC bajo un período de luz de 16 h.
NOTA: Puede ser necesario vernalizar las plántulas en la etapa de dos hojas durante 6 semanas a 4 oC, si el genotipo de interés tiene un hábito de crecimiento de tipo invierno. - Permita que las plantas M1 se autopolinen, y cosecha las semillas M2 por separado para cada planta fértil M1.
NOTA: Para evitar posibles posibilidades de un posible cruce, cubra los picos de las plantas M1 con bolsas de polinización antes de la antesis. - Plantar una sola semilla M2 de cada planta M1 para evitar la redundancia genética.
- Recoger tejido de plantas M2 en la etapa de dos hojas en 1,1 ml de 96 microtubos de pozo sorgados. Recoger alrededor de 80 mm de tejido de hoja de cada planta y registrar la identificación de cada muestra en un plan de recolección de tejidos.
- Secar con gel de hoja con un liofilizador y almacenarlo a -80 oC.
- Mantener las plantas M2 a 20–25 oC bajo un período de luz de 16 h.
- Registre datos sobre fenotipos mutantes de las plantas M2 a intervalos regulares. Los fenotipos esperados son albino, clorina, brote herbácico, variado, parcialmente fértil, estéril, etc.
- Permita que las plantas M2 se auto-fertilizan y maduren. Cosecha por separado y guarda las semillas M3 de las plantas M2 (Figura1).
NOTA: Las semillas M se utilizan para validar el fenotipo en estudios de genética inversa. Por lo tanto, M debe catalogarse cuidadosamente y almacenarse en condiciones frías y secas. Alternativamente, si la semilla debe aumentarse en el campo, se pueden plantar filas de cabeza para cada planta M, y las semillas M de cada fila se pueden cosechar y guardar por separado como el recurso de población TILLING.
3. Ensayo Cel-1 para la caracterización genética de mutantes
- Extraer ADN del tejido de la hoja de M2 utilizando un kit de extracción de ADN vegetal con un sistema de purificación de ADN (ver Tabla de Materiales)siguiendo las recomendaciones del fabricante.
- Cuantifique el ADN utilizando un espectrofotómetro y normalice las concentraciones de ADN a 25 ng/-L con agua libre de nucleasas en 96 bloques de pozos.
NOTA: Es importante comprobar la calidad del ADN ejecutándolo con un gel, ya que el ADN de baja calidad (ADN manchado) puede dar lugar a falsos negativos en muestras agrupadas. - Cree 4 grupos de ADN de x combinando ADN de cuatro 96 bloques de pozos en una sola placa, manteniendo la identidad de fila y columna de cada muestra. Añadir 50 l de ADN de cada muestra individual en la placa de la piscina para que cada pozo de la placa de la piscina contenga un total de 200 l de ADN de cuatro bloques de 96 pozos diferentes.
- Cataloge la identidad del ADN agrupado en el formato de Pool Plate-Row-Column (por ejemplo, Pool 1 A1 - Box1A1 + Box2A1 + Box3A1 + Box4A1).
- Imprima imprimadores específicos del genoma [A1] para el gen de interés utilizando la página Genome Specific Primers (GSP) con ajustes predeterminados para especies de poliploides. Para los diploides, utilice Primer 3 para diseñar imprimaciones utilizando la configuración predeterminada. Diseñar múltiples imprimaciones, si es necesario, para cubrir toda la región de codificación del gen de interés.
NOTA: El último ensamblaje de IWGSC se puede utilizar para obtener secuencias para el gen del interés en el trigo utilizando la herramienta URGI BLAST . La longitud óptima del amplicon está en el rango de 800–1,500 bp. La Tabla 1 muestra ejemplos de imprimaciones para genes cerosos en trigo hexaploide. - Ejecute un PCR para imprimaciones específicas de genes en el ADN agrupado de la siguiente manera:
- Añadir 5 l de tampón de PCR, 2 l (cada uno) de imprimaciones de 4 oM hacia delante y hacia atrás, 0,1 l de polimerasa de ADN (ver Tabla de materiales),5 l de plantilla de ADN agrupado, luego aumentar el volumen a 25 ol utilizando agua libre de nucleasas.
- Utilice un perfil de touch down para ejecutar la reacción de PCR en un ciclode ciclo térmico de la siguiente manera: 95 oC durante 1 min, siete ciclos de 95 oC durante 1 min, 67 oC a 60 oC durante 1 min con disminuciones de temperatura de 1 oC por ciclo y 72 oC durante 2 min , seguido de 30 ciclos de 95 oC durante 1 min, 60 oC durante 1 min, 72 oC para 2 min, y la extensión final a 72 oC durante 7 min.
NOTA: Por encima del perfil de PCR funciona en la plantilla de ADN de trigo para la mayoría de las imprimaciones diseñadas usando la configuración predeterminada de imprimación 3. En caso de amplificación no específica, el perfil de PCR debe ser estricto antes de pasar a los siguientes pasos.
- Generar heteroduplexes entre ADN no coincidente mediante la incubación de productos de PCR en ciclor térmico utilizando el perfil de la siguiente manera: 95 oC durante 2 min, cinco ciclos de 95 oC para 1 s, 95 oC a 85 oC durante 1 min con disminuciones de temperatura de 2 oC por ciclo, y 60 ciclos de 85 oC a 25 oC con disminuciones de 1 oC por ciclo.
- Añadir 2,5 ml de endonucleasa Cel-1 casera a los productos de PCR heteroduplexed e incubar durante 45 min a 45oC. Terminar la reacción Cel-1 añadiendo 2,5 ml de 0,5 M EDTA (pH 8.0).
NOTA: Cel-1 endonucleasa se puede extraer de tallos de apio fresco utilizando el protocolo realizado por Till et al.19 Es muy importante probar la actividad y la cantidad óptima de Cel-1 endonucleasa, que se puede probar utilizando mutantes previamente caracterizados o un kit de detección de mutaciones disponible comercialmente. - Ejecute los productos tratados con Cel-1 en un gel de agarosa del 3,0% a 100 V durante 2,5 h. Los pozos que contienen bandas de cleaved más pequeñas y únicas, además de bandas sin deslatir de longitud completa, contendrán la muestra de ADN mutante.
- Piscinas mutantes denconveres.
- Siga los pasos 3.6, 3.7, 3.8 y 3.9 para muestras individuales de ADN M2 que constituyen las agrupaciones mutantes identificadas en el paso 3.9.
- Para determinar la cigosidad de los mutantes, ejecute dos PCR (como se describe en el paso 3.6) para el ADN M2 individual, en el que la primera reacción contiene 2,5 l de ADN M2 y 2,5 l de ADN de tipo salvaje y la segunda reacción contiene sólo 5 l de ADN M2. Si la mutación es heterocigota, habrá una banda de cleaved adicional en ambas reacciones. Por otro lado, bandas de cleaved adicionales se encontrarán sólo en la primera reacción si el mutante es homocigoto.
- Para identificar la naturaleza de la mutación, secuencia los productos de PCR de los mutantes confirmados usando una plataforma de secuenciación de Sanger siguiendo las instrucciones del fabricante.
4. Cálculo de la frecuencia de mutación
NOTA: La frecuencia de mutación de una población TILLING se refiere a la distancia física media en la que se produce una mutación en los individuos de esa población. Por ejemplo, una frecuencia de mutación de 1/35 kb en una población TILLING significa que un individuo promedio de esa población posee 1 mutación por cada 35 kb en el genoma.
- Para determinar la frecuencia de mutación de una población TILLING, calcule el número total de bases examinadas.
- Para calcular el número total de bases examinadas, multiplique el tamaño del producto PCR por el número total de personas examinadas.
- Divida el número total de bases examinadas por el número de mutaciones únicas observadas utilizando la siguiente ecuación, que producirá la región física que posee 1 mutación en la población TILLING dada:
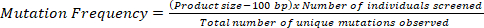
NOTA: Para tener en cuenta la limitación en la resolución de 50 bp en ambos extremos basado en una plataforma basada en gel de agarosa, reste 100 bp del tamaño del producto en el cálculo.
Resultados
La Figura 2 muestra la curva de dosificación del cultivar de trigo de pan hexaploide Jagger, trigo diploide Triticum monococcum6, y un donante genoma de trigo Aegilops tauschii7. Las dosis de EMS para las tasas deseadas de supervivencia del 50% fueron de aproximadamente 0,25%, 0,6% y 0,7% para T. monococcum, Ae. tauschiiy T. aestivum, respectivamente. La mayor tolera...
Discusión
TILLING es una herramienta genética inversa muy valiosa para la validación de genes, especialmente para granos pequeños donde los enfoques basados en la transformación tienen serios cuellos de botella11. El desarrollo de una población mutageneizada con una alta frecuencia de mutación es uno de los pasos críticos en la realización de estudios de genómica funcional. El paso más importante en el desarrollo de una población robusta de TILLING es determinar la concentración óptima de EMS. ...
Divulgaciones
Los autores no declaran intereses financieros en competencia.
Agradecimientos
Este trabajo fue apoyado por el Instituto Nacional de Alimentación y Agricultura del USDA, el proyecto Hatch 1016879 y la Estación Experimental Agrícola de Maryland a través de la Subvención MAES No. 2956952.
Materiales
| Name | Company | Catalog Number | Comments |
| 96 well 1.1 ml microtubes in microracks | National Scientific | TN0946-08R | For collecting leaf tissues |
| Agarose I biotechnology grade | VWR | 0710-500G | |
| Biosprint 96 DNA Plant Kit | Qiagen | 941558 | Kit for DNA extraction |
| Cel-1 endonuclease | Extracted as described by Till et al 2006 | Single strand specific endonuclease | |
| Centrifuge 5430 R | Eppendorf | ||
| Ethyl methanesulfonate | Sigma Aldrich | M-0880-25G | EMS, Chemical mutagen |
| Freeze Dry/Shell freeze system | Labconco | For lyophilization of leaf tissue | |
| Kingfisher Flex purification system | Thermo fisher scientific | 5400610 | High throughput DNA extraction robot |
| My Taq DNA Polymerase | Bioline | BIO-21107 | |
| Nuclease free water | Sigma aldrich | W4502-1L | |
| NuGenius gel imaging system | Syngene | ||
| Orbit Environ-shaker | Lab-line | ||
| SPECTROstar Nano | BMG LABTECH | Nano drop for DNA quantification | |
| T100 Thermal cycler | BIO-RAD | 1861096 |
Referencias
- McCallum, C. M., Comai, L., Greene, E. A., Henikoff, S. Targeted screening for induced mutations. Nature Biotechnology. 18 (4), 455-457 (2000).
- Bentley, A., MacLennan, B., Calvo, J., Dearolf, C. R. Targeted Recovery of Mutations in Drosophila. Genetics. 156 (3), 1169-1173 (2000).
- Tsai, H., et al. Discovery of Rare Mutations in Populations: TILLING by Sequencing. Plant Physiology. 156 (3), 1257-1268 (2011).
- Caldwell, D. G., et al. A structured mutant population for forward and reverse genetics in Barley (Hordeum vulgare L.). The Plant Journal. 40 (1), 143-150 (2004).
- Hazard, B., et al. Induced Mutations in the Starch Branching Enzyme II ( SBEII ) Genes Increase Amylose and Resistant Starch Content in Durum Wheat. Crop Science. 52 (4), 1754-1766 (2012).
- Rawat, N., et al. A diploid wheat TILLING resource for wheat functional genomics. BMC Plant Biology. 12, 205 (2012).
- Rawat, N., et al. TILL-D: An Aegilops tauschii TILLING Resource for Wheat Improvement. Frontiers in Plant Science. 9, (2018).
- Rawat, N., et al. Wheat Fhb1 encodes a chimeric lectin with agglutinin domains and a pore-forming toxin-like domain conferring resistance to Fusarium head blight. Nature Genetics. 48 (12), 1576-1580 (2016).
- Kippes, N., Chen, A., Zhang, X., Lukaszewski, A. J., Dubcovsky, J. Development and characterization of a spring hexaploid wheat line with no functional VRN2 genes. Theoretical and Applied Genetics. 129 (7), 1417-1428 (2016).
- Greene, E. A., et al. Spectrum of Chemically Induced Mutations From a Large-Scale Reverse-Genetic Screen in Arabidopsis. Genetics. 164 (2), 731-740 (2003).
- Harwood, W. A. Advances and remaining challenges in the transformation of barley and wheat. Journal of Experimental Botany. 63 (5), 1791-1798 (2012).
- Henikoff, S., Comai, L. Single-Nucleotide Mutations for Plant Functional Genomics. Annual Review of Plant Biology. 54 (1), 375-401 (2003).
- Uauy, C., et al. A modified TILLING approach to detect induced mutations in tetraploid and hexaploid wheat. BMC Plant Biology. 9 (1), 115 (2009).
- Uauy, C., Wulff, B. B. H., Dubcovsky, J. Combining Traditional Mutagenesis with New High-Throughput Sequencing and Genome Editing to Reveal Hidden Variation in Polyploid Wheat. Annual Review of Genetics. 51 (1), 435-454 (2017).
- Li, G., et al. The Sequences of 1504 Mutants in the Model Rice Variety Kitaake Facilitate Rapid Functional Genomic Studies. The Plant Cell. 29 (6), 1218-1231 (2017).
- Jiao, Y., et al. A Sorghum Mutant Resource as an Efficient Platform for Gene Discovery in Grasses. The Plant Cell. 28 (7), 1551-1562 (2016).
- Krasileva, K. V., et al. Uncovering hidden variation in polyploid wheat. Proceedings of the National Academy of Sciences. , 201619268 (2017).
- Dong, C., Dalton-Morgan, J., Vincent, K., Sharp, P. A Modified TILLING Method for Wheat Breeding. The Plant Genome. 2 (1), 39-47 (2009).
- Till, B. J., Zerr, T., Comai, L., Henikoff, S. A protocol for TILLING and Ecotilling in plants and animals. Nature Protocols. 1 (5), 2465-2477 (2006).
- Wu, J. -. L., et al. Chemical- and Irradiation-induced Mutants of Indica Rice IR64 for Forward and Reverse Genetics. Plant Molecular Biology. 59 (1), 85-97 (2005).
- Feldman, M., Levy, A. A. Genome Evolution Due to Allopolyploidization in Wheat. Genetics. 192 (3), 763-774 (2012).
- Comai, L. The advantages and disadvantages of being polyploid. Nature Reviews Genetics. 6 (11), 836-846 (2005).
- Guo, H., et al. Development of a High-Efficient Mutation Resource with Phenotypic Variation in Hexaploid Winter Wheat and Identification of Novel Alleles in the TaAGP.L-B1 Gene. Frontiers in Plant Science. 8, (2017).
- Rakszegi, M., et al. Diversity of agronomic and morphological traits in a mutant population of bread wheat studied in the Healthgrain program. Euphytica. 174 (3), 409-421 (2010).
- Tsai, H., Ngo, K., Lieberman, M., Missirian, V., Comai, L. Tilling by Sequencing. Plant Functional Genomics: Methods and Protocols. , 359-380 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados