Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Développement de lésions locales induites par le ciblage dans les génomes (TILLING) Populations dans les cultures de petits grains par l'éthylique Methanesulfonate Mutagenesis
Dans cet article
Résumé
Il est décrit comme un protocole pour le développement d'une population de lésions locales induites par le ciblage dans les génomes (TILLING) dans les petites cultures céréalières avec l'utilisation de l'éthyl methanesulfonate (EMS) comme mutagène. Un protocole de détection des mutations utilise l'analyse Cel-1.
Résumé
Cibler les lésions locales induites dans les génomes (TILLING) est un puissant outil de génétique inverse qui comprend la mutagénèse chimique et la détection de la variation de séquence dans les gènes cibles. TILLING est un outil de génomique fonctionnel très précieux pour la validation des gènes, en particulier dans les petits grains où les approches basées sur la transformation ont de sérieuses limites. Le développement d'une population mutagénaire robuste est essentiel pour déterminer l'efficacité d'une étude de validation génétique basée sur TILLING. Une population de TILLING avec une faible fréquence globale de mutation indique qu'une population peu grande doit être examinée pour trouver les mutations désirées, tandis qu'une concentration de mutagène élevée conduit à une mortalité élevée dans la population, conduisant à une insuffisance nombre d'individus mutagérisés. Une fois qu'une population efficace est développée, il existe de multiples façons de détecter les mutations dans un gène d'intérêt, et le choix de la plate-forme dépend de l'échelle expérimentale et la disponibilité des ressources. L'approche à base de gel Cel-1 et de gel d'agarose pour l'identification des mutants est pratique, reproductible et une plate-forme moins gourmande en ressources. Il est avantageux en ce qu'il est simple, ne nécessitant aucune connaissance de calcul, et il est particulièrement approprié pour la validation d'un petit nombre de gènes avec l'équipement de laboratoire de base. Dans le présent article, décrits sont les méthodes pour le développement d'une bonne population DE TILLING, y compris la préparation de la courbe de dosage, la mutagénèse et le maintien de la population mutante, et le dépistage de la population mutante en utilisant le PCR-basé Cel-1 test .
Introduction
Les mutations ponctuelles dans les génomes peuvent servir à de nombreux fins utiles pour les chercheurs. Selon leur nature et leur emplacement, ces mutations peuvent être utilisées pour attribuer des fonctions à des gènes ou même des domaines distincts de protéines d'intérêt. D'autre part, comme source de nouvelles variations génétiques, des mutations utiles peuvent être sélectionnées pour les caractères souhaités à l'aide d'écrans de phénotypage et davantage utilisées dans l'amélioration des cultures. TILLING est un puissant outil de génétique inverse qui comprend la mutagénèse chimique et la détection de la variation de séquence dans le gène cible. D'abord développé dans Arabidopsis1 et Drosophilia melanogaster2, les populations DE TILLING ont été développées et utilisées dans de nombreuses cultures de petits grains telles que le blé à pain hexaploïde (Triticum aestivum)3, orge (Hordeum vulgare)4, blé dur tétraploïde (T. dicoccoides durum)5, blé diploïde (T. monococcum)6 et l'ancêtre du génome "D" du blé Aegilops tauschii7 . Ces ressources ont été utilisées pour valider les rôles des gènes dans la régulation de la tolérance au stress abiotique et biotique8, la régulation du temps de floraison9, et le développement de variétés de cultures nutritionnellement supérieure5.
TILLING, avec l'utilisation d'agents mutagènes alkylants tels que le méthane sulfonate d'éthyle (EMS), l'azide de sodium, N-méthyl-N-nitrosourea (MNU), et le méthanesulfonate de méthyle (MMS), a des avantages sur d'autres outils de génétique inverse pour plusieurs raisons. Tout d'abord, la mutagénèse peut être menée sur pratiquement n'importe quelle espèce ou variété de plante10 et est indépendante du goulot d'étranglement de transformation, qui est particulièrement difficile dans le cas des petits grains11. Deuxièmement, en plus de générer des mutations knock-out qui peuvent être obtenues par d'autres approches de validation des gènes, une gamme de mutations de mauvais sens et d'épissage peut être induite, ce qui peut discerner les fonctions des domaines individuels des protéines d'intérêt12. En outre, TILLING génère une collection immortelle de mutations dans tout le génome; ainsi, une seule population peut être utilisée pour la validation fonctionnelle de plusieurs gènes. En revanche, d'autres outils de génétique inversée génèrent des ressources spécifiques uniquement au gène à l'étude13. Les mutations utiles identifiées par TILLING peuvent être déployées à des fins de reproduction et ne sont pas soumises à la réglementation, contrairement à l'édition génétique, dont la classification non transgénique est encore incertaine dans de nombreux pays. Cela devient particulièrement pertinent pour les petits grains qui sont négociés à l'échelle internationale14.
TILLING est une stratégie de validation génétique simple et efficace qui nécessite le développement de populations mutagénaires pour étudier les gènes d'intérêt. Le développement d'une population mutagénaire efficace est essentiel pour déterminer l'efficacité d'une étude de validation génétique basée sur TILLING. Une population de TILLING avec une faible fréquence globale de mutation indique qu'une population peu large doit être examinée pour les mutations désirées, tandis qu'une concentration de mutagène élevée conduit à une mortalité élevée dans la population et un nombre insuffisant de mutagénaires. Une fois qu'une bonne population est développée, il existe de multiples façons de détecter les mutations dans les gènes d'intérêt, et le choix de la plate-forme dépend de l'échelle expérimentale et la disponibilité des ressources. Le séquençage du génome entier et le séquençage de l'exome ont été utilisés pour caractériser toutes les mutations dans les populations de TILLING chez les plantes ayant de petits génomes15,16. Le séquençage de l'exome de deux populations de TILLING a été effectué dans le pain et le blé dur et est disponible au public pour identifier les mutations souhaitables et commander des lignes mutantes d'intérêt17. Il s'agit d'une grande ressource publique en termes de disponibilité de mutations souhaitables; cependant, dans les études de validation de gène, la ligne sauvage-type devrait posséder le gène d'intérêt de candidat. Malheureusement, il est encore prohibitif de séquencer l'exome de toute la population de TILLING pour la validation inversée basée sur la génétique de quelques gènes candidats dans un autre contexte. Le séquençage d'amplicon et les essais à base de Cel-1 ont été utilisés pour détecter les mutations dans les populations ciblées dans le blé, et les essais cel-1 sont plus simples, ne nécessitant aucune connaissance computationnelle, et sont particulièrement appropriés pour la validation d'un petit nombre de gènes avec des matériel de laboratoire6,18.
Dans le présent article, décrits sont des méthodes pour le développement d'une bonne population DE TILLING, y compris la préparation de la courbe de dosage, la mutagénèse et le maintien de la population mutante, et le dépistage de la population mutante en utilisant le PCR-basé Cel-1 test . Ce protocole a déjà été mis en œuvre avec succès dans le développement et l'utilisation des populations mutagénaires de Triticum aestivum, Triticum monoccocum6, orge, Aegilops tauchii7, et plusieurs autres. On y trouve des détails explicites de ces méthodes ainsi que des conseils utiles qui aideront les chercheurs à développer des populations de TILLING, en utilisant le SME comme mutagène dans n'importe quelle petite usine céréalière de choix.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Préparation de la courbe de dosage pour la mutagénèse efficace
- Faire tremper 100 graines avec le génotype d'intérêt dans six flacons de verre de 250 ml (100 dans chaque flacon) contenant 50 ml d'eau distillée. Agiter à 100 tr/min pendant 8 h à température ambiante (RT) pour l'imbibition par les graines.
- Dans une hotte de fumée, préparez 50 mL de 0,4 %, 0,6 %, 0,8 %, 1,0 % et 1,2 % (w/v) solution de méthane d'éthyle (EMS) en dissolvant 0,167, 0,249, 0,331, 0,415 et 0,498 mL de SME dans de l'eau distillée, respectivement.
REMARQUE : Le SME est liquide à RT avec une densité de 1,206 g/mL.
CAUTION : Utilisez l'équipement de protection individuelle (EPI) approprié tout en manipulant le SME. - Décant l'eau de cinq flacons et ajouter 50 ml de solution EMS dans chaque flacon contenant des graines imbibées de sorte qu'il y ait six traitements différents avec 0,0 %, 0,4 %, 0,6 %, 0,8 %, 1,0 % et 1,2 % de solution EMS. Agiter les flacons pendant 16 h à 75 tr/min et RT.
- Décant la solution EMS et collectez les graines traitées séparément pour chaque traitement à l'aide d'un chiffon à fromage. Inactiver la solution EMS utilisée en ajoutant un volume de solution inactivante EMS (0,1 M NaOH, 20% w/v Na2S2O3) pour 24 h. Traiter également les flacons contaminés et les pointes de pipette avec la solution inactivante EMS pendant 24 h.
- Laver les graines traitées au SME sous l'eau courante du robinet pendant 2 h. Transplanter chaque graine individuellement dans des dresseurs de racines contenant du sol de mise en pot.
- Cultivez des plantes à 20 à 25 oC sous une période de lumière de 16 h.
- Enregistrez des données sur la survie des plantes après 15 jours de transplantation. Utilisez l'équation suivante pour calculer le taux de survie pour chaque traitement :
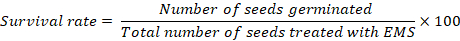
REMARQUE : Si le taux de germination est inférieur à 100 % dans les contrôles, des taux de survie précis dans tous les traitements devraient être calculés après avoir soustrait le nombre de graines qui n'ont pas germé dans les contrôles. Un taux de survie de 40% à 60% est souhaitable pour une mutagénèse efficace. Il peut être nécessaire d'effectuer une deuxième série d'optimisation de dosage avec une concentration modifiée en fonction de la survie des graines traitées jusqu'à atteindre le taux de létalité souhaitable de 40%-60%.
2. Mutagénèse et maintien de la population mutante
- Faire tremper le lot final d'au moins 3 000 graines en répartissant également (600 graines chacune) dans cinq flacons de 1 000 ml contenant 300 ml d'eau distillée. Secouez pendant 8 h à 100 tr/min sous RT pour l'imbibition.
REMARQUE : La taille finale de la population désirable dépendra de la fréquence de mutation et du niveau de ploidy du génotype, mais il est conseillé d'utiliser au moins 3 000 graines pour les héxaploïdes, 4 000 graines pour les tétraploïdes et plus de 7 000 graines pour les diploïdes. - Dans une hotte de fumée, préparer 1 500 ml de la solution optimisée de concentration EMS dans de l'eau distillée.
- Décant l'eau des flacons et ajoutez 300 ml de la concentration optimale de la solution EMS dans chaque flacon contenant 600 graines imbibées. Agiter les flacons pendant 16 h à 75 tr/min et RT.
- Décant le SME et collectez les graines traitées dans du tissu à fromage. Inactivez la solution EMS et les conteneurs de traitement avec la solution inactivante EMS comme cela a été fait à l'étape 1.4.
- Laver les graines traitées au SME sous l'eau courante du robinet pendant 2 h. Transplanter chaque graine M1 traitée par le SME individuellement dans des dresseurs de racines.
- Cultivez des plantes M1 (dérivées de graines M0) à 20'25 oC sous une période de lumière de 16 h.
REMARQUE : Il peut être nécessaire de vernaliser les semis à l'étape des deux feuilles pendant 6 semaines à 4 oC, si le génotype d'intérêt a une habitude de croissance de type hivernal. - Laisser les plantes M1 s'autopolliniser et récolter les graines M2 séparément pour chaque plante fertile M1.
REMARQUE : Pour éviter les risques de croisement séviscissant, recouvrez les pointes des plantes M1 de sacs de pollinisation avant l'anthèse. - Plantez une seule graine M2 de chaque plante M1 pour éviter la redondance génétique.
- Recueillir les tissus des plantes M2 à l'étape à deux feuilles dans 1,1 ml de 96 microtubes de puits. Recueillir environ 80 mm de tissu foliaire de chaque plante et enregistrer l'iD de chaque échantillon dans un plan de collecte de tissus.
- Faire sécher le tissu foliaire à l'aide d'un lyophilisateur et le conserver à -80 oC.
- Maintenir les plants de M2 à 20 x 25 oC sous une période de lumière de 16 h.
- Enregistrez des données sur les phénotypes mutants des plantes M2 à intervalles réguliers. Les phénotypes attendus sont les albinos, la chlorine, la pousse herbeuse, variée, partiellement fertile, stérile, etc.
- Laisser les plantes M2 s'autofertiliser et mûrir. Récoltez et sauvez séparément les graines M3 des plantes M2 (Figure 1).
REMARQUE : Les graines de M sont employées pour valider le phénotype dans des études de génétique inverse. Par conséquent, M doit être soigneusement catalogué et stocké dans des conditions fraîches et sèches. Alternativement, si la graine doit être augmentée dans le champ, des rangées de têtes peuvent être plantées pour chaque plante De, et les graines m de chaque ligne peuvent être récoltées et sauvées séparément en tant que ressource de population de TILLING.
3. Cel-1 test pour la caractérisation génétique des mutants
- Extraire l'ADN du tissu foliaire de M2 à l'aide d'un kit d'extraction d'ADN végétal avec un système de purification de l'ADN (voir Tableau des matériaux) suivant les recommandations du fabricant.
- Quantifier l'ADN à l'aide d'un spectrophotomètre et normaliser les concentrations d'ADN à 25 ng/L avec de l'eau sans nauséail dans 96 blocs de puits.
REMARQUE : Il est important de vérifier la qualité de l'ADN en l'exécutant sur un gel, car l'ADN de mauvaise qualité (ADN enduit) peut entraîner de faux négatifs dans des échantillons mis en commun. - Créez des pools d'ADN 4x en combinant l'ADN de quatre blocs de puits 96 dans une seule plaque, tout en conservant l'identité de la ligne et de la colonne de chaque échantillon. Ajoutez 50 L d'ADN de chaque échantillon individuel dans la plaque de billard afin que chaque plaque de billard contienne un total de 200 L d'ADN provenant de quatre blocs de puits différents de 96.
- Cataloguez l'identité de l'ADN mis en commun dans le format de Pool Plate-Row-Column (p. ex., Piscine 1 A1 - Box1A1 - Box2A1 - Box3A1 et Box4A1).
- Concevoir des amorces spécifiques au génome [A1] pour le gène d'intérêt à l'aide de la page Genome Specific Primers (GSP) (lt;https://probes.pw.usda.gov/GSP-gt; avec des paramètres par défaut pour les espèces polyploïdes. Pour les diploïdes, utilisez Primer 3 'lt;http://primer3.ut.ee/'gt; pour la conception d'amorces en utilisant les paramètres par défaut. Concevoir plusieurs amorces, si nécessaire, pour couvrir toute la région de codage du gène d'intérêt.
REMARQUE: Le dernier assemblage IWGSC peut être utilisé pour obtenir des séquences pour le gène d'intérêt pour le blé à l'aide de l'outil URGI BLAST 'lt;https://wheat-urgi.versailles.inra.fr/Seq-Repository/BLAST.gt;. La longueur optimale de l'amplicon est de l'ordre de 800 à 1 500 pb. Le tableau 1 montre des exemples d'amorces pour les gènes cireuses dans le blé hexaploïde. - Exécutez un PCR pour les amorces spécifiques aux gènes sur l'ADN mis en commun comme suit :
- Ajouter 5 'L de tampon PCR, 2 'L (chacun) de 4 amorces avant et inversées, 0,1 'L de polymérase d'ADN (voir tableau des matériaux),5 'L de modèle d'ADN mis en commun, puis augmenter le volume à 25 'L en utilisant de l'eau sans nucléane.
- Utilisez un profil de toucher vers le bas pour exécuter la réaction PCR sur un cycleur thermique comme suit : 95 oC pour 1 min, sept cycles de 95 oC pour 1 min, 67 oC à 60 oC pour 1 min avec des baisses de température de 1 oC par cycle et 72 oC pour 2 min , suivi de 30 cycles de 95 oC pour 1 min, de 60 oC pour 1 min, de 72 oC pour 2 min, et d'une extension finale à 72 oC pour 7 min.
REMARQUE : Le profil CI-dessus PCR fonctionne sur le modèle d'ADN de blé pour la plupart des amorces conçues à l'aide des paramètres par défaut de l'amorce 3. En cas d'amplification non spécifique, le profil PCR doit être strict avant de passer aux étapes suivantes.
- Générer des hétéroduplexes entre l'ADN dépareillé en incubant des produits PCR dans le cycle thermique en utilisant le profil suivant : 95 oC pendant 2 min, cinq cycles de 95 oC pour 1 s, 95 oC à 85 oC pour 1 min avec des baisses de température de 2 oC par cycle, et 60 cycles de 85 à 25 oC avec diminutions de 1 oC par cycle.
- Ajoutez 2,5 l d'endonucalage Cel-1 maison aux produits PCR hétéroduplexés et incubez 45 min à 45 oC. Terminez la réaction Cel-1 en ajoutant 2,5 L de 0,5 M EDTA (pH 8,0).
REMARQUE : L'endouclence Cel-1 peut être extraite des tiges fraîches de céleri utilisant le protocole exécuté par Till et autres19 Il est très important de tester l'activité et la quantité optimale de Cel-1 endonuclease, qui peut être testée utilisant des mutants précédemment caractérisés ou un kit de détection de mutation disponible dans le commerce. - Exécuter les produits traités Cel-1 sur un gel d'agarose de 3,0% à 100 V pendant 2,5 h. Les puits contenant des bandes clivées plus petites et uniques, en plus des bandes non clivées pleine longueur, contiendront l'échantillon d'ADN mutant.
- Piscines mutantes denconvolute.
- Suivez les étapes 3.6, 3.7, 3.8 et 3.9 pour les échantillons d'ADN M2 individuels constituant les pools mutants identifiés à l'étape 3.9.
- Pour déterminer la zygosité des mutants, exécutez deux PCR (comme décrit à l'étape 3.6) pour l'ADN M2 individuel, dans lequel la première réaction contient 2,5 L d'ADN M2 et 2,5 L d'ADN de type sauvage et la deuxième réaction ne contient que 5 L d'ADN M2. Si la mutation est hétérozygote, une bande clivée supplémentaire sera présente dans les deux réactions. D'autre part, d'autres bandes clivées ne seront trouvées que dans la première réaction si le mutant est homozygote.
- Pour identifier la nature de la mutation, séquencez les produits PCR des mutants confirmés à l'aide d'une plate-forme de séquençage Sanger suivant les instructions du fabricant.
4. Calcul de la fréquence des mutations
REMARQUE : La fréquence de mutation d'une population de TILLING se rapporte à la distance physique moyenne dans laquelle une mutation se produit dans les individus de cette population. Par exemple, une fréquence de mutation de 1/35 kb dans une population de TILLING signifie qu'un individu moyen de cette population possède 1 mutation par 35 kb dans le génome.
- Pour déterminer la fréquence de mutation d'une population de TILLING, calculez le nombre total de bases examinées.
- Pour calculer le nombre total de bases examinées, multipliez la taille du produit PCR par le nombre total d'individus examinés.
- Divisez le nombre total de bases examinées par le nombre de mutations uniques observées à l'aide de l'équation suivante, ce qui donnera à la région physique possédant 1 mutation dans la population donnée de TILLING :
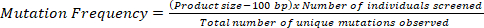
REMARQUE : Pour tenir compte de la limitation de la résolution de 50 pb aux deux extrémités basée sur une plate-forme à base de gel d'agarose, soustrayez 100 pb de la taille du produit dans le calcul.
Access restricted. Please log in or start a trial to view this content.
Résultats
La figure 2 montre la courbe de dosage du cultivar de blé de pain hexaploïde Jagger, du blé diploïde Triticum monococcum6, et d'un donneur de génome de blé Aegilops tauschii7. Les doses de SME pour les taux de survie souhaités de 50% étaient d'environ 0,25%, 0,6% et 0,7% pour T. monococcum, Ae. tauschii, et T. aestivum, respectivement. La tolérance plus élev...
Access restricted. Please log in or start a trial to view this content.
Discussion
TILLING est un outil de génétique inversée très précieux pour la validation des gènes, en particulier pour les petits grains où les approches basées sur la transformation ont de sérieux goulots d'étranglement11. Le développement d'une population mutagénaire avec une fréquence de mutation élevée est l'une des étapes critiques dans la conduite des études de génomique fonctionnelle. L'étape la plus importante dans le développement d'une population robuste de TILLING est de déterm...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt financier concurrent.
Remerciements
Ce travail a été soutenu par l'USDA National Institute of Food and Agriculture, le projet Hatch 1016879 et le Maryland Agricultural Experiment Station par l'intermédiaire de la subvention MAES no 2956952.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 96 well 1.1 ml microtubes in microracks | National Scientific | TN0946-08R | For collecting leaf tissues |
| Agarose I biotechnology grade | VWR | 0710-500G | |
| Biosprint 96 DNA Plant Kit | Qiagen | 941558 | Kit for DNA extraction |
| Cel-1 endonuclease | Extracted as described by Till et al 2006 | Single strand specific endonuclease | |
| Centrifuge 5430 R | Eppendorf | ||
| Ethyl methanesulfonate | Sigma Aldrich | M-0880-25G | EMS, Chemical mutagen |
| Freeze Dry/Shell freeze system | Labconco | For lyophilization of leaf tissue | |
| Kingfisher Flex purification system | Thermo fisher scientific | 5400610 | High throughput DNA extraction robot |
| My Taq DNA Polymerase | Bioline | BIO-21107 | |
| Nuclease free water | Sigma aldrich | W4502-1L | |
| NuGenius gel imaging system | Syngene | ||
| Orbit Environ-shaker | Lab-line | ||
| SPECTROstar Nano | BMG LABTECH | Nano drop for DNA quantification | |
| T100 Thermal cycler | BIO-RAD | 1861096 |
Références
- McCallum, C. M., Comai, L., Greene, E. A., Henikoff, S. Targeted screening for induced mutations. Nature Biotechnology. 18 (4), 455-457 (2000).
- Bentley, A., MacLennan, B., Calvo, J., Dearolf, C. R. Targeted Recovery of Mutations in Drosophila. Genetics. 156 (3), 1169-1173 (2000).
- Tsai, H., et al. Discovery of Rare Mutations in Populations: TILLING by Sequencing. Plant Physiology. 156 (3), 1257-1268 (2011).
- Caldwell, D. G., et al. A structured mutant population for forward and reverse genetics in Barley (Hordeum vulgare L.). The Plant Journal. 40 (1), 143-150 (2004).
- Hazard, B., et al. Induced Mutations in the Starch Branching Enzyme II ( SBEII ) Genes Increase Amylose and Resistant Starch Content in Durum Wheat. Crop Science. 52 (4), 1754-1766 (2012).
- Rawat, N., et al. A diploid wheat TILLING resource for wheat functional genomics. BMC Plant Biology. 12, 205(2012).
- Rawat, N., et al. TILL-D: An Aegilops tauschii TILLING Resource for Wheat Improvement. Frontiers in Plant Science. 9, (2018).
- Rawat, N., et al. Wheat Fhb1 encodes a chimeric lectin with agglutinin domains and a pore-forming toxin-like domain conferring resistance to Fusarium head blight. Nature Genetics. 48 (12), 1576-1580 (2016).
- Kippes, N., Chen, A., Zhang, X., Lukaszewski, A. J., Dubcovsky, J. Development and characterization of a spring hexaploid wheat line with no functional VRN2 genes. Theoretical and Applied Genetics. 129 (7), 1417-1428 (2016).
- Greene, E. A., et al. Spectrum of Chemically Induced Mutations From a Large-Scale Reverse-Genetic Screen in Arabidopsis. Genetics. 164 (2), 731-740 (2003).
- Harwood, W. A. Advances and remaining challenges in the transformation of barley and wheat. Journal of Experimental Botany. 63 (5), 1791-1798 (2012).
- Henikoff, S., Comai, L. Single-Nucleotide Mutations for Plant Functional Genomics. Annual Review of Plant Biology. 54 (1), 375-401 (2003).
- Uauy, C., et al. A modified TILLING approach to detect induced mutations in tetraploid and hexaploid wheat. BMC Plant Biology. 9 (1), 115(2009).
- Uauy, C., Wulff, B. B. H., Dubcovsky, J. Combining Traditional Mutagenesis with New High-Throughput Sequencing and Genome Editing to Reveal Hidden Variation in Polyploid Wheat. Annual Review of Genetics. 51 (1), 435-454 (2017).
- Li, G., et al. The Sequences of 1504 Mutants in the Model Rice Variety Kitaake Facilitate Rapid Functional Genomic Studies. The Plant Cell. 29 (6), 1218-1231 (2017).
- Jiao, Y., et al. A Sorghum Mutant Resource as an Efficient Platform for Gene Discovery in Grasses. The Plant Cell. 28 (7), 1551-1562 (2016).
- Krasileva, K. V., et al. Uncovering hidden variation in polyploid wheat. Proceedings of the National Academy of Sciences. , 201619268(2017).
- Dong, C., Dalton-Morgan, J., Vincent, K., Sharp, P. A Modified TILLING Method for Wheat Breeding. The Plant Genome. 2 (1), 39-47 (2009).
- Till, B. J., Zerr, T., Comai, L., Henikoff, S. A protocol for TILLING and Ecotilling in plants and animals. Nature Protocols. 1 (5), 2465-2477 (2006).
- Wu, J. -L., et al. Chemical- and Irradiation-induced Mutants of Indica Rice IR64 for Forward and Reverse Genetics. Plant Molecular Biology. 59 (1), 85-97 (2005).
- Feldman, M., Levy, A. A. Genome Evolution Due to Allopolyploidization in Wheat. Genetics. 192 (3), 763-774 (2012).
- Comai, L. The advantages and disadvantages of being polyploid. Nature Reviews Genetics. 6 (11), 836-846 (2005).
- Guo, H., et al. Development of a High-Efficient Mutation Resource with Phenotypic Variation in Hexaploid Winter Wheat and Identification of Novel Alleles in the TaAGP.L-B1 Gene. Frontiers in Plant Science. 8, (2017).
- Rakszegi, M., et al. Diversity of agronomic and morphological traits in a mutant population of bread wheat studied in the Healthgrain program. Euphytica. 174 (3), 409-421 (2010).
- Tsai, H., Ngo, K., Lieberman, M., Missirian, V., Comai, L. Tilling by Sequencing. Plant Functional Genomics: Methods and Protocols. , 359-380 (2015).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon