È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Sviluppo di popolazioni indotte di lesioni locali indotte dal targeting NEI genomi (TILLING) in colture di grano piccolo mediante mutagenesi metannata etilareale
In questo articolo
Riepilogo
Descritto è un protocollo per lo sviluppo di una popolazione di lesioni locali indotte di targeting IN genomi (TILLING) in piccole colture di grano con l'uso di metanosofato etilico (EMS) come mutageno. È inoltre previsto un protocollo per il rilevamento delle mutazioni utilizzando il saggio Cel-1.
Abstract
Targeting Induced Local Lesions IN Genomes (TILLING) è un potente strumento di genetica inversa che include la mutagenesi chimica e il rilevamento della variazione della sequenza nei geni bersaglio. TILLING è uno strumento di genomica funzionale di grande valore per la convalida genica, specialmente in piccoli grani in cui gli approcci basati sulla trasformazione hanno gravi limitazioni. Lo sviluppo di una solida popolazione mutagenizzata è fondamentale per determinare l'efficienza di uno studio sulla convalida genica basata su TILLING. Una popolazione TILLING con una bassa frequenza di mutazione complessiva indica che una popolazione impraticabile di grandi dimensioni deve essere sottoposta a screening per trovare le mutazioni desiderate, mentre un'elevata concentrazione di mutageni porta ad un'elevata mortalità nella popolazione, che porta a un'insufficiente numero di individui mutagenizzati. Una volta sviluppata una popolazione efficace, ci sono diversi modi per rilevare le mutazioni in un gene di interesse, e la scelta della piattaforma dipende dalla scala sperimentale e dalla disponibilità delle risorse. L'approccio basato sul gel Cel-1 e agarose per l'identificazione dei mutanti è conveniente, riproducibile e una piattaforma meno ad alta intensità di risorse. È vantaggioso in quanto è semplice, non richiede alcuna conoscenza computazionale, ed è particolarmente adatto per la convalida di un piccolo numero di geni con attrezzature di laboratorio di base. Nel presente articolo, descritti sono i metodi per lo sviluppo di una buona popolazione TILLING, compresa la preparazione della curva di dosaggio, la mutagenesi e il mantenimento della popolazione mutante, e lo screening della popolazione mutante utilizzando il saggio Cel-1 basato su PCR .
Introduzione
Le mutazioni puntiformi nei genomi possono servire a molti scopi utili per i ricercatori. A seconda della loro natura e della loro posizione, queste mutazioni possono essere utilizzate per assegnare funzioni ai geni o anche a domini distinti di proteine di interesse. D'altra parte, come fonte di una nuova variazione genetica, possono essere selezionate mutazioni utili per i tratti desiderati utilizzando schermi fenotipizzazione e ulteriormente utilizzati nel miglioramento delle colture. TILLING è un potente strumento di genetica inversa che include la mutagenesi chimica e il rilevamento della variazione di sequenza nel gene bersaglio. Sviluppati per la prima volta in Arabidopsis1 e Drosophilia melanogaster2, le popolazioni TILLING sono state sviluppate e utilizzate in molte piccole colture di grano come il grano exaploide (Triticum aestivum)3, orzo (Ordeum vulgare)4, grano tetraploiddur (T. dicoccoides durum)5, grano diploide (T. monococcum)6 e il progenitore del genoma "D" del grano Aegilops tauschii7 . Queste risorse sono state utilizzate per convalidare i ruoli dei geninella regolazione della tolleranza allo stress abiotico e biotico 8, regolando il tempo di fioritura9e sviluppando varietà di colture nutrizionalmente superiori5.
TILLING, insieme all'uso di agenti mutagenici alchilanti come il metaenesonero etilico (EMS), l'azide di sodio, N-metile-N-nitrosourea (MNU) e il metetanonero (MMS) hanno vantaggi rispetto ad altri strumenti di genetica inversa per diversi motivi. In primo luogo, la mutagenesi può essere condotta praticamente su qualsiasi specie o varietà di piante10 ed è indipendente dal collo di bottiglia di trasformazione, che è particolarmente impegnativo nel caso di piccoli grani11. In secondo luogo, oltre a generare mutazioni da knockout che possono essere ottenute con altri approcci di convalida genica, si può indotta una serie di mutazioni errate e splicing, che possono discernere le funzioni dei singoli domini delle proteine di interesse12. Inoltre, TILLING genera una raccolta immortale di mutazioni in tutto il genoma; pertanto, una singola popolazione può essere utilizzata per la convalida funzionale di più geni. Al contrario, altri strumenti di genetica inversa generano risorse specifiche solo per il gene oggetto dello studio13. Le mutazioni utili identificate tramite TILLING possono essere impiegate per scopi riproduttivi e non sono soggette a regolazione, a differenza dell'editing genetico, la cui classificazione non transgenica è ancora incerta in molti paesi. Questo diventa particolarmente rilevante per i piccoli grani che sono scambiati a livello internazionale14.
TILLING è una strategia di convalida genica semplice ed efficiente e richiede lo sviluppo di popolazioni mutagenizzate per studiare i geni di interesse. Lo sviluppo di una popolazione mutagenizzata efficace è fondamentale per determinare l'efficienza di uno studio di convalida genica basato su TILLING. Una popolazione TILLING con una bassa frequenza di mutazione complessiva indica che una popolazione impraticabile di grandi dimensioni deve essere sottoposta a screening per le mutazioni desiderate, mentre un'elevata concentrazione di mutageni porta ad un'elevata mortalità nella popolazione e a un numero insufficiente di mutantizzati. Una volta sviluppata una buona popolazione, ci sono diversi modi per rilevare le mutazioni nei geni di interesse, e la scelta della piattaforma dipende dalla scala sperimentale e dalla disponibilità delle risorse. Il sequenziamento dell'intero genoma e il sequenziamento degli esomi sono stati utilizzati per caratterizzare tutte le mutazioni nelle popolazioni di TILLING in piante con piccoli genomi15,16. Il sequenziamento dell'esoma di due popolazioni di TILLING è stato eseguito nel pane e nel grano durum ed è a disposizione del pubblico per identificare mutazioni desiderabili e ordinare linee mutanti di interesse17. È una grande risorsa pubblica in termini di disponibilità di mutazioni desiderabili; tuttavia, negli studi di convalida genica, la linea wild-type dovrebbe possedere il gene candidato di interesse. Sfortunatamente, è ancora proibitivo sequenziare l'esoma dell'intera popolazione TILLING per la convalida inversa basata sulla genetica di alcuni geni candidati in un altro background. Il sequenziamento dell'amplicone e i saggi basati su Cel-1 sono stati utilizzati per rilevare le mutazioni nelle popolazioni mirate nel grano, e i test Cel-1 sono più semplici, non richiedono alcuna conoscenza computazionale e sono particolarmente adatti per la convalida di un piccolo numero di geni con attrezzature di laboratorio6,18.
Nel presente articolo, descritti sono metodi per lo sviluppo di una buona popolazione TILLING, compresa la preparazione della curva di dosaggio, la mutagenesi e il mantenimento della popolazione mutante, e lo screening della popolazione mutante utilizzando il saggio Cel-1 basato su PCR . Questo protocollo è già stato implementato con successo nello sviluppo e nell'utilizzo di popolazioni mutagenizzate di Triticum aestivum, Triticum monoccocum6, orzo, Aegilops tauchii7,e diversi Altri. Sono inclusi dettagli espliciti di questi metodi insieme a suggerimenti utili che aiuteranno i ricercatori a sviluppare popolazioni TILLING, utilizzando EMS come mutageno in qualsiasi piccola pianta di grano di scelta.
Protocollo
1. Preparazione della curva di dosaggio per una mutagenesi efficace
- Immergere 100 semi con il genotipo di interesse in sei flaconi di vetro da 250 mL (100 in ogni flacone) contenenti 50 mL di acqua distillata. Agitare a 100 giri/min per 8 ore a temperatura ambiente (RT) per l'imbibizione dai semi.
- In un cofano di fumi, preparare una soluzione di 50 mL dello 0,4%, 0,6%, 0,8%, 1,0% e 1,2% (w/v) di metanoesonato (EMS) sciogliendo rispettivamente 0,167, 0,249, 0,331, 0,415 e 0,498 mL di EMS in acqua distillata.
NOTA: EMS è liquido a RT con una densità di 1,206 g/mL.
AVVISO: utilizzare adeguate dispositivi di protezione personale (PPE) durante la manipolazione dello SME. - Decant l'acqua da cinque flaconi e aggiungere 50 mL di soluzione EMS in ogni fiaschetta contenente semi imbibed in modo che ci siano sei diversi trattamenti con 0.0%, 0.4%, 0.6%, 0.8%, 1.0%, e 1.2% soluzione EMS. Agitare i flaconi per 16 h a 75 giri/min e RT.
- Decant la soluzione EMS e raccogliere i semi trattati separatamente per ogni trattamento utilizzando panno di formaggio. Disattivare la soluzione EMS utilizzata aggiungendo un volume di soluzione di inattivazione EMS (0,1 M NaOH, 20% w/v Na2S2O3) per 24 h. Trattare anche i flaconi contaminati e le punte di pipetta con la soluzione di disattivazione EMS per 24 ore.
- Lavare i semi trattati con EMS sotto l'acqua corrente del rubinetto per 2 h. Trapiantare ogni seme singolarmente in forine radice contenenti terriccio.
- Coltivare piante a 20-25 gradi centigradi in un periodo di luce di 16 h.
- Registrare i dati sulla sopravvivenza delle piante dopo 15 giorni di trapianto. Utilizzare la seguente equazione per calcolare il tasso di sopravvivenza per ogni trattamento:
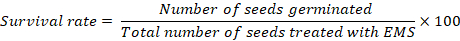
NOTA: Se il tasso di germinazione è inferiore al 100% nei controlli, i tassi di sopravvivenza precisi in tutti i trattamenti devono essere calcolati dopo aver sottratto il numero di semi che non sono riusciti a germinare nei controlli. Un tasso di sopravvivenza del 40%–60% è auspicabile per una mutagenesi efficace. Potrebbe essere richiesto di eseguire un secondo round di ottimizzazione del dosaggio con una concentrazione modificata in base alla sopravvivenza dei semi trattati fino a raggiungere il tasso di letalità desiderabile del 40%-60%.
2. Mutagenesi e mantenimento della popolazione mutante
- Immergere l'ultimo lotto di almeno 3.000 semi dividendosi equamente (600 semi ciascuno) in cinque flaconi da 1.000 mL contenenti 300 mL di acqua distillata. Agitare per 8 h a 100 rpm sotto RT per l'imbibizione.
NOTA: La dimensione finale della popolazione desiderabile dipenderà dalla frequenza di mutazione e dal livello ploidiy del genotipo, ma è consigliabile utilizzare almeno 3.000 semi per gli esaploidi, 4.000 semi per i tetraploidi e più di 7.000 semi per i diploidi. - In un cofano di fumi, preparare 1.500 mL della soluzione ottimizzata di concentrazione EMS in acqua distillata.
- Decantare l'acqua dai flaconi e aggiungere 300 ml della concentrazione ottimale della soluzione EMS in ogni fiaschetta contenente 600 semi imbevisti. Agitare i flaconi per 16 h a 75 giri/min e RT.
- Decant lo SME e raccogliere i semi trattati in panno di formaggio. Disattivare la soluzione EMS e i contenitori di trattamento con la soluzione di disattivazione EMS come completato nel passaggio 1.4.
- Lavare i semi trattati con EMS sotto l'acqua corrente del rubinetto per 2 h. Trapiantare ogni seme M1 trattato con EMS singolarmente in addestratori di radice.
- Coltivare le piante di M1 (derivate dai semi di M0) a 20-25 gradi centigradi in un periodo di luce di 16 h.
NOTA: Potrebbe essere necessario vernalizzare le piantine nella fase a due foglie per 6 settimane a 4 gradi centigradi, se il genotipo di interesse ha un'abitudine di crescita di tipo invernale. - Lasciare che le piante M1 si autompollinano e raccogliete separatamente i semi M2 per ogni pianta fertile di M1.
NOTA: Per evitare la possibilità di potenziali outcrossing, coprire le punte delle piante di M1 con sacchetti di impollinazione prima dell'anthesi. - Piantare un singolo seme M2 da ogni pianta di M1 per evitare la ridondanza genetica.
- Raccogliere il tessuto da piante M2 nella fase a due foglie in 1,1 mL di 96 ben travasato microtubi. Raccogliere circa 80 mm di tessuto fogliare da ogni pianta e registrare l'ID di ogni campione in un piano di raccolta dei tessuti.
- Asciugare a secco il tessuto fogliare con un liofilizzatore e conservarlo a -80 gradi centigradi.
- Mantenere gli impianti M2 a 20-25 gradi centigradi in un periodo di luce di 16 h.
- Registrare i dati sui fenotipi mutanti delle piante M2 a intervalli regolari. I fenotipi attesi sono albini, clorina, germogli erbosi, variegati, parzialmente fertili, sterili, ecc.
- Lasciare che le piante M2 si autofertilizzino e maturino. Raccogliere separatamente e salvare i semi M3 delle piante M2 (Figura 1).
NOTA: I semi M vengono utilizzati per convalidare il fenotipo negli studi di genetica inversa. Pertanto, M deve essere accuratamente catalogato e conservato in condizioni fredde e asciutte. In alternativa, se il seme deve essere aumentato in campo, le file della testa possono essere piantate per ogni pianta M e i semi M di ogni fila possono essere raccolti e salvati separatamente come risorsa della popolazione TILLING.
3. Analisi Cel-1 per la caratterizzazione genetica dei mutanti
- Estrarre il DNA dal tessuto fogliare di M2 utilizzando un kit di estrazione del DNA vegetale con un sistema di purificazione del DNA (vedi Tabella deimateriali) seguendo le raccomandazioni del produttore.
- Quantificare il DNA utilizzando uno spettrofotometro e normalizzare le concentrazioni di DNA a 25 ng/L con acqua priva di nucleatosi in 96 blocchi di pozzi.
NOTA: È importante controllare la qualità del DNA eseguendolo su un gel, poiché il DNA di bassa qualità (DNA spalmato) può provocare falsi negativi nei campioni raggruppati. - Crea bobine di DNA 4x combinando il DNA da quattro 96 blocchi di pozzo in una piastra, mantenendo l'identità di riga e colonna di ogni campione. Aggiungere 50 l di DNA da ogni singolo campione nella piastra della piscina in modo che ogni piastra della piscina contenga un totale di 200 L DiCI da quattro diversi 96 blocchi di pozzo.
- Catalogare l'identità del DNA raggruppato nel formato Pool Plate-Row-Column (ad esempio, Pool 1 A1 , Box1A1 , Box2A1 , Box3A1 , Box4A1 , Box4A1).
- Progettare primer specifici del genoma [A1] per il gene di interesse utilizzando la pagina Genome Specific Primers con impostazioni predefinite per le specie poliploidi. Per i diploidi, utilizzare Primer 3 per progettare i primer utilizzando le impostazioni predefinite. Progettare più primer, se necessario, per coprire l'intera regione di codifica del gene di interesse.
NOTA: l'ultimo assembly IWGSC può essere utilizzato per ottenere sequenze per il gene di interesse per il grano utilizzando lo strumento URGI BLAST . La lunghezza ottimale dell'amplificatore è compresa tra 800 e 1.500 bp. La tabella 1 mostra esempi di primer per geni cerosi nel grano esaploide. - Eseguire una PCR per primer gene-specifici su DNA raggruppato come segue:
- Aggiungete 5 ll l di buffer PCR, 2 L (ciascuno) di 4 M di primer in avanti e inverso, 0,1 l di polimerasi di DNA (vedi Tabella deimateriali), 5 l del modello di DNA raggruppato, quindi aumentate il volume a 25L utilizzando acqua priva di nuclenon.
- Utilizzare un profilo touch down per eseguire la reazione PCR su un ciclore termico come segue: 95 C per 1 min, sette cicli di 95 s per 1 min, 67 c a 60 c per 1 min con diminuzioni di temperatura di 1 oC per ciclo , seguito da 30 cicli di 95 gradi centigradi per 1 min, 60 gradi centigradi per 1 min, 72 gradi centigradi per 2 min, e da estensione finale a 72 gradi centigradi per 7 min.
NOTA: Sopra il profilo PCR funziona sul modello di DNA di grano per la maggior parte dei primer progettati utilizzando le impostazioni predefinite primer 3. In caso di amplificazione non specifica, il profilo PCR deve essere reso rigoroso prima di passare ai passaggi successivi.
- Generare eterodulessi tra il DNA non corrispondente incubando i prodotti PCR nel ciclore termico utilizzando il profilo come segue: 95 c per 2 min, cinque cicli di 95 gradi centigradi per 1 s, 95 c a 85 gradi centigradi per 1 min con diminuzioni di temperatura di 2 gradi centigradi per ciclo e 60 cicli di 85 s a 25 gradi con diminuzioni di 1oC per ciclo.
- Aggiungere 2,5 l di endonucleoce cel-1 fatto in casa ai prodotti PCR eterodelorosi e incubare per 45 min a 45 gradi centigradi. Terminare la reazione Cel-1 aggiungendo 2,5 L di 0,5 M EDTA (pH 8,0).
NOTA: Cel-1 endonuclease può essere estratto da gambi di sedano freschi utilizzando il protocollo eseguito da Till et al.19 È molto importante testare l'attività e la quantità ottimale di cel-1 endonuclease, che può essere testato utilizzando mutanti precedentemente caratterizzati o un kit di rilevamento delle mutazioni disponibile in commercio. - Eseguire i prodotti trattati con Cel-1 su un gel di agarose 3.0% a 100 V per 2,5 h. I pozzi contenenti bande scisse più piccole e uniche, oltre a bande zienare a lunghezza intera, conterranno il campione di DNA mutante.
- Denconvolute piscine mutanti.
- Seguire i passaggi 3.6, 3.7, 3.8 e 3.9 per singoli campioni di DNA M2 che si inventano i pool mutanti identificati nel passaggio 3.9.
- Per determinare la zigosità dei mutanti, eseguire due PCR (come descritto al passo 3.6) per il singolo DNA M 2, in cui la prima reazione contiene 2,5 L di DNA M2 e 2,5 L di DNA wild-type e la seconda reazione contiene solo 5 .L di DNA M2. Se la mutazione è eterozigo, una banda di zolle sarà presente in entrambe le reazioni. D'altra parte, ulteriori bande sciste si troveranno solo nella prima reazione se il mutante è omozio.
- Per identificare la natura della mutazione, sequenziare i prodotti PCR dei mutanti confermati utilizzando una piattaforma di sequenziamento di Sanger seguendo le istruzioni del produttore.
4. Calcolo della frequenza di mutazione
NOTA: la frequenza di mutazione di una popolazione TILLING si riferisce alla distanza fisica media in cui si verifica una mutazione negli individui di quella popolazione. Ad esempio, una frequenza di mutazione di 1/35 kb in una popolazione TILLING significa che un individuo medio di tale popolazione possiede 1 mutazione ogni 35 kb nel genoma.
- Per determinare la frequenza di mutazione di una popolazione TILLING, calcolare il numero totale di basi esaminate.
- Per calcolare il numero totale di basi esaminate, moltiplicare la dimensione del prodotto PCR per il numero totale di individui esaminati.
- Dividere il numero totale di basi esaminate per il numero di mutazioni uniche osservate utilizzando la seguente equazione, che produrrà la regione fisica che possiede 1 mutazione nella popolazione TILLING data:
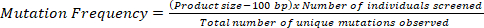
NOTA: per tenere conto della limitazione della risoluzione di 50 bp a entrambe le estremità sulla base di una piattaforma agarose a base di gel, sottrarre 100 bp dalla dimensione del prodotto nel calcolo.
Risultati
Figura 2 mostra la curva di dosaggio di cultivar di grano esaploide Jagger, grano diploide Triticum monococcum6e un donatore di genoma di grano Aegilops tauschii7. Le dosi di EMS per i tassi di sopravvivenza desiderati del 50% erano rispettivamente dello 0,25%, dello 0,6% e dello 0,7% per T. monococcum, Ae. tauschiie T. aestivum. La maggiore tolleranza EMS del grano e...
Discussione
TILLING è uno strumento di genetica inversa molto prezioso per la convalida genica, in particolare per i cereali di piccole dimensioni in cui gli approcci basati sulla trasformazione presentano gravi colli di bottiglia11. Lo sviluppo di una popolazione mutagenizzata con un'alta frequenza di mutazione è uno dei passi critici nello svolgimento di studi genomici funzionali. Il passo più importante nello sviluppo di una popolazione TILLING robusta è quello di determinare la concentrazione ottimale...
Divulgazioni
Gli autori non dichiarano interessi finanziari concorrenti.
Riconoscimenti
Questo lavoro è stato sostenuto dall'USDA National Institute of Food and Agriculture, progetto Hatch 1016879 e Maryland Agricultural Experiment Station tramite MAES Grant N. 2956952.
Materiali
| Name | Company | Catalog Number | Comments |
| 96 well 1.1 ml microtubes in microracks | National Scientific | TN0946-08R | For collecting leaf tissues |
| Agarose I biotechnology grade | VWR | 0710-500G | |
| Biosprint 96 DNA Plant Kit | Qiagen | 941558 | Kit for DNA extraction |
| Cel-1 endonuclease | Extracted as described by Till et al 2006 | Single strand specific endonuclease | |
| Centrifuge 5430 R | Eppendorf | ||
| Ethyl methanesulfonate | Sigma Aldrich | M-0880-25G | EMS, Chemical mutagen |
| Freeze Dry/Shell freeze system | Labconco | For lyophilization of leaf tissue | |
| Kingfisher Flex purification system | Thermo fisher scientific | 5400610 | High throughput DNA extraction robot |
| My Taq DNA Polymerase | Bioline | BIO-21107 | |
| Nuclease free water | Sigma aldrich | W4502-1L | |
| NuGenius gel imaging system | Syngene | ||
| Orbit Environ-shaker | Lab-line | ||
| SPECTROstar Nano | BMG LABTECH | Nano drop for DNA quantification | |
| T100 Thermal cycler | BIO-RAD | 1861096 |
Riferimenti
- McCallum, C. M., Comai, L., Greene, E. A., Henikoff, S. Targeted screening for induced mutations. Nature Biotechnology. 18 (4), 455-457 (2000).
- Bentley, A., MacLennan, B., Calvo, J., Dearolf, C. R. Targeted Recovery of Mutations in Drosophila. Genetics. 156 (3), 1169-1173 (2000).
- Tsai, H., et al. Discovery of Rare Mutations in Populations: TILLING by Sequencing. Plant Physiology. 156 (3), 1257-1268 (2011).
- Caldwell, D. G., et al. A structured mutant population for forward and reverse genetics in Barley (Hordeum vulgare L.). The Plant Journal. 40 (1), 143-150 (2004).
- Hazard, B., et al. Induced Mutations in the Starch Branching Enzyme II ( SBEII ) Genes Increase Amylose and Resistant Starch Content in Durum Wheat. Crop Science. 52 (4), 1754-1766 (2012).
- Rawat, N., et al. A diploid wheat TILLING resource for wheat functional genomics. BMC Plant Biology. 12, 205 (2012).
- Rawat, N., et al. TILL-D: An Aegilops tauschii TILLING Resource for Wheat Improvement. Frontiers in Plant Science. 9, (2018).
- Rawat, N., et al. Wheat Fhb1 encodes a chimeric lectin with agglutinin domains and a pore-forming toxin-like domain conferring resistance to Fusarium head blight. Nature Genetics. 48 (12), 1576-1580 (2016).
- Kippes, N., Chen, A., Zhang, X., Lukaszewski, A. J., Dubcovsky, J. Development and characterization of a spring hexaploid wheat line with no functional VRN2 genes. Theoretical and Applied Genetics. 129 (7), 1417-1428 (2016).
- Greene, E. A., et al. Spectrum of Chemically Induced Mutations From a Large-Scale Reverse-Genetic Screen in Arabidopsis. Genetics. 164 (2), 731-740 (2003).
- Harwood, W. A. Advances and remaining challenges in the transformation of barley and wheat. Journal of Experimental Botany. 63 (5), 1791-1798 (2012).
- Henikoff, S., Comai, L. Single-Nucleotide Mutations for Plant Functional Genomics. Annual Review of Plant Biology. 54 (1), 375-401 (2003).
- Uauy, C., et al. A modified TILLING approach to detect induced mutations in tetraploid and hexaploid wheat. BMC Plant Biology. 9 (1), 115 (2009).
- Uauy, C., Wulff, B. B. H., Dubcovsky, J. Combining Traditional Mutagenesis with New High-Throughput Sequencing and Genome Editing to Reveal Hidden Variation in Polyploid Wheat. Annual Review of Genetics. 51 (1), 435-454 (2017).
- Li, G., et al. The Sequences of 1504 Mutants in the Model Rice Variety Kitaake Facilitate Rapid Functional Genomic Studies. The Plant Cell. 29 (6), 1218-1231 (2017).
- Jiao, Y., et al. A Sorghum Mutant Resource as an Efficient Platform for Gene Discovery in Grasses. The Plant Cell. 28 (7), 1551-1562 (2016).
- Krasileva, K. V., et al. Uncovering hidden variation in polyploid wheat. Proceedings of the National Academy of Sciences. , 201619268 (2017).
- Dong, C., Dalton-Morgan, J., Vincent, K., Sharp, P. A Modified TILLING Method for Wheat Breeding. The Plant Genome. 2 (1), 39-47 (2009).
- Till, B. J., Zerr, T., Comai, L., Henikoff, S. A protocol for TILLING and Ecotilling in plants and animals. Nature Protocols. 1 (5), 2465-2477 (2006).
- Wu, J. -. L., et al. Chemical- and Irradiation-induced Mutants of Indica Rice IR64 for Forward and Reverse Genetics. Plant Molecular Biology. 59 (1), 85-97 (2005).
- Feldman, M., Levy, A. A. Genome Evolution Due to Allopolyploidization in Wheat. Genetics. 192 (3), 763-774 (2012).
- Comai, L. The advantages and disadvantages of being polyploid. Nature Reviews Genetics. 6 (11), 836-846 (2005).
- Guo, H., et al. Development of a High-Efficient Mutation Resource with Phenotypic Variation in Hexaploid Winter Wheat and Identification of Novel Alleles in the TaAGP.L-B1 Gene. Frontiers in Plant Science. 8, (2017).
- Rakszegi, M., et al. Diversity of agronomic and morphological traits in a mutant population of bread wheat studied in the Healthgrain program. Euphytica. 174 (3), 409-421 (2010).
- Tsai, H., Ngo, K., Lieberman, M., Missirian, V., Comai, L. Tilling by Sequencing. Plant Functional Genomics: Methods and Protocols. , 359-380 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon