Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Развитие ориентации индуцированных местных поражений в геномах (TILLING) Популяции в малых зерновых культур по этил метанолметанофената мутагенеза
В этой статье
Резюме
Описано это протокол для разработки ориентации индуцированных местных поражений в геномах (TILLING) населения в мелких зерновых культур с использованием этилового метанасульфоната (EMS) в качестве мутагена. Также предусмотрен протокол для обнаружения мутаций с помощью асссея Cel-1.
Аннотация
Ориентация на индуцированные локальные поражения в геномах (TILLING) является мощным инструментом обратной генетики, который включает в себя химический мутагенез и обнаружение вариации последовательности в генах-мишенях. TILLING является очень ценным функциональным инструментом геномики для проверки генов, особенно в небольших зернах, в которых подходы, основанные на трансформации, имеют серьезные ограничения. Разработка надежной мутагенизированной популяции является ключом к определению эффективности исследования генной валидации на основе TILLING. Популяция TILLING с низкой общей частотой мутаций указывает на то, что непрактично большая популяция должна быть проверена, чтобы найти желаемые мутации, в то время как высокая концентрация мутагена приводит к высокой смертности в популяции, что приводит к недостаточной мутагенизированных особей. После того, как эффективная популяция будет разработана, существует несколько способов обнаружения мутаций в интересуемом гене, и выбор платформы зависит от экспериментального масштаба и наличия ресурсов. Cel-1 ассса и агарозный гель-подход для идентификации мутантов является удобным, воспроизводимым и менее ресурсоемким платформой. Это выгодно тем, что оно простое, не требующее вычислительных знаний, и особенно подходит для проверки небольшого количества генов с базовым лабораторным оборудованием. В настоящей статье описаны методы развития хорошей популяции TILLING, включая подготовку кривой дозировки, мутагенеза и поддержание популяции мутантов, а также скрининг популяции мутантов с использованием анализов Cel-1 на основе ПЦР .
Введение
Точечные мутации в геномах могут служить исследователям многим полезными целями. В зависимости от их характера и местоположения, эти мутации могут быть использованы для присвоения функций генам или даже различным областям белков, представляющих интерес. С другой стороны, в качестве источника новых генетических вариаций, полезные мутации могут быть выбраны для желаемых признаков с помощью фенотипирования экранов и далее используется в улучшении урожая. TILLING является мощным инструментом обратной генетики, который включает в себя химический мутагенез и обнаружение изменения последовательности в гене-мишени. Впервые разработан в Arabidopsis1 и Drosophilia melanogaster2, TILLING популяций были разработаны и использованы во многих небольших зерновых культур, таких как гексаплоидный хлеб пшеницы (Triticum aestivum)3, ячмень (Hordeum vulgare)4, тетраплоидная пшеница дурума (T. dicoccoides durum)5, диплоидная пшеница (T. monococcum)6 и "D" геном прародитель пшеницы Aegilops tauschii7 . Эти ресурсы были использованы для проверки роли генов в регулировании абиотических и биотических стрессоустойчивость8, регулирующие время цветения9, и развивающихся питательно превосходных сортов сельскохозяйственных культур5.
TILLING, наряду с использованием алкилирующих мутагенных агентов, таких как этил метанесульфонат (EMS), азид натрия, N-метил-N-нитросурея (MNU), и метилметанесульфонат (MMS), имеет преимущества по сравнению с другими инструментами обратной генетики по нескольким причинам. Во-первых, мутагенез может проводиться практически на любом виде или сорте растения10 и не зависит от узкого места трансформации, что особенно сложно в случае мелких зерен11. Во-вторых, в дополнение к генерации нокаут мутации, которые могут быть получены с помощью других подходов проверки генов, ряд неправильности и сплайсинга мутации могут быть вызваны, которые могут различить функции отдельных областей белков, представляющих интерес12. Кроме того, TILLING генерирует бессмертную коллекцию мутаций по всему геному; таким образом, одна популяция может быть использована для функциональной проверки нескольких генов. В отличие от этого, другие инструменты обратной генетики генерировать ресурсы, характерные только для гена в исследовании13. Полезные мутации, выявленные с помощью TILLING, могут быть развернуты в целях размножения и не подлежат регулированию, в отличие от редактирования генов, чья нетрансгенная классификация по-прежнему не определена во многих странах. Это становится особенно актуальным для мелких зерен, которые на международном рынке торгуются14.
TILLING является простой и эффективной стратегией проверки генов и требует разработки мутагенизированных популяций для исследования генов, представляющих интерес. Разработка эффективной мутагенизированной популяции является ключом к определению эффективности исследования генной валидации на основе TILLING. Популяция TILLING с низкой общей частотой мутаций указывает на то, что непрактично большая популяция должна быть проверена на желаемые мутации, в то время как высокая концентрация мутагенов приводит к высокой смертности в популяции и недостаточному числу мутагенизированных особей. После того, как хорошая популяция будет разработана, существует несколько способов обнаружения мутаций в интересуемых генах, а выбор платформы зависит от экспериментального масштаба и наличия ресурсов. Полное секвенирование генома и секвенирование экзома было использовано для характеристики всех мутаций в популяциях TILLING в растениях с небольшими геномами15,16. Exome секвенирование двух популяций TILLING было выполнено в хлебе и пшенице durum и доступно для общественности для выявления желательных мутаций и заказа мутантных линий интереса17. Это большой общественный ресурс с точки зрения наличия желательных мутаций; однако, в исследованиях проверки гена, линия дикого типа должна обладать интересующим геном-кандидатом. К сожалению, это все еще непомерно затратный секвенировать экзома всей популяции TILLING для обратной генетики на основе проверки нескольких генов-кандидатов в другом фоне. Ампликон секвенирования и Cel-1 основе анализы были использованы в обнаружении мутаций в целевых популяций в пшенице, и Cel-1 анализы проще, не требуя вычислительных знаний, и особенно подходят для проверки небольшого числа генов с основными лабораторное оборудование6,18.
В настоящей статье описаны методы развития хорошей популяции TILLING, включая подготовку кривой дозировки, мутагенеза и поддержание популяции мутантов, а также скрининг популяции мутантов с использованием анализов Cel-1 на основе ПЦР . Этот протокол уже успешно реализован в разработке и использовании мутагенизированных популяций Triticum aestivum, Triticum monoccocum6, ячменя, Aegilops tauchii7, и несколько Другие. Включены явные детали этих методов наряду с полезными советами, которые помогут исследователям развивать популяции TILLING, используя EMS в качестве мутагена в любом небольшом зерновом заводе выбора.
Access restricted. Please log in or start a trial to view this content.
протокол
1. Подготовка кривой дозировки для эффективного мутагенеза
- Замочите 100 семян с генотипом интереса в шести 250 мл стеклянных колб (100 в каждой колбе), содержащей 50 мл дистиллированной воды. Встряхните при 100 об/мин в течение 8 ч при комнатной температуре (RT) для впитывания семенами.
- В дымовой капот, подготовить 50 мл 0,4%, 0,6%, 0,8%, 1,0%, и 1,2% (w/v) этилметанесульфонат (EMS) решение растворения 0,167, 0,249, 0,331, 0,415, и 0,498 мл ЭМС в дистиллированной воде, соответственно.
ПРИМЕЧАНИЕ: EMS является жидким на RT с плотностью 1,206 г/мл.
ВНИМАНИЕ: Используйте соответствующее индивидуальное защитное оборудование (PPE) при обращении с EMS. - Декант воды из пяти колб и добавить 50 мл раствора EMS в каждой колбе, содержащей впитанные семена так, что Есть шесть различных процедур с 0,0%, 0,4%, 0,6%, 0,8%, 1,0%, и 1,2% EMS решение. Встряхните колбы для 16 ч при 75 об/мин и RT.
- Декант раствор EMS и собирать обработанные семена отдельно для каждой обработки с помощью сырной ткани. Инактивируйте использованное решение EMS, добавив один том раствора EMS-инактивирования (0,1 M NaOH, 20% w/v Na2S2O3) за 24 ч. Также обработайте загрязненные колбы и пипетки с помощью раствора, инактивирующие EMS, в течение 24 ч.
- Вымойте EMS обработанных семян под проточной водопроводной водой в течение 2 ч. Трансплантация каждого семени индивидуально в корневые тренажеры, содержащие почву заливки.
- Выращивайте растения при 20-25 градусах Цельсия при 16-м л.
- Запись данных о выживаемости растений после 15 дней трансплантации. Используйте следующее уравнение для расчета выживаемости для каждого лечения:
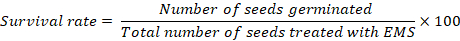
ПРИМЕЧАНИЕ: Если скорость прорастания ниже 100% в элементах управления, точные показатели выживаемости во всех лечения должны быть рассчитаны после вычитания количество семян, которые не прорастают в элементах управления. Выживаемость 40%-60% желательно для эффективного мутагенеза. Это может потребоваться для выполнения второго раунда оптимизации дозы с измененной концентрацией в зависимости от выживания обработанных семян до достижения желаемого коэффициента летальности 40%-60%.
2. Мутагенез и содержание популяции мутантов
- Замочите последнюю партию, по крайней мере 3000 семян, разделяющих поровну (600 семян каждый) в пяти 1000 мл колбы, содержащие 300 мл дистиллированной воды. Встряхните в течение 8 ч при 100 об/мин при RT для впитывания.
ПРИМЕЧАНИЕ: Окончательный размер желаемой популяции будет зависеть от частоты мутации и уровня флоиди генотипа, но желательно использовать не менее 3000 семян для гексаплоидов, 4000 семян для тетраплоидов и более 7000 семян для диплоидов. - В дымящийся капот приготовьте 1500 мл оптимизированного раствора концентрации EMS в дистиллированной воде.
- Декант воды из колбы и добавить 300 мл оптимальной концентрации раствора EMS в каждой колбе, содержащей 600 впитанных семян. Встряхните колбы на 16 ч при 75 об/мин и RT.
- Декант EMS и собирать обработанные семена в сырной ткани. Инактивируйте раствор EMS и контейнеры для обработки с помощью решения, инактивируемого EMS, как это делается на этапе 1.4.
- Вымойте EMS обработанных семян под проточной водопроводной водой в течение 2 ч. Трансплантация каждого EMS-обработанных M1 семян индивидуально в корневые тренажеры.
- Выращивайте растения M1 (производные от семян M0) при 20-25 градусах Цельсия при 16-м л.
ПРИМЕЧАНИЕ: Это может потребоваться для vernalize саженцы на стадии двух листьев в течение 6 недель при 4 градусах Цельсия, если генотип интереса имеет зимой типа роста привычки. - Разрешить M1 растений для самостоятельного опывания, и урожай M2 семена отдельно для каждого плодородного растения М1.
ПРИМЕЧАНИЕ: Чтобы избежать шансов потенциального перехиппинга, накройте шипы растений M1 пакетами опыления перед anthesis. - Завод одного М2 семян от каждого растения M1, чтобы избежать генетической избыточности.
- Соберите ткани из растений M2 на двухлистной стадии в 1,1 мл из мучил 96 хорошо микротруб. Соберите около 80 мм листовой ткани с каждого растения и запишите идентификатор каждого образца в плане сбора тканей.
- Заморозить-сухой ткани листьев с помощью лиофилизатора и хранить при -80 градусах Цельсия.
- Поддерживайте растения M2 при 20-25 градусах Цельсия при 16-м л.
- Запись данных о фенотипах мутантов растений M2 регулярно. Ожидаемые фенотипы альбиносы, хлорины, травянистые побеги, пестрые, частично плодородные, стерильные и т.д.
- Разрешить M2 растений для самоудобрения и созревания. Отдельно урожай и сохранить Семена M3 из M2 растений (рисунок1).
ПРИМЕЧАНИЕ: M семена используются для проверки фенотипа в обратном исследования генетики. Поэтому M следует тщательно каталогизировать и хранить в прохладных и сухих условиях. Кроме того, если семена должны быть увеличены в поле, головные ряды могут быть посажены для каждого растения M, и Семена M из каждого ряда могут быть собраны и сохранены отдельно в качестве ресурса населения TILLING.
3. Cel-1 асссидлявый для генетической характеристики мутантов
- Извлеките ДНК из листовой ткани M2 с помощью комплекта для извлечения ДНК растений с системой очистки ДНК (см. ТаблицуМатериалов) в соответствии с рекомендациями производителя.
- Количественная ДНК с помощью спектрофотометра и нормализуйте концентрацию ДНК до 25 нг/Л с нуклеазой-свободной водой в 96 колодцах.
ПРИМЕЧАНИЕ: Важно проверить качество ДНК, запустив ее на гель, так как низкокачественная ДНК (смазанная ДНК) может привести к ложным негативам в объединенных образцах. - Создайте 4x ДНК пулы, объединив ДНК из четырех 96 блоков скважины в одну пластину, сохраняя при этом ряд и столбец идентичности каждого образца. Добавьте 50 кЛ ДНК из каждого отдельного образца в бильярдную тарелку, чтобы каждая тарелка бассейна хорошо содержит в общей сложности 200 л ДНК из четырех различных 96 блоков скважин.
- Каталог итог объединенной ДНК в формате Pool Plate-Row-Column (например, пул 1 A1 и Box1A1 - Box2A1 - Box2A1 - Box3A1 - Box4A1 - Box4A1 ).
- Дизайн генома конкретных праймеров «A1» для гена, представляющих интерес, используя геном конкретных праймеров (GSP) страница lt;https://probes.pw.usda.gov/GSP gt; с настройками по умолчанию для полиплоидных видов. Для диплоидов используйте Primer 3 lt;http://primer3.ut.ee/'gt; для проектирования праймеров с использованием настроек по умолчанию. Дизайн нескольких грунтовок, если это необходимо, чтобы охватить всю область кодирования гена интереса.
ПРИМЕЧАНИЕ: Последняя сборка IWGSC может быть использована для получения последовательностей для гена, интересуемого пшеницей, используя инструмент URGI BLAST lt;https://wheat-urgi.versailles.inra.fr/Seq-Repository/BLAST-gt.gt.. Оптимальная длина ампликона находится в диапазоне 800-1500 б.п. Таблица 1 показывает примеры грунтовок для восковых генов в гексаплоидной пшенице. - Выполнить ПЦР для генных грунтовков на объединенной ДНК следующим образом:
- Добавьте 5 qL буфера ПЦР, 2 qL (каждый) из 4 мкм вперед и обратные грунтовки, 0,1 л полимеразы ДНК (см. Таблица материалов), 5 зл ел объединенного шаблона ДНК, затем увеличьте объем до 25 qL с помощью безнужной воды.
- Используйте профиль касания вниз для того чтобы побежать реакция PCR на тепловом цикле следующим образом: 95 C для 1 min, 7 циклов 95 C для 1 мин, 67 C до 60 C для 1 min с понижением температуры 1 C в цикл е и 72 C для 2 мин , а затем 30 циклов 95 градусов по Цельсию в течение 1 мин, 60 c в течение 1 мин, 72 C в течение 2 мин, и окончательное расширение на 72 C в течение 7 мин.
ПРИМЕЧАНИЕ: Выше пЦР профиль работает на шаблоне ДНК пшеницы для большинства грунтовок, разработанных с использованием грунтовки 3 параметров по умолчанию. В случае неспецифического усиления профиль ПЦР следует сделать строгим, прежде чем переходить к следующим шагам.
- Генерировать гетеродуплексы между несовпадением ДНК путем инкубации продуктов ПЦР в тепловом цикле с использованием профиля следующим образом: 95 градусов по Цельсию в течение 2 мин, пять циклов 95 градусов по Цельсию на 1 с, 95 градусов по Цельсию в течение 1 мин с понижением температуры от 2 градусов по Цельсию за цикл, и 60 циклов от 85 до 25 градусов с уменьшается на 1 градус цельсия за цикл.
- Добавьте 2,5 злителк домашней эндонуклеазы Cel-1 в неоздоровую продукцию ПЦР и инкубировать в течение 45 мин при 45 градусах Цельсия. Прекратите реакцию Cel-1, добавив 2,5 л 0,5 М ЭДТА (pH 8.0).
ПРИМЕЧАНИЕ: Эндонуклеаза Cel-1 может быть извлечена из свежих стеблей сельдерея с помощью протокола, выполненного Till et al.19 Очень важно проверить активность и оптимальное количество эндонуклеазы Cel-1, которая может быть протестирована с использованием ранее охарактеризованных мутантов или коммерчески доступный набор обнаружения мутаций. - Запустите Cel-1 обработанных продуктов на 3,0% агарозный гель на 100 В за 2,5 ч. Скважины, содержащие меньшие и уникальные расщепленные полосы (ы), в дополнение к полнометражным дядедными полосами, будут содержать образец ДНК мутантов.
- Денконволутные бассейны-мутанты.
- Выполните шаги 3.6, 3.7, 3.8 и 3.9 для отдельных образцов ДНК M 2, составляющих пулы мутантов, идентифицированные в шаге 3.9.
- Чтобы определить zygosity мутантов, запустить два ПЦР (как описано в шаге 3.6) для отдельных M2 ДНК, в котором первая реакция содержит 2,5 злл Из M2 ДНК и 2,5 л ДНК дикого типа и вторая реакция содержит только 5 Зл М2 ДНК. Если мутация гетерозиготная, в обеих реакциях будет присутствовать дополнительная расщепляемая полоса. С другой стороны, дополнительные расщепляемые полосы будут найдены только при первой реакции, если мутант гомозигот.
- Чтобы определить характер мутации, последовательность ПЦР продуктов подтвержденных мутантов с помощью платформы секвенирования Sanger в соответствии с инструкциями производителя.
4. Расчет частоты мутаций
ПРИМЕЧАНИЕ: Частота мутации популяции TILLING относится к среднему физическому расстоянию, на котором одна мутация происходит у индивидуумов этой популяции. Например, частота мутаций 1/35 кб в популяции TILLING означает, что средний человек этой популяции обладает 1 мутацией на каждые 35 кб в геноме.
- Чтобы определить частоту мутаций популяции TILLING, вычислите общее количество проверенных баз.
- Чтобы вычислить общее количество проверенных баз, умножьте размер продукта ПЦР на общее количество проверенных лиц.
- Разделите общее количество оснований, скрининговых на количество уникальных мутаций, наблюдаемых с помощью следующего уравнения, что даст физический регион, обладающий 1 мутацией в данной популяции TILLING:
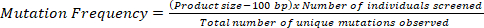
ПРИМЕЧАНИЕ: Для учета ограничения в разрешении 50 bp на обоих концах на основе агарозного геля на основе платформы, вычесть 100 bp от размера продукта в расчете.
Access restricted. Please log in or start a trial to view this content.
Результаты
На рисунке 2 показана кривая дозировки гексаплоидного хлеба пшеницы сорт Джаггер, диплоидная пшеница Triticum monococcum6, и геном донор пшеницы Aegilops tauschii7. Дозы EMS для желаемых 50% выживаемости составили около 0,25%, 0,6% и 0,7% для T...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
TILLING является весьма ценным инструментом обратной генетики для проверки генов, особенно для мелких зерен, где трансформационные подходы имеют серьезные узкие места11. Развитие мутагенизированной популяции с высокой частотой мутаций является одним из важнейших этапов в пр...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Авторы не заявляют о конкурирующих финансовых интересах.
Благодарности
Эта работа была поддержана Министерством сельского хозяйства США Национальным институтом продовольствия и сельского хозяйства, Проектом Hatch 1016879 и Мэрилендской сельскохозяйственной экспериментальной станцией через MAES Grant No 2956952.
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| 96 well 1.1 ml microtubes in microracks | National Scientific | TN0946-08R | For collecting leaf tissues |
| Agarose I biotechnology grade | VWR | 0710-500G | |
| Biosprint 96 DNA Plant Kit | Qiagen | 941558 | Kit for DNA extraction |
| Cel-1 endonuclease | Extracted as described by Till et al 2006 | Single strand specific endonuclease | |
| Centrifuge 5430 R | Eppendorf | ||
| Ethyl methanesulfonate | Sigma Aldrich | M-0880-25G | EMS, Chemical mutagen |
| Freeze Dry/Shell freeze system | Labconco | For lyophilization of leaf tissue | |
| Kingfisher Flex purification system | Thermo fisher scientific | 5400610 | High throughput DNA extraction robot |
| My Taq DNA Polymerase | Bioline | BIO-21107 | |
| Nuclease free water | Sigma aldrich | W4502-1L | |
| NuGenius gel imaging system | Syngene | ||
| Orbit Environ-shaker | Lab-line | ||
| SPECTROstar Nano | BMG LABTECH | Nano drop for DNA quantification | |
| T100 Thermal cycler | BIO-RAD | 1861096 |
Ссылки
- McCallum, C. M., Comai, L., Greene, E. A., Henikoff, S. Targeted screening for induced mutations. Nature Biotechnology. 18 (4), 455-457 (2000).
- Bentley, A., MacLennan, B., Calvo, J., Dearolf, C. R. Targeted Recovery of Mutations in Drosophila. Genetics. 156 (3), 1169-1173 (2000).
- Tsai, H., et al. Discovery of Rare Mutations in Populations: TILLING by Sequencing. Plant Physiology. 156 (3), 1257-1268 (2011).
- Caldwell, D. G., et al. A structured mutant population for forward and reverse genetics in Barley (Hordeum vulgare L.). The Plant Journal. 40 (1), 143-150 (2004).
- Hazard, B., et al. Induced Mutations in the Starch Branching Enzyme II ( SBEII ) Genes Increase Amylose and Resistant Starch Content in Durum Wheat. Crop Science. 52 (4), 1754-1766 (2012).
- Rawat, N., et al. A diploid wheat TILLING resource for wheat functional genomics. BMC Plant Biology. 12, 205(2012).
- Rawat, N., et al. TILL-D: An Aegilops tauschii TILLING Resource for Wheat Improvement. Frontiers in Plant Science. 9, (2018).
- Rawat, N., et al. Wheat Fhb1 encodes a chimeric lectin with agglutinin domains and a pore-forming toxin-like domain conferring resistance to Fusarium head blight. Nature Genetics. 48 (12), 1576-1580 (2016).
- Kippes, N., Chen, A., Zhang, X., Lukaszewski, A. J., Dubcovsky, J. Development and characterization of a spring hexaploid wheat line with no functional VRN2 genes. Theoretical and Applied Genetics. 129 (7), 1417-1428 (2016).
- Greene, E. A., et al. Spectrum of Chemically Induced Mutations From a Large-Scale Reverse-Genetic Screen in Arabidopsis. Genetics. 164 (2), 731-740 (2003).
- Harwood, W. A. Advances and remaining challenges in the transformation of barley and wheat. Journal of Experimental Botany. 63 (5), 1791-1798 (2012).
- Henikoff, S., Comai, L. Single-Nucleotide Mutations for Plant Functional Genomics. Annual Review of Plant Biology. 54 (1), 375-401 (2003).
- Uauy, C., et al. A modified TILLING approach to detect induced mutations in tetraploid and hexaploid wheat. BMC Plant Biology. 9 (1), 115(2009).
- Uauy, C., Wulff, B. B. H., Dubcovsky, J. Combining Traditional Mutagenesis with New High-Throughput Sequencing and Genome Editing to Reveal Hidden Variation in Polyploid Wheat. Annual Review of Genetics. 51 (1), 435-454 (2017).
- Li, G., et al. The Sequences of 1504 Mutants in the Model Rice Variety Kitaake Facilitate Rapid Functional Genomic Studies. The Plant Cell. 29 (6), 1218-1231 (2017).
- Jiao, Y., et al. A Sorghum Mutant Resource as an Efficient Platform for Gene Discovery in Grasses. The Plant Cell. 28 (7), 1551-1562 (2016).
- Krasileva, K. V., et al. Uncovering hidden variation in polyploid wheat. Proceedings of the National Academy of Sciences. , 201619268(2017).
- Dong, C., Dalton-Morgan, J., Vincent, K., Sharp, P. A Modified TILLING Method for Wheat Breeding. The Plant Genome. 2 (1), 39-47 (2009).
- Till, B. J., Zerr, T., Comai, L., Henikoff, S. A protocol for TILLING and Ecotilling in plants and animals. Nature Protocols. 1 (5), 2465-2477 (2006).
- Wu, J. -L., et al. Chemical- and Irradiation-induced Mutants of Indica Rice IR64 for Forward and Reverse Genetics. Plant Molecular Biology. 59 (1), 85-97 (2005).
- Feldman, M., Levy, A. A. Genome Evolution Due to Allopolyploidization in Wheat. Genetics. 192 (3), 763-774 (2012).
- Comai, L. The advantages and disadvantages of being polyploid. Nature Reviews Genetics. 6 (11), 836-846 (2005).
- Guo, H., et al. Development of a High-Efficient Mutation Resource with Phenotypic Variation in Hexaploid Winter Wheat and Identification of Novel Alleles in the TaAGP.L-B1 Gene. Frontiers in Plant Science. 8, (2017).
- Rakszegi, M., et al. Diversity of agronomic and morphological traits in a mutant population of bread wheat studied in the Healthgrain program. Euphytica. 174 (3), 409-421 (2010).
- Tsai, H., Ngo, K., Lieberman, M., Missirian, V., Comai, L. Tilling by Sequencing. Plant Functional Genomics: Methods and Protocols. , 359-380 (2015).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены