A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
פיתוח מיקוד המושרה נגעים מקומיים בשנת Genomes (לעיבוד קרקע) אוכלוסיות בגידולים גרעינים קטנים על ידי אתיל מתיונין מוטאנזיס
In This Article
Summary
המתואר הוא פרוטוקול לפיתוח נגעים מקומיים המושרה הנגרמת בידי האוכלוסייה Genomes (לעיבוד קרקע) בגידולי תבואה קטנים עם שימוש אתיל מתיונין (EMS) כמו מוטגן. כמו כן, מסופק הוא פרוטוקול לגילוי מוטציה באמצעות שיטת Cel-1.
Abstract
פילוח המושרה נגעים מקומיים Genomes (לעיבוד קרקע) הוא כלי הגנטיקה הפוכה רבת עוצמה הכוללת מוטזיס כימי וגילוי של וריאציה רצף בגנים היעד. לעיבוד קרקע הוא כלי גנומיקה פונקציונלי רב ערך עבור אימות גנים, במיוחד בגרגרים קטנים בהם גישות מבוססות שינוי להחזיק מגבלות חמורות. פיתוח אוכלוסיית מוטייציה חזקה הוא המפתח לקביעת היעילות של מחקר ואימות גנים מבוססי לעיבוד קרקע. אוכלוסיית לעיבוד קרקע עם תדר נמוך של מוטציה כוללת מצביעה על כך שאוכלוסיה גדולה מאוד צריכה להיות מוקרן כדי למצוא מוטציות רצויות, בעוד שריכוז מוטפן גבוה מוביל לתמותה גבוהה באוכלוסייה, המובילה ל ספר אנשים מוטניים. לאחר שפותחה אוכלוסייה יעילה, קיימות מספר דרכים לזיהוי מוטציות בגנים של עניין, והבחירה בפלטפורמה תלויה בקנה המידה הניסיוני ובזמינות המשאבים. שיטת Cel-1 וגישה מבוססת ג'ל לזיהוי מוטציה היא נוחה, מנויית ופלטפורמה פחות אינטנסיבית למשאבים. זה יתרון בכך שהוא פשוט, שאינו דורש ידע מעמיק, והוא מתאים במיוחד לאימות של מספר קטן של גנים עם ציוד מעבדה בסיסי. במאמר הנוכחי, מתוארים השיטות לפיתוח של אוכלוסיית לעיבוד טוב, כולל הכנת עקומת המינון, מוטגנזה ותחזוקה של אוכלוסיית המוטציות, והקרנת אוכלוסיית המוטציות באמצעות הטיפול המבוסס על ה-PCR-1 .
Introduction
מוטציות נקודה גנום יכול לשרת מטרות שימושיות רבות עבור חוקרים. בהתאם הטבע והמיקום שלהם, מוטציות אלה ניתן להשתמש כדי להקצות פונקציות לגנים או תחומים שונים של חלבונים של הריבית. מצד שני, כמקור של וריאציה גנטית הרומן, מוטציות שימושיות ניתן לבחור תכונות הרצוי באמצעות מסכי פנוטיפים ובשימוש נוסף שיפור ביבול. לעיבוד קרקע הוא כלי הגנטיקה הפוכה רבת עוצמה הכוללת מוטזיס כימי ואיתור וריאציה של רצף בגן היעד. פותח לראשונה ב arabidopsis1 ו droסופיזם מלאנוסטר2, אוכלוסיות לעיבוד לקרקע פותחו ומנוצל בגידולי תבואה קטנים רבים כגון חיטה של לחם הקסאפואיד (triticum חיטת)3, שעורה ( הורדאוםוולגאר)4, מחיטה הטטרדואיד (t. חיטת דורום)5, חיטה דידואיד (t. monococcum)6 ו-"D" הגנום מחולל חיטה של אייליאופ . משאבים אלה שימשו כדי לאמת את התפקידים של גנים בוויסות העמידות הביוטית וביוטיים ללחץ8, ויסות הפריחה זמן9, ופיתוח זנים היבול מעולה מבחינה תזונתית5.
לעיבוד קרקע, יחד עם השימוש בסוכני מוטאריים אלקיגונים כגון אתיל מתיונין (EMS), נתרן azide, N-מתיל-N-ניטרוסוראה (MNU), ו מתיל מתיונין (MMS), יש יתרונות על פני כלים אחרים גנטיקה הפוכה מכמה סיבות. ראשית, ניתן לנהל מוטגנזה על כל מין או מגוון של צמחים10 והוא בלתי תלוי בצוואר המרה, שהוא מאתגר במיוחד במקרה של גרגרים קטנים11. שנית, בנוסף ליצירת מוטציות הסתרה שניתן להשיג על ידי גישות אחרות אימות גנים, מגוון של מוטציות מוטעית ושחבור יכול להיגרם, אשר יכול להבחין פונקציות של תחומים בודדים של חלבונים של ריבית12. יתר על כן, לעיבוד קרקע מייצרת אוסף בן אלמוות של מוטציות ברחבי הגנום; לפיכך, ניתן להשתמש באוכלוסיה בודדת לאימות פונקציונלי של גנים מרובים. לעומת זאת, כלים אחרים גנטיקה הפוכה ליצור משאבים ספציפיים רק הגן תחת לימוד13. מוטציות שימושיות שזוהו באמצעות לעיבוד קרקע ניתן לפרוס למטרות רבייה אינם כפופים רגולציה, בניגוד לעריכת גנים, שסיווג לא הטרנסגניים עדיין לא בטוח במדינות רבות. הדבר הופך להיות רלוונטי במיוחד לגרגרים קטנים הנסחרים ברחבי העולם14.
לעיבוד קרקע היא אסטרטגיה פשוטה ויעילה לאימות גנים ודורשת אוכלוסיות מוטשות להתפתח לחקירת גנים מעניינים. פיתוח אוכלוסיה מוטבית יעילה היא המפתח לקביעת היעילות של מחקר ואימות גנים מבוססי לעיבוד קרקע. אוכלוסיית לעיבוד קרקע עם תדר נמוך של מוטציה כוללת מצביעה על כך שאוכלוסיה גדולה מאוד צריכה להיות מוקרן למוטציות הרצויות, בעוד שריכוז מוטאני גבוה מוביל לתמותה גבוהה באוכלוסיה ולמספר לא מספיק של אנשים מוטניים. לאחר התפתחות האוכלוסייה הטובה, קיימות מספר דרכים לזיהוי מוטציות בגנים המעניינים, ובחירת הפלטפורמה תלויה בקנה המידה הניסיוני ובזמינות המשאבים. רצף הגנום כולו רצף exome שימשו כדי לאפיין את כל מוטציות באוכלוסיות לעיבוד הצמחים עם קטן גנום15,16. רצף exome של שתי אוכלוסיות לעיבוד קרקע בוצעה בלחם וחיטה דורום זמין לציבור לזיהוי מוטציות רצויות והזמנת קווי מוטציה של עניין17. זהו משאב ציבורי גדול במונחים של זמינות של מוטציות רצויות; עם זאת, בלימודי האימות הגנטי, קו הטיפוס הפראי צריך להחזיק את גן המועמדים המעניין. למרבה הצער, זה עדיין עלות-אוסרני לרצף את האוכלוסייה של כל אוכלוסיית לעיבוד האוכלוסין כולו עבור הפוך גנטיקה מבוססי אימות של כמה גנים מועמד ברקע אחר. Amplicon רצף ו-Cel-1 מבוססי בחני שימשו בזיהוי מוטציות באוכלוסיות ממוקדות חיטה, ו Cel-1 בחני הם פשוט, לא דורש ידע מחשבתי, והם מתאימים במיוחד לאימות של מספר קטן של גנים עם בסיסי ציוד מעבדה6,18.
במאמר הנוכחי, מתוארים שיטות להתפתחות של אוכלוסיית לעיבוד טוב, כולל הכנת עקומת המינון, מוטגנזה ותחזוקה של אוכלוסיית המוטציות, והקרנת אוכלוסיית המוטציות באמצעות השיטה המבוססת על ה-PCR-1 . פרוטוקול זה כבר יושם בהצלחה בפיתוח וניצול אוכלוסיות מוטשות של triticum חיטת, triticum מונובהצטיינות6, שעורה, אגינה tauchii7, ומספר אחרים. כלולים פרטים מפורשת של שיטות אלה יחד עם טיפים שימושיים שיסייעו לחוקרים לפתח אוכלוסיות לעיבוד קרקע, באמצעות EMS כמוטגן בכל צמח דגן קטן של בחירה.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. הכנת עקומת מינון למוטזיס אפקטיבי
- להשרות 100 זרעים עם גנוטיפ של הריבית 6 250 mL מבחנות זכוכית (100 בכל בקבוקון) המכיל 50 mL של מים מזוקקים. נענעי ב 100 סל ד עבור 8 h בטמפרטורת החדר (RT) לקבלת שתיית הזרעים.
- בתוך משקה, להכין 50 mL של 0.4%, 0.6%, 0.8%, 1.0%, ו 1.2% (w/v) אתיל מתיאנסולולאט (EMS) פתרון על ידי המסת 0.167, 0.249, 0.331, 0.415, ו 0.498 mL של EMS במים מזוקקים, בהתאמה.
הערה: EMS הוא נוזל ב-RT עם צפיפות של 1.206 g/mL.
התראה: השתמש בציוד הגנה אישי מתאים (PPE) בעת טיפול ב-EMS. - Decant המים מתוך חמש מבחנות ולהוסיף 50 mL של פתרון EMS בכל בקבוקון המכיל את הזרע לשתות כך יש שישה טיפולים שונים עם 0.0%, 0.4%, 0.6%, 0.8%, 1.0%, ו 1.2% EMS פתרון. לנער מבחנות עבור 16 h ב 75 סל ד ו-RT.
- Decant פתרון EMS ולאסוף את הזרעים שטופלו בנפרד עבור כל טיפול באמצעות מטלית גבינה. הפוך את פתרון EMS המשמש לשימוש על-ידי הוספת אמצעי אחסון אחד של פתרון EMS-הפעלה (0.1 M NaOH, 20% w/v Na2-2 O3) עבור 24 h. כמו כן לטפל בצלוחיות מזוהמים ועצות פיפטה עם פתרון EMS-inactivating פעלה עבור 24 שעות.
- לשטוף את הזרעים שטופלו EMS תחת הפעלת מי ברז עבור 2 h. להשתלות כל זרע בנפרד לתוך מאמנים שורש המכילים אדמת השתילה.
- לגדל צמחים ב 20-25 ° צ' תחת הזמן אור 16 h.
- הקלטת נתונים על הישרדות הצמח לאחר 15 ימים של השתלת. השתמש במשוואה הבאה כדי לחשב את שיעור ההישרדות עבור כל טיפול:
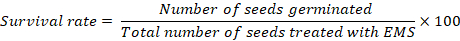
הערה: אם קצב הנביטה נמוך מ-100% בפקדים, יש לחשב שיעורי הישרדות מדויקים בכל הטיפולים לאחר הפחתת מספר הזרעים שנכשלו בלנבוט בפקדים. שיעור הישרדות של 40% – 60% רצוי למוטזיס אפקטיבי. ייתכן שתידרש לבצע סיבוב שני של מינון אופטימיזציה עם ריכוז שונה על פי ההישרדות של הזרעים שטופלו עד השגת שיעור באופן הקטלני הרצוי של 40% – 60%.
2. מוטגנזה ואחזקה של אוכלוסיית מוטציות
- להשרות את האצווה הסופית של לפחות 3,000 זרעים לחילוק באופן שווה (600 זרעים כל אחד) ב 5 1,000 mL מבחנות ה300 מכילות מים מזוקקים משנת mL. נענע עבור 8 h ב 100 סל"ד תחת RT לקבלת שתיית.
הערה: הגודל הסופי של האוכלוסיה הרצויה יהיה תלוי בתדר המוטציה וברמה הפלואידי של ה-גנוטיפ, אך מומלץ להשתמש ב-3,000 לפחות זרעים עבור hexaploids, 4,000 זרעים לטטרדואידים, ויותר מ-7,000 זרעים לדיפלואידים. - במנוע, להכין 1,500 mL של פתרון ריכוז EMS אופטימיזציה מים מזוקקים.
- Decant את המים מתוך מבחנות ולהוסיף 300 ml של ריכוז אופטימלי של פתרון EMS בכל בקבוקון המכיל 600 לשתות זרעי. לנער את מבחנות עבור 16 h ב 75 סל ד ו-RT.
- Decant ה-EMS ולאסוף את הזרעים שטופלו במטלית גבינה. הפוך את פתרון ה-EMS ומיכלי הטיפול באמצעות פתרון EMS-inactivate כפי שנעשה בשלב 1.4.
- לשטוף את הזרעים שטופלו EMS תחת הפעלת מי ברז עבור 2 h. השתלת כל מטופל מידי EMS1 זרע בנפרד לתוך מדריכים השורש.
- לגדל M1 צמחים (נגזר m0 זרעים) ב 20-25 ° צ' תחת הזמן אור 16 h.
הערה: ייתכן שתידרש להפוך את השתילים בשלב שני העלים במשך 6 שבועות ב -4 ° c, אם לסוג ה-גנוטיפ של הריבית יש הרגל צמיחה מסוג חורף. - לאפשר את הצמחים M1 כדי המאביק עצמית, והקציר m2 זרעים בנפרד עבור כל צמח m פורה1 .
הערה: כדי למנוע הזדמנויות לחציית הגבול הפוטנציאלית, יש לכסות את הקוצים של צמחים בעלי1 עם שקי האבקה לפני התזה. - הצמח M2 זרע אחד מכל מפעל1 m כדי למנוע יתירות גנטית.
- לאסוף רקמה מ2 צמחים בשלב שני עלים ב 1.1 mL של שתייסר 96 היטב microtubes. לאסוף סביב 80 מ"מ של רקמת עלה מכל צמח ולהקליט את המזהה של כל מדגם בתוכנית אוסף רקמות.
- להקפיא את רקמת העלה באמצעות ליאופליזר ולאחסן ב-80 ° c.
- שמרו על2 הצמחים בגובה של 20 עד 25 ° c בזמן אור של 16 שעות.
- הקלט נתונים על פנוטיפים של מוטציות של2 הצמחים במרווחי זמן קבועים. הפנוטיפים הצפויים הם לבקנים, כלורנה, דשא לירות, מגוון, פורה באופן חלקי, סטרילי, וכו '.
- לאפשר2 צמחים M להפרות עצמית בוגרת. הקציר בנפרד ולשמור את הזרעים M3 של2 צמחים M (איור 1).
הערה: זרעי M משמשים לאימות הפנוטיפים בלימודי גנטיקה הפוכה. לכן, M יש לקטלג בקפידה ולאחסן בתנאים קריר ויבש. לחילופין, אם יש להגדיל את הזרע בשדה, ניתן לשתול שורות ראש עבור כל צמח M, ו-M הזרעים מכל שורה ניתן לקצור ולשמור בנפרד כמשאב האוכלוסייה לעיבוד לקרקע.
3. Cel-1 שיטת לאפיון גנטי של מוטציות
- חלץ DNA מרקמת העלה של M2 באמצעות ערכת החילוץ dna צמח עם מערכת טיהור dna (ראה טבלת חומרים) בעקבות המלצות היצרן.
- לכמת DNA באמצעות ספקטרוסקופיה ולנרמל את ריכוזי ה-DNA ל -25 ng/μL עם nuclease-מים ללא תשלום 96 בלוקים.
הערה: חשוב לבדוק את איכות ה-DNA על ידי הפעלת אותו על ג'ל, כמו DNA באיכות נמוכה (DNA מרוח) עלול לגרום לתשלילים שווא בדגימות במאגר. - יצירת 4x בריכות DNA ידי שילוב ה-DNA מ 4 96 בלוקים גם לתוך צלחת אחת, תוך שמירה על זהות השורה והעמודה של כל מדגם. הוסף 50 μL של דנ א מכל מדגם בודד לתוך צלחת הבריכה, כך שכל צלחת הבריכה מכילה סך 200 μL של דנ א מארבעה שונים 96 היטב בלוקים.
- קטלוג הזהות של ה-DNA במאגר בפורמט צלחת הבריכה-טור שורה (למשל, בריכה 1 A1 = Box1A1 + Box2A1 + Box3A1 + Box4A1).
- עיצוב הגנום-ספציפיים היסוד [A1] עבור הגן של העניין באמצעות הגנום הספציפי התחל (GSP) עמוד < https://probes.pw.usda.gov/GSP > עם הגדרות ברירת מחדל עבור מינים פוליפואיד. עבור diploids, להשתמש פריימר 3 < http://primer3.ut.ee/> לעיצוב התחל באמצעות הגדרות ברירת המחדל. עצב מספר צבעי יסוד, במידת הצורך, כדי לכסות את כל אזור הקידוד של הגן המעניין.
הערה: האסיפה העדכנית ביותר של IWGSC ניתן להשתמש כדי להשיג רצפים עבור הגן של עניין חיטה באמצעות כלי הפיצוץ URGI < https://wheat-urgi.versailles.inra.fr/Seq-Repository/BLAST >. האורך אמפליקון האופטימלי הוא בטווח של 800 – 1500 bp. Table 1 מראה דוגמאות של שעווה לגנים של הקסאפואיד חיטה. - הפעל את ה-PCR לצבעי היסוד הספציפיים לגנים על דנ א במאגר כדלקמן:
- להוסיף 5 μL של מאגר PCR, 2 μL (כל אחד) של 4 μM קדימה והפוך התחל, 0.1 μL של DNA פולימראז (ראה טבלת חומרים), 5 μl של תבנית dna במאגר, ולאחר מכן להגדיל את עוצמת הקול ל 25 μl באמצעות מים חינם nuclease.
- השתמש בפרופיל מגע למטה כדי להפעיל את תגובת ה-PCR על הציקלור התרמי כדלקמן: 95 ° c עבור 1 דקות, שבעה מחזורים של 95 ° c עבור 1 דקות, 67 ° צ' עד 60 ° c עבור 1 דקות עם טמפרטורה פוחתת 1 ° c לכל מחזור ו-72 ° צלזיוס עבור 2 דקות , ואחריו 30 מחזורים של 95 ° c עבור 1 דקות, 60 ° צ' עבור 1 דקות, 72 ° צ' עבור 2 דקות, ו הארכה הסופית ב 72 ° c עבור 7 דקות.
הערה: מעל פרופיל ה-PCR עובד על תבנית DNA של חיטה עבור רוב העיצוב התחל באמצעות פריימר 3 הגדרות ברירת מחדל. במקרה של הגברה לא ספציפית, יש להחמיר את פרופיל ה-PCR לפני המעבר לשלבים הבאים.
- צור הטרודופלקסים בין דנ א לא תואם על ידי מוצרי PCR מוצרים ב ציקלer תרמית באמצעות פרופיל כדלקמן: 95 ° c עבור 2 דקות, חמישה מחזורים של 95 ° צ' עבור 1 s, 95 ° צ' עד 85 ° c עבור 1 דקות עם טמפרטורה פוחתת של 2 ° צ' למחזור, ו-60 מחזורים של 85 ° c עד 25 ° צ' עם פוחתת של 1 ° c לכל מחזור.
- הוסף 2.5 μL של מוצרים ביתיים Cel-1 endonuclease למוצרי ה-PCR המאפיי והמנדטה עבור 45 דקות ב-45 ° c. לסיים את התגובה Cel-1 על ידי הוספת 2.5 μL של 0.5 M EDTA (pH 8.0).
הערה: Cel-1 endonuclease ניתן לחלץ גבעולי סלרי טרי באמצעות פרוטוקול שבוצעה על ידי עד et al.19 זה חשוב מאוד לבדוק את הפעילות ואת כמות אופטימלית של Cel-1 endonuclease, אשר ניתן נבדק באמצעות מוטציות שאפיינו בעבר או ערכת זיהוי מוטציה זמינה מסחרית. - הפעל את מוצרים מטופלים Cel-1 על 3.0% agarose ג'ל ב 100 V עבור 2.5 h. הבארות המכילות להקות קטנות וייחודיות, בנוסף ללהקות באורך מלא, יכילו את דגימת ה-DNA של המוטציות.
- . בריכות המוטציות הDenconvolute
- בצע את השלבים 3.6, 3.7, 3.8, ו 3.9 עבור בודד M2 דגימות DNA המהווים את בריכות המוטציות שזוהו בשלב 3.9.
- כדי לקבוע את הזיניות של מוטציות, להריץ שני PCR (כמתואר בשלב 3.6) עבור היחיד M2 dna, שבו התגובה הראשונה מכילה 2.5 μl של m2 Dna ו 2.5 μl של ה-dna סוג פראי ואת התגובה השנייה מכיל רק 5 μl של M2 dna. אם המוטציה היא heterozygous, להקה נוספת ביקתה יהיה נוכח בשתי התגובות. מצד שני, להקות ביקקות נוספות ימצאו רק בתגובה הראשונה אם המוטציה מhomozygous.
- כדי לזהות את טבעו של המוטציה, רצף את מוצרי ה-PCR של המוטציות המאושר באמצעות פלטפורמת רצף שימוש בשיטת Sanger בעקבות הוראות היצרן.
4. חישוב תדר מוטציה
הערה: תדירות מוטציה של אוכלוסיית לעיבוד קרקע מתייחסת למרחק הפיזי הממוצע שבו מוטציה אחת מתרחשת אצל האנשים של אוכלוסיה זו. לדוגמה, תדירות מוטציה של 1/35 kb באוכלוסיית לעיבוד קרקע פירושו שאדם ממוצע של אוכלוסיה זו בעל מוטציה אחת לכל 35 kb בגנום.
- כדי לקבוע את תדירות המוטציה של אוכלוסיית לעיבוד קרקע, חשב את המספר הכולל של בסיסים שיוקרנו.
- כדי לחשב את המספר הכולל של בסיסים שיוקרנו, הכפל את גודל המוצר של ה-PCR לפי המספר הכולל של אנשים שיוקרנו.
- חלק את המספר הכולל של בסיסים הוקרן על ידי מספר מוטציות ייחודיות שנצפו באמצעות המשוואה הבאה, אשר תניב את האזור הפיזי בעל 1 מוטציה באוכלוסיית לעיבוד הקרקע נתון:
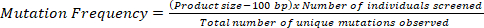
הערה: כדי להסביר את המגבלה ברזולוציה של 50 bp בשני הקצוות בהתבסס על פלטפורמה המבוססת על ג'ל agarose, להחסיר 100 bp מגודל המוצר בחישוב.
Access restricted. Please log in or start a trial to view this content.
תוצאות
איור 2 מראה את עקומת המינון של לחם הקסואיד לזנים חיטה ג ' וליד, דיפלואידי חיטה triticum מmonococcum6, ותורם הגנום של החיטה אייילאופים tauschii7. מינונים EMS עבור הרצוי 50% שיעורי ההישרדות היו כ 0.25%, 0.6% ו 0.7% עבור T. monococcum, Ae. tauschii, ו- <...
Access restricted. Please log in or start a trial to view this content.
Discussion
לעיבוד קרקע הוא כלי גנטיקה הפוכה בעל ערך רב עבור אימות גנים, במיוחד עבור דגנים קטנים שבהם גישות מבוססות שינוי יש צווארי בקבוק רציני11. פיתוח אוכלוסיה מוטדית עם תדר מוטציה גבוהה הוא אחד השלבים הקריטיים בניהול לימודי גנומיקה תפקודית. הצעד החשוב ביותר בפיתוח אוכלוסיית לעיבוד קרק...
Access restricted. Please log in or start a trial to view this content.
Disclosures
מחברים לא מצהירים על אינטרסים פיננסיים מתחרים.
Acknowledgements
עבודה זו נתמכת על ידי המכון הלאומי של משרד החקלאות של מזון וחקלאות, הצוהר פרויקט 1016879 ו מרילנד תחנת הניסוי החקלאי דרך MAES גרנט מס ' 2956952.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 96 well 1.1 ml microtubes in microracks | National Scientific | TN0946-08R | For collecting leaf tissues |
| Agarose I biotechnology grade | VWR | 0710-500G | |
| Biosprint 96 DNA Plant Kit | Qiagen | 941558 | Kit for DNA extraction |
| Cel-1 endonuclease | Extracted as described by Till et al 2006 | Single strand specific endonuclease | |
| Centrifuge 5430 R | Eppendorf | ||
| Ethyl methanesulfonate | Sigma Aldrich | M-0880-25G | EMS, Chemical mutagen |
| Freeze Dry/Shell freeze system | Labconco | For lyophilization of leaf tissue | |
| Kingfisher Flex purification system | Thermo fisher scientific | 5400610 | High throughput DNA extraction robot |
| My Taq DNA Polymerase | Bioline | BIO-21107 | |
| Nuclease free water | Sigma aldrich | W4502-1L | |
| NuGenius gel imaging system | Syngene | ||
| Orbit Environ-shaker | Lab-line | ||
| SPECTROstar Nano | BMG LABTECH | Nano drop for DNA quantification | |
| T100 Thermal cycler | BIO-RAD | 1861096 |
References
- McCallum, C. M., Comai, L., Greene, E. A., Henikoff, S. Targeted screening for induced mutations. Nature Biotechnology. 18 (4), 455-457 (2000).
- Bentley, A., MacLennan, B., Calvo, J., Dearolf, C. R. Targeted Recovery of Mutations in Drosophila. Genetics. 156 (3), 1169-1173 (2000).
- Tsai, H., et al. Discovery of Rare Mutations in Populations: TILLING by Sequencing. Plant Physiology. 156 (3), 1257-1268 (2011).
- Caldwell, D. G., et al. A structured mutant population for forward and reverse genetics in Barley (Hordeum vulgare L.). The Plant Journal. 40 (1), 143-150 (2004).
- Hazard, B., et al. Induced Mutations in the Starch Branching Enzyme II ( SBEII ) Genes Increase Amylose and Resistant Starch Content in Durum Wheat. Crop Science. 52 (4), 1754-1766 (2012).
- Rawat, N., et al. A diploid wheat TILLING resource for wheat functional genomics. BMC Plant Biology. 12, 205(2012).
- Rawat, N., et al. TILL-D: An Aegilops tauschii TILLING Resource for Wheat Improvement. Frontiers in Plant Science. 9, (2018).
- Rawat, N., et al. Wheat Fhb1 encodes a chimeric lectin with agglutinin domains and a pore-forming toxin-like domain conferring resistance to Fusarium head blight. Nature Genetics. 48 (12), 1576-1580 (2016).
- Kippes, N., Chen, A., Zhang, X., Lukaszewski, A. J., Dubcovsky, J. Development and characterization of a spring hexaploid wheat line with no functional VRN2 genes. Theoretical and Applied Genetics. 129 (7), 1417-1428 (2016).
- Greene, E. A., et al. Spectrum of Chemically Induced Mutations From a Large-Scale Reverse-Genetic Screen in Arabidopsis. Genetics. 164 (2), 731-740 (2003).
- Harwood, W. A. Advances and remaining challenges in the transformation of barley and wheat. Journal of Experimental Botany. 63 (5), 1791-1798 (2012).
- Henikoff, S., Comai, L. Single-Nucleotide Mutations for Plant Functional Genomics. Annual Review of Plant Biology. 54 (1), 375-401 (2003).
- Uauy, C., et al. A modified TILLING approach to detect induced mutations in tetraploid and hexaploid wheat. BMC Plant Biology. 9 (1), 115(2009).
- Uauy, C., Wulff, B. B. H., Dubcovsky, J. Combining Traditional Mutagenesis with New High-Throughput Sequencing and Genome Editing to Reveal Hidden Variation in Polyploid Wheat. Annual Review of Genetics. 51 (1), 435-454 (2017).
- Li, G., et al. The Sequences of 1504 Mutants in the Model Rice Variety Kitaake Facilitate Rapid Functional Genomic Studies. The Plant Cell. 29 (6), 1218-1231 (2017).
- Jiao, Y., et al. A Sorghum Mutant Resource as an Efficient Platform for Gene Discovery in Grasses. The Plant Cell. 28 (7), 1551-1562 (2016).
- Krasileva, K. V., et al. Uncovering hidden variation in polyploid wheat. Proceedings of the National Academy of Sciences. , 201619268(2017).
- Dong, C., Dalton-Morgan, J., Vincent, K., Sharp, P. A Modified TILLING Method for Wheat Breeding. The Plant Genome. 2 (1), 39-47 (2009).
- Till, B. J., Zerr, T., Comai, L., Henikoff, S. A protocol for TILLING and Ecotilling in plants and animals. Nature Protocols. 1 (5), 2465-2477 (2006).
- Wu, J. -L., et al. Chemical- and Irradiation-induced Mutants of Indica Rice IR64 for Forward and Reverse Genetics. Plant Molecular Biology. 59 (1), 85-97 (2005).
- Feldman, M., Levy, A. A. Genome Evolution Due to Allopolyploidization in Wheat. Genetics. 192 (3), 763-774 (2012).
- Comai, L. The advantages and disadvantages of being polyploid. Nature Reviews Genetics. 6 (11), 836-846 (2005).
- Guo, H., et al. Development of a High-Efficient Mutation Resource with Phenotypic Variation in Hexaploid Winter Wheat and Identification of Novel Alleles in the TaAGP.L-B1 Gene. Frontiers in Plant Science. 8, (2017).
- Rakszegi, M., et al. Diversity of agronomic and morphological traits in a mutant population of bread wheat studied in the Healthgrain program. Euphytica. 174 (3), 409-421 (2010).
- Tsai, H., Ngo, K., Lieberman, M., Missirian, V., Comai, L. Tilling by Sequencing. Plant Functional Genomics: Methods and Protocols. , 359-380 (2015).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved