需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
加载编码RNA核酸适体和核糖开关的DNA质粒的多功能丝基微胶囊的制备
摘要
该协议描述了稳健且生物相容的富含DNA的微胶囊的形成,作为能够跟踪多种配体的多重体 外 生物传感器。
摘要
我们介绍了一种通过牺牲球形核心上的逐层(LbL)组装方法制备富含DNA的丝素蛋白微胶囊的方案。在吸附质层和DNA质粒后,在单个蚕丝层的急性脱水过程中诱导蚕丝二级结构中的β片,促进了健壮微胶囊的形成。因此,分层是通过多次氢键和疏水相互作用发生的。在吸附多层壳后,核壳结构可以用金纳米颗粒(AuNPs)和/或抗体(IgG)进一步功能化,用于遥感和/或靶向递送。在二氧化硅核心上顺序沉积关键大分子的过程中调整几个关键参数,例如聚合物引物的存在、DNA 和丝蛋白的浓度以及许多吸附层,从而产生具有可变渗透性和 DNA 负载量的生物相容性、富含 DNA 的微胶囊。在二氧化硅核心溶解后,该方案证明了空心和坚固的微胶囊的形成,其中DNA质粒固定在胶囊膜的内表面。在DNA质粒和外部环境之间建立选择性渗透的生物相容性膜,在长期储存期间保存DNA,并在改善空间受限质粒的输出响应中发挥重要作用。在 体外 转录和翻译反应(无细胞系统)期间测试DNA模板的活性及其可及性。编码RNA发光适配体和核糖开关的DNA质粒被相应的分析物成功激活,如在壳膜中定位荧光标记的RNA转录物或GFPa1蛋白时可视化的那样。
引言
合成生物学领域通过利用微生物进化的自然机制来监测其环境和潜在威胁,为发展传感能力提供了独特的机会。重要的是,这些传感机制通常与保护这些微生物免受有害暴露的反应有关,调节基因表达以减轻负面影响或防止摄入有毒物质。人们已经付出了巨大的努力来设计这些微生物,以利用这些自然反应来创建全细胞传感器,但重新定向它们以识别新靶标和/或产生可测量的信号,该信号可用于定量目的(通常是荧光)1,2。目前,由于整个细胞或其某些遗传物质的泄漏,即使封装在聚合物基质中,对使用转基因微生物(GMO)的担忧,特别是在环境或人体中释放时,也表明需要利用这些传感方法的替代方法3。
利用基于微生物的传感的好处而不用担心转基因生物部署的一种强大方法是使用体外转录/翻译(IVTT)系统。从实践的角度来看,IVTT系统由含有大部分处于活性状态的细胞成分的混合物组成,这些成分是通过不同方式从细胞中"提取"的,包括超声处理,珠子跳动或其他方式4。该过程的最终产品是已经优化为执行转录和翻译的生化反应混合物,可用于以"开放容器"格式测试不同的传感器,而不受与使用整个细胞相关的限制(膜扩散、转化效率、细胞毒性等)。重要的是,可以定量添加不同的传感器组件,并通过不同的光学和光谱技术研究它们的影响,正如我们所展示的5。已经注意到,IVTT系统的性能可能不一致;然而,最近的研究已经显示了标准化其制备和表征的方法,这对于研究它们在传感器设计中的性能有很大帮助6。最近,已经证明了许多IVTT系统通过冻干纸基质中的组分来创建纸质测定的例子,包括检测重金属离子,药物,群体传感元件等7,8,9。基于IVTT的传感器的一个令人兴奋的应用领域是它们在不同类型的环境中的传感应用,包括土壤、水和人体。为了将这些IVTT系统部署到这些具有挑战性的环境中,需要实施一种封装方法来包含IVTT组件并保护它们免受降解。
IVTT系统最常见的封装方法包括使用脂质胶囊,胶束,聚合物体和其他紧密封闭的微容器10,11,12。这种方法的一个缺点是需要结合被动或主动机制来将材料运入和运出容器,以允许与外部环境通信并提供传感功能。为了克服其中的一些问题,本文报告了一种方法,该方法提供了一种简单而有效的方法来封装要在IVTT系统中表达的不同传感器设计的编码材料。该方法基于在目标质粒存在下使用生物聚合物的逐层(LbL)沉积来创建具有高孔隙率的空心微胶囊,这使得受保护的遗传物质与所选IVTT的不同组分相互作用。该研究表明,封装的质粒在该聚合物基质中激活时可以指导转录和翻译,如质粒编码的适配体和核糖开关对其相应靶标的响应所示。此外,这种LbL包被可在没有任何特殊储存条件的情况下保护质粒数月。
研究方案
1.质粒载体的构建。
- 通过扩增茶碱核糖开关 (ThyRS) 的编码序列与来自 pJ201:23976-RS-GFPa1 载体(由 DNA2.0 设计和创建)的 GFPa1 偶联并插入大 肠杆菌 表达载体 pSAL13,构建质粒载体 (pSALv-RS-GFPa1, 3.4 kb)。使用正向 (5'-CGTGGTACCGGTGGTGATGATACCCATCGTCTTGATG-3') 和反向 (5'-CGTGCTCAGCTTAAGCCAGCTCGTAG-3') 引物扩增 ThyRS 与 GFPa1 偶联的编码序列,并根据制造商的方案14 使用 DNA 聚合酶在 50 μL 体积中进行 PCR 反应。
- 从 0.5 g 琼脂糖、50 mL TAE 缓冲液(40 mM 乙酸三酯、1 mM EDTA、pH 8.0)和 3 μL DNA 染色剂制备 1% 琼脂糖凝胶。
- 将 5 μL 等分试样的 PCR 扩增产物与 5 μL 无 RNase/DNase 的水和 2 μL 6x 凝胶上样染料混合,并通过琼脂糖凝胶电泳进行分析。加载DNA分子量标准(0.1-10.0 kb)作为参考。在120 V下运行凝胶,直到染料线几乎到达凝胶底部。
- 使用紫外透射仪成像系统可视化 DNA 片段,以检查 DNA15 的正确大小。
- 根据制造商的方案16使用PCR纯化试剂盒纯化PCR产物。
- 用 KpnI 和 BlpI 限制性内切酶在 15 μL 反应中消化 PCR 产物和 pSAL 表达载体,该反应含有 10 μL PCR 产物或质粒载体(浓度 20-50 ng/μL)、1.5 μL 10x 酶缓冲液、1 μL 每种酶和 1.5 μL 无 RNase/DNase 的水,在 37 °C 下消解 2 小时。
- 向反应混合物中加入 3 μL 6x 凝胶上样染料,并按照步骤 1.3-1.5 所述在 1% 琼脂糖凝胶上分离消化的片段。
- 根据制造商的方案16使用凝胶提取试剂盒纯化DNA片段。
- 使用 T4 DNA 连接酶和补充的连接酶缓冲液,将消化的 PCR 产物连接到酶解的线性化质粒载体 pSAL 中,在 10 μL 反应中,该反应含有 3-20 fmol 的消化载体、9-60 fmol 的消化 PCR 产物、2 μL 连接酶缓冲液、1 μL(1 单位)T4 DNA 连接酶和不含 DNase/RNase 的水。将连接反应在25°C孵育3小时。
注意:确保反应混合物中的总DNA含量为0.01-0.1μg。 - 根据制造商的方案17,用10ng的连接反应混合物转化大肠杆菌DH5α感受态细胞。
- 在补充有氨苄青霉素(100μg/ mL)的LB-琼脂平板上在37°C下培养转化的细胞过夜。
- 从平板中挑选 3-4 个细菌菌落,并将每个菌落无菌转移到补充有氨苄青霉素 (100 μg/mL) 的 5 mL LB 培养基中。在37°C下以225rpm的振荡培养箱中培养过夜。
- 通过在室温下以11× g 离心3分钟来沉淀过夜培养物。
- 使用纯化试剂盒根据制造商的方案16纯化质粒。
- 通过DNA测序验证纯化质粒的序列。质粒图谱和所得构建体的序列(pSALv-RS-GFPa1)如图 1所示。
2.大规模DNA纯化。
- 根据制造商的方案将质粒载体pSALv-RS-GFPa1(3.4kb)(编码茶碱核糖开关与GFPa1报告基因偶联)或pET28c-F30-2x西兰花(5.4 kb)(编码西兰花适体)转化为 大肠杆菌 DH5α感受态细胞17。
- 对于用pSALv-RS-GFPa1转化的细胞或卡那霉素(50μg/ mL)转化的细胞,在补充有氨苄青霉素(100μg/ mL)的LB-琼脂平板上生长转化的细胞过夜,用于用pET28c-F30-2x西兰花转化的细胞。
- 从平板中挑选 3-4 个细菌菌落,并将每个菌落无菌转移到补充有适当抗生素(100 μg/mL 氨苄西林或 50 μg/mL 卡那霉素)的 LB 培养基中。在37°C下以225rpm的振荡培养箱中培养过夜。
- 使用3mL过夜培养物接种到补充有适当抗生素(100μg/ mL氨苄青霉素或50μg/ mL卡那霉素)的150mLLB中,并在37°C下以225rpm的振荡培养箱中培养培养培养过夜。
- 通过在4°C下以≥3400× g 离心10分钟来沉淀细胞。
- 使用纯化试剂盒根据制造商的方案16纯化质粒。
- 用 0.5 mL 纯 DNA 酶/无 RNase 的水洗脱 DNA。测量 DNA 浓度并制备 1 mL DNA 储备溶液 (100 ng/μL)。将带有DNA的试管储存在4°C直至进一步使用。
3.丝素蛋白的提取和初始材料的制备。
- 根据其他地方详细描述的程序,从 Bombyx mori 蚕茧中制备重组丝素蛋白(SF)蛋白的水溶液,以占Silk-LiBr溶液的10%。
- 确定SF水溶液的最终浓度。将0.5mL丝溶液移液到60mm培养皿中,使其在60°C下干燥,并测量干燥丝膜的重量。将干重除以 0.5 mL 以计算每体积百分比的重量。
- 通过血清移液管缓慢加水,用不含 DNase/RNase 的蒸馏水稀释浓缩丝溶液,以获得 1 mg/mL 的终浓度。将溶液储存在4°C以备将来使用。
- 使用抗体标记试剂盒制备荧光标记的丝素蛋白。根据制造商的方案,使用 1 mL 的 2 mg/mL 丝素蛋白溶液将蛋白质的 N 末端 α-氨基基团与 NHS 酯激活的衍生染料偶联19。
- 制备 50 mL 浓度为 6 mg/mL 的聚乙烯亚胺 (PEI) 水溶液,用 HCl (1 M) 将 pH 值调节至 4。通过无菌0.2μm膜过滤溶液。可以在环境条件下储存数月。
- 准备 SiO2 内核。将 300 μL SiO 2 颗粒移液到2 mL 微量离心管中。用 1 mL 无 DNase/RNase 的水洗涤微粒两次,以 0.2 x g 离心 1 分钟。
4. 对质素层、DNA 质粒和丝层进行逐层沉积。
- 要将PEI主层沉积到SiO2 微粒上,将1mL PEI溶液添加到步骤3.6的离心沉淀中,并在环境条件下在温度混合器上以800rpm搅拌混合物15分钟。用 1 mL 不含 DNase/RNase 的去离子水以 0.2 x g 离心 1 分钟洗涤颗粒四次。
- 为了进行DNA层的沉积,将步骤2.7中的1mLDNA质粒水溶液添加到PEI引发的微粒中,并在4°C的温度混合器上以800rpm轻轻搅拌混合物15分钟。要制备具有不同 DNA 负载的微胶囊,请使用不含 DNase/RNase 的蒸馏水将 DNA 质粒的浓度调节为 50-200 ng/μL,并使用 1 mL 这些溶液沉积 DNA。通过以0.2× g 离心1分钟来收集微粒。
- 将编码茶碱核糖开关与GFPa1偶联的DNA质粒的试管标记为ThyRS-GFPa1,将编码西兰花适配体的DNA质粒标记为BrocApt。
注意:将带有DNA的微量离心管放在冰上。 - 小心地除去上清液,并用1mL无DNase / RNase的蒸馏水洗涤微粒四次,每次以0.2× g 离心1分钟后弃去上清液。除非另有说明,否则在室温(RT)下进行所有实验。
- 为了进行丝素蛋白层的沉积,将步骤3.3中的1mL重构SF水溶液加入DNA吸附的微粒中,在10°C的温度混合器上以750rpm轻轻涡旋和搅拌混合物15分钟。通过在4°C下以0.2× g 离心1分钟来收集微粒,除去上清液,然后用1mL无DNase / RNase的蒸馏水洗涤一次。重复离心并弃去上清液。
注意:在实验过程中,将真丝溶液保持在冰上以避免温度引起的凝胶化。 - 逐渐用甲醇处理颗粒,以诱导丝蛋白结构中β片的形成。首先,加入 0.5 mL 的 DNase/RNase 蒸馏水,涡旋微量离心管,然后加入 0.5 mL 的 100% 甲醇。在10°C下轻轻摇动热混合器上的颗粒5分钟。通过以0.2× g 离心1分钟来收集颗粒。除去上清液。
- 用甲醇处理颗粒以促进β片的形成,并确保丝层的强烈物理吸附。加入 1 mL 100% 甲醇。在10°C下以750rpm轻轻摇动热混合器上的颗粒10分钟。
- 通过在4°C下以0.2× g 离心1分钟来收集颗粒,每次用1mL无DNase / RNAase的蒸馏水洗涤两次,丢弃上清液并在下一次离心前轻轻涡旋。
- 重复步骤4.5-4.8 20次,得到丝绸多层核壳结构。对于最后一个沉积步骤,使用步骤 3.4 中的荧光标记丝绸(Silk-DyLight550,1 mL)。
- 执行最后的洗涤步骤,并在环境条件下将微粒保持在 1 mL 不含 DNase/RNase 的蒸馏水中。
注意:为避免在丝绸层沉积过程中颗粒聚集,请对颗粒悬浮液进行目视检查,并使用 1 mL 移液器吸头上下移液以促进均匀的颗粒分布。 - 使用公式1计算封装在每个微胶囊中的DNA质粒拷贝数, NDNA :
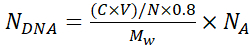 (1)
(1)
其中 N = 6.769 × 1011 - 用于封装的 SiO2 内核的数量。从已知浓度的二氧化硅颗粒的标准曲线计算它,使用连续稀释和吸收A 320在λ =320nm 处;
C-用于吸附的DNA的初始浓度
V-用于吸附的DNA体积
0.8- 核上的DNA吸附效率
Mw- DNA质粒的分子量
NA- 阿伏伽德罗数 (6.022 × 1023)
5.溶解芯,得到蚕丝微胶囊。
- 用蒸馏水稀释储备溶液(48%),制备8%氢氟酸(HF)溶液,pH 5.5。获取 50 mL 离心管。小心移液 5 mL HF 并加入 25 mL 蒸馏水以获得 8% HF 溶液。
注意:HF是一种高腐蚀性酸,可能会对组织造成严重烧伤。在处理和使用HF进行实验时必须格外小心。遵守组织制定的标准操作程序(SOP),以正确使用和处理HF,以避免不良泄漏事故。请勿使用玻璃容器稀释HF酸。使用化学罩执行协议的此步骤。 - 通过将 1.5 mL 的 8% HF 溶液添加到步骤 4.10 中的颗粒核壳微粒中来溶解 SiO2 核心。轻轻涡旋,让核心在环境条件下溶解过夜,并以 450 rpm 的速度轻轻摇动。
注意:为避免HF溢出,请使用接枝胶带密封微量离心管。使用化学罩执行协议的此步骤。 - 准备一个装满 2 L 去离子水的 2 L 玻璃烧杯。将微胶囊溶液转移到透析装置(50kDa MWCO)中,并在接下来的3天内每3小时重复更换一次水,对去离子水进行透析。
注意:在前三次换水期间收集上清液,并根据危险废物的既定方案丢弃溶液。 - 使用 1 mL 移液器将悬浮液从透析设备转移到新的 2 mL 微量离心管中以收集微胶囊。
注意:将微胶囊的水溶液在环境条件下储存数年。
6.使用共聚焦激光扫描显微镜(CLSM)对丝素蛋白微胶囊进行成像。
- 使用 DNA 染料进行 DNA 染色。
- 将 300 μL 空心丝素蛋白微胶囊转移到新鲜的 1 mL 微量离心管中。加入 500 μL 不含 RNase/DNase 的蒸馏水。
- 加入 5 μL 的 DNA 染色染料,短暂涡旋,并在室温下避光孵育 2 小时。
- 通过每次在4°C下以0.1× g 离心20分钟来执行四个洗涤步骤,小心地除去400μL上清液并用400μL无RNase / DNase的蒸馏水补充。
- 使用100倍油浸物镜(NA 1.49)在配备三个主要激光器(405 nm,488 nm,561 nm)的倒置共聚焦系统上对丝胶囊进行成像。将 100 μL 胶囊样品转移到 8 孔室载玻片的单孔中,让胶囊在成像前沉淀 20-30 分钟。
注意:染料对光漂白非常敏感。用铝箔覆盖载玻片来保护样品。
7.使用分子量截止(MWCO)法估计空心微胶囊的渗透性。
- 制备不同Mw(4 kDa、20 kDa、40 kDa、70 kDa、150 kDa、250 kDa、500 kDa和2 MDa)的FITC标记葡聚糖荧光团溶液(20 μM,diH2O)各2 mL。
- 将100μL胶囊悬浮液移液到腔室载玻片的单个孔中。分别分析每个微胶囊设计(PEI浓度,DNA质粒上样数,丝素蛋白浓度和层数)。
- 向每个孔中加入 300 μL 特定荧光团溶液,从最低的 Mw 到最高,因此每个孔将对应于特定的荧光团溶液。通过上下移液混合,让混合物在室温下孵育1小时,直到荧光团溶液的扩散达到平衡。
- 将载玻片转移到共聚焦激光扫描显微镜(CLSM)上,并在激发λ = 488nm下使用100倍油浸物镜对每个孔成像。
- 通过调整焦平面来识别感兴趣的区域,以确保胶囊以最大直径的圆的形式出现。这通常发生在观察靠近井底的样品时,当胶囊由于重力而沉淀时。
- 通过在XY方向上移动载玻片来收集微胶囊样品的几张图像。捕获图像,每个样品最多可捕获 100-150 个胶囊。
- 使用 ImageJ 软件通过比较胶囊内外的荧光强度来分析胶囊膜在每个 Mw 荧光团溶液中的渗透性。为此,以圆圈的形式绘制一个感兴趣区域(ROI)以勾勒胶囊的周长,然后单击 分析/ 测量以测量内部的荧光强度。将数据制成表格到电子表格中。对每个微胶囊执行此操作,总共200-300个胶囊。
- 通过概述ROI并测量远离胶囊的强度,以相同的方式评估外部荧光强度。执行 3-5 次测量以进行统计分析。
- 为了进行统计分析,使用配对 t检验(p < 0.05)比较胶囊内外的荧光强度。
- 使用换算 表2 根据可变 Mw的FITC-葡聚糖的流体动力学半径估算微胶囊的渗透率。
8.合成茶碱核糖开关在丝微胶囊中的 体外 活化
- 准备 1 mL 茶碱储备溶液 (100 mM,DMSO)。通过在冰上解冻组分40分钟来制备用于环状DNA的大 肠杆菌 S30提取物系统。
- 获得 0.5 mL 无 DNase/RNase 的微量离心管。进行 体外 转录/翻译反应,按以下顺序(50 μL 总体积)将无细胞组分与微胶囊样品混合:不含氨基酸的 S30 预混料(20 μL);S30提取物,圆形(15μL);完全氨基酸混合物(5μL);含有来自步骤4.10(9μL)的ThyRS-GFPa1质粒的空心微胶囊;和茶碱,100 mM DMSO(1 μL)。
注意:添加所有组分后,短暂涡旋管并在0.2 × g 的短暂离心中收集样品几秒钟。 - 将管在30°C孵育4小时,并使用λ = 488nm的激发和GFP / FITC滤光片(510nm±20nm)的发射检查读板器上的荧光。
- 使用 488 nm 和 561 nm 激光器在任何 LCSM 系统上对胶囊进行成像。使用100倍油浸物镜和8孔腔载玻片获得最佳质量的图像。
9. 蚕丝微胶囊中西兰花适配体的 体外 活化
- 准备 1 mL DFHBI-1T 染料储备溶液(30 μM,diH2O)。通过在冰上解冻组分40分钟来制备PURE(使用重组元件的蛋白质合成)无细胞系统反应试剂盒。
- 获得 0.5 mL 无 DNase/RNase 的微量离心管。通过将无细胞反应组分与微胶囊样品按以下顺序(50 μL 总体积)混合来进行 体外 转录反应:溶液 A (20 μL);溶液B(15μL);含有步骤4.10(14μL)的BrocApt质粒的空心微胶囊;和 DFHBI-1T 染料 (1 μL)。
注意:添加所有组分后,短暂涡旋管并在0.2 × g 的短暂离心中收集样品几秒钟。 - 将管在37°C孵育6小时,并使用λex = 470nm的激发和λem = 510nm±20nm的发射在读板器上检查荧光。
- 使用 488 nm 和 561 nm 激光器在任何 LCSM 系统上对胶囊进行成像。使用100倍油浸物镜和8孔盖玻片腔室载玻片获得最佳质量的图像。
结果
在这里,该研究解决了编码不同传感器设计(两种类型的RNA调控转录/翻译元件)的DNA模板在封装在丝蛋白胶囊中的功能。通过模板化逐层(LbL)组装制备关键组分的微胶囊:素数层,编码传感器设计的DNA质粒和丝素蛋白生物聚合物(图2)。以分层方式沉积大分子允许根据吸收层之间的分子间和内相互作用与壳的厚度来控制胶囊膜的渗透性。该系统的可调通透性提供了控制基?...
讨论
可以按照该协议制备装有各种类型的DNA编码传感器设计的选择性渗透水凝胶微胶囊。LbL方法的一个显着特点是能够在自下而上的组装过程中定制微胶囊的复杂性,这通常从牺牲模板上分子物质的吸附开始。通过仔细调整初始组分的浓度、pH条件和层数,可以制备具有不同DNA上样参数、功能和可调通透性的微胶囊23。为了增强胶囊的多功能性,可以使用AuNPs和IgG实现壳表面的进一步?...
披露声明
此处提出的观点和意见是作者的观点和意见,不一定代表国防部或其组件的观点
致谢
这项工作得到了空军科学研究办公室的LRIR 16RH3003J资助,以及美国国防部副部长办公室负责研究和工程的军事环境应用研究促进科技优先事项(ARAP)计划的支持。
ThyRS 的质粒载体序列 (pSALv-RS-GFPa1, 3.4 kb) 由 J. Gallivan 博士慷慨提供。 来自Bombyx mori 的蚕茧由马萨诸塞州塔夫茨大学的D.L. Kaplan博士慷慨捐赠。
材料
| Name | Company | Catalog Number | Comments |
| (Z)-4-(3,5-difluoro-4-hydroxybenzylidene)-2-methyl-1-(2,2,2-trifluoroethyl)-1H-imidazol-5(4 H)-one (DFHBI-1T) | Lucerna | DFHBI-1T | |
| 5x T4 DNA Ligase Buffer | ThermoFisher Scientific | 46300-018 | |
| 6x Blue Gel Loading Dye | New England BioLabs | B7021S | |
| 96-well plates, black circular | Corning | 3601 | |
| Agarose | Sigma-Aldrich | A9539 | BioReagent, for molecular biology, low EEO |
| Ampicillin sodium salt | Sigma-Aldrich | A0166 | powder or crystals, BioReagent, suitable for cell culture |
| BlpI restriction enzymes | New England BioLabs | R0585S | |
| Corning Disposable Vacuum Filter/Storage Systems | FisherScientific | 09-761-1 | |
| Dimethyl sulfoxide, DMSO | Sigma-Aldrich | 472301 | ACS reagent, ≥99.9% |
| DNA Plasmid, pET28c-F30-2x Broccoli (5.4 kb), BrocApt. | Addgene | Plasmid #66788 | |
| DyLightTM550 Antibody Labeling kit (Invitrogen) | ThermoFisher Scientific | 84530 | |
| E. coli S30 extract system for circular DNA | Promega | L1020 | |
| Falcon Conical centrifuge tubes, 15 mL | FisherScientific | 14-959-53A | |
| Falcon Conical centrifuge tubes, 50 mL | 14-432-22 | ||
| Fisherbrand Microcentrifuge tubes, 1.5 mL | FisherScientific | 05-408-129 | |
| Hydrofluoric acid, HF | Sigma-Aldrich | 695068 | ACS reagent, 48% |
| Kanamycin sulfate | Sigma-Aldrich | 60615 | mixture of Kanamycin A (main component) and Kanamycin B and C |
| KpnI restriction enzymes | New England BioLabs | R0142S | |
| LB agar plate supplemented with 100 µg/mL ampicillin | Sigma-Aldrich | L5667 | pre-poured agar plates with 100 µg/mL ampicillin |
| LB agar plate supplemented with 50 µg/mL kanamycin | Sigma-Aldrich | L0543 | pre-poured agar plates with 50 µg/mL kanamycin |
| LB broth (Lennox grade) | Sigma-Aldrich | L3022 | |
| Lithium bromide, LiBr | Sigma-Aldrich | 213225 | ReagentPlus, ≥99% |
| Max Efficiency DH5-α competent E. coli strain | ThermoFisher Scientific | 18258012 | |
| Methanol | MilliporeSigma | 322415 | anhydrous, 99.8% |
| MilliQ-water | EMD MilliPore | Milli-Q Reference Water Purification System | |
| MinElute PCR Purification Kit | Qiagen | 28004 | |
| N-(3-Dimethylaminopropyl)-N′-ethylcarbodiimide hydrochloride, EDC | Sigma-Aldrich | E1769 | |
| PBS (phosphate buffered saline) | ThermoFisher Scientific | 10010023 | 1x PBS, pH 7.4 |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530S | |
| Polyethylenimine, branched | Sigma-Aldrich | 408727 | average Mw ~25,000 |
| PURExpress In Vitro Protein Synthesis Kit | New England BioLabs | E6800S | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | |
| Quick-Load 2-Log DNA Ladder (0.1-10.0 kb) | New England BioLabs | N0469S | |
| SiO? silica microspheres, 4.0 µm | Polysciences, Inc. | 24331-15 | 10% aqueous solution |
| Slide-A-Lyzer G2 Dialysis Cassettes, 3.5K MWCO, 15 mL | ThermoFisher Scientific | 87724 | |
| Sodium carbonate, Na?CO? | Sigma-Aldrich | 222321 | ACS reagent, anhydrous, ≥99.5%, powder |
| Spectrum Spectra/Por Float-A-Lyzer G2 Dialysis Devices | FisherScientific | 08-607-008 | Spectrum G235058 |
| SYBR Safe DNA gel stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase (5 U/µL) | ThermoFisher Scientific | EL0011 | |
| Theophylline | Sigma-Aldrich | T1633 | anhydrous, ≥99%, powder |
| Tris Acetate-EDTA buffer (TAE buffer) | Sigma-Aldrich | T6025 | Contains 40 mM Tris-acetate and 1 mM EDTA, pH 8.3. |
| UltraPure DNase/RNase-Free Distilled Water | FisherScientific | 10-977-023 | |
| ZymoPURE II Plasmid MaxiPrep kit | ZymoResearch | D4202 |
参考文献
- Slomovic, S., Pardee, K., Collins, J. J. Synthetic biology devices for in vitro and in vivo diagnostics. Proceedings of the National Academy of Sciences of the United States of America. 112 (47), 14429-14435 (2015).
- Harbaugh, S. V., Goodson, M. S., Dillon, K., Zabarnick, S., Kelley-Loughnane, N. Riboswitch-based reversible dual-color sensor. ACS Synthetic Biology. 6 (5), 766-781 (2017).
- König, H., Frank, D., Heil, R., Coenen, C. Synthetic genomics and synthetic biology applications between hopes and concerns. Current Genomics. 14 (1), 11-24 (2013).
- Silverman, A. D., Karim, A. S., Jewett, M. C. Cell-free gene expression: An expanded repertoire of applications. Nature Reviews Genetics. 21, 151-170 (2020).
- Chushak, Y., et al. Characterization of synthetic riboswitch in cell-free protein expression systems. RNA Biology. , 1-12 (2021).
- Cole, S. D., et al. Quantification of interlaboratory cell-free protein synthesis variability. ACS Synthetic Biology. 8 (9), 2080-2091 (2019).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- Grӓwe, A., et al. A paper-based, cell-free biosensor system for the detection of heavy metals and date rape drugs. PLoS One. 14 (3), 0210940 (2019).
- Lin, X., et al. Portable environment-signal detection biosensors with cell-free synthetic biosystems. RSC Advances. 10 (64), 39261-39265 (2020).
- Caschera, F., Lee, J. W., Ho, K. K. Y., Liu, A. P., Jewett, M. C. Cell-free compartmentalized protein synthesis inside double emulsion templated liposomes with in vitro synthesized and assembled ribosomes. Chemical Communications. 52 (31), 5467-5469 (2016).
- Niederholtmeyer, H., Chaggan, C., Devaraj, N. K. Communication and quorum sensing in non-living mimics of eukaryotic cells. Nature Communications. 9, 5027 (2018).
- Timin, A. S., Gould, D. J., Sukkhorukov, G. B. Multi-layer microcapsules: Fresh insights and new applications. Expert Opinion on Drug Delivery. 14 (5), 583-587 (2017).
- Bomati, E. K., Haley, J. E., Noel, J. P., Deheyn, D. D. Spectral and structural comparison between bright and dim green fluorescent proteins in Amphioxus. Scientific Reports. 4, 5469 (2014).
- Frey, B., Reischl, U. Amplification of Genomic DNA by PCR. Molecular Diagnosis of Infectious Diseases. Methods in Molecular Medicine. 13, 143-156 (1998).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. Journal of Visualized Experiments. (62), e3923 (2012).
- Zhou, Y., et al. Rapid regeneration and reuse of silica columns from PCR purification and gel extraction kits. Scientific Reports. 8, 12870 (2018).
- Kostylev, M., Otwell, A. E., Richardson, R. E., Suzuki, Y. Cloning should be simple: Escherichia coli DH5α-mediated assembly of multiple DNA fragments with short end homologies. PLoS One. 10 (9), 0137466 (2015).
- Rockwood, D. N., et al. Materials fabrication from Bombyx mori silk fibroin. Nature Protocols. 6 (10), 1612-1631 (2011).
- Drachuk, I., et al. Silk macromolecules with amino acid-Poly(Ethylene Glycol) grafts for controlling layer-by-layer encapsulation and aggregation of recombinant bacterial cells. ACS Nano. 9 (2), 1219-1235 (2015).
- Antipov, A. A., Sukhorukov, G. B. Polyelectrolyte multilayer capsules as vehicles with tunable permeability. Advances in Colloid and Interface Science. 111 (1-2), 49-61 (2004).
- Drachuk, I., Harbaugh, S., Chávez, J. L., Kelley-Loughnane, N. Improving the activity of DNA-encoded sensing elements through confinement in silk microcapsules. ACS Applied Materials & Interfaces. 12 (43), 48329-48339 (2020).
- Melnikov, S., Ben-Shem, A., Garreau de Loubresse, N., Jenner, L., Yusupova, G., Yusupov, M. Structural basis for the inhibition of the eukaryotic ribosome. Nature Structural & Molecular Biology. 19 (6), 560-567 (2012).
- Zhao, S., et al. The future of layer-by-layer assembly: A tribute to ACS Nano associate editor Helmuth Möhwald. ACS Nano. 13 (6), 6151-6169 (2019).
- Main, K. H. S., Provan, J. I., Haynes, P. J., Wells, G., Hartley, J. A., Pyne, A. L. B. Atomic force microscopy-A tool for structural and translational DNA research. APL Bioengineering. 5, 031504 (2021).
- Riera, R., Feiner-Gracia, N., Fornaguera, C., Cascante, A., Borrós, S., Albertazzi, L. Tracking the DNA complexation state of pBAE polyplexes in cells with super resolution microscopy. Nanoscale. 11 (38), 17869-17877 (2019).
- Bilokapic, S., Strauss, M., Halic, M. Cryo-EM of nucleosome core particle interactions in trans. Scientific Reports. 8, 7046 (2018).
- Pritchard, E. M., Dennis, P. B., Omenetto, F., Naik, R. R., Kaplan, D. L. Physical and chemical aspects of stabilization of compounds in silk. Biopolymers. 97 (6), 479-498 (2012).
- Fritz, B. R., Jamil, O. K., Jewett, M. C. Implications of macromolecular crowding and reducing conditions for in vitro ribosome construction. Nucleic Acids Research. 43 (9), 4774-4784 (2015).
- Ge, X., Luo, D., Xu, J. Cell-free protein expression under macromolecular crowding conditions. PLoS One. 6 (12), 28707 (2011).
- Cawte, A. D., Unrau, P. J., Rueda, D. S. Live cell imaging of single RNA molecules with fluorogenic mango II arrays. Nature Communications. 11, 1283 (2020).
- Chen, X., et al. Visualizing RNA dynamics in live cells with bright and stable fluorescent RNAs. Nature Biotechnology. 37 (11), 1287-1293 (2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。