JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
RNA 압타머 및 리보스위치를 인코딩하는 DNA 플라스미드가 로딩된 다기능 실크 기반 마이크로캡슐의 제조
요약
이 프로토콜은 여러 리간드를 추적할 수 있는 다중화된 시험관 내 바이오센서로서 견고하고 생체적합성 DNA가 함유된 마이크로캡슐의 형성을 설명합니다.
초록
우리는 희생 구형 코어에서 층별(LbL) 조립 방법을 통해 DNA가 함유된 실크 피브로인 마이크로캡슐을 제조하기 위한 프로토콜을 소개합니다. 프라임층 및 DNA 플라스미드의 흡착에 이어, 단일 실크층의 급성 탈수 동안 실크 2차 구조에서 β-시트를 유도함으로써 견고한 마이크로캡슐의 형성을 촉진하였다. 따라서 층상은 다중 수소 결합과 소수성 상호 작용을 통해 발생했습니다. 다층 쉘의 흡착 시, 코어-쉘 구조는 원격 감지 및/또는 표적 전달에 사용되는 금 나노입자(AuNP) 및/또는 항체(IgG)로 추가로 기능화될 수 있습니다. 폴리머 프라이머의 존재, DNA 및 실크 단백질의 농도, 다수의 흡착된 층과 같은 실리카 코어에 주요 거대분자를 순차적으로 증착하는 동안 몇 가지 주요 매개변수를 조정하면 다양한 투과성 및 DNA 로딩을 갖는 생체 적합성, DNA 함유 마이크로캡슐이 생성되었습니다. 실리카 코어의 용해 시, 프로토콜은 캡슐 멤브레인의 내부 표면에 고정된 DNA 플라스미드를 갖는 중공 및 견고한 마이크로캡슐의 형성을 입증하였다. DNA 플라스미드와 외부 환경 사이에 선택적으로 투과할 수 있는 생체적합성 막을 만들어 장기 보관 시 DNA를 보존하고 공간적으로 제한된 플라스미드의 출력 반응을 개선하는 데 중요한 역할을 했습니다. DNA 템플릿의 활성과 접근성은 시험관 내 전사 및 번역 반응(cell-free system) 동안 테스트되었습니다. RNA 라이트업 앱타머 및 리보스위치를 암호화하는 DNA 플라스미드는 쉘 막에서 형광 표지된 RNA 전사체 또는 GFPa1 단백질의 국소화 중에 시각화된 바와 같이 해당 분석물과 함께 성공적으로 활성화되었습니다.
서문
합성 생물학 분야는 미생물에 의해 진화된 자연적 메커니즘을 활용하여 환경과 잠재적 위협을 모니터링함으로써 감지 기능을 개발할 수 있는 독특한 기회를 제공합니다. 중요하게도, 이러한 감지 메커니즘은 일반적으로 이러한 미생물을 유해한 노출로부터 보호하고 유전자 발현을 조절하여 부정적인 영향을 완화하거나 독성 물질의 섭취를 방지하는 반응과 관련이 있습니다. 이러한 자연 반응을 활용하지만 새로운 표적을 인식하거나 정량화 목적으로 측정할 수 있는 측정 가능한 신호(일반적으로 형광)를 생성하도록 방향을 바꾸도록 전체 세포 센서를 만들기 위해 이러한 미생물을 설계하려는 상당한 노력이 있었습니다1,2. 현재, 유전자 변형 미생물(GMO)의 사용에 대한 우려, 특히 환경이나 인체에서 방출될 때, 전체 세포 또는 일부 유전 물질의 누출로 인해, 폴리머 매트릭스에 캡슐화되어 있더라도 이러한 감지 접근법을 활용하기 위한 대안적인 방법이 필요함을 시사한다3.
GMO 배포에 대한 걱정 없이 미생물 기반 감지의 이점을 활용하는 강력한 접근 방식은 체외 전사/번역(IVTT) 시스템을 사용하는 것입니다. 실용적인 관점에서 IVTT 시스템은 초음파 처리, 비드 박동 또는 기타를 포함한 다양한 수단으로 세포에서 "추출"된 활성 상태의 대부분의 세포 구성 요소를 포함하는 혼합물로 구성됩니다4. 이 공정의 최종 산물은 전사 및 번역을 수행하도록 이미 최적화된 생화학 반응 혼합물로, 전체 세포 사용과 관련된 제약 없이(막 확산, 형질전환 효율, 세포 독성 등) "개방형 용기" 형식으로 다양한 센서를 테스트하는 데 사용할 수 있습니다. 중요한 것은, 서로 다른 센서 구성 요소를 정량적으로 추가할 수 있고, 우리가 설명한 바와 같이 서로 다른 광학 및 분광 기술로 그 효과를 연구할 수 있다는것입니다 5. IVTT 시스템의 성능이 일관되지 않을 수 있음이 밝혀졌습니다. 그러나 최근 연구에서는 준비 및 특성화를 표준화하는 접근 방식을 보여주었으며, 이는 센서 설계6에서 성능을 연구할 때 큰 도움이 됩니다. 최근에, 중금속 이온, 약물, 정족수 감지 요소및 기타의 검출을 포함하여, 종이 매트릭스에서 그 성분들의 동결건조를 통해 종이-기반 분석법을 생성하기 위해 사용되는 IVTT 시스템의 많은 예들이 입증되었다 7,8,9. IVTT 기반 센서의 흥미로운 응용 분야는 토양, 물, 인체를 포함한 다양한 유형의 환경에서 감지 응용 분야에 사용된다는 것입니다. 이러한 IVTT 시스템을 이러한 까다로운 환경에 배포하려면 IVTT 구성 요소를 포함하고 성능 저하로부터 보호하기 위한 캡슐화 접근 방식을 구현해야 합니다.
IVTT 시스템에 대한 가장 일반적인 캡슐화 접근법은 지질 캡슐, 미셀, 폴리머솜 및 기타 밀접하게 밀폐된 마이크로컨테이너(10,11,12)의 사용을 포함한다. 이 접근 방식의 한 가지 단점은 외부 환경과의 통신을 허용하고 감지 기능을 제공하기 위해 컨테이너 안팎으로 재료를 운반하기 위해 수동 또는 능동 메커니즘을 통합해야 한다는 것입니다. 이러한 문제 중 일부를 극복하기 위해 이 연구에서는 IVTT 시스템에서 표현할 다양한 센서 설계에 대한 인코딩 재료를 캡슐화하는 간단하면서도 효과적인 접근 방식을 제공하는 방법을 보고합니다. 이 접근법은 관심 플라스미드의 존재 하에 바이오폴리머의 층별(LbL) 증착을 사용하여 높은 다공성을 가진 중공 마이크로캡슐을 생성함으로써 보호된 유전 물질이 선택한 IVTT의 다양한 구성 요소와 상호 작용할 수 있도록 합니다. 이 연구는 캡슐화된 플라스미드가 플라스미드 인코딩된 앱타머 및 해당 표적에 대한 리보스위치의 반응에서 볼 수 있듯이 이 고분자 매트릭스 내에서 활성화될 때 전사 및 번역을 지시할 수 있음을 입증했습니다. 또한 이 LbL 코팅은 특별한 보관 조건 없이 몇 달 동안 플라스미드를 보호합니다.
프로토콜
1. 플라스미드 벡터의 구성.
- pJ201:23976-RS-GFPa1 벡터(DNA2.0에 의해 설계 및 생성)에서 GFPa1과 결합된 테오필린 리보스위치(ThyRS)의 코딩 서열을 증폭하여 플라스미드 벡터(pSALv-RS-GFPa1, 3.4kb)를 구성하고 대장균 발현 벡터인 pSAL13에 삽입합니다. 정방향(5'-CGTGGTACCGGTGATACCAGCATCGTCTTGATG-3') 및 역방향(5'-CGTGCTCAGCTTAAGCCAGCTCGTAG-3') 프라이머를 사용하여 GFPa1과 결합된 ThyRS의 코딩 서열을 증폭하고 제조업체의 프로토콜14에 따라 DNA 중합효소를 사용하여 50μL 부피의 PCR 반응을 수행합니다.
- 0.5 g의 아가로스, 50 mL의 TAE 완충액(40 mM Tris Acetate, 1 mM EDTA, pH 8.0) 및 3 μL의 DNA 염색으로부터 1% 아가로스 겔을 제조하였다.
- PCR 증폭 산물 5μL의 분취액을 RNase/DNase가 없는 물 5μL 및 6x 겔 로딩 염료 2μL와 혼합하고 아가로스 겔 전기영동으로 분석합니다. DNA 사다리(0.1-10.0kb)를 참조로 로드합니다. 염료 라인이 젤 바닥에 거의 도달할 때까지 120V에서 젤을 실행합니다.
- UV transilluminator 이미징 시스템을 사용하여 DNA 단편을 시각화하여 DNA15의 정확한 크기를 확인합니다.
- PCR 정제 키트를 사용하여 PCR 산물을 제조자의 프로토콜16에 따라 정제한다.
- PCR 산물 또는 플라스미드 벡터(농도 20-50 ng/μL) 1.5 μL, 10x 효소 완충액 1.5 μL, 각 효소 1 μL 및 RNase/DNase-free 물 1.5 μL를 포함하는 15 μL 반응에서 KpnI 및 BlpI 제한 효소로 PCR 산물 및 pSAL 발현 벡터를 37°C에서 2시간 동안 분해합니다.
- 3 μL의 6x 겔 로딩 염료를 반응 혼합물에 첨가하고, 단계 1.3-1.5에 설명된 바와 같이 1% 아가로스 겔 상에서 분해된 단편을 분리한다.
- 제조자의 프로토콜16에 따라 겔 추출 키트를 사용하여 DNA 단편을 정제한다.
- 분해된 PCR 산물을 분해된 선형화된 플라스미드 벡터인 pSAL로 Ligate, T4 DNA 리가제 및 보충된 리가제 완충액을 사용하여 분해된 벡터 3-20fmol, 분해된 PCR 산물 9-60fmol, 리가아제 완충액 2μL, T4 DNA 리가아제 1μL(1단위) 및 DNase/RNase가 없는 물. 결찰 반응을 25°C에서 3시간 동안 배양합니다.
참고: 반응 혼합물의 총 DNA 함량이 0.01-0.1μg인지 확인합니다. - 대장 균 DH5α 컴피턴트 세포를 제조사의 프로토콜17에 따라 10 ng의 라이게이션 반응 혼합물로 형질전환시킨다.
- 암피실린(100μg/mL)이 보충된 LB-한천 플레이트에서 37°C에서 하룻밤 동안 형질전환된 세포를 성장시킵니다.
- 플레이트에서 3-4개의 박테리아 콜로니를 골라내고 각각을 암피실린(100μg/mL)이 보충된 LB 배지 5mL에 무균적으로 옮깁니다. 배양물을 225 rpm의 진탕 인큐베이터에서 37°C에서 밤새 성장시킨다.
- 실온에서 3분 동안 11 x g 에서 원심분리하여 밤새 배양물을 펠렛화합니다.
- 정제 키트를 사용하여 제조자의 프로토콜16에 따라 플라스미드를 정제한다.
- DNA 시퀀싱을 통해 정제된 플라스미드의 서열을 확인합니다. 플라스미드 맵과 생성된 구축물(pSALv-RS-GFPa1)의 서열을 도 1에 나타내었다.
2. 대규모 DNA 정제.
- 플라스미드 벡터 pSALv-RS-GFPa1 (3.4 kb) (GFPa1 리포터 유전자와 결합된 테오필린 리보스위치 암호화) 또는 pET28c-F30-2x브로콜리 (5.4 kb) (브로콜리 앱타머 암호화)를 제조사의 프로토콜17에 따라 대장균 DH5α 적격 세포로 형질전환시킨다.
- pSALv-RS-GFPa1로 형질전환된 세포용 암피실린(100μg/mL) 또는 pET28c-F30-2x 브로콜리로 형질전환된 세포용 카나마이신(50μg/mL)이 보충된 LB-한천 플레이트에서 37°C에서 하룻밤 동안 형질전환된 세포를 성장시킵니다.
- 플레이트에서 3-4개의 박테리아 콜로니를 선택하고 각 콜로니를 적절한 항생제(100μg/mL 암피실린 또는 50μg/mL 카나마이신)가 보충된 5mL의 LB 배지에 무균적으로 옮깁니다. 배양물을 225 rpm의 진탕 인큐베이터에서 37°C에서 밤새 성장시킨다.
- 하룻밤 배양액 3mL를 사용하여 적절한 항생제(100μg/mL 암피실린 또는 50μg/mL 카나마이신)가 보충된 LB 150mL에 접종하고 225rpm의 진탕 배양기에서 37°C에서 밤새 배양액을 배양합니다.
- 세포를 4°C에서 10분 동안 ≥3400 x g 에서 원심분리하여 펠렛화한다.
- 정제 키트를 사용하여 제조자의 프로토콜16에 따라 플라스미드를 정제한다.
- 0.5mL의 순수한 DNase/RNase가 없는 물로 DNA를 용리합니다. DNA 농도를 측정하고 1mL의 DNA 원액(100ng/μL)을 준비합니다. 추가 사용이 가능할 때까지 DNA가 있는 튜브를 4°C에서 보관하십시오.
3. 실크 피브로인 추출 및 초기 재료 준비.
- Silk-LiBr 용액18의 10%를 차지하기 위해 다른 곳에서 상세히 기술된 절차에 따라 봄빅스 모리 누에 고치로부터 재구성된 실크 피브로인(SF) 단백질의 수용액을 제조한다.
- 수성 SF 용액의 최종 농도를 결정하십시오. 60 mm 페트리 접시에 0.5 mL의 실크 용액을 피펫팅하고, 60°C에서 건조시켜, 건조된 실크 필름의 무게를 측정하였다. 건조 중량을 0.5mL로 나누어 부피당 중량 백분율을 계산합니다.
- 혈청학적 피펫을 통해 물을 천천히 첨가하여 농축된 실크 용액을 DNase/RNase가 없는 증류수로 희석하여 1mg/mL 최종 농도를 얻습니다. 나중에 사용할 수 있도록 용액을 4°C에서 보관하십시오.
- 항체 표지 키트를 사용하여 형광 표지된 실크 피브로인을 준비합니다. 1 mL의 2 mg / mL 실크 피브로인 용액을 사용하여 제조업체의 프로토콜19에 따라 단백질의 N- 말단 α- 아미노 그룹을 NHS 에스테르 활성화 유도체 염료와 결합시킵니다.
- 50mg/mL 농도의 폴리에틸렌이민(PEI) 수용액 6mL를 준비하고 HCl(4M)로 pH를 1로 조정합니다. 멸균된 0.2μm 멤브레인을 통해 용액을 여과합니다. 몇 달 동안 주변 조건에서 보관할 수 있습니다.
- SiO2 코어를 준비합니다. 300 μL의SiO2 입자를 2 mL 미세원심분리 튜브에 피펫팅한다. 0.2 x g 에서 1분 동안 원심분리하여 1mL의 DNase/RNase가 없는 물로 미세 입자를 두 번 세척합니다.
4. 프라임층, DNA 플라스미드 및 실크층의 층별 증착을 수행합니다.
- PEI 프라임층을SiO2 미세입자에 증착하기 위해, 3.6단계의 스펀다운 펠릿에 PEI 용액 1mL를 첨가하고, 800rpm의 열혼합기에서 15분 동안 상온 조건에서 혼합물을 교반한다. 0.2 x g 에서 1분 동안 원심분리하여 1mL의 DNase/RNase가 없는 탈이온수로 입자를 4회 세척합니다.
- DNA 층의 증착을 수행하기 위해, 2.7 단계의 DNA 플라스미드 수용액 1 mL를 PEI 프라이밍 된 미세 입자에 첨가하고 혼합물을 4 °C에서 800 rpm으로 15 분 동안 열 혼합기에서 부드럽게 교반한다. DNA 부하가 다른 마이크로 캡슐을 준비하려면 DNase / RNase가없는 증류수를 사용하여 DNA 플라스미드의 농도를 50-200 ng / μL로 조정하고 이러한 용액 1mL를 사용하여 DNA를 증착합니다. 0.2 x g 에서 1분 동안 원심분리하여 미립자를 수집합니다.
- GFPa1과 결합된 테오필린 리보스위치를 암호화하는 DNA 플라스미드를 ThyRS-GFPa1로, 브로콜리 압타머를 BrocApt로 인코딩하는 DNA 플라스미드를 튜브에 표시합니다.
알림: DNA가 있는 미세 원심분리기 튜브를 얼음 위에 보관하십시오. - 상층액을 조심스럽게 제거하고 1mL의 DNase/RNase가 없는 증류수로 미립자를 4회 세척하고, 매번 0.2 x g 에서 1분 동안 원심분리 후 상층액을 버립니다. 달리 명시되지 않는 한 실온(RT)에서 모든 실험을 수행합니다.
- 실크 피브로인 층의 증착을 수행하기 위해 3.3단계에서 재구성된 SF 수용액 1mL를 DNA가 흡착된 미세입자에 첨가하고 혼합물을 10°C에서 750rpm으로 15분 동안 부드럽게 소용돌이 및 교반합니다. 4°C에서 1분 동안 0.2 x g 에서 원심분리하여 미세 입자를 수집하고 상층액을 제거한 다음 DNase/RNase가 없는 증류수 1mL로 1회 세척합니다. 원심 분리를 반복하고 상청액을 버립니다.
알림: 실험 중에 온도로 인한 겔화를 피하기 위해 실크 용액을 얼음 위에 보관하십시오. - 입자를 메탄올로 점진적으로 처리하여 실크 단백질 구조에서 β장 형성을 유도합니다. 먼저 DNase/RNase 증류수 0.5mL를 넣고 미세원심분리기 튜브를 소용돌이치게 한 다음 100% 메탄올 0.5mL를 넣습니다. 10°C에서 5분 동안 열혼합기에서 입자를 부드럽게 흔듭니다. 0.2 x g 에서 1분 동안 원심분리하여 입자를 수집합니다. 상청액을 제거합니다.
- 입자를 메탄올로 처리하여 β 시트의 형성을 촉진하고 실크 층의 강력한 물리적 흡착을 보장합니다. 100 % 메탄올 1mL를 넣는다. 10°C에서 10분 동안 750rpm으로 열혼합기에서 입자를 부드럽게 흔듭니다.
- 4°C에서 1분 동안 0.2 x g 에서 원심분리하여 입자를 수집하고 매번 1mL의 DNase/RNase가 없는 증류수로 두 번 세척하고 상층액을 버리고 다음 원심분리 전에 부드럽게 볼텍싱합니다.
- 4.5-4.8 단계를 20 번 반복하여 실크 다층 코어 쉘 구조를 얻습니다. 마지막 증착 단계에서는 3.4 단계의 형광 표지 실크(Silk-DyLight550, 1mL)를 사용합니다.
- 마지막 세척 단계를 수행하고 주변 조건에서 DNase/RNase가 없는 증류수 1mL에 미립자를 보관합니다.
참고: 실크 층을 증착하는 동안 입자 응집을 방지하려면 입자 현탁액을 육안으로 검사하고 1mL 피펫 팁을 사용하여 위아래로 피펫하여 균일한 입자 분포를 촉진합니다. - 식 1을 사용하여 각 마이크로캡슐, N DNA에 캡슐화된 DNA 플라스미드 사본의 수를 계산한다:
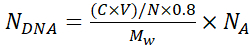 (1)
(1)
여기서 N = 6.769 × 1011 - 캡슐화에 사용되는 SiO2 코어의 수. λ = 320 nm에서 연속 희석 및 흡수 A320 을 사용하여 실리카 입자의 알려진 농도에 대한 표준 곡선에서 계산합니다.
흡착에 사용된 DNA의 C-초기 농도
V- 흡착에 사용되는 DNA의 부피
0.8- 코어의 DNA 흡착 효율
Mw- DNA 플라스미드의 분자량
NA- 아보가드로 수 (6.022 × 1023)
5. 실크 마이크로 캡슐을 얻기 위해 코어를 용해시킨다.
- 원액(8%)을 증류수로 희석하여 pH 5.5의 48% 불산(HF) 용액을 준비합니다. 50mL 원심분리기 튜브를 구입합니다. 5mL의 HF를 조심스럽게 피펫팅하고 25mL의 증류수를 첨가하여 8% HF 용액을 얻습니다.
주의 : HF는 부식성이 강한 산이며 조직에 심각한 화상을 입힐 수 있습니다. 실험을 위해 HF를 취급하고 사용하는 동안 각별한 주의를 기울여야 합니다. 바람직하지 않은 유출 사고를 방지하기 위해 조직에서 개발한 HF의 적절한 사용 및 취급을 위해 표준 운영 절차(SOP)를 준수합니다. HF 산을 희석하기 위해 유리 용기를 사용하지 마십시오. 화학 후드를 사용하여 프로토콜의 이 단계를 수행합니다. - 4.10 단계로부터 펠릿화된 코어-쉘 마이크로입자에 8% HF 용액 1.5 mL를 첨가하여SiO2 코어를 용해시킨다. 부드럽게 소용돌이치고 코어가 주변 조건에서 450rpm에서 부드럽게 흔들리면서 밤새 용해되도록 합니다.
알림: HF의 유출을 방지하려면 접목 테이프를 사용하여 미세 원심분리기 튜브를 밀봉하십시오. 화학 후드를 사용하여 프로토콜의 이 단계를 수행합니다. - 2L의 탈이온수로 채워진 2L 유리 비커를 준비합니다. 마이크로캡슐 용액을 투석 장치(50 kDa MWCO)로 옮기고, 다음 3일 동안 매 3시간마다 물을 반복적으로 교체하면서 탈이온수에 대해 투석한다.
알림: 처음 세 번의 물 교환 중에 상청액을 수집하고 유해 폐기물에 대해 확립된 프로토콜에 따라 용액을 폐기하십시오. - 1mL 피펫을 사용하여 투석 장치의 현탁액을 새로운 2mL 미세원심분리기 튜브로 옮겨 마이크로캡슐을 수집합니다.
참고: 마이크로캡슐의 수용액을 주변 조건에서 몇 년 동안 보관하십시오.
6. 공초점 레이저 스캐닝 현미경(CLSM)을 사용한 실크 피브로인 마이크로캡슐의 이미징.
- DNA 염료를 사용하여 DNA 염색을 수행합니다.
- 300 μL의 중공 실크 피브로인 마이크로캡슐을 신선한 1 mL 마이크로원심분리기 튜브로 옮깁니다. RNase/DNase가 없는 증류수 500μL를 추가합니다.
- DNA 염색 염료 5μL를 넣고 잠시 소용돌이치고 빛으로부터 보호되는 2시간 동안 실온에서 배양합니다.
- 매번 4°C에서 20분 동안 0.1 x g 에서 원심분리하여 4개의 세척 단계를 수행하고 400μL의 상층액을 조심스럽게 제거하고 400μL의 RNase/DNase가 없는 증류수로 보충합니다.
- 100x 오일 이멀젼 대물렌즈(NA 1.49)를 사용하여 3개의 주요 레이저(405nm, 488nm, 561nm)가 장착된 도립 컨포칼 시스템에서 실크 캡슐의 이미징을 수행합니다. 캡슐 샘플 100μL를 8웰 챔버 유리 슬라이드의 단일 웰로 옮기고 이미징 전에 캡슐을 20-30분 동안 침전시킵니다.
참고: 염료는 광표백에 매우 민감합니다. 슬라이드를 알루미늄 호일로 덮어 샘플을 보호하십시오.
7. 분자량 차단 (MWCO) 방법을 사용한 중공 마이크로 캡슐의 투과성 추정.
- 서로다른 Mw(4 kDa, 20 kDa, 40 kDa, 70 kDa, 150 kDa, 250 kDa, 500 kDa, and 2 MDa)의 FITC-표지된 덱스트란 형광단 용액(20 μM, diH2O)을 각각 2 mL씩 준비한다.
- 캡슐의 현탁액 100 μL를 챔버형 유리 슬라이드의 단일 웰에 피펫팅한다. 각 마이크로 캡슐 설계(PEI 농도, DNA 플라스미드 로딩 수, 실크 피브로인 농도 및 층 수)를 개별적으로 분석합니다.
- 각 웰에 가장 낮은Mw에서 시작하여 가장 높은 것까지 300μL의 특정 형광단 용액을 추가하여 각 웰이 특정 형광단 용액에 해당하도록 합니다. 위아래로 피펫팅하여 혼합하고 형광단 용액의 확산이 평형에 도달할 때까지 RT에서 1시간 동안 혼합물을 배양합니다.
- 슬라이드를 컨포칼 레이저 스캐닝 현미경(CLSM)으로 옮기고 여기 λ = 488nm에서 100x 오일 이멀젼 대물렌즈를 사용하여 각 웰을 이미지화합니다.
- 캡슐이 가장 큰 직경의 원 형태로 나타나도록 초점면을 조정하여 관심 영역을 식별합니다. 이것은 일반적으로 캡슐이 중력으로 인해 침전될 때 우물 바닥에 더 가까운 샘플을 볼 때 발생합니다.
- 마이크로 캡슐의 여러 이미지를 수집amp슬라이드를 XY 방향으로 움직여 les. 각 샘플에 대해 최대 100-150개의 캡슐을 설명하도록 이미지를 캡처합니다.
- ImageJ 소프트웨어를 사용하여 캡슐 내부와 외부의 형광 강도를 비교하여 각Mw 형광단 용액에서 캡슐 멤브레인의 투과성을 분석합니다. 이를 위해 원 형태로 관심 영역(ROI)을 그려 캡슐의 둘레를 나타내고 분석/측정을 클릭하여 내부 형광 강도를 측정합니다. 데이터를 스프레드시트로 표로 만듭니다. 총 200-300 캡슐에 대해 각 마이크로 캡슐에 대해이 작업을 수행하십시오.
- ROI를 설명하고 캡슐에서 떨어진 강도를 측정하여 동일한 방식으로 외부 형광 강도를 평가합니다. 통계 분석을 위해 3-5 측정을 수행합니다.
- 통계 분석을 수행하려면 쌍체 t-검정(p < 0.05)을 사용하여 캡슐 내부와 외부의 형광 강도를 비교합니다.
- 가변 Mw를 갖는 FITC-Dextran에 대한 유체역학적 반경에 기초하여 마이크로캡슐의 투과성을 추정하기 위해 변환 표 2를 사용한다.
8. 실크 마이크로캡슐에서 합성 테오필린 리보스위치의 시험관내 활성화
- 테오필린 원액(100 mM, DMSO) 1mL를 준비한다. 40분 동안 얼음 위에서 성분을 해동하여 원형 DNA를 위한 대장균 S30 추출물 시스템을 준비합니다.
- 0.5mL DNase/RNase가 없는 미세 원심분리기 튜브를 얻습니다. 시험관내 전사/번역 반응을 수행하고, 무세포 성분을 다음 순서로 마이크로캡슐 샘플과 조합한다 (총 부피 50 μL): 아미노산이 없는 S30 프리믹스 (20 μL); S30 추출물, 원형(15 μL); 완전 아미노산 혼합물(5μL); 단계 4.10으로부터의 ThyRS-GFPa1 플라스미드를 함유하는 속이 빈 마이크로캡슐 (9 μL); 및 테오필린, 100 mM DMSO (1 μL).
알림: 모든 구성 요소를 추가한 후 튜브를 잠시 와동시키고 샘플을 수집하십시오.amp0.2× g 에서 몇 초 동안 짧은 원심분리 동안. - 튜브를 30°C에서 4시간 동안 배양하고 λ = 488nm의 여기와 GFP/FITC 필터(510nm ± 20nm)의 방출을 사용하여 플레이트 리더에서 형광을 확인합니다.
- 488nm 및 561nm 레이저를 사용하여 모든 LCSM 시스템에서 캡슐을 이미지화합니다. 100x 오일 이멀젼 대물렌즈와 8웰 챔버 슬라이드를 사용하여 최고 품질의 이미지를 얻을 수 있습니다.
9. 실크 마이크로캡슐 중의 브로콜리 압타머의 시험관 내 활성화
- DFHBI-1T 염료 (30 μM, diH2O)의 원액 1 mL를 준비한다. 40분 동안 얼음에서 성분을 해동하여 PURE(재조합 요소를 사용한 단백질 합성) cell-free system 반응 키트를 준비합니다.
- 0.5mL DNase/RNase가 없는 미세 원심분리기 튜브를 얻습니다. 무세포 반응 성분을 다음 순서로 마이크로캡슐의 샘플과 조합함으로써 시험관내 전사 반응을 수행한다 (총 부피 50 μL): 용액 A (20 μL); 용액 B (15 μL); 단계 4.10으로부터의 BrocApt 플라스미드를 함유하는 중공 마이크로캡슐 (14 μL); 및 DFHBI-1T 염료 (1 μL).
알림: 모든 구성 요소를 추가한 후 튜브를 잠시 와동시키고 샘플을 수집하십시오.amp0.2× g 에서 몇 초 동안 짧은 원심분리 동안. - 튜브를 37°C에서 6시간 동안 배양하고λex = 470nm에서의 여기 및λem = 510nm ± 20nm에서의 방출을 사용하여 플레이트 리더에서 형광을 확인합니다.
- 488nm 및 561nm 레이저를 사용하여 모든 LCSM 시스템에서 캡슐을 이미지화합니다. 100x 오일 이멀젼 대물렌즈와 8웰 커버유리 챔버 슬라이드를 사용하여 최고 품질의 이미지를 얻을 수 있습니다.
결과
여기에서 이 연구는 실크 단백질 캡슐에 캡슐화한 후 다양한 센서 디자인(두 가지 유형의 RNA 조절 전사/번역 요소)을 인코딩하는 DNA 템플릿의 기능을 다룹니다. 마이크로캡슐은 주요 구성 요소인 프라임층, 센서 설계를 인코딩하는 DNA 플라스미드 및 실크 피브로인 바이오폴리머의 템플릿화된 층별(LbL) 조립을 통해 제조되었습니다(그림 2). 층상 방식으로 거대분자를 증착하?...
토론
다양한 유형의 DNA-인코딩된 센서 설계로 로딩된 선택적 투과성 하이드로겔 마이크로캡슐은 이 프로토콜에 따라 제조될 수 있다. LbL 접근법의 특징 중 하나는 상향식 조립 동안 마이크로 캡슐의 복잡성을 조정할 수 있다는 것인데, 이는 일반적으로 희생 템플릿에 분자 종의 흡착으로 시작됩니다. 초기 성분의 농도, pH 조건 및 층수를 조심스럽게 조절함으로써, 상이한 DNA 로딩 파라미터, 기능성 및 ?...
공개
여기에 제시된 견해와 의견은 저자의 견해이며 반드시 DoD 또는 그 구성 요소의 견해를 나타내는 것은 아닙니다
감사의 말
이 연구는 공군 과학 연구실의 LRIR 16RH3003J 보조금과 미국 국방부 연구 및 공학 차관실의 ARAP (Synthetic Biology for Military Environments Applied Research for the Advancement of S & T Priorities) 프로그램의 지원을 받았습니다.
ThyRS에 대한 플라스미드 벡터 서열(pSALv-RS-GFPa1, 3.4kb)은 J. Gallivan 박사에 의해 아낌없이 제공되었습니다. Bombyx mori 의 누에 고치는 매사추세츠주 터프츠 대학교의 D.L. Kaplan 박사가 아낌없이 기증했습니다.
자료
| Name | Company | Catalog Number | Comments |
| (Z)-4-(3,5-difluoro-4-hydroxybenzylidene)-2-methyl-1-(2,2,2-trifluoroethyl)-1H-imidazol-5(4 H)-one (DFHBI-1T) | Lucerna | DFHBI-1T | |
| 5x T4 DNA Ligase Buffer | ThermoFisher Scientific | 46300-018 | |
| 6x Blue Gel Loading Dye | New England BioLabs | B7021S | |
| 96-well plates, black circular | Corning | 3601 | |
| Agarose | Sigma-Aldrich | A9539 | BioReagent, for molecular biology, low EEO |
| Ampicillin sodium salt | Sigma-Aldrich | A0166 | powder or crystals, BioReagent, suitable for cell culture |
| BlpI restriction enzymes | New England BioLabs | R0585S | |
| Corning Disposable Vacuum Filter/Storage Systems | FisherScientific | 09-761-1 | |
| Dimethyl sulfoxide, DMSO | Sigma-Aldrich | 472301 | ACS reagent, ≥99.9% |
| DNA Plasmid, pET28c-F30-2x Broccoli (5.4 kb), BrocApt. | Addgene | Plasmid #66788 | |
| DyLightTM550 Antibody Labeling kit (Invitrogen) | ThermoFisher Scientific | 84530 | |
| E. coli S30 extract system for circular DNA | Promega | L1020 | |
| Falcon Conical centrifuge tubes, 15 mL | FisherScientific | 14-959-53A | |
| Falcon Conical centrifuge tubes, 50 mL | 14-432-22 | ||
| Fisherbrand Microcentrifuge tubes, 1.5 mL | FisherScientific | 05-408-129 | |
| Hydrofluoric acid, HF | Sigma-Aldrich | 695068 | ACS reagent, 48% |
| Kanamycin sulfate | Sigma-Aldrich | 60615 | mixture of Kanamycin A (main component) and Kanamycin B and C |
| KpnI restriction enzymes | New England BioLabs | R0142S | |
| LB agar plate supplemented with 100 µg/mL ampicillin | Sigma-Aldrich | L5667 | pre-poured agar plates with 100 µg/mL ampicillin |
| LB agar plate supplemented with 50 µg/mL kanamycin | Sigma-Aldrich | L0543 | pre-poured agar plates with 50 µg/mL kanamycin |
| LB broth (Lennox grade) | Sigma-Aldrich | L3022 | |
| Lithium bromide, LiBr | Sigma-Aldrich | 213225 | ReagentPlus, ≥99% |
| Max Efficiency DH5-α competent E. coli strain | ThermoFisher Scientific | 18258012 | |
| Methanol | MilliporeSigma | 322415 | anhydrous, 99.8% |
| MilliQ-water | EMD MilliPore | Milli-Q Reference Water Purification System | |
| MinElute PCR Purification Kit | Qiagen | 28004 | |
| N-(3-Dimethylaminopropyl)-N′-ethylcarbodiimide hydrochloride, EDC | Sigma-Aldrich | E1769 | |
| PBS (phosphate buffered saline) | ThermoFisher Scientific | 10010023 | 1x PBS, pH 7.4 |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530S | |
| Polyethylenimine, branched | Sigma-Aldrich | 408727 | average Mw ~25,000 |
| PURExpress In Vitro Protein Synthesis Kit | New England BioLabs | E6800S | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | |
| Quick-Load 2-Log DNA Ladder (0.1-10.0 kb) | New England BioLabs | N0469S | |
| SiO₂ silica microspheres, 4.0 µm | Polysciences, Inc. | 24331-15 | 10% aqueous solution |
| Slide-A-Lyzer G2 Dialysis Cassettes, 3.5K MWCO, 15 mL | ThermoFisher Scientific | 87724 | |
| Sodium carbonate, Na₂CO₃ | Sigma-Aldrich | 222321 | ACS reagent, anhydrous, ≥99.5%, powder |
| Spectrum Spectra/Por Float-A-Lyzer G2 Dialysis Devices | FisherScientific | 08-607-008 | Spectrum G235058 |
| SYBR Safe DNA gel stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase (5 U/µL) | ThermoFisher Scientific | EL0011 | |
| Theophylline | Sigma-Aldrich | T1633 | anhydrous, ≥99%, powder |
| Tris Acetate-EDTA buffer (TAE buffer) | Sigma-Aldrich | T6025 | Contains 40 mM Tris-acetate and 1 mM EDTA, pH 8.3. |
| UltraPure DNase/RNase-Free Distilled Water | FisherScientific | 10-977-023 | |
| ZymoPURE II Plasmid MaxiPrep kit | ZymoResearch | D4202 |
참고문헌
- Slomovic, S., Pardee, K., Collins, J. J. Synthetic biology devices for in vitro and in vivo diagnostics. Proceedings of the National Academy of Sciences of the United States of America. 112 (47), 14429-14435 (2015).
- Harbaugh, S. V., Goodson, M. S., Dillon, K., Zabarnick, S., Kelley-Loughnane, N. Riboswitch-based reversible dual-color sensor. ACS Synthetic Biology. 6 (5), 766-781 (2017).
- König, H., Frank, D., Heil, R., Coenen, C. Synthetic genomics and synthetic biology applications between hopes and concerns. Current Genomics. 14 (1), 11-24 (2013).
- Silverman, A. D., Karim, A. S., Jewett, M. C. Cell-free gene expression: An expanded repertoire of applications. Nature Reviews Genetics. 21, 151-170 (2020).
- Chushak, Y., et al. Characterization of synthetic riboswitch in cell-free protein expression systems. RNA Biology. , 1-12 (2021).
- Cole, S. D., et al. Quantification of interlaboratory cell-free protein synthesis variability. ACS Synthetic Biology. 8 (9), 2080-2091 (2019).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- Grӓwe, A., et al. A paper-based, cell-free biosensor system for the detection of heavy metals and date rape drugs. PLoS One. 14 (3), 0210940 (2019).
- Lin, X., et al. Portable environment-signal detection biosensors with cell-free synthetic biosystems. RSC Advances. 10 (64), 39261-39265 (2020).
- Caschera, F., Lee, J. W., Ho, K. K. Y., Liu, A. P., Jewett, M. C. Cell-free compartmentalized protein synthesis inside double emulsion templated liposomes with in vitro synthesized and assembled ribosomes. Chemical Communications. 52 (31), 5467-5469 (2016).
- Niederholtmeyer, H., Chaggan, C., Devaraj, N. K. Communication and quorum sensing in non-living mimics of eukaryotic cells. Nature Communications. 9, 5027 (2018).
- Timin, A. S., Gould, D. J., Sukkhorukov, G. B. Multi-layer microcapsules: Fresh insights and new applications. Expert Opinion on Drug Delivery. 14 (5), 583-587 (2017).
- Bomati, E. K., Haley, J. E., Noel, J. P., Deheyn, D. D. Spectral and structural comparison between bright and dim green fluorescent proteins in Amphioxus. Scientific Reports. 4, 5469 (2014).
- Frey, B., Reischl, U. Amplification of Genomic DNA by PCR. Molecular Diagnosis of Infectious Diseases. Methods in Molecular Medicine. 13, 143-156 (1998).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. Journal of Visualized Experiments. (62), e3923 (2012).
- Zhou, Y., et al. Rapid regeneration and reuse of silica columns from PCR purification and gel extraction kits. Scientific Reports. 8, 12870 (2018).
- Kostylev, M., Otwell, A. E., Richardson, R. E., Suzuki, Y. Cloning should be simple: Escherichia coli DH5α-mediated assembly of multiple DNA fragments with short end homologies. PLoS One. 10 (9), 0137466 (2015).
- Rockwood, D. N., et al. Materials fabrication from Bombyx mori silk fibroin. Nature Protocols. 6 (10), 1612-1631 (2011).
- Drachuk, I., et al. Silk macromolecules with amino acid-Poly(Ethylene Glycol) grafts for controlling layer-by-layer encapsulation and aggregation of recombinant bacterial cells. ACS Nano. 9 (2), 1219-1235 (2015).
- Antipov, A. A., Sukhorukov, G. B. Polyelectrolyte multilayer capsules as vehicles with tunable permeability. Advances in Colloid and Interface Science. 111 (1-2), 49-61 (2004).
- Drachuk, I., Harbaugh, S., Chávez, J. L., Kelley-Loughnane, N. Improving the activity of DNA-encoded sensing elements through confinement in silk microcapsules. ACS Applied Materials & Interfaces. 12 (43), 48329-48339 (2020).
- Melnikov, S., Ben-Shem, A., Garreau de Loubresse, N., Jenner, L., Yusupova, G., Yusupov, M. Structural basis for the inhibition of the eukaryotic ribosome. Nature Structural & Molecular Biology. 19 (6), 560-567 (2012).
- Zhao, S., et al. The future of layer-by-layer assembly: A tribute to ACS Nano associate editor Helmuth Möhwald. ACS Nano. 13 (6), 6151-6169 (2019).
- Main, K. H. S., Provan, J. I., Haynes, P. J., Wells, G., Hartley, J. A., Pyne, A. L. B. Atomic force microscopy-A tool for structural and translational DNA research. APL Bioengineering. 5, 031504 (2021).
- Riera, R., Feiner-Gracia, N., Fornaguera, C., Cascante, A., Borrós, S., Albertazzi, L. Tracking the DNA complexation state of pBAE polyplexes in cells with super resolution microscopy. Nanoscale. 11 (38), 17869-17877 (2019).
- Bilokapic, S., Strauss, M., Halic, M. Cryo-EM of nucleosome core particle interactions in trans. Scientific Reports. 8, 7046 (2018).
- Pritchard, E. M., Dennis, P. B., Omenetto, F., Naik, R. R., Kaplan, D. L. Physical and chemical aspects of stabilization of compounds in silk. Biopolymers. 97 (6), 479-498 (2012).
- Fritz, B. R., Jamil, O. K., Jewett, M. C. Implications of macromolecular crowding and reducing conditions for in vitro ribosome construction. Nucleic Acids Research. 43 (9), 4774-4784 (2015).
- Ge, X., Luo, D., Xu, J. Cell-free protein expression under macromolecular crowding conditions. PLoS One. 6 (12), 28707 (2011).
- Cawte, A. D., Unrau, P. J., Rueda, D. S. Live cell imaging of single RNA molecules with fluorogenic mango II arrays. Nature Communications. 11, 1283 (2020).
- Chen, X., et al. Visualizing RNA dynamics in live cells with bright and stable fluorescent RNAs. Nature Biotechnology. 37 (11), 1287-1293 (2019).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유