É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Preparação de Microcápsulas Multifuncionais à Base de Seda Carregadas com Plasmídeos de DNA Codificadores de RNA Aptamers e Riboswitches
Neste Artigo
Resumo
O protocolo descreve a formação de microcápsulas carregadas de DNA robustas e biocompatíveis como biossensores in vitro multiplexados capazes de rastrear vários ligantes.
Resumo
Apresentamos um protocolo para a preparação de microcápsulas de fibroína de seda carregadas de DNA através do método de montagem Layer-by-Layer (LbL) em núcleos esféricos sacrificiais. Após a adsorção de uma camada prima e plasmídeos de DNA, a formação de microcápsulas robustas foi facilitada pela indução de folhas de β na estrutura secundária da seda durante a desidratação aguda de uma única camada de seda. Assim, a estratificação ocorreu via ligações múltiplas de hidrogênio e interações hidrofóbicas. Após a adsorção de cascas multicamadas, as estruturas núcleo-casca podem ser funcionalizadas com nanopartículas de ouro (AuNPs) e/ou anticorpos (IgG) para serem usadas para sensoriamento remoto e/ou entrega direcionada. O ajuste de vários parâmetros-chave durante a deposição sequencial de macromoléculas-chave em núcleos de sílica, como a presença de um primer polimérico, a concentração de DNA e proteína da seda, bem como um número de camadas adsorvidas, resultou em microcápsulas biocompatíveis, carregadas de DNA com permeabilidade variável e cargas de DNA. Após a dissolução dos núcleos de sílica, o protocolo demonstrou a formação de microcápsulas ocas e robustas com plasmídeos de DNA imobilizados na superfície interna da membrana da cápsula. A criação de uma membrana biocompatível seletivamente permeável entre os plasmídeos de DNA e o ambiente externo preservou o DNA durante o armazenamento de longo prazo e desempenhou um papel importante na resposta de saída melhorada de plasmídeos espacialmente confinados. A atividade dos modelos de DNA e sua acessibilidade foram testadas durante reações de transcrição e tradução in vitro (sistemas livres de células). Plasmídeos de DNA que codificam aptâmeros e riboswitches de RNA foram ativados com sucesso com analitos correspondentes, como foi visualizado durante a localização de transcritos de RNA marcados fluorescentemente ou proteína GFPa1 nas membranas da casca.
Introdução
O campo da biologia sintética oferece oportunidades únicas para desenvolver capacidades de sensoriamento, explorando mecanismos naturais desenvolvidos por microrganismos para monitorar seu ambiente e ameaças potenciais. É importante ressaltar que esses mecanismos de sensoriamento estão tipicamente ligados a uma resposta que protege esses microrganismos da exposição nociva, regulando a expressão gênica para mitigar efeitos negativos ou prevenir a ingestão de materiais tóxicos. Tem havido esforços significativos para projetar esses microrganismos para criar sensores de células inteiras, aproveitando essas respostas naturais, mas redirecionando-os para reconhecer novos alvos e/ou produzir um sinal mensurável que possa ser medido para fins de quantificação (tipicamente fluorescência)1,2. Atualmente, a preocupação com o uso de microrganismos geneticamente modificados (OGMs), principalmente quando liberados no ambiente ou no corpo humano, devido ao vazamento de células inteiras ou de parte de seu material genético, mesmo que encapsulado em matriz polimérica, sugere que são necessárias formas alternativas de exploração dessas abordagens desensoriamento3.
Uma abordagem poderosa para explorar os benefícios do sensoriamento baseado em microrganismos sem a preocupação com a implantação de OGMs é o uso de sistemas de transcrição/tradução in vitro (IVTT). Do ponto de vista prático, os sistemas de TIVP consistem em uma mistura contendo a maioria dos componentes celulares em estado ativo que foi "extraída" das células por diferentes meios, incluindo sonicação, batimento de contas ou outros4. O produto final deste processo é uma mistura de reação bioquímica já otimizada para realizar transcrição e tradução que pode ser usada para testar diferentes sensores em formato de "vaso aberto", sem as restrições associadas ao uso de células inteiras (difusão de membrana, eficiência de transformação, toxicidade celular, etc.). É importante ressaltar que diferentes componentes do sensor podem ser adicionados quantitativamente, e seu efeito estudado por diferentes técnicas ópticas e espectrométricas, como demonstramos5. Tem-se notado que o desempenho dos sistemas IVTT pode ser inconsistente; No entanto, estudos recentes têm mostrado abordagens para padronizar sua preparação e caracterização, o que é de grande ajuda quando se estuda seu desempenho no projeto desensores6. Recentemente, muitos exemplos de sistemas de TICV utilizados para criar ensaios baseados em papel através da liofilização de seus componentes em matrizes de papel têm sido demonstrados, incluindo a detecção de íons de metais pesados, drogas, elementos de quorum sensing e outros 7,8,9. Um espaço de aplicação interessante para sensores baseados em IVTT é seu uso em aplicações de sensoriamento em diferentes tipos de ambientes, incluindo solo, água e corpo humano. Para implantar esses sistemas IVTT nesses ambientes desafiadores, uma abordagem de encapsulamento precisa ser implementada para conter os componentes IVTT e protegê-los da degradação.
As abordagens de encapsulação mais comuns para sistemas de TTIV incluem o uso de cápsulas lipídicas, micelas, polissomas e outros microrecipientes bem fechados10,11,12. Uma desvantagem dessa abordagem é a necessidade de incorporar mecanismos passivos ou ativos para transportar materiais dentro e fora dos contêineres para permitir a comunicação com o ambiente externo e fornecer recursos de sensoriamento. Para superar algumas dessas questões, o estudo aqui relata um método que fornece uma abordagem simples, mas eficaz, para encapsular os materiais de codificação para diferentes projetos de sensores a serem expressos em sistemas IVTT. Esta abordagem baseia-se no uso da deposição Layer-by-Layer (LbL) de um biopolímero na presença dos plasmídeos de interesse para criar microcápsulas ocas com alta porosidade, o que permite que o material genético protegido interaja com os diferentes componentes da TIDIV de escolha. O estudo demonstrou que plasmídeos encapsulados podem direcionar a transcrição e a tradução quando ativados dentro dessa matriz polimérica, como mostrado com a resposta de um aptâmero codificado por plasmídio e um riboswitch para seus alvos correspondentes. Além disso, este revestimento LbL protege os plasmídeos por meses sem quaisquer condições especiais de armazenamento.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Construção do vetor plasmidial.
- Construir um vetor plasmidial (pSALv-RS-GFPa1, 3,4 kb) por amplificação da sequência codificante de um riboswitch teofilina (ThyRS) acoplado com GFPa1 do vetor pJ201:23976-RS-GFPa1 (projetado e criado por DNA2.0) e inserção no vetor de expressão de E. coli , pSAL13. Utilizar iniciadores forward (5'-CGTGGTACCGGTGATACCAGCATCGTCTTGATG-3') e reverse (5'-CGTGCTCAGCTTAAGCCAGCTCGTAG-3') para amplificar a sequência codificadora de ThyRS acoplada à GFPa1 e realizar uma reação de PCR em volume de 50 μL utilizando DNA polimerase de acordo com o protocolo do fabricante14.
- Preparar um gel de agarose a 1% a partir de 0,5 g de agarose, 50 ml de tampão TAE (40 mM Tris Acetato, 1 mM EDTA, pH 8,0) e 3 μL de corante de ADN.
- Misturar alíquota de 5 μL do produto amplificado por PCR com 5 μL de água livre de RNase/DNase e 2 μL de corante de carga de gel 6x e analisar por eletroforese em gel de agarose. Carregue uma escada de DNA (0,1-10,0 kb) como referência. Passe o gel a 120 V até que a linha do corante tenha quase atingido o fundo do gel.
- Visualize os fragmentos de DNA usando um sistema de imagem por transiluminador UV para verificar o tamanho correto do DNA15.
- Purificar o produto de PCR usando um kit de purificação de PCR de acordo com o protocolo do fabricante16.
- Digerir o produto da PCR e o vetor de expressão do pSAL com as enzimas de restrição KpnI e BlpI em uma reação de 15 μL, contendo 10 μL do produto da PCR ou vetor plasmidial (concentração 20-50 ng/μL), 1,5 μL de tampão enzimático 10x, 1 μL de cada enzima e 1,5 μL de água livre de RNase/DNase, a 37 °C por 2 h.
- Adicionar 3 μL de corante de carga de gel 6x à mistura de reação e separar os fragmentos digeridos em um gel de agarose a 1%, conforme descrito nas etapas 1.3-1.5.
- Purificar os fragmentos de DNA utilizando um kit de extração em gel de acordo com o protocolo do fabricante16.
- Ligar o produto da PCR digerida em um vetor plasmidial linearizado digerido, pSAL, usando T4 DNA ligase e tampão ligase suplementado em uma reação de 10 μL contendo 3-20 fmol do vetor digerido, 9-60 fmol do produto da PCR digerido, 2 μL de tampão ligase, 1 μL (1 unidade) de T4 DNA Ligase e água livre de DNase/RNase. Incubar a reação de ligadura a 25 °C durante 3 h.
NOTA: Certifique-se de que o teor total de ADN na mistura de reacção é de 0,01-0,1 μg. - Transformar células competentes de E. coli DH5α com 10 ng da mistura de reação de ligadura de acordo com o protocolo do fabricante17.
- Cultivar as células transformadas a 37 °C durante a noite em placas de ágar-LB suplementadas com ampicilina (100 μg/mL).
- Escolher 3-4 colônias bacterianas da placa e transferir assepticamente cada uma delas para 5 mL de meio LB suplementado com ampicilina (100 μg/mL). Cultivar as culturas durante a noite a 37 °C em uma incubadora de agitação a 225 rpm.
- Pellet as culturas noturnas por centrifugação a 11 x g por 3 min à temperatura ambiente.
- Utilizar kit purificador para purificação dos plasmídeos de acordo com o protocolo do fabricante16.
- Verificar as sequências dos plasmídeos purificados por sequenciamento de DNA. O mapa do plasmídeo e a sequência do construto resultante (pSALv-RS-GFPa1) são mostrados na Figura 1.
2. Purificação do DNA em larga escala.
- Transformar o vetor plasmidial pSALv-RS-GFPa1 (3,4 kb) (codificando riboswitch teofilina acoplado ao gene repórter GFPa1) ou pET28c-F30-2xBrócolis (5,4 kb) (codificando aptâmero de brócolis) em células competentes de E. coli DH5α de acordo com o protocolo do fabricante17.
- Cultivar as células transformadas a 37 °C durante a noite em placas de ágar-LB suplementadas com ampicilina (100 μg/mL) para células transformadas com pSALv-RS-GFPa1 ou canamicina (50 μg/mL) para células transformadas com brócolis pET28c-F30-2x.
- Escolher 3-4 colônias bacterianas da placa e transferir assepticamente cada colônia para 5 mL de meio LB suplementado com antibiótico apropriado (100 μg/mL ampicilina ou 50 μg/mL canamicina). Cultivar as culturas durante a noite a 37 °C em uma incubadora de agitação a 225 rpm.
- Use 3 mL da cultura noturna para inocular em 150 mL de LB suplementado com antibiótico apropriado (100 μg/mL ampicilina ou 50 μg/mL canamicina) e cultivar as culturas durante a noite a 37 °C em uma incubadora de agitação a 225 rpm.
- Pellet as células por centrifugação a ≥3400 x g durante 10 min a 4 °C.
- Utilizar kit purificador para purificação dos plasmídeos de acordo com o protocolo do fabricante16.
- Eluir o DNA com 0,5 mL de água pura livre de DNase/RNase. Medir a concentração de ADN e preparar 1 ml de soluções de stock de ADN (100 ng/μL). Conservar os tubos com ADN a 4 °C até nova utilização.
3. Extração de fibroína de seda e preparação de materiais iniciais.
- Preparar uma solução aquosa de proteína reconstituída de fibroína da seda (SF) a partir de casulos de bicho-da-seda Bombyx mori , de acordo com o procedimento descrito em pormenor noutra parte, para representar 10% da solução de Silk-LiBr18.
- Determinar a concentração final da solução aquosa de SF. Pipetar 0,5 ml de solução de seda para uma placa de Petri de 60 mm, deixar secar a 60 °C e medir o peso da película de seda seca. Divida o peso seco por 0,5 mL para calcular a porcentagem de peso por volume.
- Diluir a solução concentrada de seda com água destilada isenta de DNase/RNase, adicionando lentamente água através de pipeta serológica para obter uma concentração final de 1 mg/ml. Conservar a solução a 4 °C para utilização futura.
- Prepare fibroína de seda fluorescentemente marcada usando um kit de marcação de anticorpos. Utilizar 1 mL de solução de fibroína de seda 2 mg/mL para acoplar os grupos N-terminal α-amino da proteína com um corante derivado ativado por éster do NHS, de acordo com o protocolo do fabricante19.
- Preparar 50 mL de solução aquosa de polietilenoimina (PEI) com uma concentração de 6 mg/mL, ajustar o pH para 4 com HCl (1 M). Filtrar a solução através de uma membrana estéril de 0,2 μm. O armazenamento é possível em condições ambientais por meses.
- Prepare núcleos SiO2 . Pipetar 300 μL de partículas de SiO 2 para um tubo de microcentrífuga de2 mL. Lavar as micropartículas duas vezes com 1 mL de água livre de DNase/RNase por centrifugação a 0,2 x g por 1 min.
4. Realizar deposição camada por camada de uma camada primária, plasmídeos de DNA e camadas de seda.
- Para depositar a camada prima de PEI sobre as micropartículas de SiO2 , adicione 1 mL de solução de PEI ao pellet girado a partir da etapa 3.6 e agite a mistura em condições ambientais em um termomisturador a 800 rpm por 15 min. Lavar as partículas quatro vezes com 1 mL de água deionizada livre de DNase/RNase por centrifugação a 0,2 x g por 1 min.
- Para realizar a deposição da camada de DNA, adicionar 1 mL da solução aquosa de plasmídeos de DNA da etapa 2.7 às micropartículas preparadas com PEI e agitar suavemente a mistura a 4 °C em um termomisturador a 800 rpm por 15 min. Para preparar microcápsulas com diferentes cargas de DNA, ajustar a concentração de plasmídeos de DNA de 50-200 ng/μL usando água destilada livre de DNase/RNase e usar 1 mL dessas soluções para depositar o DNA. Coletar as micropartículas por centrifugação a 0,2 x g por 1 min.
- Marcar os tubos para plasmídeos de DNA que codificam o riboswitch teofilina acoplado com GFPa1 como ThyRS-GFPa1, e plasmídeos de DNA que codificam o aptâmero de Brócolis como BrocApt.
NOTA: Mantenha os tubos de microcentrífuga com DNA no gelo. - Retirar cuidadosamente o sobrenadante e lavar as micropartículas quatro vezes com 1 mL de água destilada sem DNase/RNase, descartando cada vez o sobrenadante após centrifugação a 0,2 x g por 1 min. Realizar todos os experimentos à temperatura ambiente (TR), a menos que seja especificado de outra forma.
- Para efectuar a deposição da camada de fibroína da seda, adicionar 1 ml da solução aquosa de SF reconstituída do passo 3.3 às micropartículas adsorvidas no ADN, agitar suavemente a mistura a 10 °C no termomisturador a 750 rpm durante 15 minutos. Recolher as micropartículas por centrifugação a 0,2 x g durante 1 min a 4 °C, remover o sobrenadante e, em seguida, lavá-las uma vez com 1 ml de água destilada sem DNase/RNase. Repita a centrifugação e descarte o sobrenadante.
NOTA: Durante o experimento, manter a solução de seda sobre gelo para evitar gelificação induzida por temperatura. - Tratar gradualmente as partículas com metanol para induzir a formação de folhas de β na estrutura da proteína da seda. Primeiro, adicionar 0,5 mL de água destilada de DNase/RNase, agitar o tubo de microcentrífuga e, em seguida, adicionar 0,5 mL de metanol a 100%. Agite suavemente as partículas no termomisturador a 10 °C durante 5 minutos. Recolher as partículas por centrifugação a 0,2 x g durante 1 min. Remova o sobrenadante.
- Trate as partículas com metanol para promover a formação de folhas de β e garantir uma forte adsorção física da camada de seda. Adicionar 1 mL de metanol a 100%. Agite suavemente as partículas no termomisturador a 750 rpm durante 10 min a 10 °C.
- Recolher as partículas por centrifugação a 0,2 x g durante 1 min a 4 °C e lavá-las duas vezes com 1 ml de água destilada sem DNase/RNase de cada vez, descartando o sobrenadante e agitando suavemente antes da próxima centrifugação.
- Repita as etapas 4.5-4.8 20 vezes para obter estruturas de casca de núcleo multicamadas de seda. Para a última etapa de deposição, use seda fluorescentemente marcada da etapa 3.4 (Silk-DyLight550, 1 mL).
- Realizar a última etapa de lavagem e manter as micropartículas em 1 mL de água destilada livre de DNase/RNase em condições ambientais.
NOTA: Para evitar a agregação de partículas durante a deposição de camadas de seda, realizar uma inspeção visual da suspensão de partículas e pipetá-la para cima e para baixo usando uma ponta de pipeta de 1 mL para promover uma distribuição homogênea de partículas. - Calcular o número de cópias plasmidiais de DNA encapsuladas em cada microcápsula, NDNA usando a Equação 1:
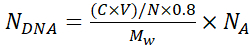 (1)
(1)
Onde N = 6,769 × 1011 - o número de núcleos SiO2 usados para encapsulamento. Calculá-lo a partir de uma curva padrão para concentrações conhecidas de partículas de sílica utilizando diluições seriadas e absorção A 320 a λ =320 nm;
C- concentração inicial de DNA utilizada para adsorção
V - o volume de DNA utilizado para adsorção
0.8- Eficiência de adsorção de DNA nos núcleos
Mw- Peso molecular do plasmídeo de DNA
NA- Número de Avogadro (6.022 × 1023)
5. Dissolução de núcleos para obtenção de microcápsulas de seda.
- Preparar solução de ácido fluorídrico (HF) a 8%, pH 5,5, diluindo a solução-mãe (48%) com água destilada. Adquira um tubo centrífugo de 50 mL. Pipetar cuidadosamente 5 mL de HF e adicionar 25 mL de água destilada para obter solução de HF a 8%.
CUIDADO: A HC é um ácido altamente corrosivo e pode causar queimaduras graves nos tecidos. Extremo cuidado deve ser tomado durante o manuseio e uso de IC para os experimentos. Aderir ao Procedimento Operacional Padrão (POP) para o uso e manuseio adequado do HF desenvolvido pela organização para evitar acidentes indesejáveis por derramamento. Não utilize recipientes de vidro para diluir o ácido HF. Use a coifa química para realizar esta etapa do protocolo. - Dissolver núcleos de SiO2 adicionando 1,5 mL de solução HF a 8% a micropartículas peletizadas core-shell a partir da etapa 4.10. Vórtice suavemente e deixe os núcleos dissolverem-se durante a noite em condições ambientais com agitação suave a 450 rpm.
OBS: Para evitar o derramamento de IC, utilizar fita adesiva para vedação do tubo da microcentrífuga. Use uma capa química para realizar esta etapa do protocolo. - Prepare um copo de vidro de 2 L preenchido com 2 L de água deionizada. Transfira a solução de microcápsulas para dispositivos de diálise (50 kDa MWCO) e dialise-as contra água deionizada com troca repetida da água a cada 3 h pelos próximos 3 dias.
OBS: Recolher o sobrenadante durante as três primeiras trocas de água e descartar a solução de acordo com o protocolo estabelecido para resíduos perigosos. - Use uma pipeta de 1 mL para transferir a suspensão dos dispositivos de diálise para novos tubos de microcentrífuga de 2 mL para coletar as microcápsulas.
NOTA: Conservar as soluções aquosas de microcápsulas em condições ambientais durante vários anos.
6. Imagem de microcápsulas de fibroína da seda usando microscópio confocal de varredura a laser (CLSM).
- Realizar coloração de DNA usando um corante de DNA.
- Transfira 300 μL de microcápsulas ocas de fibroína de seda para um tubo fresco de microcentrífuga de 1 mL. Adicionar 500 μL de água destilada isenta de RNase/DNase.
- Adicionar 5 μL do corante corante de DNA, brevemente vórtice e incubar em RT por 2 h protegido da luz.
- Executar quatro etapas de lavagem por centrifugação a 0,1 x g durante 20 min a 4 °C de cada vez, removendo cuidadosamente 400 μL do sobrenadante e reabastecendo-o com 400 μL de água destilada sem RNase/DNase.
- Realizar a obtenção de imagens de cápsulas de seda em sistemas confocais invertidos equipados com três lasers principais (405 nm, 488 nm, 561 nm) usando objetiva de imersão em óleo de 100x (NA 1.49). Transfira 100 μL da amostra da cápsula para um único poço de lâminas de vidro com câmara de 8 poços, permita que as cápsulas sedimentem por 20-30 minutos antes da obtenção de imagens.
NOTA: Os corantes são muito sensíveis ao fotoclareamento. Proteja as amostras cobrindo as lâminas com papel alumínio.
7. Estimativa da permeabilidade de microcápsulas ocas pelo método de corte de peso molecular (MWCO).
- Preparar 2 mL de cada solução de fluoróforo de dextran marcada com FITC (20 μM, diH 2 O) de diferentes Mw (4 kDa, 20 kDa, 40 kDa, 70 kDa, 150 kDa, 250 kDa, 500 kDa e2MDa).
- Pipetar 100 μL da suspensão das cápsulas para um único poço de uma lâmina de vidro com câmara. Analise cada desenho de microcápsula (concentração de PEI, número de plasmídeos de DNA, concentração de fibroína de seda e número de camadas) separadamente.
- A cada poço, adicione 300 μL da solução específica de fluoróforo a partir do menor Mw até o mais alto, de modo que cada poço corresponda à solução específica de fluoróforo. Misturar pipetando para cima e para baixo e deixar a mistura incubar por 1 h em TR até que a difusão das soluções de fluoróforo atinja o equilíbrio.
- Transfira a lâmina para um microscópio confocal de varredura a laser (CLSM) e obtenha imagens de cada poço usando objetiva de imersão em óleo de 100x na excitação λ = 488 nm.
- Identifique a área de interesse ajustando o plano focal para garantir que as cápsulas apareçam na forma de círculos de maior diâmetro. Isso normalmente acontece ao visualizar as amostras mais próximas do fundo do poço quando as cápsulas sedimentam devido à gravidade.
- Coletar várias imagens de amostras de microcápsulas movendo a lâmina na direção XY. Capture imagens para contabilizar até 100-150 cápsulas para cada amostra.
- Use o software ImageJ para analisar a permeabilidade da membrana da cápsula em cada solução de fluoróforo Mw , comparando as intensidades de fluorescência dentro e fora das cápsulas. Para isso, desenhe uma região de interesse (ROI) em forma de círculo para delinear a circunferência da cápsula e clique em Analisar/Medir para medir a intensidade da fluorescência em seu interior. Tabular os dados em uma planilha. Execute esta operação para cada microcápsula para um total de 200-300 cápsulas.
- Avalie a intensidade da fluorescência externa da mesma forma, delineando a ROI e medindo a intensidade longe das cápsulas. Realizar 3-5 medições para análise estatística.
- Para a análise estatística, comparou-se as intensidades de fluorescência dentro e fora das cápsulas usando o teste t pareado (p < 0,05).
- Utilizar a Tabela de conversão 2 para estimar a permeabilidade de microcápsulas com base nos raios hidrodinâmicos para FITC-Dextran com variável Mw.
8. Ativação in vitro do riboswitch teofilina sintético em microcápsulas de seda
- Preparar 1 mL de solução estoque de teofilina (100 mM, DMSO). Preparar o sistema de extrato de E. coli S30 para DNA circular descongelando os componentes no gelo por 40 min.
- Obter um tubo de microcentrífuga sem DNase/RNase de 0,5 mL. Realizar reação de transcrição/tradução in vitro , combinar os componentes livres de células com uma amostra de microcápsulas na seguinte ordem (volume total de 50 μL): pré-mistura S30 sem aminoácidos (20 μL); Extrato circular de S30 (15 μL); mistura completa de aminoácidos (5 μL); microcápsulas ocas contendo plasmídeos ThyRS-GFPa1 da etapa 4.10 (9 μL); e teofilina, 100 mM DMSO (1 μL).
NOTA: Depois de adicionar todos os componentes, agite brevemente o tubo e colete a amostra durante uma breve centrifugação a 0,2 × g por alguns segundos. - Incubar o tubo a 30 °C durante 4 h e verificar a fluorescência num leitor de placas utilizando excitação a λ = 488 nm e emissão para o filtro GFP/FITC (510 nm ± 20 nm).
- Obtenha imagens das cápsulas em qualquer sistema LCSM usando lasers de 488 nm e 561 nm. Obtenha imagens da melhor qualidade usando objetiva de imersão em óleo de 100x e lâminas com câmara de 8 poços.
9. Ativação in vitro de aptâmero de brócolis em microcápsulas de seda
- Preparar 1 ml de solução-mãe de corante DFHBI-1T (30 μM, diH2O). Prepare o kit de reação do sistema livre de células PURE (síntese de proteínas usando elementos recombinantes) descongelando os componentes no gelo por 40 min.
- Obter um tubo de microcentrífuga sem DNase/RNase de 0,5 mL. Realizar reação de transcrição in vitro combinando componentes da reação livre de células com uma amostra de microcápsulas na seguinte ordem (volume total de 50 μL): solução A (20 μL); solução B (15 μL); microcápsulas ocas contendo plasmídeos BrocApt da etapa 4.10 (14 μL); e corante DFHBI-1T (1 μL).
NOTA: Depois de adicionar todos os componentes, agite brevemente o tubo e colete a amostra durante uma breve centrifugação a 0,2 × g por alguns segundos. - Incubar o tubo a 37 °C durante 6 h e verificar a fluorescência num leitor de placas utilizando excitação a λex = 470 nm e emissão a λem = 510 nm ± 20 nm.
- Obtenha imagens das cápsulas em qualquer sistema LCSM usando lasers de 488 nm e 561 nm. Obtenha as imagens de melhor qualidade usando objetiva de imersão em óleo de 100x e lâminas com câmara de vidro de 8 poços.
Access restricted. Please log in or start a trial to view this content.
Resultados
Aqui, o estudo aborda a funcionalidade de modelos de DNA que codificam diferentes designs de sensores (dois tipos de elementos de transcrição/tradução regulados por RNA) após o encapsulamento em cápsulas de proteína de seda. As microcápsulas foram preparadas através da montagem Layer-by-Layer (LbL) dos componentes-chave: uma camada prime, plasmídeos de DNA que codificam desenhos de sensores e biopolímero de fibroína de seda (Figura 2). A deposição de macromoléculas de forma la...
Access restricted. Please log in or start a trial to view this content.
Discussão
Microcápsulas de hidrogel seletivamente permeáveis carregadas com vários tipos de projetos de sensores codificados por DNA podem ser preparadas seguindo este protocolo. Uma das características distintivas da abordagem LbL é a capacidade de adaptar a complexidade das microcápsulas durante a montagem de baixo para cima, que geralmente começa com a adsorção de espécies moleculares em moldes de sacrifício. Ajustando cuidadosamente as concentrações dos componentes iniciais, as condições de pH e o número de cam...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os pontos de vista e opiniões aqui apresentados são dos autores e não representam necessariamente os pontos de vista do DoD ou de seus Componentes
Agradecimentos
Este trabalho foi apoiado pela bolsa LRIR 16RH3003J do Escritório de Pesquisa Científica da Força Aérea, bem como pelo programa de Pesquisa Aplicada em Biologia Sintética para Ambientes Militares para o Avanço das Prioridades de C&T (ARAP) do Escritório do Subsecretário de Defesa dos EUA para Pesquisa e Engenharia.
A sequência de vetores plasmidiais para ThyRS (pSALv-RS-GFPa1, 3,4 kb) foi generosamente fornecida pelo Dr. J. Gallivan. Casulos de bicho-da-seda de Bombyx mori foram generosamente doados pelo Dr. D.L. Kaplan da Tufts University, MA.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| (Z)-4-(3,5-difluoro-4-hydroxybenzylidene)-2-methyl-1-(2,2,2-trifluoroethyl)-1H-imidazol-5(4 H)-one (DFHBI-1T) | Lucerna | DFHBI-1T | |
| 5x T4 DNA Ligase Buffer | ThermoFisher Scientific | 46300-018 | |
| 6x Blue Gel Loading Dye | New England BioLabs | B7021S | |
| 96-well plates, black circular | Corning | 3601 | |
| Agarose | Sigma-Aldrich | A9539 | BioReagent, for molecular biology, low EEO |
| Ampicillin sodium salt | Sigma-Aldrich | A0166 | powder or crystals, BioReagent, suitable for cell culture |
| BlpI restriction enzymes | New England BioLabs | R0585S | |
| Corning Disposable Vacuum Filter/Storage Systems | FisherScientific | 09-761-1 | |
| Dimethyl sulfoxide, DMSO | Sigma-Aldrich | 472301 | ACS reagent, ≥99.9% |
| DNA Plasmid, pET28c-F30-2x Broccoli (5.4 kb), BrocApt. | Addgene | Plasmid #66788 | |
| DyLightTM550 Antibody Labeling kit (Invitrogen) | ThermoFisher Scientific | 84530 | |
| E. coli S30 extract system for circular DNA | Promega | L1020 | |
| Falcon Conical centrifuge tubes, 15 mL | FisherScientific | 14-959-53A | |
| Falcon Conical centrifuge tubes, 50 mL | 14-432-22 | ||
| Fisherbrand Microcentrifuge tubes, 1.5 mL | FisherScientific | 05-408-129 | |
| Hydrofluoric acid, HF | Sigma-Aldrich | 695068 | ACS reagent, 48% |
| Kanamycin sulfate | Sigma-Aldrich | 60615 | mixture of Kanamycin A (main component) and Kanamycin B and C |
| KpnI restriction enzymes | New England BioLabs | R0142S | |
| LB agar plate supplemented with 100 µg/mL ampicillin | Sigma-Aldrich | L5667 | pre-poured agar plates with 100 µg/mL ampicillin |
| LB agar plate supplemented with 50 µg/mL kanamycin | Sigma-Aldrich | L0543 | pre-poured agar plates with 50 µg/mL kanamycin |
| LB broth (Lennox grade) | Sigma-Aldrich | L3022 | |
| Lithium bromide, LiBr | Sigma-Aldrich | 213225 | ReagentPlus, ≥99% |
| Max Efficiency DH5-α competent E. coli strain | ThermoFisher Scientific | 18258012 | |
| Methanol | MilliporeSigma | 322415 | anhydrous, 99.8% |
| MilliQ-water | EMD MilliPore | Milli-Q Reference Water Purification System | |
| MinElute PCR Purification Kit | Qiagen | 28004 | |
| N-(3-Dimethylaminopropyl)-N′-ethylcarbodiimide hydrochloride, EDC | Sigma-Aldrich | E1769 | |
| PBS (phosphate buffered saline) | ThermoFisher Scientific | 10010023 | 1x PBS, pH 7.4 |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530S | |
| Polyethylenimine, branched | Sigma-Aldrich | 408727 | average Mw ~25,000 |
| PURExpress In Vitro Protein Synthesis Kit | New England BioLabs | E6800S | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | |
| Quick-Load 2-Log DNA Ladder (0.1-10.0 kb) | New England BioLabs | N0469S | |
| SiO? silica microspheres, 4.0 µm | Polysciences, Inc. | 24331-15 | 10% aqueous solution |
| Slide-A-Lyzer G2 Dialysis Cassettes, 3.5K MWCO, 15 mL | ThermoFisher Scientific | 87724 | |
| Sodium carbonate, Na?CO? | Sigma-Aldrich | 222321 | ACS reagent, anhydrous, ≥99.5%, powder |
| Spectrum Spectra/Por Float-A-Lyzer G2 Dialysis Devices | FisherScientific | 08-607-008 | Spectrum G235058 |
| SYBR Safe DNA gel stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase (5 U/µL) | ThermoFisher Scientific | EL0011 | |
| Theophylline | Sigma-Aldrich | T1633 | anhydrous, ≥99%, powder |
| Tris Acetate-EDTA buffer (TAE buffer) | Sigma-Aldrich | T6025 | Contains 40 mM Tris-acetate and 1 mM EDTA, pH 8.3. |
| UltraPure DNase/RNase-Free Distilled Water | FisherScientific | 10-977-023 | |
| ZymoPURE II Plasmid MaxiPrep kit | ZymoResearch | D4202 |
Referências
- Slomovic, S., Pardee, K., Collins, J. J. Synthetic biology devices for in vitro and in vivo diagnostics. Proceedings of the National Academy of Sciences of the United States of America. 112 (47), 14429-14435 (2015).
- Harbaugh, S. V., Goodson, M. S., Dillon, K., Zabarnick, S., Kelley-Loughnane, N. Riboswitch-based reversible dual-color sensor. ACS Synthetic Biology. 6 (5), 766-781 (2017).
- König, H., Frank, D., Heil, R., Coenen, C. Synthetic genomics and synthetic biology applications between hopes and concerns. Current Genomics. 14 (1), 11-24 (2013).
- Silverman, A. D., Karim, A. S., Jewett, M. C. Cell-free gene expression: An expanded repertoire of applications. Nature Reviews Genetics. 21, 151-170 (2020).
- Chushak, Y., et al. Characterization of synthetic riboswitch in cell-free protein expression systems. RNA Biology. , 1-12 (2021).
- Cole, S. D., et al. Quantification of interlaboratory cell-free protein synthesis variability. ACS Synthetic Biology. 8 (9), 2080-2091 (2019).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- Grӓwe, A., et al. A paper-based, cell-free biosensor system for the detection of heavy metals and date rape drugs. PLoS One. 14 (3), 0210940(2019).
- Lin, X., et al. Portable environment-signal detection biosensors with cell-free synthetic biosystems. RSC Advances. 10 (64), 39261-39265 (2020).
- Caschera, F., Lee, J. W., Ho, K. K. Y., Liu, A. P., Jewett, M. C. Cell-free compartmentalized protein synthesis inside double emulsion templated liposomes with in vitro synthesized and assembled ribosomes. Chemical Communications. 52 (31), 5467-5469 (2016).
- Niederholtmeyer, H., Chaggan, C., Devaraj, N. K. Communication and quorum sensing in non-living mimics of eukaryotic cells. Nature Communications. 9, 5027(2018).
- Timin, A. S., Gould, D. J., Sukkhorukov, G. B. Multi-layer microcapsules: Fresh insights and new applications. Expert Opinion on Drug Delivery. 14 (5), 583-587 (2017).
- Bomati, E. K., Haley, J. E., Noel, J. P., Deheyn, D. D. Spectral and structural comparison between bright and dim green fluorescent proteins in Amphioxus. Scientific Reports. 4, 5469(2014).
- Frey, B. Amplification of Genomic DNA by PCR. Molecular Diagnosis of Infectious Diseases. Methods in Molecular Medicine. Reischl, U. 13, Humana Press. Totowa, NJ. 143-156 (1998).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. Journal of Visualized Experiments. (62), e3923(2012).
- Zhou, Y., et al. Rapid regeneration and reuse of silica columns from PCR purification and gel extraction kits. Scientific Reports. 8, 12870(2018).
- Kostylev, M., Otwell, A. E., Richardson, R. E., Suzuki, Y. Cloning should be simple: Escherichia coli DH5α-mediated assembly of multiple DNA fragments with short end homologies. PLoS One. 10 (9), 0137466(2015).
- Rockwood, D. N., et al. Materials fabrication from Bombyx mori silk fibroin. Nature Protocols. 6 (10), 1612-1631 (2011).
- Drachuk, I., et al. Silk macromolecules with amino acid-Poly(Ethylene Glycol) grafts for controlling layer-by-layer encapsulation and aggregation of recombinant bacterial cells. ACS Nano. 9 (2), 1219-1235 (2015).
- Antipov, A. A., Sukhorukov, G. B. Polyelectrolyte multilayer capsules as vehicles with tunable permeability. Advances in Colloid and Interface Science. 111 (1-2), 49-61 (2004).
- Drachuk, I., Harbaugh, S., Chávez, J. L., Kelley-Loughnane, N. Improving the activity of DNA-encoded sensing elements through confinement in silk microcapsules. ACS Applied Materials & Interfaces. 12 (43), 48329-48339 (2020).
- Melnikov, S., Ben-Shem, A., Garreau de Loubresse, N., Jenner, L., Yusupova, G., Yusupov, M. Structural basis for the inhibition of the eukaryotic ribosome. Nature Structural & Molecular Biology. 19 (6), 560-567 (2012).
- Zhao, S., et al. The future of layer-by-layer assembly: A tribute to ACS Nano associate editor Helmuth Möhwald. ACS Nano. 13 (6), 6151-6169 (2019).
- Main, K. H. S., Provan, J. I., Haynes, P. J., Wells, G., Hartley, J. A., Pyne, A. L. B. Atomic force microscopy-A tool for structural and translational DNA research. APL Bioengineering. 5, 031504(2021).
- Riera, R., Feiner-Gracia, N., Fornaguera, C., Cascante, A., Borrós, S., Albertazzi, L. Tracking the DNA complexation state of pBAE polyplexes in cells with super resolution microscopy. Nanoscale. 11 (38), 17869-17877 (2019).
- Bilokapic, S., Strauss, M., Halic, M. Cryo-EM of nucleosome core particle interactions in trans. Scientific Reports. 8, 7046(2018).
- Pritchard, E. M., Dennis, P. B., Omenetto, F., Naik, R. R., Kaplan, D. L. Physical and chemical aspects of stabilization of compounds in silk. Biopolymers. 97 (6), 479-498 (2012).
- Fritz, B. R., Jamil, O. K., Jewett, M. C. Implications of macromolecular crowding and reducing conditions for in vitro ribosome construction. Nucleic Acids Research. 43 (9), 4774-4784 (2015).
- Ge, X., Luo, D., Xu, J. Cell-free protein expression under macromolecular crowding conditions. PLoS One. 6 (12), 28707(2011).
- Cawte, A. D., Unrau, P. J., Rueda, D. S. Live cell imaging of single RNA molecules with fluorogenic mango II arrays. Nature Communications. 11, 1283(2020).
- Chen, X., et al. Visualizing RNA dynamics in live cells with bright and stable fluorescent RNAs. Nature Biotechnology. 37 (11), 1287-1293 (2019).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados