A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
הכנת מיקרוקפסולות רב תכליתיות על בסיס משי עמוסות בפלסמידים DNA המקודדים RNA Aptamers ו-Riboswitches
In This Article
Summary
הפרוטוקול מתאר את היווצרותן של מיקרו-קפסולות חזקות ועמוסות DNA תואמות ביולוגית כחיישנים ביולוגיים מרובי מבחנה המסוגלים לעקוב אחר מספר ליגנדות.
Abstract
אנו מציגים פרוטוקול להכנת מיקרו-קפסולות משי פיברואין עמוסות DNA באמצעות שיטת ההרכבה Layer-by-Layer (LbL) על ליבות כדוריות להקרבה. לאחר ספיחה של שכבה ראשונית ופלסמידים של DNA, היווצרות מיקרוקפסולות חזקות התאפשרה על ידי השראת יריעות β במבנה משני משי במהלך התייבשות חריפה של שכבת משי יחידה. לפיכך, השכבות התרחשו באמצעות קשרי מימן מרובים ואינטראקציות הידרופוביות. עם ספיחה של פגזים רב-שכבתיים, מבני מעטפת הליבה יכולים לתפקד עוד יותר עם ננו-חלקיקי זהב (AuNPs) ו / או נוגדנים (IgG) שישמשו לחישה מרחוק ו / או משלוח ממוקד. התאמת מספר פרמטרים מרכזיים במהלך שיקוע רציף של מקרומולקולות מפתח על ליבות סיליקה כגון נוכחות פריימר פולימרי, ריכוז הדנ"א וחלבון המשי, כמו גם מספר שכבות נספגות, הביאה למיקרו-קפסולות תואמות ביולוגית, עמוסות DNA עם חדירות משתנה ועומסי DNA. עם המסת ליבות סיליקה, הפרוטוקול הדגים היווצרות של מיקרוקפסולות חלולות וחזקות עם פלסמידים DNA משותקים על פני השטח הפנימיים של קרום הקפסולה. יצירת קרום תואם ביולוגית חדיר באופן סלקטיבי בין פלסמידים של הדנ"א לסביבה החיצונית שימרה את הדנ"א במהלך אחסון ארוך טווח ומילאה תפקיד חשוב בתגובת פלט משופרת מפלסמידים מוגבלים מרחבית. פעילותן של תבניות DNA ונגישותן נבדקו במהלך שעתוק חוץ גופי ותגובות תרגום (מערכות נטולות תאים). פלסמידים של DNA המקודדים RNA Light Light aptamers ו riboswitches הופעלו בהצלחה עם אנליטים מתאימים, כפי שהודגם במהלך לוקליזציה של תעתיקי RNA המסומנים באופן פלואורסצנטי או חלבון GFPa1 בקרומי הקליפה.
Introduction
תחום הביולוגיה הסינתטית מציע הזדמנויות ייחודיות לפיתוח יכולות חישה על ידי ניצול מנגנונים טבעיים שפותחו על ידי מיקרואורגניזמים כדי לנטר את סביבתם ואת האיומים הפוטנציאליים שלהם. חשוב לציין שמנגנוני חישה אלה קשורים בדרך כלל לתגובה המגנה על מיקרואורגניזמים אלה מפני חשיפה מזיקה, ומווסתת את ביטוי הגנים כדי למתן השפעות שליליות או למנוע צריכה של חומרים רעילים. נעשו מאמצים משמעותיים להנדס מיקרואורגניזמים אלה כדי ליצור חיישנים של תאים שלמים המנצלים את התגובות הטבעיות הללו, אך מכוונים אותן מחדש לזהות מטרות חדשות ו/או לייצר אות מדיד שניתן למדוד למטרות כימות (בדרך כלל פלואורסצנטיות)1,2. כיום, חששות לגבי השימוש במיקרואורגניזמים מהונדסים גנטית (GMOs), במיוחד כאשר הם משוחררים בסביבה או בגוף האדם, עקב דליפה של תאים שלמים או חלק מהחומר הגנטי שלהם, גם אם הם עטופים במטריצה פולימרית, מציעים כי יש צורך בדרכים חלופיות לנצל גישות חישה אלה3.
גישה רבת עוצמה לניצול היתרונות של חישה מבוססת מיקרואורגניזמים ללא דאגה לפריסה של הנדסה גנטית היא השימוש במערכות שעתוק / תרגום חוץ גופי (IVTT). מבחינה מעשית, מערכות IVTT מורכבות מתערובת המכילה את רוב מרכיבי התא במצב פעיל ש"חולץ" מהתאים באמצעים שונים, כולל סוניקציה, הכאת חרוזים או אחרים4. התוצר הסופי של תהליך זה הוא תערובת תגובה ביוכימית שכבר הותאמה לביצוע שעתוק ותרגום, שניתן להשתמש בה כדי לבדוק חיישנים שונים בפורמט "כלי פתוח", ללא האילוצים הקשורים לשימוש בתאים שלמים (דיפוזיה של ממברנה, יעילות טרנספורמציה, רעילות תאים וכו '). חשוב לציין, ניתן להוסיף רכיבי חיישנים שונים באופן כמותי, והשפעתם נחקרת בטכניקות אופטיות וספקטרומטריות שונות, כפי שהדגמנו5. זה כבר שם לב כי הביצועים של מערכות IVTT יכול להיות עקבי; עם זאת, מחקרים אחרונים הראו גישות לסטנדרטיזציה של ההכנה והאפיון שלהם, וזה עוזר מאוד כאשר לומדים את הביצועים שלהם בעיצוב חיישנים6. לאחרונה הודגמו דוגמאות רבות של מערכות IVTT המשמשות ליצירת בדיקות מבוססות נייר באמצעות ליופיליזציה של מרכיביהן במטריצות נייר, כולל זיהוי יוני מתכות כבדות, תרופות, אלמנטים של חישת מניין ואחרים 7,8,9. מרחב יישומים מרגש עבור חיישנים מבוססי IVTT הוא השימוש בהם ביישומי חישה בסוגים שונים של סביבות, כולל אדמה, מים וגוף האדם. על מנת לפרוס מערכות IVTT אלה בסביבות מאתגרות אלה, יש ליישם גישת אנקפסולציה כדי להכיל את רכיבי IVTT ולהגן עליהם מפני התפרקות.
גישות האנקפסולציה הנפוצות ביותר עבור מערכות IVTT כוללות שימוש בכמוסות שומנים, מיצלים, פולימרזומים ומיקרו-מיכלים סגורים אחרים10,11,12. חיסרון אחד של גישה זו הוא הצורך לשלב מנגנונים פסיביים או אקטיביים להובלת חומרים פנימה והחוצה מהמכולות כדי לאפשר תקשורת עם הסביבה החיצונית ולספק יכולות חישה. כדי להתגבר על חלק מבעיות אלה, המחקר כאן מדווח על שיטה המספקת גישה פשוטה אך יעילה לתמצת את חומרי הקידוד עבור עיצובי חיישנים שונים שיבואו לידי ביטוי במערכות IVTT. גישה זו מבוססת על שימוש בתצהיר שכבה אחר שכבה (LbL) של ביופולימר בנוכחות הפלסמידים המעניינים ליצירת מיקרוקפסולות חלולות עם נקבוביות גבוהה, המאפשרת לחומר הגנטי המוגן לקיים אינטראקציה עם המרכיבים השונים של ה- IVTT לפי בחירה. המחקר הראה כי פלסמידים עטופים יכולים לכוון שעתוק ותרגום כאשר הם מופעלים בתוך מטריצה פולימרית זו, כפי שמוצג עם התגובה של אפטמר מקודד פלסמיד וריבומתג למטרות המתאימות שלהם. בנוסף, ציפוי LbL זה מגן על הפלסמידים למשך חודשים ללא תנאי אחסון מיוחדים.
Protocol
1. בניית וקטור פלסמיד.
- בניית וקטור פלסמיד (pSALv-RS-GFPa1, 3.4 kb) על ידי הגברה של רצף הקידוד של ריבומתג תיאופילין (ThyRS) יחד עם GFPa1 מווקטור pJ201:23976-RS-GFPa1 (תוכנן ונוצר על ידי DNA2.0) והחדרה לווקטור ביטוי E. coli , pSAL13. השתמש בפריימרים קדימה (5'-CGTGGTACCGGTGATACCAGCATCGTCTTGATG-3') והפוך (5'-CGTGCTCAGCTTAAGCCAGCTCGTAG-3') כדי להגביר את רצף הקידוד של ThyRS יחד עם GFPa1 ולבצע תגובת PCR בנפח 50 μL באמצעות DNA פולימראז בהתאם לפרוטוקולהיצרן 14.
- הכינו ג'ל אגרוז 1% מ-0.5 גרם אגרוז, 50 מ"ל של חיץ TAE (40 מ"מ טריס אצטט, 1 מ"מ EDTA, pH 8.0) ו-3 מיקרוליטר של כתם DNA.
- יש לערבב 5 μL aliquot של המוצר המוגבר PCR עם 5 μL של מים נטולי RNase/DNase ו-2 μL של 6x צבע טעינת ג'ל ולנתח על ידי אלקטרופורזה של ג'ל אגרוז. טען סולם DNA (0.1-10.0 kb) כהפניה. הפעל את הג'ל ב 120 V עד קו הצבע כמעט הגיע לתחתית הג'ל.
- דמיינו את מקטעי הדנ"א באמצעות מערכת הדמיה של טרנסילומיטור UV כדי לבדוק את הגודל הנכון של DNA15.
- יש לטהר את מוצר ה-PCR באמצעות ערכת טיהור PCR בהתאם לפרוטוקול היצרן16.
- לעכל את מוצר ה-PCR ואת וקטור ביטוי ה-pSAL עם אנזימי הגבלה KpnI ו-BlpI בתגובה של 15 μL, המכילה 10 μL של מוצר PCR או וקטור פלסמיד (ריכוז 20-50 ng/μL), 1.5 μL של מאגר אנזימים 10x, 1 μL של כל אנזים, ו-1.5 μL של מים נטולי RNase/DNase, ב-37°C למשך שעתיים.
- הוסף 3 μL של צבע העמסת ג'ל 6x לתערובת התגובה והפרד את המקטעים המעוכלים על ג'ל אגרוז 1% כמתואר בשלבים 1.3-1.5.
- לטהר את מקטעי הדנ"א באמצעות ערכת מיצוי ג'ל בהתאם לפרוטוקולהיצרן 16.
- חבר את מוצר ה- PCR המעוכל לווקטור פלסמיד ליניארי מעוכל, pSAL, באמצעות ליגאז DNA T4 ומאגר ליגאז משלים בתגובה של 10 μL המכילה 3-20 fmol של הווקטור המעוכל, 9-60 fmol של מוצר PCR מעוכל, 2 μL של מאגר ליגאז, 1 μL (יחידה אחת) של T4 DNA Ligase, ומים נטולי DNase/RNase. לדגור על תגובת הקשירה ב 25 ° C במשך 3 שעות.
הערה: ודא שתכולת הדנ"א הכוללת בתערובת התגובה היא 0.01-0.1 מיקרוגרם. - התמירו תאים מוכשרים E. coli DH5α עם 10 ng של תערובת תגובת הקשירה בהתאם לפרוטוקולהיצרן 17.
- גדל את התאים מותמרים ב 37 ° C במשך הלילה על לוחות LB-אגר בתוספת ampicillin (100 מיקרוגרם / מ"ל).
- בחרו 3-4 מושבות חיידקים מהצלחת והעבירו כל אחת מהן ל-5 מ"ל של חומר LB בתוספת אמפיצילין (100 מיקרוגרם/מ"ל). לגדל את התרביות בן לילה ב 37 מעלות צלזיוס באינקובטור רועד ב 225 סל"ד.
- מטילים את תרביות הלילה באמצעות צנטריפוגה במהירות של 11XG למשך 3 דקות בטמפרטורת החדר.
- השתמש בערכת טיהור כדי לטהר את הפלסמידים בהתאם לפרוטוקול היצרן16.
- אמת את הרצפים של פלסמידים מטוהרים על ידי ריצוף DNA. מפת הפלסמיד ורצף המבנה שנוצר (pSALv-RS-GFPa1) מוצגים באיור 1.
2. טיהור DNA בקנה מידה גדול.
- הפוך את וקטור הפלסמיד pSALv-RS-GFPa1 (3.4 kb) (קידוד ריבומתג תאופילין יחד עם גן כתב GFPa1) או pET28c-F30-2xBroccoli (5.4 kb) (קידוד אפטמר ברוקולי) לתאים מוסמכים E. coli DH5α על פי פרוטוקולהיצרן 17.
- גדלו את התאים שעברו טרנספורמציה ב-37°C למשך הלילה על צלחות LB-אגר בתוספת אמפיצילין (100 מיקרוגרם/מ"ל) עבור תאים שעברו טרנספורמציה עם pSALv-RS-GFPa1 או קנמיצין (50 מיקרוגרם/מ"ל) עבור תאים שעברו טרנספורמציה עם pET28c-F30-2x ברוקולי.
- בחרו 3-4 מושבות חיידקים מהצלחת והעבירו כל מושבה ל-5 מ"ל של חומר LB בתוספת אנטיביוטיקה מתאימה (100 מיקרוגרם/מ"ל אמפיצילין או 50 מק"ג/מ"ל קנמיצין). לגדל את התרביות בן לילה ב 37 מעלות צלזיוס באינקובטור רועד ב 225 סל"ד.
- השתמש 3 מ"ל של תרבית לילה כדי לחסן לתוך 150 מ"ל של LB בתוספת אנטיביוטיקה מתאימה (100 מיקרוגרם / מ"ל אמפיצילין או 50 מיקרוגרם / מ"ל kanamycin) ולגדל את התרביות לילה ב 37 ° C באינקובטור רועד ב 225 סל"ד.
- גלולה את התאים על ידי צנטריפוגה ב ≥3400 x גרם במשך 10 דקות ב 4 ° C.
- השתמש בערכת טיהור כדי לטהר את הפלסמידים בהתאם לפרוטוקול היצרן16.
- יש להקפיד על הדנ"א עם 0.5 מ"ל של מים טהורים נטולי DNase/RNase. למדוד את ריכוז הדנ"א ולהכין 1 מ"ל של תמיסות מלאי DNA (100 ng/μL). אחסנו את הצינורות עם DNA בטמפרטורה של 4°C עד לשימוש נוסף.
3. מיצוי פיברואין משי והכנת חומרים ראשוניים.
- הכינו תמיסה מימית של חלבון פיברואין משי משוחזר (SF) מפקעות תולעי משי של בומביקס מורי בהתאם להליך המתואר בפירוט במקומות אחרים כדי להוות 10% מתמיסת משי-ליבר18.
- לקבוע את הריכוז הסופי של תמיסת SF מימית. פיפטה 0.5 מ"ל של תמיסת משי לצלחת פטרי 60 מ"מ, לתת לו להתייבש ב 60 מעלות צלזיוס, ולמדוד את המשקל של סרט משי יבש. חלק את המשקל היבש ב- 0.5 מ"ל כדי לחשב את המשקל לאחוז נפח.
- דללו את תמיסת המשי המרוכזת במים מזוקקים ללא DNase/RNase על ידי הוספה איטית של מים דרך פיפטה סרולוגית לקבלת ריכוז סופי של 1 מ"ג/מ"ל. אחסנו את התמיסה בטמפרטורה של 4°C לשימוש עתידי.
- הכינו פיברואין משי עם תווית פלואורסצנטית באמצעות ערכת תיוג נוגדנים. השתמש 1 מ"ל של 2 מ"ג / מ"ל תמיסת פיברואין משי כדי להתאים את קבוצות N-terminal α-amino של החלבון עם צבע נגזרת מופעל אסטר NHS על פי פרוטוקולהיצרן 19.
- הכן 50 מ"ל של תמיסה מימית פוליאתילנימין (PEI) עם ריכוז של 6 מ"ג / מ"ל, להתאים pH ל 4 עם HCl (1 M). מסננים את התמיסה דרך קרום סטרילי של 0.2 מיקרומטר. אחסון אפשרי בתנאי סביבה במשך חודשים.
- הכינו SiO2 ליבות. פיפטה 300 μL של SiO2 חלקיקים לתוך צינור מיקרוצנטריפוגה 2 מ"ל. שטפו את המיקרו-חלקיקים פעמיים עם 1 מ"ל של מים נטולי DNase/RNase באמצעות צנטריפוגה במהירות של 0.2 x גרם למשך דקה אחת.
4. בצעו שיקוע שכבה אחר שכבה של שכבה ראשונית, פלסמידים של DNA ושכבות משי.
- כדי להפקיד את השכבה הראשונית של PEI על מיקרו-חלקיקי SiO2 , הוסף 1 מ"ל של תמיסת PEI לכדורית המסובבת למטה משלב 3.6 והתסיס את התערובת בתנאי סביבה על תרמומיקסר במהירות 800 סל"ד למשך 15 דקות. שטפו את החלקיקים ארבע פעמים עם 1 מ"ל של מים נטולי DNase/RNase באמצעות צנטריפוגה במהירות של 0.2 x גרם למשך דקה אחת.
- כדי לבצע שיקוע של שכבת ה- DNA, הוסף 1 מ"ל של התמיסה המימית של פלסמידים DNA משלב 2.7 למיקרו-חלקיקים הדרוכים ב- PEI ועורר בעדינות את התערובת ב- 4 ° C על תרמומיקסר ב- 800 סל"ד למשך 15 דקות. כדי להכין מיקרו-קפסולות עם עומסי דנ"א שונים, התאימו את ריכוז פלסמידים של DNA מ-50-200 ננוגרם/מיקרוליטר באמצעות מים מזוקקים נטולי DNase/RNase והשתמשו ב-1 מ"ל של תמיסות אלה כדי להפקיד את הדנ"א. אסוף את המיקרו-חלקיקים באמצעות צנטריפוגה במהירות של 0.2 x גרם למשך דקה אחת.
- סמן את הצינורות עבור פלסמידים DNA המקודדים ריבומתג תיאופילין יחד עם GFPa1 כמו ThyRS-GFPa1, ופלסמידים DNA המקודדים ברוקולי aptamer כמו BrocApt.
הערה: שמור את צינורות המיקרוצנטריפוגות עם DNA על קרח. - הסר בזהירות את הסופרנאטנט ושטוף את המיקרו-חלקיקים ארבע פעמים עם 1 מ"ל מים מזוקקים ללא DNase/RNase, ובכל פעם השליכו את הסופרנאטנט לאחר צנטריפוגה ב 0.2 x גרם למשך דקה אחת. בצע את כל הניסויים בטמפרטורת החדר (RT) אלא אם צוין אחרת.
- כדי לבצע שיקוע של שכבת פיברואין משי להוסיף 1 מ"ל של תמיסת SF מימית משוחזרת משלב 3.3 למיקרו-חלקיקים נספגים DNA, מערבלים בעדינות ולעורר את התערובת ב 10 ° C על thermomixer ב 750 סל"ד במשך 15 דקות. אספו את המיקרו-חלקיקים באמצעות צנטריפוגה בטמפרטורה של 0.2 x גרם למשך דקה אחת ב-4°C, הסירו את הסופר-נטנט ולאחר מכן שטפו אותם פעם אחת עם 1 מ"ל של מים מזוקקים ללא DNase/RNase. חזור על הצנטריפוגה והשליך את הסופרנאטנט.
הערה: במהלך הניסוי, שמור את תמיסת המשי על קרח כדי למנוע ג'לציה הנגרמת על ידי טמפרטורה. - טפלו בהדרגה בחלקיקים עם מתנול כדי לגרום להיווצרות β יריעות במבנה חלבון משי. ראשית, להוסיף 0.5 מ"ל של מים מזוקקים DNase/RNase, מערבל את צינור microcentrifuge, ולאחר מכן להוסיף 0.5 מ"ל של 100% מתנול. נערו בעדינות את החלקיקים על התרמומיקסר בטמפרטורה של 10°C למשך 5 דקות. לאסוף את החלקיקים על ידי צנטריפוגה ב 0.2 x גרם במשך 1 דקה. הסר את supernatant.
- טפלו בחלקיקים עם מתנול כדי לקדם היווצרות יריעות β ולהבטיח ספיחה פיזית חזקה של שכבת המשי. הוסף 1 מ"ל של 100% מתנול. נערו בעדינות את החלקיקים על התרמיקסר ב-750 סל"ד למשך 10 דקות ב-10°C.
- אספו את החלקיקים באמצעות צנטריפוגה בטמפרטורה של 0.2 x גרם למשך דקה אחת ב-4°C ושטפו אותם פעמיים עם 1 מ"ל של מים מזוקקים נטולי DNase/RNase בכל פעם, השליכו את הסופרנאטנט וערברו בעדינות לפני הצנטריפוגה הבאה.
- חזור על שלבים 4.5-4.8 20 פעמים כדי להשיג מבני מעטפת ליבה רב-שכבתיים ממשי. לשלב התצהיר האחרון, השתמש במשי עם תווית ניאון משלב 3.4 (Silk-DyLight550, 1 מ"ל).
- בצעו את שלב השטיפה האחרון ושמרו את המיקרו-חלקיקים ב-1 מ"ל של מים מזוקקים נטולי DNase/RNase בתנאי הסביבה.
הערה: כדי למנוע הצטברות של חלקיקים במהלך שיקוע שכבות משי, בצע בדיקה חזותית של תרחיף החלקיקים ופיפטה אותו למעלה ולמטה באמצעות קצה פיפטה 1 מ"ל כדי לקדם פיזור חלקיקים הומוגני. - חשב את מספר עותקי פלסמיד הדנ"א העטופים בכל מיקרו-כמוסה, NDNA באמצעות משוואה 1:
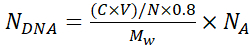 (1)
(1)
כאשר N = 6.769 × 1011 - מספר ליבות SiO2 המשמשות לכימוס. חישוב זה מתוך עקומה סטנדרטית עבור ריכוזים ידועים של חלקיקי סיליקה באמצעות דילול סדרתי ובליעה A 320 ב λ =320 ננומטר;
C- ריכוז ראשוני של DNA המשמש לספיחה
V- נפח הדנ"א המשמש לספיחה
0.8- יעילות ספיחת DNA על הליבות
Mw- משקל מולקולרי של פלסמיד DNA
NA- מספרו של אבוגדרו (6.022 × 1023)
5. פירוק ליבות להשגת מיקרוקפסולות משי.
- הכינו תמיסת 8% חומצה הידרופלואורית (HF), pH 5.5, על ידי דילול תמיסת ציר (48%) במים מזוקקים. לרכוש צינור צנטריפוגה 50 מ"ל. בזהירות פיפטה 5 מ"ל של HF ולהוסיף 25 מ"ל של מים מזוקקים כדי לקבל 8% תמיסת HF.
אזהרה: HF היא חומצה מאכל מאוד ועלולה לגרום לכוויות חמורות ברקמות. יש לנקוט משנה זהירות במהלך הטיפול והשימוש ב-HF לצורך הניסויים. יש לדבוק בנוהל הפעלה סטנדרטי (SOP) לשימוש וטיפול נאותים ב- HF שפותח על ידי הארגון כדי למנוע תאונות שפיכה בלתי רצויות. אין להשתמש במיכלי זכוכית כדי לדלל חומצה HF. השתמש במכסה המנוע הכימי כדי לבצע שלב זה של הפרוטוקול. - המסת ליבות SiO2 על ידי הוספת 1.5 מ"ל של תמיסת HF 8% למיקרו-חלקיקי מעטפת ליבה כדוריים משלב 4.10. מערבלים בעדינות ומניחים לליבות להתמוסס במהלך הלילה בתנאי סביבה עם רעידות עדינות ב-450 סל"ד.
הערה: כדי למנוע שפיכה של HF, השתמש בסרט השתלה כדי לאטום את צינור המיקרוצנטריפוגה. השתמש במכסה מנוע כימי כדי לבצע שלב זה של הפרוטוקול. - הכינו זכוכית בנפח 2 ליטר מלאה ב-2 ליטר מים שעברו דה-יוניזציה. להעביר פתרון microcapsules למכשירי דיאליזה (50 kDa MWCO) ו dialyze אותם נגד מים deionized עם שינוי חוזר של המים כל 3 שעות במשך 3 ימים הבאים.
הערה: יש לאסוף את הסופרנאטנט במהלך שלושת חילופי המים הראשונים ולהשליך את התמיסה בהתאם לפרוטוקול שנקבע לחומרי פסולת מסוכנים. - השתמש פיפטה 1 מ"ל כדי להעביר את המתלה ממכשירי דיאליזה לתוך צינורות מיקרוצנטריפוגות 2 מ"ל חדשים לאסוף את microcapsules.
הערה: יש לאחסן את התמיסות המימיות של מיקרו-קפסולות בתנאי סביבה למשך מספר שנים.
6. הדמיה של מיקרוקפסולות משי פיברואין באמצעות מיקרוסקופ סריקת לייזר קונפוקלי (CLSM).
- בצעו צביעת DNA באמצעות צבע DNA.
- מעבירים 300 μL של מיקרוקפסולות פיברואין משי חלולות לתוך צינור מיקרוצנטריפוגה טרי של 1 מ"ל. הוסף 500 μL של מים מזוקקים ללא RNase/DNase.
- הוסיפו 5 μL של צבע מכתים DNA, מערבולות קצרות, ודגרו ב-RT למשך שעתיים מוגנות מפני אור.
- בצע ארבעה שלבי שטיפה באמצעות צנטריפוגה במהירות של 0.1 x גרם למשך 20 דקות ב-4°C בכל פעם, תוך הסרה זהירה של 400 μL של הסופרנאטנט וחידושו ב-400 μL של מים מזוקקים נטולי RNase/DNase.
- בצע הדמיה של קפסולות משי על מערכות קונפוקליות הפוכות המצוידות בשלושה לייזרים עיקריים (405 ננומטר, 488 ננומטר, 561 ננומטר) באמצעות יעד טבילה בשמן 100x (NA 1.49). העבירו 100 μL של דגימת הקפסולה לבאר אחת של שקופיות זכוכית עם 8 תאים היטב, אפשרו לקפסולות לשקוע במשך 20-30 דקות לפני ההדמיה.
הערה: צבעים רגישים מאוד לפוטו-אקונומיקה. הגן על הדגימות על ידי כיסוי המגלשות ברדיד אלומיניום.
7. הערכת החדירות של מיקרו-קפסולות חלולות בשיטת MWCO (Molecular Weight Cut-Off).
- הכינו 2 מ"ל כל אחת מתמיסות הפלואורופורים של דקסטרן (20 μM, diH 2 O) של Mw שונים (4 kDa, 20 kDa, 40 kDa, 70 kDa, 150 kDa, 250 kDa, 500 kDaו-2MDa).
- פיפטה 100 μL של השעיית קפסולות לתוך באר אחת של מגלשת זכוכית קאמרית. נתח כל עיצוב מיקרוקפסולה (ריכוז PEI, מספר טעינת פלסמידים DNA, ריכוז סיבי משי ומספר השכבות) בנפרד.
- לכל באר, הוסף 300 μL של תמיסת פלואורופור ספציפית החל מ Mw הנמוך ביותר עד הגבוה ביותר, כך שכל באר תתאים לתמיסת פלואורופור ספציפית. מערבבים על ידי פיפטינג למעלה ולמטה ונותנים לתערובת לדגור במשך שעה אחת ב- RT עד שהדיפוזיה של תמיסות פלואורופור מגיעה לשיווי משקל.
- העבר את השקופית למיקרוסקופ סריקת לייזר קונפוקלי (CLSM) וצלם כל באר באמצעות יעד טבילת שמן 100x בעירור λ = 488 ננומטר.
- זהה את אזור העניין על ידי התאמת מישור המוקד כדי לוודא שהקפסולות מופיעות בצורה של מעגלים בקוטר הגדול ביותר. זה קורה בדרך כלל כאשר מסתכלים על הדגימות קרוב יותר לתחתית הבאר כאשר קפסולות משקעים עקב כוח הכבידה.
- אסוף מספר תמונות של דגימות מיקרוקפסולה על-ידי הזזת השקופית בכיוון XY. צלם תמונות כדי לקחת בחשבון עד 100-150 כמוסות עבור כל דגימה.
- השתמש בתוכנת ImageJ כדי לנתח את החדירות של קרום הקפסולה בכל תמיסת Mw fluorophore על ידי השוואת עוצמות פלואורסצנטיות בתוך ומחוץ לקפסולות. לשם כך, ציירו אזור עניין (ROI) בצורת עיגול כדי לתאר את היקף הקפסולה ולחצו על אנליזה/מדידה כדי למדוד את עוצמת הפלואורסצנטיות בפנים. טבלה של הנתונים בגיליון אלקטרוני. בצע פעולה זו עבור כל microcapsule עבור סך של 200-300 כמוסות.
- הערך את עוצמת הפלואורסצנטיות החיצונית באותו אופן על ידי מתאר החזר ההשקעה ומדידת העוצמה הרחק מהכמוסות. בצע 3-5 מדידות לניתוח סטטיסטי.
- כדי לבצע ניתוח סטטיסטי, השווה את עוצמות הפלואורסצנטיות בתוך ומחוץ לקפסולות באמצעות מבחן t זוגי (p < 0.05).
- השתמש בטבלת ההמרה 2 כדי להעריך את החדירות של מיקרוקפסולות בהתבסס על הרדיוסים ההידרודינמיים עבור FITC-Dextran עם משתנה Mw.
8. הפעלה חוץ גופית של ריבומתג תיאופילין סינתטי במיקרו-קפסולות משי
- הכן 1 מ"ל של תמיסת מלאי תיאופילין (100 mM, DMSO). הכינו את מערכת תמצית E. coli S30 לדנ"א מעגלי על ידי הפשרת הרכיבים על קרח למשך 40 דקות.
- השג צינור מיקרוצנטריפוגה נטול DNase/RNase של 0.5 מ"ל. בצע שעתוק חוץ גופי /תגובת תרגום, שלב את הרכיבים נטולי התאים עם דגימה של מיקרוקפסולות בסדר הבא (נפח כולל של 50 מיקרוליטר): S30 premix ללא חומצות אמינו (20 μL); תמצית S30, מעגלית (15 מיקרוליטר); תערובת חומצות אמינו מלאה (5 מיקרוליטר); מיקרוקפסולות חלולות המכילות פלסמידים ThyRS-GFPa1 משלב 4.10 (9 μL); ותאופילין, 100 mM DMSO (1 μL).
הערה: לאחר הוספת כל הרכיבים, מערבלים בקצרה את הצינור ואוספים את הדגימה במהלך צנטריפוגה קצרה ב 0.2 × גרם למשך מספר שניות. - לדגור על הצינור ב 30 ° C במשך 4 שעות ולבדוק את הפלואורסצנטיות על קורא לוחות באמצעות עירור ב λ = 488 ננומטר פליטה עבור מסנן GFP/FITC (510 ננומטר ± 20 ננומטר).
- דמיינו את הקפסולות בכל מערכת LCSM באמצעות לייזרים של 488 ננומטר ו-561 ננומטר. השג את התמונות באיכות הטובה ביותר באמצעות המטרה טבילת שמן 100x ושקופיות עם 8 תאים היטב.
9. הפעלה חוץ גופית של אפטמר ברוקולי במיקרוקפסולות משי
- הכן 1 מ"ל של תמיסת מלאי של צבע DFHBI-1T (30 מיקרומטר, diH2O). הכינו את ערכת תגובת המערכת נטולת התאים PURE (סינתזת חלבונים באמצעות אלמנטים רקומביננטיים) על ידי הפשרת הרכיבים על קרח למשך 40 דקות.
- השג צינור מיקרוצנטריפוגה נטול DNase/RNase של 0.5 מ"ל. בצע תגובת שעתוק חוץ גופית על ידי שילוב רכיבי תגובה ללא תאים עם מדגם של מיקרוקפסולות בסדר הבא (נפח כולל של 50 מיקרוליטר): תמיסה A (20 μL); פתרון B (15 מיקרוליטר); מיקרוקפסולות חלולות המכילות פלסמידים BrocApt משלב 4.10 (14 μL); וצבע DFHBI-1T (1 μL).
הערה: לאחר הוספת כל הרכיבים, מערבלים בקצרה את הצינור ואוספים את הדגימה במהלך צנטריפוגה קצרה ב 0.2 × גרם למשך מספר שניות. - לדגור על הצינור ב 37 ° C במשך 6 שעות ולבדוק את הפלואורסצנטיות על קורא צלחת באמצעות עירור ב λex = 470 ננומטר פליטה ב λem = 510 ננומטר ± 20 ננומטר.
- דמיינו את הקפסולות בכל מערכת LCSM באמצעות לייזרים של 488 ננומטר ו-561 ננומטר. השג את התמונות באיכות הטובה ביותר באמצעות מטרה טבילת שמן של 100x, ושקופיות עם 8 בארות מזכוכית כיסוי.
תוצאות
כאן, המחקר מתייחס לפונקציונליות של תבניות DNA המקודדות עיצובי חיישנים שונים (שני סוגים של רכיבי שעתוק/תרגום מווסתים של RNA) לאחר אנקפסולציה בקפסולות חלבון משי. מיקרו-קפסולות הוכנו באמצעות הרכבה של שכבה אחר שכבה (LbL) של רכיבי המפתח: שכבה ראשונית, פלסמידים של דנ"א המקודדים עיצובים של חיישנים, ובי...
Discussion
ניתן להכין מיקרו-קפסולות הידרוג'ל חדירות באופן סלקטיבי העמוסות בסוגים שונים של עיצובי חיישנים המקודדים בדנ"א בעקבות פרוטוקול זה. אחד המאפיינים הייחודיים של גישת LbL הוא היכולת להתאים את המורכבות של מיקרוקפסולות במהלך ההרכבה מלמטה למעלה, אשר בדרך כלל מתחיל עם ספיחה של מינים מולקולריים על ת?...
Disclosures
הדעות והדעות המובאות במסמך זה הן של המחברים ואינן מייצגות בהכרח את השקפותיו של משרד ההגנה או מרכיביו
Acknowledgements
עבודה זו נתמכה על ידי מענק LRIR 16RH3003J ממשרד חיל האוויר למחקר מדעי, כמו גם על ידי ביולוגיה סינתטית לסביבות צבאיות מחקר יישומי לקידום סדרי עדיפויות S&T (ARAP) של המשרד האמריקני של תת מזכיר ההגנה למחקר והנדסה.
רצף וקטור הפלסמיד עבור ThyRS (pSALv-RS-GFPa1, 3.4 kb) סופק בנדיבות על ידי ד"ר ג'יי גליבן. פקעות תולעי משי מבומביקס מורי נתרמו בנדיבות על ידי ד"ר ד.ל. קפלן מאוניברסיטת טאפטס, מסצ'וסטס.
Materials
| Name | Company | Catalog Number | Comments |
| (Z)-4-(3,5-difluoro-4-hydroxybenzylidene)-2-methyl-1-(2,2,2-trifluoroethyl)-1H-imidazol-5(4 H)-one (DFHBI-1T) | Lucerna | DFHBI-1T | |
| 5x T4 DNA Ligase Buffer | ThermoFisher Scientific | 46300-018 | |
| 6x Blue Gel Loading Dye | New England BioLabs | B7021S | |
| 96-well plates, black circular | Corning | 3601 | |
| Agarose | Sigma-Aldrich | A9539 | BioReagent, for molecular biology, low EEO |
| Ampicillin sodium salt | Sigma-Aldrich | A0166 | powder or crystals, BioReagent, suitable for cell culture |
| BlpI restriction enzymes | New England BioLabs | R0585S | |
| Corning Disposable Vacuum Filter/Storage Systems | FisherScientific | 09-761-1 | |
| Dimethyl sulfoxide, DMSO | Sigma-Aldrich | 472301 | ACS reagent, ≥99.9% |
| DNA Plasmid, pET28c-F30-2x Broccoli (5.4 kb), BrocApt. | Addgene | Plasmid #66788 | |
| DyLightTM550 Antibody Labeling kit (Invitrogen) | ThermoFisher Scientific | 84530 | |
| E. coli S30 extract system for circular DNA | Promega | L1020 | |
| Falcon Conical centrifuge tubes, 15 mL | FisherScientific | 14-959-53A | |
| Falcon Conical centrifuge tubes, 50 mL | 14-432-22 | ||
| Fisherbrand Microcentrifuge tubes, 1.5 mL | FisherScientific | 05-408-129 | |
| Hydrofluoric acid, HF | Sigma-Aldrich | 695068 | ACS reagent, 48% |
| Kanamycin sulfate | Sigma-Aldrich | 60615 | mixture of Kanamycin A (main component) and Kanamycin B and C |
| KpnI restriction enzymes | New England BioLabs | R0142S | |
| LB agar plate supplemented with 100 µg/mL ampicillin | Sigma-Aldrich | L5667 | pre-poured agar plates with 100 µg/mL ampicillin |
| LB agar plate supplemented with 50 µg/mL kanamycin | Sigma-Aldrich | L0543 | pre-poured agar plates with 50 µg/mL kanamycin |
| LB broth (Lennox grade) | Sigma-Aldrich | L3022 | |
| Lithium bromide, LiBr | Sigma-Aldrich | 213225 | ReagentPlus, ≥99% |
| Max Efficiency DH5-α competent E. coli strain | ThermoFisher Scientific | 18258012 | |
| Methanol | MilliporeSigma | 322415 | anhydrous, 99.8% |
| MilliQ-water | EMD MilliPore | Milli-Q Reference Water Purification System | |
| MinElute PCR Purification Kit | Qiagen | 28004 | |
| N-(3-Dimethylaminopropyl)-N′-ethylcarbodiimide hydrochloride, EDC | Sigma-Aldrich | E1769 | |
| PBS (phosphate buffered saline) | ThermoFisher Scientific | 10010023 | 1x PBS, pH 7.4 |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530S | |
| Polyethylenimine, branched | Sigma-Aldrich | 408727 | average Mw ~25,000 |
| PURExpress In Vitro Protein Synthesis Kit | New England BioLabs | E6800S | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | |
| Quick-Load 2-Log DNA Ladder (0.1-10.0 kb) | New England BioLabs | N0469S | |
| SiO? silica microspheres, 4.0 µm | Polysciences, Inc. | 24331-15 | 10% aqueous solution |
| Slide-A-Lyzer G2 Dialysis Cassettes, 3.5K MWCO, 15 mL | ThermoFisher Scientific | 87724 | |
| Sodium carbonate, Na?CO? | Sigma-Aldrich | 222321 | ACS reagent, anhydrous, ≥99.5%, powder |
| Spectrum Spectra/Por Float-A-Lyzer G2 Dialysis Devices | FisherScientific | 08-607-008 | Spectrum G235058 |
| SYBR Safe DNA gel stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase (5 U/µL) | ThermoFisher Scientific | EL0011 | |
| Theophylline | Sigma-Aldrich | T1633 | anhydrous, ≥99%, powder |
| Tris Acetate-EDTA buffer (TAE buffer) | Sigma-Aldrich | T6025 | Contains 40 mM Tris-acetate and 1 mM EDTA, pH 8.3. |
| UltraPure DNase/RNase-Free Distilled Water | FisherScientific | 10-977-023 | |
| ZymoPURE II Plasmid MaxiPrep kit | ZymoResearch | D4202 |
References
- Slomovic, S., Pardee, K., Collins, J. J. Synthetic biology devices for in vitro and in vivo diagnostics. Proceedings of the National Academy of Sciences of the United States of America. 112 (47), 14429-14435 (2015).
- Harbaugh, S. V., Goodson, M. S., Dillon, K., Zabarnick, S., Kelley-Loughnane, N. Riboswitch-based reversible dual-color sensor. ACS Synthetic Biology. 6 (5), 766-781 (2017).
- König, H., Frank, D., Heil, R., Coenen, C. Synthetic genomics and synthetic biology applications between hopes and concerns. Current Genomics. 14 (1), 11-24 (2013).
- Silverman, A. D., Karim, A. S., Jewett, M. C. Cell-free gene expression: An expanded repertoire of applications. Nature Reviews Genetics. 21, 151-170 (2020).
- Chushak, Y., et al. Characterization of synthetic riboswitch in cell-free protein expression systems. RNA Biology. , 1-12 (2021).
- Cole, S. D., et al. Quantification of interlaboratory cell-free protein synthesis variability. ACS Synthetic Biology. 8 (9), 2080-2091 (2019).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- Grӓwe, A., et al. A paper-based, cell-free biosensor system for the detection of heavy metals and date rape drugs. PLoS One. 14 (3), 0210940 (2019).
- Lin, X., et al. Portable environment-signal detection biosensors with cell-free synthetic biosystems. RSC Advances. 10 (64), 39261-39265 (2020).
- Caschera, F., Lee, J. W., Ho, K. K. Y., Liu, A. P., Jewett, M. C. Cell-free compartmentalized protein synthesis inside double emulsion templated liposomes with in vitro synthesized and assembled ribosomes. Chemical Communications. 52 (31), 5467-5469 (2016).
- Niederholtmeyer, H., Chaggan, C., Devaraj, N. K. Communication and quorum sensing in non-living mimics of eukaryotic cells. Nature Communications. 9, 5027 (2018).
- Timin, A. S., Gould, D. J., Sukkhorukov, G. B. Multi-layer microcapsules: Fresh insights and new applications. Expert Opinion on Drug Delivery. 14 (5), 583-587 (2017).
- Bomati, E. K., Haley, J. E., Noel, J. P., Deheyn, D. D. Spectral and structural comparison between bright and dim green fluorescent proteins in Amphioxus. Scientific Reports. 4, 5469 (2014).
- Frey, B., Reischl, U. Amplification of Genomic DNA by PCR. Molecular Diagnosis of Infectious Diseases. Methods in Molecular Medicine. 13, 143-156 (1998).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. Journal of Visualized Experiments. (62), e3923 (2012).
- Zhou, Y., et al. Rapid regeneration and reuse of silica columns from PCR purification and gel extraction kits. Scientific Reports. 8, 12870 (2018).
- Kostylev, M., Otwell, A. E., Richardson, R. E., Suzuki, Y. Cloning should be simple: Escherichia coli DH5α-mediated assembly of multiple DNA fragments with short end homologies. PLoS One. 10 (9), 0137466 (2015).
- Rockwood, D. N., et al. Materials fabrication from Bombyx mori silk fibroin. Nature Protocols. 6 (10), 1612-1631 (2011).
- Drachuk, I., et al. Silk macromolecules with amino acid-Poly(Ethylene Glycol) grafts for controlling layer-by-layer encapsulation and aggregation of recombinant bacterial cells. ACS Nano. 9 (2), 1219-1235 (2015).
- Antipov, A. A., Sukhorukov, G. B. Polyelectrolyte multilayer capsules as vehicles with tunable permeability. Advances in Colloid and Interface Science. 111 (1-2), 49-61 (2004).
- Drachuk, I., Harbaugh, S., Chávez, J. L., Kelley-Loughnane, N. Improving the activity of DNA-encoded sensing elements through confinement in silk microcapsules. ACS Applied Materials & Interfaces. 12 (43), 48329-48339 (2020).
- Melnikov, S., Ben-Shem, A., Garreau de Loubresse, N., Jenner, L., Yusupova, G., Yusupov, M. Structural basis for the inhibition of the eukaryotic ribosome. Nature Structural & Molecular Biology. 19 (6), 560-567 (2012).
- Zhao, S., et al. The future of layer-by-layer assembly: A tribute to ACS Nano associate editor Helmuth Möhwald. ACS Nano. 13 (6), 6151-6169 (2019).
- Main, K. H. S., Provan, J. I., Haynes, P. J., Wells, G., Hartley, J. A., Pyne, A. L. B. Atomic force microscopy-A tool for structural and translational DNA research. APL Bioengineering. 5, 031504 (2021).
- Riera, R., Feiner-Gracia, N., Fornaguera, C., Cascante, A., Borrós, S., Albertazzi, L. Tracking the DNA complexation state of pBAE polyplexes in cells with super resolution microscopy. Nanoscale. 11 (38), 17869-17877 (2019).
- Bilokapic, S., Strauss, M., Halic, M. Cryo-EM of nucleosome core particle interactions in trans. Scientific Reports. 8, 7046 (2018).
- Pritchard, E. M., Dennis, P. B., Omenetto, F., Naik, R. R., Kaplan, D. L. Physical and chemical aspects of stabilization of compounds in silk. Biopolymers. 97 (6), 479-498 (2012).
- Fritz, B. R., Jamil, O. K., Jewett, M. C. Implications of macromolecular crowding and reducing conditions for in vitro ribosome construction. Nucleic Acids Research. 43 (9), 4774-4784 (2015).
- Ge, X., Luo, D., Xu, J. Cell-free protein expression under macromolecular crowding conditions. PLoS One. 6 (12), 28707 (2011).
- Cawte, A. D., Unrau, P. J., Rueda, D. S. Live cell imaging of single RNA molecules with fluorogenic mango II arrays. Nature Communications. 11, 1283 (2020).
- Chen, X., et al. Visualizing RNA dynamics in live cells with bright and stable fluorescent RNAs. Nature Biotechnology. 37 (11), 1287-1293 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved