Method Article
通过元条形码分析监测智利有害藻华的标准化程序
摘要
该协议引入了元条形码分析的步骤,靶向16S rRNA和18S rRNA基因,用于监测海水样品中的有害藻华及其相关微生物组。它是一个功能强大的基于分子的工具,但需要几个程序,在这里逐步直观地解释。
摘要
有害藻类大量繁殖(HABs)监测已在全球范围内实施,智利是一个以渔业和水产养殖而闻名的国家,几十年来一直为此目的密集使用显微镜和毒素分析。分子生物学方法,如高通量DNA测序和基于细菌组装的方法,刚刚开始在智利的HAB监测中引入,并且程序尚未标准化。在这里,逐步介绍了用于监测智利HAB的16S rRNA和18S rRNA元条形码分析。根据最近的一项假设,藻类 - 细菌互利关联在水华开始,维持和回归方面起着关键的协同或拮抗关系。因此,从藻类细菌的角度监测HAB可以提供对HAB机制的更广泛理解和早期预警的基础。元条形码分析是最适合用于此目的的基于分子的工具之一,因为它可以检测样品中的大量藻类细菌分类信息。本文中从采样到元条形码分析的可视化程序提供了具体的说明,旨在减少错误和收集可靠的数据。
引言
已知许多海洋浮游植物物种会产生内源性毒素,当这些物种积累足够数量时,它们对海洋环境有害。这种有害藻华(HABs)今天在世界大多数大陆的海岸上被观察到1。有毒的浮游植物首先在双壳类组织中积累,导致包括人类在内的更高营养水平的生物在消化时患病和死亡。随后,这些事件对当地经济,社会经济和公共卫生产生了严重影响2。据估计,HABs每年对全球经济造成的损害为数百万至数十亿美元3。智利是经常遭受有害藻华的许多国家之一。
智利是一个长国,南北绵延4,300多公里。细长的西部陆地面向太平洋,这自然增加了智利经历HAB的机会。特别是在智利南部,有许多世界著名的鲑鱼养殖,每次该地区发生HAB时,藻类毒素都会导致大量养殖鲑鱼生病并死亡4,5,6。在智利,HAB对经济打击最严重的一年是2016年,估计每年损失8亿美元7,8。致病性有毒藻类因年份和地点而异。对于2016年的病例,亚历山大卡塔内拉和疣假单胞菌的复合体在智利南部沿海大部分地区引起广泛的HAB7,8。智利最近的HAB发生在2021年3月在智利南部的Camanchaca的Heterosigma Akashiwo的致病藻类物种中,该地区有一个大型鲑鱼养殖场。
智利多年来一直使用两种主要方法进行沿海监测;用显微镜观察海水,定期识别有毒藻类物种,并通过生化测定法测量贝类中的毒素水平9。早期发现贝类中的有毒藻类和毒素水平并不能预防有害藻类;然而,这些分析可以立即引发对策,并减少对当地社区的损害。为了进一步加强该策略的有效性,我们最近在传统的智利HAB监测计划中增加了一种基于分子的分析方法,以检测海水样品中的藻类和相关细菌群落。具体而言,选择了一种使用元条形码的大规模并行测序方法,靶向16S rRNA和18S rRNA基因。虽然这种技术需要复杂的程序和昂贵的机械和试剂,但它是一种先进的技术,可以同时检测海水样品中存在的数千种藻类和细菌属/种。
HAB的原因推测是多种多样的,例如温度和季节,但不可能一概而论。这是因为HAB物种和频率取决于区域和时空条件,涉及自然现象,例如地理独特性,上升流营养混合以及由于错误2,10,11,12而从陆地产生的元素径流。此外,富营养化等人为因素影响局部HAB12,13。由于复杂的多因素,做出准确的HAB预测并不容易。近年来,有一种观点认为,特定的细菌种群可能与HAB的发展有关,作为因素之一,支持这一假设的研究已经越来越明显14,15,16,17,18。分子生物学技术通常用于研究细菌组装;然而,这种标准化的方法尚未在智利建立HAB监测9。为了研究藻类细菌与有害藻类的关联,必须同时对当前的智利沿海监测计划进行元条形码分析。因此,该协议直观地介绍了我们的智利HAB监测程序,重点是使用元条形码分析分析海水样品中藻类和细菌种类检测的逐步程序。
描述我们的智利HAB监测计划的完整协议可在Yarimizu等人9中找到。它包括海水采样,微观藻类物种检测,藻类细菌基因检测,色素分析,气象数据收集以及物理和化学水性质测定的程序。用于藻类和细菌物种检测的16S rRNA和18S rRNA元条形码分析的逐步方案可作为预印本19提供。该协议特别展示了元条形码分析步骤,因为它是我们HAB监测程序中最复杂的部分和亮点。该协议还包括引入程序和藻类物种的显微镜检测。当通过元条形码分析藻类物种时,同时进行显微镜检查以验证两种方法的结果至关重要。此协议不包括如何使用软件进行分类分配,但数据库建议将在下一节末尾简要说明。
研究方案
1. 样品采集和预处理
- 从目标点收集约3L水样。
- 通过串联过滤(1 μm和0.2 μm孔径膜)过滤1 L水样进行16S rRNA分析,以分离自由生活和附着细菌。
- 过滤另外1升水样,通过0.2μm膜的单次过滤进行18S-rRNA分析(浮游植物检测)。
注意:水样的过滤必须在取样后12小时内完成。 - 用无菌手术剪刀将过滤后的膜切成两半,并用铝箔包裹。在-20°C下储存长达1个月或继续下一步。
- 用Chelex方法提取DNA,如9所述。在-20°C下储存长达1个月。
2. 显微镜分析
- 通过移液器将1 mL水样转移到1 mL网格载玻片上。
- 在显微镜下观察样品。
- 记录浮游植物物种的名称和数量。
3. 16S rRNA和18S rRNA代谢条形码分析
注:该过程由七个部分组成:制备、第一次PCR扩增子生成、第一次扩增子纯化、第二次PCR索引、第二次PCR扩增子验证和纯化、DNA浓度调整以及DNA变性和测序。整个过程至少需要5天(40小时),由经验丰富的实验室人员完成。请参阅 材料表 ,了解产品编号和制造商。
- 制备
- 用70%乙醇清洁移液器和层流罩柜,然后经常紫外线照射30分钟。对要使用的材料进行消毒。
- 在室温下解冻DNA样品,以100×g离心2分钟,并将每个样品的100μL上清液转移到8管条中。
- 首次生成荧光定量 PCR 扩增子
注:在层流罩柜中执行以下过程。始终用PCR级水将引物从1μM储备稀释至目标浓度,以避免引物污染。- 在无菌的1.5 mL管中制备第一个PCR预混液进行反应(表1,表2)。
- 在8管条中等分22.5μL预混液,并加入2.5μLDNA样品。使用2.5μL PCR级水进行阴性对照。
- 运行第一个PCR循环(表3)。
- 准备100 mL的2%琼脂糖-TBE凝胶,其中包含10μL的1x核酸凝胶染色剂。
- 在琼脂糖凝胶上加载1.5μL1x DNA上样染料和4μLPCR产物的混合物。此外,在凝胶上加载100 bp DNA分子量标准品。
- 在100 V下进行电泳30分钟。
- 确保在紫外光图像捕获下存在500-600 bp范围的波段。引物二聚体条带为圆形80 bp。
注:海洋水样含有PCR抑制剂。缺失的扩增子有时可以通过用PCR级水稀释样品1:100或1:1000来解决。 - 将第一批PCR产物储存在-20°C,直到下一步。
注意:储存时间不要超过一周。
- 首次 PCR 扩增子纯化
注:此部分可在层流罩柜外进行。- 使用磁珠DNA纯化系统去除PCR反应残基,包括引物二聚体产物。
- 将每个清洁的第一个PCR产物的20μL转移到新的96孔板中。用微密封膜密封板。储存在-20°C,直到下一步继续。
注意:存放时间不要超过一周。
- 通过第二次 PCR 进行索引
注:在本节中,纯化的首次PCR产物将使用各种指数引物组合进行扩增。- 将所有指数1和指数2引物(表4)稀释至1μM,用PCR级水放入8管PCR条中,置于层流罩柜中。
注:本节今后的步骤可以在层流罩柜外进行,因为分度引物特定于第一个PCR反应悬垂适配器。 - 将索引1引物放在水平行中 索引2引物放在垂直行中(表5)。
- 在新的96孔板中,向每个孔中加入12.5μL热启动就绪配方。
- 使用多通道移液器向每个孔中加入每个索引引物(1μM)的2.5μL,如 表4 所示。
- 加入7.5μL纯化的首次PCR产物。
- 通过上下移液10次轻轻混合。用微密封膜盖住板。
- 运行第二个PCR循环(表3)。
- 将板保持在-20°C。
注意:储存时间不要超过一周。
- 将所有指数1和指数2引物(表4)稀释至1μM,用PCR级水放入8管PCR条中,置于层流罩柜中。
- 第二荧光定量 PCR 扩增子验证和纯化
- 使用片段分析仪和相关试剂。使用前涡旋和旋转试剂。
- 让样品缓冲液和DNA筛选胶带在室温下平衡30分钟。然后,将DNA筛选胶带放入片段分析仪中。
- 将2 μL样品缓冲液和3 μL第二PCR扩增子混合在新的8管条中。将8管条插入片段分析仪。按 "运行" 开始。
注意:必须避免形成气泡。 - 确保16S和18S rRNA基因的第二个PCR扩增子约为613 bp(600 - 630 bp)。
- 使用磁珠DNA纯化系统纯化第二个PCR产物。
- DNA浓度调节
- 使用核酸定量分光光度计测量纯化的第二PCR产物中的DNA浓度。
- 计算目标基因浓度(以 nM 为单位),将 16S 和 18S rRNA 基因的平均最终文库大小计为 613 bp:
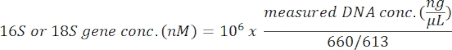
- 在新的0.2 mL 96孔板中,用无菌PCR水将每个纯化的第二PCR产物稀释至4 nM。
注意:该板可以储存在-20°C。否则,必须在不停止的情况下执行其余过程。 - 等分每个4 nM第二PCR产物的3μL,并全部混合在新的无菌1.5mL管中作为混合文库。始终将管子保持在4°C或冰浴中。
- 测量合并文库的浓度,以便使用核酸定量分光光度计进行确认。如果浓度高于4 nM,则将浓度调节至4 nM。
注意:过度浓缩的DNA会产生高估的读取数字,从而妨碍分析。
- DNA 变性和测序
注:本节使用Illumina MiSeq系统与特定试剂。请参阅 材料表中的产品编号 。
注意:严格遵守时间。在测序当天解冻除墨盒以外的所有试剂。- 测序前一天,从-20°C取出预填充的即用型试剂盒,并储存在4°C进行解冻。
- 将适用于 1.5 mL 离心管的加热块设置为 96 °C。
- 将杂交缓冲液放在冰上。
- 用PCR级水将分子级NaOH从1 N稀释到0.2 N的新管中。
- 在新试管中,用 TE 缓冲液 (pH 8.0) 将即用型对照库从 10 nM 原液稀释至 4 nM(即 2 μL 对照库 + 3 μL TE 缓冲液)。
- 将 16 μL 的 4 nM 池化样品库与 4 μL 的 4 nM 对照库混合在标有"1"的新管中。
- 在标有"2"的新管中混合管"1"中的10μL样品与10μL0.2 N NaOH混合。
- 涡旋管25秒,短暂旋转,并在室温下孵育5分钟。
- 将980μL杂交缓冲液加入试管2中。
- 在标有"3"的新试管中,将试管2中的样品与390μL杂交缓冲液混合260μL。通过倒置管子混合。
- 将管3在96°C下孵育2分钟,并立即将其放在冰上最多2分钟。
- 从4°C冰箱中取出墨盒。
- 设置样本表,以便使用每个相应的索引 1 和索引 2 适配器进行排序,如 表 6所示。
- 从 MiSeq v3 套件中卸通池。用无菌分子级PCR水轻轻清洁流通池。
注意:不要将水倒在流通池的毛细管点上。用非纤维纸轻轻擦去流筒上的水。 - 将全体积的管3(650μL)加载到墨盒中。
- 在仪器操作软件中,选择排序并按照说明进行操作。插入流通池、掺入缓冲液和滤芯。加载样品表。按 "运行" 开始反应。
注意:此序列运行将需要 3-5 天。数据将自动上传到Basespace平台。原始数据将存储在计算机的两个文件夹中:包含 fastq 文件的分析文件夹和包含 bcl 文件和 jpeg 图片的输出。
4.测序数据的分类分配
- 快速文件处理
注意:R24 的 DADA2 包23 是推荐用于处理 FASTQ 文件的数据库之一。该指南见[https://github.com/mickeykawai/exec_dada2]。本指南是采用最新版本的 DADA2 管道教程 [https://benjjneb.github.io/dada2/tutorial.html] 编写的。- 处理原始 FASTQ 配对端序列,用于修剪、质量过滤、除去和计数唯一序列、样本推断、合并为重叠群和去除嵌合序列。
- 对16S rRNA和18S rRNA基因片段使用以下DADA2指定参数;trimLefts=0, 0, truncLens=[检查序列质量并将其设置为适当的长度]。
注意:其他参数的默认值为 maxN=0、maxEE=2,2、trancQ=2。
- 分类分配
注意:建议将 SILVA rRNA 数据库用于 DADA2 的分类分配 [silva_nr_v132_train_set.fa.gz设置为默认值]。- 去除叶绿体和线粒体OTU进行16S rRNA基因片段分析。从16S rRNA和18S rRNA分析中去除单例。
- 根据具有最低测序深度的样品,将所有样品耙定到均匀的测序深度。
- 记录读取次数、分配的 OTU 和级别、筛选的读取次数以及排除的单例数。
- 统计分析
- 使用phyloseq25 和素食26的R包对微生物数据进行统计分析。
结果
该协议使用18S rRNA基因元条形码分析来鉴定海水样品中的藻类物种。图1显示了一个具有代表性的结果,其中使用该协议分析了2019年2月19日从智利蒙特港梅特里(−41.597; -72.7056)收集的海水。结果显示,海水样本中共有13,750个藻类物种,超过30种藻类。该样品中的主要藻类是Navicula spp。相对丰度为70.77%。此外,观察到Micromonas(6.40%),Chaetoceros spp.(4.44%),Scripsiella spp.(2.44%)和Prorocentrum spp.的丰度足够。 (1.28%).假性胆菌属是智利HAB中毒性最高的藻类致病因素之一,从该海水样本中检测到0.52%。
为了验证数据的可靠性,将18S rRNA基因分析鉴定的藻类物种与同一海水样品中通过显微镜获得的藻类物种进行了比较(图2)。与18S rRNA基因分析一致,显微镜显示优势物种为 Navicula spp。相对丰度为74.1%, 前中心属 (0.60%)为次要物种。相反,从显微镜观察中记录的 异荚膜属 (9.04%)未通过该样本中的18S rRNA基因分析鉴定。通过显微镜记录的周围有12.6%的无法识别的小型浮游植物细胞。这可能是 微单胞菌,根据18S rRNA的结果。
该协议使用16S rRNA基因元条形码分析来鉴定海水样品中的细菌物种。一个具有代表性的结果如图 3所示,其中用于18S rRNA基因分析的相同海水与16S rRNA基因分析相同。结果显示,海水样本中共有31,758个读数,其中有超过30种细菌物种。应该概述的是,该海水样品通过串联过滤膜(1μm和0.2μm孔径)以将自由活细菌与附着细菌分开。然后,对每个滤膜捕获的细胞进行DNA提取处理,然后进行16S rRNA基因分析。 图3 中的代表性结果显示了从0.2μm孔径膜中鉴定出的细菌物种,这些细菌被定义为自由生活的细菌。占主导地位的自由生活细菌是 淀粉杆菌属, 相对丰度为20.02%,其次是 进化枝杆菌 (13.53%)和 Aligiphilus spp.。(7.06%).从该海水样本中检测到的其余细菌种类分布相对均匀。对于由1μm孔径膜捕获的细胞,可以进行与附着细菌种类检测相同的分析。
当这些元条形码测定在一定研究周期内的预定时间点进行时,结果可以总结为时间序列分析。一种方法是绘制特定藻类和细菌物种的相对丰度作为时间的函数,以找到独特的生长模式。 图 4 显示了智利梅特里的 亚历山大 和 假单胞菌 的代表性时间序列图。总结时间序列元条形码分析的另一种方法是将所有已识别的藻类和细菌绘制为时间的函数,表示某些生物群的种群变化。 图5 和 图6 分别总结了五个月内从Metri海水中检测到的所有细菌属和目细菌的相对丰度。
表1:第一PCR预混液含量: 该表显示了16S rRNA和18S rRNA分析的每个反应的预混液含量。引物序列列于表3中。 请点击此处下载此表格。
表2:引物序列: 首次PCR的引物被列在16S rRNA和18S rRNA分析中。 请点击此处下载此表格。
表3:PCR循环: 列出了第一次 PCR 和第二次 PCR 的热循环。 请点击此处下载此表格。
表 4:索引序列: 列出了用于第二次PCR的索引1(i7)和索引2(i5)引物。 请点击此处下载此表格。
表 5:索引 1 和索引 2 定位示例: 为了减少误差,必须首先将分度引物放置在该位置,并使用多通道移液器将每个引物的等分试样转移到96孔板中。 请点击此处下载此表格。
表6:样品表示例: 在测序之前,必须创建与索引 1 和索引 2 适配器相对应的样本表。 请点击此处下载此表格。
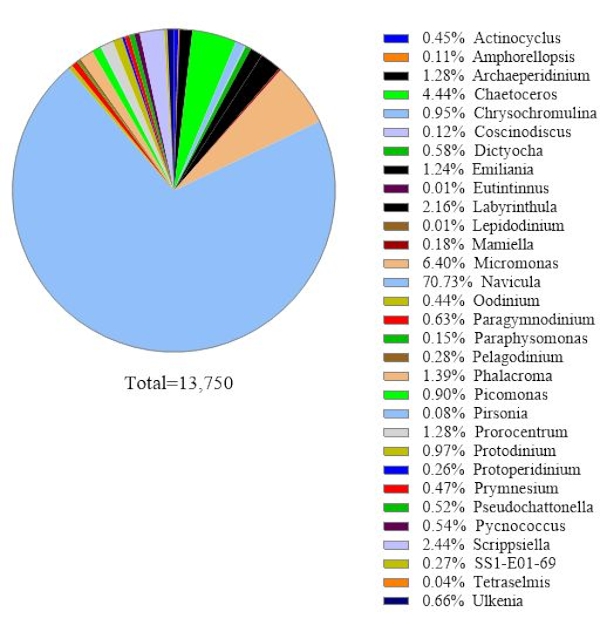
图1:18S rRNA元条形码分析的代表性结果:2019 年2月19日从智利洛斯拉各斯梅特里收集的海水样本中存在的藻类物种通过18S rRNA元条形码测定法鉴定。从中删除了分配"未知"的序列,并绘制了每个已识别物种的相对丰度。 请点击此处查看此图的放大版本。
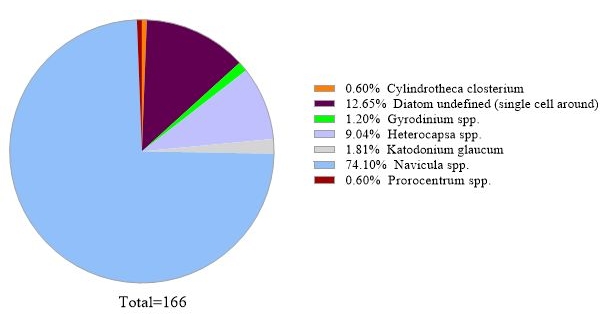
图2:显微镜分析的代表性结果: 藻类物种于2019年2月19日通过显微镜从智利洛斯拉各斯Metri的水样中鉴定出来。手动计算每个物种的数量并绘制。 请点击此处查看此图的放大版本。
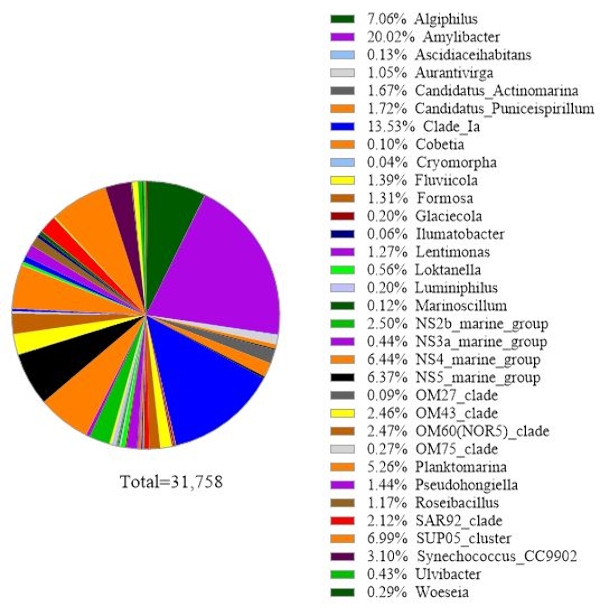
图3:16S rRNA元条形码分析的代表性结果 :2019年2月19日,智利洛斯拉各斯Metri的水样中存在的细菌物种通过16S rRNA元条形码测定法鉴定。绘制了每个已识别物种的相对丰度图。 请点击此处查看此图的放大版本。
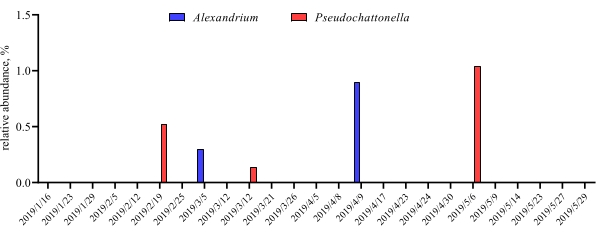
图4:从智利Metri的18S rRNA元条形码蛋白获得的Alexandrium属和Pseudochattonella属的代表性时间序列图:图转载自Yarimizu等人9。通过18S rRNA分析选择性监测了亚历山大藻和假单胞菌这两种有毒藻类,并绘制了相对丰度作为时间点的函数。请点击此处查看此图的放大版本。
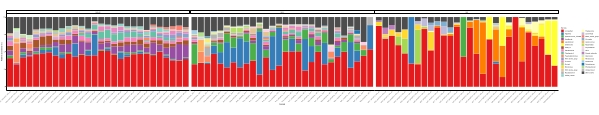
图5:从16S rRNA和18S rRNA元条形码中获得的代表性属时间序列图: 在智利梅特里的水中发现的所有细菌和真核属,监测了五个月以上。 请点击此处查看此图的放大版本。
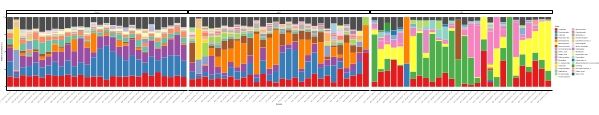
图6:从16S rRNA和18S rRNA元条形码中获得的代表性顺序时间序列图: 在智利Metri水中发现的所有细菌和真核目,监测了五个月以上。 请点击此处查看此图的放大版本。
表 S1 和 表 S2:
表 S1:图5和图 6的原始数据。表 S2:对图 5和图6的原始数据的 QC 检查结果。请点击此处下载这些表格。
讨论
该协议成功进行了18S rRNA和16S rRNA基因元条形码分析,以鉴定海水样品中的藻类和细菌物种,以监测智利HABs。该协议检测到的显性藻类和一些次要藻类与通过显微镜获得的藻类一致,证实了该协议的可靠性。该协议的亮点是,该分析从Metri的海水中检测到Alexandrium spp.和Pseudochattonella spp.,这是智利南部引起HAB的两种最有问题的物种,即使在低丰度下也是如此。特别是假单胞菌属。在显微镜下识别具有挑战性的物种,因为细胞很小,在光下或使用固定剂很容易破裂27,28。元条形码可以提供藻类物种信息,这些信息在海水样品中很少存在,显微镜无法识别。该协议还检测到30多种细菌物种。虽然这些细菌如何与藻类生长有关目前尚不清楚,但在时间序列分析中收集藻类细菌种类的大量数据可能会提供如此重要的发现。因此,在传统的智利HAB监测计划中增加元条形码分析无疑将加强目前的HAB监测效率。事实上,这种用于元条形码分析的视觉方案不仅有利于智利水的藻类细菌监测,而且有利于全球其他受HAB影响地区的海岸监测计划。
尽管该协议提供了上述优点,但应讨论该方法的缺点。如视觉协议所示,元条形码方法耗时且复杂,需要昂贵的机械和试剂。实验室人员必须经过专门培训,否则会浪费材料,劳动力和时间。此外,通过元条形码进行的藻类检测必须与显微镜配对,以验证从两种正交方法中鉴定出相同的优势物种29。显微镜是一种非侵入性工具,在大多数情况下,用于识别藻类物种,这意味着当藻类物种具有独特而明显的形状时,更难犯错误。当然,如果物种彼此具有非常相似的形状,则可能会发生人为错误。另一方面,由于元条形码分析需要多个步骤,因此自然会增加错误。它可能是样品混淆,错误的试剂添加或缺少某些程序。因此,将元条形码结果与通过显微镜获得的结果进行比较至关重要。最后,该协议仅在定性上适用,并且必须计算结果的相对丰度。正如Lamb等人在2019年所说,目前的元编码作为定量性能仍然有限,在元条形码可以自信地用于定量应用之前,还需要进行额外的研究30。
该协议针对Illumina Inc.发布的16S宏基因组测序文库制备手册中每次运行140 - 170个样本进行了优化。优化的协议经过多次测试,并进一步修改以发布此最终版本。因此,强烈建议精确地遵循每个步骤。该协议最关键的部分是需要格外小心以避免样品污染。移液器和层流罩必须始终用70%的酒精和紫外线清洗,并且应高压灭菌材料。在每次新运行时,应直接从原液中稀释引物和试剂,否则重复使用试剂并暴露于多个稀释液中可能会造成样品污染。该协议规定了何时可以在层流罩外完成操作。除非另有说明,否则样品应在清洁的层流罩中处理。当同时处理多个8管条带时,建议在第一个8管条带上工作,并在移动到下一个8管条之前将其盖上盖子。长时间打开盖子会导致样品污染,因为16S和18S rRNA基因在周围环境中高度普遍。所述时间(如混合时间和孵育时间)应严格按照描述进行,因为每个步骤的最佳持续时间是根据多次测试运行选择的。例如,时间过长或不足会降低样品产量。该过程应该仅在协议声明可以停止的部分停止。进行阴性控制至关重要,因为它确认阳性结果不是人为的假阳性。最后,强烈建议计划日期,因为它至少需要五天来处理所有步骤,并且某些部分在下一个停止标志之前无法停止。
这里简要指出了测序数据的分类分配的局限性。新发现的细菌和藻类的核苷酸序列每天都在数据库中更新。虽然经过充分研究的藻类和细菌物种被可靠地登记,但数据库中也更新了许多未知物种的序列。它表明,注册核苷酸序列的分辨率并不总是足以进行物种鉴定,而仅用于属鉴定。因此,微观物种鉴定必须正交进行。为这项工作建立开放获取管道在同时处理大量序列数据以及在发生错误时有效调查方面具有显着的优势。此外,DADA2 的输出位于文本或 csv 文件中,便于进一步进行统计分析。另一方面,需要大型服务器来执行分类分配,尤其是当数据集很大时。要设置管道,需要工程师来设置管道,专业人员必须了解参数,操作,Linux和生物信息学。
除了作为HAB监测工具的范围外,该协议的使用可以扩展到调查研究目的。例如,智利政府与当地渔民以及环境和平协会之间就当地HAB31,32的数量增加进行了持续的辩论。政府声称这是由于全球变暖和厄尔尼诺等自然现象造成的,而后来的各方则声称鲑鱼养殖是原因。鲑鱼不是智利的本土物种,半个世纪前也不在智利水域。当时的智利政府旨在通过发展鲑鱼业务来发展经济并为贫困地区创造就业机会33.随着海外对资本利润的干预,智利在围栏式水产养殖发展方面取得了巨大成功,鲑鱼的数量在过去几十年中显着增加31,32,33。随后,大量鲑鱼的食物被扔进海里,导致当地人怀疑鲑鱼养殖是当地HABs频繁的原因31、32.真相现在还不得而知,但最终必须理解,以便制定保护当地海洋环境,经济和人类健康免受HAB攻击的策略。对局部藻类 - 细菌关系的基于分子的分析可能有助于为此类主题向前迈进一步。例如,该技术可以搜索以前在目标海洋区域不存在但近年来急剧增加的藻类和细菌物种,这与Sakai等人通过GIS和eDNA分析34发现未记录的大和蝾螈(Hynobius vandenburghi)种群的研究有些相似。因此,除了HAB监测工具之外,这种元条形码协议还具有其他潜在客户的潜在用途。
披露声明
作者声明没有利益冲突。资助者在研究的设计、数据收集、分析或解释方面没有任何作用;相反,它们没有参与任何工作。在撰写手稿时,或在决定发表结果时。
致谢
这项研究得到了赠款(JPMJSA1705)的支持,用于智利可持续发展监测藻类的科学和技术研究伙伴关系(SATREPS-MACH)的研究。我们感谢Sandra Rios博士(洛斯拉各斯大学)允许我们使用电影剪辑。我们感谢尼尔·安德鲁·霍兰德(Neal Andrew Holland)对手稿的勤奋校对。我们衷心感谢智利蒙特港CREAN-IFOP实验室小组成员就浮游植物鉴定向我们提供建议。
材料
| Name | Company | Catalog Number | Comments |
| 1.5 mL single tube | ThermoFisher | Q32856 | sterile |
| 1.5 mL tubes | ThermoFisher | 2150N | sterile |
| 8-strip 0.2 mL PCR tubes and flat cap | ThermoFisher | AB0451 & 4323032 | sterile |
| 96-well 0.2 mL PCR plate | ThermoFisher | N8010560 | |
| AccuRuler 100 bp DNA Ladder | MaestroGen | 02001-500 | |
| Chelex 100 Chelating Resin | Bio-Rad | 1432832 | |
| Chemical Duty Vacuum Pressure Pump | MilliporeSigma | WP6122050 | |
| D1000 Reagents | Agilent Technologies | 5067-5583 | |
| D1000 sample buffer | Agilent Technologies | 5067-5602 | |
| DNA loading dye (Blue/Orange Loading Dye 6X) | Promega | G1881 | |
| Ethanol Molecular Biology Grade | E7023 | Merck KGaA | |
| Filtration device | ThermoFisher | 09-740-36H, 09-740-23E | |
| Fluorescent DNA concentration measurement kit (Qubit dsDNA HS Assay kit) | Thermo Fisher Scientific | Q32851 | |
| Fluorometer (Qubit4 Fluorometer) | ThermoFisher | Q33238 | |
| Fragment analysis verification matrix (D1000 ScreenTape) | Agilent Technologies | 5067-5582 | |
| Fragment Analyzer (Agilent Tapestation 4150) | Agilent | G2992AA | |
| GelRed Nucleic Acid Gel Stain | Biotium | 41002 | |
| Generic bench vortexer (Vortex Genie 2) | Scientific Industries | SI-0236 | |
| Heat block | |||
| High performance sequencing-grade DNA polymerase (KAPA HiFi Hotstart ReadyMix), 2X | Roche | KK2602 | |
| HT1 Hybridization buffer | Illumina | 20015892 | |
| Illumina Nextera XT v2 Index Kit set A, B, C and D | Illumina | FC-131-2001, -2002, -2003, -2004 | |
| Laminar hood cabinet | Biobase Co. | Biobase PCR-800 PCR Cabinet | |
| Loading tips (Specific for Agilent TapeStation systems) | Agilent Technologies | 5067-5598 | |
| Magnetic beads DNA cleanup system (ProNex® Size-Selective Purification System) | Promega | NG2002 | |
| Magnetic stand for 96 wells (Magnetic Stand-96) | ThermoFisher | AM10027 | |
| Micro-seal film for 96-well plate | ThermoFisher | 4311971 | sterile |
| MiSeq Reagent Kit v3 | Illumina | MS-102-3003 | includes pre-filled, ready-to-use reagent cartridges. |
| MiSeq system | Illumina | SY-410-1003 | includes pre-installed software |
| Molecular grade nuclease-free water | Integrated DNA Technologies | 11-05-01-04 | |
| Molecular grade sodium hydroxide (NaOH) 1M | Merck KGaA | 1091371000 | |
| Multichannel pipettes | Not specified | Not specified | |
| Multi-Purpose Agarose | Cleaver Scientific | CSL-AG500 | |
| Ow D2 Wide-Gel Electrophoresis System | ThermoFisher | D2 | |
| Ow EC-105 Compact Power Supply | ThermoFisher | 105ECA-115 | |
| Pellet Pestle— Cordless Motor | ThermoFisher | K749540-0000 | |
| PhiX control Kit v3 | Illumina | FC-110-300 | ready-to-use control library for Illumina sequencing |
| Pipette tips | Not specified | Not specified | sterile |
| Pipettes | Not specified | Not specified | 2, 10, 20, 100, 1000 μL |
| SterivexTM GP 0.22 μm filter unit | MilliporeSigma | SVGP01050 | |
| Tabletop centrifuge | Eppendorf 5427R Centrifuge | Eppendorf AG | |
| TBE buffer | ThermoFisher | B52 | |
| TE buffer (pH 8.0) | ThermoFisher | AM9849 | |
| Terr PCR Direct FFPE Kit | Takara Bio USA | 639284 | includes 2X Terra PCR Direct Buffer |
| Terra PCR Direct Polymerase Mix, 1.25 U/μL | Takara Bio USA | 639271 | High performance Inhibitor-tolerant DNA polymerase |
| ThermalCycler (MiniAMP Plus Thermal Cycler) | ThermoFisher | A37835 | |
| TransIlluminator and image capture system | Analytik Jena, AG | GelDoc-ItTS2 Imager | |
| Whatman 1.0 m pore-sized membrane | MilliporeSigma | WHA111110 |
参考文献
- Hallegraeff, G., Enevoldsen, H., Zingone, A. Global harmful algal bloom status reporting. Harmful Algae. 102, 101992 (2021).
- Anderson, D. M. Red Tides. Scientific American. 271 (2), 52-58 (1994).
- Sanseverino, I., Conduto, A. D. S., Pozzoli, L., Dobricic, S., Lettieri, T. . Algal Bloom and its Economic Impact. , (2016).
- Molinet, C., Niklitschek, E., Seguel, M., Díaz, P. Trends of natural accumulation and detoxification of paralytic shellfish poison in two bivalves from the Norwest Patagonian inland sea. Revista de Biologia Marina Y Oceanografia. 45, 195-204 (2010).
- Mardones, J., Clement, A., Rojas, X., Aparicio, C. Alexandrium catenella during 2009 in Chilean waters, and recent expansion to coastal ocean. Harmful Algae News. 41, 8-9 (2010).
- Alvarez, G., et al. Paralytic shellfish toxins in surf clams Mesodesma donacium during a large bloom of Alexandrium catenella dinoflagellates associated to an intense shellfish mass mortality. Toxins (Basel). 11 (4), (2019).
- Clément, A., et al. Exceptional summer conditions and HABs of Pseudochattonella in Southern Chile create record impacts on salmon farms. Harmful Algae News. 53, 1-3 (2016).
- Trainer, V. L., et al. Pelagic harmful algal blooms and climate change: Lessons from nature's experiments with extremes. Harmful Algae. 91, 101591 (2020).
- Yarimizu, K., et al. Protocols for monitoring harmful algal blooms for sustainable aquaculture and coastal fisheries in Chile. International Journal of Environmental Research and Public Health. 17 (20), (2020).
- Roegner, G. C., Hickey, B. M., Newton, J. A., Shanks, A. L., Armstrong, D. A. Wind-induced plume and bloom intrusions into Willapa Bay, Washington. Limnology & Oceanography. 47 (4), 1033-1042 (2002).
- Tweddle, J. F., et al. Relationships among upwelling, phytoplankton blooms, and phycotoxins in coastal Oregon shellfish. Marine Ecology Progress Series. 405, 131-145 (2010).
- Glibert, P. M., Anderson, D. M., Gentien, P., Graneli, E., Sellner, K. G. The global complex phenomena of harmful algae blooms. Oceanography. 18 (2), 130-141 (2005).
- Paredes-Mella, J., Varela, D., Fernández, P., Espinoza-González, O. Growth performance of Alexandrium catenella from the Chilean fjords under different environmental drivers: Plasticity as a response to a highly variable environment. Journal of Plankton Research. 42 (2), 119-134 (2020).
- Azam, F., Malfatti, F. Microbial structuring of marine ecosystems. Nature Reviews Microbiology. 5, 782-791 (2007).
- Amin, S. A., et al. Interaction and signaling between a cosmopolitan phytoplankton and associated bacteria. Nature. 522, 98-101 (2015).
- Berdalet, E., et al. Marine harmful algal blooms, human health, and wellbeing: Challenges and opportunities in the 21st century. Journal of Marine Biology Association. 2015, 1-31 (2015).
- Ramanan, R., Kim, B. -. H., Cho, D. -. H., Oh, H. -. M., Kim, H. -. S. Algae-bacteria interactions: Evolution, ecology, and emerging applications. Biotechnology Advances. 34 (1), 14-29 (2016).
- Seymour, J. R., Amin, S. A., Raina, J. B., Stocker, R. Zooming in on the phycosphere: The ecological interface for phytoplankton-bacteria relationships. Nature Reviews Microbiology. 2, 17065 (2017).
- Maruyama, F., et al. 16S and 18S Metabarcoding analysis for Chilean coastal waters harmful algal blooms. protocol.io. , (2020).
- Tanabe, A. S., et al. Comparative study of the validity of three regions of the 18S-rRNA gene for massively parallel sequencing-based monitoring of the planktonic eukaryote community. Molecular Ecology Resources. 16, 402-414 (2016).
- Nishitani, G., et al. Multiple plastids collected by the Dinoflagellate Dinophysis mitra through Kleptoplastidy. Applied and Environmental Microbiology. 78 (3), 813-821 (2012).
- Klindworth, A., et al. Evaluation of general 16S ribosomal RNA gene PCR primers for classical and next-generation sequencing-based diversity studies. Nucleic Acids Research. 41 (1), 1 (2013).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nature Methods. 13 (7), 581-583 (2016).
- McMurdie, P. J., Holmes, S. phyloseq: An R Package for Reproducible Interactive Analysis and Graphics of Microbiome Census Data. PLoS One. 8 (4), 61217 (2013).
- Dixon, P. VEGAN, a package of R functions for community ecology. Journal of Vegetation Science. 14 (6), 927-930 (2003).
- Dittami, S. M., Riisberg, I., Edvardsen, B. Molecular probes for the detection and identification of ichthyotoxic marine microalgae of the genus Pseudochattonella (Dictyochophyceae, Ochrophyta). Environmental Science and Pollution Research. 20 (10), 6824-6837 (2013).
- Mardones, J. I., et al. Salinity-growth response and ichthyotoxic potency of the Chilean Pseudochattonella verruculosa. Frontiers in Marine Science. 6 (24), (2019).
- Santi, I., Kasapidis, P., Karakassis, I., Pitta, P. A comparison of DNA metabarcoding and microscopy methodologies for the study of aquatic microbial eukaryotes. Diversity. 13 (5), 180 (2021).
- Lamb, P. D., et al. How quantitative is metabarcoding: A meta-analytical approach. Molecular Ecology. 28 (2), 420-430 (2019).
- Mascareño, A., et al. Controversies in social-ecological systems: lessons from a major red tide crisis on Chiloe Island, Chile. Ecology and Society. 23, (2018).
- Armijo, J., Oerder, V., Auger, P. -. A., Bravo, A., Molina, E. The 2016 red tide crisis in southern Chile: Possible influence of the mass oceanic dumping of dead salmons. Marine Pollution Bulletin. 150, 110603 (2020).
- Swanson, H. A. . Caught in Comparisons: Japanese Salmon in an Uneven World. , (2013).
- Sakai, Y., et al. Discovery of an unrecorded population of Yamato salamander (Hynobius vandenburghi) by GIS and eDNA analysis. Environmental DNA. 1 (3), 281-289 (2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。