Method Article
Un procedimiento estandarizado para monitorear las floraciones de algas nocivas en Chile mediante el análisis de metacódigo de barras
En este artículo
Resumen
Este protocolo introduce pasos de análisis de metacódigo de barras, dirigidos a los genes 16S rRNA y 18S rRNA, para monitorear las floraciones de algas nocivas y su microbioma asociado en muestras de agua de mar. Es una poderosa herramienta de base molecular, pero requiere varios procedimientos, que se explican visualmente aquí paso a paso.
Resumen
El monitoreo de floraciones de algas nocivas (HAB) se ha implementado en todo el mundo, y Chile, un país famoso por su pesca y acuicultura, ha utilizado intensamente análisis microscópicos y de toxinas durante décadas para este propósito. Los métodos biológicos moleculares, como la secuenciación de ADN de alto rendimiento y los enfoques basados en ensamblajes bacterianos, apenas están comenzando a introducirse en el monitoreo de HAB chileno, y los procedimientos aún no se han estandarizado. Aquí, los análisis de metacódigo de barras de ARNr 16S y ARNr 18S para monitorear haBs chilenos se introducen paso a paso. Según una hipótesis reciente, la asociación mutualista algas-bacterianas juega una relación sinérgica o antagónica crítica que explica el inicio, el mantenimiento y la regresión de la floración. Por lo tanto, el monitoreo de HAB desde perspectivas de algas-bacterianas puede proporcionar una comprensión más amplia de los mecanismos de HAB y la base para la alerta temprana. El análisis de metacódigo de barras es una de las herramientas moleculares más adecuadas para este propósito, ya que puede detectar información taxonómica masiva de algas y bacterias en una muestra. Los procedimientos visuales de muestreo para el análisis de metacódigo de barras en este documento proporcionan instrucciones específicas, con el objetivo de reducir los errores y la recopilación de datos confiables.
Introducción
Se sabe que muchas especies de fitoplancton marino producen toxinas endógenas, y cuando estas especies se acumulan en cantidades suficientes, son perjudiciales para el medio ambiente marino. Tales floraciones de algas nocivas (HAB) se observan hoy en día en las costas de la mayoría de los continentes del mundo1. El fitoplancton tóxico se acumula primero en los tejidos bivalvos, lo que lleva a la enfermedad y la muerte en niveles tróficos más altos de organismos, incluidos los humanos, después de la digestión. Posteriormente, estos eventos traen graves implicaciones para la economía local, la socioeconomía y la salud pública2. El daño a la economía global causado por los HAB se estima en millones a miles de millones de dólares cada año3. Chile es uno de los muchos países que sufren de HAB frecuentes.
Chile es un país de tierra larga, que se extiende a lo largo de 4.300 km al norte y al sur. La tierra occidental alargada se enfrenta al Océano Pacífico, lo que naturalmente aumenta las posibilidades de que Chile experimente HABs. Especialmente en el sur de Chile, hay muchas acuiculturas de salmón de fama mundial, y cada vez que se produce un HAB en la región, las toxinas de algas resultan en que el salmón de piscifactoría masivo se enferme y muera4,5,6. En Chile, el año en que el HAB golpeó más fuerte a la economía fue 2016, con una pérdida anual estimada de US$ 800 millones7,8. Las especies de algas tóxicas causales varían según el año y la ubicación. Para el caso de 2016, un complejo de Alexandrium catanella y Pseudochattonella verruclosa causó un HAB generalizado en la mayor parte de la costa sur de Chile7,8. El HAB más reciente en Chile ocurrió con la especie de algas causantes de Heterosigma Akashiwo en marzo de 2021 en Camanchaca, ubicada en el sur de Chile, donde el área tiene una gran granja de salmón.
Chile ha estado realizando monitoreo costero durante muchos años utilizando dos métodos principales; observar el agua de mar con microscopios para identificar especies de algas tóxicas regularmente y medir los niveles de toxinas en mariscos mediante ensayos bioquímicos9. La detección temprana de algas tóxicas y niveles de toxinas en mariscos no previene las HAB; sin embargo, estos análisis pueden desencadenar contramedidas inmediatas y reducir los daños a las comunidades locales. Para fortalecer aún más la efectividad de esta estrategia, recientemente se agregó un método analítico de base molecular a nuestro programa convencional de monitoreo de HAB chileno para detectar algas y comunidades bacterianas relacionadas en muestras de agua de mar. Específicamente, se eligió un método de secuenciación paralela masiva que utiliza metacódigo de barras que se dirige a los genes 16S rRNA y 18S rRNA. Aunque esta técnica requiere procedimientos complicados y maquinaria y reactivos costosos, es una tecnología avanzada que puede detectar miles de géneros / especies de algas y bacterias presentes en una muestra de agua de mar a la vez.
Se especula que las causas de la HAB son varias, como la temperatura y la estación, pero es imposible generalizarlas. Esto se debe a que las especies y frecuencias de HAB dependen de la región y las condiciones espaciotemporales, que involucran fenómenos naturales como la singularidad geográfica, la mezcla de nutrientes de surgencia y la escorrentía de elementos de la tierra debido a la errosión2,10,11,12. Además, factores artificiales como la eutrofización influyen en los HAB locales12,13. Debido al complejo multifactor, no es fácil hacer una predicción HAB precisa. En los últimos años, existe la opinión de que poblaciones bacterianas específicas pueden estar relacionadas con el desarrollo de HABs como uno de los factores, y la investigación para apoyar esta hipótesis ha sido cada vez más evidente14,15,16,17,18. Las técnicas de biología molecular se utilizan generalmente para estudiar el ensamblaje bacteriano; sin embargo, dicho método estandarizado aún no se ha establecido en el monitoreo de haB chileno9. Para estudiar la asociación argalo-bacteriana con haBs, es imperativo realizar simultáneamente análisis de metacódigo de barras para los actuales programas de monitoreo costero chileno. Por lo tanto, este protocolo presenta visualmente nuestro programa chileno de monitoreo de HAB, centrándose en un procedimiento gradual para analizar la detección de especies de algas y bacterias en muestras de agua de mar utilizando el análisis de metacódigo de barras.
El protocolo completo que describe nuestro programa chileno de monitoreo de HAB está disponible en Yarimizu et al.9. Incluye los procedimientos de muestreo de agua de mar, detección microscópica de especies de algas, detección de genes de algas-bacterianas, análisis de pigmentos, recopilación de datos meteorológicos y ensayos físicos y químicos de propiedades del agua. El protocolo paso a paso del análisis de metacódigo de barras de ARNr 16S y ARNr 18S para la detección de especies de algas y bacterias está disponible como preimpresión19. Este protocolo demuestra particularmente los pasos de análisis de metacódigo de barras, ya que es la parte más complicada y el punto culminante de nuestro programa de monitoreo HAB. Este protocolo también incluye una introducción del programa y microscopía de detección de especies de algas. Al analizar especies de algas por metacódigo de barras, es crucial realizar simultáneamente microscopía para verificar los resultados de los dos métodos. Este protocolo no incluye cómo utilizar el software para la asignación de taxonomía, pero la recomendación de la base de datos se indica brevemente al final de la siguiente sección.
Protocolo
1. Recogida de muestras y pretratamiento
- Recolecte aproximadamente 3 L de muestra de agua del punto objetivo.
- Filtre 1 L de muestra de agua para el análisis de ARNr 16S a través de una filtración en tándem (membrana de tamaño de poro de 1 μm y 0,2 μm) para separar las bacterias de vida libre y las adheridas.
- Filtre otra muestra de 1 L de agua para el análisis de ARNr 18S (detección de fitoplancton) a través de una sola filtración con una membrana de 0,2 μm.
NOTA: La filtración de la muestra de agua debe completarse dentro de las 12 h posteriores al muestreo. - Cortar la membrana filtrada por la mitad con tijeras quirúrgicas estériles y envolverla con papel de aluminio. Conservar a -20 °C durante un máximo de 1 mes o continuar con el siguiente paso.
- Extraer ADN con el método Chelex como se describe9. Conservar a -20 °C durante un máximo de 1 mes.
2. Análisis de microscopio
- Transfiera 1 ml de la muestra de agua por una pipeta a un tobogán de rejilla de 1 ml.
- Observe la muestra bajo un microscopio.
- Registre los nombres y la cantidad de especies de fitoplancton.
3. Análisis de metacódigo de barras de ARNr 16S y ARNr 18S
NOTA: Este proceso consta de siete secciones: preparación, primera generación de amplicón PCR, primera limpieza de amplicones, indexación por segunda PCR, segunda verificación y limpieza de amplicones PCR, ajuste de concentración de ADN y desnaturalización y secuenciación de ADN. Todo el proceso toma un mínimo de 5 días (40 h) por un personal de laboratorio experimentado. Consulte la Tabla de materiales para conocer los números de producto y los fabricantes.
- Preparación
- Limpie las pipetas y el gabinete de la campana laminar con 70% de etanol seguido de exposición a los rayos UV durante 30 minutos con frecuencia. Esterilizar los materiales a utilizar.
- Descongele las muestras de ADN a temperatura ambiente, centrífuga a 100 x g durante 2 min y transfiera 100 μL de cada sobrenadante de muestra a tiras de 8 tubos.
- Primera generación de amplicones PCR
NOTA: Realice los siguientes procedimientos en un gabinete de campana laminar. Diluya siempre los cebadores de 1 μM a la concentración objetivo con agua de grado PCR para evitar la contaminación de la imprimación.- Preparar la primera mezcla maestra de PCR en un tubo estéril de 1,5 ml para las reacciones (Tabla 1, Tabla 2).
- Alícuota 22,5 μL de la mezcla maestra en una tira de 8 tubos y añade 2,5 μL de muestra de ADN. Utilice 2,5 μL de agua de grado PCR para el control negativo.
- Ejecutar el primer ciclo de PCR (Tabla 3).
- Preparar 100 ml de gel de agarosa-TBE al 2% que contenga 10 μL de 1x tinción de gel de ácido nucleico.
- Cargue una mezcla de 1,5 μL de 1x colorante de carga de ADN y 4 μL de producto pcrista en el gel de agarosa. Además, cargue una escalera de ADN de 100 pb en el gel.
- Realizar electroforesis a 100 V durante 30 min.
- Asegúrese de que haya una banda en el rango de 500-600 pb bajo una captura de imagen con luz UV. La banda del dímero de imprimación es redonda de 80 pb.
NOTA: Las muestras de agua marina contienen inhibidores de PCR. Los amplicones faltantes a veces se pueden resolver diluyendo muestras 1:100 o 1:1000 con agua de grado PCR. - Guarde los primeros productos de PCR a -20 °C hasta el siguiente paso.
PRECAUCIÓN: No exceda una semana de almacenamiento.
- Primera limpieza de amplicon PCR
NOTA: Esta sección se puede realizar fuera de un gabinete de campana laminar.- Utilice el sistema de limpieza de ADN de perlas magnéticas para eliminar los residuos de reacción de PCR, incluidos los productos de dímero de imprimación.
- Transfiera 20 μL de cada primer producto de PCR limpiado a una nueva placa de 96 pocillos. Selle la placa con una película de microsellado. Conservar a -20 °C hasta que se proceda al siguiente paso.
PRECAUCIÓN: No exceda más de una semana de almacenamiento.
- Indexación por segunda PCR
NOTA: En esta sección, los primeros productos de PCR purificados se amplificarán con varias combinaciones de imprimación de índice.- Diluya todos los cebadores de Índice 1 e Índice 2(Tabla 4)a 1 μM con agua de grado PCR en tiras de PCR de 8 tubos colocadas en un gabinete de campana laminar.
NOTA: Los pasos en adelante en esta sección se pueden realizar fuera de un gabinete de campana laminar, ya que los cebadores de índice son específicos del primer adaptador de voladizo de reacciones de PCR. - Posición Índice 1 imprimación en una fila horizontal Índice 2 imprimaciones en una fila vertical (Tabla 5).
- En una nueva placa de 96 pocillos, agregue 12.5 μL de formulación lista para el arranque en caliente a cada pozo.
- Agregue 2.5 μL de cada imprimación de índice (1 μM) a cada pozo como en la Tabla 4 usando una pipeta multicanal.
- Añadir 7,5 μL del primer producto de PCR purificado.
- Mezclar suavemente mediante pipeteo hacia arriba y hacia abajo 10 veces. Cubra la placa con una película de microsellado.
- Ejecutar el segundo ciclo de PCR (Tabla 3).
- Mantenga la placa a -20 °C.
PRECAUCIÓN: No exceda una semana de almacenamiento.
- Diluya todos los cebadores de Índice 1 e Índice 2(Tabla 4)a 1 μM con agua de grado PCR en tiras de PCR de 8 tubos colocadas en un gabinete de campana laminar.
- Segunda verificación y limpieza de amplicones PCR
- Utilice un analizador de fragmentos y un reactivo asociado. Reactivo de vórtice y spin down antes de su uso.
- Permita que el búfer de muestra y la cinta de detección de ADN se equilibren a temperatura ambiente durante 30 minutos. Luego, coloque la cinta de pantalla de ADN en un analizador de fragmentos.
- Mezclar 2 μL de tampón de muestra y 3 μL de segundo amplicón PCR en nuevas tiras de 8 tubos. Inserte las 8 tiras de tubo en el analizador de fragmentos. Presione Ejecutar para comenzar.
PRECAUCIÓN: Se debe evitar la formación de burbujas de aire. - Asegúrese de que los segundos amplicones de PCR sean de aproximadamente 613 pb (600 - 630 pb) para los genes 16S y 18S rRNA.
- Purificar los productos de segunda PCR utilizando un sistema de limpieza de ADN de perlas magnéticas.
- Ajuste de la concentración de ADN
- Mida la concentración de ADN en los segundos productos de PCR purificados utilizando un espectrofotómetro de cuantificación de ácido nucleico.
- Calcule la concentración de genes objetivo en nM, teniendo en cuenta el tamaño promedio de la biblioteca final como 613 pb para los genes 16S y 18S rRNA:
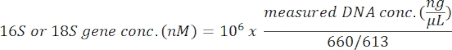
- Diluya cada segundo producto de PCR purificado con agua estéril de PCR a 4 nM en una nueva placa de pozo de 0,2 ml 96.
NOTA: La placa se puede almacenar a -20 °C aquí. De lo contrario, el resto de procedimientos deben realizarse sin detenerse. - Alícuota 3 μL de cada segundo producto de PCR de 4 nM y mezcle todo en un nuevo tubo estéril de 1,5 ml como biblioteca agrupada. Mantenga el tubo a 4 °C o en baño de hielo en todo momento.
- Mida la concentración de la biblioteca agrupada para su confirmación utilizando un espectrofotómetro de cuantificación de ácidos nucleicos. Ajuste la concentración a 4 nM si es superior a 4 nM.
NOTA: El ADN sobreconcentrado produce números de lectura sobreestimados, lo que dificulta el análisis.
- Desnaturalización y secuenciación del ADN
NOTA: Esta sección utiliza el sistema Illumina MiSeq con reactivos específicos. Ver números de producto en Tabla de Materiales.
NOTA: Siga estrictamente el tiempo. Descongele todos los reactivos excepto los cartuchos el día de la secuenciación.- Un día antes de la secuenciación, retire un cartucho de reactivo listo para usar precargado de -20 °C y guárdelo a 4 °C para descongelarlo.
- Ajuste un bloque de calor adecuado para tubos de centrífuga de 1,5 ml a 96 °C.
- Coloque el tampón de hibridación sobre hielo.
- Diluir El NaOH de grado molecular de 1 N a 0,2 N en un tubo nuevo con agua de grado PCR.
- Diluya una biblioteca de control lista para usar con tampón TE (pH 8.0) de 10 nM de stock a 4 nM en un tubo nuevo (es decir, 2 μL de biblioteca de control + 3 μL de tampón TE).
- Mezcle 16 μL de la biblioteca de muestras agrupadas de 4 nM con 4 μL de la biblioteca de control de 4 nM en un nuevo tubo etiquetado como "1".
- Mezclar 10 μL de muestra en el tubo "1" con 10 μL de 0,2 N NaOH en un tubo nuevo etiquetado como "2".
- Vórtice el tubo 2 durante 5 s, gire brevemente e incube a temperatura ambiente durante 5 min.
- Añadir 980 μL de tampón de hibridación al tubo 2.
- En un nuevo tubo etiquetado como "3", mezcle 260 μL la muestra del tubo 2 con 390 μL de tampón de hibridación. Mezclar invirtiendo el tubo.
- Incubar el tubo 3 a 96 °C durante 2 min e inmediatamente colocarlo en hielo durante un máximo de 2 min.
- Retire el cartucho del refrigerador a 4 °C.
- Configure la hoja de muestra para la secuenciación con cada adaptador de índice 1 e índice 2 correspondiente como en la Tabla 6.
- Retire la celda de flujo del kit MiSeq v3. Limpie suavemente la célula de flujo con agua estéril de PCR de grado molecular.
NOTA: No vierta agua sobre los puntos capilares de la célula de flujo. Limpie suavemente el agua de la célula de flujo con papel no fibroso. - Cargue el volumen completo del tubo 3 (650 μL) en el cartucho.
- En el software de operación del instrumento, seleccione la secuenciación y siga las instrucciones. Inserte la celda de flujo, el búfer de incorporación y el cartucho. Cargar hoja de muestra. Presione Ejecutar para iniciar la reacción.
NOTA: Esta ejecución de secuencia tardará de 3 a 5 días. Los datos se cargarán automáticamente en la plataforma Basespace. Los datos sin procesar se almacenarán en dos carpetas en la computadora: la carpeta de análisis que contiene archivos fastq y la salida que contiene archivos bcl e imágenes jpeg.
4.Asignación taxonómica a datos de secuenciación
- Procesamiento de archivos FASTQ
NOTA: DADA2 paquete23 de R24 es una de las bases de datos recomendadas para procesar archivos FASTQ. La guía está disponible en [https://github.com/mickeykawai/exec_dada2]. Esta guía se preparó adoptando la versión reciente del tutorial de canalización DADA2 [https://benjjneb.github.io/dada2/tutorial.html].- Procese secuencias de extremo emparejado FASTQ sin procesar para recortar, filtrar de calidad, desreplicar y contar secuencias únicas, inferencia de muestras, fusionarse en contigs y eliminar secuencias quiméricas.
- Utilice los siguientes parámetros especificados por DADA2 para los fragmentos de genes de ARNr 16S y ARNr 18S; trimLefts=0, 0, truncLens=[Compruebe la calidad de la secuencia y configúrela en una longitud adecuada].
NOTA: Los valores predeterminados para los otros parámetros son maxN=0, maxEE=2,2, trancQ=2.
- Asignación de taxonomía
NOTA: Se recomienda la base de datos SILVA rRNA para la asignación de taxonomía para DADA2 [silva_nr_v132_train_set.fa.gz se establece como predeterminado].- Elimine el cloroplasto y las OTU mitocondriales para el análisis de fragmentos del gen 16S rRNA. Elimine los singletons del análisis de ARNr 16S y ARNr 18S.
- Enrarifique todas las muestras a una profundidad de secuenciación uniforme basada en la muestra que tiene la profundidad de secuenciación más baja.
- Registre el número de lecturas, las OTU asignadas y en qué nivel, las lecturas filtradas y el número de singletons excluidos.
- Análisis estadístico
- Realizar análisis estadísticos sobre datos microbianos utilizando paquetes R de phyloseq25 y vegano26.
Resultados
Este protocolo utiliza el análisis de metacódigo de barras del gen 18S rRNA para identificar especies de algas en muestras de agua de mar. Un resultado representativo se muestra en la Figura 1,en la que se analizó con este protocolo el agua de mar recolectada de Metri, Puerto Montt, Chile (−41.597; −72.7056) el 19 de febrero de 2019. El resultado mostró un total de 13.750 lecturas con más de 30 especies de algas en la muestra de agua de mar. El alga dominante en esta muestra fue Navicula spp. con una abundancia relativa del 70,77%. Además, se observó suficiente abundancia para Micromonas (6,40%), Chaetoceros spp. (4,44%), Scripsiella spp. (2,44%) y Prorocentrum spp. (1.28%). Se detectó pseudochattonella spp.,uno de los causantes de algas tóxicas más altas de la HAB chilena, con un 0,52% de esta muestra de agua de mar.
Para verificar la fiabilidad de los datos, se compararon las especies de algas identificadas mediante análisis del gen 18S rRNA con las obtenidas por microscopía en la misma muestra de agua de mar(Figura 2). Consistente con el análisis del gen 18S rRNA, la microscopía mostró que la especie dominante era Navicula spp. con una abundancia relativa del 74,1% y Prorocentrum spp. (0,60%) como especie menor. Por el contrario, Heterocapsa spp. (9,04%) documentada a partir de la observación microscópica no fue identificada por el análisis del gen 18S rRNA en esta muestra. Hubo un 12,6% de células de fitoplancton pequeñas alrededor de no identificables registradas por microscopía. Esto podría ser Micromonas,según los resultados del ARNr 18S.
El protocolo utiliza el análisis de metacódigo de barras del gen 16S rRNA para identificar especies bacterianas en muestras de agua de mar. Un resultado representativo se muestra en la Figura 3,en la que se analizó el mismo agua de mar utilizada para el análisis del gen 18S rRNA con el análisis del gen 16S rRNA. El resultado mostró un total de 31.758 lecturas con más de 30 especies bacterianas en la muestra de agua de mar. Debe describirse que esta muestra de agua de mar se pasó a través de membranas de filtro en tándem (tamaños de poro de 1 μm y 0,2 μm) para separar las bacterias de vida libre de las bacterias adheridas. Luego, las células capturadas por cada membrana filtrante fueron tratadas para la extracción de ADN, seguida del análisis del gen 16S rRNA. El resultado representativo de la Figura 3 muestra especies de bacterias identificadas a partir de una membrana de tamaño de poro de 0,2 μm, que se definen como bacterias de vida libre. La bacteria de vida libre dominante fue Amylibacter spp. con una abundancia relativa del 20,02%, seguida por clade Ia (13,53%) y Aligiphilus spp. (7.06%). El resto de las especies bacterianas detectadas a partir de esta muestra de agua de mar se distribuyeron de manera relativamente equitativa. El mismo análisis se puede hacer para las células capturadas por una membrana del tamaño de un poro de 1 μm como detección de especies de bacterias adheridas.
Cuando estos ensayos de metacódigo de barras se realizan en puntos de tiempo programados durante un cierto período de estudio, los resultados se pueden resumir como análisis de series temporales. Una forma de hacerlo es trazar la abundancia relativa de especies particulares de algas y bacterias en función del tiempo para encontrar un patrón de crecimiento único. La Figura 4 muestra una gráfica representativa de series temporales de Alexandrium y Pseudochattonella en Metri, Chile. Otra forma de resumir un análisis de metacódigo de barras de series temporales es trazar todas las algas y bacterias identificadas en función del tiempo, representando el cambio poblacional de ciertos grupos de organismos. La Figura 5 y la Figura 6 resumen la abundancia relativa de todos los géneros y órdenes bacterianos, respectivamente, que se detectan en el agua de mar de Metri durante cinco meses.
Tabla 1: Primer contenido de mezcla maestra de PCR: La tabla muestra el contenido de mezcla maestra por reacción para los análisis de ARNr 16S y ARNr 18S. Las secuencias de imprimación se enumeran en la Tabla 3. Haga clic aquí para descargar esta tabla.
Tabla 2: Secuencias de imprimación: Los cebadores para la primera PCR se enumeran para los análisis de ARNr 16S y ARNr 18S. Haga clic aquí para descargar esta tabla.
Tabla 3: Ciclos de PCR: Se enumeran los ciclos térmicos para la primera PCR y la segunda PCR. Haga clic aquí para descargar esta tabla.
Tabla 4: Secuencias de índice: Se enumeran los cebadores de índice 1 (i7) e índice 2 (i5) que se utilizarán para la segunda PCR. Haga clic aquí para descargar esta tabla.
Tabla 5: Ejemplo de posicionamiento del Índice 1 y del Índice 2: Para reducir el error, los cebadores de índice deben colocarse primero en la posición, y la alícuota de cada uno se transfiere a una placa de 96 pocillos utilizando pipeta multicanal. Haga clic aquí para descargar esta tabla.
Tabla 6: Ejemplo de hoja de muestra: Antes de la secuenciación, se debe crear una hoja de muestra correspondiente a los adaptadores de índice 1 e índice 2. Haga clic aquí para descargar esta tabla.
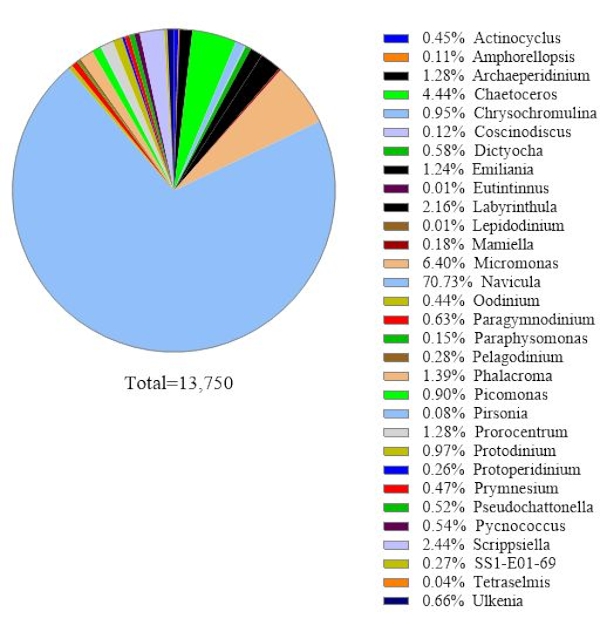
Figura 1: Resultado representativo del análisis de metabarcoding de ARNr 18S: Las especies de algas presentes en una muestra de agua de mar recolectada en Metri, Los Lagos, Chile el 19 de febrero de 2019, fueron identificadas por el ensayo de metacódigo de barras de ARNr 18S. Se eliminaron las secuencias asignadas como "desconocidas" y se trazó la abundancia relativa de cada especie identificada. Haga clic aquí para ver una versión más grande de esta figura.
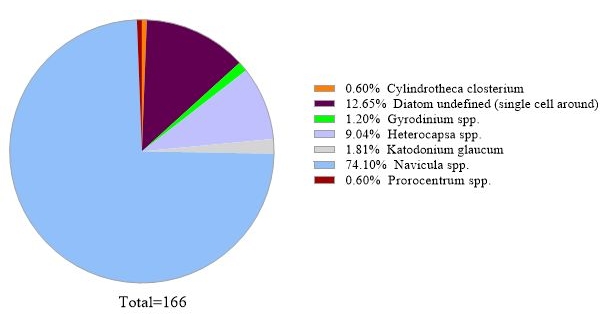
Figura 2: Resultado representativo del análisis microscópico: Las especies de algas fueron identificadas a partir de una muestra de agua de Metri, Los Lagos, Chile el 19 de febrero de 2019, por microscopía. La cantidad de cada especie se contó manualmente y se trazó. Haga clic aquí para ver una versión más grande de esta figura.
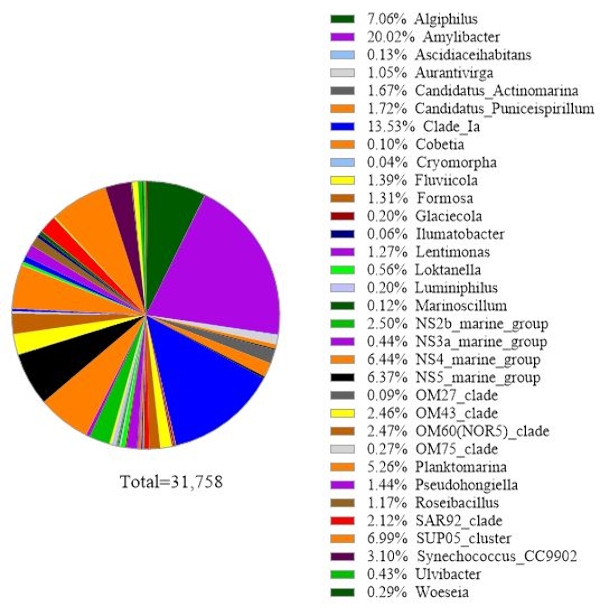
Figura 3: Resultado representativo del análisis de metabarcoding 16S rRNA: Las especies bacterianas presentes en una muestra de agua de Metri, Los Lagos, Chile el 19 de febrero de 2019, fueron identificadas por el ensayo de metacódigo de barras 16S rRNA. Se trazó la abundancia relativa de cada especie identificada. Haga clic aquí para ver una versión más grande de esta figura.
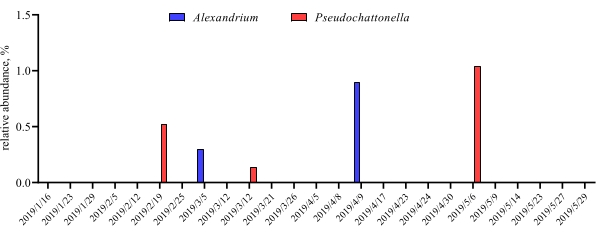
Figura 4: Gráfica representativa de series temporales de Alexandrium spp. y Pseudochattonella spp. obtenida a partir del metacódigo de barras 18S rRNA en Metri, Chile: Figura reimpresa de Yarimizu et al9. Las dos especies de algas tóxicas, Alexandrium y Pseudochattonella fueron monitoreadas selectivamente por análisis de ARNr 18S a lo largo del tiempo, y la abundancia relativa se trazó en función del punto de tiempo. Haga clic aquí para ver una versión más grande de esta figura.
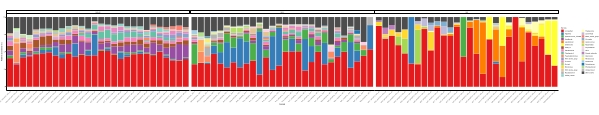
Figura 5: Diagrama de series temporales de género representativo obtenido a partir del metacódigo de barras de ARNr 16S y ARNr 18S: Todos los géneros bacterianos y eucariotas identificados en el agua de Metri, Chile, monitoreados durante cinco meses. Haga clic aquí para ver una versión más grande de esta figura.
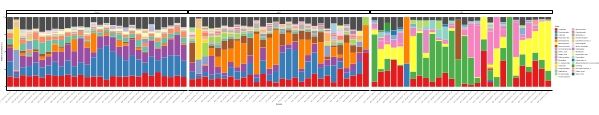
Figura 6: Gráfico de series temporales de orden representativo obtenido a partir del metacódigo de barras de ARNr 16S y ARNr 18S: Todos los órdenes bacterianos y eucariotas identificados en el agua de Metri, Chile, monitoreados durante cinco meses. Haga clic aquí para ver una versión más grande de esta figura.
Cuadro S1 y Cuadro S2:
Tabla S1: Datos brutos para la Figura 5 y la Figura 6. Tabla S2: Resultado de la verificación del control de calidad de los datos brutos de la Figura 5 y la Figura 6. Haga clic aquí para descargar estas tablas.
Discusión
Este protocolo realizó con éxito el análisis de metacódigo de barras de genes 18S rRNA y 16S rRNA para identificar especies de algas y bacterias en muestras de agua de mar para el monitoreo de HABs chilenos. Las algas dominantes y algunas algas menores detectadas por este protocolo fueron consistentes con las obtenidas por microscopía, confirmando la fiabilidad del protocolo. Lo más destacado del protocolo es que el análisis detectó Alexandrium spp. y Pseudochattonella spp.,las dos especies causantes de HAB más problemáticas en el sur de Chile, a partir del agua de mar de Metri incluso en una abundancia baja. Especialmente, Pseudochattonella spp. son especies difíciles de identificar bajo un microscopio porque las células son pequeñas y se rompen fácilmente bajo la luz o el uso de fijadores27,28. El metacódigo de barras puede proporcionar información sobre especies de algas que apenas está presente en muestras de agua de mar que la microscopía no puede identificar. El protocolo también detectó más de 30 especies bacterianas. Aunque se desconoce cómo estas bacterias se relacionan con el crecimiento de algas en este momento, la recopilación masiva de datos de especies de algas y bacterias en el análisis de series temporales posiblemente proporcionará descubrimientos tan importantes. Por lo tanto, agregar el análisis de metacódigo de barras a los programas convencionales de monitoreo de HAB chilenos sin duda fortalecerá la eficiencia actual de monitoreo de HAB. De hecho, este protocolo visual para el análisis de metacódigos de barras puede ser beneficioso no solo para el monitoreo de algas y bacterias del agua chilena, sino también para los programas de monitoreo de costas en otras áreas afectadas por HAB en todo el mundo.
Aunque este protocolo proporcionó las ventajas mencionadas anteriormente, se deben discutir los inconvenientes del método. Como se ve en el protocolo visual, el método de metacódigo de barras consume mucho tiempo y es complejo, y requiere maquinaria y reactivos costosos. El personal del laboratorio debe estar especialmente capacitado, o desperdiciará material, mano de obra y tiempo. Además, la detección de algas por metacódigo de barras debe emparejarse con microscopía para verificar que las mismas especies dominantes se identifican a partir de los dos métodos ortogonales29. La microscopía es una herramienta no invasiva, en su mayor parte, para identificar especies de algas, lo que significa que es más difícil cometer errores cuando las especies de algas tienen formas únicas y aparentes. Por supuesto, el error humano puede ocurrir si las especies tienen formas muy similares entre sí. Por otro lado, dado que el análisis de metacódigo de barras requiere múltiples pasos, naturalmente aumenta los errores. Podría ser una muestra mezclada, una adición incorrecta de reactivos o la falta de algunos procedimientos. Por lo tanto, es fundamental comparar los resultados del metacódigo de barras con los obtenidos por microscopía. Por último, el protocolo es aplicable sólo cualitativamente, y los resultados deben calcularse para la abundancia relativa. Como Lamb et al. declararon en 2019, el metacódigo de barras actual como rendimiento cuantitativo aún es limitado, y se requiere investigación adicional antes de que el metacódigo de barras pueda utilizarse con confianza para aplicaciones cuantitativas30.
Este protocolo fue optimizado para 140 - 170 muestras por ejecución del manual de preparación de la biblioteca de secuenciación metagenómica 16S publicado por Illumina Inc. El protocolo optimizado fue probado muchas veces y modificado para emitir esta versión final. Por lo tanto, es muy recomendable seguir cada paso con precisión. La parte más crítica del protocolo es que se requiere un cuidado adicional para evitar la contaminación de la muestra. Las pipetas y la campana de flujo laminar deben limpiarse con alcohol al 70% y exposición a los rayos UV en todo momento, y los materiales esterilizables deben ser esterilizados en autoclave. Los cebadores y reactivos deben diluirse directamente de la cepa en cada nueva tirada, o la reutilización de reactivos y la exposición a varias diluciones pueden ser una causa de contaminación de la muestra. El protocolo especifica cuándo se puede realizar la operación fuera de la campana de flujo laminar. A menos que sea lo contrario, las muestras deben tratarse en una campana de flujo laminar limpia. Cuando se trata de múltiples tiras de 8 tubos simultáneamente, se recomienda trabajar en la primera tira de 8 tubos y taparla antes de pasar a la siguiente tira de 8 tubos. Dejar la tapa abierta durante mucho tiempo puede causar contaminación de la muestra, ya que los genes 16S y 18S rRNA son altamente omnipresentes en los alrededores. El tiempo indicado, como el tiempo de mezcla y el tiempo de incubación, debe seguirse como se describe exactamente porque la mejor duración para cada paso se seleccionó en función de las múltiples ejecuciones de prueba. El exceso o la insuficiencia de tiempo pueden, por ejemplo, reducir el rendimiento de la muestra. El proceso debe detenerse solo en la parte que el protocolo dice que puede detenerse. Es crucial tener un control negativo, ya que confirma que los resultados positivos no son falsos positivos artificiales. Por último, es muy recomendable planificar los días porque se requieren al menos cinco días para procesar todos los pasos, y algunas partes no se pueden detener hasta la siguiente señal de parada.
La limitación de la asignación taxonómica para datos secuenciados se señala brevemente aquí. Las secuencias de nucleótidos recientemente descubiertas para especies bacterianas y de algas se actualizan diariamente en el banco de datos. Si bien las especies de algas y bacterias bien estudiadas se registran de manera confiable, también hay muchas secuencias para especies desconocidas actualizadas en el banco de datos. Indica que la resolución de las secuencias de nucleótidos registradas no siempre es suficiente para la identificación de especies, sino solo para la identificación de géneros. Por lo tanto, la identificación microscópica de especies debe realizarse ortogonalmente. Establecer una canalización de acceso abierto para este trabajo tiene una ventaja significativa al tratar con un gran conjunto de datos secuenciados a la vez e investigar de manera eficiente cuándo ocurre un error. Además, la salida de DADA2 está en un archivo de texto o csv, lo que facilita el análisis estadístico adicional. Por otro lado, se requiere un servidor grande para realizar asignaciones taxonómicas, especialmente cuando un conjunto de datos es grande. Para configurar la tubería, se necesitan ingenieros para configurar la tubería, y los profesionales deben comprender los parámetros, la operación, Linux y la bioinformática.
Aparte del alcance como herramienta de monitoreo de HAB, el uso de este protocolo se puede ampliar con fines de investigación. Por ejemplo, hay debates en curso entre el gobierno chileno y los pescadores locales junto con las asociaciones de paz ambiental con respecto al aumento del número de HABlocales 31,32. El gobierno afirma que se debe a un fenómeno natural como el calentamiento global y El Niño, mientras que las partes posteriores afirman que la acuicultura del salmón es la causa. El salmón no es una especie autóctona de Chile y no estaba en aguas chilenas hace medio siglo. El gobierno chileno en ese momento tenía como objetivo hacer crecer la economía y crear empleos para las zonas pobres mediante el desarrollo de un negocio de salmón33. Con la intervención del extranjero para obtener ganancias de capital, Chile tuvo un gran éxito en el desarrollo de la acuicultura estilo corral, y el número de salmones ha aumentado notablemente en las últimas décadas31,32,33. Posteriormente, una gran cantidad de alimentos para el salmón ha sido arrojada al mar, lo que lleva a la población local a sospechar que la acuicultura del salmón es la razón de los frecuentes HABs locales31,32. La verdad se desconoce ahora, pero debe entenderse eventualmente para que se puedan idear estrategias para proteger el medio ambiente marino local, la economía y la salud humana de las HAB. El análisis molecular de la relación local entre algas y bacterias puede contribuir a una aclaración de un paso adelante para dicho tema. Por ejemplo, la técnica puede buscar las especies de algas y bacterias que no estaban presentes en el área oceánica objetivo antes, pero que han aumentado dramáticamente en los últimos años, lo que es algo similar al estudio realizado por Sakai et al. para el descubrimiento de una población no registrada de salamandra Yamato(Hynobius vandenburghi)mediante análisis GIS y eDNA34. Por lo tanto, más allá de la herramienta de monitoreo HAB, este protocolo de metacódigo de barras tiene el uso potencial para otros prospectos.
Divulgaciones
Los autores declaran no tener conflicto de intereses. Los financiadores no tuvieron ningún papel en el diseño del estudio, en la recopilación, análisis o interpretación de datos; en la redacción del manuscrito, o en la decisión de publicar los resultados.
Agradecimientos
Este estudio fue apoyado por la subvención (JPMJSA1705) para un estudio sobre la Asociación de Investigación en Ciencia y Tecnología para el Desarrollo Sostenible-Monitoreo de Algas en Chile (SATREPS-MACH). Agradecemos a la Dra. Sandra Ríos (Universidad de Los Lagos) por permitirnos usar un clip de película. Agradecemos a Neal Andrew Holland por su diligente revisión del manuscrito. Expresamos nuestro sincero agradecimiento a los miembros del grupo de laboratorio de CREAN-IFOP en Puerto Montt, Chile, por asesorarnos en la identificación del fitoplancton.
Materiales
| Name | Company | Catalog Number | Comments |
| 1.5 mL single tube | ThermoFisher | Q32856 | sterile |
| 1.5 mL tubes | ThermoFisher | 2150N | sterile |
| 8-strip 0.2 mL PCR tubes and flat cap | ThermoFisher | AB0451 & 4323032 | sterile |
| 96-well 0.2 mL PCR plate | ThermoFisher | N8010560 | |
| AccuRuler 100 bp DNA Ladder | MaestroGen | 02001-500 | |
| Chelex 100 Chelating Resin | Bio-Rad | 1432832 | |
| Chemical Duty Vacuum Pressure Pump | MilliporeSigma | WP6122050 | |
| D1000 Reagents | Agilent Technologies | 5067-5583 | |
| D1000 sample buffer | Agilent Technologies | 5067-5602 | |
| DNA loading dye (Blue/Orange Loading Dye 6X) | Promega | G1881 | |
| Ethanol Molecular Biology Grade | E7023 | Merck KGaA | |
| Filtration device | ThermoFisher | 09-740-36H, 09-740-23E | |
| Fluorescent DNA concentration measurement kit (Qubit dsDNA HS Assay kit) | Thermo Fisher Scientific | Q32851 | |
| Fluorometer (Qubit4 Fluorometer) | ThermoFisher | Q33238 | |
| Fragment analysis verification matrix (D1000 ScreenTape) | Agilent Technologies | 5067-5582 | |
| Fragment Analyzer (Agilent Tapestation 4150) | Agilent | G2992AA | |
| GelRed Nucleic Acid Gel Stain | Biotium | 41002 | |
| Generic bench vortexer (Vortex Genie 2) | Scientific Industries | SI-0236 | |
| Heat block | |||
| High performance sequencing-grade DNA polymerase (KAPA HiFi Hotstart ReadyMix), 2X | Roche | KK2602 | |
| HT1 Hybridization buffer | Illumina | 20015892 | |
| Illumina Nextera XT v2 Index Kit set A, B, C and D | Illumina | FC-131-2001, -2002, -2003, -2004 | |
| Laminar hood cabinet | Biobase Co. | Biobase PCR-800 PCR Cabinet | |
| Loading tips (Specific for Agilent TapeStation systems) | Agilent Technologies | 5067-5598 | |
| Magnetic beads DNA cleanup system (ProNex® Size-Selective Purification System) | Promega | NG2002 | |
| Magnetic stand for 96 wells (Magnetic Stand-96) | ThermoFisher | AM10027 | |
| Micro-seal film for 96-well plate | ThermoFisher | 4311971 | sterile |
| MiSeq Reagent Kit v3 | Illumina | MS-102-3003 | includes pre-filled, ready-to-use reagent cartridges. |
| MiSeq system | Illumina | SY-410-1003 | includes pre-installed software |
| Molecular grade nuclease-free water | Integrated DNA Technologies | 11-05-01-04 | |
| Molecular grade sodium hydroxide (NaOH) 1M | Merck KGaA | 1091371000 | |
| Multichannel pipettes | Not specified | Not specified | |
| Multi-Purpose Agarose | Cleaver Scientific | CSL-AG500 | |
| Ow D2 Wide-Gel Electrophoresis System | ThermoFisher | D2 | |
| Ow EC-105 Compact Power Supply | ThermoFisher | 105ECA-115 | |
| Pellet Pestle— Cordless Motor | ThermoFisher | K749540-0000 | |
| PhiX control Kit v3 | Illumina | FC-110-300 | ready-to-use control library for Illumina sequencing |
| Pipette tips | Not specified | Not specified | sterile |
| Pipettes | Not specified | Not specified | 2, 10, 20, 100, 1000 μL |
| SterivexTM GP 0.22 μm filter unit | MilliporeSigma | SVGP01050 | |
| Tabletop centrifuge | Eppendorf 5427R Centrifuge | Eppendorf AG | |
| TBE buffer | ThermoFisher | B52 | |
| TE buffer (pH 8.0) | ThermoFisher | AM9849 | |
| Terr PCR Direct FFPE Kit | Takara Bio USA | 639284 | includes 2X Terra PCR Direct Buffer |
| Terra PCR Direct Polymerase Mix, 1.25 U/μL | Takara Bio USA | 639271 | High performance Inhibitor-tolerant DNA polymerase |
| ThermalCycler (MiniAMP Plus Thermal Cycler) | ThermoFisher | A37835 | |
| TransIlluminator and image capture system | Analytik Jena, AG | GelDoc-ItTS2 Imager | |
| Whatman 1.0 m pore-sized membrane | MilliporeSigma | WHA111110 |
Referencias
- Hallegraeff, G., Enevoldsen, H., Zingone, A. Global harmful algal bloom status reporting. Harmful Algae. 102, 101992 (2021).
- Anderson, D. M. Red Tides. Scientific American. 271 (2), 52-58 (1994).
- Sanseverino, I., Conduto, A. D. S., Pozzoli, L., Dobricic, S., Lettieri, T. . Algal Bloom and its Economic Impact. , (2016).
- Molinet, C., Niklitschek, E., Seguel, M., Díaz, P. Trends of natural accumulation and detoxification of paralytic shellfish poison in two bivalves from the Norwest Patagonian inland sea. Revista de Biologia Marina Y Oceanografia. 45, 195-204 (2010).
- Mardones, J., Clement, A., Rojas, X., Aparicio, C. Alexandrium catenella during 2009 in Chilean waters, and recent expansion to coastal ocean. Harmful Algae News. 41, 8-9 (2010).
- Alvarez, G., et al. Paralytic shellfish toxins in surf clams Mesodesma donacium during a large bloom of Alexandrium catenella dinoflagellates associated to an intense shellfish mass mortality. Toxins (Basel). 11 (4), (2019).
- Clément, A., et al. Exceptional summer conditions and HABs of Pseudochattonella in Southern Chile create record impacts on salmon farms. Harmful Algae News. 53, 1-3 (2016).
- Trainer, V. L., et al. Pelagic harmful algal blooms and climate change: Lessons from nature's experiments with extremes. Harmful Algae. 91, 101591 (2020).
- Yarimizu, K., et al. Protocols for monitoring harmful algal blooms for sustainable aquaculture and coastal fisheries in Chile. International Journal of Environmental Research and Public Health. 17 (20), (2020).
- Roegner, G. C., Hickey, B. M., Newton, J. A., Shanks, A. L., Armstrong, D. A. Wind-induced plume and bloom intrusions into Willapa Bay, Washington. Limnology & Oceanography. 47 (4), 1033-1042 (2002).
- Tweddle, J. F., et al. Relationships among upwelling, phytoplankton blooms, and phycotoxins in coastal Oregon shellfish. Marine Ecology Progress Series. 405, 131-145 (2010).
- Glibert, P. M., Anderson, D. M., Gentien, P., Graneli, E., Sellner, K. G. The global complex phenomena of harmful algae blooms. Oceanography. 18 (2), 130-141 (2005).
- Paredes-Mella, J., Varela, D., Fernández, P., Espinoza-González, O. Growth performance of Alexandrium catenella from the Chilean fjords under different environmental drivers: Plasticity as a response to a highly variable environment. Journal of Plankton Research. 42 (2), 119-134 (2020).
- Azam, F., Malfatti, F. Microbial structuring of marine ecosystems. Nature Reviews Microbiology. 5, 782-791 (2007).
- Amin, S. A., et al. Interaction and signaling between a cosmopolitan phytoplankton and associated bacteria. Nature. 522, 98-101 (2015).
- Berdalet, E., et al. Marine harmful algal blooms, human health, and wellbeing: Challenges and opportunities in the 21st century. Journal of Marine Biology Association. 2015, 1-31 (2015).
- Ramanan, R., Kim, B. -. H., Cho, D. -. H., Oh, H. -. M., Kim, H. -. S. Algae-bacteria interactions: Evolution, ecology, and emerging applications. Biotechnology Advances. 34 (1), 14-29 (2016).
- Seymour, J. R., Amin, S. A., Raina, J. B., Stocker, R. Zooming in on the phycosphere: The ecological interface for phytoplankton-bacteria relationships. Nature Reviews Microbiology. 2, 17065 (2017).
- Maruyama, F., et al. 16S and 18S Metabarcoding analysis for Chilean coastal waters harmful algal blooms. protocol.io. , (2020).
- Tanabe, A. S., et al. Comparative study of the validity of three regions of the 18S-rRNA gene for massively parallel sequencing-based monitoring of the planktonic eukaryote community. Molecular Ecology Resources. 16, 402-414 (2016).
- Nishitani, G., et al. Multiple plastids collected by the Dinoflagellate Dinophysis mitra through Kleptoplastidy. Applied and Environmental Microbiology. 78 (3), 813-821 (2012).
- Klindworth, A., et al. Evaluation of general 16S ribosomal RNA gene PCR primers for classical and next-generation sequencing-based diversity studies. Nucleic Acids Research. 41 (1), 1 (2013).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nature Methods. 13 (7), 581-583 (2016).
- McMurdie, P. J., Holmes, S. phyloseq: An R Package for Reproducible Interactive Analysis and Graphics of Microbiome Census Data. PLoS One. 8 (4), 61217 (2013).
- Dixon, P. VEGAN, a package of R functions for community ecology. Journal of Vegetation Science. 14 (6), 927-930 (2003).
- Dittami, S. M., Riisberg, I., Edvardsen, B. Molecular probes for the detection and identification of ichthyotoxic marine microalgae of the genus Pseudochattonella (Dictyochophyceae, Ochrophyta). Environmental Science and Pollution Research. 20 (10), 6824-6837 (2013).
- Mardones, J. I., et al. Salinity-growth response and ichthyotoxic potency of the Chilean Pseudochattonella verruculosa. Frontiers in Marine Science. 6 (24), (2019).
- Santi, I., Kasapidis, P., Karakassis, I., Pitta, P. A comparison of DNA metabarcoding and microscopy methodologies for the study of aquatic microbial eukaryotes. Diversity. 13 (5), 180 (2021).
- Lamb, P. D., et al. How quantitative is metabarcoding: A meta-analytical approach. Molecular Ecology. 28 (2), 420-430 (2019).
- Mascareño, A., et al. Controversies in social-ecological systems: lessons from a major red tide crisis on Chiloe Island, Chile. Ecology and Society. 23, (2018).
- Armijo, J., Oerder, V., Auger, P. -. A., Bravo, A., Molina, E. The 2016 red tide crisis in southern Chile: Possible influence of the mass oceanic dumping of dead salmons. Marine Pollution Bulletin. 150, 110603 (2020).
- Swanson, H. A. . Caught in Comparisons: Japanese Salmon in an Uneven World. , (2013).
- Sakai, Y., et al. Discovery of an unrecorded population of Yamato salamander (Hynobius vandenburghi) by GIS and eDNA analysis. Environmental DNA. 1 (3), 281-289 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados