Method Article
메타바코딩 분석을 통해 칠레의 유해한 조류 꽃을 모니터링하기 위한 표준화된 절차
요약
이 프로토콜은 해수 샘플에서 유해한 조류 꽃과 관련 미생물군유전체를 모니터링하기 위해 16S rRNA 및 18S rRNA 유전자를 대상으로 메타바코딩 분석 단계를 소개합니다. 그것은 강력한 분자 기반 도구이지만 시각적으로 단계별로 여기에 설명되는 몇 가지 절차가 필요합니다.
초록
유해한 조류 꽃 (HABs) 모니터링은 전 세계적으로 구현되었으며, 수산과 양식으로 유명한 칠레는 이러한 목적을 위해 수십 년 동안 현미경 및 독소 분석을 집중적으로 사용했습니다. 고처리량 DNA 염기서열 분석 및 세균 조립 기반 접근법과 같은 분자 생물학적 방법은 칠레 HAB 모니터링에서 도입되기 시작했으며 절차는 아직 표준화되지 않았습니다. 여기서, 칠레 HABs를 모니터링하기 위한 16S rRNA 및 18S rRNA 메타바코딩 분석은 단계적으로 도입된다. 최근 가설에 따르면, 조류 세균 상호 연관은 꽃 개시, 유지 보수 및 회귀를 차지하는 중요한 시너지 또는 적대적 관계를 재생합니다. 따라서, 조류 세균성 관점에서 HAB를 모니터링하는 것은 HAB 메커니즘과 조기 경고의 기초를 더 폭넓게 이해할 수 있다. 메타바코딩 분석은 샘플에서 거대한 조류 세균 분류증 정보를 감지할 수 있기 때문에 이 목적을 위해 가장 적합한 분자 기반 도구 중 하나입니다. 본 원에서 메타바코딩 분석에 대한 샘플링의 시각적 절차는 신뢰할 수 있는 데이터의 오류 및 수집을 줄이는 것을 목표로 하는 구체적인 지침을 제공합니다.
서문
많은 해양 식물성 식물성 플랑크톤 종은 내인성 독소를 생산하는 것으로 알려져 있으며,이 종은 충분한 수로 축적되면 해양 환경에 해롭습니다. 이러한 유해한 조류 꽃 (HABs)는 세계1의대부분의 대륙의 해안에서 오늘 관찰된다. 독성 식물성 플랑크톤은 먼저 쌍발판 조직에 축적되어 소화 시 인간을 포함한 더 높은 열대 지방 체 수준에서 질병과 사망으로 이어집니다. 그 후, 이러한 사건은 지역 경제, 사회 경제학 및 공중 보건2에심각한 영향을 미칩니다. HAB로 인한 세계 경제의 피해는매년수백만~수십억 달러로 추산됩니다 3. 칠레는 빈번한 HABs로 고통받는 많은 국가 중 하나입니다.
칠레는 북쪽과 남쪽으로 4,300km가 넘는 긴 땅의 나라입니다. 길쭉한 서부 땅은 태평양을 마주하고 있으며, 이는 칠레가 HAB를 경험할 확률을 자연스럽게 증가시킵니다. 특히 칠레 남부에는 세계적으로 유명한 연어 양식이 많으며, HAB가 이 지역에서 발생할 때마다 조류 독소는 거대한 양식 연어가 아프고4,5,6로죽는다. 칠레에서는 HAB가 경제를 가장 힘들게 강타한 해는 2016년으로, 연간 8억 달러의7,8달러의손실을 기록했습니다. 인과 성 독성 조류 종은 해마다 다양합니다. 2016 년 경우, 알렉산드리움 카타넬라와 슈도차토넬라 verruclosa의 단지는 남부 칠레 해안7의대부분에 광범위한 HAB를 발생7,8. 칠레에서 가장 최근의 HAB는 2021년 3월 칠레 남부카만차카에 위치한 이테로시그마 아카시우의 조류종과 함께 이 지역에 큰 연어 농장이 있는 곳에서 일어났습니다.
칠레는 두 가지 주요 방법을 사용하여 수년 동안 해안 모니터링을 수행해 왔습니다. 현미경으로 바닷물을 관찰하여 독성 조류 종을 정기적으로 식별하고 생화학적 분석을 통해 조개에서 독소 수준을측정합니다 9. 조개에서 독성 조류 및 독소 수준의 조기 검출은 HAB를 방지하지 않습니다; 그러나 이러한 분석은 즉각적인 대책을 촉발하고 지역 사회에 대한 피해를 줄일 수 있습니다. 이 전략의 효과를 더욱 강화하기 위해 최근 기존의 칠레 HAB 모니터링 프로그램에 분자 기반 분석 방법이 추가되어 해수 샘플에서 조류 및 관련 세균 지역 사회를 검출했습니다. 구체적으로, 16S rRNA 및 18S rRNA 유전자를 표적으로 하는 메타바코딩을 이용한 대규모 병렬 시퀀싱 방법이 선택되었다. 이 기술은 복잡한 절차와 비싼 기계 및 시약을 필요로하지만, 한 번에 해수 샘플에 존재하는 조류와 박테리아 제네라 / 종의 수천을 감지 할 수있는 고급 기술이다.
HAB의 원인은 온도와 계절 등 다양한 것으로 추측되지만 일반화하는 것은 불가능합니다. 이는 HAB 종및 주파수가 지역 및 현면 상태에 의존하기 때문에, 지리적 고유성, 영양 혼합 및 원소 유출과 같은 자연적인 현상을 수반하는 것은 2,10,11,12로인한 토지로부터의 원소 유출이다. 또한, 부영양화와 같은 인공적 인자는 지역 HABs12,13에영향을 미친다. 복잡한 다인자로 인해 정확한 HAB 예측을 하기가 쉽지 않습니다. 최근 몇 년 동안, 특정 세균 성 인구가 요인 중 하나로서 HABs의 발달과 관련이있을 수 있다는 견해가 있으며,이 가설을 지원하기 위한 연구는14,15,16,17,18로점점 더 분명해지고 있다. 분자 생물학 기술은 일반적으로 세균 성 조립을 연구하는 데 사용됩니다. 그러나, 이러한 표준화된 방법은 칠레 HAB 모니터링9에아직 확립되지 않았다. HAB와 조류 세균 성 협회를 공부하기 위해, 동시에 현재 칠레 해안 모니터링 프로그램에 대한 메타 바코딩 분석을 수행하는 것이 필수적이다. 따라서, 이 프로토콜은 메타바코딩 분석을 사용하여 해수 샘플에서 조류 및 박테리아 종 검출을 분석하기 위한 단계별 절차에 초점을 맞춘 칠레 HAB 모니터링 프로그램을 시각적으로 소개합니다.
우리의 칠레 HAB 모니터링 프로그램을 설명하는 전체 프로토콜은 야리미즈 외9에서사용할 수 있습니다. 여기에는 해수 샘플링, 미세한 조류 종 검출, 조류 세균 유전자 검출, 안료 분석, 기상 데이터 수집 및 물리적 및 화학적 물 특성 분석 절차가 포함됩니다. 조류 및 세균성 종 검출을 위한 16S rRNA 및 18S rRNA 메타바코딩 분석의 단계별 프로토콜은프리프린트(19)로이용된다. 이 프로토콜은 HAB 모니터링 프로그램의 가장 복잡한 부분이자 하이라이트이기 때문에 특히 메타바코딩 분석 단계를 보여 줍니다. 이 프로토콜은 또한 조류 종의 프로그램 및 현미경 검출의 소개를 포함합니다. 대사코딩에 의해 조류 종을 분석할 때, 동시에 현미경 검사를 수행하여 두 가지 방법으로부터 결과를 확인하는 것이 중요합니다. 이 프로토콜에는 분류 할당에 소프트웨어를 사용하는 방법은 포함되지 않지만 데이터베이스 권장 사항은 다음 섹션의 끝에 간략하게 명시되어 있습니다.
프로토콜
1. 샘플 수집 및 전처리
- 대상 지점에서 약 3L의 물 샘플을 수집합니다.
- 16S rRNA 분석을 위한 1L의 물 샘플을 탠덤 여과(1 μm 및 0.2 μm 및 0.2 μm 모공 크기 멤브레인)을 통해 자유 생활 및 부착된 박테리아를 분리합니다.
- 0.2 μm 멤브레인을 가진 단일 여과를 통해 18S-rRNA 분석(식물성 플랑크톤 검출)을 위한 또 다른 1 L의 물 샘플을 필터링합니다.
참고: 물 샘플의 여과는 샘플링의 12 시간 이내에 완료해야합니다. - 멸균 수술 가위로 여과 된 멤브레인을 반으로 자르고 알루미늄 호일로 감쌉니까. -20°C에 최대 1개월 동안 보관하거나 다음 단계로 진행하십시오.
- 설명된9와같이 Chelex 방법으로 DNA를 추출합니다. 최대 1개월 동안 -20°C에 보관하십시오.
2. 현미경 분석
- 1mL 그리드 슬라이드에 파이펫으로 물 샘플 1mL를 전송합니다.
- 현미경으로 샘플을 관찰하십시오.
- 피토플랑크톤 종의 이름과 양을 기록합니다.
3. 16S rRNA 및 18S rRNA 메타바코딩 분석
참고: 이 과정은 제제, 제1 PCR 앰플리턴 생성, 제1 앰플리턴 클렌징, 제2 PCR 증폭기별 색인화, 제2 PCR 증폭기 검증 및 정리, DNA 농도 조정, DNA 절전 및 시퀀싱의 일곱 부분으로 구성됩니다. 전체 프로세스는 숙련된 실험실 직원이 최소 5일(40시간)이 소요됩니다. 제품 번호 및 제조를 위한 재료 표를 참조하십시오.
- 준비
- 70%에탄올이 있는 깨끗한 파이펫과 라미나르 후드 캐비닛이 30분 동안 자외선 노출을 자주 합니다. 사용할 재료를 살균합니다.
- 실온에서 DNA 샘플을 해동하고, 원심분리기는 100 x g에서 2분 동안 해동하고, 각 시료의 100 μL을 8관 스트립으로 옮긴다.
- 최초의 PCR 앰플리턴 생성
참고: 라미나르 후드 캐비닛에서 다음 절차를 수행합니다. 프라이머 오염을 피하기 위해 항상 1 μM 스톡에서 목표 농도로 프라이머를 희석시 PCR 등급의 물로 희석하십시오.- 반응(표 1, 표 2)에대한 멸균 1.5 mL 튜브에서 첫 번째 PCR 마스터 믹스를 준비합니다.
- 8튜브 스트립에 마스터 믹스의 Aliquot 22.5 μL을 추가하고 DNA 샘플의 2.5 μL을 추가합니다. 음의 제어를 위해 PCR 등급 물의 2.5 μL을 사용합니다.
- 첫 번째 PCR주기(표 3)를실행합니다.
- 1x 핵산젤 얼룩의 10μL을 함유한 아가로즈-TBE 젤 의 100mL를 준비한다.
- 아가로즈 젤에 1x DNA 적재 염료의 1.5 μL과 PCR 제품의 4 μL의 혼합물을 적재한다. 또한, 젤에 100 bp DNA 사다리를 적재하십시오.
- 30 분 동안 100 V에서 전기 전도를 수행합니다.
- UV 라이트 이미지 캡처 아래에 500-600 bp 범위의 밴드가 있는지 확인합니다. 프라이머-디머 밴드는 라운드 80 bp입니다.
참고: 해양 수분 샘플에는 PCR 억제제가 포함되어 있습니다. 누락 된 앰플리통은 때때로 PCR 등급의 물로 샘플 1:100 또는 1:1000을 희석하여 해결할 수 있습니다. - 다음 단계까지 -20°C에 첫 번째 PCR 제품을 저장합니다.
주의: 1주일의 저장 공간을 초과하지 마십시오.
- 첫 번째 PCR 앰플리턴 정리
참고: 이 섹션은 라미나르 후드 캐비닛 외부에서 수행할 수 있습니다.- 마그네틱 비즈 DNA 정리 시스템을 사용하여 프라이머-이머 제품을 포함한 PCR 반응 잔류물을 제거합니다.
- 각 청소된 첫 번째 PCR 제품의 20 μL을 새로운 96 웰 플레이트로 옮기습니다. 마이크로 씰 필름으로 접시를 밀봉합니다. 다음 단계가 진행될 때까지 -20°C에 보관하십시오.
주의: 1주일 이상의 저장소를 초과하지 마십시오.
- 두 번째 PCR별 색인
참고: 이 섹션에서는 정제된 첫 번째 PCR 제품이 다양한 인덱스 프라이머 조합으로 증폭됩니다.- 모든 인덱스 1 및 인덱스 2 프라이머(표4)를라미나르 후드 캐비닛에 배치된 8개의 튜브 PCR 스트립에 PCR 등급의 물로 1 μM에서 1 μM로 희석합니다.
참고: 인덱스 프라이머가 첫 번째 PCR 반응 오버행 어댑터에 특정하므로 이 섹션의 단계는 라미나르 후드 캐비닛 외부에서 수행할 수 있습니다. - 세로행(표 5)의수평 행 인덱스 2 프라이머에서 위치 인덱스 1 프라이머.
- 새로운 96웰 플레이트에 각 웰에 12.5 μL의 핫 스타트 준비 제형을 추가합니다.
- 멀티채널 파이펫을 사용하여 표 4에 각 인덱스 프라이머(1 μM)의 2.5 μL을 추가합니다.
- 정제된 첫 번째 PCR 제품의 7.5 μL을 추가합니다.
- 10번 위아래로 피펫을 하여 부드럽게 섞습니다. 마이크로 씰 필름으로 접시를 덮습니다.
- 두 번째 PCR주기(표 3)를실행합니다.
- 플레이트를 -20°C로 유지합니다.
주의: 1주일의 저장 공간을 초과하지 마십시오.
- 모든 인덱스 1 및 인덱스 2 프라이머(표4)를라미나르 후드 캐비닛에 배치된 8개의 튜브 PCR 스트립에 PCR 등급의 물로 1 μM에서 1 μM로 희석합니다.
- 두 번째 PCR 앰플리턴 검증 및 정리
- 조각 분석기 및 관련 시약을 사용합니다. 사용 전에 소용돌이와 시약을 회전하십시오.
- 샘플 버퍼 및 DNA 스크린 테이프가 30분 동안 실내 온도에서 평형화되도록 합니다. 그런 다음 DNA 스크린 테이프를 단편 분석기에 배치합니다.
- 새로운 8 튜브 스트립에 샘플 버퍼 2 μL과 두 번째 PCR 앰플리턴의 3 μL을 혼합합니다. 8개의 튜브 스트립을 조각 분석기에 삽입합니다. 실행을 눌러 시작합니다.
주의: 기포형성은 피해야 합니다. - 두 번째 PCR 앰플리컨이 16S 및 18S rRNA 유전자 모두에 대해 약 613 bp (600 - 630 bp)인지 확인하십시오.
- 마그네틱 비즈 DNA 정화 시스템을 사용하여 두 번째 PCR 제품을 정화합니다.
- DNA 농도 조정
- 핵산 정량화 분광광계를 사용하여 정제된 제2 PCR 제품에서 DNA 농도를 측정합니다.
- 16S 및 18S rRNA 유전자 모두에 대해 평균 최종 라이브러리 크기를 613 bp로 계산하는 nM에서 표적 유전자 농도를 계산합니다.
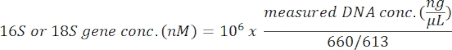
- 멸균 PCR 물로 정제된 두 번째 PCR 제품을 새로운 0.2mL 96 웰 플레이트에 4nM로 희석합니다.
참고 : 플레이트는 여기에 -20 °C에서 저장할 수 있습니다. 그렇지 않으면 나머지 절차는 중단 없이 수행해야 합니다. - 각 4nM 초 PCR 제품의 Aliquot 3 μL을 풀라이브러리로 새로운 멸균 1.5mL 튜브에 모두 섞는다. 튜브를 4°C 또는 얼음 욕조에 항상 보관하십시오.
- 핵산 정량화 분광광계를 이용하여 확인을 위해 풀로 풀로 된 라이브러리의 농도를 측정한다. 4nM보다 높은 경우 농도를 4nM로 조정합니다.
참고: 과도하게 집중된 DNA는 과대 평가된 읽기 번호를 생성하여 분석을 방해합니다.
- DNA 변성 및 시퀀싱
참고: 이 섹션에서는 특정 시약이 있는 일루미나 미세크 시스템을 사용합니다. 재료 표에서제품 번호를 참조하십시오.
참고: 엄격하게 시간을 따르십시오. 시퀀싱 당일 카트리지를 제외한 모든 시약을 해동합니다.- 시퀀싱 하루 전에 -20°C에서 미리 채워진 즉시 사용 시약 카트리지를 제거하고 해동을 위해 4°C에 보관하십시오.
- 1.5mL 원심분리기 튜브에 적합한 열 블록을 96°C로 설정합니다.
- 혼성화 버퍼를 얼음 위에 놓습니다.
- PCR 등급의 물을 가진 새로운 튜브에서 분자 등급 NaOH를 1N에서 0.2 N으로 희석시하십시오.
- TE 버퍼(pH 8.0)로 사용 식 제어 라이브러리를 10nM 스톡에서 4nM로 희석시 새로운 튜브(예: 제어 라이브러리의 2μL + TE 버퍼3 μL)를 희석시보세요.
- 4nM 풀처리된 샘플 라이브러리의 16μL과 4nM 제어 라이브러리의 4μL을 "1"이라고 표시된 새로운 튜브에 혼합합니다.
- 튜브 "1"에 샘플 10 μL을 0.2 N NaOH의 10 μL과 혼합하여 "2"라고 표시된 새로운 튜브에 섞습니다.
- 튜브 2를 5초동안 소용돌이치게 하고, 잠시 회전하고, 실온에서 5분 동안 배양합니다.
- 튜브 2에 하이브리드화 버퍼 980 μL을 추가합니다.
- "3"이라고 표시된 새로운 튜브에서 260 μL의 샘플을 튜브 2에서 390 μL의 혼성화 버퍼와 혼합합니다. 튜브를 반전시켜 섞는다.
- 96°C에서 3번의 튜브를 2분 동안 즉시 얼음 위에 최대 2분 동안 배치합니다.
- 4°C 냉장고에서 카트리지를 제거합니다.
- 각 해당 인덱스 1및 인덱스 2 어댑터를 표 6과같이 시퀀싱하기 위한 샘플 시트를 설정합니다.
- MiSeq v3 키트에서 플로셀을 제거합니다. 멸균 분자 등급 PCR 물로 유동세포를 부드럽게 청소하십시오.
참고: 유량셀의 모세관 점에 물을 붓지 마십시오. 비 섬유 종이로 유량세포에서 물을 부드럽게 닦아냅니다. - 튜브 3(650 μL)의 전체 부피를 카트리지에 적재합니다.
- 계측기 작동 소프트웨어에서 시퀀싱을 선택하고 지침을 따릅니다. 흐름셀, 통합 버퍼 및 카트리지를 삽입합니다. 샘플 시트를 로드합니다. 실행을 눌러 반응을 시작합니다.
참고: 이 시퀀스 실행은 3-5일이 소요됩니다. 데이터는 Basespace 플랫폼에 자동으로 업로드됩니다. 원시 데이터는 컴퓨터의 두 폴더에 저장됩니다 : 패스트 크 파일을 포함하는 분석 폴더와 bcl 파일 및 jPEG 사진을 포함하는 출력.
4. 데이터 시퀀싱에 대한 분류학 할당
- FASTQ 파일 처리
참고: R24의 DADA2 패키지23은 FASTQ 파일을 처리하는 데 권장되는 데이터베이스 중 하나입니다. 지침은 [https://github.com/mickeykawai/exec_dada2]에서 사용할 수 있습니다. 이 지침은 DADA2 파이프라인 자습서 [https://benjjneb.github.io/dada2/tutorial.html]의 최근 버전을 채택하여 준비되었습니다.- 고유한 시퀀스 트리밍, 품질 필터링, 복제 및 계산, 샘플 추론, 연속으로 병합 및 키메라 시퀀스를 제거하기 위한 raw FASTQ 페어링 엔드 시퀀스를 처리합니다.
- 16S rRNA 및 18S rRNA 유전자 단편모두에 대해 DADA2 지정된 매개 변수를 다음과 같은 사용; trimLefts=0, 0, truncLens=[시퀀스 품질을 확인하고 적절한 길이로 설정].
참고: 다른 매개 변수의 기본 값은 maxN=0, maxEE=2, trancQ=2입니다.
- 분류 할당
참고: SILVA rRNA 데이터베이스는 DADA2 [silva_nr_v132_train_set.fa.gz 기본값으로 설정]에 대한 분류 할당에 권장됩니다.- 16S rRNA 유전자 단편 분석을 위한 엽록소 및 미토콘드리아 OTUs를 제거하십시오. 16S rRNA 및 18S rRNA 분석에서 싱글톤을 제거합니다.
- 모든 샘플을 가장 낮은 시퀀싱 깊이를 갖는 시료를 기반으로 시퀀싱 깊이로 리리피합니다.
- 읽기 수, 할당된 OtUs 및 수준, 필터링된 읽기 및 제외된 싱글톤 수를 기록합니다.
- 통계 분석
- phyloseq25 및 비건26의R 패키지를 사용하여 미생물 데이터에 대한 통계 분석을 수행합니다.
결과
이 프로토콜은 18S rRNA 유전자 대사 코딩 분석을 사용하여 해수 샘플에서 조류 종을 식별합니다. 대표적인 결과는 2019년 2월 19일 칠레 메트리(-41.597; -72.7056)에서 수집된 해수가 이 프로토콜로 분석된 그림1에 나와 있다. 그 결과 해수 샘플에서 30종 이상의 조류 종을 읽는 총 13,750마리의 판독이 나타났습니다. 이 샘플에서 지배적 인 조류는 나비 불라 SPP이었다. 상대적인 풍요로움은 70.77%입니다. 또한, 마이크로모나스(6.40%), 채토세로스(4.44%), 스크립시엘라(2.44%), 프로로센트럼 스프에대한 충분한 풍요로움이 관찰되었다. (1.28%). 칠레 HAB의 가장 높은 독성 조류 원인 중 하나인 슈도차트토넬라 spp.는이 해수 샘플에서 0.52%로 검출되었습니다.
데이터 신뢰성을 확인하기 위해, 18S rRNA 유전자 분석에 의해 확인된 조류 종은 동일한 해수 샘플에서 현미경 검사법에 의해 얻어진 것과 비교하였다(도2). 18S rRNA 유전자 분석과 일치하여, 현미경 검사는 지배적 인 종이 Navicula spp이었다는것을 보여 주었다. 상대적인 풍요로움은 74.1%, 프로로센트럼(0.60%)은 소종으로 나타났다. 반대로, 현미경 관찰으로부터 문서화된 헤테로캡사 spp.(9.04%)는 이 샘플에서 18S rRNA 유전자 분석에 의해 확인되지 않았다. 현미경 검사법에 의해 기록된 식별할 수 없는 식물성 플랑크톤 세포 의 주위에 작은의 12.6%가 있었습니다. 이것은 18S rRNA 결과에 따라 마이크로모나스가될 수 있었습니다.
이 프로토콜은 16S rRNA 유전자 대사 코딩 분석을 사용하여 해수 샘플에서 세균종을 식별합니다. 대표적인 결과는 도 3에서나타내며, 18S rRNA 유전자 분석에 사용된 동일한 바닷물이 16S rRNA 유전자 분석으로 분석되었다. 그 결과 해수 샘플에서 30종 이상의 세균종을 읽는 총 31,758마리가 나타났습니다. 이 해수 샘플은 부착 된 박테리아에서 자유 살아있는 박테리아를 분리하기 위해 탠덤 필터 멤브레인 (1 μm 및 0.2 μm 모공 크기)을 통과했다는 것을 설명해야합니다. 이어서, 각 필터 막에 의해 포획된 세포는 DNA 추출을 위해 처리되었고, 그 다음으로 16S rRNA 유전자 분석이 뒤따랐다. 도 3의 대표적인 결과는 0.2 μm 모공 크기 막에서 확인된 박테리아 종을 나타내며, 이는 자유 살아있는 박테리아로 정의된다. 지배적인 자유살아있는 박테리아는 20.02%의 상대적인 풍부를 가진 Amylibacter spp, Clade Ia (13.53%)와 알리지필루스 스프가그 뒤를 이었다. (7.06%). 이 해수 샘플에서 검출된 세균종의 나머지 는 비교적 균등하게 분포하였다. 부착박테리아 종 검출과 같이 1 μm 모공 크기의 멤브레인에 의해 포획된 세포에 대해 동일한 분석을 수행할 수 있다.
이러한 메타바코딩 분석이 특정 연구 기간 동안 예약된 시간 지점에서 수행되는 경우 결과를 타임 시리즈 분석으로 요약할 수 있습니다. 그것을 할 수있는 한 가지 방법은 독특한 성장 패턴을 찾기 위해 시간의 함수로 특정 조류와 세균 종의 상대적 풍부를 플롯하는 것입니다. 그림 4는 칠레 메트리에서 알렉산드리움과 슈도차트토넬라의 대표적인 타임시리즈 플롯을 보여줍니다. 타임 시리즈 메타바코딩 분석을 요약하는 또 다른 방법은 특정 유기체 그룹의 인구 변화를 나타내는 시간 함수로 확인된 모든 조류 및 세균을 플롯하는 것입니다. 도 5 및 도 6은 5개월 이상 메트리의 바닷물에서 검출되는 모든 세균 속과 질서의 상대적 풍요로움을 요약한다.
표 1: 첫 번째 PCR 마스터 믹스 콘텐츠: 표는 16S rRNA 및 18S rRNA 분석에 대한 반응당 마스터 믹스 콘텐츠를 보여줍니다. 프라이머 시퀀스는 표 3에 나열됩니다. 이 테이블을 다운로드하려면 여기를 클릭하십시오.
표 2: 프라이머 시퀀스: 첫 번째 PCR에 대한 프라이머는 16S rRNA 및 18S rRNA 분석을 위해 나열됩니다. 이 테이블을 다운로드하려면 여기를 클릭하십시오.
표 3: PCR 주기: 첫 번째 PCR 및 두 번째 PCR의 열 주기가 나열됩니다. 이 테이블을 다운로드하려면 여기를 클릭하십시오.
표 4: 인덱스 시퀀스: 두 번째 PCR에 사용할 인덱스 1(i7) 및 인덱스 2(i5) 프라이머가 나열됩니다. 이 테이블을 다운로드하려면 여기를 클릭하십시오.
표 5: 인덱스 1 및 인덱스 2 위치 지정의 예: 오류를 줄이기 위해 인덱스 프라이머를 먼저 위치에 배치해야 하며, 각 의 알리쿼트는 멀티채널 파이펫을 사용하여 96웰 플레이트로 전송됩니다. 이 테이블을 다운로드하려면 여기를 클릭하십시오.
표 6: 샘플 시트의 예: 시퀀싱 전에 인덱스 1 및 인덱스 2 어댑터에 해당하는 샘플 시트를 만들어야 합니다. 이 테이블을 다운로드하려면 여기를 클릭하십시오.
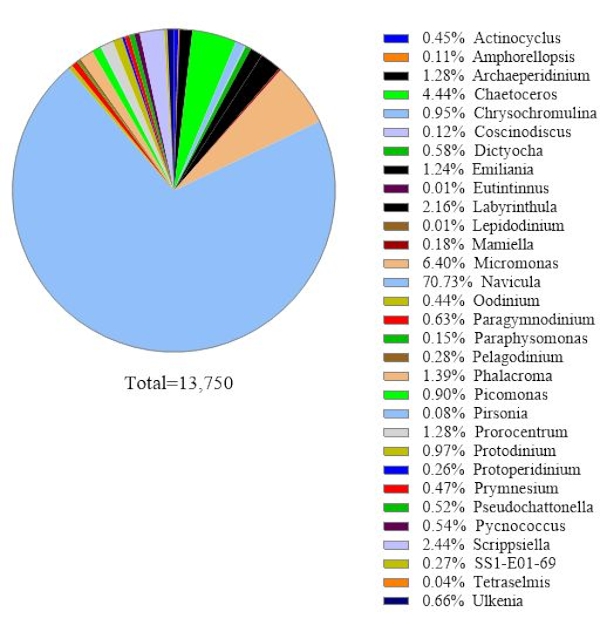
그림 1: 18S rRNA 메타바코딩 분석의 대표적인 결과: 2019년 2월 19일 칠레 주 트트리, 로스라고스에서 수집된 해수 샘플에 존재하는 조류 종은 18S rRNA 대사코딩 분석으로 확인되었다. "알 수 없는" 시퀀스가 제거되었고, 확인된 각 종의 상대적 풍요로움이 그려졌다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
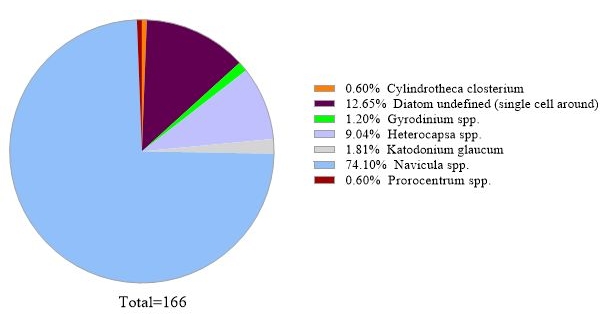
그림 2: 현미경 분석의 대표적인 결과: 조류 종은 2019년 2월 19일 칠레 주 미트리에서 현미경 검사법에 의해 수분 샘플에서 확인되었다. 각 종의 수량은 수동으로 계산되고 플롯되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
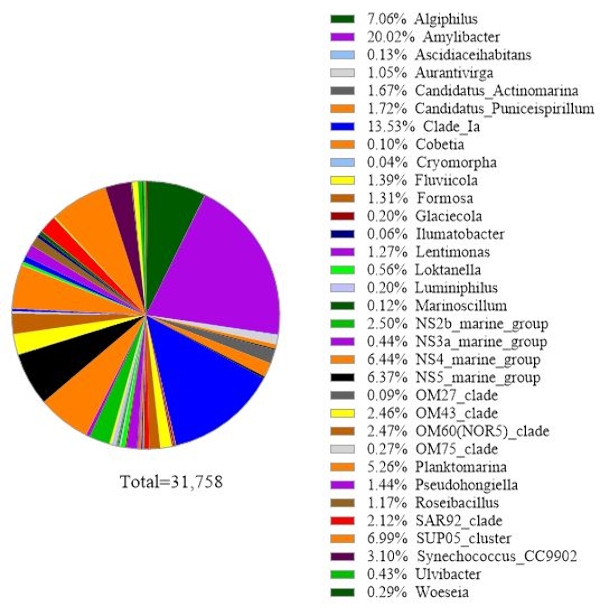
그림 3: 16S rRNA 메타바코딩 분석의 대표적인 결과: 2019년 2월 19일 칠레 주 트트리, 로스라고스에서 수질 샘플에 존재하는 세균종은 16S rRNA 대사코딩 분석으로 확인되었다. 확인된 각 종의 상대적 풍요로움이 그려졌습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
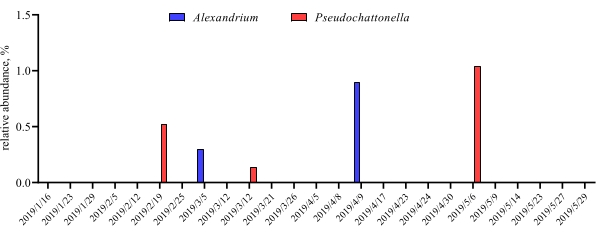
그림 4: 알렉산드리움 spp. 및 슈도차토넬라 spp의 대표 타임 시리즈 플롯. 18S rRNA 메타바코킹인 멧트리에서 얻은, 칠레: 그림 야리미즈 외9에서재인쇄. 두 개의 독성 조류 종, 알렉산드리움과 슈도차트토넬라는 시간이 지남에 따라 18S rRNA 분석에 의해 선택적으로 모니터링되었고, 상대적 풍부는 시간 점의 함수로 플롯되었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
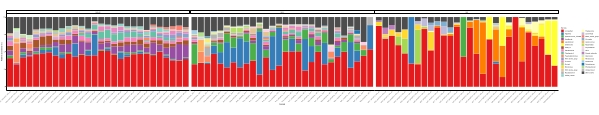
그림 5: 16S rRNA 및 18S rRNA 대사로부터 얻은 대표적인 속 시계열 플롯: 칠레 멧트리의 물에서 확인된 모든 세균 및 진핵 속은 5개월 이상 모니터링하였다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
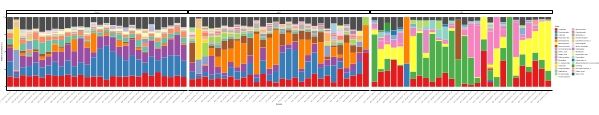
그림 6: 16S rRNA 및 18S rRNA 대사로부터 얻은 대표적인 주문 타임 시리즈 플롯: 칠레 멧트리의 물에서 확인된 모든 세균 및 진핵 주문은 5개월 이상 모니터링되었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
표 S1 및 테이블 S2:
표 S1: 그림 5 및 그림 6에대한 원시 데이터 . 표 S2: 그림 5 및 그림 6의원시 데이터에 대한 QC 검사 결과 . 이 테이블을 다운로드하려면 여기를 클릭하십시오.
토론
이 프로토콜은 칠레 HABs를 모니터링하기 위한 해수 샘플에서 조류 및 세균 종을 식별하기 위해 18S rRNA 및 16S rRNA 유전자 대사 코딩 분석을 성공적으로 수행했습니다. 이 프로토콜에 의해 검출된 지배적인 조류 및 몇몇 경미한 조류는 현미경 검사법에 의해 얻어진 것과 일치하여 프로토콜의 신뢰성을 확인했습니다. 프로토콜의 하이라이트는 분석 알렉산드리움 spp. 및 슈도차토넬라 spp.,남부 칠레에서 종을 일으키는 두 개의 가장 문제가 HAB, 심지어 낮은 풍부에 메트트리의 바닷물에서 검출. 특히, 슈도차트토넬라 spp. 세포가 가볍거나고정제(27,28)의사용 하에서 작고 쉽게 파열되기 때문에 현미경으로 식별하는 데 어려움을 겪고 있다. 메타바코딩은 현미경 검사가 식별할 수 없는 해수 샘플에 거의 존재하는 조류 종 정보를 제공할 수 있습니다. 프로토콜은 또한 30 개 이상의 세균종을 검출했습니다. 이 박테리아가 조류 성장과 관련이 있는 방법은 이 시점에서 알려지지 않았지만, 타임시리즈 분석에서 조류 균 종의 대규모 데이터 수집은 아마도 그러한 중요한 발견을 제공 할 것입니다. 따라서 기존의 칠레 HAB 모니터링 프로그램에 메타바코딩 분석을 추가하면 의심할 여지 없이 현재HAB 모니터링 효율성을 강화할 수 있습니다. 사실, 메타바코딩 분석을 위한 이 시각적 프로토콜은 칠레물의 조류 세균 모니터링뿐만 아니라 전 세계 다른 HAB 영향 지역의 해안 모니터링 프로그램에도 도움이 될 수 있습니다.
이 프로토콜은 위에서 언급한 이점을 제공했지만 메서드의 단점을 설명해야 합니다. 시각적 프로토콜에서 볼 수 있듯이 메타바코딩 방법은 시간이 많이 걸리고 복잡하므로 고가의 기계 및 시약이 필요합니다. 실험실 직원은 특별히 교육을 받아야하며 재료, 노동 및 시간을 낭비합니다. 또한, 대사코드에 의한 조류 검출은 두 개의 직교방법(29)으로부터동일한 지배적인 종들이 식별되는지 확인하기 위해 현미경 검사법과 결합되어야 한다. 현미경 검사는 조류 종을 식별하는 비 침습적 도구입니다, 조류 종은 독특하고 명백한 모양을 가질 때 실수를하기 어렵다는 것을 의미. 물론, 종들이 서로 매우 유사한 모양을 가지고 있다면 인간의 실수가 발생할 수 있습니다. 반면에 메타바코딩 해석에는 여러 단계가 필요하므로 자연스럽게 오류가 증가합니다. 샘플이 혼합되거나 잘못된 시약 추가 또는 일부 절차가 누락될 수 있습니다. 따라서, 현미경 검사법에 의해 얻어진 결과와 메타바코딩 결과를 비교하는 것이 중요합니다. 마지막으로 프로토콜은 질적으로만 적용되며 상대적 풍부를 위해 결과를 계산해야 합니다. 2019년에 언급한 바와 같이, 현재의 메타바코딩은 정량적 성능이 여전히 제한되어 있으며, 메타바코딩이 정량적응용(30)에자신있게 활용되기 전에 추가 연구가 필요하다.
이 프로토콜은 일루미나 Inc.가 발행한 16S 메타게노믹 시퀀싱 라이브러리 준비 매뉴얼에서 실행당 140-170 개의 샘플에 최적화되었습니다. 최적화된 프로토콜은 여러 번 테스트되었으며 이 최종 버전을 발급하도록 추가 수정되었습니다. 따라서 각 단계를 정확하게 따르는 것이 좋습니다. 프로토콜의 가장 중요한 부분은 샘플 오염을 피하기 위해 추가 주의가 필요하다는 것입니다. 파이펫과 라미나르 플로우 후드는 항상 70%의 알코올과 자외선 노출로 세척해야 하며, 멸균 가능한 물질은 자동 분해되어야 합니다. 프라이머와 시약은 새로운 모든 실행에서 주식에서 직접 희석되어야하며 시약과 여러 희석에 대한 노출을 재사용은 샘플 오염 원인이 될 수 있습니다. 프로토콜은 라미나르 흐름 후드 외부에서 작업을 수행할 수 있는 시기를 지정합니다. 그렇지 않으면 샘플을 세척된 라미나르 플로우 후드로 처리해야 합니다. 여러 개의 8튜브 스트립을 동시에 다룰 때, 다음 8튜브 스트립으로 이동하기 전에 첫 번째 8튜브 스트립에서 작업하고 캡을 씌는 것이 좋습니다. 뚜껑을 장시간 열어 두면 16S 및 18S rRNA 유전자가 주변에서 매우 유비쿼터스이기 때문에 샘플 오염을 일으킬 수 있습니다. 혼합 시간 및 인큐베이션 시간과 같은 명시된 시간은 여러 테스트 실행을 기반으로 각 단계에 대한 최상의 지속 시간을 선택했기 때문에 정확하게 설명된 대로 따라야 합니다. 예를 들어, 초과 또는 시간이 부족하면 샘플 수율을 줄일 수 있습니다. 이 프로세스는 프로토콜이 중지할 수 있다고 말하는 부분에서만 중지해야 합니다. 긍정적인 결과가 인위적인 거짓 긍정이 아니라는 것을 확인함에 따라 부정적인 제어를 갖는 것이 중요합니다. 마지막으로 모든 단계를 처리하는 데 최소 5일이 필요하고 다음 중단 기호가 있을 때까지 일부 부품을 중지할 수 없으므로 일을 계획하는 것이 좋습니다.
시퀀스 데이터에 대한 분류학적 할당의 제한은 여기에서 간략하게 지적됩니다. 세균성 및 조류 종에 대한 새로 발견 된 뉴클레오티드 서열은 매일 데이터 뱅크에서 업데이트됩니다. 잘 연구 된 조류와 세균성 종은 안정적으로 등록되어 있지만 데이터 뱅크에서 업데이트 된 알 수없는 종에 대한 많은 시퀀스도 있습니다. 등록된 뉴클레오티드 서열의 해상도가 종 식별을 위해항상 충분하지는 않지만 속 식별에만 충분하지 않다는 것을 나타냅니다. 따라서, 현미경 종 식별은 직교적으로 수행되어야 합니다. 이 작업에 대한 개방형 액세스 파이프라인을 설정하면 대규모 시퀀싱 된 데이터 집합을 한 번에 처리하고 오류가 발생할 때 효율적으로 조사하는 데 상당한 이점이 있습니다. 또한 DADA2의 출력은 텍스트 또는 csv 파일에 있으므로 추가 통계 분석이 용이합니다. 반면에 특히 데이터 집합이 큰 경우 분류 할당을 수행하기 위해 큰 서버가 필요합니다. 파이프라인을 설정하려면 파이프라인을 설정하려면 엔지니어가 필요하며 전문가는 매개 변수, 운영, Linux 및 생물 정보학을 이해해야 합니다.
HAB 모니터링 도구로서의 범위를 제외하고, 조사 연구를 위해 이 프로토콜의 사용을 확장할 수 있습니다. 예를 들어 칠레 정부와 지역 어부들 사이에는 지역 HAB31,32의증가에 관한 환경 평화 협회간의 논의가 계속되고 있습니다. 정부는 지구 온난화와 엘니뇨와 같은 자연 현상 때문이라고 주장하고 있으며, 이후 당사자들은 연어 양식이 원인이라고 주장합니다. 연어는 칠레의 토착 종이 아니며 반세기 전에 칠레 물에 있지 않았습니다. 당시 칠레 정부는 연어 사업33을개발하여 경제를 성장시키고 가난한 지역의 일자리를 창출하는 것을 목표로 삼았습니다. 자본이익을 위해 해외에서 개입한 칠레는 펜스타일의 양식업 개발에 큰 성공을 거두었으며, 연어의 수는 지난 수십 년 동안31,32,33로크게 증가했습니다. 그 후, 연어에 대한 많은 양의 음식이 바다에 던져져, 연어 양식이 자주 지역HABs31,32의이유라고 의심하는 지역 사람들을 선도하는. 진실은 지금 알려지지 않았지만, HAB로부터 지역 해양 환경, 경제 및 인간의 건강을 보호하기위한 전략이 고안 될 수 있도록 결국 이해되어야한다. 국소 조류 세균 관계에 대한 분자 기반 분석은 그러한 주제에 대한 단계 전진 해명에 기여할 수 있다. 예를 들어, 이 기술은 표적 해양 지역에 존재하지 않았지만 최근 몇 년 동안 극적으로 증가한 조류 및 세균종을 검색할 수 있으며, 이는 GIS 및 eDNA분석에의해 야마토 도롱뇽(Hynobiusvandenburghi)의기록되지 않은 집단의 발견을 위해 사카이 등의 연구와 다소 유사하다. 따라서 HAB 모니터링 도구를 넘어 이 메타바코딩 프로토콜은 다른 잠재 고객에게 잠재적인 사용이 있습니다.
공개
저자는 이해 상충을 선언하지 않습니다. 기금은 연구의 디자인, 수집, 분석 또는 데이터의 해석에 아무런 역할이 없었다; 원고를 작성하거나 결과를 게시하기로 결정합니다.
감사의 말
이 연구는 칠레의 지속 가능한 개발 모니터링 조류 (SATREPS-MACH)를위한 과학 기술 연구 파트너십에 대한 연구를위한 보조금 (JPMJSA1705)에 의해 지원되었습니다. 샌드라 리오스 박사(유니버시다드 데 로스 라고스)에게 영화 클립을 사용할 수 있게 해 준 것에 감사드립니다. 우리는 원고의 그의 부지런한 교정에 대한 닐 앤드류 네덜란드 감사합니다. 우리는 식물플랑크톤 식별에 대한 조언을 위해 칠레 푸에르토 몽트의 CREAN-IFOP의 실험실 그룹 구성원에게 진심으로 감사를 표합니다.
자료
| Name | Company | Catalog Number | Comments |
| 1.5 mL single tube | ThermoFisher | Q32856 | sterile |
| 1.5 mL tubes | ThermoFisher | 2150N | sterile |
| 8-strip 0.2 mL PCR tubes and flat cap | ThermoFisher | AB0451 & 4323032 | sterile |
| 96-well 0.2 mL PCR plate | ThermoFisher | N8010560 | |
| AccuRuler 100 bp DNA Ladder | MaestroGen | 02001-500 | |
| Chelex 100 Chelating Resin | Bio-Rad | 1432832 | |
| Chemical Duty Vacuum Pressure Pump | MilliporeSigma | WP6122050 | |
| D1000 Reagents | Agilent Technologies | 5067-5583 | |
| D1000 sample buffer | Agilent Technologies | 5067-5602 | |
| DNA loading dye (Blue/Orange Loading Dye 6X) | Promega | G1881 | |
| Ethanol Molecular Biology Grade | E7023 | Merck KGaA | |
| Filtration device | ThermoFisher | 09-740-36H, 09-740-23E | |
| Fluorescent DNA concentration measurement kit (Qubit dsDNA HS Assay kit) | Thermo Fisher Scientific | Q32851 | |
| Fluorometer (Qubit4 Fluorometer) | ThermoFisher | Q33238 | |
| Fragment analysis verification matrix (D1000 ScreenTape) | Agilent Technologies | 5067-5582 | |
| Fragment Analyzer (Agilent Tapestation 4150) | Agilent | G2992AA | |
| GelRed Nucleic Acid Gel Stain | Biotium | 41002 | |
| Generic bench vortexer (Vortex Genie 2) | Scientific Industries | SI-0236 | |
| Heat block | |||
| High performance sequencing-grade DNA polymerase (KAPA HiFi Hotstart ReadyMix), 2X | Roche | KK2602 | |
| HT1 Hybridization buffer | Illumina | 20015892 | |
| Illumina Nextera XT v2 Index Kit set A, B, C and D | Illumina | FC-131-2001, -2002, -2003, -2004 | |
| Laminar hood cabinet | Biobase Co. | Biobase PCR-800 PCR Cabinet | |
| Loading tips (Specific for Agilent TapeStation systems) | Agilent Technologies | 5067-5598 | |
| Magnetic beads DNA cleanup system (ProNex® Size-Selective Purification System) | Promega | NG2002 | |
| Magnetic stand for 96 wells (Magnetic Stand-96) | ThermoFisher | AM10027 | |
| Micro-seal film for 96-well plate | ThermoFisher | 4311971 | sterile |
| MiSeq Reagent Kit v3 | Illumina | MS-102-3003 | includes pre-filled, ready-to-use reagent cartridges. |
| MiSeq system | Illumina | SY-410-1003 | includes pre-installed software |
| Molecular grade nuclease-free water | Integrated DNA Technologies | 11-05-01-04 | |
| Molecular grade sodium hydroxide (NaOH) 1M | Merck KGaA | 1091371000 | |
| Multichannel pipettes | Not specified | Not specified | |
| Multi-Purpose Agarose | Cleaver Scientific | CSL-AG500 | |
| Ow D2 Wide-Gel Electrophoresis System | ThermoFisher | D2 | |
| Ow EC-105 Compact Power Supply | ThermoFisher | 105ECA-115 | |
| Pellet Pestle— Cordless Motor | ThermoFisher | K749540-0000 | |
| PhiX control Kit v3 | Illumina | FC-110-300 | ready-to-use control library for Illumina sequencing |
| Pipette tips | Not specified | Not specified | sterile |
| Pipettes | Not specified | Not specified | 2, 10, 20, 100, 1000 μL |
| SterivexTM GP 0.22 μm filter unit | MilliporeSigma | SVGP01050 | |
| Tabletop centrifuge | Eppendorf 5427R Centrifuge | Eppendorf AG | |
| TBE buffer | ThermoFisher | B52 | |
| TE buffer (pH 8.0) | ThermoFisher | AM9849 | |
| Terr PCR Direct FFPE Kit | Takara Bio USA | 639284 | includes 2X Terra PCR Direct Buffer |
| Terra PCR Direct Polymerase Mix, 1.25 U/μL | Takara Bio USA | 639271 | High performance Inhibitor-tolerant DNA polymerase |
| ThermalCycler (MiniAMP Plus Thermal Cycler) | ThermoFisher | A37835 | |
| TransIlluminator and image capture system | Analytik Jena, AG | GelDoc-ItTS2 Imager | |
| Whatman 1.0 m pore-sized membrane | MilliporeSigma | WHA111110 |
참고문헌
- Hallegraeff, G., Enevoldsen, H., Zingone, A. Global harmful algal bloom status reporting. Harmful Algae. 102, 101992 (2021).
- Anderson, D. M. Red Tides. Scientific American. 271 (2), 52-58 (1994).
- Sanseverino, I., Conduto, A. D. S., Pozzoli, L., Dobricic, S., Lettieri, T. . Algal Bloom and its Economic Impact. , (2016).
- Molinet, C., Niklitschek, E., Seguel, M., Díaz, P. Trends of natural accumulation and detoxification of paralytic shellfish poison in two bivalves from the Norwest Patagonian inland sea. Revista de Biologia Marina Y Oceanografia. 45, 195-204 (2010).
- Mardones, J., Clement, A., Rojas, X., Aparicio, C. Alexandrium catenella during 2009 in Chilean waters, and recent expansion to coastal ocean. Harmful Algae News. 41, 8-9 (2010).
- Alvarez, G., et al. Paralytic shellfish toxins in surf clams Mesodesma donacium during a large bloom of Alexandrium catenella dinoflagellates associated to an intense shellfish mass mortality. Toxins (Basel). 11 (4), (2019).
- Clément, A., et al. Exceptional summer conditions and HABs of Pseudochattonella in Southern Chile create record impacts on salmon farms. Harmful Algae News. 53, 1-3 (2016).
- Trainer, V. L., et al. Pelagic harmful algal blooms and climate change: Lessons from nature's experiments with extremes. Harmful Algae. 91, 101591 (2020).
- Yarimizu, K., et al. Protocols for monitoring harmful algal blooms for sustainable aquaculture and coastal fisheries in Chile. International Journal of Environmental Research and Public Health. 17 (20), (2020).
- Roegner, G. C., Hickey, B. M., Newton, J. A., Shanks, A. L., Armstrong, D. A. Wind-induced plume and bloom intrusions into Willapa Bay, Washington. Limnology & Oceanography. 47 (4), 1033-1042 (2002).
- Tweddle, J. F., et al. Relationships among upwelling, phytoplankton blooms, and phycotoxins in coastal Oregon shellfish. Marine Ecology Progress Series. 405, 131-145 (2010).
- Glibert, P. M., Anderson, D. M., Gentien, P., Graneli, E., Sellner, K. G. The global complex phenomena of harmful algae blooms. Oceanography. 18 (2), 130-141 (2005).
- Paredes-Mella, J., Varela, D., Fernández, P., Espinoza-González, O. Growth performance of Alexandrium catenella from the Chilean fjords under different environmental drivers: Plasticity as a response to a highly variable environment. Journal of Plankton Research. 42 (2), 119-134 (2020).
- Azam, F., Malfatti, F. Microbial structuring of marine ecosystems. Nature Reviews Microbiology. 5, 782-791 (2007).
- Amin, S. A., et al. Interaction and signaling between a cosmopolitan phytoplankton and associated bacteria. Nature. 522, 98-101 (2015).
- Berdalet, E., et al. Marine harmful algal blooms, human health, and wellbeing: Challenges and opportunities in the 21st century. Journal of Marine Biology Association. 2015, 1-31 (2015).
- Ramanan, R., Kim, B. -. H., Cho, D. -. H., Oh, H. -. M., Kim, H. -. S. Algae-bacteria interactions: Evolution, ecology, and emerging applications. Biotechnology Advances. 34 (1), 14-29 (2016).
- Seymour, J. R., Amin, S. A., Raina, J. B., Stocker, R. Zooming in on the phycosphere: The ecological interface for phytoplankton-bacteria relationships. Nature Reviews Microbiology. 2, 17065 (2017).
- Maruyama, F., et al. 16S and 18S Metabarcoding analysis for Chilean coastal waters harmful algal blooms. protocol.io. , (2020).
- Tanabe, A. S., et al. Comparative study of the validity of three regions of the 18S-rRNA gene for massively parallel sequencing-based monitoring of the planktonic eukaryote community. Molecular Ecology Resources. 16, 402-414 (2016).
- Nishitani, G., et al. Multiple plastids collected by the Dinoflagellate Dinophysis mitra through Kleptoplastidy. Applied and Environmental Microbiology. 78 (3), 813-821 (2012).
- Klindworth, A., et al. Evaluation of general 16S ribosomal RNA gene PCR primers for classical and next-generation sequencing-based diversity studies. Nucleic Acids Research. 41 (1), 1 (2013).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nature Methods. 13 (7), 581-583 (2016).
- McMurdie, P. J., Holmes, S. phyloseq: An R Package for Reproducible Interactive Analysis and Graphics of Microbiome Census Data. PLoS One. 8 (4), 61217 (2013).
- Dixon, P. VEGAN, a package of R functions for community ecology. Journal of Vegetation Science. 14 (6), 927-930 (2003).
- Dittami, S. M., Riisberg, I., Edvardsen, B. Molecular probes for the detection and identification of ichthyotoxic marine microalgae of the genus Pseudochattonella (Dictyochophyceae, Ochrophyta). Environmental Science and Pollution Research. 20 (10), 6824-6837 (2013).
- Mardones, J. I., et al. Salinity-growth response and ichthyotoxic potency of the Chilean Pseudochattonella verruculosa. Frontiers in Marine Science. 6 (24), (2019).
- Santi, I., Kasapidis, P., Karakassis, I., Pitta, P. A comparison of DNA metabarcoding and microscopy methodologies for the study of aquatic microbial eukaryotes. Diversity. 13 (5), 180 (2021).
- Lamb, P. D., et al. How quantitative is metabarcoding: A meta-analytical approach. Molecular Ecology. 28 (2), 420-430 (2019).
- Mascareño, A., et al. Controversies in social-ecological systems: lessons from a major red tide crisis on Chiloe Island, Chile. Ecology and Society. 23, (2018).
- Armijo, J., Oerder, V., Auger, P. -. A., Bravo, A., Molina, E. The 2016 red tide crisis in southern Chile: Possible influence of the mass oceanic dumping of dead salmons. Marine Pollution Bulletin. 150, 110603 (2020).
- Swanson, H. A. . Caught in Comparisons: Japanese Salmon in an Uneven World. , (2013).
- Sakai, Y., et al. Discovery of an unrecorded population of Yamato salamander (Hynobius vandenburghi) by GIS and eDNA analysis. Environmental DNA. 1 (3), 281-289 (2019).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유