Method Article
Una procedura standardizzata per il monitoraggio delle fioriture algali dannose in Cile mediante analisi metabarcodifica
In questo articolo
Riepilogo
Questo protocollo introduce fasi di analisi metabarcodifica, mirate ai geni rRNA 16S e rRNA 18S, per monitorare le fioriture algali dannose e il microbioma associato nei campioni di acqua di mare. È un potente strumento basato su molecole, ma richiede diverse procedure, che sono spiegate visivamente qui passo dopo passo.
Abstract
Il monitoraggio delle fioriture di alghe nocive (HAB) è stato implementato in tutto il mondo e il Cile, un paese famoso per la pesca e l'acquacoltura, ha utilizzato intensamente analisi microscopiche e tossiche per decenni per questo scopo. I metodi biologici molecolari, come il sequenziamento del DNA ad alto rendimento e gli approcci basati sull'assemblaggio batterico, stanno appena iniziando a essere introdotti nel monitoraggio HAB cileno e le procedure non sono ancora state standardizzate. Qui, le analisi di metabarcoding dell'rRNA 16S e dell'rRNA 18S per il monitoraggio degli HAB cileni vengono introdotte gradualmente. Secondo una recente ipotesi, l'associazione mutualistica algale-batterica svolge una relazione sinergica o antagonistica critica che rappresenta l'inizio, il mantenimento e la regressione della fioritura. Pertanto, il monitoraggio dell'HAB da prospettive algali-batteriche può fornire una comprensione più ampia dei meccanismi HAB e la base per l'allerta precoce. L'analisi metabarcoding è uno degli strumenti molecolari più adatti a questo scopo perché può rilevare massicce informazioni tassonomiche algali-batteriche in un campione. Le procedure visive di campionamento per l'analisi metabarcoding qui forniscono istruzioni specifiche, con l'obiettivo di ridurre gli errori e la raccolta di dati affidabili.
Introduzione
Molte specie di fitoplancton marino sono note per produrre tossine endogene e quando queste specie si accumulano in numero sufficiente, sono dannose per l'ambiente marino. Tali fioriture algali dannose (HAB) sono osservate oggi sulle coste della maggior parte dei continenti del mondo1. Il fitoplancton tossico si accumula per la prima volta nei tessuti bivalvi, portando a malattie e morte in livelli trofici più elevati di organismi, compresi gli esseri umani, dopo la digestione. Successivamente, questi eventi portano gravi implicazioni per l'economia locale, la socioeconomia e la salute pubblica2. Il danno all'economia globale causato dagli HAB è stimato in milioni o miliardi di dollari ogni anno3. Il Cile è uno dei tanti paesi che soffrono di frequenti HAB.
Il Cile è un paese di lunga terra, che si estende per oltre 4.300 km a nord e a sud. La terra occidentale allungata si affaccia sull'Oceano Pacifico, il che aumenta naturalmente le possibilità che il Cile sperimenti HAB. Soprattutto nel sud del Cile, ci sono molte acquacolture di salmone di fama mondiale, e ogni volta che si verifica un HAB nella regione, le tossine algali provocano la malattia di un massiccio salmone d'allevamento e muore4,5,6. In Cile, l'anno in cui l'HAB ha colpito più duramente l'economia è stato il 2016, con una perdita annua stimata di 800 milioni di dollariUSA 7,8. Le specie algali tossiche causative variano in base all'anno e alla posizione. Per il caso del 2016, un complesso di Alexandrium catanella e Pseudochattonella verruclosa ha causato un HAB diffuso in gran parte della costa cilena meridionale7,8. L'HAB più recente in Cile si è verificato con la specie algale causativa di Heterosigma Akashiwo nel marzo 2021 a Camanchaca, situata nel sud del Cile, dove l'area ha un grande allevamento di salmoni.
Il Cile conduce da molti anni il monitoraggio costiero utilizzando due metodi principali; osservare l'acqua di mare con microscopi per identificare regolarmente le specie di alghe tossiche e misurare i livelli di tossine nei molluschi mediante saggi biochimici9. La diagnosi precoce delle alghe tossiche e dei livelli di tossine nei molluschi non previene gli HAB; tuttavia, queste analisi possono innescare contromisure immediate e ridurre i danni alle comunità locali. Per rafforzare ulteriormente l'efficacia di questa strategia, un metodo analitico a base molecolare è stato recentemente aggiunto al nostro programma di monitoraggio HAB cileno convenzionale per rilevare alghe e comunità batteriche correlate nei campioni di acqua di mare. In particolare, è stato scelto un metodo di sequenziamento parallelo massiccio che utilizza il metabarcoding che si rivolge ai geni rRNA 16S e rRNA 18S. Sebbene questa tecnica richieda procedure complicate e costosi macchinari e reagenti, è una tecnologia avanzata in grado di rilevare migliaia di alghe e batteri / specie presenti in un campione di acqua di mare contemporaneamente.
Si ipotizza che le cause dell'HAB siano varie, come la temperatura e la stagione, ma è impossibile generalizzarle. Questo perché le specie e le frequenze HAB dipendono dalla regione e dalle condizioni spazio-temporali, coinvolgendo fenomeni naturali come l'unicità geografica, la miscelazione dei nutrienti in risalita e il deflusso degli elementi dalla terra a causa dell'errosione2,10,11,12. Inoltre, fattori artificiali come l'eutrofizzazione influenzano gli HAB locali12,13. A causa del complesso multifattore, non è facile fare una previsione HAB accurata. Negli ultimi anni, c'è un'opinione secondo cui specifiche popolazioni batteriche possono essere correlate allo sviluppo di HAB come uno dei fattori, e la ricerca a sostegno di questa ipotesi è stata sempre più evidente14,15,16,17,18. Le tecniche di biologia molecolare sono generalmente utilizzate per studiare l'assemblaggio batterico; tuttavia, tale metodo standardizzato non è stato ancora stabilito nel monitoraggio HAB cileno9. Per studiare l'associazione algale-batterica con gli HAB, è imperativo eseguire contemporaneamente analisi di metabarcoding per gli attuali programmi di monitoraggio costiero cileno. Pertanto, questo protocollo introduce visivamente il nostro programma di monitoraggio HAB cileno, concentrandosi su una procedura graduale per analizzare il rilevamento di alghe e specie di batteri nei campioni di acqua di mare utilizzando l'analisi metabarcoding.
Il protocollo completo che descrive il nostro programma di monitoraggio HAB cileno è disponibile in Yarimizu et al.9. Include le procedure di campionamento dell'acqua di mare, rilevamento di specie algali microscopiche, rilevamento di geni algali-batterici, analisi dei pigmenti, raccolta di dati meteorologici e saggi di proprietà fisiche e chimiche dell'acqua. Il protocollo graduale di analisi metabarcoding di rRNA 16S e rRNA 18S per il rilevamento di specie algali e batteriche è disponibile come prestampa19. Questo protocollo dimostra in particolare le fasi di analisi del metabarcoding poiché è la parte più complicata e il punto culminante del nostro programma di monitoraggio HAB. Questo protocollo include anche un'introduzione del programma e il rilevamento al microscopio di specie algali. Quando si analizzano le specie algali mediante metabarcoding, è fondamentale eseguire contemporaneamente la microscopia per verificare i risultati dei due metodi. Questo protocollo non include come utilizzare il software per l'assegnazione della tassonomia, ma la raccomandazione del database è brevemente indicata alla fine della sezione seguente.
Protocollo
1. Raccolta e pretrattamento dei campioni
- Raccogliere circa 3 L di campione d'acqua dal punto di destinazione.
- Filtrare 1 L di campione d'acqua per l'analisi dell'rRNA 16S attraverso una filtrazione tandem (membrana da 1 μm e 0,2 μm) per separare i batteri liberi e attaccati.
- Filtrare un altro campione di 1 L di acqua per l'analisi 18S-rRNA (rilevamento del fitoplancton) attraverso una singola filtrazione con una membrana da 0,2 μm.
NOTA: La filtrazione del campione d'acqua deve essere completata entro 12 ore dal campionamento. - Tagliare la membrana filtrata a metà con forbici chirurgiche sterili e avvolgerla con un foglio di alluminio. Conservare a -20 °C per un massimo di 1 mese o procedere al passaggio successivo.
- Estrarre il DNA con il metodo Chelex come descritto9. Conservare a -20 °C per un massimo di 1 mese.
2. Analisi al microscopio
- Trasferire 1 mL del campione d'acqua con una pipetta su una griglia-scivolo da 1 mL.
- Osservare il campione al microscopio.
- Registrare i nomi e la quantità di specie di fitoplancton.
3. Analisi metabarcoding di rRNA 16S e rRNA 18S
NOTA: Questo processo è composto da sette sezioni: preparazione, prima generazione di amplicon PCR, prima pulizia amplicon, indicizzazione mediante seconda PCR, seconda verifica e pulizia dell'amplicon PCR, regolazione della concentrazione del DNA e denaturazione e sequenziamento del DNA. L'intero processo richiede un minimo di 5 giorni (40 ore) da parte di personale di laboratorio esperto. Vedere la Tabella dei materiali per i numeri di prodotto e i produttori.
- Preparazione
- Pulire pipette e cappa laminare con etanolo al 70% seguito frequentemente dall'esposizione ai raggi UV per 30 minuti. Sterilizzare i materiali da utilizzare.
- Scongelare i campioni di DNA a temperatura ambiente, centrifugare a 100 x g per 2 minuti e trasferire 100 μL di ciascun surnatante campione a strisce a 8 tubi.
- Prima generazione di amplicon PCR
NOTA: eseguire le seguenti procedure in un armadio con cappuccio laminare. Diluire sempre i primer da 1 μM alla concentrazione target con acqua di grado PCR per evitare la contaminazione del primer.- Preparare la prima miscela master PCR in un tubo sterile da 1,5 ml per le reazioni (Tabella 1, Tabella 2).
- Aliquota 22,5 μL della miscela master in una striscia a 8 tubi e aggiungere 2,5 μL di campione di DNA. Utilizzare 2,5 μL di acqua di grado PCR per il controllo negativo.
- Eseguire il primo ciclo di PCR (Tabella 3).
- Preparare 100 mL di gel di agarosio-TBE al 2% contenente 10 μL di 1x colorazione di gel di acido nucleico.
- Caricare una miscela di 1,5 μL di 1x colorante di carico del DNA e 4 μL di prodotto PCR sul gel di agarosio. Inoltre, caricare la scala del DNA da 100 bp sul gel.
- Eseguire l'elettroforesi a 100 V per 30 min.
- Assicurarsi che vi sia una banda a 500-600 bp sotto un'acquisizione di immagini a luce UV. La banda primer-dimero è rotonda di 80 bp.
NOTA: I campioni di acqua marina contengono inibitori della PCR. Gli ampliconi mancanti a volte possono essere risolti diluendo i campioni 1:100 o 1:1000 con acqua di grado PCR. - Conservare i primi prodotti PCR a -20 °C fino al passaggio successivo.
ATTENZIONE: non superare una settimana di conservazione.
- Prima pulizia dell'amplicon PCR
NOTA: questa sezione può essere eseguita all'esterno di un armadio con cappuccio laminare.- Utilizzare il sistema di pulizia del DNA delle perline magnetiche per rimuovere i residui di reazione PCR, compresi i prodotti primer-dimero.
- Trasferire 20 μL di ogni primo prodotto PCR pulito su una nuova piastra da 96 pozzetti. Sigillare la piastra con un film di microsigillatura. Conservare a -20 °C fino al passaggio successivo.
ATTENZIONE: non superare più di una settimana di conservazione.
- Indicizzazione per seconda PCR
NOTA: In questa sezione, i primi prodotti PCR purificati saranno amplificati con varie combinazioni di primer indice.- Diluire tutti i primer Index 1 e Index 2 (Tabella 4) a 1 μM con acqua di grado PCR in strisce PCR a 8 tubi collocate in un armadio con cappa laminare.
NOTA: i passaggi successivi in questa sezione possono essere eseguiti al di fuori di un armadio con cappuccio laminare, poiché i primer indice sono specifici per il primo adattatore di sporgenza delle reazioni PCR. - Indice di posizione 1 primer in una riga orizzontale Indice 2 primer in una riga verticale (Tabella 5).
- In una nuova piastra da 96 pozzetti, aggiungere 12,5 μL di formulazione pronta per l'avvio a caldo a ciascun pozzetto.
- Aggiungere 2,5 μL di ciascun primer indice (1 μM) a ciascun pozzetto come nella Tabella 4 utilizzando una pipetta multicanale.
- Aggiungere 7,5 μL di primo prodotto PCR purificato.
- Mescolare delicatamente pipettando su e giù 10 volte. Coprire la piastra con un film di microsigilli.
- Eseguire il secondo ciclo di PCR (Tabella 3).
- Tenere la piastra a -20 °C.
ATTENZIONE: non superare una settimana di conservazione.
- Diluire tutti i primer Index 1 e Index 2 (Tabella 4) a 1 μM con acqua di grado PCR in strisce PCR a 8 tubi collocate in un armadio con cappa laminare.
- Seconda verifica e pulizia dell'amplicon PCR
- Utilizzare un analizzatore di frammenti e il reagente associato. Vortice e spin down reagente prima dell'uso.
- Lasciare che il tampone campione e il nastro dello schermo del DNA si equilibrino a temperatura ambiente per 30 minuti. Quindi, posizionare il nastro dello schermo del DNA in un analizzatore di frammenti.
- Miscelare 2 μL di tampone campione e 3 μL di secondo amplicon PCR in nuove strisce a 8 tubi. Inserire le 8 strisce di tubo nell'analizzatore di frammenti. Premere Esegui per iniziare.
ATTENZIONE: La formazione di bolle d'aria deve essere evitata. - Assicurarsi che i secondi ampliconi PCR siano circa 613 bp (600 - 630 bp) per entrambi i geni rRNA 16S e 18S.
- Purificare i secondi prodotti PCR utilizzando un sistema di pulizia del DNA con perline magnetiche.
- Regolazione della concentrazione del DNA
- Misurare la concentrazione di DNA nei secondi prodotti PCR purificati utilizzando uno spettrofotometro di quantificazione dell'acido nucleico.
- Calcola la concentrazione del gene bersaglio in nM, tenendo conto della dimensione media della libreria finale come 613 bp per entrambi i geni rRNA 16S e 18S:
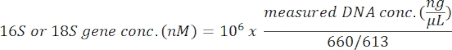
- Diluire ogni secondo prodotto PCR purificato con acqua PCR sterile a 4 nM in una nuova piastra da 0,2 mL 96.
NOTA: La piastra può essere conservata a -20 °C qui. In caso contrario, il resto delle procedure deve essere eseguito senza fermarsi. - Aliquota 3 μL di ogni prodotto PCR da 4 nM di secondo e miscelare il tutto in un nuovo tubo sterile da 1,5 ml come libreria in pool. Tenere il tubo a 4 °C o in bagno di ghiaccio in ogni momento.
- Misurare la concentrazione della libreria aggregata per la conferma utilizzando uno spettrofotometro di quantificazione dell'acido nucleico. Regolare la concentrazione a 4 nM se è superiore a 4 nM.
NOTA: il DNA iperconcentrato produce numeri di lettura sovrastimati, ostacolando l'analisi.
- Denaturazione e sequenziamento del DNA
NOTA: Questa sezione utilizza il sistema Illumina MiSeq con reagenti specifici. Vedere i numeri di prodotto in Tabella dei materiali.
NOTA: seguire rigorosamente l'ora. Scongelare tutti i reagenti tranne le cartucce il giorno del sequenziamento.- Un giorno prima del sequenziamento, rimuovere una cartuccia di reagente preriempita pronta all'uso da -20 °C e conservare a 4 °C per lo scongelamento.
- Impostare un blocco termico adatto per tubi centrifuga da 1,5 mL a 96 °C.
- Posizionare il buffer di ibridazione sul ghiaccio.
- Diluire il grado molecolare NaOH da 1 N a 0,2 N in un nuovo tubo con acqua di grado PCR.
- Diluire una libreria di controllo pronta all'uso con tampone TE (pH 8,0) da 10 nM stock a 4 nM in un nuovo tubo (cioè 2 μL di libreria di controllo + 3 μL di buffer TE).
- Mescolare 16 μL di una libreria di campioni in pool da 4 nM con una libreria di controllo da 4 μL di 4 nM in un nuovo tubo etichettato "1".
- Miscelare 10 μL di campione in provetta "1" con 10 μL di 0,2 N NaOH in un nuovo tubo etichettato "2".
- Ruotare il tubo 2 per 5 s, girare brevemente verso il basso e incubare a temperatura ambiente per 5 minuti.
- Aggiungere 980 μL di tampone di ibridazione al tubo 2.
- In un nuovo tubo etichettato "3", mescolare 260 μL il campione del tubo 2 con 390 μL di tampone di ibridazione. Mescolare invertendo il tubo.
- Incubare il tubo 3 a 96 °C per 2 minuti e metterlo immediatamente sul ghiaccio per un massimo di 2 minuti.
- Rimuovere la cartuccia dal frigorifero a 4 °C.
- Impostare il foglio di esempio per la sequenziazione con ciascun adattatore di indice 1 e indice 2 corrispondente come nella tabella 6.
- Rimuovere la cella di flusso dal kit MiSeq v3. Pulire delicatamente la cella di flusso con acqua PCR sterile di grado molecolare.
NOTA: non versare acqua sui punti capillari della cella di flusso. Pulire delicatamente l'acqua dalla cella di flusso con carta non fibrosa. - Caricare l'intero volume del tubo 3 (650 μL) nella cartuccia.
- Nel software di funzionamento dello strumento, selezionare la sequenza e seguire le istruzioni. Inserire la cella di flusso, il buffer di incorporazione e la cartuccia. Caricare il foglio di esempio. Premere Esegui per avviare la reazione.
NOTA: l'esecuzione della sequenza richiederà 3-5 giorni. I dati verranno caricati automaticamente sulla piattaforma Basespace. I dati grezzi verranno memorizzati in due cartelle nel computer: cartella di analisi contenente file fastq e output contenente file bcl e immagini jpeg.
4.Assegnazione tassonomica ai dati di sequenziamento
- Elaborazione di file FASTQ
NOTA: il pacchetto DADA223 di R24 è uno dei database consigliati per elaborare i file FASTQ. La linea guida è disponibile all'indirizzo [https://github.com/mickeykawai/exec_dada2]. Questa linea guida è stata preparata adottando la recente versione del tutorial sulla pipeline DADA2 [https://benjjneb.github.io/dada2/tutorial.html].- Elabora sequenze accoppiate FASTQ grezze per il taglio, il filtraggio di qualità, la dereplicazione e il conteggio di sequenze uniche, l'inferenza del campione, la fusione in contig e la rimozione di sequenze chimeriche.
- Utilizzare i seguenti parametri specificati DADA2 per i frammenti di geni rRNA 16S e rRNA 18S; trimLefts=0, 0, truncLens=[Controllare la qualità della sequenza e impostarla su una lunghezza appropriata].
NOTA: i valori predefiniti per gli altri parametri sono maxN=0, maxEE=2,2, trancQ=2.
- Assegnazione tassonomia
NOTA: il database silva rRNA è consigliato per l'assegnazione della tassonomia per DADA2 [silva_nr_v132_train_set.fa.gz è impostato come predefinito].- Rimuovere il cloroplasto e le OTU mitocondriali per l'analisi del frammento del gene rRNA 16S. Rimuovere i singleton dall'analisi dell'rRNA 16S e dell'rRNA 18S.
- Rarificare tutti i campioni fino alla profondità di sequenziamento uniforme in base al campione con la profondità di sequenziamento più bassa.
- Registrare il numero di letture, le OTU assegnate e a quale livello, le letture filtrate e il numero di singleton esclusi.
- Analisi statistica
- Eseguire analisi statistiche su dati microbici utilizzando pacchetti R di phyloseq25 e vegan26.
Risultati
Questo protocollo utilizza l'analisi metabarcoding del gene rRNA 18S per identificare le specie algali nei campioni di acqua di mare. Un risultato rappresentativo è mostrato nella Figura 1, in cui l'acqua di mare raccolta da Metri, Puerto Montt, Cile (-41.597; -72.7056) il 19 febbraio 2019, è stata analizzata con questo protocollo. Il risultato ha mostrato un totale di 13.750 letture con oltre 30 specie algali nel campione di acqua di mare. L'alga dominante in questo campione era Navicula spp. con un'abbondanza relativa del 70,77%. Inoltre, è stata osservata un'abbondanza sufficiente per Micromonas (6,40%), Chaetoceros spp. (4,44%), Scripsiella spp. (2,44%) e Prorocentrum spp. (1.28%). Pseudochattonella spp., uno dei più alti causativi algali tossici di HAB cileno, è stato rilevato con lo 0,52% da questo campione di acqua di mare.
Per verificare l'affidabilità dei dati, le specie algali identificate dall'analisi del gene rRNA 18S sono state confrontate con quelle ottenute al microscopio nello stesso campione di acqua di mare (Figura 2). Coerentemente con l'analisi del gene rRNA 18S, la microscopia ha mostrato che la specie dominante era Navicula spp. con un'abbondanza relativa del 74,1% e Prorocentrum spp. (0,60%) come specie minore. Al contrario, Heterocapsa spp. (9,04%) documentato dall'osservazione microscopica non è stato identificato dall'analisi del gene rRNA 18S in questo campione. C'era il 12,6% di piccole cellule di fitoplancton non identificabili registrate al microscopio. Questo potrebbe essere Micromonas, secondo i risultati dell'rRNA 18S.
Il protocollo utilizza l'analisi di metabarcoding del gene rRNA 16S per identificare le specie batteriche nei campioni di acqua di mare. Un risultato rappresentativo è mostrato nella Figura 3, in cui la stessa acqua di mare utilizzata per l'analisi del gene rRNA 18S è stata analizzata con l'analisi del gene rRNA 16S. Il risultato ha mostrato un totale di 31.758 letture con oltre 30 specie batteriche nel campione di acqua di mare. Va sottolineato che questo campione di acqua di mare è stato fatto passare attraverso membrane filtranti tandem (dimensioni dei pori di 1 μm e 0,2 μm) per separare i batteri viventi liberi dai batteri attaccati. Quindi, le cellule catturate da ciascuna membrana filtrante sono state trattate per l'estrazione del DNA, seguita dall'analisi del gene rRNA 16S. Il risultato rappresentativo nella Figura 3 mostra specie di batteri identificati da una membrana di 0,2 μm di dimensioni dei pori, che sono definiti come batteri viventi liberi. I batteri viventi liberi dominanti erano Amylibacter spp. con un'abbondanza relativa del 20,02%, seguiti da Clade Ia (13,53%) e Aligiphilus spp. (7.06%). Il resto delle specie batteriche rilevate da questo campione di acqua di mare erano relativamente equamente distribuite. La stessa analisi può essere eseguita per le cellule catturate da una membrana delle dimensioni di un poro di 1 μm come rilevamento di specie di batteri attaccati.
Quando questi test di metabarcoding vengono eseguiti in punti temporali programmati per un determinato periodo di studio, i risultati possono essere riassunti come analisi di serie temporali. Un modo per farlo è tracciare l'abbondanza relativa di particolari specie algali e batteriche in funzione del tempo per trovare un modello di crescita unico. La Figura 4 mostra un grafico rappresentativo delle serie temporali di Alexandrium e Pseudochattonella a Metri, in Cile. Un altro modo per riassumere un'analisi di metabarcoding di serie temporali è quello di tracciare tutte le alghe e i batteri identificati in funzione del tempo, rappresentando il cambiamento di popolazione di determinati gruppi di organismi. La Figura 5 e la Figura 6 riassumono l'abbondanza relativa di tutti i generi e l'ordine batterico, rispettivamente, che vengono rilevati dall'acqua di mare di Metri nell'arco di cinque mesi.
Tabella 1: Primo contenuto di master mix PCR: La tabella mostra il contenuto del mix principale per reazione per le analisi dell'rRNA 16S e dell'rRNA 18S. Le sequenze di primer sono elencate nella Tabella 3. Fare clic qui per scaricare questa tabella.
Tabella 2: Sequenze di primer: I primer per la prima PCR sono elencati per le analisi dell'rRNA 16S e dell'rRNA 18S. Fare clic qui per scaricare questa tabella.
Tabella 3: Cicli di PCR: Sono elencati i cicli termici per la prima PCR e la seconda PCR. Fare clic qui per scaricare questa tabella.
Tabella 4: Sequenze di indice: Sono elencati i primer index 1 (i7) e index 2 (i5) da utilizzare per la seconda PCR. Fare clic qui per scaricare questa tabella.
Tabella 5: Esempio di posizionamento dell'Indice 1 e dell'Indice 2: Per ridurre gli errori, i primer dell'indice devono essere posizionati prima nella posizione e l'aliquota di ciascuno viene trasferita su una piastra a 96 pozzetti utilizzando una pipetta multicanale. Fare clic qui per scaricare questa tabella.
Tabella 6: Esempio di foglio campione: Prima della sequenziazione, è necessario creare un foglio di esempio corrispondente agli adattatori di indice 1 e indice 2. Fare clic qui per scaricare questa tabella.
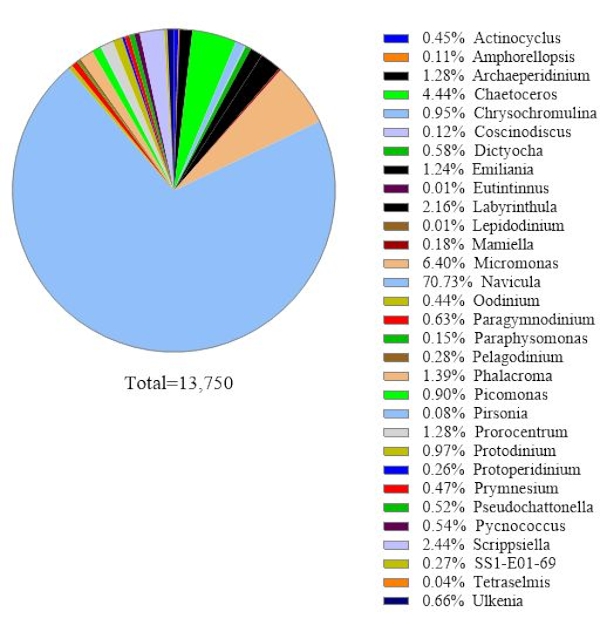
Figura 1: Risultato rappresentativo dell'analisi di metabarcoding dell'rRNA 18S: le specie algali presenti in un campione di acqua di mare raccolto da Metri, Los Lagos, Cile il 19 febbraio 2019, sono state identificate dal test di metabarcoding dell'rRNA 18S. Le sequenze assegnate a "sconosciuto" sono state eliminate e l'abbondanza relativa di ciascuna specie identificata è stata tracciata. Fare clic qui per visualizzare una versione più grande di questa figura.
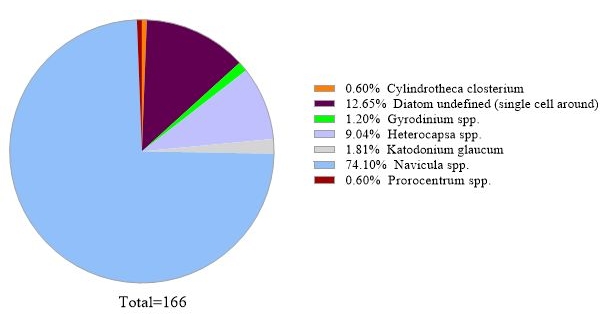
Figura 2: Risultato rappresentativo dell'analisi microscopica: le specie algali sono state identificate da un campione d'acqua di Metri, Los Lagos, Cile il 19 febbraio 2019, al microscopio. La quantità di ogni specie è stata contata manualmente e tracciata. Fare clic qui per visualizzare una versione più grande di questa figura.
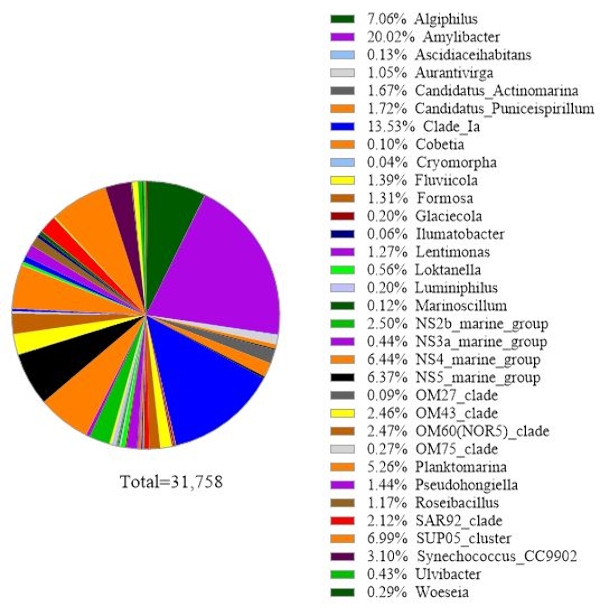
Figura 3: Risultato rappresentativo dell'analisi di metabarcoding dell'rRNA 16S: le specie batteriche presenti in un campione d'acqua di Metri, Los Lagos, Cile il 19 febbraio 2019, sono state identificate dal test di metabarcoding dell'rRNA 16S. L'abbondanza relativa di ogni specie identificata è stata tracciata. Fare clic qui per visualizzare una versione più grande di questa figura.
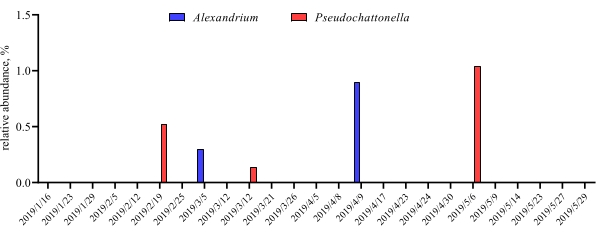
Figura 4: Grafico rappresentativo delle serie temporali di Alexandrium spp. e Pseudochattonella spp. ottenuto da 18S rRNA metabarcodingin Metri, Cile: Figura ristampata da Yarimizu et al9. Le due specie algali tossiche, Alexandrium e Pseudochattonella sono state monitorate selettivamente dall'analisi dell'rRNA 18S nel tempo e l'abbondanza relativa è stata tracciata in funzione del punto temporale. Fare clic qui per visualizzare una versione più grande di questa figura.
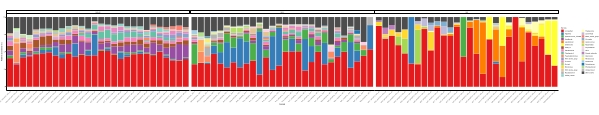
Figura 5: Grafico delle serie temporali del genere rappresentativo ottenuto dal metabarcoding dell'rRNA 16S e dell'rRNA 18S: tutti i generi batterici ed eucariotici identificati nell'acqua di Metri, Cile, monitorati per cinque mesi. Fare clic qui per visualizzare una versione più grande di questa figura.
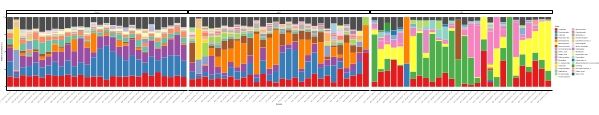
Figura 6: Grafico delle serie temporali dell'ordine rappresentativo ottenuto dal metabarcoding dell'rRNA 16S e dell'rRNA 18S: tutti gli ordini batterici ed eucariotici identificati nell'acqua di Metri, Cile, monitorati per cinque mesi. Fare clic qui per visualizzare una versione più grande di questa figura.
Tabella S1 e Tabella S2:
Tabella S1: Dati grezzi per la Figura 5 e la Figura 6. Tabella S2: Risultato del controllo di qualità sui dati grezzi della Figura 5 e della Figura 6. Fare clic qui per scaricare queste tabelle.
Discussione
Questo protocollo ha eseguito con successo l'analisi metabarcoding del gene rRNA 18S e 16S rRNA per identificare specie algali e batteriche in campioni di acqua di mare per il monitoraggio degli HAB cileni. Le alghe dominanti e alcune alghe minori rilevate da questo protocollo erano coerenti con quelle ottenute al microscopio, confermando l'affidabilità del protocollo. Il clou del protocollo è che l'analisi ha rilevato Alexandrium spp. e Pseudochattonella spp., le due specie più problematiche che causano HAB nel sud del Cile, dall'acqua di mare di Metri anche a bassa abbondanza. Soprattutto, Pseudochattonella spp. sono specie difficili da identificare al microscopio perché le cellule sono piccole e facilmente frantumabili sotto la luce o l'uso di fissativi27,28. Il metabarcoding può fornire informazioni sulle specie algali che sono scarsamente presenti nei campioni di acqua di mare che la microscopia non può identificare. Il protocollo ha anche rilevato oltre 30 specie batteriche. Sebbene il modo in cui questi batteri siano correlati alla crescita delle alghe sia sconosciuto a questo punto, la massiccia raccolta di dati sulle specie algali-batteriche nell'analisi delle serie temporali fornirà probabilmente scoperte così importanti. Pertanto, l'aggiunta dell'analisi metabarcoding ai programmi convenzionali di monitoraggio HAB cileni rafforzerà senza dubbio l'attuale efficienza di monitoraggio HAB. In effetti, questo protocollo visivo per l'analisi del metabarcoding può essere utile non solo per il monitoraggio algale-batterico dell'acqua cilena, ma anche per i programmi di monitoraggio della costa in altre aree colpite da HAB in tutto il mondo.
Sebbene questo protocollo abbia fornito i vantaggi sopra indicati, è necessario discutere gli svantaggi del metodo. Come si vede nel protocollo visivo, il metodo di metabarcoding è lungo e complesso, richiede macchinari e reagenti costosi. Il personale di laboratorio deve essere appositamente addestrato, altrimenti sprecherà materiale, manodopera e tempo. Inoltre, il rilevamento delle alghe mediante metabarcoding deve essere abbinato al microscopio per verificare che le stesse specie dominanti siano identificate dai due metodi ortogonali29. La microscopia è uno strumento non invasivo, per la maggior parte, per identificare le specie algali, il che significa che è più difficile commettere errori quando le specie algali hanno forme uniche e apparenti. Naturalmente, l'errore umano può verificarsi se le specie hanno forme molto simili tra loro. D'altra parte, poiché l'analisi della metabarcodifica richiede più passaggi, aumenta naturalmente gli errori. Potrebbe trattarsi di un campione confuso, aggiunta di reagente errata o mancante di alcune procedure. Pertanto, è fondamentale confrontare i risultati del metabarcoding con quelli ottenuti al microscopio. Infine, il protocollo è applicabile solo qualitativamente e i risultati devono essere calcolati per l'abbondanza relativa. Come Lamb et al. hanno affermato nel 2019, l'attuale metabarcoding come prestazioni quantitative è ancora limitato e sono necessarie ulteriori ricerche prima che il metabarcoding possa essere utilizzato con sicurezza per applicazioni quantitative30.
Questo protocollo è stato ottimizzato per 140 - 170 campioni per esecuzione dal manuale di preparazione della libreria di sequenziamento metagenomico 16S emesso da Illumina Inc. Il protocollo ottimizzato è stato testato molte volte e ulteriormente modificato per rilasciare questa versione finale. Pertanto, si consiglia vivamente di seguire con precisione ogni passaggio. La parte più critica del protocollo è che è necessaria una particolare attenzione per evitare la contaminazione del campione. Le pipette e la cappa a flusso laminare devono essere pulite con alcool al 70% e l'esposizione ai raggi UV in ogni momento e i materiali sterilizzabili devono essere autoclavati. I primer e i reagenti devono essere diluiti direttamente dallo stock ad ogni nuova serie, o il riutilizzo dei reagenti e l'esposizione a diverse diluizioni possono essere una causa di contaminazione del campione. Il protocollo specifica quando l'operazione può essere eseguita al di fuori della cappa a flusso laminare. Salvo diversa indicazione, i campioni devono essere trattati in una cappa a flusso laminare pulita. Quando si ha a che fare con più strisce a 8 tubi contemporaneamente, si consiglia di lavorare sulla prima striscia a 8 tubi e tapparla prima di passare alla successiva striscia a 8 tubi. Lasciare il coperchio aperto per lungo tempo può causare la contaminazione del campione poiché i geni dell'rRNA 16S e 18S sono altamente onnipresenti nell'ambiente circostante. Il tempo indicato, come il tempo di miscelazione e il tempo di incubazione, deve essere seguito come esattamente descritto perché la durata migliore per ogni fase è stata selezionata in base alle molteplici esecuzioni di test. Un tempo eccessivo o insufficiente può, ad esempio, ridurre la resa del campione. Il processo dovrebbe essere interrotto solo nella parte in cui il protocollo dice che può fermarsi. È fondamentale avere un controllo negativo in quanto conferma che i risultati positivi non sono falsi positivi artificiali. Infine, si consiglia vivamente di pianificare i giorni perché richiede almeno cinque giorni per elaborare tutti i passaggi e alcune parti non possono essere fermate fino al prossimo segnale di arresto.
La limitazione dell'assegnazione tassonomica per i dati sequenziati è brevemente notata qui. Le sequenze nucleotidiche recentemente scoperte per le specie batteriche e algali vengono aggiornate quotidianamente nella banca dati. Mentre le specie algali e batteriche ben studiate sono registrate in modo affidabile, ci sono anche molte sequenze per specie sconosciute aggiornate nella banca dati. Indica che la risoluzione delle sequenze nucleotidiche registrate non è sempre sufficiente per l'identificazione delle specie, ma solo per l'identificazione del genere. Pertanto, l'identificazione microscopica delle specie deve essere eseguita ortogonalmente. La creazione di una pipeline ad accesso aperto per questo lavoro ha un vantaggio significativo nel gestire un ampio set di dati sequenziati contemporaneamente e nell'indagare in modo efficiente quando si verifica un errore. Inoltre, l'output di DADA2 è in un file di testo o csv, il che semplifica ulteriori analisi statistiche. D'altra parte, è necessario un server di grandi dimensioni per eseguire assegnazioni tassonomiche, specialmente quando un set di dati è di grandi dimensioni. Per configurare la pipeline, sono necessari ingegneri per impostare la pipeline e i professionisti devono comprendere parametri, funzionamento, Linux e bioinformatica.
Oltre all'ambito come strumento di monitoraggio HAB, l'uso di questo protocollo può essere ampliato per scopi di ricerca investigativa. Ad esempio, ci sono dibattiti in corso tra il governo cileno e i pescatori locali insieme alle associazioni per la pace ambientale in merito all'aumento del numero di HAB31locali,32. Il governo sostiene che è dovuto a un fenomeno naturale come il riscaldamento globale e El Niño, mentre le parti successive sostengono che l'acquacoltura del salmone è la causa. Il salmone non è una specie indigena del Cile e non era nell'acqua cilena mezzo secolo fa. Il governo cileno a quel tempo mirava a far crescere l'economia e creare posti di lavoro per le aree povere sviluppando un'attività di salmone33. Con l'intervento dall'estero per profitto di capitale, il Cile ha avuto un grande successo nello sviluppo dell'acquacoltura a penna, e il numero di salmoni è aumentato notevolmente negli ultimi decenni31,32,33. Successivamente, una grande quantità di cibo per il salmone è stata gettata in mare, portando la popolazione locale a sospettare che l'acquacoltura del salmone sia la ragione dei frequenti HAB locali31,32. La verità è sconosciuta ora, ma deve essere compresa alla fine in modo che possano essere escogitate strategie per proteggere l'ambiente marino locale, l'economia e la salute umana dagli HAB. L'analisi molecolare sulla relazione algale-batterica locale può contribuire a un chiarimento in avanti per tale soggetto. Ad esempio, la tecnica può cercare le specie algali e batteriche che prima non erano presenti nell'area oceanica bersaglio ma che sono aumentate drasticamente negli ultimi anni, il che è in qualche modo simile allo studio fatto da Sakai et al. per la scoperta di una popolazione non registrata di salamandra Yamato (Hynobius vandenburghi) da GIS e eDNA analisi34. Pertanto, al di là dello strumento di monitoraggio HAB, questo protocollo di metabarcoding ha il potenziale utilizzo per altri potenziali clienti.
Divulgazioni
Gli autori non dichiarano alcun conflitto di interessi. I finanziatori non hanno avuto alcun ruolo nella progettazione dello studio, nella raccolta, nell'analisi o nell'interpretazione dei dati; nella stesura del manoscritto, o nella decisione di pubblicare i risultati.
Riconoscimenti
Questo studio è stato supportato dalla sovvenzione (JPMJSA1705) per uno studio su Science and Technology Research Partnership for Sustainable Development-Monitoring Algae in Chile (SATREPS-MACH). Ringraziamo la dottoressa Sandra Rios (Universidad de Los Lagos) per averci permesso di utilizzare un filmato. Ringraziamo Neal Andrew Holland per la sua diligente correzione di bozze del manoscritto. Esprimiamo il nostro sincero apprezzamento ai membri del gruppo di laboratorio del CREAN-IFOP di Puerto Montt, in Cile, per averci consigliato sull'identificazione del fitoplancton.
Materiali
| Name | Company | Catalog Number | Comments |
| 1.5 mL single tube | ThermoFisher | Q32856 | sterile |
| 1.5 mL tubes | ThermoFisher | 2150N | sterile |
| 8-strip 0.2 mL PCR tubes and flat cap | ThermoFisher | AB0451 & 4323032 | sterile |
| 96-well 0.2 mL PCR plate | ThermoFisher | N8010560 | |
| AccuRuler 100 bp DNA Ladder | MaestroGen | 02001-500 | |
| Chelex 100 Chelating Resin | Bio-Rad | 1432832 | |
| Chemical Duty Vacuum Pressure Pump | MilliporeSigma | WP6122050 | |
| D1000 Reagents | Agilent Technologies | 5067-5583 | |
| D1000 sample buffer | Agilent Technologies | 5067-5602 | |
| DNA loading dye (Blue/Orange Loading Dye 6X) | Promega | G1881 | |
| Ethanol Molecular Biology Grade | E7023 | Merck KGaA | |
| Filtration device | ThermoFisher | 09-740-36H, 09-740-23E | |
| Fluorescent DNA concentration measurement kit (Qubit dsDNA HS Assay kit) | Thermo Fisher Scientific | Q32851 | |
| Fluorometer (Qubit4 Fluorometer) | ThermoFisher | Q33238 | |
| Fragment analysis verification matrix (D1000 ScreenTape) | Agilent Technologies | 5067-5582 | |
| Fragment Analyzer (Agilent Tapestation 4150) | Agilent | G2992AA | |
| GelRed Nucleic Acid Gel Stain | Biotium | 41002 | |
| Generic bench vortexer (Vortex Genie 2) | Scientific Industries | SI-0236 | |
| Heat block | |||
| High performance sequencing-grade DNA polymerase (KAPA HiFi Hotstart ReadyMix), 2X | Roche | KK2602 | |
| HT1 Hybridization buffer | Illumina | 20015892 | |
| Illumina Nextera XT v2 Index Kit set A, B, C and D | Illumina | FC-131-2001, -2002, -2003, -2004 | |
| Laminar hood cabinet | Biobase Co. | Biobase PCR-800 PCR Cabinet | |
| Loading tips (Specific for Agilent TapeStation systems) | Agilent Technologies | 5067-5598 | |
| Magnetic beads DNA cleanup system (ProNex® Size-Selective Purification System) | Promega | NG2002 | |
| Magnetic stand for 96 wells (Magnetic Stand-96) | ThermoFisher | AM10027 | |
| Micro-seal film for 96-well plate | ThermoFisher | 4311971 | sterile |
| MiSeq Reagent Kit v3 | Illumina | MS-102-3003 | includes pre-filled, ready-to-use reagent cartridges. |
| MiSeq system | Illumina | SY-410-1003 | includes pre-installed software |
| Molecular grade nuclease-free water | Integrated DNA Technologies | 11-05-01-04 | |
| Molecular grade sodium hydroxide (NaOH) 1M | Merck KGaA | 1091371000 | |
| Multichannel pipettes | Not specified | Not specified | |
| Multi-Purpose Agarose | Cleaver Scientific | CSL-AG500 | |
| Ow D2 Wide-Gel Electrophoresis System | ThermoFisher | D2 | |
| Ow EC-105 Compact Power Supply | ThermoFisher | 105ECA-115 | |
| Pellet Pestle— Cordless Motor | ThermoFisher | K749540-0000 | |
| PhiX control Kit v3 | Illumina | FC-110-300 | ready-to-use control library for Illumina sequencing |
| Pipette tips | Not specified | Not specified | sterile |
| Pipettes | Not specified | Not specified | 2, 10, 20, 100, 1000 μL |
| SterivexTM GP 0.22 μm filter unit | MilliporeSigma | SVGP01050 | |
| Tabletop centrifuge | Eppendorf 5427R Centrifuge | Eppendorf AG | |
| TBE buffer | ThermoFisher | B52 | |
| TE buffer (pH 8.0) | ThermoFisher | AM9849 | |
| Terr PCR Direct FFPE Kit | Takara Bio USA | 639284 | includes 2X Terra PCR Direct Buffer |
| Terra PCR Direct Polymerase Mix, 1.25 U/μL | Takara Bio USA | 639271 | High performance Inhibitor-tolerant DNA polymerase |
| ThermalCycler (MiniAMP Plus Thermal Cycler) | ThermoFisher | A37835 | |
| TransIlluminator and image capture system | Analytik Jena, AG | GelDoc-ItTS2 Imager | |
| Whatman 1.0 m pore-sized membrane | MilliporeSigma | WHA111110 |
Riferimenti
- Hallegraeff, G., Enevoldsen, H., Zingone, A. Global harmful algal bloom status reporting. Harmful Algae. 102, 101992 (2021).
- Anderson, D. M. Red Tides. Scientific American. 271 (2), 52-58 (1994).
- Sanseverino, I., Conduto, A. D. S., Pozzoli, L., Dobricic, S., Lettieri, T. . Algal Bloom and its Economic Impact. , (2016).
- Molinet, C., Niklitschek, E., Seguel, M., Díaz, P. Trends of natural accumulation and detoxification of paralytic shellfish poison in two bivalves from the Norwest Patagonian inland sea. Revista de Biologia Marina Y Oceanografia. 45, 195-204 (2010).
- Mardones, J., Clement, A., Rojas, X., Aparicio, C. Alexandrium catenella during 2009 in Chilean waters, and recent expansion to coastal ocean. Harmful Algae News. 41, 8-9 (2010).
- Alvarez, G., et al. Paralytic shellfish toxins in surf clams Mesodesma donacium during a large bloom of Alexandrium catenella dinoflagellates associated to an intense shellfish mass mortality. Toxins (Basel). 11 (4), (2019).
- Clément, A., et al. Exceptional summer conditions and HABs of Pseudochattonella in Southern Chile create record impacts on salmon farms. Harmful Algae News. 53, 1-3 (2016).
- Trainer, V. L., et al. Pelagic harmful algal blooms and climate change: Lessons from nature's experiments with extremes. Harmful Algae. 91, 101591 (2020).
- Yarimizu, K., et al. Protocols for monitoring harmful algal blooms for sustainable aquaculture and coastal fisheries in Chile. International Journal of Environmental Research and Public Health. 17 (20), (2020).
- Roegner, G. C., Hickey, B. M., Newton, J. A., Shanks, A. L., Armstrong, D. A. Wind-induced plume and bloom intrusions into Willapa Bay, Washington. Limnology & Oceanography. 47 (4), 1033-1042 (2002).
- Tweddle, J. F., et al. Relationships among upwelling, phytoplankton blooms, and phycotoxins in coastal Oregon shellfish. Marine Ecology Progress Series. 405, 131-145 (2010).
- Glibert, P. M., Anderson, D. M., Gentien, P., Graneli, E., Sellner, K. G. The global complex phenomena of harmful algae blooms. Oceanography. 18 (2), 130-141 (2005).
- Paredes-Mella, J., Varela, D., Fernández, P., Espinoza-González, O. Growth performance of Alexandrium catenella from the Chilean fjords under different environmental drivers: Plasticity as a response to a highly variable environment. Journal of Plankton Research. 42 (2), 119-134 (2020).
- Azam, F., Malfatti, F. Microbial structuring of marine ecosystems. Nature Reviews Microbiology. 5, 782-791 (2007).
- Amin, S. A., et al. Interaction and signaling between a cosmopolitan phytoplankton and associated bacteria. Nature. 522, 98-101 (2015).
- Berdalet, E., et al. Marine harmful algal blooms, human health, and wellbeing: Challenges and opportunities in the 21st century. Journal of Marine Biology Association. 2015, 1-31 (2015).
- Ramanan, R., Kim, B. -. H., Cho, D. -. H., Oh, H. -. M., Kim, H. -. S. Algae-bacteria interactions: Evolution, ecology, and emerging applications. Biotechnology Advances. 34 (1), 14-29 (2016).
- Seymour, J. R., Amin, S. A., Raina, J. B., Stocker, R. Zooming in on the phycosphere: The ecological interface for phytoplankton-bacteria relationships. Nature Reviews Microbiology. 2, 17065 (2017).
- Maruyama, F., et al. 16S and 18S Metabarcoding analysis for Chilean coastal waters harmful algal blooms. protocol.io. , (2020).
- Tanabe, A. S., et al. Comparative study of the validity of three regions of the 18S-rRNA gene for massively parallel sequencing-based monitoring of the planktonic eukaryote community. Molecular Ecology Resources. 16, 402-414 (2016).
- Nishitani, G., et al. Multiple plastids collected by the Dinoflagellate Dinophysis mitra through Kleptoplastidy. Applied and Environmental Microbiology. 78 (3), 813-821 (2012).
- Klindworth, A., et al. Evaluation of general 16S ribosomal RNA gene PCR primers for classical and next-generation sequencing-based diversity studies. Nucleic Acids Research. 41 (1), 1 (2013).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nature Methods. 13 (7), 581-583 (2016).
- McMurdie, P. J., Holmes, S. phyloseq: An R Package for Reproducible Interactive Analysis and Graphics of Microbiome Census Data. PLoS One. 8 (4), 61217 (2013).
- Dixon, P. VEGAN, a package of R functions for community ecology. Journal of Vegetation Science. 14 (6), 927-930 (2003).
- Dittami, S. M., Riisberg, I., Edvardsen, B. Molecular probes for the detection and identification of ichthyotoxic marine microalgae of the genus Pseudochattonella (Dictyochophyceae, Ochrophyta). Environmental Science and Pollution Research. 20 (10), 6824-6837 (2013).
- Mardones, J. I., et al. Salinity-growth response and ichthyotoxic potency of the Chilean Pseudochattonella verruculosa. Frontiers in Marine Science. 6 (24), (2019).
- Santi, I., Kasapidis, P., Karakassis, I., Pitta, P. A comparison of DNA metabarcoding and microscopy methodologies for the study of aquatic microbial eukaryotes. Diversity. 13 (5), 180 (2021).
- Lamb, P. D., et al. How quantitative is metabarcoding: A meta-analytical approach. Molecular Ecology. 28 (2), 420-430 (2019).
- Mascareño, A., et al. Controversies in social-ecological systems: lessons from a major red tide crisis on Chiloe Island, Chile. Ecology and Society. 23, (2018).
- Armijo, J., Oerder, V., Auger, P. -. A., Bravo, A., Molina, E. The 2016 red tide crisis in southern Chile: Possible influence of the mass oceanic dumping of dead salmons. Marine Pollution Bulletin. 150, 110603 (2020).
- Swanson, H. A. . Caught in Comparisons: Japanese Salmon in an Uneven World. , (2013).
- Sakai, Y., et al. Discovery of an unrecorded population of Yamato salamander (Hynobius vandenburghi) by GIS and eDNA analysis. Environmental DNA. 1 (3), 281-289 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon