需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
使用循环离子淌度光谱仪进行串联离子淌度实验
摘要
离子淌度光谱法(IMS)是质谱法表征生物分子的有趣补充,特别是因为它对异构体敏感。该协议描述了串联IMS(IMS / IMS)实验,该实验允许分离分子并生成其片段的迁移率曲线。
摘要
准确表征化学结构对于了解其潜在的生物学机制和功能特性非常重要。质谱(MS)是一种流行的工具,但并不总是足以完全揭示所有结构特征。例如,尽管碳水化合物具有生物学相关性,但它们的表征因许多级别的异构体而变得复杂。离子淌度光谱(IMS)是一个有趣的补充,因为它对离子构象敏感,因此对异构体敏感。
此外,最近的进展显着改进了该技术:与线性IMS仪器相比,上一代Cyclic IMS仪器提供了额外的功能,例如增加的分辨能力或执行串联离子迁移率(IMS / IMS)实验的可能性。在IMS/IMS期间,根据其离子迁移率选择离子,碎片化并重新分析,以获得有关其片段的离子迁移率信息。最近的研究表明,这种IMS / IMS数据中包含的片段的迁移率曲线可以充当特定聚糖的指纹,并且可以用于分子网络策略中,以结构相关的方式组织糖组学数据集。
因此,该协议的目标是描述如何生成IMS / IMS数据,从样品制备到离子迁移率维度的最终碰撞截面(CCS)校准,从而产生可重复的光谱。以一种代表性聚糖为例,该协议将展示如何在循环IMS仪器上构建IMS/IMS控制序列,如何考虑该控制序列以将IMS到达时间转换为漂移时间(即应用于离子的有效分离时间),以及如何从原始数据中提取相关的迁移率信息。该协议旨在清楚地解释IMS / IMS实验的关键点,从而帮助新的Cyclic IMS用户执行直接且可重复的采集。
引言
生物分子的完整化学表征是了解其潜在生物学和功能特性的关键。为此,近年来,"组学"科学不断发展,旨在大规模表征生物浓度下的化学结构。在蛋白质组学和代谢组学中,MS已成为揭示生物介质中发现的结构异质性的核心工具 - 特别是由于其灵敏度和通过串联MS(MS / MS)提供结构信息的能力。在MS / MS策略中,根据其质量选择离子,然后片段化,最后获取其片段的质量以建立分子的指纹。MS/MS光谱尤其可用于匹配光谱数据库1,2,或试探性地重建父结构3,4。在相似光谱属于相似化合物的假设下,MS/ MS数据也可用于构建通过相似性评分连接相关物种的分子网络(MNs)5,6。
然而,由于MS检测离子的质荷比(m / z)的固有特性,该技术对落在(立体)异构体范围内的许多结构特征视而不见。例如,碳水化合物由几种单糖亚基组成,其中许多是立体异构体甚至异构体(例如,Glc与Gal或Glc与人)。这些亚基由糖苷键连接,糖苷键可以通过键的位置(区域异构体)和无构体碳的空间位阻构型(非对称性)而不同。这些特性使得独立的MS难以区分碳水化合物异构体7,并且只能使用高能活化方法解决区域异构体8,9,10。虽然衍生化是破坏立体异构群等价性的一种选择11,但它需要大量的样品制备。另一个更直接的选择是将MS与对异构体敏感的分析维度(如IMS)耦合。
由于此协议是为已经熟悉 IMS 基本概念的用户设计的,并且由于其他地方提供了详细的评论12,13,因此此处仅简要概述了 IMS 的原理。IMS是一种气相分离方法,它依赖于离子与缓冲气体和电场的相互作用,最终根据其气相构象分离离子。在商用仪器上可以找到耦合到MS的IMS的不同原理:一些在高低电场(场不对称IMS,FAIMS)的交替下工作,而大多数在低场限值内工作 - 特别是漂移管IMS(DTIMS,线性递减电场),行波IMS(TWIMS,对称电位波)和捕获IMS(TIMS,高流量缓冲气体捕获离子对抗电场)13.低场方法允许访问所谓的CCS,这是离子 - 气体对的一种性质,代表离子的表面(在Å2 或nm2中),在分离过程中与缓冲气体相互作用。CCS在理论上与仪器无关,因此可用于生成可在不同实验室之间再现的数据14。离子淌度分离可能受到各种参数的影响,特别是受迁移池中气体压力和气体温度波动的影响。CCS校准是解决这个问题的一种方法,因为校准剂和感兴趣的物种都会受到类似的影响13。但是,必须将仪器安装在温度受控的房间内,并具有可靠的气体压力控制系统。
IMS的一个有趣的演变是IMS / IMS,它于2006年由Clemmer的小组首次引入,作为MS / MS15,16的类似物。在IMS / IMS中,根据其离子迁移率选择性地分离感兴趣的离子;然后它被激活(直到可能的碎片),并对激活的离子或片段进行新的IMS分析。在第一个仪器设计中,两个IMS细胞串联放置,由激活所在的离子漏斗隔开。从那时起,尽管提出了许多IMS / IMS设置(有关审查,请参阅Eldrid和Thalassinos17),但第一台具有IMS / IMS功能的商用质谱仪直到2019年才上市18。该仪器通过将它与另一项技术突破相结合,大大改进了最初的概念:IMS单元的循环设计。
理论上,循环IMS单元允许近乎无限地增加漂移路径长度,从而增加仪器的分辨能力19。这是通过特定的仪器几何形状实现的,其中循环TWIMS单元正交放置在主离子光轴上。IMS单元入口处的多功能阵列区域允许控制离子路径的方向:(i)将离子侧向发送以进行IMS分离,(ii)向前用于MS检测,或(iii)从IMS单元向后存储到预阵列单元中。从这个预阵列存储池中,离子可以被激活,片段在IMS细胞中重新注入以进行离子迁移率测量,这种方法已成功用于表征立体异构体20。最终,收集的数据包含前体及其片段的离子迁移率和 m / z 信息。
在最近一篇使用这种循环设计进行聚糖分析的出版物(Ollivier等人21)中,我们表明,这种IMS / IMS数据中包含的片段的迁移率曲线充当生物分子的指纹,可用于分子网络策略。由此产生的网络称为IM-MN,导致糖组学数据集以结构相关的方式进行组织,而仅由MS / MS数据(MS-MN)构建的网络几乎没有透露信息。为了补充本出版物并帮助Cyclic IMS用户实现此工作流,此协议提供了用于收集数据的协议的完整描述。该协议仅关注 IMS/IMS 数据的生成,然后用户可以使用这些数据来构建 IM-MN 网络(请参阅 21),或用于他们选择的任何其他应用程序。本文将不考虑IM-MN的构建,因为分子网络的方案已经可用22。强调了为产生有价值和可重复的IMS / IMS收购而必须遵循的关键点。以Ollivier等人研究的寡糖之一 为例。21、以下步骤详述:(i)样品制备,(ii)循环IMS仪器的调谐,(iii)数据的自动峰值拾取,以及(iv)CCS校准。
研究方案
注意: 图 1 提供了该协议的概述。用于本方案中描述的实验的参数可以在 补充表S1 和 补充表S2中找到。
1. 样品溶液的制备
注意:该协议是使用阿拉伯木聚糖五糖(23-α-L-阿拉伯呋喃糖基木基四糖或XA2XX;见材料表)作为示例来描述的。
- 溶剂的制备:500μM LiCl在50:50 H2O:MeOH(体积/体积)中。
- 通过称量212mg LiCl,在H2 O中制备100mM氯化锂(LiCl)储备溶液,并在50mL聚丙烯锥形管中加入50mL高纯度去离子水(H2O)。摇晃直至完全溶解。
注意:溶剂掺杂锂盐以促进光谱仪离子源中[M + Li]+ 加合物的形成,因为与其他碱加合物相比,它通常产生质量更好的碎裂光谱。建议使用LiCl,因为以前已经发现有机酸(以及它们的盐)会影响IMS配置文件23。 - 在玻璃瓶中,将LiCl储备溶液稀释200x:加入250μL储备溶液,加入24.75mL H2O.加入25 mL甲醇(MeOH)以达到500μM的LiCl在50:50 H2O:MeOH(v / v)中的最终浓度。超声处理2分钟以脱气溶剂。
注:MeOH存在健康危害(H225,H301,H311,H331,H370);在拔出器罩下操作,穿着实验室外套,手套和护目镜。比例为50:50 MeOH / H2O(v / v)似乎是寡糖电离的最佳溶剂;然而,如果需要,MeOH可以被乙腈(ACN)取代。
- 通过称量212mg LiCl,在H2 O中制备100mM氯化锂(LiCl)储备溶液,并在50mL聚丙烯锥形管中加入50mL高纯度去离子水(H2O)。摇晃直至完全溶解。
- 在1.5 mL聚丙烯管中,称取1mg碳水化合物。以适当体积的500μM LiCl溶解,以达到1mg / mL的浓度。在50:50 MeOH / H 2 O + 500μM LiCl中稀释至10μg/ mL的终浓度。储存在4°C。
注意:选择10μg/mL的浓度是为了优化IMS/IMS-MS期间所有片段离子的信号(这是针对纯化合物;在处理混合物时增加浓度)。为了获取参考IMS / IMS光谱,不要进一步稀释样品:尽管仪器提供了校正它的选项,但MS检测器在碎片化之前预计会饱和(参见步骤3.2)。
2. 循环IMS质谱仪的调谐
注:与软件相关的说明(窗口、菜单和命令)以 粗体突出显示。
- 从仪器控制软件(MS调谐 页面,请参阅 材料表中的软件详细信息)打开仪器控制台,然后将仪器置于 操作 模式。等待至少3小时,以使IMS电池中的高压稳定下来。
注意:为了获得最佳的再现性,IMS电池中的电压需要完全稳定。打开高压,让仪器在一夜之间稳定下来,然后再进行任何循环IMS分析。此外,离子淌度池中的压力和温度必须尽可能保持恒定。尽管" 真空 "选项卡中提供了压力的回读,但对温度没有回读可用。将仪器保存在恒温实验室中。这项工作中使用的仪器在20°C恒温的实验室中以1.75毫巴的速度运行。 - 循环 IMS 仪器设置
注:标准溶液必须使用内置流体系统进行仪器设置。- 将装满制造商提供的相应标准的流体容器放在流体系统上:储液槽B("锁质"):10 pg / μL亮氨酸脑啡肽(LEU ENK)在50:50 ACN / H2O + 0.1%甲酸中;水库 C("Calibrant"):MajorMix。
注:在此协议中,MajorMix校准溶液将用于校准 m/z 和CCS尺寸。出于实际原因,将进行外部CCS校准(参见协议的步骤5);因此,也可以将内部校准剂混合物用于CCS,另一种校准剂用于 m / z (例如甲酸钠或碘化钠)。 - 在 Quartz 控制台的 "调谐 "页面上,转到" 流体 "选项卡。将样品流体学设置为储液槽C,将参考流体学设置为储液槽B.将两种溶液连续注入离子源以检查MS信号。
- 根据制造商的说明,从仪器设置页面执行ADC设置、检测器设置(使用LEU ENK)和质量校准(参见校准解决方案的材料表)。
- 将装满制造商提供的相应标准的流体容器放在流体系统上:储液槽B("锁质"):10 pg / μL亮氨酸脑啡肽(LEU ENK)在50:50 ACN / H2O + 0.1%甲酸中;水库 C("Calibrant"):MajorMix。
- 通过单次分离记录校准溶液的 IMS 采集(将其用于外部 IMS 校准)。
注:在所有采集(校准和采集)期间,离子源和行波(TW)参数(静态波高和波速)必须保持恒定。如果用户对其样品的最佳参数没有先验知识,则可以在方案的步骤3之后执行该步骤(对于中性寡糖的[M + Li]+ 加合物,代表性结果使用16 V的TW高度和350 m / s的TW速度,这给出了最佳结果)。- 从 "流体 学"选项卡中,选择挡板位置 样品 ,然后通过"样品"探头以10μL/min的流速将校准剂(参见 材料表)注入离子源(使用内置流体系统)。
- 设置单通道 IMS 序列。在 "调谐 "页面中,将乐器置于 移动 模式,然后打开" 循环序列控制 "窗口。选择" 高级 模式"。从此新窗口的循环 函数 选项卡中,选择 添加捆绑包,然后选择 单/多通道。等待一系列移动事件出现在同一窗口的" 序列 "选项卡中。
注意:要激活实时显示,用户必须应用仪器参数:单击"在 TOF 模式下调谐"或"在移动模式下运行"。在 TOF 和 Mobility 模式之间切换仪器之前,必须中止任何正在运行的采集(包括 Tune 页面显示)。由于离子传输参数的变化,TOF模式和迁移率模式之间的相对丰度可能会有所不同。 - 调整顺序,使所有校准离子在循环IMS赛道上进行一次通过。不要更改注射时间或弹出并获取时间;但是,请将"分离时间"降低到 1 毫秒(在"序列"选项卡中)。如果校准混合物的某些离子不适合显示的到达时间窗口,请在ADC设置选项卡中增加每个箱的推送次数,以更改IMS与正交加速度TOF分析仪的推杆的同步。
注:控制序列中的时间仅控制用于离子浇注的多功能阵列。只要离子在赛道上进行第一次(或第n次)传递,即使TW的方向在此期间在阵列中发生了变化,它们也会完成所述传递。将分离时间降低到 1 毫秒意味着阵列将在 1 毫秒后切换到弹出模式。这确保了较快的离子将没有足够的时间通过阵列并在较慢的离子完成第一次通过之前进行第二次传递。因此,所有离子都将经历相同次数的通过(即一次通过),这是执行IMS校准所必需的。 - 记录 2 分钟的采集。在" 循环序列控制 "窗口中,单击" 采集 "以打开 "采集设置" 弹出窗口。输入 文件名、 描述和 采集长度(分钟), 然后单击 保存。
- 在与步骤2.3相同的条件下,再采集2分钟的校准溶液(用它来检查CCS校准的质量)。在" 循环序列控制 "窗口中,单击" 采集 "以打开 "采集设置" 弹出窗口。输入 文件名、 描述和 采集长度(分钟), 然后单击 保存。
- 用50:50 H 2 O / ACN彻底清洗流体系统,以避免吸音管中的校准剂结晶。
3. IMS/IMS-MS 采集
- 使用注射泵,以10μg/mL的流速将(锂掺杂的)样品以10μg/mL的流速通过样品探针注入。
- 将仪器切换到 TOF 模式(从 MS调谐 页面)以检查信号的稳定性。记录样品的完整MS采集(1分钟),这对于检查同位素模式和潜在污染物的存在非常有用。
注意:由于选择样品浓度是为了获得片段的良好离子信号,因此在此步骤中可以观察到TOF饱和度。TOF饱和度可以使用以下伪影来识别:(i)人为增加的MS分辨率,(ii)同位素比的变化,以及(iii)同位素之间的大量低丰度峰。使用 DRE 透镜(动态范围增强,调谐主页面的四分频/MS 配置文件/DRE 选项卡)在 TOF 模式下衰减离子的传输并丢弃饱和度(图 2A,B)。 - 将仪器置于 MSMS 模式("调谐"主页面的"四分仪/质谱"选项卡),并在"MSMS 质量"字段中选择目标离子的质量,以便在四极杆中进行分离(例如:m/z 为 685.2,对应于阿拉伯木聚糖五糖的 [M+Li]+ 离子种)。记录1分钟的采集,以在处理数据时检查前体隔离。
注意:锂加合物在同位素峰的-1 Da处有同位素,需要从MS / MS选择窗口中移除,以免干扰处理步骤。可以通过使用四分形/毫秒轮廓选项卡中的LM分辨率和HM分辨率参数缩小选择范围来删除它(图2C)。 - 设置"切片"IMS序列,以对目标异构体执行基于迁移率的选择。
- 将仪器切换到 移动 模式(请参阅步骤 2.3.2)。在" 循环序列控制 "窗口中,从" 循环函数 "选项卡中,选择 "添加捆绑包 ",然后选择" 切片"。等待复杂的移动事件序列出现在" 序列 "选项卡中(图 3)。
注意:可以可视化 IMS/IMS 过程的每个步骤:单击"序列"选项卡中的"弹出并获取"事件。以红色突出显示后,使用"向上"和"向下"按钮将其移动到序列中的适当位置。 - 将 "弹出"和"获取" 事件放在第一个 "单独" 事件之后(即,在 如图 3 所示的序列中,将其移动到第 3 行而不是第 8 行),然后单击" 运行"。查找要实时显示的初始分离结果。通过更改序列中此事件的时间值,直到 IMS 峰值的分辨率令人满意,来增加多通道分离的第一个 Separate 事件的持续时间。记录1分钟的采集以供参考。
注:请注意" ADC设置 "选项卡中的 ADC 启动延迟值:检查隔离质量会很有用。 - 点击 暂停。请注意,将显示初始分离的结果,尽管在用户再次单击" 运行 "之前,不会应用控件序列中的修改。将" 弹出并获取" 事件放在" 弹出"、" 弹出到预存储"和 "保留并弹出" 事件下方。调整事件的持续时间,使目标峰值位于 "弹出到预存储" 区域中,而任何其他离子位于 "弹出" 或 "保留并弹出" 区域。
注:与到达时间分布 (ATD) 相比,这三个事件的持续时间可以使用" 移动图 "选项卡中移动频谱下方的颜色编码条进行可视化(图 3)。 - 将 "弹出并获取" 事件放在序列末尾的 "从预存储重新输入" 和第二 个"单独" 事件的下方。单击 "运行 "以显示所选人口。
注意:由于选定的群体已离开 IMS 单元,因此所有先前的分离都已丢失,并且将返回到单通道分离(这是必需的)。 - 检查隔离的质量。要验证是否只选择了感兴趣的峰值,请在重新注射后执行与重新注射前相同的分离(即相同的 分离 时间),如图 4所示。记录1分钟的采集以供参考。
注意:鼓励用户检查弹出的群体; "弹出到预存储" 时间窗口应为基线水平(图 4B)。要检查这一点,请将 ADC启动延迟 置于 手动 模式的 ADC设置 选项卡中,然后输入步骤3.4.2中记录的延迟时间。记录1分钟的采集以供参考。 - 在"序列"选项卡的用户定义事件时间旁边的列("时间 Abs"列,以红色突出显示)中,查找所有事件的求和时间。请注意在"从预存储重新注射"事件行中找到的 Time Abs,用于执行 CCS 校准。
- 将仪器切换到 移动 模式(请参阅步骤 2.3.2)。在" 循环序列控制 "窗口中,从" 循环函数 "选项卡中,选择 "添加捆绑包 ",然后选择" 切片"。等待复杂的移动事件序列出现在" 序列 "选项卡中(图 3)。
- 在两轮 IMS 之间对目标峰值进行分段。改变回射步骤的电压以增加离子的动能,并在与离子迁移率气体碰撞时将其碎片化。
- 将"弹出并获取"之前的"分离"事件的持续时间设置为 1 毫秒(请参阅步骤 2.3.3 中的说明)。
- 在 "从预存储重新输入 "行中,勾选 "启用激活 "框,并使用内置控件优化碎片。如果光谱令人满意(例如,基峰是片段),请直接进入步骤3.5.4。
注意:启用激活时,线路上的三个电压将变为灰色:如果需要手动优化电压,这些是用户需要更改的电压(请参阅下一步)。这三个电压(预阵列梯度、预阵列偏置和阵列偏移)形成用于激活离子的梯度。离子的动能将随着预阵列偏置和阵列偏移之间的斜率而增加(见图5)。梯度→偏置→偏移值的默认值为:不带激活 85 → 70 → 45 V;内置功能的最大激活电压为 185 → 170 → -5 V (+150 V)。分片后,不要忘记使用DRE透镜重新调整离子传输(减少信号的衰减)(见步骤3.2)。 - 如果分段与内置控件不满意,请取消选中 启用激活 框,然后继续手动优化重新注入电压。增加 阵列前梯度 电压( 阵列前偏置 电压必须始终保持在比 阵列前梯度低15 V),并降低 阵列失调 电压(可设置为负),直到结果令人满意。
注:手动调谐多功能阵列的电压时,用户可以从"Mobilogram"视图切换到多功能阵列中施加的电压的交互式原理图(PE图),以更好地可视化电压设置(图5A)。 - 记录 2 分钟的采集。在采集弹出窗口中,勾选 "保留漂移时间 "选项以生成仅包含到达时间与 m/z 的文件(用于色谱分析的采集时间 - 保留时间 - 已从文件中删除)。请注意,此文件标记为 *_dt。原始。
注意:如果用户忘记选中 "保留漂移时间 "选项,仍然可以使用 Driftscope 2.9 软件(文件|提取 IMS 维度。导出到 MassLynx |保留漂移时间)。
- 在"调谐"主页面中将仪器转回 TOF 模式,并用 50:50 MeOH/H2O 彻底冲洗系统,然后再继续下一个样品。
4. 使用 MZmine 224 进行 IMS/IMS-MS 处理
注:MZmine 2 可从 材料表中给出的 URL 获得。建议使用MZmine 2.51。在准备本手稿时,由于导入功能的更改,后期版本无法从Cyclic IMS仪器打开RAW文件。
- 导入仅包含 IMS 和 m/z 尺寸 (*_dt的原始文件。RAW) 使用 Raw 数据方法|原始数据导入。
注意:Raw 文件将显示在 MZmine 主窗口的左侧。不要导入原始 *。 仍包含保留时间维度的 RAW 文件。MZmine不会将保留时间与IMS到达时间区分开来,并且两个维度的数据点会重叠。 - 优化代表性文件上的工作流参数,方法是在 原始数据文件 列表。
- 评估数据中的噪声水平。右键单击" 原始数据文件"列表中的文件 ,选择" 显示 TIC "并显示基峰"色谱图"(BPC)。双击眼睛可观察到的最小峰值以显示其质谱。将数据中的噪声水平考虑为该光谱中基峰的第二个同位素的水平,并在以下处理步骤中对所有强度阈值使用相同的值。
注:数据是使用四极杆分离获得的,因此MZmine将其视为MS / MS.在整个MZmine处理过程中,请确保在 MS级别= 2下工作。 - 使用 原始数据方法执行质量检测|特征检测|质量检测。对于在配置文件模式下采集的数据,请使用 小波变换 算法。要在 MZmine 中设置算法的参数,请单击算法旁边的 [...] 按钮,然后使用 "显示预览 "选项在优化参数时可视化数据。
注意:在此阶段,算法选择的峰值将在预览窗口中以红色显示。在专有RAW文件上使用 小波变换 算法时,MZmine有时会将轮廓数据点误认为是质心峰。软件将显示一条消息,指出用户正在质心光谱上运行配置文件算法:忽略此消息并单击 确定。 - 使用原始数据方法为每个片段质量重建提取的离子淌度谱(EIM ),|特征检测|ADAP色谱图构建器 位于上一步生成的"质量"质量列表中。由于此阶段的 m/z 容差 输入是扫描到扫描容差,因此请确保使其至少比整体预期精度高 3-4 倍。
- 由于上一步没有预览选项,因此请使用 MZmine 主窗口右侧面板上显示 的特征列表 直接检查峰值拾取的质量。打开 "功能"列表,选择所有行,单击鼠标右键,然后选择" 显示/XIC(对话框)"。单击" 全部 "以显示迁移率谱上的所有离子。检查以彩色显示的选取的峰,以确保没有明显的错过峰。
- 对 EIM 进行积分,以分割在多个要素中包含不同峰的 m/z 。 使用功能列表方法|特征检测|色谱图反卷积,然后选择 小波 (ADAP) 算法。使用 "显示预览 "选项和以下关键参数优化数据算法: 信噪比阈值、系数/面积阈值和 RT 小波范围。
注:建议检查反卷积光谱的方面。使用色谱图可视化工具,如步骤 4.2.4 中所述。解卷积的峰将以彩色显示,并且应拆分相同质量的峰,如图 6A所示。 - 使用特征列表方法对非卷积的 EIM 去同位素 |同位素|同位素峰石斑鱼。使用仪器的预期精度作为 m / z 公差值,并将到达时间公差设置为0.1 ms(在MZmine中显示为 保留时间公差0.1分钟),因为在IMS分离期间不会解析同位素。检查特征列表:如果有任何同位素残留,请增加公差值。
注意:虽然理论上可以在特征列表处理的任何时刻执行去同位素,但重要的是要最后进行,以便可以导出电荷值(用于其他步骤的算法有时会删除电荷状态信息)。
- 评估数据中的噪声水平。右键单击" 原始数据文件"列表中的文件 ,选择" 显示 TIC "并显示基峰"色谱图"(BPC)。双击眼睛可观察到的最小峰值以显示其质谱。将数据中的噪声水平考虑为该光谱中基峰的第二个同位素的水平,并在以下处理步骤中对所有强度阈值使用相同的值。
- 如果处理多个 IMS/IMS-MS 光谱,请使用这些优化的参数重复处理。为所有光谱保持相同的参数。
- 在多光谱的情况下,将它们分组到一个表中以导出它们;如果没有,请直接跳到步骤 4.5。要对光谱进行分组,请使用 特征列表方法|对齐|加入矫正器。由于目标不是实际对齐峰值,因此对 m/z 和到达时间都使用限制性公差值。为两个维度赋予相同的权重。
- 将最终特征列表导出到 *.csv 文件。 使用功能列表方法|出口/进口|导出到 CSV 文件 并导出以下值:" 导出行 m/z"、"导出行保留时间 (实际 IMS 到达时间)、 峰值 m/z 和 峰值高度。使用逗号作为字段分隔符。
5. 质心IMS/IMS光谱的TWCCSN2
注意:在此协议中,将使用对数拟合校准25,26 ,这往往比线性校准提供更好的结果,并且易于在电子表格或内部处理脚本中实现。内部脚本(用R编写)可在 材料表中给出的URL上找到。
- 从校准器采集中选取参考到达时间值(请参阅步骤 2.3)。使用构造器软件手动执行此操作(请参阅 材料表)以检查所有IMS校准峰的方面。
- 在 色谱图 窗口中,打开 *_dt。与 校准器对应的 RAW 文件。
- 对于每个校准点,使用显示|生成 EIM 质量 选项。
- 检查 EIM 的配置文件。如果某些内容定义不明确,请使用 "流程"对它们进行平滑处理|平滑 选项(因为最佳结果通常使用Savitzky-Golay算法获得,在3个箱子上平滑2次)。在电子表格中报告顶点值。
注意:由于参考点通常是使用低分辨率 DTIMS 设备获取的,因此根据校准器的不同,循环 IMS 中可能会出现一些多模式分布。从校准列表中删除任何呈现此类分布的峰值。
- 根据校准器计算对数拟合参数。
- 对于所有校准点,请计算以下内容。
- 使用等式 (1) 计算漂移时间:
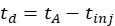 (1)
(1)
td漂移时间,tA测量的到达时间,tinj在IMS单元中的注射时间(均以毫秒为单位)。
注意:对于低聚糖片段等小分子,不同质量之间的死区时间(从IMS细胞和检测器出口之间的飞行时间)变化在CCS校准的误差范围内,可以忽略不计。 - 使用等式(2)计算离子的中性质量:
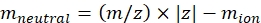 (二)
(二)
z为离子的电荷态,mion为反离子的质量(以Da为单位)。使用精确的质量以避免引入不确定性。如果存在原子损耗而不是反离子,请使用负 mion 值(例如,对于 [M-H]-mneutral = (m/z) * |z| - (- 1.007276) = (m/z) * |z| + 1.007276)。 - 使用公式 (3) 计算 CCS 参数:
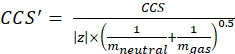 (3)
(3)
使用CCS时,参考漂移管DTCCSN2值(以nm2为单位),mgas为漂移气体的质量(以Da为单位;例如对于氮气:mgas = 28.01 Da)。 - 使用方程 (4) 计算 td' 参数:
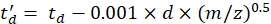 (4)
(4)
与 d 一起,检测器启动延迟通过实验用于校正死区时间(通常约为1.5 ms)。 - 计算上述参数的对数:
ln (CCS') 和 ln (t'd)
- 使用等式 (1) 计算漂移时间:
- 使用 Eq (5) 执行线性回归以确定 R2 系数以及对数拟合的 x 和 y 参数(x 为斜率,ln(y) 为截距):
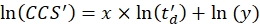 (5)
(5)
注意:用户可以绘制 ln(CCS') 与 ln(td') 值,以直观地检查校准结果,尽管这是可选的。
- 对于所有校准点,请计算以下内容。
- 将校准应用于实验数据,以校准MZmine为导出到*.csv文件的每个IMS / IMS频谱选择的峰值。对于每个点,计算如下。
- 使用等式 (6) 计算漂移时间:
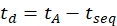 (6)
(6)
使用 tseq 表示最终 IMS 分离之前的时间(步骤 3.4.6 中记下的"时间 Abs"值)。
注:如果校准使用不同序列采集的多个 IMS/IMS 光谱,请仔细检查 tseq 值。 - 使用等式(7)计算离子的中性质量:
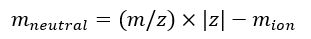 (7)
(7) - 使用均衡器 (8) 和均衡器 (9) 计算 td' 和 td'' 参数:
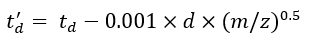 (8)
(8)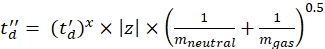 (9)
(9) - 使用等式 (10) 计算最终校准的 CCS 值(TWCCSN2,以 nm2 为单位):
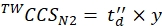 (10)
(10)
注意:虽然步骤 5.2.2.给出 ln(y) 作为截距, y 必须用于获取最终的 CCS 值。不要忘记应用指数函数。
- 使用等式 (6) 计算漂移时间:
- 通过将校准应用于步骤2.4中获取的校准溶液的第二次采集来检查校准的准确性。
注意:校准应产生误差约为1-2%的结果。
结果
选择阿拉伯木聚糖五糖XA2XX作为示例来说明该协议。该化合物是市售的,但仅作为与另一种阿拉伯木聚糖五糖XA3XX的混合物(纯XA3XX也是市售的)。XA2XX和XA3XX的结构在 补充图S1中给出。由于商业混合物中XA2XX和XA3XX的比例约为50:50,因此制备20μg/mL混合物的溶液,在50:50 MeOH/H2O + 500μM LiCl中达到约10μg/mL的XA2XX浓度。...
讨论
SELECT系列循环IMS是一个功能强大的工具,允许选择给定的 m / z 和离子迁移率的已定义离子群,而无需上游色谱分离。该仪器提供了生成该离子群的双次碎片图的可能性,可以从中提取MS / MS和IMS / IMS光谱。但是,用户必须注意在实验过程中需要注意的几个关键点。
首先,用户应仔细检查MS隔离窗口是否存在可能的等压污染物。实际上,四极杆的隔离窗口相对较宽,并且?...
披露声明
作者没有利益冲突要披露。
致谢
S.O.感谢法国国家研究机构资助他的博士学位(授予ANR-18-CE29-0006)。
材料
| Name | Company | Catalog Number | Comments |
| 33-α-L- plus 23-α-L-Arabinofuranosyl-xylotetraose (XA3XX/XA2XX) mixture | Megazyme Ltd., Wicklow, Ireland | O-XAXXMIX | XA2XX + XA3XX mixture |
| 33-α-L-Arabinofuranosyl-xylotetraose (XA3XX) | Megazyme Ltd., Wicklow, Ireland | O-XA3XX | Pure XA3XX standard |
| Eppendorf Safe-Lock Tubes, 1.5 mL, Eppendorf Quality, colorless, 1,000 tubes | Eppendorf, Hamburg, Germany | 0030120086 | Used to prepare the carbohydrate stock solution and dilution |
| FALCON 50 mL Polypropylene Conical Tube 30 x 115 mm | Corning Science México S.A. de C.V., Reynosa, Tamaulipas, Mexico | 352070 | Used to prepare the aqueous stock solution of 100 mM LiCl |
| Lithium Chloride (ACS reagent, ≥99 %) | Sigma-Aldrich Inc., Saint Quentin Fallavier, France | 310468 | Used to dope the sample with lithium |
| Major Mix IMS/Tof Calibration Kit | Waters Corp., Wilmslow, UK | 186008113 | Calibration solution for MS and IMS |
| MassLynx 4.2 SCN1016 Release 6 (Waters Embedded Analyser Platform for Cyclic IMS 2.9.1 Release 9) | Waters Corp., Wilmslow, UK | 721022377 | Cyclic IMS vendor software for instrument control and data processing |
| Methanol for HPLC PLUS Gradient grade | Carlo-Erba Reagents, Val de Reuil, France | 412383 | High-purity solvent |
| MS Leucine Enkephaline Kit | Waters Corp., Wilmslow, UK | 700002456 | Reference compound used for tuning of the mass spectrometer |
| SCHOTT DURAN 100 mL borosilicate glass bottle | VWR INTERNATIONAL, Radnor, Pennsylvania, US | 218012458 | Used to prepare the solution of 500 µM LiCl in 50:50 MeOH/Water |
| SELECT SERIES Cyclic IMS | Waters Corp., Wilmslow, UK | 186009432 | Ion mobility-mass spectrometer equipped with a cylic IMS cell |
| Website: http://mzmine.github.io/ | MZmine Development Team | - | Link to download the MZmine software |
| Website: https://github.com/siollivier/IM-MN | INRAE, UR BIA, BIBS Facility, Nantes, France | - | Link to an in-house R script containing a CCS calibration function |
参考文献
- Allard, P. -. M., et al. Integration of molecular networking and in-silico MS/MS fragmentation for natural products dereplication. Analytical Chemistry. 88 (6), 3317-3323 (2016).
- Wang, M., et al. Mass spectrometry searches using MASST. Nature Biotechnology. 38 (1), 23-26 (2020).
- David, M., Fertin, G., Rogniaux, H., Tessier, D. SpecOMS: a full open modification search method performing all-to-all spectra comparisons within minutes. Journal of Proteome Research. 16 (8), 3030-3038 (2017).
- Dührkop, K., et al. SIRIUS 4: a rapid tool for turning tandem mass spectra into metabolite structure information. Nature Methods. 16 (4), 299-302 (2019).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Nothias, L. -. F., et al. Feature-based molecular networking in the GNPS analysis environment. Nature Methods. 17 (9), 905-908 (2020).
- Gray, C. J., et al. Advancing solutions to the Carbohydrate Sequencing Challenge. Journal of the American Chemical Society. 141 (37), 14463-14479 (2019).
- Ropartz, D., et al. Online coupling of high-resolution chromatography with extreme UV photon activation tandem mass spectrometry: Application to the structural investigation of complex glycans by dissociative photoionization. Analytica Chimica Acta. 933, 1-9 (2016).
- Wolff, J. J., et al. Negative electron transfer dissociation of glycosaminoglycans. Analytical Chemistry. 82 (9), 3460-3466 (2010).
- Ropartz, D., et al. Charge transfer dissociation of complex oligosaccharides: comparison with collision-induced dissociation and extreme ultraviolet dissociative photoionization. Journal of the American Society for Mass Spectrometry. 27 (10), 1614-1619 (2016).
- Morelle, W., et al. Fragmentation characteristics of permethylated oligosaccharides using a matrix-assisted laser desorption/ionization two-stage time-of-flight (TOF/TOF) tandem mass spectrometer. Rapid Communications in Mass Spectrometry. 18 (22), 2637-2649 (2004).
- Gabelica, V., Marklund, E. Fundamentals of ion mobility spectrometry. Current Opinion in Chemical Biology. 42, 51-59 (2018).
- Gabelica, V., et al. Recommendations for reporting ion mobility mass spectrometry measurements. Mass Spectrometry Reviews. 38 (3), 291-320 (2019).
- Hernandez-Mesa, M., et al. Interlaboratory and interplatform study of steroids collision cross section by traveling wave ion mobility spectrometry. Analytical Chemistry. 92 (7), 5013-5022 (2020).
- Koeniger, S. L., et al. An IMS-IMS analogue of MS-MS. Analytical Chemistry. 78 (12), 4161-4174 (2006).
- Merenbloom, S. I., Koeniger, S. L., Valentine, S. J., Plasencia, M. D., Clemmer, D. E. IMS−IMS and IMS−IMS−IMS/MS for separating peptide and protein fragment ions. Analytical Chemistry. 78 (8), 2802-2809 (2006).
- Eldrid, C., Thalassinos, K. Developments in tandem ion mobility mass spectrometry. Biochemical Society Transactions. 48 (6), 2457-2466 (2020).
- Giles, K., et al. A cyclic ion mobility-mass spectrometry system. Analytical Chemistry. 91 (13), 8564-8573 (2019).
- Merenbloom, S. I., Glaskin, R. S., Henson, Z. B., Clemmer, D. E. High-resolution ion cyclotron mobility spectrometry. Analytical Chemistry. 81 (4), 1482-1487 (2009).
- Ollivier, S., et al. Anomeric retention of carbohydrates in multistage cyclic ion mobility (IMSn): de novo structural elucidation of enzymatically produced mannosides. Analytical Chemistry. 93 (15), 6254-6261 (2021).
- Ollivier, S., Fanuel, M., Rogniaux, H., Ropartz, D. Molecular networking of high-resolution tandem ion mobility spectra: a structurally relevant way of organizing data in glycomics. Analytical Chemistry. 93 (31), 10871-10878 (2021).
- Aron, A. T., et al. Reproducible molecular networking of untargeted mass spectrometry data using GNPS. Nature Protocols. 15 (6), 1954-1991 (2020).
- McKenna, K. R., Li, L., Krishnamurthy, R., Liotta, C. L., Fernández, F. M. Organic acid shift reagents for the discrimination of carbohydrate isobars by ion mobility-mass spectrometry. The Analyst. 145 (24), 8008-8015 (2021).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11, 395 (2010).
- Ruotolo, B. T., Benesch, J. L. P., Sandercock, A. M., Hyung, S. -. J., Robinson, C. V. Ion mobility-mass spectrometry analysis of large protein complexes. Nature Protocols. 3 (7), 1139-1152 (2008).
- Bush, M. F., Hall, Z., Giles, K., Hoyes, J., Robinson, C. V., Ruotolo, B. T. Collision cross sections of proteins and their complexes: a calibration framework and database for gas-phase structural biology. Analytical Chemistry. 82 (22), 9557-9565 (2010).
- Ropartz, D., et al. Structure determination of large isomeric oligosaccharides of natural origin through multipass and multistage cyclic traveling-wave ion mobility mass spectrometry. Analytical Chemistry. 91 (18), 12030-12037 (2019).
- Tolmachev, A. V., et al. Characterization of ion dynamics in structures for lossless ion manipulations. Analytical Chemistry. 86 (18), 9162-9168 (2014).
- Arndt, J. R., et al. High-resolution ion-mobility-enabled peptide mapping for high-throughput critical quality attribute monitoring. Journal of the American Society for Mass Spectrometry. 32 (8), 2019-2032 (2021).
- Le Fèvre, A., Dugourd, P., Chirot, F. Exploring conformational landscapes using trap and release tandem ion mobility spectrometry. Analytical Chemistry. 93 (9), 4183-4190 (2021).
- Ohshimo, K., He, X., Ito, R., Misaizu, F. Conformer separation of dibenzo-crown-ether complexes with Na+ and K+ ions studied by cryogenic ion mobility-mass spectrometry. The Journal of Physical Chemistry A. 125 (17), 3718-3725 (2021).
- Purves, R. W., Barnett, D. A., Ells, B., Guevremont, R. Gas-phase conformers of the [M + 2H]2+ ion of bradykinin investigated by combining high-field asymmetric waveform ion mobility spectrometry, hydrogen/deuterium exchange, and energy-loss measurements. Rapid Communications in Mass Spectrometry. 15 (16), 1453-1456 (2001).
- Ujma, J., et al. Cyclic ion mobility mass spectrometry distinguishes anomers and open-ring forms of pentasaccharides. Journal of the American Society for Mass Spectrometry. 30 (6), 1028-1037 (2019).
- Warnke, S., Faleh, A. B., Scutelnic, V., Rizzo, T. R. Separation and identification of glycan anomers using ultrahigh-resolution ion-mobility spectrometry and cryogenic ion spectroscopy. Journal of The American Society for Mass Spectrometry. 30 (11), 2204-2211 (2019).
- Williamson, D. L., Bergman, A. E., Nagy, G. Investigating the structure of α/β carbohydrate linkage isomers as a function of group I metal adduction and degree of polymerization as revealed by cyclic ion mobility separations. Journal of the American Society for Mass Spectrometry. 32 (10), 2573-2582 (2021).
- Myers, O. D., Sumner, S. J., Li, S., Barnes, S., Du, X. One step forward for reducing false positive and false negative compound identifications from mass spectrometry metabolomics data: new algorithms for constructing extracted ion chromatograms and detecting chromatographic peaks. Analytical Chemistry. 89 (17), 8696-8703 (2017).
- Marchand, A., Livet, S., Rosu, F., Gabelica, V. Drift tube ion mobility: how to reconstruct collision cross section distributions from arrival time distributions. Analytical Chemistry. 89 (23), 12674-12681 (2017).
- Davis, D. M., et al. Analysis of ion mobility spectra for mixed vapors using Gaussian deconvolution. Analytica Chimica Acta. 289 (3), 263-272 (1994).
- Polasky, D. A., Dixit, S. M., Fantin, S. M., Ruotolo, B. T. CIUSuite 2: next-generation software for the analysis of gas-phase protein unfolding data. Analytical Chemistry. 91 (4), 3147-3155 (2019).
- Salbo, R., et al. Traveling-wave ion mobility mass spectrometry of protein complexes: accurate calibrated collision cross-sections of human insulin oligomers. Rapid Communications in Mass Spectrometry. 26 (10), 1181-1193 (2012).
- Gelb, A. S., Jarratt, R. E., Huang, Y., Dodds, E. D. A study of calibrant selection in measurement of carbohydrate and peptide ion-neutral collision cross sections by traveling wave ion mobility spectrometry. Analytical Chemistry. 86 (22), 11396-11402 (2014).
- Richardson, K., Langridge, D., Dixit, S. M., Ruotolo, B. T. An improved calibration approach for traveling wave ion mobility spectrometry: robust, high-precision collision cross sections. Analytical Chemistry. 93 (7), 3542-3550 (2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。