È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Utilizzo di uno spettrometro ciclico per la mobilità ionica per esperimenti di mobilità ionica tandem
In questo articolo
Riepilogo
La spettrometria di mobilità ionica (IMS) è un interessante complemento alla spettrometria di massa per la caratterizzazione delle biomolecole, in particolare perché è sensibile all'isomeria. Questo protocollo descrive un esperimento IMS (IMS/IMS) in tandem, che permette l'isolamento di una molecola e la generazione dei profili di mobilità dei suoi frammenti.
Abstract
Una caratterizzazione accurata delle strutture chimiche è importante per comprenderne i meccanismi biologici sottostanti e le proprietà funzionali. La spettrometria di massa (MS) è uno strumento popolare, ma non è sempre sufficiente per svelare completamente tutte le caratteristiche strutturali. Ad esempio, sebbene i carboidrati siano biologicamente rilevanti, la loro caratterizzazione è complicata da numerosi livelli di isomeria. La spettrometria di mobilità ionica (IMS) è un complemento interessante perché è sensibile alle conformazioni ioniche e, quindi, all'isomeria.
Inoltre, i recenti progressi hanno migliorato significativamente la tecnica: l'ultima generazione di strumenti IMS ciclici offre funzionalità aggiuntive rispetto agli strumenti IMS lineari, come un maggiore potere risolutivo o la possibilità di eseguire esperimenti di mobilità ionica tandem (IMS/ IMS). Durante IMS/IMS, uno ione viene selezionato in base alla sua mobilità ionica, frammentato e rianalizzato per ottenere informazioni sulla mobilità ionica sui suoi frammenti. Lavori recenti hanno dimostrato che i profili di mobilità dei frammenti contenuti in tali dati IMS / IMS possono agire come un'impronta digitale di un particolare glicano e possono essere utilizzati in una strategia di rete molecolare per organizzare set di dati glicemici in modo strutturalmente rilevante.
L'obiettivo di questo protocollo è quindi quello di descrivere come generare dati IMS/IMS, dalla preparazione del campione alla calibrazione finale della sezione trasversale di collisione (CCS) della dimensione di mobilità ionica che produce spettri riproducibili. Prendendo l'esempio di un glicano rappresentativo, questo protocollo mostrerà come costruire una sequenza di controllo IMS / IMS su uno strumento IMS ciclico, come tenere conto di questa sequenza di controllo per tradurre il tempo di arrivo IMS in tempo di deriva (cioè il tempo di separazione effettivo applicato agli ioni) e come estrarre le informazioni di mobilità rilevanti dai dati grezzi. Questo protocollo è progettato per spiegare chiaramente i punti critici di un esperimento IMS/IMS e quindi aiutare i nuovi utenti IMS ciclici a eseguire acquisizioni semplici e riproducibili.
Introduzione
La caratterizzazione chimica completa delle biomolecole è fondamentale per comprendere le loro proprietà biologiche e funzionali sottostanti. A tal fine, le scienze "omiche" si sono sviluppate negli ultimi anni, mirando alla caratterizzazione su larga scala di strutture chimiche a concentrazioni biologiche. In proteomica e metabolomica, la SM è diventata uno strumento fondamentale per svelare l'eterogeneità strutturale che si trova nei mezzi biologici, in particolare grazie alla sua sensibilità e capacità di fornire informazioni strutturali attraverso la SM tandem (MS / MS). Nelle strategie MS/MS, uno ione viene selezionato in base alla sua massa, quindi frammentato e, infine, le masse dei suoi frammenti vengono acquisite per stabilire un'impronta digitale della molecola. Gli spettri MS/MS possono, in particolare, essere utilizzati per abbinare database spettrali1,2 o ricostruire provvisoriamente le strutture madri3,4. Nell'ipotesi che spettri simili appartengano a composti simili, i dati MS/MS possono anche essere utilizzati per costruire reti molecolari (MN) che collegano specie correlate attraverso un punteggio di somiglianza5,6.
Tuttavia, a causa della proprietà intrinseca della SM di rilevare il rapporto massa-carica (m / z) degli ioni, la tecnica è cieca a una serie di caratteristiche strutturali che rientrano nell'intervallo dell'isomeria (stereo). Ad esempio, i carboidrati sono costituiti da diverse subunità monosaccaridiche, molte delle quali sono stereoisomeri o addirittura epimeri (ad esempio, Glc vs. Gal o Glc vs. Man). Queste subunità sono collegate da legami glicosidici, che possono differire per la posizione del legame (regioisomerismo) e la configurazione sterica del carbonio anomerico (anomerismo). Queste caratteristiche rendono difficile per la SM autonoma distinguere tra isomeri di carboidrati7 e solo il regioisomerismo può essere affrontato utilizzando metodi di attivazione ad alta energia8,9,10. Sebbene la derivatizzazione sia un'opzione per interrompere l'equivalenza dei gruppi stereoisomerici11, richiede un'ampia preparazione del campione. Un'altra opzione più semplice è quella di accoppiare la SM con una dimensione analitica sensibile all'isomeria, come l'IMS.
Poiché questo protocollo è progettato per gli utenti che hanno già familiarità con i concetti di base di IMS e poiché le recensioni dettagliate sono disponibili altrove12,13, qui viene fornita solo una breve panoramica dei principi di IMS. IMS è un metodo di separazione in fase gassosa che si basa sull'interazione di ioni con un gas tampone e un campo elettrico, separando infine gli ioni in base alle loro conformazioni in fase gassosa. Diversi principi di IMS accoppiati a MS possono essere trovati su strumenti commerciali: alcuni operano alternando campi elettrici alti e bassi (IMS asimmetrico di campo, FAIMS), mentre la maggior parte opera entro il limite di campo basso, in particolare il tubo di deriva IMS (DTIMS, campo elettrico decrescente lineare), l'onda mobile IMS (TWIMS, onde di potenziale simmetrico) e l'IMS intrappolato (TIMS, alto flusso di ioni di intrappolamento del gas tampone contro i campi elettrici)13 . I metodi a basso campo consentono l'accesso a un cosiddetto CCS, una proprietà della coppia ione-gas che rappresenta la superficie (in Å2 o nm2) dello ione che interagisce con il gas tampone durante la separazione. La CCS è teoricamente indipendente dallo strumento ed è quindi utile per generare dati riproducibili tra diversi laboratori14. Le separazioni di mobilità ionica possono essere influenzate da vari parametri e, in particolare, dalle fluttuazioni della pressione del gas e della temperatura del gas nella cella di mobilità. La calibrazione CCS è un modo per rimediare a questo, poiché sia il calibrante che la specie di interesse saranno influenzati in modo simile13. Tuttavia, è obbligatorio installare lo strumento in una stanza a temperatura controllata e disporre di un sistema di controllo della pressione del gas affidabile.
Un'evoluzione interessante dell'IMS è IMS/IMS, che è stato introdotto per la prima volta nel 2006 dal gruppo di Clemmer come analogo di MS/MS15,16. In IMS/IMS, uno ione di interesse è isolato selettivamente in base alla sua mobilità ionica; viene quindi attivato (fino a possibile frammentazione) e viene eseguita una nuova analisi IMS dello ione o dei frammenti attivati. Nel primo progetto strumentale, due celle IMS sono state messe in serie, separate da un imbuto ionico dove si trovava l'attivazione. Da allora, sebbene siano state proposte diverse configurazioni IMS/ IMS (per una revisione, vedi Eldrid e Thalassinos17), il primo spettrometro di massa commerciale con capacità IMS / IMS è diventato disponibile solo nel 201918. Questo strumento ha sostanzialmente migliorato il concetto iniziale combinandolo con un'altra svolta tecnologica: un design ciclico della cella IMS.
La cella IMS ciclica consente teoricamente di aumentare quasi infinitamente la lunghezza del percorso di deriva e, quindi, il potere risolutivo dello strumento19. Ciò è stato ottenuto per mezzo di una particolare geometria dello strumento, in cui la cella ciclica TWIMS è posizionata ortogonalmente all'asse ottico ionico principale. Una regione di matrice multifunzione all'ingresso della cella IMS consente di controllare la direzione del percorso ionico: (i) inviare ioni lateralmente per la separazione IMS, (ii) in avanti per il rilevamento MS o (iii) indietro dalla cella IMS per essere memorizzati in una cella prearray. Da questa cella di conservazione prearray, gli ioni possono essere attivati e i frammenti reiniettati nella cella IMS per la misurazione della mobilità ionica, un approccio che è stato utilizzato con successo per caratterizzare gli stereoisomeri20. In definitiva, i dati raccolti contengono mobilità ionica e informazioni m/z per il precursore e i suoi frammenti.
In una recente pubblicazione che ha utilizzato questo disegno ciclico per le analisi dei glicani (Ollivier et al.21), abbiamo dimostrato che il profilo di mobilità dei frammenti contenuti in tali dati IMS / IMS agisce come un'impronta digitale di una biomolecola che può essere utilizzata in una strategia di rete molecolare. La rete risultante, chiamata IM-MN, ha portato all'organizzazione di set di dati glicemici in modo strutturalmente rilevante, mentre la rete costruita esclusivamente da dati MS / MS (MS-MN) ha rivelato poche informazioni. Per completare questa pubblicazione e aiutare gli utenti di Cyclic IMS a implementare questo flusso di lavoro, questo protocollo fornisce una descrizione completa del protocollo utilizzato per raccogliere i dati. Questo protocollo si concentra solo sulla generazione dei dati IMS/IMS che gli utenti possono quindi utilizzare per costruire reti IM-MN (vedi21) o per qualsiasi altra applicazione di loro scelta. La costruzione di IM-MN non sarà presa in considerazione nel presente documento, poiché i protocolli per il networking molecolare sono già disponibili22. Vengono evidenziati i punti cruciali che devono essere seguiti per generare acquisizioni IMS/IMS preziose e riproducibili. Prendendo l'esempio di uno degli oligosaccaridi studiati da Ollivier et al. 21, i seguenti passaggi sono dettagliati: (i) preparazione del campione, (ii) messa a punto dello strumento IMS ciclico, (iii) peak-picking automatizzato dei dati e (iv) calibrazione CCS.
Protocollo
NOTA: una panoramica del protocollo è fornita nella Figura 1. I parametri utilizzati per gli esperimenti descritti nel presente protocollo sono riportati nella Tabella supplementare S1 e nella Tabella supplementare S2.
1. Preparazione della soluzione campione
NOTA: Il protocollo è descritto utilizzando un pentasaccaride arabinoxilano (23-α-L-arabinofuranosil-xilotetraosio o XA2XX; vedere la Tabella dei materiali) come esempio.
- Preparazione del solvente: 500 μM LiCl in 50:50 H2O:MeOH (vol./vol.).
- Preparare una soluzione madre da 100 mM di cloruro di litio (LiCl) in H2O pesando 212 mg di LiCl e aggiungere 50 mL di acqua deionizzata ad alta purezza (H2O) in un tubo conico in polipropilene da 50 mL. Agitare fino a completa dissoluzione.
NOTA: Il solvente viene drogato con un sale di litio per promuovere la formazione di addotti [M + Li] + nella fonte ionica dello spettrometro, poiché di solito produce spettri di frammentazione di migliore qualità rispetto ad altri addotti alcalini. L'uso di LiCl è raccomandato perché gli acidi organici (e quindi i loro sali) hanno precedentemente un impatto sui profili IMS23. - In una bottiglia di vetro, diluire la soluzione madre LiCl 200x: a 250 μL della soluzione madre, aggiungere 24,75 mL di H2O. Aggiungere 25 mL di metanolo (MeOH) per raggiungere una concentrazione finale di LiCl di 500 μM in 50:50 H2O:MeOH (v/v). Sonicare per 2 minuti per degasare il solvente.
NOTA: MeOH presenta un pericolo per la salute (H225, H301, H311, H331, H370); manipolare sotto un cappuccio estrattore indossando un cappotto da laboratorio, guanti e protezione per gli occhi. Una proporzione di 50:50 MeOH/H2O (v/v) sembra essere il miglior solvente per la ionizzazione degli oligosaccaridi; tuttavia, MeOH può essere sostituito da acetonitrile (ACN) se necessario.
- Preparare una soluzione madre da 100 mM di cloruro di litio (LiCl) in H2O pesando 212 mg di LiCl e aggiungere 50 mL di acqua deionizzata ad alta purezza (H2O) in un tubo conico in polipropilene da 50 mL. Agitare fino a completa dissoluzione.
- In un tubo di polipropilene da 1,5 ml, pesare 1 mg di carboidrati. Sciogliere con un volume appropriato di 500 μM LiCl per raggiungere una concentrazione di 1 mg/mL. Diluire fino a una concentrazione finale di 10 μg/mL in 50:50 MeOH/H2O + 500 μM LiCl. Conservare a 4 °C.
NOTA: La concentrazione di 10 μg/mL è stata scelta per ottimizzare il segnale su tutti gli ioni frammento durante IMS/IMS-MS (questo è per un composto puro; aumentare la concentrazione quando si lavora su miscele). Per l'acquisizione degli spettri IMS/IMS di riferimento, non diluire ulteriormente il campione: è prevista la saturazione del rivelatore MS prima della frammentazione, sebbene lo strumento offra opzioni per correggerlo (vedere il punto 3.2.).
2. Messa a punto dello spettrometro di massa IMS ciclico
NOTA: le istruzioni relative al software (finestre, menu e comandi) sono evidenziate in grassetto.
- Aprire la console dello strumento dal software di controllo dello strumento (pagina MS tune , vedere i dettagli del software nella Tabella dei materiali) e mettere lo strumento in modalità Operativa . Attendere almeno 3 ore affinché le alte tensioni si stabilizzino nella cella IMS.
NOTA: Per la migliore riproducibilità, le tensioni nella cella IMS devono essere completamente stabilizzate. Accendere le alte tensioni e lasciare che lo strumento si stabilizzi durante la notte prima di qualsiasi analisi IMS ciclica. Inoltre, la pressione e la temperatura nella cella di mobilità ionica devono essere mantenute il più costanti possibile. Sebbene nella scheda Vuoto sia disponibile un readback per la pressione, non è disponibile alcun readback per la temperatura. Conservare lo strumento in un laboratorio termostatato. Lo strumento utilizzato in questo lavoro funziona a 1,75 mbar in un laboratorio termostatato a 20 °C. - Configurazione ciclica dello strumento IMS
NOTA: le soluzioni standard devono essere infuse utilizzando il sistema fluidico integrato per la configurazione dello strumento.- Posizionare i contenitori di fluidica riempiti con le norme appropriate fornite dal produttore sul sistema di fluidica: Serbatoio B ("Lockmass"): 10 pg/μL di leucina encefalina (LEU ENK) in 50:50 ACN/H2O + 0,1% di acido formico; Serbatoio C ('Calibrante'): MajorMix.
NOTA: in questo protocollo, la soluzione di calibrazione MajorMix verrà utilizzata per calibrare sia le dimensioni m/z che CCS. Per motivi pratici, verrà eseguita una calibrazione CCS esterna (vedi step 5 del protocollo); quindi, è anche possibile utilizzare una miscela di calibrante interna per il CCS e un altro calibrante per il m/z (ad esempio, formiato di sodio o ioduro di sodio). - Nella pagina Sintonizzazione della console Quartz, vai alla scheda Fluidics . Impostare la fluidica del campione sul serbatoio C e la fluidica di riferimento sul serbatoio B. Infondere entrambe le soluzioni consecutivamente nella sorgente ionica per controllare il segnale MS.
- Eseguire la configurazione ADC, la configurazione del rilevatore (utilizzando LEU ENK) e la calibrazione della massa (vedere la Tabella dei materiali per la soluzione di calibrazione) dalla pagina Configurazione dello strumento in base alle istruzioni del produttore.
- Posizionare i contenitori di fluidica riempiti con le norme appropriate fornite dal produttore sul sistema di fluidica: Serbatoio B ("Lockmass"): 10 pg/μL di leucina encefalina (LEU ENK) in 50:50 ACN/H2O + 0,1% di acido formico; Serbatoio C ('Calibrante'): MajorMix.
- Registrare un'acquisizione IMS della soluzione di calibrazione con una separazione a singolo passaggio (utilizzarla per la calibrazione IMS esterna).
NOTA: La sorgente di ioni e i parametri dell'onda mobile (TW) (altezza dell'onda statica e velocità dell'onda) devono essere mantenuti costanti durante tutte le acquisizioni (calibrazione e acquisizioni). Se l'utente non ha una conoscenza preliminare dei parametri ottimali per il suo campione, questo passaggio può essere eseguito dopo la fase 3 del protocollo (per gli addotti [M + Li] + di oligosaccaridi neutri, i risultati rappresentativi utilizzano un'altezza TW di 16 V e una velocità TW di 350 m / s, che danno i migliori risultati).- Dalla scheda Fluidica , selezionare la posizione del deflettore Campione e infondere il calibrante (vedere la Tabella dei materiali) nella sorgente di ioni (utilizzando il sistema fluidico integrato) attraverso la sonda "Campione" a una portata di 10 μL/min.
- Impostare una sequenza IMS a passaggio singolo. Dalla pagina Tune , metti lo strumento in modalità Mobility e apri la finestra Cyclic Sequence Control . Seleziona Modalità avanzata . Dalla scheda Funzioni cicliche di questa nuova finestra, selezionare Aggiungi bundle, quindi Single/Multipass. Attendere che una sequenza di eventi per dispositivi mobili venga visualizzata nella scheda Sequenza della stessa finestra.
NOTA: Per attivare la visualizzazione in tempo reale, l'utente deve applicare i parametri dello strumento: fare clic su Sintonizza in modalità TOF o Esegui in modalità Mobilità . Prima di passare lo strumento tra le modalità TOF e Mobility , è necessario interrompere qualsiasi acquisizione in esecuzione (inclusa la visualizzazione della pagina Tune ). L'abbondanza relativa di ioni può variare tra la modalità TOF e la modalità Mobility a causa dei cambiamenti nei parametri di trasmissione degli ioni. - Adattare la sequenza in modo che tutti gli ioni calibranti facciano un singolo passaggio intorno alla pista IMS ciclica. Non modificare il tempo di iniezione o l'espulsione e acquisire tempo; tuttavia, abbassare il tempo separato a 1 ms (nella scheda Sequenza ). Se alcuni ioni della miscela di calibrazione non rientrano nella finestra temporale di arrivo visualizzata, modificare la sincronizzazione dell'IMS con il pusher dell'analizzatore TOF di accelerazione ortogonale aumentando il numero di push per bin nella scheda Impostazioni ADC .
NOTA: i tempi nella sequenza di controllo controllano solo l'array multifunzione per il gating ionico. Finché gli ioni sono impegnati nel loro primo (o ennesimo) passaggio intorno alla pista, finiranno detto passaggio anche se la direzione del TW è cambiata nell'array nel frattempo. Abbassando il tempo di separazione a 1 ms significa che l'array passerà alla modalità di espulsione dopo 1 ms. Ciò garantisce che gli ioni più veloci non avranno abbastanza tempo per passare attraverso l'array e impegnarsi in un secondo passaggio prima che gli ioni più lenti finiscano il loro primo passaggio. Pertanto, tutti gli ioni saranno sottoposti allo stesso numero di passaggi (cioè un passaggio), necessario per eseguire la calibrazione IMS. - Registra un'acquisizione di 2 minuti. Nella finestra Controllo sequenza ciclica , fare clic su Acquisisci per aprire la finestra popup Impostazioni acquisizione . Immettere il nome file, la descrizione e la durata dell'acquisizione (min) e fare clic su Salva.
- Registrare un'altra acquisizione di 2 minuti della soluzione di calibrazione nelle stesse condizioni del passaggio 2.3 (utilizzare questa opzione per verificare la qualità della calibrazione CCS). Nella finestra Controllo sequenza ciclica , fare clic su Acquisisci per aprire la finestra popup Impostazioni acquisizione . Immettere il nome file, la descrizione e la durata dell'acquisizione (min) e fare clic su Salva.
- Lavare accuratamente il sistema fluidico con 50:50 H2O/ACN per evitare la cristallizzazione del calibrante nel tubo peek.
3. Acquisizione IMS/IMS-MS
- Utilizzando una pompa a siringa, infondere il campione (drogato al litio) a 10 μg/mL attraverso la sonda campione ad una portata di 10 μL/min.
- Passare lo strumento alla modalità TOF (dalla pagina MS tune ) per verificare la stabilità del segnale. Registrare un'acquisizione completa della SM (1 min) del campione, che sarà utile per verificare il pattern isotopico e la presenza di potenziali contaminanti.
NOTA: Poiché la concentrazione del campione viene scelta per ottenere un buon segnale ionico per i frammenti, in questa fase si può osservare una saturazione TOF. La saturazione DEL TOF può essere identificata utilizzando i seguenti artefatti: (i) una risoluzione MS aumentata artificialmente, (ii) un cambiamento nei rapporti isotopici e (iii) una moltitudine di picchi a bassa abbondanza tra gli isotopi. Utilizzare l'obiettivo DRE (miglioramento della gamma dinamica, scheda Quad/MS Profile/DRE della pagina tune principale) per attenuare la trasmissione degli ioni ed eliminare la saturazione in modalità TOF (Figura 2A,B). - Metti lo strumento in modalità MSMS (scheda Quad/MS Profile della pagina principale Tune ) e seleziona la massa dello ione bersaglio nel campo MSMS Mass per l'isolamento nel quadrupolo (nell'esempio: m/z di 685.2, corrispondente alla specie ionica [M+Li]+ del pentasaccaride arabinoxilano). Registrare un'acquisizione di 1 minuto per verificare l'isolamento del precursore durante l'elaborazione dei dati.
NOTA: gli addotti di litio hanno un isotopo a -1 Da del picco monoisotopico, che deve essere rimosso dalla finestra di selezione MS/MS in modo che non interferisca con le fasi di elaborazione. Può essere rimosso restringendo l'intervallo di selezione utilizzando i parametri Risoluzione LM e Risoluzione HM nella scheda Profilo Quad/MS (Figura 2C). - Impostare una sequenza IMS di "slicing" per eseguire una selezione basata sulla mobilità dell'isomero di interesse.
- Passare lo strumento alla modalità Mobilità (vedere il punto 2.3.2). Nella finestra Controllo sequenza ciclica , dalla scheda Funzioni cicliche , selezionare Aggiungi bundle e quindi Slicing. Attendere che nella scheda Sequenza venga visualizzata una sequenza complessa di eventi di mobilità (Figura 3).
NOTA: è possibile visualizzare ogni fase del processo IMS/IMS: fare clic sull'evento Eject and Acquire nella scheda Sequenza . Una volta evidenziato in rosso, spostatelo nella posizione appropriata all'interno della sequenza utilizzando i pulsanti Su e Giù . - Posizionare l'evento Eject and Acquire subito dopo il primo evento Separate (ovvero spostarlo alla riga 3 anziché alla riga 8 nella sequenza come mostrato nella Figura 3), quindi fare clic su Esegui. Cerca i risultati della separazione iniziale da visualizzare in tempo reale. Aumentare la durata del primo evento separato per una separazione multipass modificando il valore temporale per questo evento nella sequenza fino a quando la risoluzione dei picchi IMS non è soddisfacente. Registrare un'acquisizione di 1 minuto come riferimento.
NOTA: Prendere nota del valore ADC Start Delay nella scheda ADC Setup : sarà utile verificare la qualità dell'isolamento. - Fai clic su Pausa. Si noti che vengono visualizzati i risultati della separazione iniziale, anche se le modifiche nella sequenza di controllo non verranno applicate finché l'utente non fa nuovamente clic su Esegui . Posizionate l'evento Eject and Acquire sotto gli eventi Eject, Eject to Pre-Store e Hold e Eject . Regolare la durata degli eventi in modo che il picco di destinazione si trovi nell'area Espelli in pre-archiviazione e qualsiasi altro ione si trovi nell'area Espelli o Blocca ed espelli .
NOTA: la durata di questi tre eventi rispetto alle distribuzioni del tempo di arrivo (ATD) può essere visualizzata utilizzando la barra codificata a colori sotto lo spettro di mobilità nella scheda Mobilogramma (Figura 3). - Posizionate l'evento Eject and Acquire alla fine della sequenza, sotto gli eventi Reinject from Pre-Store e i secondi separate . Fare clic su Esegui per visualizzare la popolazione selezionata.
NOTA: poiché la popolazione selezionata ha lasciato la cella IMS, tutta la separazione precedente è stata persa ed è tornata a una separazione a passaggio singolo (desiderata). - Controllare la qualità dell'isolamento. Per verificare che sia stato selezionato solo il picco di interesse, eseguire la stessa separazione dopo la reiniezione di prima della reiniezione (cioè lo stesso tempo separato ) come mostrato nella Figura 4. Registrare un'acquisizione di 1 minuto come riferimento.
NOTA: Gli utenti sono incoraggiati a controllare la popolazione espulsa; la finestra temporale Espulsione in pre-archiviazione deve essere di livello basale (Figura 4B). Per verificarlo, inserire il ritardo di avvio ADC in modalità manuale nella scheda Impostazioni ADC e immettere il tempo di ritardo indicato nel passaggio 3.4.2. Registrare un'acquisizione di 1 minuto come riferimento. - Nella scheda Sequenza , nella colonna accanto agli orari degli eventi definiti dall'utente (la colonna Time Abs , evidenziata in rosso), cerca i tempi sommati di tutti gli eventi. Prendi nota del Time Abs trovato sulla linea dell'evento Reinject from Pre-Store per eseguire la calibrazione CCS.
- Passare lo strumento alla modalità Mobilità (vedere il punto 2.3.2). Nella finestra Controllo sequenza ciclica , dalla scheda Funzioni cicliche , selezionare Aggiungi bundle e quindi Slicing. Attendere che nella scheda Sequenza venga visualizzata una sequenza complessa di eventi di mobilità (Figura 3).
- Frammentare il picco di destinazione tra i due round di IMS. Modificare le tensioni della fase di reiniezione per aumentare l'energia cinetica degli ioni e frammentarli in caso di collisione con il gas di mobilità ionica.
- Impostare la durata dell'evento Separate che precede direttamente Eject and Acquire su 1 ms (vedere la spiegazione nel passaggio 2.3.3).
- Nella riga Reinject from Pre-Store selezionare la casella Abilita attivazione e ottimizzare la frammentazione con il controllo incorporato. Se lo spettro è soddisfacente (ad esempio, il picco di base è un frammento), procedere direttamente al passaggio 3.5.4.
NOTA: Quando si abilita l'attivazione, tre tensioni sulla linea diventano grigie: queste sono le tensioni che l'utente deve modificare se è richiesta l'ottimizzazione manuale delle tensioni (vedere il passaggio successivo). Queste tre tensioni (Pre-Array Gradient, Pre-Array Bias e Array Offset) formano il gradiente utilizzato per attivare gli ioni. L'energia cinetica degli ioni aumenterà con la pendenza tra il Pre-Array Bias e l'Array Offset (vedere la Figura 5). I valori predefiniti dei valori gradiente → bias → offset sono: senza attivazione 85 → 70 → 45 V; attivazione massima della funzione integrata 185 → 170 → -5 V (+150 V). Dopo la frammentazione, non dimenticare di riadattare la trasmissione ionica usando la lente DRE (diminuire l'attenuazione del segnale) (vedere il punto 3.2.). - Se la frammentazione non è soddisfacente con il controllo integrato, deselezionare la casella Abilita attivazione e procedere all'ottimizzazione manuale delle tensioni di reiniezione. Aumentare la tensione del gradiente pre-array (la tensione di polarizzazione pre-array deve essere mantenuta sempre 15 V al di sotto del gradiente pre-array) e abbassare la tensione di offset dell'array (che può essere impostata come negativa) fino a quando i risultati non sono soddisfacenti.
NOTA: quando si sintonizzano manualmente le tensioni dell'array multifunzione, l'utente può passare dalla vista 'Mobilogram' a schemi interattivi delle tensioni applicate nell'array multifunzione (diagramma PE) per visualizzare meglio le impostazioni di tensione (Figura 5A). - Registra un'acquisizione di 2 minuti. Nella finestra pop-up di acquisizione, selezionare l'opzione Mantieni tempo di deriva per generare un file contenente solo i tempi di arrivo rispetto a m/z (il tempo di acquisizione utilizzato per le analisi cromatografiche - il tempo di ritenzione - viene rimosso dal file). Si noti che questo file è etichettato con *_dt. CRUDO.
NOTA: se l'utente dimentica di selezionare l'opzione Mantieni tempo di deriva , è comunque possibile estrarre la quota IMS utilizzando il software Driftscope 2.9 (File | Esporta in MassLynx | Mantieni il tempo di deriva).
- Riportare lo strumento in modalità TOF nella pagina principale della melodia e risciacquare accuratamente il sistema con 50:50 MeOH/H2O prima di procedere con il campione successivo.
4. Elaborazione IMS/IMS-MS con MZmine 224
NOTA: MZmine 2 è disponibile dall'URL indicato nella Tabella dei materiali. Si raccomanda l'uso di MZmine 2.51. Al momento della preparazione di questo manoscritto, le versioni successive non possono aprire file RAW da strumenti IMS ciclici a causa di un cambiamento nella funzione di importazione.
- Importate i file raw contenenti solo le dimensioni IMS e m/z (*_dt. RAW) utilizzando metodi di dati grezzi | Importazione di dati grezzi.
NOTA: i file raw appariranno sul lato sinistro della finestra principale di MZmine. Non importare l'originale *. File RAW che contengono ancora la dimensione del tempo di conservazione. MZmine non distingue il tempo di ritenzione dall'ora di arrivo IMS e i punti dati di entrambe le dimensioni si sovrapporrebbero. - Ottimizzare i parametri del flusso di lavoro in un file rappresentativo selezionandolo nella casella File di dati grezzi lista.
- Valutare il livello di rumore nei dati. Fare clic con il pulsante destro del mouse sul file nell'elenco File di dati grezzi, selezionare Mostra TIC e visualizzare il picco di base "cromatogramma" (BPC). Fare doppio clic sul picco più piccolo osservabile a occhio per visualizzarne lo spettro di massa. Considerare che il livello di rumore nei dati è intorno a quello del secondo isotopo del picco di base in questo spettro e utilizzare lo stesso valore per tutte le soglie di intensità nelle seguenti fasi di elaborazione.
NOTA: I dati sono stati acquisiti utilizzando l'isolamento del quadrupolo e sono quindi considerati da MZmine come MS/MS. Durante l'intera elaborazione di MZmine, assicurarsi di lavorare a un livello MS = 2. - Eseguire il rilevamento di massa utilizzando i metodi dei dati grezzi | | di rilevamento delle funzionalità Rilevamento di massa. Per i dati acquisiti in modalità profilo, utilizzare l'algoritmo di trasformazione Wavelet . Per impostare i parametri degli algoritmi in MZmine, fare clic sul pulsante [...] accanto all'algoritmo e utilizzare l'opzione Mostra anteprima per visualizzare i dati ottimizzando i parametri.
NOTA: in questa fase, i picchi selezionati dall'algoritmo appariranno in rosso nella finestra di anteprima. Quando si utilizza l'algoritmo di trasformazione wavelet su file RAW proprietari, MZmine a volte confonde i punti dati del profilo per picchi centroidi. Il software visualizzerà un messaggio che indica che l'utente sta eseguendo un algoritmo di profilo su spettri centrati: ignora questo messaggio e fai clic su OK. - Ricostruire gli spettri di mobilità ionica estratti (EIM) per ogni massa di frammento utilizzando metodi di dati grezzi | | di rilevamento delle funzionalità Costruttore di cromatogrammi ADAP nell'elenco di massa "masse" generato dal passaggio precedente. Poiché l'input di tolleranza m/z in questa fase è una tolleranza scan-to-scan, assicurarsi di lasciarlo almeno 3-4 volte superiore alla precisione complessiva prevista.
- Poiché il passaggio precedente non dispone di un'opzione di anteprima, controllare direttamente la qualità del picco di prelievo utilizzando l'elenco Funzionalità visualizzato nel pannello di destra della finestra principale di MZmine. Aprire l'elenco Feature, selezionare tutte le righe, fare clic con il pulsante destro del mouse e scegliere Mostra/XIC (finestra di dialogo). Fare clic su Tutto per visualizzare tutti gli ioni nello spettro di mobilità. Ispeziona i picchi scelti che appaiono a colori per assicurarti che non ci siano picchi mancati evidenti.
- Deconvolvere gli EIM per dividere gli m/z che contengono picchi diversi in più feature. Utilizzare i metodi dell'elenco delle funzionalità | | di rilevamento delle funzionalità Deconvoluzione del cromatogramma e scegliere l'algoritmo Wavelets (ADAP ). Ottimizzare l'algoritmo per i dati utilizzando l'opzione Mostra anteprima e i seguenti parametri chiave: soglia S/N, soglia coefficiente/area e intervallo wavelet RT.
NOTA: si consiglia di controllare l'aspetto dello spettro deconvolto. Utilizzare lo strumento di visualizzazione del cromatogramma, come descritto nel passaggio 4.2.4. I picchi deconvolti appariranno a colori e i picchi della stessa massa dovrebbero essere divisi, come presentato nella Figura 6A. - Deisotope gli EIM deconvoluti utilizzando i metodi dell'elenco delle funzionalità | Isotopi | Picchi isotopici cernia. Utilizzare la precisione prevista dello strumento per il valore di tolleranza m/z e impostare la tolleranza del tempo di arrivo su 0,1 ms (visualizzata in MZmine come Tolleranza del tempo di ritenzione 0,1 min), poiché gli isotopi non vengono risolti durante la separazione IMS. Controllare l'elenco delle caratteristiche: se rimangono degli isotopi, aumentare i valori di tolleranza.
NOTA: sebbene il deisotoping possa teoricamente essere eseguito in qualsiasi momento dell'elaborazione dell'elenco delle funzionalità, è importante farlo per ultimo in modo che i valori di carica possano essere esportati (gli algoritmi utilizzati per gli altri passaggi a volte rimuovono le informazioni sullo stato di carica).
- Valutare il livello di rumore nei dati. Fare clic con il pulsante destro del mouse sul file nell'elenco File di dati grezzi, selezionare Mostra TIC e visualizzare il picco di base "cromatogramma" (BPC). Fare doppio clic sul picco più piccolo osservabile a occhio per visualizzarne lo spettro di massa. Considerare che il livello di rumore nei dati è intorno a quello del secondo isotopo del picco di base in questo spettro e utilizzare lo stesso valore per tutte le soglie di intensità nelle seguenti fasi di elaborazione.
- Se si elaborano più spettri IMS/IMS-MS, ripetere l'elaborazione con questi parametri ottimizzati. Mantenere gli stessi parametri per tutti gli spettri.
- Nel caso di spettri multipli, raggrupparli in un'unica tabella per esportarli; in caso contrario, andare direttamente al passaggio 4.5. Per raggruppare gli spettri, utilizzare i metodi dell'elenco delle caratteristiche | Allineamento | Join aligner. Poiché l'obiettivo non è quello di allineare effettivamente i picchi, utilizzare valori di tolleranza restrittivi sia per m/z che per l'ora di arrivo. Dare lo stesso peso a entrambe le dimensioni.
- Esportare l'elenco delle funzionalità finale in un file *.csv . Utilizzare i metodi dell'elenco delle funzionalità | | di esportazione/importazione Esporta in file CSV ed esporta i seguenti valori: Esporta riga m/z, Esporta tempo di conservazione riga (l'ora di arrivo IMS effettiva), Picco m/z e Altezza picco. Utilizzare una virgola come separatore di campo.
5. TWCCSN2 degli spettri IMS/IMS centroidi
NOTA: In questo protocollo verrà utilizzata una calibrazione logaritmica di adattamento25,26, che tende a dare risultati migliori rispetto alla calibrazione lineare ed è facile da implementare in un foglio di calcolo o in uno script di elaborazione interno. Uno script interno (scritto in R) è disponibile all'URL indicato nella Tabella dei materiali.
- Selezionare i valori del tempo di arrivo di riferimento dall'acquisizione del calibrante (vedere il passaggio 2.3). Fallo manualmente utilizzando il software di costruzione (vedi la Tabella dei materiali) per verificare l'aspetto di tutti i picchi calibranti IMS.
- Nella finestra Cromatogramma , aprire la *_dt. RAW corrispondente al calibrante.
- Per ogni punto di calibrazione, generare l'EIM utilizzando il display | Opzione di massa .
- Controllare il profilo degli EIM. Se alcuni sono mal definiti, attenuarli utilizzando il processo | Opzione liscia (poiché i migliori risultati si ottengono in genere con l'algoritmo Savitzky-Golay, liscio 2 volte su 3 contenitori). Segnala i valori apex in un foglio di calcolo.
NOTA: poiché i punti di riferimento vengono generalmente acquisiti utilizzando dispositivi DTIMS a bassa risoluzione, alcune distribuzioni multimodali possono apparire in IMS ciclico a seconda dei calibranti. Rimuovere qualsiasi picco che presenti tale distribuzione dall'elenco di calibrazione.
- Calcolare i parametri di adattamento logaritmico dai calibranti.
- Per tutti i punti di calibrazione, calcolare quanto segue.
- Calcola il tempo di deriva usando Eq (1):
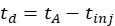 (1)
(1)
con td il tempo di deriva, tA il tempo di arrivo misurato e tinj il tempo di iniezione nella cella IMS (tutto in ms).
NOTA: Per piccole molecole, come i frammenti di oligosaccaridi, la variazione del tempo morto (tempo di volo tra l'uscita dalla cella IMS e dal rivelatore) tra masse diverse rientra nell'intervallo di errore della calibrazione CCS e può essere ignorata. - Calcola la massa neutra degli ioni usando Eq (2):
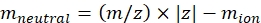 (2)
(2)
con z lo stato di carica dello ione e mion la massa del controione (in Da). Usa masse esatte per evitare di introdurre incertezza. Se c'è una perdita di atomo invece di un controione, usa valori di mioni negativi (ad esempio, per [M-H]-mneutral = (m/z) * |z| - (- 1.007276) = (m/z) * |z| + 1.007276). - Calcola il parametro CCS usando Eq (3):
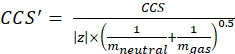 (3)
(3)
con CCS il valore DTCCSN2 del tubo di deriva di riferimento (in nm2), e mgas la massa del gas di deriva (in Da; es. per l'azoto: mgas = 28,01 Da). - Calcola il parametro td' usando Eq (4):
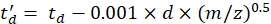 (4)
(4)
con d il ritardo di avvio del rivelatore utilizzato sperimentalmente per correggere il tempo morto (in genere ~ 1,5 ms). - Calcola il logaritmo dei parametri di cui sopra:
ln (CCS') e ln (t'd)
- Calcola il tempo di deriva usando Eq (1):
- Eseguire una regressione lineare per determinare il coefficiente R2 e i parametri x e y dell'adattamento logaritmico (con x la pendenza e ln(y) l'intercetta ) usando Eq (5):
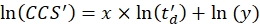 (5)
(5)
NOTA: l'utente può tracciare i valori ln(CCS') vs ln(td') per controllare visivamente i risultati della calibrazione, anche se questo è facoltativo.
- Per tutti i punti di calibrazione, calcolare quanto segue.
- Applicare la calibrazione ai dati sperimentali per calibrare i picchi selezionati da MZmine per ogni spettro IMS/IMS esportato nel file *.csv. Per ogni punto, calcola quanto segue.
- Calcola il tempo di deriva usando Eq (6):
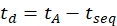 (6)
(6)
con tseq il tempo che precede la separazione IMS finale (il valore «Time Abs» indicato al punto 3.4.6).
NOTA: se si calibrano più spettri IMS/IMS acquisiti con sequenze diverse, controllare attentamente i valori tseq. - Calcola la massa neutra degli ioni usando Eq (7):
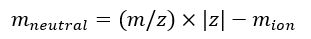 (7)
(7) - Calcola i parametri td' e td'' usando Eq (8) ed Eq (9):
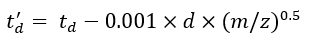 (8)
(8)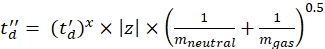 (9)
(9) - Calcolare i valori CCS calibrati finali (TWCCSN2 in nm2) utilizzando Eq (10):
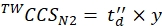 (10)
(10)
NOTA: anche se il passaggio 5.2.2. dà ln(y) come intercetta, y deve essere usato per ottenere il valore CCS finale. Non dimenticare di applicare una funzione esponenziale.
- Calcola il tempo di deriva usando Eq (6):
- Verificare l'accuratezza della taratura applicando la taratura alla seconda acquisizione della soluzione di taratura acquisita al punto 2.4.
NOTA: la calibrazione dovrebbe produrre risultati con un errore di ~ 1-2%.
Risultati
Un pentasaccaride arabinoxilano, XA2XX, è stato scelto come esempio per illustrare questo protocollo. Questo composto è disponibile in commercio, ma solo come miscela con un altro pentasaccaride arabinoxilano, XA3XX (XA3XX puro è anche disponibile in commercio). Le strutture di XA2XX e XA3XX sono riportate nella Figura supplementare S1. Poiché il rapporto tra XA2XX e XA3XX nella miscela commerciale è ~ 50: 50, è stata prep...
Discussione
L'IMS ciclico SELECT SERIES è un potente strumento che consente di selezionare una popolazione di ioni definita, di una data mobilità m/z e ionica, senza la necessità di separazione cromatografica a monte. Lo strumento offre la possibilità di generare una mappa di frammentazione bidimensionale di questa popolazione ionica, da cui è possibile estrarre sia gli spettri MS/MS che IMS/IMS. Tuttavia, l'utente deve notare diversi punti critici che richiedono attenzione durante il processo sperimentale.
Divulgazioni
Gli autori non hanno alcun conflitto di interessi da divulgare.
Riconoscimenti
S.O. è grato all'Agenzia Nazionale Francese per la Ricerca per aver finanziato il suo dottorato di ricerca (sovvenzione ANR-18-CE29-0006).
Materiali
| Name | Company | Catalog Number | Comments |
| 33-α-L- plus 23-α-L-Arabinofuranosyl-xylotetraose (XA3XX/XA2XX) mixture | Megazyme Ltd., Wicklow, Ireland | O-XAXXMIX | XA2XX + XA3XX mixture |
| 33-α-L-Arabinofuranosyl-xylotetraose (XA3XX) | Megazyme Ltd., Wicklow, Ireland | O-XA3XX | Pure XA3XX standard |
| Eppendorf Safe-Lock Tubes, 1.5 mL, Eppendorf Quality, colorless, 1,000 tubes | Eppendorf, Hamburg, Germany | 0030120086 | Used to prepare the carbohydrate stock solution and dilution |
| FALCON 50 mL Polypropylene Conical Tube 30 x 115 mm | Corning Science México S.A. de C.V., Reynosa, Tamaulipas, Mexico | 352070 | Used to prepare the aqueous stock solution of 100 mM LiCl |
| Lithium Chloride (ACS reagent, ≥99 %) | Sigma-Aldrich Inc., Saint Quentin Fallavier, France | 310468 | Used to dope the sample with lithium |
| Major Mix IMS/Tof Calibration Kit | Waters Corp., Wilmslow, UK | 186008113 | Calibration solution for MS and IMS |
| MassLynx 4.2 SCN1016 Release 6 (Waters Embedded Analyser Platform for Cyclic IMS 2.9.1 Release 9) | Waters Corp., Wilmslow, UK | 721022377 | Cyclic IMS vendor software for instrument control and data processing |
| Methanol for HPLC PLUS Gradient grade | Carlo-Erba Reagents, Val de Reuil, France | 412383 | High-purity solvent |
| MS Leucine Enkephaline Kit | Waters Corp., Wilmslow, UK | 700002456 | Reference compound used for tuning of the mass spectrometer |
| SCHOTT DURAN 100 mL borosilicate glass bottle | VWR INTERNATIONAL, Radnor, Pennsylvania, US | 218012458 | Used to prepare the solution of 500 µM LiCl in 50:50 MeOH/Water |
| SELECT SERIES Cyclic IMS | Waters Corp., Wilmslow, UK | 186009432 | Ion mobility-mass spectrometer equipped with a cylic IMS cell |
| Website: http://mzmine.github.io/ | MZmine Development Team | - | Link to download the MZmine software |
| Website: https://github.com/siollivier/IM-MN | INRAE, UR BIA, BIBS Facility, Nantes, France | - | Link to an in-house R script containing a CCS calibration function |
Riferimenti
- Allard, P. -. M., et al. Integration of molecular networking and in-silico MS/MS fragmentation for natural products dereplication. Analytical Chemistry. 88 (6), 3317-3323 (2016).
- Wang, M., et al. Mass spectrometry searches using MASST. Nature Biotechnology. 38 (1), 23-26 (2020).
- David, M., Fertin, G., Rogniaux, H., Tessier, D. SpecOMS: a full open modification search method performing all-to-all spectra comparisons within minutes. Journal of Proteome Research. 16 (8), 3030-3038 (2017).
- Dührkop, K., et al. SIRIUS 4: a rapid tool for turning tandem mass spectra into metabolite structure information. Nature Methods. 16 (4), 299-302 (2019).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Nothias, L. -. F., et al. Feature-based molecular networking in the GNPS analysis environment. Nature Methods. 17 (9), 905-908 (2020).
- Gray, C. J., et al. Advancing solutions to the Carbohydrate Sequencing Challenge. Journal of the American Chemical Society. 141 (37), 14463-14479 (2019).
- Ropartz, D., et al. Online coupling of high-resolution chromatography with extreme UV photon activation tandem mass spectrometry: Application to the structural investigation of complex glycans by dissociative photoionization. Analytica Chimica Acta. 933, 1-9 (2016).
- Wolff, J. J., et al. Negative electron transfer dissociation of glycosaminoglycans. Analytical Chemistry. 82 (9), 3460-3466 (2010).
- Ropartz, D., et al. Charge transfer dissociation of complex oligosaccharides: comparison with collision-induced dissociation and extreme ultraviolet dissociative photoionization. Journal of the American Society for Mass Spectrometry. 27 (10), 1614-1619 (2016).
- Morelle, W., et al. Fragmentation characteristics of permethylated oligosaccharides using a matrix-assisted laser desorption/ionization two-stage time-of-flight (TOF/TOF) tandem mass spectrometer. Rapid Communications in Mass Spectrometry. 18 (22), 2637-2649 (2004).
- Gabelica, V., Marklund, E. Fundamentals of ion mobility spectrometry. Current Opinion in Chemical Biology. 42, 51-59 (2018).
- Gabelica, V., et al. Recommendations for reporting ion mobility mass spectrometry measurements. Mass Spectrometry Reviews. 38 (3), 291-320 (2019).
- Hernandez-Mesa, M., et al. Interlaboratory and interplatform study of steroids collision cross section by traveling wave ion mobility spectrometry. Analytical Chemistry. 92 (7), 5013-5022 (2020).
- Koeniger, S. L., et al. An IMS-IMS analogue of MS-MS. Analytical Chemistry. 78 (12), 4161-4174 (2006).
- Merenbloom, S. I., Koeniger, S. L., Valentine, S. J., Plasencia, M. D., Clemmer, D. E. IMS−IMS and IMS−IMS−IMS/MS for separating peptide and protein fragment ions. Analytical Chemistry. 78 (8), 2802-2809 (2006).
- Eldrid, C., Thalassinos, K. Developments in tandem ion mobility mass spectrometry. Biochemical Society Transactions. 48 (6), 2457-2466 (2020).
- Giles, K., et al. A cyclic ion mobility-mass spectrometry system. Analytical Chemistry. 91 (13), 8564-8573 (2019).
- Merenbloom, S. I., Glaskin, R. S., Henson, Z. B., Clemmer, D. E. High-resolution ion cyclotron mobility spectrometry. Analytical Chemistry. 81 (4), 1482-1487 (2009).
- Ollivier, S., et al. Anomeric retention of carbohydrates in multistage cyclic ion mobility (IMSn): de novo structural elucidation of enzymatically produced mannosides. Analytical Chemistry. 93 (15), 6254-6261 (2021).
- Ollivier, S., Fanuel, M., Rogniaux, H., Ropartz, D. Molecular networking of high-resolution tandem ion mobility spectra: a structurally relevant way of organizing data in glycomics. Analytical Chemistry. 93 (31), 10871-10878 (2021).
- Aron, A. T., et al. Reproducible molecular networking of untargeted mass spectrometry data using GNPS. Nature Protocols. 15 (6), 1954-1991 (2020).
- McKenna, K. R., Li, L., Krishnamurthy, R., Liotta, C. L., Fernández, F. M. Organic acid shift reagents for the discrimination of carbohydrate isobars by ion mobility-mass spectrometry. The Analyst. 145 (24), 8008-8015 (2021).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11, 395 (2010).
- Ruotolo, B. T., Benesch, J. L. P., Sandercock, A. M., Hyung, S. -. J., Robinson, C. V. Ion mobility-mass spectrometry analysis of large protein complexes. Nature Protocols. 3 (7), 1139-1152 (2008).
- Bush, M. F., Hall, Z., Giles, K., Hoyes, J., Robinson, C. V., Ruotolo, B. T. Collision cross sections of proteins and their complexes: a calibration framework and database for gas-phase structural biology. Analytical Chemistry. 82 (22), 9557-9565 (2010).
- Ropartz, D., et al. Structure determination of large isomeric oligosaccharides of natural origin through multipass and multistage cyclic traveling-wave ion mobility mass spectrometry. Analytical Chemistry. 91 (18), 12030-12037 (2019).
- Tolmachev, A. V., et al. Characterization of ion dynamics in structures for lossless ion manipulations. Analytical Chemistry. 86 (18), 9162-9168 (2014).
- Arndt, J. R., et al. High-resolution ion-mobility-enabled peptide mapping for high-throughput critical quality attribute monitoring. Journal of the American Society for Mass Spectrometry. 32 (8), 2019-2032 (2021).
- Le Fèvre, A., Dugourd, P., Chirot, F. Exploring conformational landscapes using trap and release tandem ion mobility spectrometry. Analytical Chemistry. 93 (9), 4183-4190 (2021).
- Ohshimo, K., He, X., Ito, R., Misaizu, F. Conformer separation of dibenzo-crown-ether complexes with Na+ and K+ ions studied by cryogenic ion mobility-mass spectrometry. The Journal of Physical Chemistry A. 125 (17), 3718-3725 (2021).
- Purves, R. W., Barnett, D. A., Ells, B., Guevremont, R. Gas-phase conformers of the [M + 2H]2+ ion of bradykinin investigated by combining high-field asymmetric waveform ion mobility spectrometry, hydrogen/deuterium exchange, and energy-loss measurements. Rapid Communications in Mass Spectrometry. 15 (16), 1453-1456 (2001).
- Ujma, J., et al. Cyclic ion mobility mass spectrometry distinguishes anomers and open-ring forms of pentasaccharides. Journal of the American Society for Mass Spectrometry. 30 (6), 1028-1037 (2019).
- Warnke, S., Faleh, A. B., Scutelnic, V., Rizzo, T. R. Separation and identification of glycan anomers using ultrahigh-resolution ion-mobility spectrometry and cryogenic ion spectroscopy. Journal of The American Society for Mass Spectrometry. 30 (11), 2204-2211 (2019).
- Williamson, D. L., Bergman, A. E., Nagy, G. Investigating the structure of α/β carbohydrate linkage isomers as a function of group I metal adduction and degree of polymerization as revealed by cyclic ion mobility separations. Journal of the American Society for Mass Spectrometry. 32 (10), 2573-2582 (2021).
- Myers, O. D., Sumner, S. J., Li, S., Barnes, S., Du, X. One step forward for reducing false positive and false negative compound identifications from mass spectrometry metabolomics data: new algorithms for constructing extracted ion chromatograms and detecting chromatographic peaks. Analytical Chemistry. 89 (17), 8696-8703 (2017).
- Marchand, A., Livet, S., Rosu, F., Gabelica, V. Drift tube ion mobility: how to reconstruct collision cross section distributions from arrival time distributions. Analytical Chemistry. 89 (23), 12674-12681 (2017).
- Davis, D. M., et al. Analysis of ion mobility spectra for mixed vapors using Gaussian deconvolution. Analytica Chimica Acta. 289 (3), 263-272 (1994).
- Polasky, D. A., Dixit, S. M., Fantin, S. M., Ruotolo, B. T. CIUSuite 2: next-generation software for the analysis of gas-phase protein unfolding data. Analytical Chemistry. 91 (4), 3147-3155 (2019).
- Salbo, R., et al. Traveling-wave ion mobility mass spectrometry of protein complexes: accurate calibrated collision cross-sections of human insulin oligomers. Rapid Communications in Mass Spectrometry. 26 (10), 1181-1193 (2012).
- Gelb, A. S., Jarratt, R. E., Huang, Y., Dodds, E. D. A study of calibrant selection in measurement of carbohydrate and peptide ion-neutral collision cross sections by traveling wave ion mobility spectrometry. Analytical Chemistry. 86 (22), 11396-11402 (2014).
- Richardson, K., Langridge, D., Dixit, S. M., Ruotolo, B. T. An improved calibration approach for traveling wave ion mobility spectrometry: robust, high-precision collision cross sections. Analytical Chemistry. 93 (7), 3542-3550 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon