Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Использование спектрометра циклической подвижности ионов для экспериментов с тандемной подвижностью ионов
В этой статье
Резюме
Спектрометрия подвижности ионов (IMS) является интересным дополнением к масс-спектрометрии для характеристики биомолекул, особенно потому, что она чувствительна к изомерии. Этот протокол описывает тандемный эксперимент IMS (IMS/IMS), который позволяет выделить молекулу и генерировать профили подвижности ее фрагментов.
Аннотация
Точная характеристика химических структур важна для понимания их основных биологических механизмов и функциональных свойств. Масс-спектрометрия (МС) является популярным инструментом, но не всегда достаточна для полного раскрытия всех структурных особенностей. Например, хотя углеводы биологически значимы, их характеристика осложняется многочисленными уровнями изомерии. Спектрометрия подвижности ионов (IMS) является интересным дополнением, поскольку она чувствительна к конформациям ионов и, таким образом, к изомерии.
Кроме того, последние достижения значительно улучшили технику: последнее поколение инструментов Cyclic IMS предлагает дополнительные возможности по сравнению с линейными инструментами IMS, такие как повышенная разрешающая способность или возможность выполнять эксперименты с тандемной подвижностью ионов (IMS / IMS). Во время IMS/IMS ион выбирается на основе его подвижности ионов, фрагментируется и повторно анализируется для получения информации о подвижности ионов о его фрагментах. Недавняя работа показала, что профили мобильности фрагментов, содержащиеся в таких данных IMS/IMS, могут действовать как отпечаток конкретного гликана и могут использоваться в стратегии молекулярных сетей для организации наборов данных гликомики структурно значимым образом.
Таким образом, цель этого протокола состоит в том, чтобы описать, как генерировать данные IMS/IMS, от подготовки образца до окончательной калибровки поперечного сечения столкновения (CCS) измерения подвижности ионов, которое дает воспроизводимые спектры. На примере одного репрезентативного гликана этот протокол покажет, как построить управляющую последовательность IMS/IMS на приборе Cyclic IMS, как учитывать эту контрольную последовательность для преобразования времени прибытия IMS во время дрейфа (т.е. эффективное время разделения, применяемое к ионам), и как извлекать соответствующую информацию о мобильности из необработанных данных. Этот протокол предназначен для четкого объяснения критических моментов эксперимента IMS/IMS и, таким образом, помогает новым пользователям Cyclic IMS выполнять простые и воспроизводимые приобретения.
Введение
Полная химическая характеристика биомолекул является ключом к пониманию их основных биологических и функциональных свойств. С этой целью в последние годы развиваются «омические» науки, нацеленные на широкомасштабную характеристику химических структур в биологических концентрациях. В протеомике и метаболомике РС стал основным инструментом для разгадки структурной гетерогенности, обнаруженной в биологических средах, особенно благодаря его чувствительности и способности предоставлять структурную информацию через тандемный РС (РС / МС). В стратегиях MS / MS ион выбирается в соответствии с его массой, затем фрагментируется, и, наконец, массы его фрагментов приобретаются, чтобы установить отпечаток молекулы. Спектры MS/MS могут, в частности, использоваться для сопоставления спектральных баз данных1,2 или предварительной реконструкции родительских структур3,4. Исходя из предположения, что сходные спектры принадлежат аналогичным соединениям, данные MS/MS также могут быть использованы для построения молекулярных сетей (MNs), соединяющих родственные виды через оценку сходства5,6.
Однако из-за присущего МС свойства обнаруживать отношение массы к заряду (m/z) ионов, методика слепа к ряду структурных особенностей, которые попадают в диапазон (стерео)изомерии. Например, углеводы состоят из нескольких моносахаридных субъединиц, многие из которых являются стереоизомерами или даже эпимерами (например, Glc против Gal или Glc против Man). Эти субъединицы связаны гликозидными связями, которые могут отличаться положением связи (региоизомеризм) и стерической конфигурацией аномерного углерода (аномеризм). Эти характеристики затрудняют для автономного рассеянного склероза различение изомеров углеводов7, и только региоизомеризм может быть устранен с использованием высокоэнергетических методов активации8,9,10. Хотя дериватизация является вариантом нарушения эквивалентности стереоизомерных групп11, она требует обширной пробоподготовки. Другим, более простым вариантом является соединение MS с аналитическим измерением, чувствительным к изомерии, таким как IMS.
Поскольку этот протокол предназначен для пользователей, которые уже знакомы с основными концепциями IMS, и поскольку подробные обзоры доступны в других местах12,13, здесь приведен только краткий обзор принципов IMS. IMS - это метод разделения газовой фазы, который опирается на взаимодействие ионов с буферным газом и электрическим полем, в конечном итоге разделяя ионы в соответствии с их газофазными конформациями. Различные принципы IMS в сочетании с MS можно найти на коммерческих приборах: некоторые работают при переменных высоких и низких электрических полях (асимметричные поля IMS, FAIMS), в то время как большинство работают в пределах предела низкого поля, в частности дрейфующая трубка IMS (DTIMS, линейно уменьшающееся электрическое поле), бегущая волна IMS (TWIMS, симметричные потенциальные волны) и захваченные IMS (TIMS, высокий поток буферных газов, улавливающих ионы против электрических полей)13 . Низкопольные методы обеспечивают доступ к так называемому CCS, свойству пары ион-газ, которая представляет поверхность (в Å2 или нм2) иона, который взаимодействует с буферным газом во время разделения. CCS теоретически не зависит от приборов и, таким образом, полезен для получения данных, которые могут быть воспроизведены между различными лабораториями14. На разделение подвижности ионов могут влиять различные параметры и, в частности, колебания давления газа и температуры газа в ячейке подвижности. Калибровка CCS является способом исправить это, поскольку как калибрант, так и интересующие виды будут затронуты аналогичным образом13. Тем не менее, обязательно установить прибор в помещении с контролируемой температурой и иметь надежную систему контроля давления газа.
Интересной эволюцией IMS является IMS/IMS, которая была впервые представлена в 2006 году группой Клеммера как аналог MS/MS15,16. В IMS/IMS ион, представляющий интерес, выборочно изолируется на основе его подвижности ионов; затем он активируется (до возможной фрагментации) и выполняется новый анализ IMS активированного иона или фрагментов. В первой инструментальной конструкции две ячейки IMS были помещены последовательно, разделенные ионной воронкой, где стояла активация. С тех пор, хотя был предложен ряд установок IMS / IMS (для обзора см. Eldrid и Thalassinos17), первый коммерческий масс-спектрометр с возможностью IMS / IMS стал доступен только в 201918 году. Этот инструмент существенно улучшил первоначальную концепцию, объединив ее с другим технологическим прорывом: циклической конструкцией ячейки IMS.
Циклическая ячейка IMS теоретически позволяет почти бесконечно увеличивать длину дрейфующего пути и, таким образом, разрешающую способность прибора19. Это было достигнуто с помощью определенной геометрии прибора, где циклическая ячейка TWIMS размещена ортогонально к основной оптической оси ионов. Многофункциональная область массива на входе в ячейку IMS позволяет контролировать направление ионного пути: (i) отправлять ионы вбок для разделения IMS, (ii) вперед для обнаружения MS или (iii) назад от ячейки IMS для хранения в ячейке prearray. Из этой ячейки предварительного хранилища ионы могут быть активированы, а фрагменты повторно введены в ячейку IMS для измерения подвижности ионов, подход, который был успешно использован для характеристики стереоизомеров20. В конечном счете, собранные данные содержат информацию о подвижности ионов и m/z для предшественника и его фрагментов.
В недавней публикации, которая использовала этот циклический дизайн для анализа гликанов (Ollivier et al.21), мы показали, что профиль подвижности фрагментов, содержащихся в таких данных IMS / IMS, действует как отпечаток биомолекулы, который может быть использован в стратегии молекулярной сети. Полученная сеть, называемая IM-MN, привела к организации наборов данных гликомики структурно значимым образом, тогда как сеть, построенная исключительно из данных MS / MS (MS-MN), выявила мало информации. Чтобы дополнить эту публикацию и помочь пользователям Cyclic IMS реализовать этот рабочий процесс, этот протокол предоставляет полное описание протокола, используемого для сбора данных. Этот протокол фокусируется только на генерации данных IMS/IMS, которые пользователи могут затем использовать для построения сетей IM-MN (см. 21) или для любого другого приложения по своему выбору. Построение IM-MN не будет рассматриваться в настоящем документе, так как протоколы для молекулярных сетей уже доступны22. Подчеркиваются важнейшие моменты, которые необходимо соблюдать для получения ценных и воспроизводимых приобретений СУИ/СУИ. Возьмем пример одного из олигосахаридов, изученных Ollivier et al. На фиг.21 подробно описаны следующие этапы: i) подготовка проб, ii) настройка прибора циклической ИСМ, iii) автоматический пиковый отбор данных и iv) калибровка УХУ.
протокол
ПРИМЕЧАНИЕ: Обзор протокола приведен на рисунке 1. Параметры, используемые для экспериментов, описанных в настоящем протоколе, можно найти в Дополнительной таблице S1 и Дополнительной таблице S2.
1. Подготовка образца раствора
ПРИМЕЧАНИЕ: Протокол описан с использованием арабиноксилана пентасахарида (23-α-L-арабинофуранозил-ксилотетраоза или XA2XX; см. Таблицу материалов) в качестве примера.
- Приготовление растворителя: 500 мкМ LiCl в 50:50 H2O:MeOH (об./об.).
- Приготовьте 100 мМ раствора хлорида лития (LiCl) в H2O путем взвешивания 212 мг LiCl и добавьте 50 мл деионизированной воды высокой чистоты (H2O) в коническую трубку из полипропилена объемом 50 мл. Встряхните до полного растворения.
ПРИМЕЧАНИЕ: Растворитель легируется солью лития для содействия образованию аддуктов [M + Li] + в источнике ионов спектрометра, поскольку он обычно дает более качественные спектры фрагментации по сравнению с другими щелочными аддуктами. Использование LiCl рекомендуется, поскольку ранее было обнаружено, что органические кислоты (и, следовательно, их соли) влияют на профили IMS23. - В стеклянном флаконе разводят раствор запаса LiCl в 200x: до 250 мкл исходного раствора, добавляют 24,75 мл H2O. Добавляют 25 мл метанола (MeOH) для достижения конечной концентрации LiCl 500 мкМ в 50:50 H2O:MeOH (v/v). Соникат в течение 2 мин для дегазации растворителя.
ПРИМЕЧАНИЕ: MeOH представляет опасность для здоровья (H225, H301, H311, H331, H370); манипулировать под капюшоном экстрактора в лабораторном халате, перчатках и защите глаз. Пропорция 50:50 MeOH/H2O (v/v), по-видимому, является лучшим растворителем для ионизации олигосахаридов; однако MeOH может быть заменен ацетонитрилом (ACN), если это необходимо.
- Приготовьте 100 мМ раствора хлорида лития (LiCl) в H2O путем взвешивания 212 мг LiCl и добавьте 50 мл деионизированной воды высокой чистоты (H2O) в коническую трубку из полипропилена объемом 50 мл. Встряхните до полного растворения.
- В полипропиленовой трубке объемом 1,5 мл взвесьте 1 мг углевода. Растворяют с соответствующим объемом 500 мкМ LiCl до концентрации 1 мг/мл. Разбавляют до конечной концентрации 10 мкг/мл в 50:50 MeOH/H2O + 500 мкМ LiCl. Хранить при температуре 4 °C.
ПРИМЕЧАНИЕ: Концентрация 10 мкг/мл была выбрана для оптимизации сигнала по всем фрагментам ионов во время IMS/IMS-MS (это для чистого соединения; увеличение концентрации при работе на смесях). Для получения эталонных спектров IMS/IMS не разбавляйте образец дальше: ожидается насыщение детектора MS до фрагментации, хотя прибор предлагает варианты его коррекции (см. этап 3.2.).
2. Настройка циклического масс-спектрометра IMS
ПРИМЕЧАНИЕ: Инструкции, связанные с программным обеспечением (окна, меню и команды), выделены жирным шрифтом.
- Откройте приборную консоль из программного обеспечения управления прибором (страница MS tune , см. сведения о программном обеспечении в Таблице материалов) и переведите прибор в режим работы . Подождите не менее 3 часов, пока высокие напряжения стабилизируются в ячейке IMS.
ПРИМЕЧАНИЕ: Для лучшей воспроизводимости напряжения в ячейке IMS должны быть полностью стабилизированы. Включите высокие напряжения и дайте инструменту стабилизироваться в течение ночи перед любым циклическим анализом IMS. Кроме того, давление и температура в ячейке подвижности ионов должны поддерживаться как можно более постоянными. Хотя обратное считывание давления доступно на вкладке Вакуум , обратное считывание для температуры недоступно. Храните прибор в термостатированной лаборатории. Прибор, используемый в этой работе, работает при 1,75 мбар в лаборатории с термостатом при 20 °C. - Циклическая настройка приборов IMS
ПРИМЕЧАНИЕ: Стандартные растворы должны вводиться с использованием встроенной жидкостной системы для установки прибора.- Поместите контейнеры с жидкостями, заполненные соответствующими стандартами, предоставленными заводом-изготовителем, в систему жидкости: Резервуар B ('Lockmass'): 10 пг/мкл лейцин-энкефалина (LEU ENK) в 50:50 ACN/H2O + 0,1% муравьиной кислоты; Резервуар C ('Калибрант'): MajorMix.
ПРИМЕЧАНИЕ: В этом протоколе калибровочное решение MajorMix будет использоваться для калибровки размеров m/z и CCS. По практическим соображениям будет выполнена внешняя калибровка CCS (см. шаг 5 протокола); следовательно, можно также использовать собственную смесь калибранта для CCS и другой калибрант для m/z (например, формиат натрия или йодид натрия). - На странице Настройка консоли Quartz перейдите на вкладку Fluidics . Установите флюидику образца в резервуар C, а эталонную флюидику в резервуар B. Вливайте оба раствора последовательно в источник ионов для проверки сигнала MS.
- Выполните настройку АЦП, настройку детектора (с помощью НОУ ENK) и калибровку массы (см. Таблицу материалов для калибровочного раствора) на странице Настройка прибора в соответствии с инструкциями производителя.
- Поместите контейнеры с жидкостями, заполненные соответствующими стандартами, предоставленными заводом-изготовителем, в систему жидкости: Резервуар B ('Lockmass'): 10 пг/мкл лейцин-энкефалина (LEU ENK) в 50:50 ACN/H2O + 0,1% муравьиной кислоты; Резервуар C ('Калибрант'): MajorMix.
- Запишите получение IMS калибровочного раствора с однопроходным разделением (используйте это для внешней калибровки IMS).
ПРИМЕЧАНИЕ: Параметры источника ионов и бегущей волны (TW) (высота статической волны и скорость волны) должны быть постоянными во время всех сборов (калибровка и приобретение). Если пользователь не обладает предварительными знаниями об оптимальных параметрах для своего образца, этот шаг может быть выполнен после шага 3 протокола (для аддуктов [M+Li]+ нейтральных олигосахаридов репрезентативные результаты используют высоту TW 16 В и скорость TW 350 м/с, которые дают наилучшие результаты).- На вкладке Fluidics выберите положение перегородки Sample и вливайте калибрант (см. Таблицу материалов) в источник ионов (с использованием встроенной системы флюидиков) через зонд «Sample» со скоростью потока 10 мкл/мин.
- Настройте однопроходную последовательность IMS. На странице Настройка переведите инструмент в режим мобильности и откройте окно Управление циклической последовательностью . Выберите Расширенный режим. На вкладке Циклические функции этого нового окна выберите Добавить связку, а затем Single/Multipass. Дождитесь появления последовательности событий мобильности на вкладке Sequence того же окна.
ПРИМЕЧАНИЕ: Для активации отображения в режиме реального времени пользователь должен применить параметры инструмента: нажать настроить в режиме TOF или Запустить в режиме мобильности . Перед переключением инструмента между режимами TOF и Mobility необходимо прервать любое запущенное приобретение (включая отображение страницы Tune ). Относительное обилие ионов может варьироваться между режимами TOF и Mobility из-за изменения параметров передачи ионов. - Адаптируйте последовательность таким образом, чтобы все калибрантные ионы делали один проход вокруг циклической гоночной трассы IMS. Не изменяйте время впрыска или выброса и приобретайте время; однако уменьшите время разделения до 1 мс (на вкладке Последовательность ). Если некоторые ионы калибровочной смеси не помещаются в отображаемое окно времени прибытия, измените синхронизацию IMS с толкателем анализатора ортогонального ускорения TOF, увеличив количество Pushes Per Bin во вкладке Настройки АЦП .
ПРИМЕЧАНИЕ: Время в управляющей последовательности управляет только многофункциональным массивом для ионного нагнетания. До тех пор, пока ионы участвуют в своем первом (или n-м) прохождении вокруг гоночной трассы, они завершат указанный проход, даже если направление TW изменилось в массиве за это время. Снижение времени разделения до 1 мс означает, что массив перейдет в режим выброса через 1 мс. Это гарантирует, что у более быстрых ионов не будет достаточно времени, чтобы пройти через массив и сделать второй проход, прежде чем более медленные ионы завершат свой первый проход. Поэтому все ионы будут подвергаться одинаковому количеству проходов (т.е. одному проходу), которое необходимо для выполнения калибровки IMS. - Запишите 2-минутное приобретение. В окне Управление циклической последовательностью нажмите « Получить », чтобы открыть всплывающее окно «Параметры получения ». Введите имя файла, описание и продолжительность получения (мин) и нажмите кнопку Сохранить.
- Запишите еще 2 минуты получения калибровочного раствора в тех же условиях, что и на шаге 2.3 (используйте это для проверки качества калибровки CCS). В окне Управление циклической последовательностью нажмите « Получить », чтобы открыть всплывающее окно «Параметры получения ». Введите имя файла, описание и продолжительность получения (мин) и нажмите кнопку Сохранить.
- Тщательно промывайте систему жидкости с помощью 50:50 H2O/ACN, чтобы избежать кристаллизации калибранта в трубке.
3. Приобретение IMS/IMS-MS
- Используя шприцевой насос, вливайте (легированный литием) образец со скоростью 10 мкг/мл через пробоотборник со скоростью потока 10 мкл/мин.
- Переключите прибор в режим TOF (со страницы настроек MS ), чтобы проверить стабильность сигнала. Запишите полное получение МС (1 мин) образца, что будет полезно для проверки изотопного рисунка и наличия потенциальных загрязняющих веществ.
ПРИМЕЧАНИЕ: Поскольку концентрация образца выбирается для получения хорошего ионного сигнала для фрагментов, на этом этапе может наблюдаться насыщение TOF. Насыщение TOF может быть идентифицировано с помощью следующих артефактов: (i) искусственно увеличенное разрешение MS, (ii) изменение изотопных соотношений и (iii) множество пиков низкой плотности между изотопами. Используйте объектив DRE (расширение динамического диапазона, вкладка Quad/MS Profile/DRE на главной странице Tune) для ослабления передачи ионов и отбрасывания насыщения в режиме TOF (рисунок 2A,B). - Переведите прибор в режим MSMS (вкладка Quad/MS Profile на главной странице Tune ) и выберите массу целевого иона в поле MSMS Mass для выделения в квадруполе (в примере: m/z 685.2, соответствующий [M+Li]+ ионному виду арабиноксилана пентасахарида). Запишите 1 минуту сбора, чтобы проверить изоляцию предшественника при обработке данных.
ПРИМЕЧАНИЕ: Аддукты лития имеют изотоп в -1 Да моноизотопного пика, который необходимо удалить из окна выбора MS/MS, чтобы он не мешал этапам обработки. Его можно удалить, сузив диапазон выбора с помощью параметров LM Resolution и HM Resolution на вкладке Quad/MS Profile (рисунок 2C). - Настройте последовательность IMS «нарезки» для выполнения выбора интересующего изомера на основе мобильности.
- Переключите прибор в режим Mobility Mode (см. шаг 2.3.2). В окне Управление циклической последовательностью на вкладке Циклические функции выберите Добавить связку , а затем Нарезка. Дождитесь появления сложной последовательности событий мобильности на вкладке Sequence (рисунок 3).
ПРИМЕЧАНИЕ: Можно визуализировать каждый шаг процесса IMS/IMS: нажмите на событие «Извлечь и получить» на вкладке «Последовательность». После выделения красным цветом переместите его в соответствующее положение в последовательности с помощью кнопок «Вверх» и «Вниз». - Поместите событие Eject and Acquire сразу после первого события Separate (т. е. переместите его в строку 3 вместо строки 8 в последовательности, как показано на рисунке 3), а затем нажмите кнопку Выполнить. Ищите результаты начального разделения, которые будут отображаться в режиме реального времени. Увеличьте продолжительность первого отдельного события для многопроходного разделения, изменив значение времени для этого события в последовательности до тех пор, пока разрешение пиков IMS не станет удовлетворительным. Запишите 1 минуту приобретения для справки.
ПРИМЕЧАНИЕ: Обратите внимание на значение ADC Start Delay на вкладке Настройка АЦП : будет полезно проверить качество изоляции. - Нажмите кнопку Пауза. Обратите внимание, что отображаются результаты начального разделения, хотя изменения в последовательности элементов управления не будут применяться до тех пор, пока пользователь не нажмет кнопку Выполнить еще раз. Поместите событие «Извлечение и получение» под событиями «Извлечение», «Извлечение в предварительное хранилище» и «Удержание и извлечение». Отрегулируйте продолжительность событий таким образом, чтобы целевой пик находился в области «Извлечь в предварительное хранилище», а любой другой ион — в области извлечения или удержания и извлечения.
ПРИМЕЧАНИЕ: Продолжительность этих трех событий по сравнению с распределением времени прибытия (ATD) может быть визуализирована с помощью цветовой полосы под спектром мобильности на вкладке Mobilogram (рисунок 3). - Поместите событие Eject and Acquire в конце последовательности, под событием Reinject from Pre-Store и вторым отдельным событием. Нажмите кнопку Выполнить , чтобы отобразить выбранное население.
ПРИМЕЧАНИЕ: Поскольку выбранная популяция покинула ячейку IMS, все предыдущее разделение было потеряно, и она вернулась к однопроходному разделению (что является желательным). - Проверьте качество изоляции. Чтобы убедиться, что выбран только пик интереса, выполните то же разделение после повторной закачки, что и до повторной закачки (т.е. то же отдельное время), как показано на рисунке 4. Запишите 1 минуту приобретения для справки.
ПРИМЕЧАНИЕ: Пользователям рекомендуется проверять выброшенную популяцию; Временное окно Eject to Pre-Store должно быть базового уровня (рисунок 4B). Чтобы проверить это, переведите задержку запуска АЦП в ручной режим на вкладке Настройки АЦП и введите время задержки, указанное на шаге 3.4.2. Запишите 1 минуту приобретения для справки. - На вкладке Последовательность в столбце рядом с заданным пользователем временем события (столбец Time Abs выделен красным цветом) найдите суммированное время всех событий. Обратите внимание на Time Abs , найденное в строке события Reinject from Pre-Store для выполнения калибровки CCS.
- Переключите прибор в режим Mobility Mode (см. шаг 2.3.2). В окне Управление циклической последовательностью на вкладке Циклические функции выберите Добавить связку , а затем Нарезка. Дождитесь появления сложной последовательности событий мобильности на вкладке Sequence (рисунок 3).
- Фрагментируйте целевой пик между двумя раундами IMS. Изменяйте напряжения ступени обратной закачки, чтобы увеличить кинетическую энергию ионов, и фрагментируйте их при столкновении с газом подвижности ионов.
- Установите продолжительность отдельного события, непосредственно предшествующего извлечению и получению , равным 1 мс (см. объяснение в шаге 2.3.3).
- В строке Повторное добавление из предварительного хранилища установите флажок Включить активацию и оптимизируйте фрагментацию с помощью встроенного элемента управления. Если спектр является удовлетворительным (например, базовый пик является фрагментом), перейдите непосредственно к шагу 3.5.4.
ПРИМЕЧАНИЕ: При включении активации три напряжения на линии станут серыми: это напряжения, которые пользователь должен изменить, если требуется ручная оптимизация напряжений (см. следующий шаг). Эти три напряжения (Pre-Array Gradient, Pre-Array Bias и Array Offset) образуют градиент, используемый для активации ионов. Кинетическая энергия ионов будет увеличиваться с наклоном между смещением pre-Array и смещением массива (см. рисунок 5). Значения по умолчанию для значений смещения → смещения градиента → смещения по умолчанию: без активации 85 → 70 → 45 В; максимальная активация встроенной функции 185 → 170 → -5 В (+150 В). После фрагментации не забудьте перенастроить ионную передачу с помощью линзы DRE (уменьшить затухание сигнала) (см. шаг 3.2.). - Если фрагментация не удовлетворяет встроенному элементу управления, снимите флажок Включить активацию и перейдите к ручной оптимизации напряжений обратной установки. Увеличьте напряжение градиента предварительного массива (напряжение смещения перед массивом всегда должно поддерживаться на 15 В ниже градиента предварительного массива) и понизьте напряжение смещения массива (которое может быть установлено как отрицательное) до тех пор, пока результаты не будут удовлетворительными.
ПРИМЕЧАНИЕ: При ручной настройке напряжений многофункционального массива пользователь может переключиться с представления «Mobilogram» на интерактивные схемы напряжений, подаваемых в многофункциональном массиве (PE-диаграмма), чтобы лучше визуализировать настройки напряжения (рисунок 5A). - Запишите 2-минутное приобретение. Во всплывающем окне сбора установите флажок Сохранить время дрейфа , чтобы создать файл, содержащий только время прибытия против m/z (время получения, используемое для хроматографического анализа - время хранения - удаляется из файла). Обратите внимание, что этот файл помечен как *_dt. СЫРОЙ.
ПРИМЕЧАНИЕ: Если пользователь забыл проверить опцию Сохранить время дрейфа , все равно можно извлечь измерение IMS с помощью программного обеспечения Driftscope 2.9 (Файл | Экспорт в | MassLynx Сохранить время дрейфа).
- Верните инструмент в режим TOF на главной странице Tune и тщательно промойте систему с помощью 50:50 MeOH/H2O, прежде чем переходить к следующему сэмплу.
4. Обработка IMS/IMS-MS с помощью MZmine 224
ПРИМЕЧАНИЕ: MZmine 2 доступен по URL-адресу, указанному в Таблице материалов. Рекомендуется применение МЗмина 2.51. На момент подготовки данной рукописи более поздние версии не могут открывать файлы RAW из инструментов Cyclic IMS из-за изменения функции импорта.
- Импортируйте необработанные файлы, содержащие только размеры IMS и m/z (*_dt. RAW) с использованием методов Raw data | Импорт необработанных данных.
ПРИМЕЧАНИЕ: Необработанные файлы появятся в левой части главного окна MZmine. Не импортируйте оригинал *. Raw-файлы , которые по-прежнему содержат измерение времени хранения. MZmine не отличает время хранения от времени прибытия IMS, и точки данных обоих измерений будут перекрываться. - Оптимизируйте параметры рабочего процесса в репрезентативном файле, выбрав его в поле Файлы необработанных данных список.
- Оцените уровень шума в данных. Щелкните правой кнопкой мыши файл в списке Файлы необработанных данных , выберите Показать TIC и отобразите базовый пик «Хроматограмма» (BPC). Дважды щелкните по наименьшему пику, наблюдаемому глазом, чтобы отобразить его массовый спектр. Считайте, что уровень шума в данных равен уровню второго изотопа базового пика в этом спектре, и используйте это же значение для всех пороговых значений интенсивности на следующих этапах обработки.
ПРИМЕЧАНИЕ: Данные были получены с использованием квадрупольной изоляции и, таким образом, рассматриваются MZmine как MS / MS. На протяжении всей обработки MZmine обязательно работайте на уровне MS = 2. - Выполните массовое обнаружение с помощью методов raw data | | обнаружения функций Массовое обнаружение. Для данных, полученных в режиме профиля, используйте алгоритм вейвлет-преобразования . Чтобы настроить параметры алгоритмов в MZmine, нажмите на кнопку [...] рядом с алгоритмом и используйте опцию Show preview для визуализации данных при оптимизации параметров.
ПРИМЕЧАНИЕ: На этом этапе пики, выбранные алгоритмом, будут отображаться красным цветом в окне предварительного просмотра. При использовании алгоритма вейвлет-преобразования на проприетарных файлах RAW MZmine иногда ошибочно принимает точки данных профиля за центроидированные пики. Программа отобразит сообщение о том, что пользователь запускает алгоритм профиля на центроидных спектрах: проигнорируйте это сообщение и нажмите OK. - Реконструируйте извлеченные спектры подвижности ионов (EIM) для каждой массы фрагмента, используя методы raw data | | обнаружения функций Конструктор хроматограммы ADAP в списке масс «масс», сгенерированном предыдущим шагом. Поскольку входной допуск m/z на этом этапе является допуском от сканирования к сканированию, убедитесь, что он по крайней мере в 3-4 раза выше общей ожидаемой точности.
- Поскольку предыдущий шаг не имеет опции предварительного просмотра, проверьте качество пикового выбора непосредственно с помощью списка функций , который появился на правой панели главного окна MZmine. Откройте список Компоненты, выделите все строки, щелкните правой кнопкой мыши и выберите Show/XIC (диалоговое окно).. Нажмите кнопку Все , чтобы отобразить все ионы в спектре подвижности. Осмотрите выбранные пики, которые выглядят в цвете, чтобы убедиться, что нет очевидных пропущенных пиков.
- Разверните EIM, чтобы разделить m/z , которые содержат различные пики, на несколько объектов. Использование методов списка компонентов | | обнаружения функций Деконволюция хроматограммы и выбор алгоритма вейвлетов (ADAP ). Оптимизируйте алгоритм для данных с помощью опции Показать предварительный просмотр и следующих ключевых параметров: порог S/N, порог коэффициента/площади и диапазон вейвлетов RT.
ПРИМЕЧАНИЕ: Рекомендуется проверить аспект деконволюционного спектра. Используйте инструмент визуализации хроматограммы, как описано в шаге 4.2.4. Деконволюционные пики будут отображаться в цвете, а пики той же массы должны быть разделены, как показано на рисунке 6А. - Деизотоп деконволюционных EIM с помощью методов списка компонентов | Изотопы | Изотопные пики групера. Используйте ожидаемую точность прибора для значения допуска m/z и установите допуск времени прибытия на уровне 0,1 мс (отображается в MZmine как допуск времени удержания 0,1 мин), так как изотопы не разрешаются во время разделения IMS. Проверьте список характеристик: если какие-либо изотопы остались, увеличьте значения допусков.
ПРИМЕЧАНИЕ: Хотя деизотопирование теоретически может быть выполнено в любой момент обработки списка функций, важно сделать это последним, чтобы значения заряда можно было экспортировать (алгоритмы, используемые для других шагов, иногда удаляют информацию о состоянии заряда).
- Оцените уровень шума в данных. Щелкните правой кнопкой мыши файл в списке Файлы необработанных данных , выберите Показать TIC и отобразите базовый пик «Хроматограмма» (BPC). Дважды щелкните по наименьшему пику, наблюдаемому глазом, чтобы отобразить его массовый спектр. Считайте, что уровень шума в данных равен уровню второго изотопа базового пика в этом спектре, и используйте это же значение для всех пороговых значений интенсивности на следующих этапах обработки.
- При обработке нескольких спектров IMS/IMS-MS повторите обработку с этими оптимизированными параметрами. Сохраняйте одинаковые параметры для всех спектров.
- В случае нескольких спектров сгруппируйте их в одну таблицу для их экспорта; если нет, перейдите непосредственно к шагу 4.5. Чтобы сгруппировать спектры, используйте методы списка объектов | | выравнивания Присоединяйтесь к выравнивателю. Поскольку цель состоит не в том, чтобы фактически выровнять пики, используйте ограничительные значения допусков как для м/з , так и для времени прибытия. Придайте одинаковый вес обоим размерам.
- Экспортируйте окончательный список функций в файл *.csv . Использование методов списка компонентов | Экспорт/импорт | Экспорт в CSV-файл и экспорт следующих значений: Экспорт строки m/z, Время хранения строки экспорта (фактическое время прибытия IMS), Пик m/z и Пиковая высота. Используйте запятую в качестве разделителя полей.
5. TWCCSN2 центроидированных спектров IMS/IMS
ПРИМЕЧАНИЕ: В этом протоколе будет использоваться логарифмическая калибровка соответствия25,26, которая, как правило, дает лучшие результаты, чем линейная калибровка, и легко реализуется в электронной таблице или собственном скрипте обработки. Собственный сценарий (написанный на R) доступен по URL-адресу, указанному в таблице материалов.
- Выберите контрольные значения времени прибытия из сбора калибранта (см. шаг 2.3). Сделайте это вручную, используя программное обеспечение конструктора (см. Таблицу материалов), чтобы проверить аспект всех пиков калибранта IMS.
- В окне Хроматограмма откройте *_dt. RAW-файл , соответствующий калибранту.
- Для каждой точки калибровки сгенерируйте EIM с помощью Display | Массовый вариант.
- Проверьте профиль EIM. Если некоторые из них плохо определены, сгладьте их с помощью process | Плавный вариант (так как наилучшие результаты обычно получаются с помощью алгоритма Савицкого-Голея, плавный 2 раза на 3 бункерах). Отчет о значениях вершин в электронной таблице.
ПРИМЕЧАНИЕ: Поскольку контрольные точки обычно приобретаются с помощью устройств DTIMS с низким разрешением, некоторые мультимодальные распределения могут появляться в циклических IMS в зависимости от калибрантов. Удалите из списка калибровки все пики, представляющие такое распределение.
- Вычисление логарифмических параметров подгонки из калибрантов.
- Для всех точек калибровки рассчитайте следующее.
- Рассчитайте время дрейфа с помощью Eq (1):
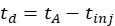 (1)
(1)
с td - время дрейфа, tA - измеренное время прибытия и tinj - время инъекции в ячейку IMS (все в мс).
ПРИМЕЧАНИЕ: Для малых молекул, таких как фрагменты олигосахаридов, время мертвого времени (время полета между выходом из ячейки IMS и детектором) вариации между различными массами находится в пределах диапазона погрешности калибровки CCS и может быть проигнорировано. - Рассчитайте нейтральную массу ионов с помощью Eq (2):
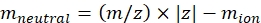 (2)
(2)
с z — состояние заряда иона, а мион — масса контриона (в Da). Используйте точные массы, чтобы избежать введения неопределенности. Если вместо контриона есть потеря атома, используйте отрицательные значения мионов (например, для [M-H]-mneutral = (m/z) * |z| - (- 1,007276) = (m/z) * |z| + 1,007276). - Рассчитайте параметр CCS с помощью Eq (3):
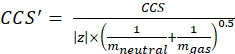 (3)
(3)
с CCS - значение эталонной дрейфующей трубки DTCCSN2 (в нм2), а mgas - масса дрейфующего газа (в Da; например, для азота: mgas = 28,01 Da). - Вычислите параметр td' с помощью Eq (4):
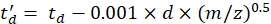 (4)
(4)
с d задержка запуска детектора используется экспериментально для коррекции мертвого времени (обычно ~1,5 мс). - Рассчитайте логарифм вышеуказанных параметров:
ln (CCS') и ln (t'd)
- Рассчитайте время дрейфа с помощью Eq (1):
- Выполните линейную регрессию для определения коэффициента R2 и параметров x и y логарифмического соответствия (с x наклоном и ln(y) перехватом) с помощью Eq (5):
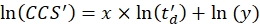 (5)
(5)
ПРИМЕЧАНИЕ: Пользователь может построить график значений ln(CCS') против ln(td') для визуальной проверки результатов калибровки, хотя это необязательно.
- Для всех точек калибровки рассчитайте следующее.
- Примените калибровку к экспериментальным данным, чтобы откалибровать пики, выбранные MZmine для каждого спектра IMS/IMS, экспортированного в файл *.csv. Для каждой точки вычислите следующее.
- Рассчитайте время дрейфа с помощью Eq (6):
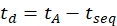 (6)
(6)
с tseq - временем, предшествующим окончательному разделению IMS (значение "Time Abs", указанное на этапе 3.4.6).
ПРИМЕЧАНИЕ: При калибровке нескольких спектров IMS/IMS, полученных с различными последовательностями, тщательно проверьте значения tseq. - Рассчитайте нейтральную массу ионов с помощью Eq (7):
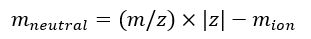 (7)
(7) - Рассчитайте параметры td' и td'' с помощью Eq (8) и Eq (9):
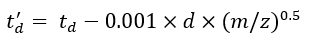 (8)
(8)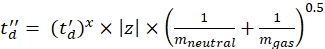 (9)
(9) - Рассчитайте конечные калиброванные значения CCS (TWCCSN2 в нм2), используя Eq (10):
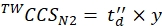 (10)
(10)
ПРИМЕЧАНИЕ: Хотя этап 5.2.2. дает ln(y) в качестве перехвата, y должен использоваться для получения конечного значения CCS. Не забудьте применить экспоненциальную функцию.
- Рассчитайте время дрейфа с помощью Eq (6):
- Проверьте точность калибровки, применив калибровку ко второму приобретению калибровочного раствора, полученного на этапе 2.4.
ПРИМЕЧАНИЕ: Калибровка должна давать результаты с погрешностью ~1-2%.
Результаты
Арабиноксилан пентасахарид, XA2XX, был выбран в качестве примера для иллюстрации этого протокола. Это соединение коммерчески доступно, но только в виде смеси с другим арабиноксилановым пентасахаридом, XA3XX (чистый XA3XX также коммерчески доступен). Структуры XA2XX и XA3...
Обсуждение
Циклический IMS серии SELECT является мощным инструментом, который позволяет выбрать определенную популяцию ионов — заданной m/z и подвижности ионов — без необходимости хроматографического разделения. Прибор предоставляет возможность создания карты бидименсионной фрагментации это...
Раскрытие информации
У авторов нет конфликта интересов для раскрытия.
Благодарности
S.O. благодарен Французскому национальному исследовательскому агентству за финансирование его докторской диссертации (грант ANR-18-CE29-0006).
Материалы
| Name | Company | Catalog Number | Comments |
| 33-α-L- plus 23-α-L-Arabinofuranosyl-xylotetraose (XA3XX/XA2XX) mixture | Megazyme Ltd., Wicklow, Ireland | O-XAXXMIX | XA2XX + XA3XX mixture |
| 33-α-L-Arabinofuranosyl-xylotetraose (XA3XX) | Megazyme Ltd., Wicklow, Ireland | O-XA3XX | Pure XA3XX standard |
| Eppendorf Safe-Lock Tubes, 1.5 mL, Eppendorf Quality, colorless, 1,000 tubes | Eppendorf, Hamburg, Germany | 0030120086 | Used to prepare the carbohydrate stock solution and dilution |
| FALCON 50 mL Polypropylene Conical Tube 30 x 115 mm | Corning Science México S.A. de C.V., Reynosa, Tamaulipas, Mexico | 352070 | Used to prepare the aqueous stock solution of 100 mM LiCl |
| Lithium Chloride (ACS reagent, ≥99 %) | Sigma-Aldrich Inc., Saint Quentin Fallavier, France | 310468 | Used to dope the sample with lithium |
| Major Mix IMS/Tof Calibration Kit | Waters Corp., Wilmslow, UK | 186008113 | Calibration solution for MS and IMS |
| MassLynx 4.2 SCN1016 Release 6 (Waters Embedded Analyser Platform for Cyclic IMS 2.9.1 Release 9) | Waters Corp., Wilmslow, UK | 721022377 | Cyclic IMS vendor software for instrument control and data processing |
| Methanol for HPLC PLUS Gradient grade | Carlo-Erba Reagents, Val de Reuil, France | 412383 | High-purity solvent |
| MS Leucine Enkephaline Kit | Waters Corp., Wilmslow, UK | 700002456 | Reference compound used for tuning of the mass spectrometer |
| SCHOTT DURAN 100 mL borosilicate glass bottle | VWR INTERNATIONAL, Radnor, Pennsylvania, US | 218012458 | Used to prepare the solution of 500 µM LiCl in 50:50 MeOH/Water |
| SELECT SERIES Cyclic IMS | Waters Corp., Wilmslow, UK | 186009432 | Ion mobility-mass spectrometer equipped with a cylic IMS cell |
| Website: http://mzmine.github.io/ | MZmine Development Team | - | Link to download the MZmine software |
| Website: https://github.com/siollivier/IM-MN | INRAE, UR BIA, BIBS Facility, Nantes, France | - | Link to an in-house R script containing a CCS calibration function |
Ссылки
- Allard, P. -. M., et al. Integration of molecular networking and in-silico MS/MS fragmentation for natural products dereplication. Analytical Chemistry. 88 (6), 3317-3323 (2016).
- Wang, M., et al. Mass spectrometry searches using MASST. Nature Biotechnology. 38 (1), 23-26 (2020).
- David, M., Fertin, G., Rogniaux, H., Tessier, D. SpecOMS: a full open modification search method performing all-to-all spectra comparisons within minutes. Journal of Proteome Research. 16 (8), 3030-3038 (2017).
- Dührkop, K., et al. SIRIUS 4: a rapid tool for turning tandem mass spectra into metabolite structure information. Nature Methods. 16 (4), 299-302 (2019).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Nothias, L. -. F., et al. Feature-based molecular networking in the GNPS analysis environment. Nature Methods. 17 (9), 905-908 (2020).
- Gray, C. J., et al. Advancing solutions to the Carbohydrate Sequencing Challenge. Journal of the American Chemical Society. 141 (37), 14463-14479 (2019).
- Ropartz, D., et al. Online coupling of high-resolution chromatography with extreme UV photon activation tandem mass spectrometry: Application to the structural investigation of complex glycans by dissociative photoionization. Analytica Chimica Acta. 933, 1-9 (2016).
- Wolff, J. J., et al. Negative electron transfer dissociation of glycosaminoglycans. Analytical Chemistry. 82 (9), 3460-3466 (2010).
- Ropartz, D., et al. Charge transfer dissociation of complex oligosaccharides: comparison with collision-induced dissociation and extreme ultraviolet dissociative photoionization. Journal of the American Society for Mass Spectrometry. 27 (10), 1614-1619 (2016).
- Morelle, W., et al. Fragmentation characteristics of permethylated oligosaccharides using a matrix-assisted laser desorption/ionization two-stage time-of-flight (TOF/TOF) tandem mass spectrometer. Rapid Communications in Mass Spectrometry. 18 (22), 2637-2649 (2004).
- Gabelica, V., Marklund, E. Fundamentals of ion mobility spectrometry. Current Opinion in Chemical Biology. 42, 51-59 (2018).
- Gabelica, V., et al. Recommendations for reporting ion mobility mass spectrometry measurements. Mass Spectrometry Reviews. 38 (3), 291-320 (2019).
- Hernandez-Mesa, M., et al. Interlaboratory and interplatform study of steroids collision cross section by traveling wave ion mobility spectrometry. Analytical Chemistry. 92 (7), 5013-5022 (2020).
- Koeniger, S. L., et al. An IMS-IMS analogue of MS-MS. Analytical Chemistry. 78 (12), 4161-4174 (2006).
- Merenbloom, S. I., Koeniger, S. L., Valentine, S. J., Plasencia, M. D., Clemmer, D. E. IMS−IMS and IMS−IMS−IMS/MS for separating peptide and protein fragment ions. Analytical Chemistry. 78 (8), 2802-2809 (2006).
- Eldrid, C., Thalassinos, K. Developments in tandem ion mobility mass spectrometry. Biochemical Society Transactions. 48 (6), 2457-2466 (2020).
- Giles, K., et al. A cyclic ion mobility-mass spectrometry system. Analytical Chemistry. 91 (13), 8564-8573 (2019).
- Merenbloom, S. I., Glaskin, R. S., Henson, Z. B., Clemmer, D. E. High-resolution ion cyclotron mobility spectrometry. Analytical Chemistry. 81 (4), 1482-1487 (2009).
- Ollivier, S., et al. Anomeric retention of carbohydrates in multistage cyclic ion mobility (IMSn): de novo structural elucidation of enzymatically produced mannosides. Analytical Chemistry. 93 (15), 6254-6261 (2021).
- Ollivier, S., Fanuel, M., Rogniaux, H., Ropartz, D. Molecular networking of high-resolution tandem ion mobility spectra: a structurally relevant way of organizing data in glycomics. Analytical Chemistry. 93 (31), 10871-10878 (2021).
- Aron, A. T., et al. Reproducible molecular networking of untargeted mass spectrometry data using GNPS. Nature Protocols. 15 (6), 1954-1991 (2020).
- McKenna, K. R., Li, L., Krishnamurthy, R., Liotta, C. L., Fernández, F. M. Organic acid shift reagents for the discrimination of carbohydrate isobars by ion mobility-mass spectrometry. The Analyst. 145 (24), 8008-8015 (2021).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11, 395 (2010).
- Ruotolo, B. T., Benesch, J. L. P., Sandercock, A. M., Hyung, S. -. J., Robinson, C. V. Ion mobility-mass spectrometry analysis of large protein complexes. Nature Protocols. 3 (7), 1139-1152 (2008).
- Bush, M. F., Hall, Z., Giles, K., Hoyes, J., Robinson, C. V., Ruotolo, B. T. Collision cross sections of proteins and their complexes: a calibration framework and database for gas-phase structural biology. Analytical Chemistry. 82 (22), 9557-9565 (2010).
- Ropartz, D., et al. Structure determination of large isomeric oligosaccharides of natural origin through multipass and multistage cyclic traveling-wave ion mobility mass spectrometry. Analytical Chemistry. 91 (18), 12030-12037 (2019).
- Tolmachev, A. V., et al. Characterization of ion dynamics in structures for lossless ion manipulations. Analytical Chemistry. 86 (18), 9162-9168 (2014).
- Arndt, J. R., et al. High-resolution ion-mobility-enabled peptide mapping for high-throughput critical quality attribute monitoring. Journal of the American Society for Mass Spectrometry. 32 (8), 2019-2032 (2021).
- Le Fèvre, A., Dugourd, P., Chirot, F. Exploring conformational landscapes using trap and release tandem ion mobility spectrometry. Analytical Chemistry. 93 (9), 4183-4190 (2021).
- Ohshimo, K., He, X., Ito, R., Misaizu, F. Conformer separation of dibenzo-crown-ether complexes with Na+ and K+ ions studied by cryogenic ion mobility-mass spectrometry. The Journal of Physical Chemistry A. 125 (17), 3718-3725 (2021).
- Purves, R. W., Barnett, D. A., Ells, B., Guevremont, R. Gas-phase conformers of the [M + 2H]2+ ion of bradykinin investigated by combining high-field asymmetric waveform ion mobility spectrometry, hydrogen/deuterium exchange, and energy-loss measurements. Rapid Communications in Mass Spectrometry. 15 (16), 1453-1456 (2001).
- Ujma, J., et al. Cyclic ion mobility mass spectrometry distinguishes anomers and open-ring forms of pentasaccharides. Journal of the American Society for Mass Spectrometry. 30 (6), 1028-1037 (2019).
- Warnke, S., Faleh, A. B., Scutelnic, V., Rizzo, T. R. Separation and identification of glycan anomers using ultrahigh-resolution ion-mobility spectrometry and cryogenic ion spectroscopy. Journal of The American Society for Mass Spectrometry. 30 (11), 2204-2211 (2019).
- Williamson, D. L., Bergman, A. E., Nagy, G. Investigating the structure of α/β carbohydrate linkage isomers as a function of group I metal adduction and degree of polymerization as revealed by cyclic ion mobility separations. Journal of the American Society for Mass Spectrometry. 32 (10), 2573-2582 (2021).
- Myers, O. D., Sumner, S. J., Li, S., Barnes, S., Du, X. One step forward for reducing false positive and false negative compound identifications from mass spectrometry metabolomics data: new algorithms for constructing extracted ion chromatograms and detecting chromatographic peaks. Analytical Chemistry. 89 (17), 8696-8703 (2017).
- Marchand, A., Livet, S., Rosu, F., Gabelica, V. Drift tube ion mobility: how to reconstruct collision cross section distributions from arrival time distributions. Analytical Chemistry. 89 (23), 12674-12681 (2017).
- Davis, D. M., et al. Analysis of ion mobility spectra for mixed vapors using Gaussian deconvolution. Analytica Chimica Acta. 289 (3), 263-272 (1994).
- Polasky, D. A., Dixit, S. M., Fantin, S. M., Ruotolo, B. T. CIUSuite 2: next-generation software for the analysis of gas-phase protein unfolding data. Analytical Chemistry. 91 (4), 3147-3155 (2019).
- Salbo, R., et al. Traveling-wave ion mobility mass spectrometry of protein complexes: accurate calibrated collision cross-sections of human insulin oligomers. Rapid Communications in Mass Spectrometry. 26 (10), 1181-1193 (2012).
- Gelb, A. S., Jarratt, R. E., Huang, Y., Dodds, E. D. A study of calibrant selection in measurement of carbohydrate and peptide ion-neutral collision cross sections by traveling wave ion mobility spectrometry. Analytical Chemistry. 86 (22), 11396-11402 (2014).
- Richardson, K., Langridge, D., Dixit, S. M., Ruotolo, B. T. An improved calibration approach for traveling wave ion mobility spectrometry: robust, high-precision collision cross sections. Analytical Chemistry. 93 (7), 3542-3550 (2021).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены