É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Usando um espectrômetro de mobilidade de íons cíclicos para experimentos de mobilidade de íons tandem
Neste Artigo
Resumo
A espectrometria de mobilidade de íons (IMS) é um complemento interessante à espectrometria de massa para a caracterização de biomoléculas, notadamente por ser sensível ao isomerismo. Este protocolo descreve um experimento tandem IMS (IMS/IMS), que permite o isolamento de uma molécula e a geração dos perfis de mobilidade de seus fragmentos.
Resumo
A caracterização precisa das estruturas químicas é importante para entender seus mecanismos biológicos subjacentes e propriedades funcionais. A espectrometria de massa (MS) é uma ferramenta popular, mas nem sempre é suficiente para desvendar completamente todas as características estruturais. Por exemplo, embora os carboidratos sejam biologicamente relevantes, sua caracterização é complicada por inúmeros níveis de isomerismo. A espectrometria de mobilidade de íons (IMS) é um complemento interessante porque é sensível às conformações de íons e, portanto, ao isomerismo.
Além disso, os avanços recentes melhoraram significativamente a técnica: a última geração de instrumentos IMS cíclicos oferece recursos adicionais em comparação com instrumentos lineares de IMS, como um aumento da potência de resolução ou a possibilidade de realizar experimentos de mobilidade de íons tandem (IMS/IMS). Durante o IMS/IMS, um íon é selecionado com base em sua mobilidade de íons, fragmentado e reanalisado para obter informações sobre mobilidade de íons sobre seus fragmentos. Trabalhos recentes mostraram que os perfis de mobilidade dos fragmentos contidos em tais dados IMS/IMS podem atuar como uma impressão digital de um glicano específico e podem ser usados em uma estratégia de rede molecular para organizar conjuntos de dados glicómicos de forma estruturalmente relevante.
O objetivo deste protocolo é, portanto, descrever como gerar dados IMS/IMS, desde a preparação da amostra até a calibração final da Seção Transversal de Colisão (CCS) da dimensão de mobilidade de íons que produz espectros reprodutíveis. Tomando o exemplo de um glycan representativo, este protocolo mostrará como construir uma sequência de controle IMS/IMS em um instrumento IMS Cíclico, como explicar essa sequência de controle para traduzir o tempo de chegada do IMS em tempo de deriva (ou seja, o tempo efetivo de separação aplicado aos íons) e como extrair as informações relevantes de mobilidade dos dados brutos. Este protocolo foi projetado para explicar claramente os pontos críticos de um experimento IMS/IMS e, assim, ajudar novos usuários de IMS cíclicos a realizar aquisições simples e reprodutíveis.
Introdução
A caracterização química completa das biomoléculas é fundamental para entender suas propriedades biológicas e funcionais subjacentes. Para isso, as ciências da "omics" têm se desenvolvido nos últimos anos, visando a caracterização em larga escala de estruturas químicas em concentrações biológicas. Em proteômica e metabolômica, a MS tornou-se uma ferramenta central para desvendar a heterogeneidade estrutural encontrada na mídia biológica - notadamente graças à sua sensibilidade e capacidade de fornecer informações estruturais através de tandem MS (MS/MS). Nas estratégias de MS/MS, um íon é selecionado de acordo com sua massa, depois fragmentado, e finalmente, as massas de seus fragmentos são adquiridas para estabelecer uma impressão digital da molécula. Os espectros de MS/MS podem, em particular, ser usados para combinar bancos de dados espectrais1,2 ou reconstruir provisoriamente as estruturas-mãe3,4. Sob a suposição de que espectros semelhantes pertencem a compostos semelhantes, os dados de MS/MS também podem ser usados para construir redes moleculares (MNs) conectando espécies relacionadas através de um escore de similaridade5,6.
No entanto, devido à propriedade inerente da EM para detectar a relação massa-carga (m/z) de íons, a técnica é cega para uma série de características estruturais que se enquadram na faixa do isomerismo (estéreo). Por exemplo, os carboidratos são feitos de várias subunidades de monossacarídeos, muitas das quais são estereólogos ou mesmo epimers (por exemplo, Glc vs. Gal ou Glc vs. Man). Essas subunidades estão ligadas por ligações glicosidicas, que podem diferir pela posição da ligação (regioisomerismo) e pela configuração estérica do carbono anomérico (anomerismo). Essas características dificultam a distinção entre isômeros de carboidratos7, e apenas o regioisomerismo pode ser abordado usando métodos de ativação de alta energia8,9,10. Embora a derivatização seja uma opção para interromper a equivalência de grupos estereosméricos11, requer uma preparação extensiva da amostra. Outra opção mais simples é acoplamento de ESM com uma dimensão analítica sensível ao isômerismo, como o IMS.
Como este protocolo é projetado para usuários que já estão familiarizados com os conceitos básicos do IMS, e como avaliações detalhadas estão disponíveis em outros lugares12,13, apenas uma breve visão geral dos princípios do IMS é dada aqui. IMS é um método de separação em fase de gás que se baseia na interação de íons com um gás tampão e um campo elétrico, separando íons de acordo com suas conformações de fase gasosa. Diferentes princípios do IMS acoplados à MS podem ser encontrados em instrumentos comerciais: alguns operam em campos elétricos alternados de alta e baixa (IMS assimétricos de campo, FAIMS), enquanto a maioria opera dentro do limite de campo baixo — notavelmente o tubo de deriva IMS (DTIMS, campo elétrico decrescente linear), imS de onda itinerante (TWIMS, ondas potenciais simétricas) e IMS preso (TIMS, alto fluxo de íons de captura de gás tampão contra campos elétricos)13 . Os métodos de campo baixo permitem o acesso a um chamado CCS, uma propriedade do par de gás íon que representa a superfície (em Å2 ou nm2) do íon que interage com o gás tampão durante a separação. O CCS é teoricamente independente de instrumentos e, portanto, é útil para gerar dados que podem ser reproduzidos entre diferentes laboratórios14. As separações de mobilidade de íons podem ser impactadas por vários parâmetros e, notadamente, por flutuações da pressão do gás e da temperatura do gás na célula de mobilidade. A calibração do CCS é uma forma de remediar isso, pois tanto o calibrante quanto a espécie de interesse serão igualmente afetados13. No entanto, é obrigatório instalar o instrumento em uma sala controlada pela temperatura e ter um sistema de controle de pressão de gás confiável.
Uma evolução interessante do IMS é o IMS/IMS, que foi introduzido pela primeira vez em 2006 pelo grupo de Clemmer como um analógico de MS/MS15,16. No IMS/IMS, um íon de interesse é isolado seletivamente com base em sua mobilidade de íons; em seguida, é ativado (até possível fragmentação), e uma nova análise do IMS do íon ativado ou fragmentos é realizada. No primeiro design instrumental, duas células IMS foram colocadas em série, separadas por um funil de íons onde a ativação estava. Desde então, embora uma série de configurações IMS/IMS tenham sido propostas (para revisão, consulte Eldrid e Thalassinos17), o primeiro espectrômetro de massa comercial com capacidade IMS/IMS só ficou disponível em 201918. Este instrumento melhorou substancialmente o conceito inicial, combinando-o com outro avanço tecnológico: um design cíclico da célula IMS.
A célula IMS cíclica teoricamente permite aumentar quase infinitamente o comprimento do caminho de deriva e, assim, o poder de resolução do instrumento19. Isso foi conseguido por meio de uma geometria de instrumento particular, onde a célula Cíclica TWIMS é colocada ortogonalmente ao eixo óptico de íon principal. Uma região de matriz multifuncional na entrada da célula IMS permite controlar a direção do caminho de íon: (i) enviar íons para os lados para a separação do IMS, (ii) para a frente para detecção de MS ou (iii) para trás da célula IMS a ser armazenada em uma célula de prearray. A partir desta célula de loja de prearray, os íons podem ser ativados e os fragmentos reinjetados na célula IMS para medição de mobilidade de íons, uma abordagem que tem sido usada com sucesso para caracterizar estereomers20. Em última análise, os dados coletados contêm mobilidade de íons e informações m/z para o precursor e seus fragmentos.
Em uma publicação recente que utilizou este desenho cíclico para análises glican (Ollivier et al.21), mostramos que o perfil de mobilidade dos fragmentos contidos nesses dados IMS/IMS age como uma impressão digital de uma biomolécula que pode ser usada em uma estratégia de rede molecular. A rede resultante, chamada IM-MN, levou à organização de conjuntos de dados glicômicos de forma estruturalmente relevante, enquanto a rede construída exclusivamente a partir de dados MS/MS (MS-MN) revelou poucas informações. Para complementar esta publicação e ajudar os usuários do IMS Cíclico a implementar este fluxo de trabalho, este protocolo fornece uma descrição completa do protocolo usado para coletar os dados. Este protocolo se concentra apenas na geração dos dados IMS/IMS que os usuários podem usar para criar redes IM-MN (ver21)— ou para qualquer outra aplicação de sua escolha. A construção do IM-MN não será considerada aqui, pois já existem protocolos para redes moleculares. Destacam-se os pontos cruciais que devem ser seguidos para gerar aquisições valiosas e reprodutíveis do IMS/IMS. Tomando o exemplo de um dos oligossacarídeos estudados por Ollivier et al. 21, as seguintes etapas são detalhadas: (i) preparação da amostra, (ii) ajuste do instrumento IMS Cíclico, (iii) pico automatizado de captação dos dados e (iv) calibração ccs.
Protocolo
NOTA: Uma visão geral do protocolo é fornecida na Figura 1. Os parâmetros utilizados para os experimentos descritos no presente protocolo podem ser encontrados na Tabela Suplementar S1 e Tabela Suplementar S2.
1. Preparação da solução amostral
NOTA: O protocolo é descrito usando um pentasacarídeo arabinoxylan (23-α-L-arabinofuranosyl-xylotetraose ou XA2XX; veja a Tabela dos Materiais) como exemplo.
- Preparação do solvente: 500 μM LiCl em 50:50 H2O:MeOH (vol./vol.).
- Prepare uma solução de estoque de 100 mM de cloreto de lítio (Cicv) em H2O pesando 212 mg de Cicílica e adicione 50 mL de água deionizada de alta pureza (H2O) em um tubo cônico de polipropileno de 50 mL. Agite até dissolver completamente.
NOTA: O solvente é dopado com um sal de lítio para promover a formação de adutos [M+Li]+ na fonte de íon do espectrômetro, pois geralmente produz espectros de fragmentação de melhor qualidade em comparação com outros adutos alcalinos. O uso de LiCl é recomendado porque os ácidos orgânicos (e, portanto, seus sais) foram encontrados anteriormente para impactar os perfis do IMS23. - Em uma garrafa de vidro, diluir a solução de estoque liCl 200x: a 250 μL da solução de estoque, adicione 24,75 mL de H2O. Adicione 25 mL de metanol (MeOH) para alcançar uma concentração final de LiCl de 500 μM em 50:50 H2O:MeOH (v/v). Sonicato por 2 minutos para desgas o solvente.
NOTA: MeOH apresenta um risco à saúde (H225, H301, H311, H331, H370); manipular sob um capuz extrator usando um jaleco, luvas e proteção ocular. Uma proporção de 50:50 MeOH/H2O (v/v) parece ser o melhor solvente para a ionização de oligossacarídeos; no entanto, o MeOH pode ser substituído por acetonitrila (ACN) se necessário.
- Prepare uma solução de estoque de 100 mM de cloreto de lítio (Cicv) em H2O pesando 212 mg de Cicílica e adicione 50 mL de água deionizada de alta pureza (H2O) em um tubo cônico de polipropileno de 50 mL. Agite até dissolver completamente.
- Em um tubo de polipropileno de 1,5 mL, pesa 1 mg do carboidrato. Dissolva-se com um volume apropriado de 500 μM LiCl para atingir uma concentração de 1 mg/mL. Diluir para uma concentração final de 10 μg/mL em 50:50 MeOH/H2O + 500 μM LiCl. Armazenar a 4 °C.
NOTA: A concentração de 10 μg/mL foi escolhida para otimizar o sinal sobre todos os íons fragmentados durante o IMS/IMS-MS (isto é para um composto puro; aumentar a concentração ao trabalhar em misturas). Para a aquisição de espectros de referência IMS/IMS, não dilua ainda mais a amostra: a saturação do detector de MS antes da fragmentação é esperada, embora o instrumento ofereça opções para corrigi-lo (ver passo 3.2.).
2. Ajuste do espectrômetro de massa Cíclico IMS
NOTA: As instruções relacionadas ao software (janelas, menus e comandos) são destacadas em negrito.
- Abra o console de instrumentos a partir do software de controle de instrumentos (página de sintonização MS , veja os detalhes do software na Tabela de Materiais) e coloque o instrumento no modo Operar . Aguarde pelo menos 3 horas para que as altas tensões se estabilizem na célula IMS.
NOTA: Para a melhor reprodutibilidade, as tensões na célula IMS precisam ser completamente estabilizadas. Ligue as altas tensões e deixe o instrumento estabilizar durante a noite antes de qualquer análise cíclica do IMS. Além disso, a pressão e a temperatura na célula de mobilidade de íons devem ser mantidas o mais constante possível. Embora um retorno para a pressão esteja disponível na guia Vácuo , não há leitura disponível para a temperatura. Mantenha o instrumento em um laboratório termostatado. O instrumento utilizado neste trabalho opera a 1,75 mbar em um termostato de laboratório a 20 °C. - Configuração do instrumento IMS cíclico
NOTA: As soluções padrão devem ser infundidas usando o sistema fluido embutido para a configuração do instrumento.- Coloque os recipientes fluidos preenchidos com as normas adequadas fornecidas pelo fabricante no sistema de fluidos: Reservatório B ('Lockmass'): 10 pg/μL leucina enkephaline (LEU ENK) em 50:50 ACN/H2O + 0,1% ácido fórmico; Reservatório C ('Calibrante'): MajorMix.
NOTA: Neste protocolo, a solução de calibração MajorMix será usada para calibrar as dimensões m/z e CCS. Por razões práticas, será realizada uma calibração externa do CCS (ver o passo 5 do protocolo); portanto, também é possível usar uma mistura calibrante interna para o CCS e outro calibrante para o m/z (por exemplo, formatar de sódio ou iodeto de sódio). - Na página Tune do console Quartz, vá para a guia Fluidics . Coloque a amostra fluidica para o reservatório C e os fluidos de referência para o reservatório B. Infundir ambas as soluções consecutivamente na fonte de íons para verificar o sinal de MS.
- Execute a configuração da ADC, a configuração do detector (usando LEU ENK) e a calibração de massa (consulte a tabela de materiais para a solução de calibração) a partir da página configuração do instrumento de acordo com as instruções do fabricante.
- Coloque os recipientes fluidos preenchidos com as normas adequadas fornecidas pelo fabricante no sistema de fluidos: Reservatório B ('Lockmass'): 10 pg/μL leucina enkephaline (LEU ENK) em 50:50 ACN/H2O + 0,1% ácido fórmico; Reservatório C ('Calibrante'): MajorMix.
- Registo uma aquisição do IMS da solução de calibração com uma separação de passe único (use-a para calibração externa do IMS).
NOTA: Os parâmetros de origem de íon e de onda itinerante (TW) (altura de onda estática e velocidade de onda) devem ser mantidos constantes durante todas as aquisições (calibração e aquisições). Se o usuário não tiver conhecimento prévio dos parâmetros ideais para sua amostra, esta etapa pode ser realizada após a etapa 3 do protocolo (para adutos [M+Li]+ de oligossacarídeos neutros, os resultados representativos utilizam uma altura TW de 16 V e velocidade TW de 350 m/s, que dão os melhores resultados).- A partir da guia Fluidics , selecione a posição de defletor Amostra e infundir o calibrante (ver a tabela de materiais) na fonte de íon (usando o sistema fluido embutido) através da sonda 'Amostra' a uma taxa de fluxo de 10 μL/min.
- Configure uma sequência IMS de passe único. Na página Tune , coloque o instrumento no modo Mobility e abra a janela controle de sequência cíclica . Selecione o modo Avançado . Na guia Funções Cíclicas desta nova janela, selecione Adicionar pacote e, em seguida, Single/Multipass. Aguarde que uma sequência de eventos de mobilidade apareça na guia Sequência da mesma janela.
NOTA: Para ativar o visor em tempo real, o usuário deve aplicar os parâmetros do instrumento: clique no modo Tune in TOF ou Run in Mobility . Antes de alternar o instrumento entre os modos TOF e Mobility , é necessário abortar qualquer aquisição em execução (incluindo a exibição da página Tune ). A abundância relativa de íons pode variar entre o modo TOF e o modo Mobilidade devido a alterações nos parâmetros de transmissão de íons. - Adapte a sequência para que todos os íons calibrantes façam uma única passagem ao redor da pista cíclica do IMS. Não altere o tempo de injetar ou o Ejetar e adquirir tempo; no entanto, reduza o tempo separado para 1 ms (na guia Sequência ). Se alguns íons da mistura de calibração não se encaixarem na janela de tempo de chegada exibida, altere a sincronização do IMS com o empurrador do analisador TOF de aceleração ortogonal aumentando o número de Pushes Per Bin na guia Configurações ADC .
NOTA: Os tempos na sequência de controle controlam apenas a matriz multifuncional para gating de íon. Enquanto os íons estiverem engajados em sua primeira (ou nth) passagem ao redor da pista, eles terminarão o referido passe mesmo que a direção do TW tenha mudado na matriz nesse meio tempo. Reduzir o tempo de separação para 1 ms significa que a matriz mudará para o modo de ejeção após 1 ms. Isso garante que os íons mais rápidos não terão tempo suficiente para passar pela matriz e se engajar em uma segunda passagem antes que os íons mais lentos terminem sua primeira passagem. Portanto, todos os íons serão submetidos ao mesmo número de passes (ou seja, um passe), o que é necessário para realizar a calibração do IMS. - Registo uma aquisição de 2 minutos. Na janela Controle de sequência cíclica , clique em Adquirir para abrir a janela popup Configurações de aquisição . Insira o nome de arquivo, descrição e comprimento de aquisição (minutos) e clique em Salvar.
- Registo a aquisição de mais 2 min da solução de calibração nas mesmas condições da etapa 2.3 (use-a para verificar a qualidade da calibração do CCS). Na janela Controle de sequência cíclica , clique em Adquirir para abrir a janela popup Configurações de aquisição . Insira o nome de arquivo, descrição e comprimento de aquisição (minutos) e clique em Salvar.
- Lave bem o sistema fluido com 50:50 H2O/ACN para evitar a cristalização do calibrante na tubulação de espia.
3. Aquisição do IMS/IMS-MS
- Utilizando uma bomba de seringa, infundir a amostra (dopada de lítio) a 10 μg/mL através da sonda amostral a uma taxa de fluxo de 10 μL/min.
- Mude o instrumento para o modo TOF (a partir da página de sintonização MS ) para verificar a estabilidade do sinal. Registo a aquisição completa de MS (1 min) da amostra, que será útil para verificar o padrão isotópico e a presença de potenciais contaminantes.
NOTA: Como a concentração amostral é escolhida para obter um bom sinal de íon para os fragmentos, uma saturação TOF pode ser observada nesta etapa. A saturação do TOF pode ser identificada usando os seguintes artefatos: (i) uma resolução de MS artificialmente aumentada, (ii) uma mudança nas proporções isotópicas e (iii) uma infinidade de picos de baixa abundância entre os isótopos. Use a lente DRE (aprimoramento do intervalo dinâmico, perfil quad/ms/dre da página principal de sintonia) para atenuar a transmissão de íons e descartar a saturação no modo TOF (Figura 2A,B). - Coloque o instrumento no modo MSMS (aba Perfil Quad/MS da página principal de sintonia ) e selecione a massa do íon-alvo no campo de massa MSMS para isolamento no quadrúpole (no exemplo: m/z de 685,2, correspondente à espécie [M+Li]+ iônica da pentasaccarida arabinoxylan). Registo uma aquisição de 1 min para verificar o isolamento precursor ao processar os dados.
NOTA: Os adutos de lítio têm um isótopo a -1 Da do pico monoisotópico, que precisa ser removido da janela de seleção de MS/MS para que não interfira nas etapas de processamento. Ele pode ser removido estreitando a faixa de seleção usando os parâmetros de Resolução LM e HM na guia Perfil Quad/MS (Figura 2C). - Configure uma sequência IMS de "fatiamento" para executar uma seleção baseada em mobilidade do isômero de interesse.
- Mude o instrumento para o modo Mobilidade (ver passo 2.3.2). Na janela Controle de sequência cíclica , da guia Funções Cíclicas , selecione Adicionar pacote e, em seguida, fatiar. Aguarde que uma sequência complexa de eventos de mobilidade apareça na guia Sequência (Figura 3).
NOTA: É possível visualizar cada etapa do processo IMS/IMS: clique no evento Ejetar e Adquirir na guia Sequência . Uma vez destacado em vermelho, mova-o para a posição apropriada dentro da sequência usando os botões Para Cima e Para Baixo . - Posicione o evento Eject and Acquire logo após o primeiro evento Separado (ou seja, mova-o na linha 3 em vez da linha 8 na sequência exibida na Figura 3) e, em seguida, clique em Executar. Procure os resultados da separação inicial a ser exibida em tempo real. Aumente a duração do primeiro evento Separado para uma separação de vários passes alterando o valor de tempo para este evento na sequência até que a resolução dos picos do IMS seja satisfatória. Registo uma aquisição de 1 min para referência.
NOTA: Anote o valor de atraso de início da ADC na guia configuração da ADC : será útil verificar a qualidade do isolamento. - Clique em Pausa. Observe que os resultados da separação inicial são exibidos, embora modificações na sequência de controle não serão aplicadas até que o usuário clique em Executar novamente. Posicione o evento Eject and Acquire abaixo dos eventos Eject, Eject to Pre-Store e Hold and Eject . Ajuste a duração dos eventos de modo que o pico alvo esteja na região do Ejetar para Pré-Loja , e qualquer outro íon esteja na região de Ejetar ou Hold e Ejetar .
NOTA: A duração desses três eventos em comparação com as distribuições de tempo de chegada (ATDs) pode ser visualizada usando a barra codificada por cores abaixo do espectro de mobilidade na guia Mobilograma (Figura 3). - Posicione o evento Eject and Acquire no final da sequência, abaixo do Reinject da Pré-Loja e do segundo Eventos Separados . Clique em Executar para exibir a população selecionada.
NOTA: Como a população selecionada deixou a célula IMS, toda a separação anterior foi perdida, e está de volta a uma separação de passe único (que é desejada). - Verifique a qualidade do isolamento. Para verificar se apenas o pico de interesse foi selecionado, realize a mesma separação após a reinjeção anterior (ou seja, mesmo tempo separado ) como mostrado na Figura 4. Registo uma aquisição de 1 min para referência.
NOTA: Os usuários são encorajados a verificar a população ejetada; a janela de tempo Eject to Pré-Store deve ser nível de linha de base (Figura 4B). Para verificar isso, coloque o ADC Start Delay no modo Manual na guia Configurações ADC e digite o tempo de atraso observado na etapa 3.4.2. Registo uma aquisição de 1 min para referência. - Na aba Sequência , na coluna ao lado dos horários de evento definidos pelo usuário (a coluna Time Abs , destacada em vermelho), procure os tempos somados de todos os eventos. Tome nota do Time Abs encontrado na linha do evento Reinject from Pre-Store para realizar a calibração CCS.
- Mude o instrumento para o modo Mobilidade (ver passo 2.3.2). Na janela Controle de sequência cíclica , da guia Funções Cíclicas , selecione Adicionar pacote e, em seguida, fatiar. Aguarde que uma sequência complexa de eventos de mobilidade apareça na guia Sequência (Figura 3).
- Fragmente o pico alvo entre as duas rodadas do IMS. Alterar as tensões da etapa de reinjeção para aumentar a energia cinética dos íons, e fragmentá-los após colidir com o gás de mobilidade de íons.
- Defina a duração do evento Separado diretamente precedendo o Ejeto e Adquira para 1 ms (veja explicação na etapa 2.3.3).
- Na linha Reinject da Pré-Loja , marque a caixa Ativar e otimize a fragmentação com o controle embutido. Se o espectro for satisfatório (por exemplo, o pico base é um fragmento), proceda diretamente à etapa 3.5.4.
NOTA: Ao ativar a ativação, três tensões na linha ficarão cinzas: estas são as tensões que o usuário precisa alterar se for necessária a otimização manual das tensões (veja o próximo passo). Essas três tensões (Pré-Array Gradient, Pre-Array Bias e Array Offset) formam o gradiente usado para ativar os íons. A energia cinética dos íons aumentará com a inclinação entre o Viés Pré-Array e o Deslocamento de Matriz (ver Figura 5). Os valores padrão dos valores de Deslocamento de Viés → Gradiente → são: sem ativação 85 → 70 → 45 V; ativação máxima da função incorporada 185 → 170 → -5 V (+150 V). Após a fragmentação, não se esqueça de reajustar a transmissão de íons usando a lente DRE (diminuir a atenuação do sinal) (ver passo 3.2.). - Se a fragmentação não for satisfatória com o controle embutido, desmarque a caixa De ativação e proceda para otimizar manualmente as tensões de reinjeção. Aumente a tensão de gradiente pré-array (a tensão de viés pré-array deve ser sempre mantida 15 V abaixo do Gradiente Pré-Array) e abaixe a tensão de deslocamento de matriz (que pode ser definida como negativa) até que os resultados sejam satisfatórios.
NOTA: Ao ajustar manualmente as tensões da matriz multifuncional, o usuário pode mudar da exibição 'Mobilograma' para esquemas interativos das tensões aplicadas na matriz multifuncional (diagrama PE) para visualizar melhor as configurações de tensão (Figura 5A). - Registo uma aquisição de 2 min. Na janela pop-up de aquisição, marque a opção Reter tempo de deriva para gerar um arquivo contendo apenas os tempos de chegada vs m/z (o tempo de aquisição usado para análises cromatográficas - o tempo de retenção - é removido do arquivo). Observe que este arquivo está rotulado de *_dt. O RAW.
NOTA: Se o usuário esquecer de verificar a opção Reter tempo de deriva , ainda é possível extrair a dimensão IMS usando o software Driftscope 2.9 (Arquivo | Exportar para o masslynx | Mantenha o tempo de deriva).
- Gire o instrumento de volta para o modo TOF na página principal do Tune e enxágue completamente o sistema com 50:50 MeOH/H2O antes de prosseguir com a próxima amostra.
4. Processamento IMS/IMS-MS com MZmine 224
NOTA: MZmine 2 está disponível a partir da URL dada na Tabela de Materiais. Recomenda-se o uso de MZmine 2.51. No momento da elaboração deste manuscrito, as versões posteriores não podem abrir arquivos RAW de instrumentos IMS cíclicos devido a uma mudança na função de importação.
- Importe os arquivos brutos contendo apenas as dimensões IMS e m/z (*_dt. RAW) utilizando métodos de dados brutos | Importação de dados brutos.
NOTA: Arquivos brutos aparecerão no lado esquerdo da janela principal do MZmine. Não importe o original *. Arquivos RAW que ainda contêm a dimensão do tempo de retenção. O MZmine não distingue o tempo de retenção do tempo de chegada do IMS, e os pontos de dados de ambas as dimensões se sobreporiam. - Otimize os parâmetros do fluxo de trabalho em um arquivo representativo, selecionando-o no Arquivos de dados brutos lista.
- Avalie o nível de ruído nos dados. Clique com o botão direito do mouse no arquivo na lista de arquivos de dados Raw , selecione Mostrar TIC e exibir o pico base "cromaatograma" (BPC). Clique duas vezes no menor pico observável olho por olho para exibir seu espectro de massa. Considere o nível de ruído nos dados em torno do segundo isótopo do pico base neste espectro, e use esse mesmo valor para todos os limiares de intensidade nas etapas seguintes de processamento.
NOTA: Os dados foram adquiridos por meio do isolamento do quadrúpole e são, portanto, considerados pela MZmine como MS/MS. Durante todo o processamento de MZmine, certifique-se de trabalhar em nível de MS = 2. - Realize a detecção em massa usando métodos de dados raw | | de detecção de recursos Detecção em massa. Para dados adquiridos no modo perfil, use o algoritmo de transformação Wavelet . Para configurar os parâmetros dos algoritmos em MZmine, clique no botão [...] ao lado do algoritmo e use a opção mostrar visualização para visualizar os dados enquanto otimiza os parâmetros.
NOTA: Nesta fase, os picos selecionados pelo algoritmo aparecerão em vermelho na janela de visualização. Ao usar o algoritmo de transformação de ondas em arquivos RAW proprietários, o MZmine às vezes confunde os pontos de dados do perfil com picos centralídeos. O software exibirá uma mensagem informando que o usuário está executando um algoritmo de perfil em espectros centralóides: ignore essa mensagem e clique em OK. - Reconstrua o espectro de mobilidade de íons extraído (EIM) para cada massa de fragmento usando métodos de dados brutos | | de detecção de recursos Construtor de cromatograma ADAP na lista de massas 'massas' gerada pela etapa anterior. Como a entrada de tolerância m/z nesta fase é uma tolerância de varredura para digitalização, certifique-se de deixá-la pelo menos 3-4 vezes maior do que a precisão global esperada.
- Como a etapa anterior não tem uma opção de visualização, verifique a qualidade da escolha de pico diretamente usando a lista de recursos que apareceu no painel direito da janela MZmine principal. Abra a lista De recurso, selecione todas as linhas, clique com o botão direito do mouse e selecione Mostrar/XIC (diálogo). Clique em Tudo para exibir todos os íons no espectro de mobilidade. Inspecione os picos escolhidos que aparecem em cores para garantir que não haja picos perdidos óbvios.
- Desconvolve os EIMs para dividir o m/z que contêm diferentes picos em múltiplas características. Use métodos de lista de recursos | | de detecção de recursos Deconvolução de cromatogramas e escolha o algoritmo Wavelets (ADAP). Otimize o algoritmo para os dados usando a opção mostrar e os seguintes parâmetros-chave: limiar S/N, limite de coeficiente/área e faixa de onda RT.
NOTA: Recomenda-se verificar o aspecto do espectro desconvolvado. Use a ferramenta de visualização de cromatograma, conforme descrito na etapa 4.2.4. Os picos desconvolvados aparecerão em cores, e picos da mesma massa devem ser divididos, como apresentado na Figura 6A. - Deisótopos os EIM desconvolved usando métodos de lista de recursos | Isótopos | Garoupeiro isotópico. Use a precisão esperada do instrumento para o valor de tolerância m/z e defina a tolerância ao tempo de chegada para 0,1 ms (exibida em MZmine como tolerância ao tempo de retenção 0,1 min), pois os isótopos não são resolvidos durante a separação do IMS. Verifique a lista de recursos: se houver isótopos, aumente os valores de tolerância.
NOTA: Embora o deisotop pode teoricamente ser realizado em qualquer momento do processamento da lista de recursos, é importante fazê-lo por último para que os valores de carga possam ser exportados (os algoritmos usados para as outras etapas às vezes removerão as informações do estado de carga).
- Avalie o nível de ruído nos dados. Clique com o botão direito do mouse no arquivo na lista de arquivos de dados Raw , selecione Mostrar TIC e exibir o pico base "cromaatograma" (BPC). Clique duas vezes no menor pico observável olho por olho para exibir seu espectro de massa. Considere o nível de ruído nos dados em torno do segundo isótopo do pico base neste espectro, e use esse mesmo valor para todos os limiares de intensidade nas etapas seguintes de processamento.
- Se processar vários espectros IMS/IMS-MS, repita o processamento com esses parâmetros otimizados. Mantenha os mesmos parâmetros para todos os espectros.
- No caso de vários espectros, agrupa-os em uma única tabela para exportá-los; se não, pule diretamente para a etapa 4.5. Para agrupar os espectros, use métodos de lista de recursos | | de alinhamento Junte-se ao alinhador. Como o objetivo não é realmente alinhar os picos, use valores de tolerância restritivos tanto para m/z quanto para o tempo de chegada. Dê o mesmo peso para ambas as dimensões.
- Exporte a lista final de recursos para um arquivo *.csv . Use métodos de lista de recursos | | de exportação/importação Exportar para o arquivo CSV e exportar os seguintes valores: Linha de exportação m/z, tempo de retenção da linha de exportação (o tempo real de chegada do IMS), pico m/z e altura do pico. Use uma círgula como separador de campo.
5. TWCCSN2 do espectro IMS/IMS centralado
NOTA: Neste protocolo, será utilizada uma calibração de ajuste logarítmico25,26, que tende a dar melhores resultados do que a calibração linear e é fácil de implementar em uma planilha ou em um script de processamento interno. Um script interno (escrito em R) está disponível na URL dada na Tabela de Materiais.
- Escolha os valores de tempo de chegada de referência da aquisição calibrante (ver passo 2.3). Faça isso manualmente usando o software de construção (veja a Tabela de Materiais) para verificar o aspecto de todos os picos calibradores do IMS.
- Na janela cromatograma , abra o _dt. Arquivo RAW correspondente ao calibrante.
- Para cada ponto de calibração, gere o EIM usando o Display | Opção de massa .
- Verifique o perfil dos EIMs. Se alguns estiverem mal definidos, suavize-os usando o Processo | Opção suave (como os melhores resultados são tipicamente obtidos com o algoritmo Savitzky-Golay, suave 2 vezes mais de 3 caixas). Denuncie os valores do ápice em uma planilha.
NOTA: Como os pontos de referência são geralmente adquiridos usando dispositivos DTIMS de baixa resolução, algumas distribuições multimodais podem aparecer no IMS Cíclico, dependendo dos calibrantes. Remova qualquer pico apresentando tal distribuição da lista de calibração.
- Calcule os parâmetros de ajuste logarítmicos dos calibrantes.
- Para todos os pontos de calibração, calcule o seguinte.
- Calcule o tempo de deriva usando Eq (1):
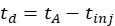 (1)
(1)
com td o tempo de deriva, tA o tempo de chegada medido, e tinj o tempo de injeção na célula IMS (tudo em ms).
NOTA: Para moléculas pequenas, como fragmentos de oligossacarídeos, a hora de voo (tempo de voo entre a saída da célula IMS e o detector) varia entre diferentes massas está dentro da faixa de erro da calibração do CCS e pode ser ignorada. - Calcular a massa neutra dos íons usando Eq (2):
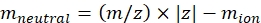 (2)
(2)
com z o estado de carga do íon, e mion a massa do contra-íon (em Da). Use massas exatas para evitar a introdução da incerteza. Se houver perda de átomo em vez de um contra-íon, use valores de mion negativos (por exemplo, para [M-H]-mneutral = (m/z) * |z| - (- 1,007276) = (m/z) * |z| + 1,007276). - Calcular o parâmetro ccs usando Eq (3):
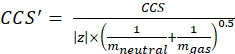 (3)
(3)
com CCS o tubo de deriva de referência valor DTCCSN2 (em nm2), e mgas a massa do gás de deriva (em Da; ex. para nitrogênio: mgas = 28,01 Da). - Calcule o parâmetro td' usando Eq (4):
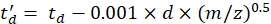 (4)
(4)
com d o detector start delay usado experimentalmente para corrigir para o tempo morto (tipicamente ~1,5 ms). - Calcular logaritmo dos parâmetros acima:
In (CCS') e in (t'd)
- Calcule o tempo de deriva usando Eq (1):
- Realize uma regressão linear para determinar o coeficiente R2 e os parâmetros x e y do ajuste logarítmico (com x a inclinação e in(y) a interceptação) usando Eq (5):
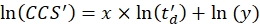 (5)
(5)
NOTA: O usuário pode traçar os valores ln(CCS) vs(td') para verificar visualmente os resultados da calibração, embora isso seja opcional.
- Para todos os pontos de calibração, calcule o seguinte.
- Aplique a calibração aos dados experimentais para calibrar os picos escolhidos pelo MZmine para cada espectro IMS/IMS exportado para o arquivo *.csv. Para cada ponto, calcule o seguinte.
- Calcule o tempo de deriva usando Eq (6):
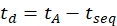 (6)
(6)
com tseq o tempo anterior à separação final do IMS (o valor 'Time Abs' observado na etapa 3.4.6).
NOTA: Se calibrar vários espectros IMS/IMS adquiridos com diferentes sequências, verifique cuidadosamente os valores do tseq . - Calcular a massa neutra dos íons usando Eq (7):
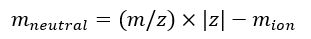 (7)
(7) - Calcule os parâmetros td' e td'' utilizando Eq (8) e Eq (9):
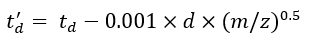 (8)
(8)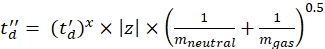 (9)
(9) - Calcule os valores ccs calibrados finais (TWCCSN2 em nm2) usando Eq (10):
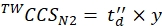 (10)
(10)
NOTA: Apesar do passo 5.2.2. dá ln(y) como a interceptação, y deve ser usado para obter o valor ccs final. Não se esqueça de aplicar uma função exponencial.
- Calcule o tempo de deriva usando Eq (6):
- Verifique a precisão da calibração aplicando a calibração à segunda aquisição da solução de calibração adquirida na etapa 2.4.
NOTA: A calibração deve produzir resultados com um erro de ~1-2%.
Resultados
Um pentasacarídeo arabinoxylan, XA2XX, foi escolhido como exemplo para ilustrar este protocolo. Este composto está comercialmente disponível, mas apenas como uma mistura com outra pentasacarida arabinoxylan, XA3XX (XA3XX puro também está disponível comercialmente). As estruturas de XA2XX e XA3XX são dadas em Figura Suplementar S1. Como a razão de XA2XX e XA3XX na mistura comercial é ~50:50, uma solução a 20 μg/mL da ...
Discussão
O SELECT SERIES Cyclic IMS é uma ferramenta poderosa que permite selecionar uma população de íons definida — de uma determinada mobilidade m/z e íon — sem a necessidade de separação cromatográfica a montante. O instrumento oferece a possibilidade de gerar um mapa de fragmentação bidimensional dessa população de íons, a partir do qual podem ser extraídos espectros de MS/MS e IMS/IMS. No entanto, o usuário deve observar vários pontos críticos que requerem atenção durante o processo experimen...
Divulgações
Os autores não têm conflito de interesses para divulgar.
Agradecimentos
S.O. agradece à Agência Nacional de Pesquisa francesa por financiar seu Doutorado (conceder ANR-18-CE29-0006).
Materiais
| Name | Company | Catalog Number | Comments |
| 33-α-L- plus 23-α-L-Arabinofuranosyl-xylotetraose (XA3XX/XA2XX) mixture | Megazyme Ltd., Wicklow, Ireland | O-XAXXMIX | XA2XX + XA3XX mixture |
| 33-α-L-Arabinofuranosyl-xylotetraose (XA3XX) | Megazyme Ltd., Wicklow, Ireland | O-XA3XX | Pure XA3XX standard |
| Eppendorf Safe-Lock Tubes, 1.5 mL, Eppendorf Quality, colorless, 1,000 tubes | Eppendorf, Hamburg, Germany | 0030120086 | Used to prepare the carbohydrate stock solution and dilution |
| FALCON 50 mL Polypropylene Conical Tube 30 x 115 mm | Corning Science México S.A. de C.V., Reynosa, Tamaulipas, Mexico | 352070 | Used to prepare the aqueous stock solution of 100 mM LiCl |
| Lithium Chloride (ACS reagent, ≥99 %) | Sigma-Aldrich Inc., Saint Quentin Fallavier, France | 310468 | Used to dope the sample with lithium |
| Major Mix IMS/Tof Calibration Kit | Waters Corp., Wilmslow, UK | 186008113 | Calibration solution for MS and IMS |
| MassLynx 4.2 SCN1016 Release 6 (Waters Embedded Analyser Platform for Cyclic IMS 2.9.1 Release 9) | Waters Corp., Wilmslow, UK | 721022377 | Cyclic IMS vendor software for instrument control and data processing |
| Methanol for HPLC PLUS Gradient grade | Carlo-Erba Reagents, Val de Reuil, France | 412383 | High-purity solvent |
| MS Leucine Enkephaline Kit | Waters Corp., Wilmslow, UK | 700002456 | Reference compound used for tuning of the mass spectrometer |
| SCHOTT DURAN 100 mL borosilicate glass bottle | VWR INTERNATIONAL, Radnor, Pennsylvania, US | 218012458 | Used to prepare the solution of 500 µM LiCl in 50:50 MeOH/Water |
| SELECT SERIES Cyclic IMS | Waters Corp., Wilmslow, UK | 186009432 | Ion mobility-mass spectrometer equipped with a cylic IMS cell |
| Website: http://mzmine.github.io/ | MZmine Development Team | - | Link to download the MZmine software |
| Website: https://github.com/siollivier/IM-MN | INRAE, UR BIA, BIBS Facility, Nantes, France | - | Link to an in-house R script containing a CCS calibration function |
Referências
- Allard, P. -. M., et al. Integration of molecular networking and in-silico MS/MS fragmentation for natural products dereplication. Analytical Chemistry. 88 (6), 3317-3323 (2016).
- Wang, M., et al. Mass spectrometry searches using MASST. Nature Biotechnology. 38 (1), 23-26 (2020).
- David, M., Fertin, G., Rogniaux, H., Tessier, D. SpecOMS: a full open modification search method performing all-to-all spectra comparisons within minutes. Journal of Proteome Research. 16 (8), 3030-3038 (2017).
- Dührkop, K., et al. SIRIUS 4: a rapid tool for turning tandem mass spectra into metabolite structure information. Nature Methods. 16 (4), 299-302 (2019).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Nothias, L. -. F., et al. Feature-based molecular networking in the GNPS analysis environment. Nature Methods. 17 (9), 905-908 (2020).
- Gray, C. J., et al. Advancing solutions to the Carbohydrate Sequencing Challenge. Journal of the American Chemical Society. 141 (37), 14463-14479 (2019).
- Ropartz, D., et al. Online coupling of high-resolution chromatography with extreme UV photon activation tandem mass spectrometry: Application to the structural investigation of complex glycans by dissociative photoionization. Analytica Chimica Acta. 933, 1-9 (2016).
- Wolff, J. J., et al. Negative electron transfer dissociation of glycosaminoglycans. Analytical Chemistry. 82 (9), 3460-3466 (2010).
- Ropartz, D., et al. Charge transfer dissociation of complex oligosaccharides: comparison with collision-induced dissociation and extreme ultraviolet dissociative photoionization. Journal of the American Society for Mass Spectrometry. 27 (10), 1614-1619 (2016).
- Morelle, W., et al. Fragmentation characteristics of permethylated oligosaccharides using a matrix-assisted laser desorption/ionization two-stage time-of-flight (TOF/TOF) tandem mass spectrometer. Rapid Communications in Mass Spectrometry. 18 (22), 2637-2649 (2004).
- Gabelica, V., Marklund, E. Fundamentals of ion mobility spectrometry. Current Opinion in Chemical Biology. 42, 51-59 (2018).
- Gabelica, V., et al. Recommendations for reporting ion mobility mass spectrometry measurements. Mass Spectrometry Reviews. 38 (3), 291-320 (2019).
- Hernandez-Mesa, M., et al. Interlaboratory and interplatform study of steroids collision cross section by traveling wave ion mobility spectrometry. Analytical Chemistry. 92 (7), 5013-5022 (2020).
- Koeniger, S. L., et al. An IMS-IMS analogue of MS-MS. Analytical Chemistry. 78 (12), 4161-4174 (2006).
- Merenbloom, S. I., Koeniger, S. L., Valentine, S. J., Plasencia, M. D., Clemmer, D. E. IMS−IMS and IMS−IMS−IMS/MS for separating peptide and protein fragment ions. Analytical Chemistry. 78 (8), 2802-2809 (2006).
- Eldrid, C., Thalassinos, K. Developments in tandem ion mobility mass spectrometry. Biochemical Society Transactions. 48 (6), 2457-2466 (2020).
- Giles, K., et al. A cyclic ion mobility-mass spectrometry system. Analytical Chemistry. 91 (13), 8564-8573 (2019).
- Merenbloom, S. I., Glaskin, R. S., Henson, Z. B., Clemmer, D. E. High-resolution ion cyclotron mobility spectrometry. Analytical Chemistry. 81 (4), 1482-1487 (2009).
- Ollivier, S., et al. Anomeric retention of carbohydrates in multistage cyclic ion mobility (IMSn): de novo structural elucidation of enzymatically produced mannosides. Analytical Chemistry. 93 (15), 6254-6261 (2021).
- Ollivier, S., Fanuel, M., Rogniaux, H., Ropartz, D. Molecular networking of high-resolution tandem ion mobility spectra: a structurally relevant way of organizing data in glycomics. Analytical Chemistry. 93 (31), 10871-10878 (2021).
- Aron, A. T., et al. Reproducible molecular networking of untargeted mass spectrometry data using GNPS. Nature Protocols. 15 (6), 1954-1991 (2020).
- McKenna, K. R., Li, L., Krishnamurthy, R., Liotta, C. L., Fernández, F. M. Organic acid shift reagents for the discrimination of carbohydrate isobars by ion mobility-mass spectrometry. The Analyst. 145 (24), 8008-8015 (2021).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11, 395 (2010).
- Ruotolo, B. T., Benesch, J. L. P., Sandercock, A. M., Hyung, S. -. J., Robinson, C. V. Ion mobility-mass spectrometry analysis of large protein complexes. Nature Protocols. 3 (7), 1139-1152 (2008).
- Bush, M. F., Hall, Z., Giles, K., Hoyes, J., Robinson, C. V., Ruotolo, B. T. Collision cross sections of proteins and their complexes: a calibration framework and database for gas-phase structural biology. Analytical Chemistry. 82 (22), 9557-9565 (2010).
- Ropartz, D., et al. Structure determination of large isomeric oligosaccharides of natural origin through multipass and multistage cyclic traveling-wave ion mobility mass spectrometry. Analytical Chemistry. 91 (18), 12030-12037 (2019).
- Tolmachev, A. V., et al. Characterization of ion dynamics in structures for lossless ion manipulations. Analytical Chemistry. 86 (18), 9162-9168 (2014).
- Arndt, J. R., et al. High-resolution ion-mobility-enabled peptide mapping for high-throughput critical quality attribute monitoring. Journal of the American Society for Mass Spectrometry. 32 (8), 2019-2032 (2021).
- Le Fèvre, A., Dugourd, P., Chirot, F. Exploring conformational landscapes using trap and release tandem ion mobility spectrometry. Analytical Chemistry. 93 (9), 4183-4190 (2021).
- Ohshimo, K., He, X., Ito, R., Misaizu, F. Conformer separation of dibenzo-crown-ether complexes with Na+ and K+ ions studied by cryogenic ion mobility-mass spectrometry. The Journal of Physical Chemistry A. 125 (17), 3718-3725 (2021).
- Purves, R. W., Barnett, D. A., Ells, B., Guevremont, R. Gas-phase conformers of the [M + 2H]2+ ion of bradykinin investigated by combining high-field asymmetric waveform ion mobility spectrometry, hydrogen/deuterium exchange, and energy-loss measurements. Rapid Communications in Mass Spectrometry. 15 (16), 1453-1456 (2001).
- Ujma, J., et al. Cyclic ion mobility mass spectrometry distinguishes anomers and open-ring forms of pentasaccharides. Journal of the American Society for Mass Spectrometry. 30 (6), 1028-1037 (2019).
- Warnke, S., Faleh, A. B., Scutelnic, V., Rizzo, T. R. Separation and identification of glycan anomers using ultrahigh-resolution ion-mobility spectrometry and cryogenic ion spectroscopy. Journal of The American Society for Mass Spectrometry. 30 (11), 2204-2211 (2019).
- Williamson, D. L., Bergman, A. E., Nagy, G. Investigating the structure of α/β carbohydrate linkage isomers as a function of group I metal adduction and degree of polymerization as revealed by cyclic ion mobility separations. Journal of the American Society for Mass Spectrometry. 32 (10), 2573-2582 (2021).
- Myers, O. D., Sumner, S. J., Li, S., Barnes, S., Du, X. One step forward for reducing false positive and false negative compound identifications from mass spectrometry metabolomics data: new algorithms for constructing extracted ion chromatograms and detecting chromatographic peaks. Analytical Chemistry. 89 (17), 8696-8703 (2017).
- Marchand, A., Livet, S., Rosu, F., Gabelica, V. Drift tube ion mobility: how to reconstruct collision cross section distributions from arrival time distributions. Analytical Chemistry. 89 (23), 12674-12681 (2017).
- Davis, D. M., et al. Analysis of ion mobility spectra for mixed vapors using Gaussian deconvolution. Analytica Chimica Acta. 289 (3), 263-272 (1994).
- Polasky, D. A., Dixit, S. M., Fantin, S. M., Ruotolo, B. T. CIUSuite 2: next-generation software for the analysis of gas-phase protein unfolding data. Analytical Chemistry. 91 (4), 3147-3155 (2019).
- Salbo, R., et al. Traveling-wave ion mobility mass spectrometry of protein complexes: accurate calibrated collision cross-sections of human insulin oligomers. Rapid Communications in Mass Spectrometry. 26 (10), 1181-1193 (2012).
- Gelb, A. S., Jarratt, R. E., Huang, Y., Dodds, E. D. A study of calibrant selection in measurement of carbohydrate and peptide ion-neutral collision cross sections by traveling wave ion mobility spectrometry. Analytical Chemistry. 86 (22), 11396-11402 (2014).
- Richardson, K., Langridge, D., Dixit, S. M., Ruotolo, B. T. An improved calibration approach for traveling wave ion mobility spectrometry: robust, high-precision collision cross sections. Analytical Chemistry. 93 (7), 3542-3550 (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados