このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
タンデムイオン移動度実験のための周期的イオン移動度分光計の使用
要約
イオン移動度分析(IMS)は、生体分子の特性評価のための質量分析を補完する興味深いものであり、特に異性化に敏感であるためです。このプロトコルは、分子の単離とその断片の移動度プロファイルの生成を可能にするタンデムIMS(IMS/IMS)実験を記述します。
要約
化学構造の正確な特性評価は、その根底にある生物学的メカニズムと機能的特性を理解するために重要です。質量分析(MS)は一般的なツールですが、すべての構造的特徴を完全に明らかにするには必ずしも十分ではありません。例えば、炭水化物は生物学的に関連性があるが、それらの特性評価は多数のレベルの異性化によって複雑である。イオン移動度分光法(IMS)は、イオン立体構造に敏感であり、したがって異性性に敏感であるため、興味深い補完物です。
さらに、最近の進歩により、この技術が大幅に改善されました:前世代のサイクリックIMS計測器は、分解能の向上やタンデムイオンモビリティ(IMS/IMS)実験の実行可能性など、リニアIMS計測器と比較して追加の機能を提供します。IMS/IMS では、イオンの移動度に基づいてイオンが選択され、断片化され、再分析されて、その断片に関するイオン移動度情報を取得します。最近の研究は、このようなIMS/IMSデータに含まれる断片の移動度プロファイルが、特定のグリカンのフィンガープリントとして機能し、構造的に関連する方法でグリコミクスデータセットを編成するための分子ネットワーキング戦略に使用できることを示した。
したがって、このプロトコルの目的は、サンプル調製から再現可能なスペクトルを生成するイオン移動度寸法の最終的な衝突断面(CCS)較正まで、IMS/IMSデータを生成する方法を記述することです。1つの代表的なグリカンの例を挙げると、このプロトコルは、サイクリックIMS機器上にIMS/IMS制御シーケンスを構築する方法、IMS到着時間をドリフト時間(すなわち、イオンに適用される有効な分離時間)に変換するためにこの制御シーケンスを考慮する方法、および生データから関連するモビリティ情報を抽出する方法を示す。このプロトコルは、IMS/IMS 実験の重要なポイントを明確に説明し、新しいサイクリック IMS ユーザーが簡単で再現可能な集録を実行するのに役立つように設計されています。
概要
生体分子の完全な化学的特性評価は、その根底にある生物学的および機能的特性を理解するための鍵です。この目的のために、近年、「オミックス」科学が発展し、生物学的濃度での化学構造の大規模な特徴付けを目指している。プロテオミクスおよびメタボロミクスにおいて、MSは、特にタンデムMS(MS/MS)を介して構造情報を提供する感度と能力のおかげで、生物学的媒体に見られる構造的不均一性を解明するためのコアツールとなっている。MS/MS戦略では、イオンはその質量に応じて選択され、次いで断片化され、そして最後に、その断片の質量が分子のフィンガープリントを確立するために獲得される。MS/MSスペクトルは、特に、スペクトルデータベース1,2を一致させるために、または親構造3,4を暫定的に再構築するために使用することができる。類似のスペクトルが類似の化合物に属するという仮定の下で、MS/MSデータは、類似性スコア5,6を介して近縁種をつなぐ分子ネットワーク(MN)を構築するためにも使用することができる5,6。
しかし、イオンの質量電荷比(m / z)を検出するMSの固有の特性のために、この技術は(立体)異性性の範囲内にある多くの構造的特徴に盲目的である。例えば、炭水化物はいくつかの単糖サブユニットからなり、その多くは立体異性体またはエピマー(例えば、Glc対GalまたはGlc対Man)である。これらのサブユニットはグリコシド結合によって連結されており、これは結合の位置(位置異性)およびアノマー炭素の立体配置(アノマー性)によって異なる可能性がある。これらの特性により、スタンドアロンMSが炭水化物異性体を区別することは困難であり7、高エネルギー活性化法を使用して位置異性のみに対処することができます8,9,10。誘導体化は立体異性体基の等価性を破壊するための選択肢ですが11、広範なサンプル調製が必要です。もう1つのより簡単な選択肢は、MSをIMSなどの異性化に敏感な分析次元と組み合わせることです。
このプロトコルは、IMS の基本概念に既に精通しているユーザー向けに設計されており、詳細なレビューは他の場所で入手できるため12,13 ここでは、IMS の原則の簡単な概要のみを示します。IMSは、イオンとバッファーガスおよび電場との相互作用に依存し、最終的に気相立体構造に従ってイオンを分離する気相分離法である。MSに結合されたIMSのさまざまな原理は、商用機器で見つけることができます:いくつかは交互の高電界と低電界(電界非対称IMS、FAIMS)で動作しますが、ほとんどは低磁界限界内で動作します - 特にドリフトチューブIMS(DTIMS、直線的に減少する電界)、進行波IMS(TWIMS、対称電位波)、およびトラップされたIMS(TIMS、電界に対してイオンをトラップするバッファガスの高流量)13.低磁場法は、分離中にバッファーガスと相互作用するイオンの表面(Å2またはnm2)を表すイオン - ガス対の特性である、いわゆるCCSへのアクセスを可能にする。CCSは理論的には機器に依存しないため、異なるラボ間で再現できるデータを生成するのに役立ちます14。イオン移動度分離は、様々なパラメータによって、特に移動度セル内のガス圧力およびガス温度の変動によって影響を受ける可能性がある。CCSキャリブレーションは、キャリブラントと対象種の両方が同様に影響を受けるため、これを改善する方法です13。ただし、温度制御された部屋に機器を設置し、信頼性の高いガス圧力制御システムを持つことが必須です。
IMSの興味深い進化はIMS/IMSであり、これは2006年にクレマーのグループによってMS/MS15,16のアナログとして初めて導入された。IMS/IMSでは、目的のイオンは、そのイオン移動度に基づいて選択的に単離される。その後、(フラグメンテーションが可能になるまで) 活動化され、活動化されたイオンまたはフラグメントの新しい IMS 分析が実行されます。最初の機器設計では、2つのIMSセルを直列に配置し、活性化が立っていたイオン漏斗によって分離した。それ以来、多くのIMS/IMSセットアップが提案されましたが(レビューについては、EldridとThalassinos17を参照)、IMS/IMS機能を備えた最初の商用質量分析計は201918年に利用可能になりました。この装置は、IMSセルの循環設計という別の技術的ブレークスルーと組み合わせることで、初期の概念を大幅に改善しました。
周期的なIMSセルは、理論的には、ドリフトパスの長さをほぼ無限に増加させ、したがって、計測器19の分解能を増加させることを可能にする。これは、環状TWIMSセルが主イオン光軸に対して直交して配置される特定の機器形状によって達成された。IMS セルの入り口にある多機能アレイ領域は、(i) IMS 分離のためにイオンを横向きに送る、(ii) MS 検出のために前方に送る、または (iii) IMS セルから後方にプリアレイ・セルに格納する、イオン経路の方向を制御することができます。このプレアレイストアセルから、イオンを活性化し、フラグメントをイオン移動度測定のためにIMSセルに再注入することができ、このアプローチは立体異性体20を特徴付けるために首尾よく使用されている。最終的に、収集されたデータには、前駆体とその断片のイオン移動度と m / z 情報が含まれています。
グリカン分析にこの環状設計を使用した最近の出版物(Ollivier et al.21)では、このようなIMS/IMSデータに含まれるフラグメントの移動度プロファイルが、分子ネットワーク戦略に使用できる生体分子のフィンガープリントとして機能することを示した。その結果、IM-MNと呼ばれるネットワークは、構造的に関連する方法でグリコミクスデータセットの組織化につながったが、MS / MSデータ(MS-MN)のみから構築されたネットワークはほとんど情報を明らかにしなかった。本書を補完し、Cyclic IMS ユーザーがこのワークフローを実装できるように、このプロトコルには、データの収集に使用されるプロトコルの完全な説明が記載されています。このプロトコルは、ユーザーが IM-MN ネットワークの構築に使用できる IMS/IMS データの生成 (21 を参照) のみに焦点を合わせています (または、ユーザーが選択したその他のアプリケーションに対して)。IM-MNの構築は、分子ネットワークのためのプロトコルがすでに利用可能であるため、ここでは考慮されません22。貴重で再現可能な IMS/IMS 集録を生成するために従わなければならない重要なポイントが強調表示されます。Ollivierらによって研究されたオリゴ糖の1つを例 にとる。図21では、(i)サンプル調製、(ii)サイクリックIMS機器のチューニング、(iii)データの自動ピークピッキング、および(iv)CCSキャリブレーションのステップが詳述されています。
プロトコル
メモ: プロトコルの概要を図 1 に示します。本プロトコールに記載される実験に使用されるパラメータは、補足表S1および補足表S2に見出すことができる。
試料溶液の調製
注:このプロトコルは、例としてアラビノキシラン五糖(23-α-L-アラビノフラノシル-キシロテトラオースまたはXA2XX; 材料表を参照)を用いて記載されている。
- 溶媒の調製:50:50 H2O:MeOH中の500μM LiCl(体積/体積)。
- 212mgのLiClを秤量してH2O中の塩化リチウム(LiCl)の100mM原液を調製し、50mLポリプロピレン製円錐管に50mLの高純度脱イオン水(H2O)を加える。完全に溶解するまで振る。
注:溶媒にはリチウム塩がドープされており、分光計のイオン源における[M + Li]+ 付加物の形成を促進し、通常は他のアルカリ付加物と比較してより高品質のフラグメンテーションスペクトルが得られる。LiClの使用が推奨されるのは、有機酸(およびその塩)がIMSプロファイルに影響を与えることが以前に判明しているためです23。 - ガラス瓶に、LiCl ストック溶液を 200x: 250 μL のストック溶液に希釈し、24.75 mL の H2O を加えます。25 mL のメタノール (MeOH) を加えて、50:50 H2O:MeOH (v/v) で 500 μM の LiCl の最終濃度に達します。溶媒を脱気するために2分間超音波処理する。
注:MeOHは健康上の危険(H225、H301、H311、H331、H370)を提示します。ラボコート、手袋、および目の保護具を着用した抽出フードの下で操作します。50:50 MeOH/H2O(v/v)の割合は、オリゴ糖のイオン化に最適な溶媒であると思われる。しかしながら、MeOHは、必要に応じてアセトニトリル(ACN)で置換することができる。
- 212mgのLiClを秤量してH2O中の塩化リチウム(LiCl)の100mM原液を調製し、50mLポリプロピレン製円錐管に50mLの高純度脱イオン水(H2O)を加える。完全に溶解するまで振る。
- 1.5mLポリプロピレンチューブに、1mgの炭水化物の重量を量る。500 μM LiClの適切な容量で溶解し、1 mg/mLの濃度に達する。50:50 MeOH/H2O + 500 μM LiCl で終濃度 10 μg/mL に希釈します。4°Cで保存してください。
注:10μg/mLの濃度は、IMS/IMS-MS中のすべてのフラグメントイオンにわたってシグナルを最適化するために選択されました(これは純粋な化合物用であり、混合物で作業する場合は濃度を上げてください)。基準IMS/IMSスペクトルを取得するには、サンプルをさらに希釈しないでください:フラグメンテーションの前にMS検出器の飽和が予想されますが、装置には補正オプションが用意されています(ステップ3.2を参照)。
2. サイクリックIMS質量分析計のチューニング
メモ: ソフトウェア関連の指示 (ウィンドウ、メニュー、およびコマンド) は 太字で強調表示されています。
- 計器制御ソフトウェアから計器コンソールを開き(MSチューニング ページ、 材料表のソフトウェアの詳細を参照)、計器を 操作 モードにします。IMS セル内で高電圧が安定するまで、少なくとも 3 時間待ちます。
メモ: 最高の再現性を得るには、IMS セル内の電圧を完全に安定させる必要があります。高電圧をオンにして、IMSの巡回解析の前に機器を一晩安定させます。さらに、イオン移動度セル内の圧力および温度は、可能な限り一定に保たれなければならない。圧力のリードバックは 真空 タブで利用できますが、温度のリードバックは利用できません。機器を熱測定された実験室に保管してください。この作業で使用された機器は、20°Cで熱測定された実験室で1.75mbarで動作します。 - サイクリック IMS 計器のセットアップ
メモ:標準ソリューションは、機器のセットアップに内蔵のフルイディクスシステムを使用して注入する必要があります。- 適切な製造業者提供の標準で満たされた流体工学容器を流体システム上に置きます:リザーバB(「ロックマス」):10pg/μLロイシンエンケファリン(LEU ENK)を50:50 ACN / H2O + 0.1%ギ酸に入れます。Reservoir C ('Calibrant'): MajorMix.
注: このプロトコルでは、MajorMix キャリブレーションソリューションを使用して、 m/z 寸法と CCS 寸法の両方をキャリブレーションします。実用的な理由から、外部CCSキャリブレーションが実行されます(プロトコルのステップ5を参照)。したがって、CCS用の社内キャリブラント混合物と m / z 用の別のキャリブラント(例えば、ギ酸ナトリウムまたはヨウ化ナトリウム)を使用することもできる。 - Quartz コンソールの [チューニング] ページで、[フルイディクス] タブに移動します。サンプル流体をリザーバCに設定し、基準フルイディクスをリザーバBに設定します。両方の溶液をイオン源に連続して注入して、MS信号を確認します。
- ADCのセットアップ、検出器のセットアップ(LEU ENKを使用)、および質量校正(校正ソリューションについては 材料表 を参照)を、製造元の指示に従って 機器のセットアップ ページから実行します。
- 適切な製造業者提供の標準で満たされた流体工学容器を流体システム上に置きます:リザーバB(「ロックマス」):10pg/μLロイシンエンケファリン(LEU ENK)を50:50 ACN / H2O + 0.1%ギ酸に入れます。Reservoir C ('Calibrant'): MajorMix.
- 1 パス分離でキャリブレーション・ソリューションの IMS アクイジションを記録します (これは外部 IMS キャリブレーションに使用します)。
メモ:イオン源と進行波(TW)パラメータ(静的波の高さと波速)は、すべての集録(キャリブレーションと集録)中に一定に保つ必要があります。ユーザーがサンプルに最適なパラメータに関する事前知識を持っていない場合、このステップはプロトコルのステップ3の後に実行することができます(中性オリゴ糖の[M + Li]+ 付加体の場合、代表的な結果は16VのTW高さと350 m/sのTW速度を使用し、最良の結果が得られます)。- 「フルイディクス」タブから、バッフル位置サンプルを選択し、「サンプル」プローブを介してイオン源にキャリバート(材料表を参照)を10μL/minの流量で注入します。
- シングルパス IMS シーケンスをセットアップします。 「チューン」 ページで、インストゥルメントを モビリティ モードにし、 サイクリックシーケンスコントロール ウィンドウを開きます。 [詳細 モード] を選択します。この新しいウィンドウの 「循環関数 」タブから、「 バンドルの追加」を選択してから、「 シングル/マルチパス」を選択します。モビリティ イベントのシーケンスが同じウィンドウの [ シーケンス] タブに表示されるのを待ちます。
メモ:リアルタイム表示を有効にするには、計測器パラメータを適用する必要があります:TOFモードでチューニングまたはモビリティモードで実行をクリックします。計測器をTOFモードとモビリティモードに切り替える前に、実行中の集録(Tuneページ表示を含む)を中止する必要があります。イオンの相対的な存在量は、イオン透過パラメータの変化のためにTOFモードとモビリティモードの間で変化し得る。 - すべてのキャリブラントイオンがサイクリックIMS競馬場の周りを1回通過するようにシーケンスを調整します。注入時間またはイジェクトおよび集録時間を変更しないでください。 ただし、[分離時間] を [シーケンス] タブの 1 ミリ秒に下げます。キャリブレーション混合気の一部のイオンが表示された到着時間枠に収まらない場合は、[ADC 設定]タブで[ビンあたりのプッシュ数]を増やして、IMSと直交加速度TOFアナライザのプッシャーとの同期を変更します。
メモ: 制御シーケンスの時刻は、イオンゲーティング用の多機能アレイのみを制御します。イオンが競馬場の周りを最初に(またはn回目)通過している限り、その間に配列内でTWの方向が変わったとしても、そのパスを終了します。分離時間を1ミリ秒に短縮すると、アレイは1ミリ秒後にイジェクトモードに切り替わります。これにより、より速いイオンは、遅いイオンが最初のパスを終える前に、アレイを通過して2回目のパスに従事するのに十分な時間を持たないことが保証されます。したがって、すべてのイオンは、IMS較正を実行するために必要な同じパス数(すなわち、1パス)を受けることになる。 - 2 分間の集録を記録します。「サイクリックシーケンスコントロール」ウィンドウで、「集録」をクリックして「集録設定」ポップアップウィンドウを開きます。 ファイル名、説明、および取得期間(分)を入力し、「保存」をクリックします。
- ステップ2.3と同じ条件で校正溶液をさらに2分間取得して記録します(CCS校正の品質を確認するためにこれを使用します)。「サイクリックシーケンスコントロール」ウィンドウで、「集録」をクリックして「集録設定」ポップアップウィンドウを開きます。 ファイル名、説明、および取得期間(分)を入力し、「保存」をクリックします。
- 流体システムを50:50 H2O/ACNで徹底的に洗浄し、ピークチューブ内のキャリブラントの結晶化を防ぎます。
3. IMS/IMS-MS の取得
- シリンジポンプを使用して、(リチウムドープ)サンプルを10 μg/mLでサンプルプローブに10 μL/minの流速で注入します。
- (MSチューニングページから)計測器をTOFモードに切り替えて、信号の安定性を確認します。サンプルの完全なMS取得(1分)を記録すると、同位体パターンと潜在的な汚染物質の存在を確認するのに役立ちます。
注:サンプル濃度はフラグメントに対して良好なイオンシグナルを得るために選択されるため、このステップでTOF飽和が観察されることがあります。TOF飽和は、(i)人為的に増加したMS分解能、(ii)同位体比の変化、および(iii)同位体間の多数の低存在量ピークのアーティファクトを使用して同定することができる。DREレンズ(ダイナミックレンジエンハンスメント、メインチューンページのクワッド/MSプロファイル/DREタブ)を使用して、イオンの透過を減衰させ、TOFモードで飽和を破棄します(図2A、B)。 - 機器をMSMSモード(メインチューンページのクワッド/MSプロファイルタブ)にし、四重極で単離するためにMSMS質量フィールドで標的イオンの質量を選択します(例では、685.2のm/z、アラビノキシラン五糖の[M + Li]+イオン種に対応)。1分間の集録を記録して、データ処理時の前駆体の分離を確認します。
注: リチウム付加物は、モノアイソトピックピークの -1 Da に同位体があるため、処理ステップに干渉しないように MS/MS 選択ウィンドウから除去する必要があります。これは、[クワッド/MSプロファイル]タブの[LM分解能]パラメータと[HM分解能]パラメータを使用して選択範囲を狭めることで削除できます(図2C)。 - IMS シーケンスを「スライス」して、目的の異性体のモビリティー・ベースの選択を実行します。
- 計測器をモビリティモードに切り替えます(ステップ2.3.2を参照)。「サイクリックシーケンスコントロール」ウィンドウの「サイクリック機能」タブで、「バンドルを追加」を選択し、「スライス」を選択します。モビリティ イベントの複雑なシーケンスが [シーケンス] タブに表示されるまで待ちます (図 3)。
注: IMS/IMS プロセスの各ステップを視覚化することができます: 「シーケンス」タブの「排出および獲得」イベントをクリックします。赤でハイライト表示したら、[上へ]ボタンと[下へ]ボタンを使用して、シーケンス内の適切な位置に移動します。 - Eject イベントと Acquire イベントを最初の Separate イベントの直後に配置し (つまり、図 3 に示すシーケンスの行 8 ではなく行 3 に移動します)、[実行] をクリックします。リアルタイムで表示される初期分離の結果を探します。IMS ピークの解決が満足のいくものになるまで、シーケンス内のこのイベントの時間値を変更して、マルチパス分離の最初の分離イベントの継続時間を長くします。参照用に1分間の集録を記録します。
メモ: [ADC のセットアップ]タブ の[ADC の開始遅延]の値に注意してください:絶縁の品質を確認すると便利です。 - 「一時停止」をクリックします。初期分離の結果が表示されますが、ユーザーが [実行] をもう一度クリックするまで、制御シーケンスの変更は適用されません。イジェクトおよび集録イベントを、イジェクト、プリストアへのイジェクト、および保留およびイジェクトの各イベントの下に配置します。ターゲットピークが「イジェクトからプリストアへ」領域にあり、その他のイオンが「イジェクト」領域または「ホールドアンドイジェクト」領域にあるように、イベントの継続時間を調整します。
注: 到着時間分布 (ATD) と比較したこれら 3 つのイベントの継続時間は、[ モービログラム ] タブのモビリティ スペクトルの下にある色分けされたバーを使用して視覚化できます (図 3)。 - Eject イベントと Acquire イベントは、シーケンスの最後に、 プレストアからの再注入 イベントと 2 番目の [個別のイベント] の下に配置します。[ 実行 ]をクリックして、選択した母集団を表示します。
注: 選択された母集団が IMS セルを離れたため、以前の分離はすべて失われ、シングルパス分離 (望ましい) に戻ります。 - 分離の品質を確認します。目的のピークのみが選択されたことを確認するには、図4に示すように、再注入前と同じ分離(すなわち、同じ分離時間)を再注入後に行う。参照用に1分間の集録を記録します。
注:ユーザーは、排出された人口を確認することをお勧めします。[イジェクトからプリストアへ]時間枠はベースラインレベルである必要があります(図4B)。これを確認するには、[ADC 設定]タブで[手動モードのADC開始遅延]を表示し、ステップ3.4.2で記録した遅延時間を入力します。参照用に1分間の集録を記録します。 - [シーケンス] タブのユーザー定義のイベント時刻の横にある列 (赤で強調表示された [時間 Abs] 列) で、すべてのイベントの合計時間を探します。CCS キャリブレーションを実行するための [プレストアから再注入] イベントの行で見つかった時間 Abs に注意してください。
- 計測器をモビリティモードに切り替えます(ステップ2.3.2を参照)。「サイクリックシーケンスコントロール」ウィンドウの「サイクリック機能」タブで、「バンドルを追加」を選択し、「スライス」を選択します。モビリティ イベントの複雑なシーケンスが [シーケンス] タブに表示されるまで待ちます (図 3)。
- IMS の 2 つのラウンド間のターゲット・ピークをフラグメント化します。再注入ステップの電圧を変更してイオンの運動エネルギーを増加させ、イオン移動度ガスとの衝突時にそれらを断片化する。
- イジェクトと集録の直前の Separate イベントの継続時間を 1 ミリ秒に設定します (ステップ 2.3.3 の説明を参照)。
- [ ストア前から再挿入 ] 行で、[ アクティブ化を有効にする] ボックスをオンにし、組み込みコントロールを使用して断片化を最適化します。スペクトルが満足のいく場合(例えば、塩基ピークがフラグメントである場合)、ステップ3.5.4に直接進む。
メモ:アクティベーションを有効にすると、ライン上の3つの電圧が灰色に変わります:これらは、電圧を手動で最適化する必要がある場合にユーザーが変更する必要がある電圧です(次の手順を参照)。これら3つの電圧(プレアレイ 勾配、プレアレイバイアス、および アレイオフセット)は、イオンを活性化するために使用される勾配を形成します。イオンの運動エネルギーは、 プレアレイバイアス と アレイオフセット の間の傾きとともに増加します( 図5参照)。グラデーション→バイアス→オフセット値のデフォルト値は、アクティブ化なし 85 → 70 → 45 V です。組み込み関数の最大アクティブ化 185 → 170 → -5 V (+150 V)。フラグメンテーション後、 DREレンズ を使用してイオン透過を再調整することを忘れないでください(信号の減衰を減らす)(ステップ3.2を参照)。 - フラグメンテーションが内蔵コントロールで十分でない場合は、[アクティブ化を有効にする]ボックスのチェックを外し、再注入電圧を手動で最適化します。アレイ前勾配電圧を上げ(プリアレイバイアス電圧は常にアレイ前勾配より15 V低くする必要があります)、結果が満足できるようになるまでアレイオフセット電圧を下げます(負に設定できます)。
メモ:多機能アレイの電圧を手動で調整する場合、ユーザは「モービログラム」ビューから多機能アレイに印加される電圧のインタラクティブな回路図(PE図)に切り替えて、電圧設定をより正確に視覚化することができます(図5A)。 - 2分間の集録を記録します。集録ポップアップウィンドウで、[ ドリフト時間を保持] オプションにチェックマークを付けて、到着時間と m/z (クロマトグラフィー分析に使用された集録時間、保持時間)のみを含むファイルを生成します。このファイルには *_dt というラベルが付けられていることに注意してください 。ラウ。
注: ユーザーが 「ドリフト時間を保持する」 オプションをチェックし忘れた場合でも、ドリフトスコープ 2.9 ソフトウェアを使用して IMS ディメンションを抽出することができます (ファイル|マスリンクス|へのエクスポートドリフト時間を保持します)。
- メインのTuneページで計測器をTOFモードに戻し、50:50 MeOH/H2Oでシステムを十分にすすぎ、次のサンプルに進みます。
4. MZmine 224によるIMS/IMS-MS処理
注:MZmine 2は、 資料表に記載されているURLから入手できます。MZmine 2.51 の使用をお勧めします。この原稿の作成時点では、インポート機能の変更により、それ以降のバージョンではサイクリックIMSインストゥルメントからRAWファイルを開くことができません。
- IMS および m/z ディメンション (*_dt のみを含むロー・ファイルをインポートします。RAW) 生データメソッドを使用して|生データのインポート。
注:RAWファイルはメインMZmineウィンドウの左側に表示されます。元の*をインポートしないでください 。 保持期間ディメンションがまだ含まれている RAW ファイル。MZmine は保持時間と IMS 到着時間を区別せず、両方のディメンションのデータ・ポイントが重複します。 - 代表的なファイルのワークフローパラメータを最適化するには、 生データファイル リスト。
- データ内のノイズレベルを評価します。「 生データ ファイル」リストでファイルを右クリックし、「 TICを表示 」を選択して、ベースピーク「クロマトグラム」(BPC)を表示します。目で観測できる最小のピークをダブルクリックすると、そのマススペクトルが表示されます。データ内のノイズレベルがこのスペクトルのベースピークの2番目の同位体のノイズレベル付近にあると考え、次の処理ステップのすべての強度しきい値にこの同じ値を使用します。
注:データは四重極分離を使用して取得されたため、MZmineはMS / MSと見なします。MZmine処理全体を通して、 MSレベル= 2で動作するようにしてください。 - 生データメソッドを使用した質量検出の実行 |機能検出|質量検出。プロファイルモードで集録したデータの場合は、 ウェーブレット変換 アルゴリズムを使用します。MZmineでアルゴリズムのパラメータを設定するには、アルゴリズムの横にある [... ]ボタンをクリックし、[ プレビューを表示] オプションを使用してパラメータを最適化しながらデータを視覚化します。
注: この段階では、アルゴリズムによって選択されたピークがプレビュー ウィンドウに赤で表示されます。独自のRAWファイルで ウェーブレット変換 アルゴリズムを使用すると、MZmineはプロファイルデータポイントを重心ピークと間違えることがあります。ソフトウェアは、ユーザーが重心スペクトルでプロファイルアルゴリズムを実行していることを示すメッセージを表示します。このメッセージを無視して[ OK]をクリックします。 - 抽出されたイオン移動度スペクトル(EIM)を、各フラグメント質量について 、生データ法を用いて再構成する|機能検出| 前のステップで生成された「質量」質量リスト上のADAPクロマトグラムビルダー。この段階でのm/z公差入力はスキャン /スキャン公差 であるため、予想される精度全体の少なくとも3~4倍高い値にしてください。
- 前の手順にはプレビューオプションがないため、メインMZmineウィンドウの右側のパネルに表示された 機能リスト を使用して、ピークピッキングの品質を直接確認します。 「フィーチャー」リストを開き、すべての行を選択して右クリックし、「 表示/XIC (ダイアログ)」を選択します。[ すべて ]をクリックして、移動度スペクトル上のすべてのイオンを表示します。色で表示される選択したピークを調べて、明らかな欠落したピークがないことを確認します。
- EIM を逆畳んで、複数のフィーチャに異なるピークを含む m/z を分割します。 機能リストのメソッドを使用する |機能検出|クロマトグラムデコンボリューション、および ウェーブレット(ADAP) アルゴリズムを選択します。 プレビュー表示 オプションと、 S/N しきい値、係数/面積しきい値、および RT ウェーブレット範囲の主要なパラメーターを使用して、データのアルゴリズムを最適化します。
メモ: デコンボリューションされたスペクトルのアスペクトを確認することをお勧めします。手順 4.2.4 で説明したクロマトグラム視覚化ツールを使用します。図 6Aに示すように、畳み取り解除されたピークは色で表示され、同じ質量のピークは分割する必要があります。 - フィーチャー・リスト・メソッドを使用して、デコンボリューションされた EIM をデアイソトープ |同位体|同位体ピークハタ。 m/z 公差値に計器の期待精度を使用し、IMS 分離中に同位体が分解されないため、到着時間公差を 0.1 ms (MZmine では 保持時間許容値 0.1 分と表示) に設定します。フィーチャ リストを確認する: 同位体が残っている場合は、公差値を増やします。
注: 理論的には、デアイソトーピングはフィーチャ リスト処理の任意の時点で実行できますが、電荷値をエクスポートできるように、最後に行うことが重要です (他の手順で使用されるアルゴリズムによって、電荷状態情報が削除されることがあります)。
- データ内のノイズレベルを評価します。「 生データ ファイル」リストでファイルを右クリックし、「 TICを表示 」を選択して、ベースピーク「クロマトグラム」(BPC)を表示します。目で観測できる最小のピークをダブルクリックすると、そのマススペクトルが表示されます。データ内のノイズレベルがこのスペクトルのベースピークの2番目の同位体のノイズレベル付近にあると考え、次の処理ステップのすべての強度しきい値にこの同じ値を使用します。
- 複数のIMS/IMS-MSスペクトルを処理する場合は、これらの最適化されたパラメータで処理を繰り返します。すべてのスペクトルに対して同じパラメータを保持します。
- 複数のスペクトルの場合は、それらを1つのテーブルにグループ化してエクスポートします。そうでない場合は、ステップ 4.5 に直接進んでください。スペクトルをグループ化するには、 フィーチャリストメソッドを使用して|整列|アライナーに参加します。目的は実際にピークを揃えることではないため、 m/z と到着時間の両方に制限的な許容値を使用します。両方の寸法に同じ重みを与えます。
- 最終的な機能リストを *.csv ファイルにエクスポートします。 機能リストのメソッドを使用する |エクスポート/インポート|CSV ファイルに エクスポートし、エクスポート行 m/z、 エクスポート行保存時間 (実際の IMS 到着時間)、 ピーク m/z、および ピーク高さの値をエクスポートします。コンマをフィールド区切り文字として使用します。
5. 重心付きIMS/IMSスペクトルのTWCCSN2
注:このプロトコルでは、対数フィットキャリブレーション25,26が使用され、線形キャリブレーションよりも優れた結果が得られる傾向があり、スプレッドシートまたは社内の処理スクリプトに簡単に実装できます。社内スクリプト(Rで書かれています)は、材料表に記載されているURLから入手できます。
- キャリブラントの集録から基準到達時間値を選択します(ステップ2.3を参照)。コンストラクター・ソフトウェア ( 材料表を参照) を使用して手動でこれを行い、すべての IMS キャリブラント・ピークの側面を確認します。
- クロマトグラムウィンドウで、*_dtを開きます。キャリバートに対応するRAWファイル。
- キャリブレーション・ポイントごとに、 ディスプレイ・|を使用して EIM を生成マスオプション 。
- EIM のプロファイルを確認してください。定義が不十分なものがある場合は、プロセス |を使用して滑らかにします。 スムーズ オプション(最良の結果は、通常、サヴィツキー・ゴレイアルゴリズムで得られるため、3ビンにわたって2回滑らかになります)。頂点値をスプレッドシートで報告します。
メモ: 基準点は通常、低分解能DTIMSデバイスを使用して取得されるため、キャリブラントによっては、サイクリックIMSに一部のマルチモーダル分布が表示される場合があります。このような分布を示すピークをキャリブレーションリストから削除します。
- キャリブラントから対数適合パラメータを計算します。
- すべてのキャリブレーションポイントについて、次の計算を行います。
- 式(1)を使用してドリフト時間を計算します。
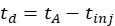 (1)
(1)
td はドリフト時間、tA は測定された到着時間、tinj は IMS セルへの注入時間です (すべてミリ秒単位)。
注: オリゴ糖フラグメントなどの小分子の場合、異なる質量間のデッドタイム(IMSセルからの出口と検出器間の飛行時間)の変動はCCSキャリブレーションの誤差範囲内にあり、無視できます。 - Eq(2)を使用してイオンの中性質量を計算します。
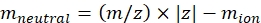 (2)
(2)
zはイオンの電荷状態、mionは対イオンの質量(Da)である。正確な質量を使用して、不確実性の発生を回避します。対イオンの代わりに原子損失がある場合は、負のミオン値を使用します(たとえば、[M-H]-mneutral = (m/z) * |z| - (- 1.007276) = (m/z) * |z| + 1.007276)。 - 式(3)を使用してCCSのパラメータを計算します。
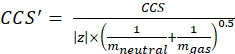 (3)
(3)
CCSでは、基準ドリフトチューブDTCCSN2値(nm2単位)、およびmgasはドリフトガスの質量(Da単位、窒素の場合:mgas = 28.01Da)。 - 式 (4) を使用して td' パラメータを計算します。
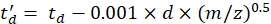 (4)
(4)
dでは、検出器の起動遅延は、デッドタイム(通常は〜1.5ms)を補正するために実験的に使用されました。 - 上記のパラメータの対数を計算します。
ln (CCS') および ln (t'd)
- 式(1)を使用してドリフト時間を計算します。
- 線形回帰を実行して、式(5)を使用して、対数適合のR2係数とxパラメータとyパラメータ(xは傾き、ln(y)は切片)を決定します。
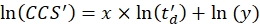 (5)
(5)
メモ: ln(CCS') vs ln(td') の値をプロットして、キャリブレーションの結果を視覚的に確認できますが、これはオプションです。
- すべてのキャリブレーションポイントについて、次の計算を行います。
- キャリブレーションを実験データに適用して、*.csv ファイルにエクスポートされたすべての IMS/IMS スペクトルについて、MZmine によって選択されたピークをキャリブレーションします。ポイントごとに、次の計算を行います。
- 式(6)を使用してドリフト時間を計算します。
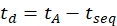 (6)
(6)
tseq を指定すると、最終的な IMS 分離に先立つ時間 (ステップ 3.4.6 で示された「時間 Abs」値)。
メモ: 異なるシーケンスで取得した複数の IMS/IMS スペクトルを較正する場合は、tseq 値を慎重に確認してください。 - Eq(7)を使用してイオンの中性質量を計算します。
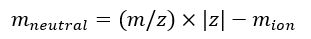 (7)
(7) - 式 (8) と式 (9) を使用して td' および td'' パラメータを計算します。
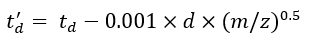 (8)
(8)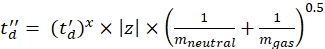 (9)
(9) - 式(10)を使用して、最終的な較正されたCCS値(nm2のTWCCSN2)を計算します。
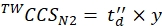 (10)
(10)
注:ステップ5.2.2ですが。はインターセプトとして ln(y) を与え、最終的な CCS 値を取得するには y を使用しなければなりません。指数関数を適用することを忘れないでください。
- 式(6)を使用してドリフト時間を計算します。
- ステップ2.4で取得した校正溶液の2回目の取得に校正を適用して、校正の精度を確認します。
注:キャリブレーションでは、約1〜2%の誤差が生じるはずです。
結果
アラビノキシラン五糖XA2XXを、このプロトコルを説明する例として選択した。この化合物は市販されているが、別のアラビノキシラン五糖との混合物としてのみ、XA3XX(純粋なXA3XXも市販されている)。XA2XX および XA3XX の構造は、補足図 S1 に示されています。市販の混合物中のXA2XXとXA3XXの比が〜50:50となるように、混合物の20μg/mLの?...
ディスカッション
SELECT SERIES サイクリックIMSは、上流のクロマトグラフィー分離を必要とせずに、所定の m/z とイオン移動度の定義済みイオン集団を選択できる強力なツールです。この装置は、このイオン集団の二次元フラグメンテーションマップを生成する可能性を提供し、そこからMS/MSスペクトルとIMS/IMSスペクトルの両方を抽出することができます。ただし、ユーザーは、実験プロセス中に注意を必...
開示事項
著者らは、開示する利益相反はありません。
謝辞
S.O.は、博士号(助成金ANR-18-CE29-0006)に資金を提供してくれたフランス国立研究庁に感謝しています。
資料
| Name | Company | Catalog Number | Comments |
| 33-α-L- plus 23-α-L-Arabinofuranosyl-xylotetraose (XA3XX/XA2XX) mixture | Megazyme Ltd., Wicklow, Ireland | O-XAXXMIX | XA2XX + XA3XX mixture |
| 33-α-L-Arabinofuranosyl-xylotetraose (XA3XX) | Megazyme Ltd., Wicklow, Ireland | O-XA3XX | Pure XA3XX standard |
| Eppendorf Safe-Lock Tubes, 1.5 mL, Eppendorf Quality, colorless, 1,000 tubes | Eppendorf, Hamburg, Germany | 0030120086 | Used to prepare the carbohydrate stock solution and dilution |
| FALCON 50 mL Polypropylene Conical Tube 30 x 115 mm | Corning Science México S.A. de C.V., Reynosa, Tamaulipas, Mexico | 352070 | Used to prepare the aqueous stock solution of 100 mM LiCl |
| Lithium Chloride (ACS reagent, ≥99 %) | Sigma-Aldrich Inc., Saint Quentin Fallavier, France | 310468 | Used to dope the sample with lithium |
| Major Mix IMS/Tof Calibration Kit | Waters Corp., Wilmslow, UK | 186008113 | Calibration solution for MS and IMS |
| MassLynx 4.2 SCN1016 Release 6 (Waters Embedded Analyser Platform for Cyclic IMS 2.9.1 Release 9) | Waters Corp., Wilmslow, UK | 721022377 | Cyclic IMS vendor software for instrument control and data processing |
| Methanol for HPLC PLUS Gradient grade | Carlo-Erba Reagents, Val de Reuil, France | 412383 | High-purity solvent |
| MS Leucine Enkephaline Kit | Waters Corp., Wilmslow, UK | 700002456 | Reference compound used for tuning of the mass spectrometer |
| SCHOTT DURAN 100 mL borosilicate glass bottle | VWR INTERNATIONAL, Radnor, Pennsylvania, US | 218012458 | Used to prepare the solution of 500 µM LiCl in 50:50 MeOH/Water |
| SELECT SERIES Cyclic IMS | Waters Corp., Wilmslow, UK | 186009432 | Ion mobility-mass spectrometer equipped with a cylic IMS cell |
| Website: http://mzmine.github.io/ | MZmine Development Team | - | Link to download the MZmine software |
| Website: https://github.com/siollivier/IM-MN | INRAE, UR BIA, BIBS Facility, Nantes, France | - | Link to an in-house R script containing a CCS calibration function |
参考文献
- Allard, P. -. M., et al. Integration of molecular networking and in-silico MS/MS fragmentation for natural products dereplication. Analytical Chemistry. 88 (6), 3317-3323 (2016).
- Wang, M., et al. Mass spectrometry searches using MASST. Nature Biotechnology. 38 (1), 23-26 (2020).
- David, M., Fertin, G., Rogniaux, H., Tessier, D. SpecOMS: a full open modification search method performing all-to-all spectra comparisons within minutes. Journal of Proteome Research. 16 (8), 3030-3038 (2017).
- Dührkop, K., et al. SIRIUS 4: a rapid tool for turning tandem mass spectra into metabolite structure information. Nature Methods. 16 (4), 299-302 (2019).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Nothias, L. -. F., et al. Feature-based molecular networking in the GNPS analysis environment. Nature Methods. 17 (9), 905-908 (2020).
- Gray, C. J., et al. Advancing solutions to the Carbohydrate Sequencing Challenge. Journal of the American Chemical Society. 141 (37), 14463-14479 (2019).
- Ropartz, D., et al. Online coupling of high-resolution chromatography with extreme UV photon activation tandem mass spectrometry: Application to the structural investigation of complex glycans by dissociative photoionization. Analytica Chimica Acta. 933, 1-9 (2016).
- Wolff, J. J., et al. Negative electron transfer dissociation of glycosaminoglycans. Analytical Chemistry. 82 (9), 3460-3466 (2010).
- Ropartz, D., et al. Charge transfer dissociation of complex oligosaccharides: comparison with collision-induced dissociation and extreme ultraviolet dissociative photoionization. Journal of the American Society for Mass Spectrometry. 27 (10), 1614-1619 (2016).
- Morelle, W., et al. Fragmentation characteristics of permethylated oligosaccharides using a matrix-assisted laser desorption/ionization two-stage time-of-flight (TOF/TOF) tandem mass spectrometer. Rapid Communications in Mass Spectrometry. 18 (22), 2637-2649 (2004).
- Gabelica, V., Marklund, E. Fundamentals of ion mobility spectrometry. Current Opinion in Chemical Biology. 42, 51-59 (2018).
- Gabelica, V., et al. Recommendations for reporting ion mobility mass spectrometry measurements. Mass Spectrometry Reviews. 38 (3), 291-320 (2019).
- Hernandez-Mesa, M., et al. Interlaboratory and interplatform study of steroids collision cross section by traveling wave ion mobility spectrometry. Analytical Chemistry. 92 (7), 5013-5022 (2020).
- Koeniger, S. L., et al. An IMS-IMS analogue of MS-MS. Analytical Chemistry. 78 (12), 4161-4174 (2006).
- Merenbloom, S. I., Koeniger, S. L., Valentine, S. J., Plasencia, M. D., Clemmer, D. E. IMS−IMS and IMS−IMS−IMS/MS for separating peptide and protein fragment ions. Analytical Chemistry. 78 (8), 2802-2809 (2006).
- Eldrid, C., Thalassinos, K. Developments in tandem ion mobility mass spectrometry. Biochemical Society Transactions. 48 (6), 2457-2466 (2020).
- Giles, K., et al. A cyclic ion mobility-mass spectrometry system. Analytical Chemistry. 91 (13), 8564-8573 (2019).
- Merenbloom, S. I., Glaskin, R. S., Henson, Z. B., Clemmer, D. E. High-resolution ion cyclotron mobility spectrometry. Analytical Chemistry. 81 (4), 1482-1487 (2009).
- Ollivier, S., et al. Anomeric retention of carbohydrates in multistage cyclic ion mobility (IMSn): de novo structural elucidation of enzymatically produced mannosides. Analytical Chemistry. 93 (15), 6254-6261 (2021).
- Ollivier, S., Fanuel, M., Rogniaux, H., Ropartz, D. Molecular networking of high-resolution tandem ion mobility spectra: a structurally relevant way of organizing data in glycomics. Analytical Chemistry. 93 (31), 10871-10878 (2021).
- Aron, A. T., et al. Reproducible molecular networking of untargeted mass spectrometry data using GNPS. Nature Protocols. 15 (6), 1954-1991 (2020).
- McKenna, K. R., Li, L., Krishnamurthy, R., Liotta, C. L., Fernández, F. M. Organic acid shift reagents for the discrimination of carbohydrate isobars by ion mobility-mass spectrometry. The Analyst. 145 (24), 8008-8015 (2021).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11, 395 (2010).
- Ruotolo, B. T., Benesch, J. L. P., Sandercock, A. M., Hyung, S. -. J., Robinson, C. V. Ion mobility-mass spectrometry analysis of large protein complexes. Nature Protocols. 3 (7), 1139-1152 (2008).
- Bush, M. F., Hall, Z., Giles, K., Hoyes, J., Robinson, C. V., Ruotolo, B. T. Collision cross sections of proteins and their complexes: a calibration framework and database for gas-phase structural biology. Analytical Chemistry. 82 (22), 9557-9565 (2010).
- Ropartz, D., et al. Structure determination of large isomeric oligosaccharides of natural origin through multipass and multistage cyclic traveling-wave ion mobility mass spectrometry. Analytical Chemistry. 91 (18), 12030-12037 (2019).
- Tolmachev, A. V., et al. Characterization of ion dynamics in structures for lossless ion manipulations. Analytical Chemistry. 86 (18), 9162-9168 (2014).
- Arndt, J. R., et al. High-resolution ion-mobility-enabled peptide mapping for high-throughput critical quality attribute monitoring. Journal of the American Society for Mass Spectrometry. 32 (8), 2019-2032 (2021).
- Le Fèvre, A., Dugourd, P., Chirot, F. Exploring conformational landscapes using trap and release tandem ion mobility spectrometry. Analytical Chemistry. 93 (9), 4183-4190 (2021).
- Ohshimo, K., He, X., Ito, R., Misaizu, F. Conformer separation of dibenzo-crown-ether complexes with Na+ and K+ ions studied by cryogenic ion mobility-mass spectrometry. The Journal of Physical Chemistry A. 125 (17), 3718-3725 (2021).
- Purves, R. W., Barnett, D. A., Ells, B., Guevremont, R. Gas-phase conformers of the [M + 2H]2+ ion of bradykinin investigated by combining high-field asymmetric waveform ion mobility spectrometry, hydrogen/deuterium exchange, and energy-loss measurements. Rapid Communications in Mass Spectrometry. 15 (16), 1453-1456 (2001).
- Ujma, J., et al. Cyclic ion mobility mass spectrometry distinguishes anomers and open-ring forms of pentasaccharides. Journal of the American Society for Mass Spectrometry. 30 (6), 1028-1037 (2019).
- Warnke, S., Faleh, A. B., Scutelnic, V., Rizzo, T. R. Separation and identification of glycan anomers using ultrahigh-resolution ion-mobility spectrometry and cryogenic ion spectroscopy. Journal of The American Society for Mass Spectrometry. 30 (11), 2204-2211 (2019).
- Williamson, D. L., Bergman, A. E., Nagy, G. Investigating the structure of α/β carbohydrate linkage isomers as a function of group I metal adduction and degree of polymerization as revealed by cyclic ion mobility separations. Journal of the American Society for Mass Spectrometry. 32 (10), 2573-2582 (2021).
- Myers, O. D., Sumner, S. J., Li, S., Barnes, S., Du, X. One step forward for reducing false positive and false negative compound identifications from mass spectrometry metabolomics data: new algorithms for constructing extracted ion chromatograms and detecting chromatographic peaks. Analytical Chemistry. 89 (17), 8696-8703 (2017).
- Marchand, A., Livet, S., Rosu, F., Gabelica, V. Drift tube ion mobility: how to reconstruct collision cross section distributions from arrival time distributions. Analytical Chemistry. 89 (23), 12674-12681 (2017).
- Davis, D. M., et al. Analysis of ion mobility spectra for mixed vapors using Gaussian deconvolution. Analytica Chimica Acta. 289 (3), 263-272 (1994).
- Polasky, D. A., Dixit, S. M., Fantin, S. M., Ruotolo, B. T. CIUSuite 2: next-generation software for the analysis of gas-phase protein unfolding data. Analytical Chemistry. 91 (4), 3147-3155 (2019).
- Salbo, R., et al. Traveling-wave ion mobility mass spectrometry of protein complexes: accurate calibrated collision cross-sections of human insulin oligomers. Rapid Communications in Mass Spectrometry. 26 (10), 1181-1193 (2012).
- Gelb, A. S., Jarratt, R. E., Huang, Y., Dodds, E. D. A study of calibrant selection in measurement of carbohydrate and peptide ion-neutral collision cross sections by traveling wave ion mobility spectrometry. Analytical Chemistry. 86 (22), 11396-11402 (2014).
- Richardson, K., Langridge, D., Dixit, S. M., Ruotolo, B. T. An improved calibration approach for traveling wave ion mobility spectrometry: robust, high-precision collision cross sections. Analytical Chemistry. 93 (7), 3542-3550 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved