需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
通过表面等离子体共振 设计 抗病毒药物
摘要
本协议描述了用于SPR结合测定的新工具,以检查CV-N与HA,S糖蛋白,相关杂交型聚糖和高甘露糖寡糖的结合。SPR用于测定将二聚体或单体CV-N与这些聚糖结合的KD 。
摘要
表面等离子体共振(SPR)用于测量血凝素(HA)与结构域交换的氰基蛋白-N(CV-N)二聚体的结合,并监测甘露糖基化肽与CV-N高亲和力结合位点之间的相互作用。据报道,病毒包膜加标 gp120、HA 和埃博拉糖蛋白 (GP) 1,2 可结合二聚体 CVN2 上的高亲和力和低亲和力结合位点。二甘露糖基化的HA肽也在两个低亲和力结合位点与CVN2的工程分子结合,CVN2具有相应配体的高亲和力位点并发生突变以取代碳水化合物结合口袋中的稳定二硫键,从而确认多价结合。HA与假抗体CVN2的一个高亲和力结合位点显示,解离常数(KD)为275 nM,通过寡聚化进一步中和人类免疫缺陷病毒1型(HIV-1)。将结构域交换的CVN2中的二硫键数量从4个减少到2个,通过将胱氨酸取代为谷氨酸和精氨酸的极性残基对,导致与HA的结合亲和力降低。在最强的相互作用中,埃博拉GP1,2与CVN2结合,使用没有跨膜结构域的包膜聚糖在较低的纳摩尔范围内具有两个高亲和力结合位点。在本研究中,多特异性单体CV-N与严重急性呼吸综合征冠状病毒2(SARS-CoV-2)刺突(S)糖蛋白的结合在K D = 18.6μM处测量,与纳摩尔KD与其他病毒尖峰相比,并通过其在中摩尔范围内的受体结合域在μ中摩尔范围内。
引言
Tetherin相关的抗病毒活性由干扰素-α诱导,它包含基于蛋白质的Tether,导致完全形成的病毒粒子保留在感染的细胞表面1。Tetherin糖基化在抑制病毒释放方面的必要性仍然不确定,这意味着糖基化模式对重组表达聚糖的重要性对于体外研究1,2,这取决于(在流感病毒的情况下)表面表达的流感血凝素HA3,4的构象.已经注意到,与N-连接糖基化相关的寡糖的修饰足以限制HIV-1型释放2,而二聚化在防止病毒释放中起着至关重要的作用,因此涉及跨膜结构域或糖基磷脂酰肌醇(GPI)锚,用于拴系出芽病毒粒子5.描述了人和鼠系留蛋白的独特功能,以阻断多种包膜病毒、逆转录病毒和丝状病毒。BST-2/tetherin是一种先天免疫1,6的干扰素诱导抗病毒蛋白,具有广谱抗病毒活性,并被包膜糖蛋白5拮抗以易位系留蛋白或破坏Tetherin6的结构。例如,表面表达的包膜糖蛋白HA和神经氨酸酶对甲型流感病毒的拮抗作用是众所周知的,以菌株特异性方式7,促进宿主受体结合位点8的识别。在与HA上快速定制的聚糖屏蔽相互作用的化学计量学中研究聚糖靶向抗体,从而与甲型H3N2和H1N1亚型4结合亲和力。
为了阐明抗病毒药物与病毒包膜尖峰(即碳水化合物配体)之间的结合机制,以及互补的免疫学和光谱学方法,对单甘露糖、二甘露糖和三甘露糖部分进行了化学合成。甘露糖基化肽是通过糖基{β}-过乙酸酯的叠氮基化为1,2-反式糖基叠氮化物转化9而创建的,模仿了在危及生命的病毒表面上通常发现的N-乙酰氨基葡萄糖和高甘露糖低聚糖。三唑生物等排体用于模拟形成 HA 肽10 甘露糖基化残基的键,并促进与 HA 头结构域上第二个 N 连接糖基化点周围的抗病毒 CV-N 衍生物的位点特异性相互作用(HA 顶部有 4 个 N-连接聚糖 N54、N97、N181、N301)8,11,12.谷氨酸(Glu)和精氨酸(Arg)之间的相互作用以及由此产生的螺旋偶极子表现出模型肽和蛋白质的良好稳定性,但使用SPR可视化。如果与通过直接抑制聚糖部分上的受体结合来识别HA10上的单个化学合成糖基化位点相比,则显示四位点突变Fc结构与其受体的更高亲和力可在体内引发效应功能,揭示连接到Fc突变体的N-连接聚糖的不相关组成有待机械确定13。
CV-N 显示出针对 HIV 14,15、流感 16 和埃博拉病毒的抗病毒活性,这是通过纳摩尔结合包膜刺突蛋白12,17,18,19 上的高甘露糖寡糖修饰介导的。确定流感HA与CV-N中的一个高亲和力碳水化合物结合位点(H)或共价连接的二聚体CVN2中的两个Hs结合分别具有平衡解离常数(K D)= 5.7 nM(图1A)和KD = 2.7 nM。CV-N和CVN2都含有另外一个或两个低亲和力碳水化合物结合位点(L)s12,17,20,21。埃博拉GP1,2与CVN2的2H结合,亲和力在较低的纳摩尔范围(KD = 26 nM)。与埃博拉GP1,2和HA结合的CV-N WT表现出从K D = 34 nM到KD = 5.7 nM的亲和力(A/New York/55/04)12。凝集素,如CV-N,专门针对病毒包膜上的高甘露糖聚糖,进一步抑制丙型肝炎病毒、SARS-CoV、疱疹病毒、马尔堡病毒和麻疹病毒22的复制。
CV-N小分子已经彻底研究了20多年,因为它的功能可以结合多种病毒以抑制病毒进入16,18。结构分析和结合亲和力测定表明,通过微摩尔范围内的二价结合,在结构域交换的CVN2二聚体中交联两个L,以增强对病毒包膜糖蛋白10,19的亲和力。Manα1-2Manα在Man(8)D1D3臂上的选择性结合,Man(9)包括位于相反蛋白质原型体20上的两个具有不同亲和力的结合位点,从而达到纳摩尔结合亲和力(图1B)。因此,CVN2被认为是一种假抗体,因为它在HIV gp120上结合表位的应用,类似于病毒中和抗体17。在本文中,作者有兴趣研究CVN2通过其受体结合域(RBD) 与 SARS-CoV-2尖峰的潜在结合。固定化人血管紧张素转换酶 (ACE)-2 与 SARS-CoV-2 RBD 的结合曲线导致这种生物学相关的结合相互作用的 KD = 4.7 nM23。
相比之下,选定的免疫球蛋白类别可识别特定且一致的结构蛋白模式,这些模式为膜锚定的HA区域赋予亲和力成熟的底物24。CV-N在几乎所有甲型和乙型流感病毒中都显示出高效的活性16,它是一种广泛的中和抗病毒剂。我们对HA1和HA2茎上靶向表位位置的了解不完整,这些表位可能涉及通过高度中和抗体靶向聚糖的表位结构,并与凝集素结合25相比。
图 1:CV-N 与病毒包膜尖峰的 SPR 结合测定示意图。 (A)用于CV-N与配体结合的SPR测定:HA全长蛋白(90kDa)。动力学数据集(5120、2560、1280、640、320、160、80、40、20、10、5、2.5、0 nM)显示与甲型流感HA/纽约/55/04(H3N2)的实时双重参照结合。(B)CVN2L0变体V2与固定配体DM结合,浓度范围为500nM至16μM。 序:L残基以黄色突出显示。H 残基以灰色突出显示。E58和R73是野生型蛋白中半胱氨酸的替代品,使V2成为稳定的蛋白质折叠,具有三个而不是四个二硫键 请单击此处查看此图的大图。
虽然膜-远端 HA 顶部的聚糖屏蔽诱导与 CV-N 12 的高亲和力结合,但在其低亲和力位点10,12 进一步观察到CVN2 与 HA 顶部二硫键相邻的 HA 结合。通过CV-N在碳水化合物结合中鉴定各种极性相互作用和相互作用位点。通过在结合位点生成敲除变体来验证这些相互作用,以将结合亲和力与计算机预测的糖基化相关联12。因此,该项目旨在比较先前测试的化学甘露糖基化HA肽与SARS相关2019-nCoV尖峰和SARS-CoV-2的短肽序列的结合亲和力和特异性,这些短肽序列由少量不同的N-连接糖基化位点和O-连接糖基化修饰。使用冷冻电子显微镜和结合测定,Pinto及其同事报告了一种单克隆抗体S309,该抗体可能识别SARS-CoV-2刺突蛋白上的表位,该表位含有沙贝病毒亚属内的保守聚糖,而不会与受体附着竞争26。本研究的方案描述了设计、表达和表征 CV-N 变体对于使用 SPR 技术研究 CV-N 和 CVN2 如何与糖基化蛋白质和合成甘露糖基化肽结合的重要性10,12.
串联连接的二聚体CVN2L027和结合位点变体(V2-V5)重组表达,变体具有二硫键替代物(C58E和C73R)(图2A)。此外,制备具有单点突变E41A的突变体,因为该位置已被视为分子间交叉接触残基。这种突变体是另一种有趣的分子,用于凝集素和高甘露糖寡糖之间的SPR结合测量,破译结合结构域,并允许与二聚体形式进行比较。CVN2的结构域交换晶体结构显示出一个灵活的接头,其延伸在49到54个残基之间。这两个结构域可以作为刚体继续围绕铰链移动,通过分子内结构域相互作用(结构域A-残基1-39;90-101-与结构域B-残基40-89)或通过分子间结构域交换产生二聚体[域A(第一个单体)与结构域B(第二个),以及域B(第一个单体)与结构域A(第二个拷贝)]。除了Glu4128之外,两个原型的A和B结构域之间没有密切的相互作用。CV-N的基因可以使用重复PCR方法与40-mer合成的寡核苷酸29进行开发,然后亚克隆到pET11a的NdeI和BamHI位点中,以转化为(电穿孔)为电感受态细胞,如Keeffe,J.R.27所述。该蛋白质用于实现各自的晶体结构(PDB ID 3S3Y),包括一个N-末端6-组氨酸纯化标签,后跟一个因子Xa蛋白酶切割位点。定点诱变用于进行点突变、切换密码子以及插入或删除单个或多个碱基或密码子以进行氨基酸交换。这些转化为蛋白质功能和结构提供了宝贵的见解。重组表达和纯化的CV-N、CVN2和CVN3已经过生物物理研究20,21,27,生产成本低廉,因此用于表征固定在SPR传感器芯片上的聚糖的结合测定。传统的酶联免疫吸附测定(ELISA)在聚糖配体的固定化技术方面提供了较低的重现性,并将各种结合位点变体的实时结合(如SPR所示)转化为终点测定。
结合亲和变体CVN2L0-V2(具有二硫键取代10的同源二聚体CV-N的完整折叠)在大肠杆菌(大肠杆菌)中用His标签表达,在Ni-NTA 柱上应用亲和色谱纯化,并使用SPR测试与HA(H3N2),单甘露糖基化HA肽和二甘露糖基化HA肽的结合。 化学甘露糖基化肽或HA和S蛋白都是配体和胺偶联到亲水芯片表面通过反应性酯或生物素-链霉亲和素蛋白工程。将相同的顺序运行程序应用于这些配体,注入CV-N的各种稀释液和CV-N(和CVN2)的变体,以获得分子相互作用分析的动力学信息,如下所述30。RBD固定化SPR传感器芯片用于CV-N与S肽的结合研究,并将亲和力与SARS-CoV-2与人ACE2的结合进行比较。
研究方案
在本研究中,CVN小泛素样修饰剂(SUMO)融合蛋白已用于酶联免疫吸附测定而不是CV-N,适用于基于细胞的测定。根据标准方案12,商业获得重组全长甲型流感病毒HA H3蛋白(见材料表)或在哺乳动物HEK293细胞系和杆状病毒感染的昆虫细胞中表达。武汉-1刺突蛋白在哺乳动物HEK293细胞中表达。单甘露糖基化肽(MM)和二甘露糖基化肽(DM)的合成允许检测CVN2和单甘露糖基化小分子的均相配体10。
1. 创建 CV-N 构造
- 对于每个CVN2变体和CVN2L0蛋白(PDB ID 3S3Y),从商业来源获得具有N末端pelB前导序列和pET27b(+)载体中的His标签的基因构建体(参见 材料表,补充文件1)。
- 获取CVN2L0及其变体(V2,V3,V4和V5;图2A,C)在CVN2L0模板基因的背景中,该基因由每个CV-N重复的两个不同的DNA序列组成。
- 将冻干质粒DNA溶解在无菌去离子蒸馏水(ddH2O)中至终浓度为100ng / μL。
2. 质粒DNA转化细胞制备LB-琼脂平板
- 通过将 10 g/L 蛋白胨、5 g/L 酵母提取物和 5 g/L NaCl 溶解在 ddH2O 中来制备培养基 LB-Lennox(参见材料表),并将 pH 值调节至 7.4。按照先前发表的报告10,通过化学方法将每种变体(V2-V5)转化为感受态大肠杆菌BL21(DE3)。
- 拆分溶液(900 μL 和 100 μL),在 LB-琼脂平板(50 μg/mL 卡那霉素)上转移 100 μL,然后轻轻使用无菌细胞扩散器。将琼脂平板在37°C孵育过夜。
3. 克隆
- 按照参考文献27将CV-N基因亚克隆到pET11a的NdeI和BamHI位点(见材料表)中,以转化为(电穿孔)到电感受态细胞中。
4. 定点诱变
- 在CVN2L0模板基因的背景中产生CVN2L0 29 和突变CVN-E41A,该基因包含每个CV-N重复序列27的两个可区分的DNA序列。
- 使用定点诱变试剂盒(参见材料表)和特异性诱变引物5'-gagaaccgtcaacgtttgcgataacagagttcagg-3'和5'-cctgaactcttatcgcaaacgttgacggttc-3'进行突变,用于运行PCR31。
- 使用多种浓度的双链 DNA (dsDNA) 模板(范围为 5-50 ng)(例如,5、10、20 和 50 ng 的 dsDNA 模板)开始一系列样品反应。保持引物浓度恒定。
注意:PCR混合物和热循环方案通常按照定点诱变试剂盒32的说明手册中所述使用。
- 使用多种浓度的双链 DNA (dsDNA) 模板(范围为 5-50 ng)(例如,5、10、20 和 50 ng 的 dsDNA 模板)开始一系列样品反应。保持引物浓度恒定。
- 在矿物油覆盖层下方添加 Dpn I 限制性内切酶(1 μL、10 U/μL,参见 材料表)。彻底轻轻地混合反应物,在台式微量离心机中旋转1分钟,并立即在37°C孵育1小时以消化亲本超螺旋dsDNA。
5. 细菌细胞的转化
- 将XL1-Blue超感受态细胞(见 材料表)在冰上轻轻解冻。要转化每个对照和样品反应,请将超感受态细胞 (50 μL) 等分到预冷的聚丙烯圆底管 (14 mL) 中。
- 从每个对照和样品反应(突变的 ssDNA)中转移 1 μL Dpn I 处理的单链 DNA (ssDNA),以分离合成互补链的超感受态细胞的等分试样。小心旋转转化反应,将反应在冰上混合并孵育30分钟。
注意:在将Dpn I处理的DNA转移到转化反应之前,建议小心地从移液器吸头中去除任何残留的矿物油。作为可选对照,需要通过将 0.1 ng/μL 的 pUC18 对照质粒 (1 μL) 与 50 μL 等分试样的超感受态细胞混合来检查 XL1-Blue 超感受态细胞的转化效率。
- 从每个对照和样品反应(突变的 ssDNA)中转移 1 μL Dpn I 处理的单链 DNA (ssDNA),以分离合成互补链的超感受态细胞的等分试样。小心旋转转化反应,将反应在冰上混合并孵育30分钟。
- 在42°C下对转化反应施加热脉冲45秒,然后将反应放在冰上2分钟。
注意:施加的热脉冲已针对聚丙烯圆底管(14 mL)中的上述条件进行了优化。 - 加入0.5mL NZY+肉汤(每升含有:10gNZ胺(酪蛋白水解物),5g酵母提取物,5gNaCl,12.5mL1M MgCl2,12.5mL1M MgSO4,10mL2M葡萄糖,pH 7.5,并预热至42°C),并在37°C下以225-250rpm振荡孵育转化反应1小时。将每个转化反应的正确体积(对照质粒转化5 μL;样品转化250 μL)接种在LB-氨苄西林琼脂平板上。
注意:对于转化对照和诱变,将细胞散布在具有5-溴-4-氯-3-吲哚基-β-D-吡喃半乳糖苷(X-gal,80μg/ mL)和异丙基-1-硫代-β-D-吡喃半乳糖苷(IPTG,20mM)的LB-氨苄西林琼脂平板上(参见 材料表)。用转化的 E的单个菌落接种50 mL细胞培养物。 大肠杆菌 细胞,用于纯化突变的质粒DNA进行分析。诱变通过外部设施的DNA测序来确认。
6. 表达和蛋白纯化
- 对于大规模培养,用转化板中的单个菌落接种少量LB(含有氨苄青霉素)。
- 使用过夜培养物,用添加剂接种表达培养物,例如 10 mM 的 MgCl2、10 mM 的 MgSO4 和 20 mM 的葡萄糖,将种子培养物稀释至 1/100。
- 在37°C下剧烈摇动生长细胞。 在将细胞冷却至20°C之前,将细胞生长至0.4-0.6(对数中期)之间的Abs 600nm。 用1mM IPTG诱导并生长过夜。
- 然后,通过在4°C下以4,000× g 离心15分钟来收获细胞,并用移液管弃去上清液。
- 将细胞沉淀重悬于磷酸盐缓冲盐水(PBS)缓冲液中,并在4°C下以4,000× g 重新离心15分钟。 然后,用移液管弃去上清液。将剩余的沉淀重悬于10mL裂解缓冲液中,并将悬浮液在37°C孵育1小时。
注意:裂解缓冲液的组成:(50 mM NaH 2 PO4、300 mM NaCl、2% Triton-X100、500 ng/mL 溶菌酶、1 mM 苯甲基磺酰氟 (PMSF)、1 mM 二硫苏糖醇、1 mM MgCl2,pH 8,参见材料表)。- 将混合物置于两个冻融循环(-80°C)下。通过在4°C下以4,000× g 离心15分钟来分离可溶性和不溶性部分,并使用聚丙烯酰胺凝胶电泳(PAGE)分析它们,特别是十二烷基硫酸钠(SDS)-PAGE33(图2B,C)。
注意:细胞可以通过多种方式裂解,例如冻融,超声处理,匀浆,酶裂解或这些方法的组合。建议从包涵体中纯化以收集高蛋白产量26。
- 将混合物置于两个冻融循环(-80°C)下。通过在4°C下以4,000× g 离心15分钟来分离可溶性和不溶性部分,并使用聚丙烯酰胺凝胶电泳(PAGE)分析它们,特别是十二烷基硫酸钠(SDS)-PAGE33(图2B,C)。
- 使用Ni-NTA柱色谱纯化蛋白质(见 材料表)。将可溶性级分加载到再生的Ni-NTA珠柱(1 mL / min)上。用TBS缓冲液(50mM的Tris,150mM的氯化钠,pH 7.5)洗涤系统,然后开始梯度(0-100%500mM咪唑在TBS中超过60分钟)并收集馏分(1mL / min)。透析纯化的蛋白质以针对100mM PBS进行生化表征(图2C,D)。
- 或者,在 14 mL 管中使用 His-Select Ni 2+ 亲和凝胶(参见材料表),分别用20 mM 咪唑和 250 mM 咪唑在缓冲溶液中结合并重悬 his-Zero 标记的重组表达的 CV-N。分批孵育至少30分钟。
注意:将这些半纯化的蛋白质应用于一次性预填充柱,用于缓冲液置换和生物样品(例如碳水化合物和蛋白质)的纯化,这些样品可以从固定化金属亲和色谱中加载1-1.5 mL洗脱液。 - 将蛋白质溶液转移到带有10kDa截止过滤器的离心管中(参见 材料表),并通过在4,500× g 和4°C下离心10分钟来浓缩它们。 对于 SPR 测量,将分析物溶液交换为 10 mM HEPES、150 mM 氯化钠、3 mM 乙二胺四乙酸 (EDTA) 和 0.05% 吐温20,pH 7.4 (HBS-EP(+),参见 材料表)。
- 将此SPR电泳缓冲液加入1:10的稀释因子,并在4,500× g和4 °C下离心四次至初始体积10分钟。
- 使用NanoDrop紫外-可见分光光度计(参见材料表)确定280nm处的蛋白质浓度,基于计算出的主要蛋白质CVN2L0的消光系数(20,440 M-1 cm-1),显示大小为23,474 Da34。使用PBS(100 mM,pH 7.0)或SPR缓冲液作为空白,并在三个稀释步骤(1:1,1:10和1:100)下测量蛋白质浓度。
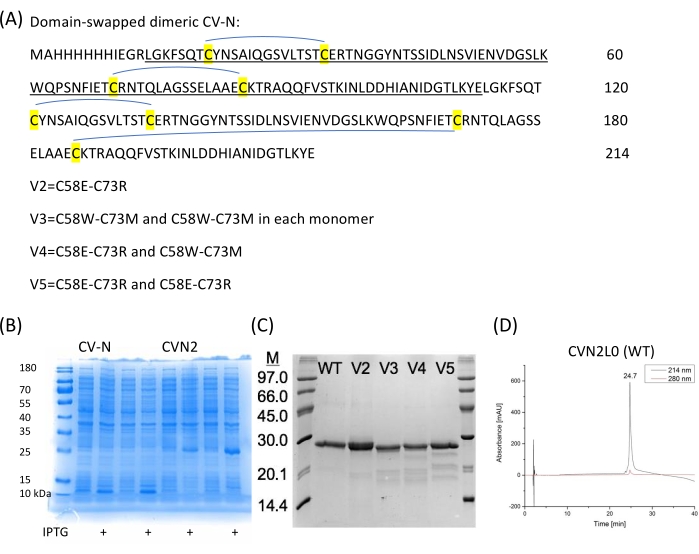
图2:CV-N序列和表达 。 (A)每个CV-N重复序列(每个101个氨基酸)和四个二硫键之间没有接头的CVN2在 E中的pET11a载体中表达。 大肠杆菌。(B)CV-N(单体)和CVN2(二聚体)两个独立菌落的表达。(C)在SDS-PAGE上纯化和分析二硫键变体。使用低分子量标记物(6 μL)作为参考。WT = CVN2L0 轴承 (A) 中标记的四个二硫键。V2 是一种变体,在位置 58 和 73 处被极性残基取代二硫键。V3-V5是具有两个剩余的S-S键的变体,并且极性(C58E-C73R)或非极性(C58W-C73M)取代氨基酸或这些残基对取代的组合。(D) 在缓冲液 A 中以 1 mL/min 的流速洗脱纯化 CVN2L0 的 HPLC 色谱图,线性梯度为 5%-65% 缓冲液 B,超过 30 分钟。 缓冲液 A 为:0.1% (v/v) 三氟乙酸的 ddH2O 溶液,缓冲液 B 为:0.08% (v/v) 三氟乙酸乙腈溶液。蛋白质在高性能硅胶300-5-C4(150 x 4.6 mm)色谱柱上以214 nm和280 nm进行分析。 请点击此处查看此图的大图。
7. SPR 光谱
- 使用双通道SPR系统(参见 材料表)和电泳缓冲液HBS-EP(+)和10 mM甘氨酸HCl pH 1.5-1.6作为再生缓冲液。打开仪器,脱气器,自动进样器,泵送并用ddH20洗涤整个系统1小时。将即用型电泳缓冲液放入单独的瓶中。
- 将艾默松油滴到检测器上,并将涂有薄金膜的玻璃传感器芯片(见 材料表)安装在三端口流通池下方的检测器上,并在上侧用羧甲基葡聚糖水凝胶功能化。通过下拉手柄来修复设置。
注意:C19RBDHC30M 200 nm链霉亲和素衍生化羧甲基葡聚糖水凝胶具有中等密度的生物素化严重急性呼吸综合征冠状病毒-2 RBD蛋白,是一种具有预固定配体的即用型传感器芯片。
8. 用于CV-N与HA,S蛋白和RBD结合的SPR结合测定
- 按照以下步骤将蛋白质配体固定到传感器芯片上。
- 通过单击菜单栏中的 "表单 "和集成的 SPRAutoLink 软件中的"运行表编辑器"打开 运行表 (请参阅 材料表)。从可用运行表列表中选择 并单击BASIC_Immobilization ,然后按照计算机屏幕上的实验程序步骤进行操作。使用的相应示例编辑器显示在右上角。
- 单击"表格"部分中的 "样品集编辑器 ",填写放置在自动进样器中的两个样品架的试剂列表,以便进一步分析。单击菜单栏中的 "工具"自动进样器直接控制 ,将样品架向前或带回原位。选择4°C作为工作温度。
注意:SPR 软件允许通过选择相应的工具以及自动进样器处理和单击"表格"来实现"SPR 仪器直接控制"和"泵直接控制";还可以选择运行表编辑器、数据绘图或后处理来执行数据分析。文件直接保存在默认目录中,并从"后处理"窗口导出为 scrubber.files。
- 通过单击工具和泵直接控制启动泵以注入 ddH20,并通过单击 SPR 仪器直接控制记录数据,并且每次在新出现的窗口中启动。将偶联试剂(步骤8.3)放入300 μL小瓶中,将其放入自动进样器架中,然后单击运行启动运行表。
注意:芯片表面要么用 10 mM 甘氨酸缓冲液 pH 9.0 调节,要么用 1 M 氯化钠、0.1 M 硼酸钠缓冲液 pH 9.0 冲洗,以调节羧基衍生化芯片表面,用于 EDC/NHS 活化混合物30。 - 对于这种简单的蛋白质-蛋白质相互作用,使用CMD500D芯片(参见 材料表)生成具有固定化HA的微折射率单位(μRIU)= 2500 - 3000流通池和具有刺突蛋白的μRIU = 400流通池。以 15 μL/min 的输注流速,通过应用以下顺序步骤,注入 0.4 M N-乙基-N'-(二甲氨基丙基)碳二亚胺盐酸盐 (EDC*HCl) 和 0.1 M N-羟基琥珀酰亚胺 (NHS) 的水性等量混合物。
- 以 25,000 μL/min 的速度重新填充泵,执行基线调整 30 秒,在 6 分钟的接触时间内注入 90 μL 样品活化溶液 (EDC/NHS),然后再保持 5 分钟。
- 基线后仅以 10 μL/min 在左侧流通池(蓝色)上运行 1 分钟后重复此循环,以 20 μg/mL 注入和固定化学合成的肽10、HA 和刺突蛋白,并允许随后用 ddH2O 进行基线调整 1.5 分钟,然后用 1 M 乙醇胺 HCl pH 8.5 淬灭活化的芯片表面。
- 将管从液体进样器切换到脱气器,从ddH20到使用HBS-EP(+)进入瓶中(补充图1)。
- 分析 SPR 传感器图。
- 要进行动力学研究,请使用各种分析物浓度(10-5-10-8 M),每次进样后进行再生步骤,并在不同的分析物后进行空白测量。将流速更改为10μL/min并开始进样4分钟接触时间,然后生成5分钟基线,以及两个再生步骤,每个步骤2分钟,间隔为30秒。
- 注入缓冲溶液进行空白测量,从样品运行中减去传感器图以标准化不同的蛋白质浓度。
- 单击 表单,向下滚动,然后单击此操作模式更改为"后处理"。单击 "添加 "以选择在数据绘图表中为每个流通池随时间生成的结合曲线,并将叠加层导出为洗涤器文件 (.ovr)。单击"文件"以打开文件保存选项。通过对齐左右曲线并从配体通道的信号中减去第二个参考通道的信号来获得响应曲线。
注意:数据在"后处理"中通过计算定义传感器图进行操作。它被放入来自左右流通池的传感器图的叠加中,或表示为来自两个通道差异的传感器图。 - 在结合测量前后,用 50-100 mM 甘氨酸缓冲液 pH 9.5、水和 20% 乙醇清洁整个流体,以去除痕量盐或任何蛋白质污染,或更严格地使用 0.5% SDS 和甘氨酸。
注意:为防止仪器损坏,如果芯片存放在缓冲液下或100%湿度下,建议在将芯片盒重新插入仪器之前检查玻璃芯片的机械稳定性。
结果
在三个独立的SPR实验中测试二聚结构域交换的CVN2L0分子是否与HA顶部区域结合,并以KD值表示结合亲和力。假设结构域B包含H结合位点,其通过取代二硫键进入离子残基而受到影响,并且结构域A形成L10,18。首先测试CVN2L0和变体V2(三个二硫键)和V5(两个二硫键)的单次进样是否与HA耦合传感器芯片结合,以估计结合能力,然后再使用自动进样器...
讨论
CV-N的结合亲和力与功能结合位点的数量相关[当工程为结构域交换二聚体时,结构域B上为2H,结构域A为2L]。在大肠杆菌中表达具有改变的结合亲和力的变体(CVN2L0-V2,CV-N的同源二聚体稳定折叠,包括二硫键敲除),纯化,并使用SPR10对HA蛋白(H3N2)结合进行阳性测试,并且在HA与H或L碳水化合物结合位点和KD1 = 49 nM和KD2 = 8μM38时显示出构象...
披露声明
作者没有什么可透露的。
致谢
作者感谢维也纳工业大学生物技术和微生物学系和维也纳医科大学肾脏病学和透析学系第三医学系的Christian Derntl博士,特别是Markus Wahrmann博士的技术和科学支持。哺乳动物细胞中的蛋白质表达得到了维也纳自然资源与生命科学大学(BOKU)生物技术系的支持。作者希望对来自德国杜塞尔多夫XanTec生物分析的Nico Dankbar博士表示深深的感谢,感谢他对进行SPR结合测定的有益科学讨论。
材料
| Name | Company | Catalog Number | Comments |
| Äkta primeplus | Cytiva | ||
| Amicon tubes | Merck | C7715 | |
| Ampillicin | Sigma-Aldrich | A5354 | |
| Beckmann Coulter Cooler Allegra X-30R centrifuge | Beckman Coulter | B06320 | |
| Cell spreader | Sigma-Aldrich | HS86655 | silver stainless steel, bar L 33 mm |
| Custom DNA Oligos | Sigma-Aldrich | OLIGO | |
| Custom Gensynthesis | GenScript | #1390661 | cloning vector: pET27b(+) |
| Cytiva HBS-EP+ Buffer 10, 4x50mL | Thermo Scientific | 50-105-5354 | |
| Dionex UlitMate 3000 | Thermo Scientific | IQLAAAGABHFAPBMBFB | |
| Dpn I restriction enzyme (10 U/μL) | Fisher Scientific | ER1701 | |
| DTT | Merck | DTT-RO | |
| EDC | Merck | 39391 | |
| EDTA | Merck | E9884 | |
| Eppendorf Safe-Lock Tubes | Eppendorf | 30120086 | |
| Eppendorf Safe-Lock Tubes | Eppendorf | 30120094 | |
| Eppendorf Minispin and MiniSpin Plus personal microcentrifuge | Sigma-Aldrich | Z606235 | |
| Ethanol | Merck | 51976 | |
| Ethanolamine HCl | Merck | E6133 | |
| Falcon 50mL Conical Centrifuge Tubes | Fisher Scientific | 14-432-22 | |
| Falcon 14 mL Round Bottom Polystyrene Test Tube, with Snap Cap, Sterile, 25/Pack | Corning | 352057 | |
| Glucose | Merck | G8270 | |
| Glycine HCl | Merck | 55097 | |
| HA H3 protein | Abcam | ab69751 | |
| HEPES | Merck | H3375 | |
| His-select Ni2+ | Merck | H0537 | |
| Imidazole | Merck | I2399 | |
| IPTG | Merck | I6758 | |
| Kanamycin A | Sigma-Aldrich | K1377 | |
| Kromasil 300-5-C4 | Nouryon | ||
| LB agar | Merck | 52062 | |
| LB agar | Merck | 19344 | |
| LB Lennox | Merck | L3022 | |
| Lysozyme | Merck | 10837059001 | |
| Magnesium chloride | Merck | M8266 | |
| Magnesium sulfate | Merck | M7506 | |
| NaH2P04 | Merck | S0751 | |
| NanoDrop UV-Vis2000c spectrophotometer | Thermo Scientific | ND2000CLAPTOP | |
| NaOH | Merck | S5881 | |
| NHS | Merck | 130672 | |
| NZ amine (casein hydrolysate) | Merck | C0626 | |
| PBS | Merck | 806552 | |
| PD MidiTrap G-10 | Sigma-Aldrich | GE28-9180-11 | |
| Peptone | Merck | 70171 | |
| pET11a | Merck Millipore (Novagen) | 69436 | |
| PMSF | Merck | PMSF-RO | |
| QIAprep Spin Miniprep Kit (1000) | Qiagen | 27106X4 | |
| Reichert Software Package Autolink1-1-9 | Reichert | ||
| Reichert SPR SR7500DC Dual Channel System | Reichert | ||
| Scrubber2-2012-09-04 for data analysis | Reichert | ||
| SDS | Merck | 11667289001 | |
| Site-directed mutagenesis kit incl pUC18 control plasmid | Stratagene | #200518 | |
| Sodim chloride | Merck | S9888 | |
| Sodium acetate.Trihydrate | Merck | 236500 | |
| SPR sensor chip C19RBDHC30M | XanTec bioanalytics | SCR C19RBDHC30M | |
| SPR sensor chip CMD500D | XanTec bioanalytics | SCR CMD500D | |
| Sterilin Standard 90mm Petri Dishes | Thermo Scientific | 101R20 | |
| TBS | Merck | T5912 | 10x, solution |
| Triton-X100 | Merck | T8787 | |
| Tryptone | Merck | 93657 | |
| Tween20 | Merck | P1379 | |
| Vortex-Genie 2 Mixer | Merck | Z258423 | |
| X-gal | Merck | XGAL-RO | |
| XL1-Blue Supercompetent Cells | Stratagene | #200236 | |
| Yeast extract | Merck | Y1625 |
参考文献
- Perez-Caballero, D., et al. Tetherin inhibits HIV-1 release by directly tethering virions to cells. Cell. 139 (3), 499-511 (2009).
- Waheed, A. A., Gitzen, A., Swiderski, M., Freed, E. O. High-mannose but not complex-type glycosylation of tetherin is required for restriction of HIV-1 release. Viruses. 10 (1), 26 (2018).
- Wilson, I. A., et al. Structure of the haemagglutinin membrane glycoprotein of influenza virus at 3 A resolution. Nature. 289 (5796), 366-373 (1981).
- Otterstrom, J. J., et al. Relating influenza virus membrane fusion kinetics to stoichiometry of neutralizing antibodies at the single-particle level. Proceedings of the National Academy of Sciences of the United States of America. 111 (48), 5143-5148 (2014).
- Kaletsky, R. L., Francica, J. R., Agrawal-Gamse, C., Bates, P. Tetherin-mediated restriction of filovirus budding is antagonized by the Ebola glycoprotein. Proceedings of the National Academy of Sciences of the United States of America. 106 (8), 2886-2891 (2009).
- Tokarev, A., Skasko, M., Fitzpatrick, K., Guatelli, J. Antiviral activity of the interferon-induced cellular protein BST-2/tetherin. AIDS Research and Human Retroviruses. 25 (12), 1197-1210 (2009).
- Gnirss, K., et al. Tetherin sensitivity of influenza A viruses is strain specific: Role of hemagglutinin and neuraminidase. Journal of Virology. 89 (18), 9178-9188 (2015).
- Fleury, D., et al. A complex of influenza hemagglutinin with a neutralizing antibody that binds outside the virus receptor binding site. Nature Structural Biology. 6 (6), 530-534 (1999).
- Salunke, S. B., et al. Iron(III) chloride as an efficient catalyst for stereoselective synthesis of glycosyl azides and a cocatalyst with Cu(0) for the subsequent click chemistry. Chemical Communication. (Camb). 47 (37), 10440-10442 (2011).
- Schilling, P. E., et al. Mannosylated hemagglutinin peptides bind cyanovirin-N independent of disulfide-bonds in complementary binding sites. RSC Advances. 10 (19), 11079-11087 (2020).
- Fleury, D., Wharton, S. A., Skehel, J. J., Knossow, M., Bizebard, T. Antigen distortion allows influenza virus to escape neutralization. Nature Structural Biology. 5 (2), 119-123 (1998).
- Maier, I., Schiestl, R. H., Kontaxis, G. Cyanovirin-N binds viral envelope proteins at the low-affinity carbohydrate binding site without direct virus neutralization ability. Molecules. 26 (12), 3621 (2021).
- Ahmed, A. J., Keremane, S. R., Vielmetter, J., Bjorkman, P. J. Structural characterization of GASDALIE Fc bound to the activating Fc receptor FcγRIIIa. Journal of Structural Biology. 194 (1), 78-89 (2016).
- Boyd, R. Discovery of cyanovirin-N, a novel human immunodeficiency virus-inactivating protein that binds viral surface envelope glycoprotein gp120: potential applications to microbicide development. Antimicrobial Agents and Chemotherapy. 41 (7), 1521-1530 (1997).
- Bolmstedt, A. J., O'Keefe, B. R., Shenoy, S. R., McMahon, J. B., Boyd, M. R. Cyanovirin-N defines a new class of antiviral agent targeting N-linked, high-mannose glycans in an oligosaccharide-specific manner. Molecular Pharmacology. 59 (5), 949-954 (2001).
- O'Keefe, B. R., et al. Potent anti-influenza activity of cyanovirin-N and interactions with viral hemagglutinin. Antimicrobial Agents and Chemotherapy. 47 (8), 2518-2525 (2003).
- Shenoy, S. R., et al. Multisite and multivalent binding between cyanovirin-N and branched oligomannosides: calorimetric and NMR characterization. Chemical Biology. 9 (10), 1109-1118 (2002).
- Bewley, C. A., Kiyonaka, S., Hamachi, I. Site-specific discrimination by cyanovirin-N for alpha-linked trisaccharides comprising the three arms of Man(8) and Man(9). Journal of Molecular Biology. 322 (4), 881-889 (2002).
- Barrientos, L. G., Matei, E., Lasala, F., Delgado, R., Gronenborn, A. M. Dissecting carbohydrate-Cyanovirin-N binding by structure-guided mutagenesis: functional implications for viral entry inhibition. Protein Engineering Design & Selection. 19 (12), 525-535 (2006).
- Bewley, C. A., Otero-Quintero, S. The potent anti-HIV protein cyanovirin-N contains two novel carbohydrate binding sites that selectively bind to Man(8) D1D3 and Man(9) with nanomolar affinity: implications for binding to the HIV envelope protein gp120. Journal of the American Chemical Society. 123 (17), 3892-3902 (2001).
- Bewley, C. A. Solution structure of a cyanovirin-N:Man alpha 1-2Man alpha complex: structural basis for high-affinity carbohydrate-mediated binding to gp120. Structure. 9 (10), 931-940 (2001).
- Jensen, S. M. R., et al. Differential inhibitory effects of Cyanovirin-N, Griffithsin, and Scytovirin on entry mediated by envelopes of gammaretroviruses and deltaretroviruses. Journal of Virology. 88 (4), 2327-2332 (2014).
- Lan, J., et al. Structure of the SARS-CoV-2 spike receptor-binding domain bound to the ACE2 receptor. Nature. 581 (7807), 215-220 (2020).
- Lingwood, D., et al. Structural and genetic basis for development of broadly neutralizing influenza antibodies. Nature. 489 (7417), 566-570 (2012).
- Ekiert, D. C., et al. Antibody recognition of a highly conserved influenza virus epitope. Science. 324 (5924), 246-251 (2009).
- Pinto, D., et al. Cross-neutralization of SARS-CoV-2 by a human monoclonal SARS-CoV antibody. Nature. 583 (7815), 290-295 (2020).
- Keeffe, J. R., et al. Designed oligomers of cyanovirin-N show enhanced HIV neutralization. Proceedings of the National Academy of Sciences of the United States of America. 108 (34), 14079-14084 (2011).
- Yang, F., et al. Crystal structure of cyanovirin-N, a potent HIV-inactivating protein, shows unexpected domain swapping. Journal of Molecular Biology. 288 (3), 403-412 (1999).
- Stemmer, W. P., Crameri, A., Ha, K. D., Brennan, T. M., Heyneker, H. L. Single-step assembly of a gene and entire plasmid from large numbers of oligodeoxyribonucleotides. Gene. 164 (1), 49-53 (1995).
- Fischer, M. J. E., Mol, N., Fischer, M. Amine coupling through EDC/NHS: A practical approach. Surface Plasmon Resonance. Methods in Molecular Biology (Methods and Protocols). 627, (2010).
- Novoradovsky, A., et al. Computational principles of primer design for site directed mutagenesis. Technical Proceedings of 2005 NSTI Nanotechnology Conference and Trade Show. , 532-535 (2005).
- . QuikChange Site-Directed MutagenesisKit, User Manual Available from: https://users.drew.edu/jliu3/Docs/Stratagene%20Quikchange%20mutagenesis.pdf#:~:text=The%20QuikChange%20sitedirected%20mutagenesis%20kit%20is%20used%20to (2005)
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- . Expasy.org Available from: https://web.expasy.org/cgi-bin/protparam/protparam (2022)
- Karlsson, R. Real-time competitive kinetic analysis of interactions between low-molecular-weight ligands in solution and surface-immobilized receptors. Analytical Biochemistry. 221 (1), 142-151 (1994).
- Schuck, P., Zhao, H. The role of mass transport limitation and surface heterogeneity in the biophysical characterization of macromolecular binding processes by SPR biosensing. Methods Molecular Biology. 627, 15-54 (2020).
- Barnes, O., et al. SARS-CoV-2 neutralizing antibody structures inform therapeutic strategies. Nature. 588 (7839), 682-687 (2020).
- . Using reichert Surface Plasmon Resonance (SPR) for Antiviral Development, Application Note 28 Available from: https://www.reichertspr.com/clientuploads/directory/application_notes/Application_Note_28__Using_Reichert_Surface_Plasmon_Resonance_for_Antiviral_Testing.pdf (2022)
- Sundberg, E. J., Andersen, P. S., Gorshkova, I. I., Schuck, P., Schuck, P. Surface plasmon resonance biosensing in the study of ternary systems of interacting proteins. Protein Interactions: Biophysical Approaches for the Study of Complex Reversible Systems. 5, 97-141 (2007).
- . Method Development Notes Available from: https://www.reichertspr.com/applications/method-development-notes/ (2022)
- Angulo, J., Enríquez-Navas, P. M., Nieto, P. M. Ligand-receptor binding affinities from saturation transfer difference (STD)-NMR spectroscopy: the binding Isotherm of STD initial growth rates. Chemistry. 16 (26), 7803-7812 (2010).
- Goldflam, M., Tarragó, T., Gairí, M., Giralt, E. NMR studies of protein-ligand interactions. Methods in Molecular Biology. 831, 233-259 (2012).
- Kumar, S., Maurya, V. K., Prasad, A. K., Bhatt, M. L. B., Saxena, S. K. Structural, glycosylation and antigenic variation between 2019 novel coronavirus (2019-nCoV) and SARS coronavirus (SARS-CoV). Virusdisease. 31 (1), 13-21 (2020).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。