このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
表面プラズモン共鳴 による 抗ウイルス剤のエンジニアリング
要約
本プロトコルは、HA、S糖タンパク質、関連するハイブリッド型グリカン、および高マンノースオリゴ糖へのCV-N結合を調べるためのSPR結合アッセイのための新しいツールについて説明しています。SPRは、これらのグリカンに二量体または単量体CV-Nのいずれかを結合するためのKD を決定するために使用される。
要約
表面プラズモン共鳴(SPR)は、ドメインスワップされたシアノビリン-N(CV-N)二量体へのヘマグルチニン(HA)結合を測定し、マンノシル化ペプチドとCV-Nの高親和性結合部位との間の相互作用を監視するために使用されます。ウイルスエンベロープスパイクgp120、HA、およびエボラ糖タンパク質(GP)1,2は、二量体CVN2上の高親和性および低親和性結合部位の両方に結合することが報告されています。ジマンノシル化HAペプチドはまた、CVN2の改変分子への2つの低親和性結合部位で結合しており、CVN2はそれぞれのリガンドに対して高親和性部位を有し、糖鎖結合ポケット内の安定化ジスルフィド結合を置き換えるために変異しているため、多価結合が確認されます。HA結合は、275 nMの解離定数(KD)で疑似抗体CVN2の1つの高親和性結合部位に示され、オリゴマー化によってヒト免疫不全ウイルス1型(HIV-1)をさらに中和します。ドメインスワップCVN2のジスルフィド架橋の数は、シスチンをグルタミン酸とアルギニンの極性残基対に置き換えることで4から2に減少し、HAに対する結合親和性が低下します。最も強い相互作用の中で、エボラGP1,2は、膜貫通ドメインのないエンベロープグリカンを使用して、低ナノモル範囲の2つの高親和性結合部位を持つCVN2によって結合されます。本研究では、重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)スパイク(S)糖タンパク質への多重特異性モノマーCV-Nの結合は、他のウイルススパイクへのナノモルKDと比較して、および中μモル範囲のその受容体結合ドメインを介して、KD = 18.6 μMで測定されます。
概要
テザリン関連抗ウイルス活性は、インターフェロンαによって誘導され、タンパク質ベースのテザーを含み、感染した細胞表面上に完全に形成されたビリオンの保持をもたらす1。ウイルス放出の阻害におけるテザリングリコシル化の必要性は依然として不明であり、in vitro研究のための組換え発現グリカン上のグリコシル化パターンの重要性を示唆しています1,2、これは(インフルエンザウイルスの場合)表面発現インフルエンザ赤血球凝集素HA 3,4の立体配座に依存します。.N結合型グリコシル化につながれたオリゴ糖の修飾は、HIVタイプ1型放出のテザリン媒介制限に十分であり、二量体化はウイルス放出の防止に不可欠な役割を果たし、それによって出芽ビリオンをつなぐための膜貫通ドメインまたはグリコシル-ホスファチジル-イノシトール(GPI)アンカーが関与することが注目されています5。.ヒトおよびマウステザリンが複数のエンベロープウイルス、レトロウイルス、およびフィロウイルスをブロックするための独自の機能が説明されています。BST-2/テザリンは、自然免疫1,6のインターフェロン誘導性抗ウイルスタンパク質であり、広域スペクトルの抗ウイルス活性で作用し、エンベロープ糖タンパク質5によって拮抗され、テザリンを転座させるか、テザリン6の構造を破壊します。例えば、A型インフルエンザウイルス上で表面発現されたエンベロープ糖タンパク質HAおよびノイラミニダーゼは、株特異的な様式7におけるテザリン拮抗作用についてよく知られており、宿主受容体結合部位の認識を容易にする8。糖鎖標的抗体は、HA上の急速にカスタマイズされた糖鎖シールドとの相互作用の化学量論で研究され、インフルエンザAのH3N2およびH1N1サブタイプ4への結合親和性をもたらします。
抗ウイルス剤とウイルスエンベロープスパイク(糖鎖リガンド)の結合機構を解明するために、モノマンノース、ジマンノース、トリマンノース部分を化学合成します。マンノシル化ペプチドは、グリコシル{β}-過酢酸塩から1,2-トランスグリコシルアジド変換9へのアジドグリコシル化によって生成され、生命を脅かすウイルスの表面に通常見られるN-アセチルグルコサミンおよび高マンノースオリゴ糖を模倣します。トリアゾールバイオアイソスターは、HAペプチド10のマンノシル化残基を形成する結合を模倣し、HAヘッドドメイン上の2番目のN結合型グリコシル化スポット(4つのN結合型糖鎖N54、N97、N181、N301を有するHAトップ)8,11,12の周りの抗ウイルスCV-N誘導体との部位特異的相互作用を促進するために利用されます。.グルタミン酸(Glu)とアルギニン(Arg)の間の相互作用と結果として生じるらせん双極子は、モデルペプチドとタンパク質の両方の良好な安定性を示しましたが、SPRを使用して視覚化されます。糖鎖部分への受容体結合を直接阻害することによってHA10上の単一の化学合成グリコシル化部位を認識することと比較した場合、その受容体に対する4部位変異Fc構造のより高い親和性がインビボでエフェクター機能を惹起することが示され、Fc変異体に結合したN結合型糖鎖の無関係な組成が機構的に決定されることが明らかになった13。
CV-Nは、HIV 14,15、インフルエンザ16、およびエボラウイルスに対する抗ウイルス活性を示し、エンベロープスパイクタンパク質12,17,18,19上の高マンノースオリゴ糖修飾へのナノモル結合によって媒介されます。CV-Nにおける1つの高親和性糖鎖結合部位(H)または共有結合した二量体CVN2における2つのHsへのインフルエンザHA結合は、それぞれ平衡解離定数(KD)= 5.7 nM(図1A)およびKD = 2.7 nMを有すると決定される。CV-NおよびCVN2はいずれも、別の1つまたは2つの低親和性炭水化物結合部位(L)12,17,20,21を有する。エボラGP1,2は、より低いナノモル範囲(KD = 26nM)の親和性を有するCVN2の2Hに結合する。エボラGP1,2およびHAに結合するCV-N WTは、KD = 34 nMからKD = 5.7 nMまでの親和性を示す(A/New York/55/04)12。ウイルスエンベロープ上の高マンノースグリカンを特異的に標的とするCV-Nなどのレクチンは、C型肝炎ウイルス、SARS-CoV、ヘルペスウイルス、マールブルグウイルス、および麻疹ウイルス22の複製をさらに阻害します。
低分子CV-N分子は、広範囲のウイルスに結合してウイルスの侵入を阻害するように機能するため、20年以上にわたって徹底的に研究されてきました16,18。構造解析および結合親和性アッセイは、ウイルスエンベロープ糖タンパク質に対する結合活性を高めるために、マイクロモル範囲の二価結合によるドメインスワップCVN2ダイマー内の2つのLの架橋を示しています10,19。Man(8)D1D3アームおよびMan(9)に対するManα1-2Manαの選択的結合は、反対のタンパク質プロトマー20上に位置する異なる親和性の2つの結合部位を含み、それによってナノモル結合親和性に達する(図1B)。したがって、CVN2は、ウイルス中和抗体と同様に、HIV gp120上のエピトープに結合するその応用に関する疑似抗体と見なされます17。ここで、著者は、CVN2の受容体結合ドメイン(RBD)を介したSARS-CoV-2スパイクへの潜在的な結合を調査することに関心があります。固定化されたヒトアンジオテンシン変換酵素(ACE)-2とSARS-CoV-2 RBDとの結合曲線は、この生物学的に関連する結合相互作用についてKD = 4.7 nMをもたらします23。
対照的に、選択された免疫グロブリンクラスは、特異的で一貫した構造タンパク質パターンを認識し、膜固定HA領域24における親和性成熟のための基質を付与する。CV-Nは、ほぼすべてのインフルエンザAおよびBウイルス16において非常に強力な活性を示し、広範に中和する抗ウイルス剤である。私たちの知識は、高中和抗体による糖鎖標的化のためのエピトープ構造を含む可能性のあるHA1およびHA2のステム上の標的エピトープの位置について、およびレクチン結合と比較して不完全です25。
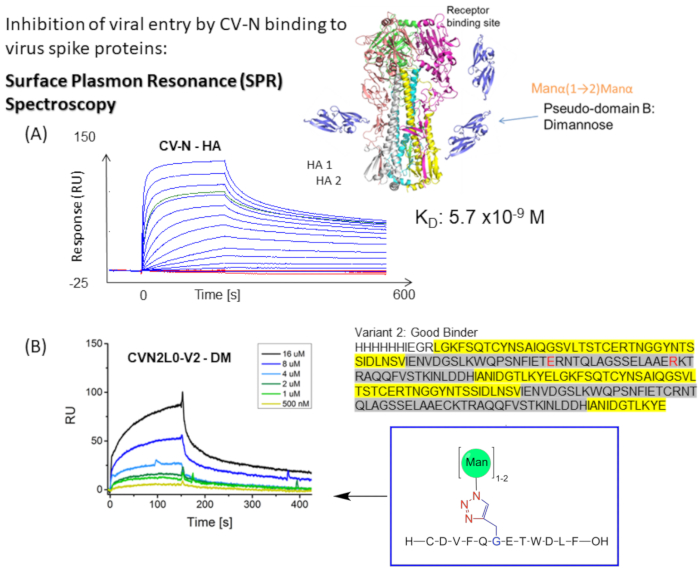
図1:CV-NからウイルスエンベロープスパイクへのSPR結合アッセイの概略図。 (A)リガンドへのCV-N結合のためのSPRアッセイ:HA全長タンパク質(90kDa)。インフルエンザHA A /ニューヨーク/55/04(H3N2)へのリアルタイムの二重参照結合を示す運動データセット(5120、2560、1280、640、320、160、80、40、20、10、5、2.5、0 nM)。(B)CVN2L0変異体V2は、500nM〜16μMの濃度範囲内で固定化リガンドDMに結合する。 配列:L残基は黄色で強調表示されています。H残基は灰色で強調表示されます。E58およびR73は野生型タンパク質のシステインの置換であり、V2を4つのジスルフィド結合ではなく3つのジスルフィド結合を有する安定なタンパク質フォールドにする。 この図の拡大版を表示するには、ここをクリックしてください。
膜遠位HA上部上の糖鎖シールドはCV-N 12への高親和性結合を誘導するのに対し、CVN2はHA上部のジスルフィド架橋に隣接するHAへの結合が、その低親和性部位でさらに観察されている10,12。様々な極性相互作用および相互作用部位がCV-Nによる糖鎖結合において同定される。これらの相互作用は、結合部位にノックアウト変異体を生成して、in silico予測グリコシル化12に対する結合親和性を相関させることによって検証される。したがって、このプロジェクトは、結合親和性と特異性において以前にテストされた化学的にマンノシル化されたHAペプチドを、少数の異なるN結合型グリコシル化部位とO結合型グリコシル化によって自然に修飾されたSARS関連の2019-nCoVスパイクおよびSARS-CoV-2からの短いペプチド配列と比較することを目的としています。今回、Pintoたちの研究グループは、クライオ電子顕微鏡法と結合アッセイを用いて、受容体付着と競合することなく、サルベコウイルス亜属内の保存された糖鎖を含むSARS-CoV-2スパイクタンパク質のエピトープを認識する可能性のあるモノクローナル抗体S309を報告している26。この研究のプロトコルは、CV-NおよびCVN2がSPR技術を使用してグリコシル化タンパク質および合成マンノシル化ペプチドにどのように結合するかを研究するために、CV-N変異体の設計、発現、および特性評価がどのように重要であるかを説明しています10,12。
タンデム結合二量体CVN2L027および結合部位変異体(V2-V5)は組換え的に発現され、変異体はジスルフィド結合置換体(C58EおよびC73R)を有する(図2A)。また、一点変異E41Aを有する変異体が調製されるのは、この位置が分子間交差接触残基として見られたからである。この変異体は、レクチンと高マンノースオリゴ糖の間のSPR結合測定のための別の興味深い分子であり、結合ドメインを解読し、二量体型との比較を可能にします。CVN2のドメインスワップ結晶構造は、49〜54残基に及ぶ柔軟なリンカーを示しています。2つのドメインは、剛体としてヒンジの周りを移動し続けることができ、分子内ドメイン相互作用を介してモノマー(ドメインA-残基1-39;90-101-とドメインB-残基40-89)または分子間ドメインスワッピングによって二量体[ドメインA(第1のモノマー)とドメインB(第2のコピー)、およびドメインB(第1のモノマー)とドメインA(第2のコピー)]のいずれかを発達させる。2つのプロトマーのAドメインとBドメインの間には、Glu4128を除いて密接な相互作用はありません。CV-Nの遺伝子は、40量体合成オリゴ29を用いた反復PCR法を用いて開発することができ、次いで、Keeffe, J.R.27によって記載されているように、電気コンピテント細胞への形質転換(エレクトロポレーション)のためにpET11aのNdeIおよびBamHI部位にサブクローニングされる。それぞれの結晶構造(PDB ID 3S3Y)を達成するために使用されるタンパク質は、N末端6-ヒスチジン精製タグとそれに続く第Xa因子プロテアーゼ切断部位を含む。部位特異的突然変異誘発は、アミノ酸交換のために、点突然変異を起こし、コドンを切り替え、単一または複数の塩基またはコドンを挿入または欠失するために利用される。これらの形質転換は、タンパク質の機能と構造に関する貴重な洞察を提供します。組換え発現および精製されたCV-N、CVN2、およびCVN3は、生物物理学的によく研究されており20、21、27、製造が安価であるため、SPRセンサーチップに固定化されたグリカンへの結合アッセイの特性評価に使用されます。従来の酵素結合免疫吸着アッセイ(ELISA)は、糖鎖リガンドの固定化技術に関する再現性が低く、SPRで示されているさまざまな結合部位変異体のリアルタイム結合をエンドポイントアッセイに変換します。
結合親和性バリアントCVN2L0-V2(ジスルフィド架橋置換10を有するホモ二量体CV-Nのインタクトフォールド)を大腸菌(大腸菌)のHisタグで発現させ、アフィニティークロマトグラフィーを適用してNi-NTAカラムで精製し、SPRを用いてHA(H3N2)、モノマンノシル化HAペプチドおよびジマンノシル化HAペプチドへの結合について試験した。 化学的にマンノシル化ペプチド、またはHAおよびSタンパク質、すべてが親水性チップ表面に結合したリガンドおよびアミン反応性エステルまたはビオチン-ストレプトアビジンタンパク質工学を介して。逐次実行の同じ手順をこれらのリガンドに適用し、CV-Nの様々な希釈液およびCV-N(およびCVN2)の変異体を注入して、以下に説明するように分子間相互作用分析のための速度論的情報を得る30。RBD固定化SPRセンサーチップは、CV-NからSペプチドへの結合研究に使用され、ヒトACE2とのSARS-CoV-2結合との親和性と比較されます。
プロトコル
本研究では、CV-Nの代わりにCVN低分子ユビキチン様修飾因子(SUMO)融合タンパク質が酵素結合免疫吸着アッセイに使用され、細胞ベースのアッセイに適しています。組換え全長インフルエンザAウイルスHA H3タンパク質は、商業的に取得されるか( 材料の表を参照されたい)、または標準プロトコール12に従って哺乳動物HEK293細胞株およびバキュロウイルス感染昆虫細胞で発現される。武漢-1スパイクタンパク質は、哺乳類のHEK293細胞で発現しています。モノマンノシル化ペプチド(MM)およびジマンノシル化ペプチド(DM)の合成により、CVN2およびモノマンノシル化低分子10に対する均一なリガンドの検出が可能になります。
1. CV-Nコンストラクトの作成
- CVN2変異体およびCVN2L0タンパク質(PDB ID 3S3Y)のそれぞれについて、市販のソースからpET27b(+)ベクター中のN末端pelBリーダー配列およびHisタグを有する遺伝子構築物を入手する( 材料表、補足ファイル1を参照)。
- CVN2L0 とそのバリアント(V2、V3、V4、および V5;図2A,C)は、CV-Nリピートごとに2つの異なるDNA配列からなるCVN2L0鋳型遺伝子の背景にある。
- 凍結乾燥プラスミドDNAを滅菌脱イオン蒸留水(ddH2O)に最終濃度100 ng/μLまで溶解します。
2. プラスミドDNA形質転換細胞を用いたLB-寒天プレートの作製
- 10 g/Lのペプトン、5 g/Lの酵母エキス、および5 g/LのNaClをddH2Oに溶解して培養液LB-レノックスを調製し(材料の表を参照)、pHを7.4に調整します。以前に発表された第10報に続いて化学的方法によって各変異体(V2-V5)についてコンピテント大腸菌BL21(DE3)への形質転換を行う。
- 溶液(900 μLと100 μL)を分割し、100 μLをLB-寒天プレート(50 μg/mLカナマイシン)に移し、滅菌セルスプレッダーを静かに使用します。寒天プレートを37°Cで一晩インキュベートします。
3.クローニング
- CV-Nの遺伝子をpET11aのNdeIおよびBamHI部位にサブクローニングし( 材料の表を参照)、以下の参照27のエレクトロコンピテントセルに形質転換(エレクトロポレーション)します。
4.部位特異的突然変異誘発
- CV-Nリピート27ごとに2つの識別されたDNA配列を含むCVN2L0鋳型遺伝子のバックグラウンドでCVN2L029および変異型CVN-E41Aを生成する。
- PCR31を実行するための部位特異的突然変異誘発キット(材料表を参照)および特異的変異原性プライマー5'-gagaaccgtcaacgtttggataacagagttcagg-3'および5'-cctgaactctgttatcgcaaacacgtggttctc-3'を使用して変異を行います。
- 5〜50 ngの範囲の複数濃度の二本鎖DNA(dsDNA)テンプレートを使用して一連のサンプル反応を開始します(例:5、10、20、および50 ngのdsDNAテンプレート)。プライマー濃度を一定に保ちます。
注:PCRミックスおよび熱サイクルプロトコルは、部位特異的突然変異誘発キット32の取扱説明書に記載されているように一般的に使用されます。
- 5〜50 ngの範囲の複数濃度の二本鎖DNA(dsDNA)テンプレートを使用して一連のサンプル反応を開始します(例:5、10、20、および50 ngのdsDNAテンプレート)。プライマー濃度を一定に保ちます。
- Dpn I制限酵素(1 μL、10 U/μL、 材料表を参照)を鉱物油オーバーレイの下に追加します。反応を完全かつ穏やかに混合し、卓上マイクロ遠心分離機で1分間スピンダウンし、すぐに37°Cで1時間インキュベートして、親スーパーコイルdsDNAを消化します。
5.細菌細胞の形質転換
- XL1-Blueスーパーコンピテントセル( 材料の表を参照)を氷上で穏やかに解凍します。各コントロールおよびサンプル反応を形質転換するには、スーパーコンピテントセル(50 μL)をプレチルドポリプロピレン丸底チューブ(14 mL)に分注します。
- 各コントロールおよびサンプル反応(変異ssDNA)から1 μLのDpn I処理一本鎖DNA(ssDNA)をスーパーコンピテントセルの分離アリコートに移し、相補鎖を合成します。形質転換反応を注意深く回転させ、氷上で反応を混合して30分間インキュベートします。
注:Dpn I処理されたDNAを形質転換反応に移す前に、ピペットチップから残っている鉱油を注意深く除去することをお勧めします。オプションのコントロールとして、0.1 ng/μLのpUC18コントロールプラスミド(1 μL)と50 μLのスーパーコンピテントセルを混合して、XL1-Blueスーパーコンピテントセルの形質転換効率を確認する必要があります。
- 各コントロールおよびサンプル反応(変異ssDNA)から1 μLのDpn I処理一本鎖DNA(ssDNA)をスーパーコンピテントセルの分離アリコートに移し、相補鎖を合成します。形質転換反応を注意深く回転させ、氷上で反応を混合して30分間インキュベートします。
- 変態反応に熱パルスを印加し、42°Cで45秒間反応させた後、氷上に2分間置きます。
注意: 適用される熱パルスは、ポリプロピレン丸底チューブ(14 mL)の上記の条件に対してすでに最適化されています。 - 0.5 mLのNZY+ブロス(1リットルあたり:10 gのNZアミン(カゼイン加水分解物)、5 gの酵母エキス、5 gのNaCl、12.5 mLの1 M MgCl 2、12.5 mLの1 M MgSO4、10 mLの2 Mグルコース、pH 7.5、および42°Cに予熱)を加え、225-250 rpmで1時間振とうしながら、37°Cで形質転換反応をインキュベートします。各形質転換反応の正しい容量(コントロールプラスミド形質転換から5 μL、サンプル形質転換から250 μL)をLB-アンピシリン寒天プレートにプレートします。
注:形質転換制御と突然変異誘発のために、5-ブロモ-4-クロロ-3-インドリル-β-D-ガラクトピラノシド(X-gal、80 μg / mL)およびイソプロピル-1-チオ-β-D-ガラクトピラノシド(IPTG、20 mM)を含むLB-アンピシリン寒天プレート上に細胞を広げます( 材料の表を参照)。50 mLの細胞培養物に形質転換 Eの単一コロニーを接種する。分析のために変異プラスミドDNAを精製するための 大腸菌 細胞。突然変異誘発は、外部施設でのDNAシーケンシングによって確認されます。
6. 発現とタンパク質精製
- 大規模な培養の場合は、形質転換プレートから少量のLB(アンピシリンを含む)に単一のコロニーを接種します。
- 一晩培養して、10 mMのMgCl2、10 mMのMgSO4、 20 mMのグルコースなどの添加剤を発現培養液に接種し、種培養液を1/100に希釈します。
- 37°Cで激しく振とうしながら細胞を増殖させる。 細胞を20°Cに冷却する前に、細胞を0.4〜0.6(中対数期)の間でAbs 600 nmに成長させます。 1 mM IPTGで誘導し、一晩で増殖させます。
- その後、4,000 x g で4°Cで15分間遠心分離して細胞を回収し、上清をピペットで廃棄した。
- 細胞ペレットをリン酸緩衝生理食塩水(PBS)バッファーに再懸濁し、4,000 x g で4°Cで15分間再遠心分離します。 その後、上清をピペットで捨てる。残りのペレットを10 mLの溶解バッファーに再懸濁し、懸濁液を37°Cで1時間インキュベートします。
注:溶解バッファーの組成:(50 mMのNaH 2 PO4、300 mMのNaCl、2%Triton-X100、500 ng / mLのリゾチーム、1 mMのフェニルメチルスルホニルフルオリド(PMSF)、1 mMのジチオスレイトール、1 mMのMgCl2、pH 8、材料の表を参照)。- 混合物を2回の凍結融解サイクル(-80°C)にかけます。4,000 x g で4°Cで15分間遠心分離することにより可溶性画分と不溶性画分を分離し、ポリアクリルアミドゲル電気泳動(PAGE)、特にドデシル硫酸ナトリウム(SDS)-PAGE33を使用して分析します(図2B、C)。
注:細胞は、凍結融解、超音波処理、均質化、酵素溶解、またはこれらの方法の組み合わせなど、いくつかの方法で溶解することができます。封入体からの精製は、高いタンパク質収率を収集するために推奨されます26。
- 混合物を2回の凍結融解サイクル(-80°C)にかけます。4,000 x g で4°Cで15分間遠心分離することにより可溶性画分と不溶性画分を分離し、ポリアクリルアミドゲル電気泳動(PAGE)、特にドデシル硫酸ナトリウム(SDS)-PAGE33を使用して分析します(図2B、C)。
- Ni-NTAカラムクロマトグラフィーを使用してタンパク質を精製します( 材料表を参照)。可溶性画分を再生Ni-NTAビーズカラム(1 mL/分)にロードします。グラジエント(TBS中の0〜100%500 mMイミダゾールを60分間)を開始し、画分を収集する前に、TBSバッファー(50 mMのトリス、150 mMの塩化ナトリウム、pH 7.5)でシステムを洗浄します。精製タンパク質を透析して、100 mM PBSに対する生化学的特性評価を行います(図2C、D)。
- あるいは、His-Select Ni 2+アフィニティーゲル(材料表を参照)を14 mLチューブで使用して、Hisタグ付き組換え発現CV-Nをそれぞれ20 mMイミダゾールおよび250 mMイミダゾールを含む緩衝溶液に結合および再懸濁します。バッチで少なくとも30分間インキュベートします。
注:これらの半精製タンパク質をシングルユースプレパックカラムに適用して、固定化金属アフィニティークロマトグラフィーから1〜1.5 mLの溶出液をロードできる炭水化物やタンパク質などの生物学的サンプルのバッファー交換とクリーンアップを行います。 - タンパク質溶液を10 kDaカットオフフィルター( 材料表を参照)を備えた遠心分離チューブに移し、4,500 x g および4°Cで10分間遠心分離して濃縮します。 SPR測定の場合は、分析対象物を10 mM HEPES、150 mM塩化ナトリウム、3 mMエチレンジアミン四酢酸(EDTA)、および0.05%Tween20、pH 7.4(HBS-EP(+)、 材料表を参照)に交換します。
- このSPRランニングバッファーを1:10の希釈係数に加え、4,500 x g および4°Cで10分間、初期容量まで4回遠心分離します。
- 23,474 Da34のサイズを示すメインタンパク質CVN2L0の計算された吸光係数(20,440 M-1 cm-1)に基づいて、NanoDrop UV-Vis分光光度計(材料の表を参照)を使用して280 nmでのタンパク質濃度を決定します。PBS(100 mM、pH 7.0)またはSPRバッファーをブランクとして使用し、3つの希釈ステップ(1:1、1:10、および1:100)でタンパク質濃度を測定します。
図2:CV-N配列および発現 。 (a)各CV-Nリピート(各101アミノ酸)と4つのジスルフィド架橋との間にリンカーを有さないCVN2 をEでpET11aベクターで発現させる。 大腸菌。(b)CV-N(モノマー)およびCVN2(二量体)についての2つの独立したコロニーの発現。(C)ジスルフィド結合変異体を精製し、SDS-PAGE上で分析する。低分子量マーカー(6μL)を基準として使用します。WT = CVN2L0は、(A)でマークされているように4つのジスルフィド架橋を持っています。V2は、58位および73位の極性残基によるジスルフィド結合置換を有する変異体である。V3-V5は、残りの2つのS-S結合と極性(C58E-C73R)または非極性(C58W-C73M)のいずれかのアミノ酸置換、またはこれらの残基対置換の組み合わせを有する変異体である。(D)精製されたCVN2L0のHPLCクロマトグラムを、バッファーA中の5%〜65%バッファーBから30分間にわたって直線勾配で1 mL/minの流速で溶出します。 バッファーAはddH2O中の0.1%(v / v)トリフルオロ酢酸、バッファーBはアセトニトリル中の0.08%(v / v)トリフルオロ酢酸です。タンパク質は、高性能シリカゲル300-5-C4(150 x 4.6 mm)カラムで214 nmおよび280 nmで分析されます。 この図の拡大版を表示するには、ここをクリックしてください。
7. SPR分光法
- 再生バッファーとしてランニングバッファーHBS-EP(+)および10 mMグリシンHCl pH 1.5-1.6を備えたデュアルチャネルSPRシステム( 材料表を参照)を使用します。機器、デガッサー、オートサンプラーの電源を入れ、ポンプでシステム全体をddH20で1時間洗浄します。すぐに使用できるランニングバッファーを別のボトルに入れます。
- エマージョンオイルを検出器に滴下し、薄い金膜でコーティングされ、カルボキシメチルデキストランヒドロゲルで官能化されたガラスセンサーチップ( 材料の表を参照)を3ポートフローセルの下の検出器に直接取り付けます。ハンドリングをプルダウンして設定を修正します。
注:C19RBDHC30M 200 nmストレプトアビジン誘導体化カルボキシメチルデキストランヒドロゲルは、中密度のビオチン化重症急性呼吸器症候群コロナウイルス-2 RBDタンパク質を含み、事前に固定化されたリガンドを備えたすぐに使用できるセンサーチップです。
8. CV-NがHA、Sタンパク質、RBDに結合するためのSPR結合アッセイ
- 以下の手順に従って、タンパク質性リガンドをセンサーチップに固定化します。
- メニューバーの フォーム をクリックし、統合されたSPRAutoLinkソフトウェアで テーブルエディタを実行して 、実行テーブルを開きます( 材料表を参照)。使用可能な実行テーブルのリストから BASIC_Immobilization を選択してクリックし、コンピューター画面で実験手順の手順に従います。使用するそれぞれのサンプルエディターは、右上隅に表示されます。
- フォームセクションのサンプル セットエディター をクリックして、オートサンプラーに配置された2つのラックの試薬リストに記入し、さらに分析します。メニューバーの「ツール」として オートサンプラーダイレクトコントロール をクリックして、ラックを前方または背面に戻します。動作温度として4°Cを選択します。
注:SPRソフトウェアでは、対応するツールを選択し、オートサンプラーを処理し、フォームをクリックすることにより、「SPR機器ダイレクトコントロール」と「ポンプダイレクトコントロール」が可能です。また、テーブルエディタの実行、データプロット、または後処理を選択して、データ分析を実行することもできます。ファイルはデフォルトのディレクトリに直接保存され、後処理ウィンドウからscrubber.filesとしてエクスポートされます。
- ツールとポンプダイレクトコントロールをクリックしてddH20を注入するためにポンプを開始し、SPR機器ダイレクトコントロールをクリックしてデータを記録し、毎回新しく表示されるウィンドウで開始します。カップリング試薬(ステップ8.3)を300 μLバイアルに入れ、オートサンプラーラックに入れ、ランをクリックしてランテーブルを開始します。
注:チップ表面は、10 mMグリシンバッファーpH 9.0で調整されているか、1 M塩化ナトリウム、0.1 Mホウ酸ナトリウムバッファーpH 9.0でフラッシュして、EDC/NHS活性化ミックス30用にカルボキシル誘導体化チップ表面を調整している可能性があります。 - この単純なタンパク質間相互作用のために、CMD500Dチップ( 材料の表を参照)を使用して、固定化されたHAを有するマイクロ屈折率単位(μRIU)= 2500 - 3000フローセルおよびスパイクタンパク質を有するμRIU = 400フローセルを生成する。15 μL/分の注入流速で、次の連続したステップを適用して、0.4 M N-エチル-N'-(ジメチルアミノプロピル)カルボジイミド塩酸塩(EDC * HCl)と0.1 M N-ヒドロキシスクシンイミド(NHS)の水性で等量の混合物を注入します。
- 補充ポンプの補充を25,000 μL/minで行い、ベースライン調整を30秒間行い、90 μLのサンプル活性化溶液(EDC/NHS)を6分間の接触時間で注入し、さらに5分間保持します。
- ベースラインを左のフローセル(青)のみで10 μL/minで1分間実行した後、このサイクルを繰り返して、化学合成ペプチド10、HA、およびスパイクタンパク質を20 μg/mLで注入および固定化し、その後のベースライン調整を可能にします ddH2O 1.5分間 活性化チップ表面を1 M エタノールアミンHCl pH 8.5でクエンチします。
- 液体サンプラーからddH20のデガッサーにチューブをHBS-EP(+)でボトルに切り替えます(補足図1)。
- SPR センサーグラムを分析します。
- 速度論的研究を行うには、さまざまな分析物濃度(10-5-10-8 M)を使用し、各注入後の再生ステップと異なる分析種の後のブランク測定を行います。流量を10 μL/minに変更し、4分間の接触時間で注入を開始し、次に5分間のベースライン生成、および30秒間隔でそれぞれ2分間の2つの再生ステップを開始します。
- サンプルランからセンサーグラムを差し引いたブランク測定用のバッファー溶液を注入して、さまざまなタンパク質濃度を正規化します。
- フォームをクリックし、下にスクロールして、この操作モードをクリックして「後処理」に変更します。追加をクリックして、各フローセルのデータプロットフォームで時間の経過とともに生成された結合曲線を選択し、オーバーレイをスクラバーファイル(.ovr)としてエクスポートします。[ファイル]をクリックして、ファイル保存オプションを開きます。左右の曲線を揃え、リガンドチャネルの信号から2番目の基準チャネルの信号を差し引くことにより、応答曲線を取得します。
注:データは、センサーグラムを計算的に定義することにより、「後処理」で操作されます。これは、左右のフローセルからのセンサーグラムのオーバーレイに入れられるか、両方のチャネルの差からセンサーグラムとして表されます。 - 結合測定の前後に、50〜100 mMのグリシンバッファーpH 9.5、水、および20%エタノールで流路系全体を洗浄し、0.5%SDSおよびグリシンで微量の塩またはタンパク質汚染、またはより厳しいものを除去します。
注意: 機器の損傷を防ぐために、チップがバッファー下または湿度100%で保管されている場合は、チップカートリッジを機器に再挿入する前に、ガラスチップの機械的安定性を確認することをお勧めします。
結果
二量体ドメインスワップCVN2L0分子は、3つの別々のSPR実験でHAトップ領域への結合についてテストされ、結合親和性はKD値で提示されます。ドメインBは、ジスルフィド結合をイオン性残基に置換することによって影響を受けるH結合部位を含むと仮定され、ドメインAはL10,18を形成する。CVN2L0およびバリアントV2(3つのジスルフィドブリッジ)お?...
ディスカッション
CV-Nの結合親和性は、機能的結合部位の数と相関しています[ドメインBでは2H、ドメインスワップダイマーとして操作された場合はドメインAでは2L]。結合親和性が変化する変異体(CVN2L0-V2、ジスルフィド架橋ノックアウトを含むCV-Nのホモ二量体安定フォールド)を大腸菌で発現させ、精製し、SPR10を用いてHAタンパク質(H3N2)への結合について積極的に試験し、HAをHまたはL?...
開示事項
著者は開示するものは何もありません。
謝辞
著者は、ウィーン工科大学のバイオテクノロジーおよび微生物学部門およびウィーン医科大学の腎臓学および透析部門の医学部IIIのクリスチャン・ダーントル博士、特に技術的および科学的支援のためのマルクス・ワールマン博士を認めています。哺乳類細胞におけるタンパク質発現は、ウィーン天然資源生命科学大学(BOKU)のバイオテクノロジー学部によってサポートされました。著者は、ドイツのデュッセルドルフにあるXanTecバイオアナリティクスのNico Dankbar博士に、SPR結合アッセイの実施に関する有益な科学的議論について深い感謝の意を表したいと考えています。
資料
| Name | Company | Catalog Number | Comments |
| Äkta primeplus | Cytiva | ||
| Amicon tubes | Merck | C7715 | |
| Ampillicin | Sigma-Aldrich | A5354 | |
| Beckmann Coulter Cooler Allegra X-30R centrifuge | Beckman Coulter | B06320 | |
| Cell spreader | Sigma-Aldrich | HS86655 | silver stainless steel, bar L 33 mm |
| Custom DNA Oligos | Sigma-Aldrich | OLIGO | |
| Custom Gensynthesis | GenScript | #1390661 | cloning vector: pET27b(+) |
| Cytiva HBS-EP+ Buffer 10, 4x50mL | Thermo Scientific | 50-105-5354 | |
| Dionex UlitMate 3000 | Thermo Scientific | IQLAAAGABHFAPBMBFB | |
| Dpn I restriction enzyme (10 U/μL) | Fisher Scientific | ER1701 | |
| DTT | Merck | DTT-RO | |
| EDC | Merck | 39391 | |
| EDTA | Merck | E9884 | |
| Eppendorf Safe-Lock Tubes | Eppendorf | 30120086 | |
| Eppendorf Safe-Lock Tubes | Eppendorf | 30120094 | |
| Eppendorf Minispin and MiniSpin Plus personal microcentrifuge | Sigma-Aldrich | Z606235 | |
| Ethanol | Merck | 51976 | |
| Ethanolamine HCl | Merck | E6133 | |
| Falcon 50mL Conical Centrifuge Tubes | Fisher Scientific | 14-432-22 | |
| Falcon 14 mL Round Bottom Polystyrene Test Tube, with Snap Cap, Sterile, 25/Pack | Corning | 352057 | |
| Glucose | Merck | G8270 | |
| Glycine HCl | Merck | 55097 | |
| HA H3 protein | Abcam | ab69751 | |
| HEPES | Merck | H3375 | |
| His-select Ni2+ | Merck | H0537 | |
| Imidazole | Merck | I2399 | |
| IPTG | Merck | I6758 | |
| Kanamycin A | Sigma-Aldrich | K1377 | |
| Kromasil 300-5-C4 | Nouryon | ||
| LB agar | Merck | 52062 | |
| LB agar | Merck | 19344 | |
| LB Lennox | Merck | L3022 | |
| Lysozyme | Merck | 10837059001 | |
| Magnesium chloride | Merck | M8266 | |
| Magnesium sulfate | Merck | M7506 | |
| NaH2P04 | Merck | S0751 | |
| NanoDrop UV-Vis2000c spectrophotometer | Thermo Scientific | ND2000CLAPTOP | |
| NaOH | Merck | S5881 | |
| NHS | Merck | 130672 | |
| NZ amine (casein hydrolysate) | Merck | C0626 | |
| PBS | Merck | 806552 | |
| PD MidiTrap G-10 | Sigma-Aldrich | GE28-9180-11 | |
| Peptone | Merck | 70171 | |
| pET11a | Merck Millipore (Novagen) | 69436 | |
| PMSF | Merck | PMSF-RO | |
| QIAprep Spin Miniprep Kit (1000) | Qiagen | 27106X4 | |
| Reichert Software Package Autolink1-1-9 | Reichert | ||
| Reichert SPR SR7500DC Dual Channel System | Reichert | ||
| Scrubber2-2012-09-04 for data analysis | Reichert | ||
| SDS | Merck | 11667289001 | |
| Site-directed mutagenesis kit incl pUC18 control plasmid | Stratagene | #200518 | |
| Sodim chloride | Merck | S9888 | |
| Sodium acetate.Trihydrate | Merck | 236500 | |
| SPR sensor chip C19RBDHC30M | XanTec bioanalytics | SCR C19RBDHC30M | |
| SPR sensor chip CMD500D | XanTec bioanalytics | SCR CMD500D | |
| Sterilin Standard 90mm Petri Dishes | Thermo Scientific | 101R20 | |
| TBS | Merck | T5912 | 10x, solution |
| Triton-X100 | Merck | T8787 | |
| Tryptone | Merck | 93657 | |
| Tween20 | Merck | P1379 | |
| Vortex-Genie 2 Mixer | Merck | Z258423 | |
| X-gal | Merck | XGAL-RO | |
| XL1-Blue Supercompetent Cells | Stratagene | #200236 | |
| Yeast extract | Merck | Y1625 |
参考文献
- Perez-Caballero, D., et al. Tetherin inhibits HIV-1 release by directly tethering virions to cells. Cell. 139 (3), 499-511 (2009).
- Waheed, A. A., Gitzen, A., Swiderski, M., Freed, E. O. High-mannose but not complex-type glycosylation of tetherin is required for restriction of HIV-1 release. Viruses. 10 (1), 26 (2018).
- Wilson, I. A., et al. Structure of the haemagglutinin membrane glycoprotein of influenza virus at 3 A resolution. Nature. 289 (5796), 366-373 (1981).
- Otterstrom, J. J., et al. Relating influenza virus membrane fusion kinetics to stoichiometry of neutralizing antibodies at the single-particle level. Proceedings of the National Academy of Sciences of the United States of America. 111 (48), 5143-5148 (2014).
- Kaletsky, R. L., Francica, J. R., Agrawal-Gamse, C., Bates, P. Tetherin-mediated restriction of filovirus budding is antagonized by the Ebola glycoprotein. Proceedings of the National Academy of Sciences of the United States of America. 106 (8), 2886-2891 (2009).
- Tokarev, A., Skasko, M., Fitzpatrick, K., Guatelli, J. Antiviral activity of the interferon-induced cellular protein BST-2/tetherin. AIDS Research and Human Retroviruses. 25 (12), 1197-1210 (2009).
- Gnirss, K., et al. Tetherin sensitivity of influenza A viruses is strain specific: Role of hemagglutinin and neuraminidase. Journal of Virology. 89 (18), 9178-9188 (2015).
- Fleury, D., et al. A complex of influenza hemagglutinin with a neutralizing antibody that binds outside the virus receptor binding site. Nature Structural Biology. 6 (6), 530-534 (1999).
- Salunke, S. B., et al. Iron(III) chloride as an efficient catalyst for stereoselective synthesis of glycosyl azides and a cocatalyst with Cu(0) for the subsequent click chemistry. Chemical Communication. (Camb). 47 (37), 10440-10442 (2011).
- Schilling, P. E., et al. Mannosylated hemagglutinin peptides bind cyanovirin-N independent of disulfide-bonds in complementary binding sites. RSC Advances. 10 (19), 11079-11087 (2020).
- Fleury, D., Wharton, S. A., Skehel, J. J., Knossow, M., Bizebard, T. Antigen distortion allows influenza virus to escape neutralization. Nature Structural Biology. 5 (2), 119-123 (1998).
- Maier, I., Schiestl, R. H., Kontaxis, G. Cyanovirin-N binds viral envelope proteins at the low-affinity carbohydrate binding site without direct virus neutralization ability. Molecules. 26 (12), 3621 (2021).
- Ahmed, A. J., Keremane, S. R., Vielmetter, J., Bjorkman, P. J. Structural characterization of GASDALIE Fc bound to the activating Fc receptor FcγRIIIa. Journal of Structural Biology. 194 (1), 78-89 (2016).
- Boyd, R. Discovery of cyanovirin-N, a novel human immunodeficiency virus-inactivating protein that binds viral surface envelope glycoprotein gp120: potential applications to microbicide development. Antimicrobial Agents and Chemotherapy. 41 (7), 1521-1530 (1997).
- Bolmstedt, A. J., O'Keefe, B. R., Shenoy, S. R., McMahon, J. B., Boyd, M. R. Cyanovirin-N defines a new class of antiviral agent targeting N-linked, high-mannose glycans in an oligosaccharide-specific manner. Molecular Pharmacology. 59 (5), 949-954 (2001).
- O'Keefe, B. R., et al. Potent anti-influenza activity of cyanovirin-N and interactions with viral hemagglutinin. Antimicrobial Agents and Chemotherapy. 47 (8), 2518-2525 (2003).
- Shenoy, S. R., et al. Multisite and multivalent binding between cyanovirin-N and branched oligomannosides: calorimetric and NMR characterization. Chemical Biology. 9 (10), 1109-1118 (2002).
- Bewley, C. A., Kiyonaka, S., Hamachi, I. Site-specific discrimination by cyanovirin-N for alpha-linked trisaccharides comprising the three arms of Man(8) and Man(9). Journal of Molecular Biology. 322 (4), 881-889 (2002).
- Barrientos, L. G., Matei, E., Lasala, F., Delgado, R., Gronenborn, A. M. Dissecting carbohydrate-Cyanovirin-N binding by structure-guided mutagenesis: functional implications for viral entry inhibition. Protein Engineering Design & Selection. 19 (12), 525-535 (2006).
- Bewley, C. A., Otero-Quintero, S. The potent anti-HIV protein cyanovirin-N contains two novel carbohydrate binding sites that selectively bind to Man(8) D1D3 and Man(9) with nanomolar affinity: implications for binding to the HIV envelope protein gp120. Journal of the American Chemical Society. 123 (17), 3892-3902 (2001).
- Bewley, C. A. Solution structure of a cyanovirin-N:Man alpha 1-2Man alpha complex: structural basis for high-affinity carbohydrate-mediated binding to gp120. Structure. 9 (10), 931-940 (2001).
- Jensen, S. M. R., et al. Differential inhibitory effects of Cyanovirin-N, Griffithsin, and Scytovirin on entry mediated by envelopes of gammaretroviruses and deltaretroviruses. Journal of Virology. 88 (4), 2327-2332 (2014).
- Lan, J., et al. Structure of the SARS-CoV-2 spike receptor-binding domain bound to the ACE2 receptor. Nature. 581 (7807), 215-220 (2020).
- Lingwood, D., et al. Structural and genetic basis for development of broadly neutralizing influenza antibodies. Nature. 489 (7417), 566-570 (2012).
- Ekiert, D. C., et al. Antibody recognition of a highly conserved influenza virus epitope. Science. 324 (5924), 246-251 (2009).
- Pinto, D., et al. Cross-neutralization of SARS-CoV-2 by a human monoclonal SARS-CoV antibody. Nature. 583 (7815), 290-295 (2020).
- Keeffe, J. R., et al. Designed oligomers of cyanovirin-N show enhanced HIV neutralization. Proceedings of the National Academy of Sciences of the United States of America. 108 (34), 14079-14084 (2011).
- Yang, F., et al. Crystal structure of cyanovirin-N, a potent HIV-inactivating protein, shows unexpected domain swapping. Journal of Molecular Biology. 288 (3), 403-412 (1999).
- Stemmer, W. P., Crameri, A., Ha, K. D., Brennan, T. M., Heyneker, H. L. Single-step assembly of a gene and entire plasmid from large numbers of oligodeoxyribonucleotides. Gene. 164 (1), 49-53 (1995).
- Fischer, M. J. E., Mol, N., Fischer, M. Amine coupling through EDC/NHS: A practical approach. Surface Plasmon Resonance. Methods in Molecular Biology (Methods and Protocols). 627, (2010).
- Novoradovsky, A., et al. Computational principles of primer design for site directed mutagenesis. Technical Proceedings of 2005 NSTI Nanotechnology Conference and Trade Show. , 532-535 (2005).
- . QuikChange Site-Directed MutagenesisKit, User Manual Available from: https://users.drew.edu/jliu3/Docs/Stratagene%20Quikchange%20mutagenesis.pdf#:~:text=The%20QuikChange%20sitedirected%20mutagenesis%20kit%20is%20used%20to (2005)
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- . Expasy.org Available from: https://web.expasy.org/cgi-bin/protparam/protparam (2022)
- Karlsson, R. Real-time competitive kinetic analysis of interactions between low-molecular-weight ligands in solution and surface-immobilized receptors. Analytical Biochemistry. 221 (1), 142-151 (1994).
- Schuck, P., Zhao, H. The role of mass transport limitation and surface heterogeneity in the biophysical characterization of macromolecular binding processes by SPR biosensing. Methods Molecular Biology. 627, 15-54 (2020).
- Barnes, O., et al. SARS-CoV-2 neutralizing antibody structures inform therapeutic strategies. Nature. 588 (7839), 682-687 (2020).
- . Using reichert Surface Plasmon Resonance (SPR) for Antiviral Development, Application Note 28 Available from: https://www.reichertspr.com/clientuploads/directory/application_notes/Application_Note_28__Using_Reichert_Surface_Plasmon_Resonance_for_Antiviral_Testing.pdf (2022)
- Sundberg, E. J., Andersen, P. S., Gorshkova, I. I., Schuck, P., Schuck, P. Surface plasmon resonance biosensing in the study of ternary systems of interacting proteins. Protein Interactions: Biophysical Approaches for the Study of Complex Reversible Systems. 5, 97-141 (2007).
- . Method Development Notes Available from: https://www.reichertspr.com/applications/method-development-notes/ (2022)
- Angulo, J., Enríquez-Navas, P. M., Nieto, P. M. Ligand-receptor binding affinities from saturation transfer difference (STD)-NMR spectroscopy: the binding Isotherm of STD initial growth rates. Chemistry. 16 (26), 7803-7812 (2010).
- Goldflam, M., Tarragó, T., Gairí, M., Giralt, E. NMR studies of protein-ligand interactions. Methods in Molecular Biology. 831, 233-259 (2012).
- Kumar, S., Maurya, V. K., Prasad, A. K., Bhatt, M. L. B., Saxena, S. K. Structural, glycosylation and antigenic variation between 2019 novel coronavirus (2019-nCoV) and SARS coronavirus (SARS-CoV). Virusdisease. 31 (1), 13-21 (2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved