Method Article
冷冻电子断层扫描远程数据收集和断层扫描平均
摘要
本协议描述了使用Tomo5的高分辨率冷冻电子断层扫描远程数据采集以及使用emClarity的后续数据处理和子断层图平均。以载铁蛋白为例,说明实现 2.86 Å 分辨率冷冻电子断层扫描结构的详细分步过程。
摘要
冷冻电子断层扫描(cryo-ET)近年来发展势头强劲,特别是自从引入直接电子探测器,改进的自动采集策略,扩展电子显微镜使用冷冻电子断层扫描进行高分辨率成像的可能性的制备技术以及新的断层图平均软件以来。此外,数据采集变得越来越简化,使许多用户更容易访问。SARS-CoV-2 大流行进一步加速了全球许多设施的远程冷冻电子显微镜 (cryo-EM) 数据收集,尤其是单颗粒冷冻电镜,使用户在大流行期间能够不间断地访问最先进的仪器。随着Tomo5(3D电子断层扫描软件)的最新进展,远程冷冻电子断层扫描数据收集已变得强大且易于从世界任何地方处理。本文旨在提供详细的演练,从断层扫描软件中的数据收集设置开始,用于(远程)冷冻电子断层扫描数据收集会话的过程,并进行详细的故障排除。(远程)数据收集协议进一步补充了以apoferritin为例,通过emClarity的亚断层图平均,以近原子分辨率进行结构测定的工作流程。
引言
众所周知,低温电子显微镜(cryo-EM)经历了一个复兴时期,加速了它成为结构生物学的核心和中心有用的工具。直接电子探测器1,2,3的开发与利用,改进的显微镜和电子源3,4,5,自动化/吞吐量的改进6,7,8,9,以及单颗粒分析的计算进展10,11,12,13,14和断层扫描15,16,17都是该技术最近成功的部分原因。这些技术驱动因素已经发展了冷冻电镜在低温和天然条件下解决生物大分子结构的能力。容易获得的分辨率足以进行原子精确的建模,并将该技术带到结构生物学领域的最前沿。表达和纯化目标生物靶标的还原论方法早已在基础生物学研究、药物发现和转化科学的大分子晶体学 (MX) 中被证明是成功的。以同样的方法,冷冻电镜现在可以提供与高分辨率MX研究平行的结果。目前在结构生物学的冷冻电镜分支中取得的主要成功称为单颗粒分析(SPA),它获取通常纯化蛋白质标本18的2D投影图像,以获得生物大分子19的数千个视图。这些图像 (1) 包含来自一系列视图的信息,这些信息完全代表了 3D 空间中目标的方向,以及 (2) 捕获物体构象异质性,以后可以对其进行分离和研究。
另一种获取生物样品的2D投影图像的方法是冷冻电子断层扫描(cryo-ET),即使是 原位 且未经纯化。冷冻电子断层扫描通过机械旋转标本,以倾斜角度拍摄同一物体的一系列图像。因此,在SPA中收集的2D投影,代表目标分子的角度姿势,本质上是作为冷冻电子断层扫描成像实验20的一部分收集的。然后将断层倾斜系列重建为断层图,其中包含成像大分子复合物的 3D 表示。断层扫描数据收集的性质在一定程度上确实减少了对平均的依赖,以实现从 2D 图像集合中实现分子的完整 3D 表示。然而,由于当前的载物台设计,试样通常从-60°倾斜到+60°,在断层扫描3D重建中留下缺失的楔形21 信息。
然后,单个断层图中的3D重建具有缺失的信息楔形和低信噪比。可以将单个大分子提取为断层图下图,并平均在一起以解决此问题。如果子断层图中的每个大分子以不同的方向找到,则在目标对象的每个子断层图中,缺失的楔形图的方向不同,因此由于缺少楔形,对许多副本进行平均会填充信息。图像处理的最新发展也试图训练人工智能神经网络,用有意义的数据填补缺失的楔子22。这种平均过程还增加了信噪比,类似于单颗粒分析中的平均目标,因此重建的质量和分辨率得到了提高。如果感兴趣的分子具有对称性,也可以在平均过程中定义和使用,从而进一步提高重建分辨率。将大分子的 3D 体积从断层图提取到一组子断层图及其后续处理中称为断层图平均 (STA)23。如果每个断层扫描代表正在研究的分子的唯一副本,则可以使用STA工作流程询问任何结构异质性。正如SPA工作流程中常用的那样,在STA期间可以采用分类技术来剖析感兴趣的复合物的构象状态。除了STA能够在冷冻电子断层扫描中实现高分辨率重建外,这种方法使该技术成为研究天然细胞环境中大分子结构机制或通常不适合SPA24,25,26的靶标的有力工具。
电子断层扫描在室温下确定细胞标本的3D超微结构方面有着悠久的历史27。通过标本的物理倾斜获取视图为在细胞长度尺度上对物体进行 3D 重建提供了足够的信息,当细胞结构缺乏平均的规律性时尤其重要。细胞也可以冷冻到基板上,以便在细胞边缘进行冷冻电子断层扫描成像,其中标本足够薄以透明电子。在这些条件下,STA可用于确定细胞环境中的大分子结构,尽管当样品足够薄以达到电子透明时28。然而,当与其他制备技术相结合时,包括冷冻相关光学和电子显微镜(cryo-光电联用显微)和聚焦离子束铣削(cryo-FIB),冷冻电子断层扫描可用于在低温条件下对整个细胞内部进行成像29。这将冷冻电子断层扫描研究细胞超微结构的能力与STA的力量相结合,原 位 确定大分子复合物的结构,同时识别其细胞位置30 并提供参与动态过程的复合物的快照31。该技术对细胞标本进行成像并在多项研究中采用STA的能力突出了该技术在 原位 解决大分子结构的能力,即使在与SPA32相当的分辨率下也是如此。另一个好处是了解大分子的原始位置,由断层图30中的最终分类3D重建表示。因此,大分子结构可以与细胞超微结构相关。这些跨长度尺度的观察可能会导致重要的发现,其中结构机制可能与功能研究中的细胞变化相关。
冷冻电子断层扫描和 STA 允许在三个主要工作流程中收集数据:分子、细胞和薄片断层扫描。纯化的大分子复合物的结构可以通过分子断层扫描通过冷冻断层扫描来确定。在细胞足够薄的细胞环境中确定蛋白质结构可描述为细胞断层扫描。最近,随着低温靶向和铣削的发展,这些相同的技术可以应用于薄片断层扫描工作流程,以确定细胞深处天然环境中的蛋白质结构,同时揭示观察到这些蛋白质的细胞环境。根据可用的软件包,最重要的是,可以根据样品的要求使用不同的数据收集策略。纯化蛋白质的铜 TEM 网格上的分子或非贴壁样品通常需要较少的处理,因此在理想情况下保持平坦且未损坏。电子断层扫描可以很容易地在孔碳网格上串联设置,以系统的方式快速获取数十到数百个断层图。用户设置蛋白质大量存在于网格上的分子断层扫描样品的最简单方法是使用Tomo5(本研究中使用的3D电子断层扫描软件,见材料表)。其他断层扫描软件,如 Leginon9 和 serialEM6 也可用;它们为更个性化的数据收集方法提供了更多的设置选项,但更复杂,因此更难导航,特别是对于刚接触断层扫描的用户和远程访问其会话的用户。对于拥有庞大而多样化的用户群的设施,Tomo5易于在远程环境中操作并培训用户。对于贴壁细胞,网格通常需要更多的处理步骤,并且使用易碎的金网格的必要性增加了在处理和数据收集策略中改进护理的需求。为了便于找到感兴趣的蜂窝区域并避免在高倾斜角度下被网格本身遮挡,使用更大的网格尺寸也是有益的,但代价是它们本质上更脆弱。对于薄片样品,样品的脆性由薄片的质量决定,这可能是可变的。这些因素增加了设置时间和考虑因素,但增加的适应性和鲁棒性再次使Tomo5适合这种类型的数据收集。但是,每个工作流都存在专门的数据收集方案。BISECT和PACE-tomo(均在SerialEM中运行)引入了在断层扫描采集过程中脚本化光束图像偏移的可能性,以提高断层扫描收集速度28,特别是在分子断层扫描中。SerialEM6,7,33中的中放大倍率蒙太奇(MMM)可以更好地识别和精确靶向所有工作流程中的分子特征,尽管在撰写本文时,这些功能已开始在Tomo5中实现。
与SPA一样,通过对采集软件的改进以及用于平均16,17,32,34,35,36,37,38的子断层图的大量可用软件包,冷冻电子断层扫描和STA变得越来越容易获得。此外,在大流行期间,能够远程访问冷冻电镜仪器对于英国钻石光源 (DLS) 的电子生物成像中心 (eBIC) 等国家设施的持续运营至关重要。这些发展使冷冻电子断层扫描对于希望利用该技术的研究人员来说更容易获得和强大。一旦获得数据,STA是分析循环对象以获得最大分辨率重建并允许对大分子异质性进行分类的重要工具。目前的协议旨在提供准备冷冻透射电镜显微镜以进行冷冻电子断层扫描数据收集的详细演练,以及如何使用emClarity对载铁蛋白的分子断层扫描数据集作为示例进行断层图平均。使用 emClarity(用于高分辨率冷冻电子断层扫描和亚断层扫描平均的软件,参见材料表)需要从命令行运行脚本,因此假定对 Linux/UNIX 系统有一定程度的熟悉。
远程连接取决于每个研究所/设施的网络环境。在 eBIC,远程系统使用允许在 Diamond 使用的特定网络配置上进行远程数据收集的程序。与显微镜的远程连接可通过两个平台实现:NoMachine和TeamViewer(参见 材料表)。使用程序NoMachine,用户可以登录到远程Windows桌面。NoMachine提供的远程Windows桌面与显微镜位于同一网络上,因此充当显微镜的虚拟支持PC。用户通过虚拟支持PC连接到显微镜,通过TeamViewer直接访问和控制运行TUI和Tomo的显微镜PC。
本协议由两部分组成(步骤1和步骤2)。第 1 步侧重于使用 Tomo5(3D 电子断层扫描软件)进行远程冷冻电子断层扫描数据采集。(远程)会话的演练以越来越高的放大倍率捕获图像,最终允许用户将断层扫描软件引导到标本区域进行断层扫描数据收集。 图 1 总结了此过程。第 2 步详细介绍了使用 emClarity(高分辨率冷冻电子断层扫描和亚断层图平均软件)进行冷冻电子断层扫描 STA 数据处理。 图 9 总结了此过程。
该协议适用于远程受众。它假设人员在显微镜下并加载样品已完成直接对准,并负责相机调整和增益参考获取。对于该协议,假设使用带有自动加载器的三聚光镜系统。有关断层扫描软件的进一步详细指南,可在加载软件的 Windows 开始按钮中找到制造商的详细手册。
研究方案
本研究中使用的软件包部分是免费提供的(见 材料表)。
1. 使用Tomo5进行远程冷冻电子断层扫描数据采集
- 如果未加载软件,请首先从TEM服务器PC启动此软件(请参阅 材料表)。
- 通过选择 预设执行初始检查并配置成像条件。
- 通过在 预设 下的"准备"选项卡中调整图像采集参数来启动会话设置(图2A,B)。
- 调整"概述放大倍率"以适应网格平方大小和剂量以获得足够的计数。
注意:如果网格类型的合适放大倍率未知,请在加载网格后返回到此步骤。 - "以心为中心的高度"和"搜索放大倍率"可以相同。单击"断层扫描"选项卡中的 搜索 以设置位置并确保放大倍率足以显示感兴趣的标志,并在视野中适合"曝光"和"跟踪/对焦"区域(图2C)。
- 将"曝光放大倍率"设置为所需的目标像素大小。如果未知,则通过将放大倍率调整到所需的视野以适合感兴趣区域来建立它。
- 调整"概述放大倍率"以适应网格平方大小和剂量以获得足够的计数。
- 将图像采集(曝光)的参数复制到所有其他高放大倍率预设(跟踪、漂移、对焦、Thon Ring、零损失)。为此,请按"设置"将"曝光"设置为显微镜,并在单独选择所有其他高放大倍率后获取所有其他高放大倍率的"曝光设置"。
- 调整曝光时间,特别是对于"聚焦"和"跟踪"以给出适当的计数数,还要考虑60°处的厚度,即在0°和60°处,以使足够的电子通过样品以提供强信号互相关。
注意:作为估计,"跟踪"和"对焦"的曝光时间等于"曝光"预设是一个很好的第一测量。有关"曝光"预设的剂量计算示例,请参见 图3。 在"准备"选项卡中设置"曝光"预设的计算曝光时间。
- 调整曝光时间,特别是对于"聚焦"和"跟踪"以给出适当的计数数,还要考虑60°处的厚度,即在0°和60°处,以使足够的电子通过样品以提供强信号互相关。
- 调整"零损失预设"以使用更亮、更大的光斑尺寸(光斑尺寸越大对应数量越小),因为这需要更高的剂量。
注意:如果显微镜上存在能量过滤器的狭缝,则此预设最适合用于对齐。
- 通过在 预设 下的"准备"选项卡中调整图像采集参数来启动会话设置(图2A,B)。
- 按照以下步骤执行图集收集。
- 要收集图集,请单击"图集"选项卡中的新会话,设置 会话 首选项,输入存储路径和输出格式,然后按 应用。选择 筛选 (图4),然后勾选所有要获取的图谱。选择 关闭科尔阀 以使显微镜无人监督;这将在收集所有选定的图集后关闭柱阀。要开始放映,请按 "开始" 按钮。
- 通过单击左侧面板中"筛选"下的 网格 (图4)来检查单个或多个目标图集,然后左键单击并拖动鼠标以移动并中键滚动以放大和缩小。选择目标设置的 网格 ,选择它,然后单击从软件内部 加载样本 。
- 对于图像偏移校准,请继续使用图集筛选选项卡在加载的网格内导航。找到在所有放大倍率预设下都可以识别的可识别特征,即与孔边缘或其他可识别特征重叠的冰晶(图 5)。通过鼠标右键单击移动到该正方形,然后"将舞台移动到网格正方形"。
注意:载物台需要处于共心高度才能正确实施图像偏移校准。
- 执行图像偏移校准。
- 在"自动功能"选项卡(图6)中,将预设设置为"真心高度",导航到"通过平台倾斜自动以心为中心",然后按 开始。监控状态窗口以确保成功找到以心为中心的高度,并密切关注正负倾斜图像;它们必须相关。
注意:从Tomo软件的5.8版本开始,可以修改以心为中心的高度接受标准;默认值为 0.25 μm,将其设置为 0.5-0.8 μm 可提供更大的回旋余地。数值取决于显微镜性能,但建议它们尽可能小。- 当处于以真心为中心的高度并处于焦点时,将舞台居中放在要素上。使用"地图集"预设收集预览。从"准备"选项卡中设置所有"预设"。随后,将放大倍率增加到 "预览">>中心功能的概述,然后重复"搜索放大倍率",最后重复"曝光放大倍率"。
- 如果特征在定位期间保持居中,请跳过图像偏移校准。否则,要校准图像偏移,请转到"准备"选项卡,选择 校准图像偏移,然后按 开始 (图5)。这将迭代地将较低的放大倍率与以曝光放大倍率为中心的特征对齐。
注意:如果初始预设"曝光"在此阶段未居中,则对于重新居中,软件将仅在此步骤中使用舞台偏移。- 按 继续 进入"曝光"预设。显示的下一个图像将是"搜索预设"。要校准预设之间的图像偏移,请在"搜索"预览中双击"曝光"预览的中心位置,然后按 重新获取 (图5)。点击 继续 移动到下一对预设。
注意:如果在此校准期间应用了图集放大倍率的图像偏移,请确保在图像偏移校准完成后重新获取图集。选择所需的图集,然后在"图集"选项卡的顶部面板中按 重置所选内容 。确认并再次开始获取该地图集。
- 按 继续 进入"曝光"预设。显示的下一个图像将是"搜索预设"。要校准预设之间的图像偏移,请在"搜索"预览中双击"曝光"预览的中心位置,然后按 重新获取 (图5)。点击 继续 移动到下一对预设。
- 在"自动功能"选项卡(图6)中,将预设设置为"真心高度",导航到"通过平台倾斜自动以心为中心",然后按 开始。监控状态窗口以确保成功找到以心为中心的高度,并密切关注正负倾斜图像;它们必须相关。
- 执行断层扫描设置。
- 在"断层扫描"选项卡中创建断层扫描数据收集设置。
注意:除非特别说明,否则设置完全在"断层扫描"选项卡中完成。 - 启动 新会话。在生物样品的"会话设置"中,选择板 状 作为样品类型,然后选择 批量 和 低剂量,选择输出格式和存储文件夹,(可选)添加电子邮件收件人,然后按 应用。
注:"批次位置"菜单项现在变为可用(图7A)。当前加载的图集会自动导入。可以获取和重新访问"概述"和"搜索图像",直到获取新图像为止。此外,从版本 5.8 开始,可以使用"获取搜索地图"获取 3 x 3、4 x 4 和 5 x 5 图块的搜索地图,以简化目标查找和设置。它们对应于中等放大倍率蒙太奇的一个版本。 - 要设置目标,请转到"Atlas箭头",找到感兴趣的区域,然后通过选择右键单击弹出的选项移动到那里。拍摄概览图像以确认以优化中心高度调整的良好位置,然后按 自动同心;这将运行 EUCENTRIC 按阶段倾斜例程。然后,重新获取新的概览图像以更新真心高度。
注意:如果位置处于共心高度,则不需要"自动对焦"按钮。如果真心高度图像看起来远远失焦,则真心高度可能不正确,必须重做(有关真心高度故障排除,请参阅讨论部分)。 - 在设置第一个位置之前,使用"概览"预设检查目标;选择"概览"预设并将载物台倾斜至±60°,以检查位置可以设置与网格条的距离(图8)。通过在"设置倾斜"窗口中输入所需的倾斜角度值来执行此操作(图 8A)。
- 检查方形"概览"或获取的"搜索地图",移动到感兴趣的区域,然后按 获取搜索。检查"搜索"图像。如果感兴趣区域未居中,请右键单击所需位置,然后重复 "将舞台移至此处 "并 获取图像。将倾斜度 (°) 设置为 0.00(或载物台可以处理的任何其他起始角度)以定义断层扫描的起始角度。
- 对于第一个断层扫描,输入所需断层影像命名 约定的名称和 散焦以为每个断层影像设置所需的 散焦 值。
- 或者,从Tomo 5.8版本开始,可以一次性系统地更改散焦值;为此,单击选择位置,选择要更改的位置或勾选复选标记以 选择所有位置,然后单击 更新散焦 并调整参数(图 7A)。
- 调整"焦点"和"跟踪"区域(图2C)。左键单击以拖动跟踪和焦点区域(黄色和蓝色圆圈)。确保跟踪/聚焦区域(大部分)位于碳或其他合适的跟踪特征上,即与感兴趣区域相似的特征;避免裂缝、冰污染、过厚的区域和空洞。
- 当选择没有许多跟踪特征的空白碳时,通过选择足够高的聚焦剂量,并确保该区域在断层扫描中途开始燃烧,并跟踪以更高的倾斜度缓慢燃烧样品。这有利于保持跟踪精度。确保跟踪/对焦区域不会暴露以后的采集区域。
注意:要小心靠近的东西和进入光束的东西。避免具有会干扰跟踪和/或曝光的特征的区域,包括网格条。
- 当选择没有许多跟踪特征的空白碳时,通过选择足够高的聚焦剂量,并确保该区域在断层扫描中途开始燃烧,并跟踪以更高的倾斜度缓慢燃烧样品。这有利于保持跟踪精度。确保跟踪/对焦区域不会暴露以后的采集区域。
- 设置所有参数并定义所需的感兴趣区域以及"焦点"和"跟踪"后,按 添加位置 。对新目标重复此操作。
注意:要检查已设置的批次位置的共心高度是否正确校准,有几种可用的策略。建议根据所进行的断层扫描类型(分子、细胞、薄片)选择一个。 - 对于分子断层扫描,假设网格相当平坦,并且在包含目标位置的每个正方形的中心都完成了真中心高度,请跳过"全部优化"(如果选择了位置,则优化所选, 图7A)过程。如果Tomo在通过舞台倾斜程序自动以心为中心方面遇到困难,可以单击 跳过以中心为中心。
- 对于细胞或薄片样品,每个批次位置可能位于不同的 z 高度。要么使用"全部优化"来谨慎,要么不要勾选"跳过以Eucentric"选项。
注意:"全部优化"将遍历通过"选择位置"设置或选择的所有断层扫描;细化以心为中心的高度,并在数据采集前执行跟踪和聚焦。此过程可以暴露来自下一个断层扫描焦点/跟踪区域的任何重叠曝光(图7B),在继续之前应考虑这一点。 - 检查并重新访问未通过优化的方块。使用鼠标右键,单击 失败的位置 以查看选项。如果共心高度失败,请找到"通过平台倾斜自动以心为中心"(步骤1.4.1.),获取新的"搜索"图像以更新z高度,然后"添加位置"。删除失败/先前初始化的位置。单击" 跳过以Eucent为中心 "选项,该选项已通过"全部优化"完成。
注意:如果未运行"全部优化",则不得选中"跳过以真心为中心"。Tomo5 将在获取每个断层图之前运行以真为中心的改进;这样,将跳过未通过以真心为中心的细化的位置。
- 对于细胞或薄片样品,每个批次位置可能位于不同的 z 高度。要么使用"全部优化"来谨慎,要么不要勾选"跳过以Eucentric"选项。
- 在"断层扫描"选项卡中创建断层扫描数据收集设置。
- 按照以下步骤执行自动功能。
- 检查设置以通过"自动功能"选项卡 执行 对齐,方法是在图集中导航到碳区域。使该区域达到真心高度,并遵循如下所述的对齐顺序。
- 使用"真心高度"预设通过载物台倾斜运行 自动真心 。
- 使用"对焦"预设运行 自动对焦例程 。
- 使用"Thon Ring"预设运行 自动污名 例程。
- 使用"Thon Ring"预设运行 自动通信 例程。
- 插入所需的物镜孔径(图6B),可能是100 μm孔径,作为提高样品对比度的良好折衷方案,并且在非常高的分辨率下信号中只有很小的截止值。
- 在光圈插入后重复 自动柱头 程序。
注意:在像素大小约为 3 Å 且分辨率更低的放大倍率下,自动彗视例程可能会失败;在这种情况下,请在 TEM 用户界面中的直接对齐下选中并对齐 Tomo 旋转中心。
- 检查 自动零损失 定心程序是否适用于您的样品。将"零损失"预设为合理的高剂量(步骤1.2.3),自动功能中的例程将更有可能成功。
- 检查设置以通过"自动功能"选项卡 执行 对齐,方法是在图集中导航到碳区域。使该区域达到真心高度,并遵循如下所述的对齐顺序。
- 执行数据收集。
- 要在"断层扫描"选项卡中开始自动采集,请选择 数据采集 板并设置所需的参数(图7C)。设置数据采集参数:倾斜步长(°)、最大正角(°)、最大负角(°)、跟踪方案(推荐: 选择采集前跟踪/对焦)。选择 关闭柱阀 以获得共心高度方案。
注意:很少使用的参数是"调整曝光时间"、"调整 ZLP"、"使用相位板"和"倾斜后暂停"。- 对不适用于 STA 的断层扫描使用"调整曝光时间",以在较高倾斜度下以指定比率增加 曝光时间 。
- 调整 ZLP 选项将在每次断层扫描后运行 ZLP 细化,无论设置的周期如何。它很慢,偶尔会失败而没有明显的原因,并且会跳过该位置获取。但是,它可用于非常窄的狭缝宽度,即Selectris(X)滤波器上的3-5 eV。
- 使用相板自从引入带有能量过滤器的DED以来很少使用。
- 倾斜后 暂停会暂停采集,但不允许对软件进行任何更改。
- 仔细检查数据收集参数(在"制备"选项卡中),同意剂量计算和所需的倾斜方案,在"曝光"预设中指定 馏分(Nr. )的数量,然后开始采集。
注意:馏分的数量取决于样品、采集参数和计划的后处理步骤,即 STA 与形态学分析,并且必须保持在运动校正和 CTF 估计获得足够信号的值。4-10 馏分的范围是一个很好的估计,其中 4 更适合较厚的样品,10 更适合较薄的分子样品。- 然后,单击"开始" 开始 数据收集并监控第一次断层扫描采集,以确保按预期采集断层影像。
注意:最近的断层扫描软件版本在"断层扫描"选项卡中有一个额外的电影播放器平板,可以在其中观看获取的断层扫描。在加载过程中要有耐心。
- 然后,单击"开始" 开始 数据收集并监控第一次断层扫描采集,以确保按预期采集断层影像。
- 要在"断层扫描"选项卡中开始自动采集,请选择 数据采集 板并设置所需的参数(图7C)。设置数据采集参数:倾斜步长(°)、最大正角(°)、最大负角(°)、跟踪方案(推荐: 选择采集前跟踪/对焦)。选择 关闭柱阀 以获得共心高度方案。
2. 使用emClarity对载铁蛋白进行冷冻电子断层扫描STA。
注意:此处,emClarity软件17 用于说明通过STA进行冷冻断层扫描结构测定。 图 9 总结了此过程。以6个倾斜系列的载铁蛋白(EMPIAR-10787)为例。应用八面体对称性,最终地图的分辨率为2.86 Å,仅从4,800个粒子中获得,接近奈奎斯特频率(2.68 Å)。
- 确保已下载并安装emClarity软件包39 (参见 材料表)。安装IMOD用于软件处理40。
注意:需要具备 Linux 命令的基本知识。更详细的教程可以在参考文献41中找到,它以核糖体数据集(EMPIAR-10304)为例,并描述了分步过程。对于不同类型的蛋白质样品,还提供先前发表的报告42。 - 准备输入文件和目录。
注:原始帧已通过 MotionCor2(补丁 5 x 5)43 进行了运动校正(请参阅 材料表)。使用newstack(IMOD)堆叠运动校正图像以生成倾斜系列,并使用Etomo手动对齐补丁跟踪(参见 材料表),然后是emClarity。为了获得更好的对齐,建议在Etomo中进行偏移和倾斜补偿。六个倾斜系列更名为TS1至TS6。以下是第一个倾斜系列 (TS1) 的说明和命令作为示例。- 建立项目文件夹。
注意:该软件的最新版本是 1.5.3.11。所有相关的软件日志都可以在本地项目文件夹(.../logFile/emClarity.logfile)中找到。 - 在项目文件夹下,创建另一个新文件夹"fixedStacks",并准备输入文件:TS1.fixed,TS1.xf和TS1.tlt。
注意:TS1.fixed:原始倾斜系列,也称为Etomo中的 TS1.st。TS1.xf:此文件包含 Etomo 倾斜对齐后的变换坐标。TS1.tlt:此文件包含倾斜角度。
- 建立项目文件夹。
- 按照以下步骤估计散焦。
- 计算散焦。使用 补充表1中的显微镜和成像参数更新参数文件。将参数文件复制到项目文件夹,将其名称更改为 param_ctf.m,然后运行: emClarity ctf 估计值param_ctf.m TS1。
注意:参数文件模板可以在 补充文件 1 或本地软件安装目录 (.../emClarity_1.5.3.11/docs/exampleParametersAndRunScript/ 中找到)。
- 计算散焦。使用 补充表1中的显微镜和成像参数更新参数文件。将参数文件复制到项目文件夹,将其名称更改为 param_ctf.m,然后运行: emClarity ctf 估计值param_ctf.m TS1。
- 检查每个堆栈的 CTF 估计结果。
- 使用 3dmod 检查对齐的堆栈(例如,aliStacks/TS1_ali1.fixed),并确保基准磁珠被正确擦除。
- 确保日志文件报告惯用手是正确的,可以在 logfile/emClarity.logfile 中找到(图 9)。
- 检查散焦值(例如,fixedStacks/ctf/TS_ali1_psRadial_1.pdf),并确保它与理论 CTF 估计值匹配。
- 定义子区域。
- 生成分箱断层扫描。在项目文件夹中,运行: sh recScript2.sh -1。
注意:每个堆栈的较小(大小)断层扫描将存储在新文件夹"bin10"中。为了加快该过程,可以在bin10断层图中定义一个或多个子区域的坐标。脚本文件将位于本地软件安装目录 (.../emClarity_1.5.3.11/docs/) 中。 - 通过选择 x最小值、x 最大值、y 分钟、y最大值、z分钟和 z最大值六个点来确定边界,以创建一个子区域。在文件夹"bin10"下,运行:3dmod TS1_bin10.rec。
注意: 如果要在一个倾斜系列中创建四个子区域,请选择 6 × 4 = 24 个点。每个堆栈必须在文件夹"bin10"下有一个模型文件 (*.mod)。 - 将模型文件转换为 emClarity 格式。这将创建新的文件夹侦察。将每个子区域的所有坐标存储在此文件夹下。在项目文件夹中,运行: sh recScript2.sh TS1。
- 生成分箱断层扫描。在项目文件夹中,运行: sh recScript2.sh -1。
- 拾取颗粒。
- 查找用于拾取颗粒的载铁蛋白模板。从电子显微镜数据库 (EMD-10101)44 下载模板。确保模板的像素大小与原始数据的像素大小匹配。在项目文件夹中,运行: emClarity 重新缩放 ApoF.mrc ApoF_Template_rescale.mrc 3.60 1.34 CPU。
- 生成用于粒子拾取的 CTF 校正断层扫描图。在项目文件夹中,运行: emClarity ctf 3d param_ts.m templateSearch。
- 为每个子区域运行粒子拾取。对于载铁蛋白数据集,请在 bin6 进行模板搜索。修改Tmp_angleSearch参数(θ 输出、Δ输出、θ 输入、Δ 输入)以确定平面内或面外搜索的角度范围和间隔(以度为单位)。在项目文件夹中,运行:emClarity templateSearch param_ts.m TS1 1 ApoF_Template_rescale.mrc O 1。
注意:此步骤将生成文件夹"convmap_wedge_Type2_bin6"。此文件夹下的 csv 文件(例如,TS1_1_bin6.csv)包含有关拾取的颗粒的所有信息。 - 使用 3dmod 删除不正确的粒子。在文件夹"convmap_wedge_Type2_bin6"下,运行:3dmod ../cache/TS1_1_bin6.rec TS1_1_bin6.mod.
注意:在这些区域的碳边缘或冰污染旁边发现错误的颗粒是很常见的。在不正确的点上单击鼠标右键,然后按键盘上的 退格 键将其删除。在此步骤中,确保删除大多数误报点(图10)。 - 将文件夹名称"convmap_wedge_Type2_bin6"更改为"convmap",因为emClarity将在以下步骤中在convmap下查找子区域信息。
- 初始化项目。在项目文件夹中,运行: emClarity init param0.m,为 emClarity 创建一个数据库, ApoF.mat。
- 在 STA 和对齐之前执行断层扫描重建。要在bin4处生成CTF校正的子区域断层扫描,请运行: emClarity ctf 3d param0.m。
注意:CTF 校正的断层扫描(例如,cache/TS1_1_bin4.rec) 将在新文件夹"cache"中生成。 - 执行 STA 和对齐。
- 从bin4开始,使用CTF校正的子断层扫描进行平均。在项目文件夹中,运行: emClarity avg param0.m 0 RawAlignment。
- 继续执行对齐并运行: emClarity alignRaw param0.m 0。
注意:平均步骤会生成一个参考,在此步骤中,emClarity 将使用该参考来对齐粒子。可以更改Raw_angleSearch参数(θ 输出、Δ输出、θ 输入、Δ 输入、Δ输入)以设置每个循环的角度范围和步长。由于大多数不正确的粒子已从数据库中删除(步骤2.6.4),因此这些对齐的角度设置可以从相对较小的程度开始。 - 更新Raw_angleSearch参数并再运行几个周期(步骤 2.9.1 和 2.9.2)。要加快该过程,请分别执行面内和面外对齐。
注意:对于每个分档,建议运行多个循环并逐渐减少角度设置。对于ApoF数据集,在bin 4处又进行了5个循环,更多详细信息见补充表2。对于 cycle001,在项目文件夹中,运行:emClarity avg param1.m 1 RawAlignment,后跟 emClarity alignRaw param1.m 1。 - 清洁重叠的颗粒。在项目文件夹中,运行: emClarity removeDuplicates param5.m 5。
- 使用断层心复苏术进行倾斜系列细化。
注意:此步骤是可选的。- 运行 tomoCPR 以优化堆栈几何形状。在项目文件夹中,运行: emClarity tomoCPR param5.m 5。
- 生成新对齐的堆栈并更新几何文件。在项目文件夹中,运行: emClarity ctf update param6.m。
注意: 确保正确生成新的几何文件(例如,fixedStacks/ctf/TS1_ali2_ctf.tlt)和新对齐的堆栈(例如,aliStacks/TS1_ali2.fixed)。 - 在 bin2 处创建新的子断层扫描。在项目文件夹中,运行: emClarity ctf 3d param6.m。
注意:接下来将在bin2(步骤2.9.1,2.9.2和2.9.3),重复清洁(步骤2.9.4)和可选的断层心复苏术(步骤2.10)上进行几个周期的平均值和对齐。由于高对称性,在bin2处对载铁蛋白进行了另外5个循环。更新每个周期的Raw_angleSearch参数。
- 执行最终重建。
- 继续在bin1和断层心肺复苏术下运行几个周期(步骤2.9和步骤2.10)。在bin1对apoferritin数据集进行了另外10个循环。有关命令和参数的更多详细信息,请参见 表 2。
- 通过组合两个半数据集来执行最终重建。在项目文件夹中,运行:emClarity avg param21.m 21 RawAlignment,然后运行 emClarity avg param21.m 21 FinalAlignment。
注意:将生成最终映射(例如,B 因子为 10,cycle021_ApoF_class0_final_bFact-10.mrc), 如图 11 所示。
结果
对于细胞和薄片样品,数据收集策略在很大程度上取决于样本和成像研究的目标(图1)。靶向方法取决于分子靶标是 原位还是 由纯化的大分子复合物制备,用于含有分子靶标的高分辨率重建标本。对于玻璃化到多孔(碳)网格上的纯化复合物,靶向可以简单地基于(碳)支撑膜孔中的成像。对于 原位 工作,靶向方法需要根据相关数据或已知的低放大倍率细胞标志物了解分子实体的位置。理想情况下,在拍摄概览图像时,蜂窝地标是可识别的,如果足以大致定位感兴趣的区域,则可以提供一种通过搜索图像确认目标身份的快速方法。但是,如果可观察到的事件很少见,则可能需要中等放大倍率的搜索图像来鉴定目标是否正确。搜索地图是搜索图像的中等放大蒙太奇,因此可以使目标查找更加容易,可以在可见感兴趣特征的放大倍率下获取目标。然后可以筛选搜索地图以查找和设置目标批次位置。对于包含用于超微结构重建的细胞特征的标本,靶向方法是相似的,但同样取决于细胞事件在不同放大倍率下的可见性及其在样品中的普遍性。
还必须考虑数据采集策略;在所有情况下,研究的目标在很大程度上决定了数据的收集方式。对于细胞超微结构的重建,低放大倍率(20-5 Å / px)和大视野可能是合适的,但需要高放大倍率来重建分子或高分辨率细节(5-1 Å / px)。在理想条件下以 1.5 Å/px 收集的数据集在物理上只能以 3.0 Å/px 的奈奎斯特频率生成重建;然而,在现实中,许多因素,包括但不限于试样厚度、尺寸和异质性,都会影响获得的重建质量。精确的成像参数还可以平衡基于研究目标的放大倍率与视野,以包含足够的信息。本文介绍了一个达到2.86 Å的断层扫描平均病例,但表1中提供了其他研究17,42,43,44,45,以说明与针对不同结局的研究相关的收集参数17,42,45,46。
一旦建立了冷冻电子断层扫描的靶向工作流程和数据采集制度,就可以收集许多不同样品类型的数据。这里展示了各种标本的代表性断层图:分子样本,如载铁蛋白(视频 1)、薄细胞过程(视频 2)和厚细胞标本的 FIB 铣削薄片(视频 3)。
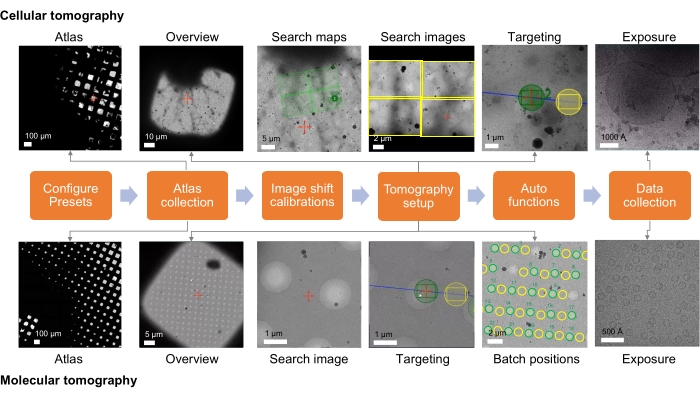
图 1:断层扫描工作流程设置概述。 协议中描述的冷冻电子断层扫描成像工作流程显示为流程图。显示了预期采集的图像,用于细胞和分子工作流程。所呈现的命名遵循Tomo5惯例,尽管大多数断层扫描采集软件共享收集这些图像的共同原则。 请点击此处查看此图的大图。
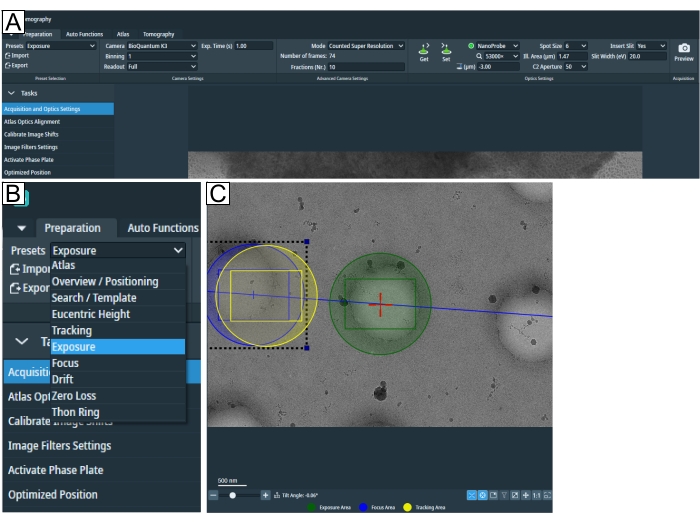
图 2:准备选项卡和搜索预设条件 。 (A) 整个选项卡的概览图像。在此选项卡中设置了成像条件预设,"校准图像偏移"和"图像过滤器设置"可以在"任务"下拉列表中找到。(B) 放大"预设"下拉菜单,其中可以为单独的成像条件设置选择每个预设。(C) 描绘足够放大倍率的图像,以将曝光和"聚焦和跟踪"区域都放入视野中。 请点击此处查看此图的大图。
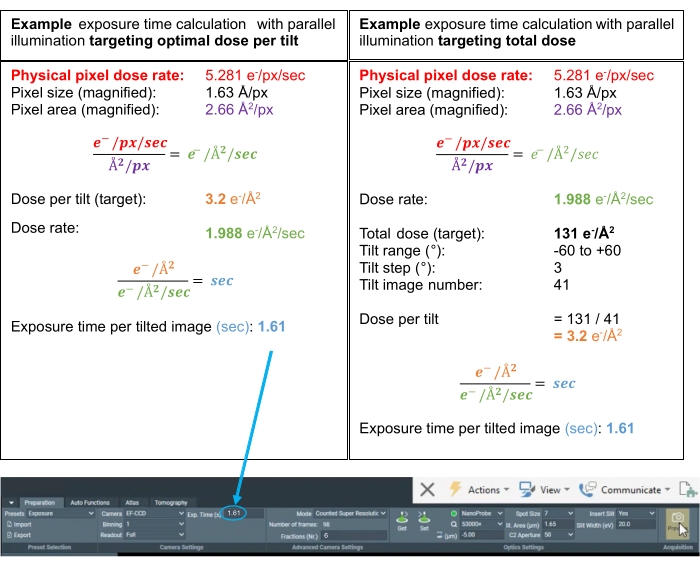
图3:剂量计算。 可能的断层扫描采集方案的剂量计算示例,其中剂量率已在真空上测量。这两个计算确定了每次倾斜的曝光时间,无论是针对每次倾斜的最佳剂量还是全倾斜系列的最佳总剂量。在断层扫描平均中,通常的做法是将每个倾斜图像的最佳剂量定在 3.0-3.5 e-/Å2 范围内。在这两种情况下,"分数(Nr.)"都设置为6,以实现每帧电影倾斜的~0.5 e-/Å2 剂量,以获得足够的信号来执行运动校正。 请点击此处查看此图的大图。
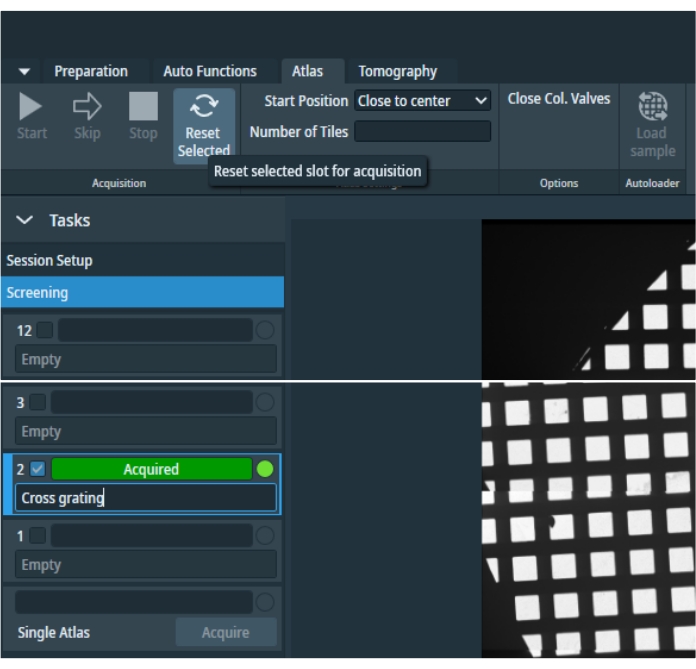
图 4:图集选项卡。"地图集"选项卡的概述图像。图像已被裁剪以避免空的网格位置空间。"任务"菜单包含会话设置首选项和网格选择空间,用于单独选择所有清点的网格,以及在从自动加载器中取出盒式磁带后获取单个图集的选项。选定的网格可以重置,然后重新获取。请点击此处查看此图的大图。
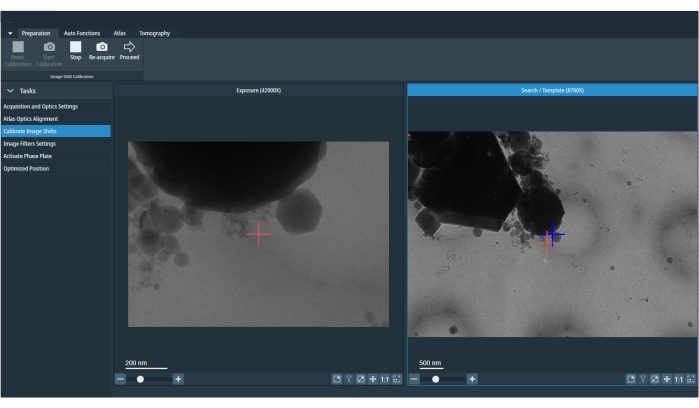
图 5:校准的图像偏移。 图像描绘了曝光和搜索图像。搜索图像中的红叉是移动标记,用于校正曝光和搜索之间的偏移。应在会话开始时或更改成像条件预设后重做图像偏移校准。 请点击此处查看此图的大图。
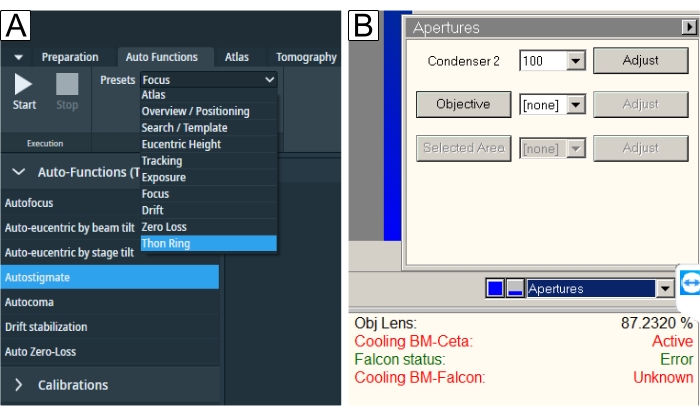
图 6:自动功能和光圈 。 (A) "自动功能"选项卡描述了"预设"下拉列表和"任务"选择。蓝色下划线是"自动柱头"(也用蓝色下划线)和"自动昏迷"所需的"Thon Ring"预设。应该为每个任务选择相应的预设,然后按 "开始" 按钮。(B) 光圈可在TEM用户界面中找到。执行自动功能后选择所需的"物镜光圈",并在打开光圈的情况下运行"自动柱头"。 请点击此处查看此图的大图。
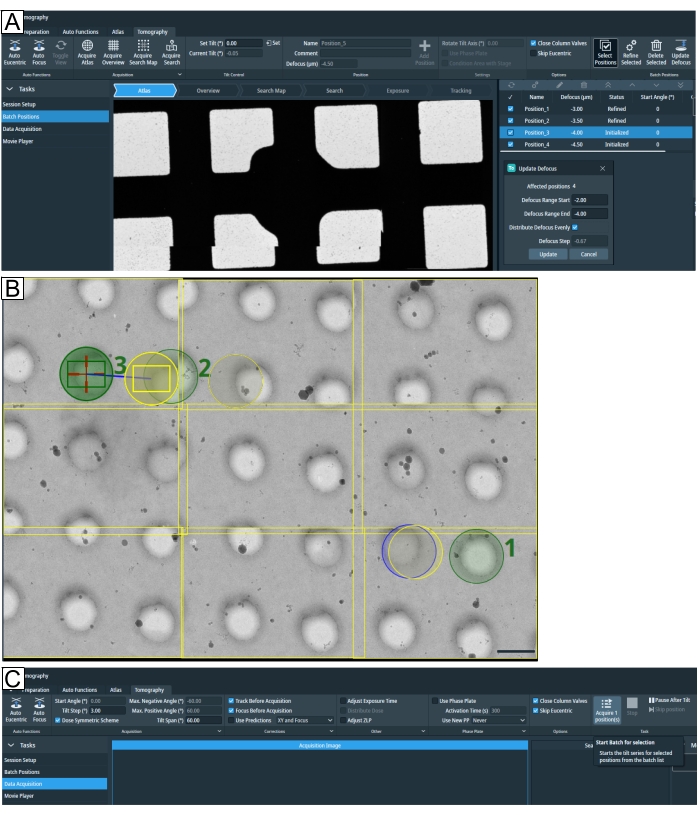
图 7:断层扫描选项卡概述。 图片显示了Tomo 5.8用户界面。(一)批次仓位。图中描述的最新功能:"获取搜索地图"选项;断层图位置显示在带有放大选项的图集视图中;右上角突出显示的是"选择职位";下面所有四个位置都已选择"更新散焦"参数。(B) 在搜索地图上选择三个位置,标记为 1、2 和 3。位置 1 在运行"全部优化"时不会造成问题。但是,如果在高目标密度设置中,例如选择薄片位置时,如位置 2 和位置 3 所示,则"全部优化"将在位置 2 的"曝光"区域上运行"跟踪"和"聚焦"例程,在获取断层扫描之前暴露目标。网格类型为 R1.2/1.3。比例尺 = 1.2 μm。 (C) 数据采集。现在可以单独获取"批次仓位"中设置的选定仓位。 请点击此处查看此图的大图。
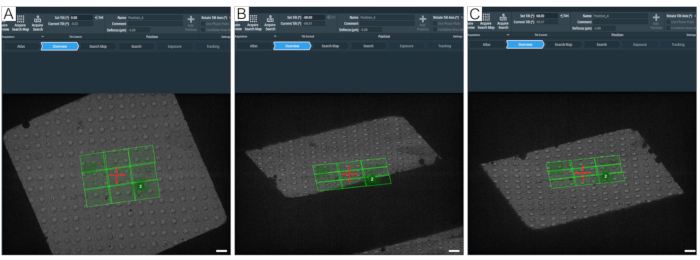
图 8:定义最大倾斜角度。 图描述了确定断层扫描采集的最大倾斜范围的分步方法。(A)0°概览和网格正方形中心的搜索地图。要测试倾斜范围,可以在断层扫描软件中使用"设置倾斜(°)"设置角度,方法是键入所需的值,然后按 Set 键。(B)载物台已倾斜到-60°,表明搜索地图的一角不会在-60°处完全获取。(C) 载物台已倾斜至60°。通过计算在±60°处消失的孔,可以了解网格正方形的倾斜范围。比例尺 = 2.5 μm。 请点击此处查看此图的大图。
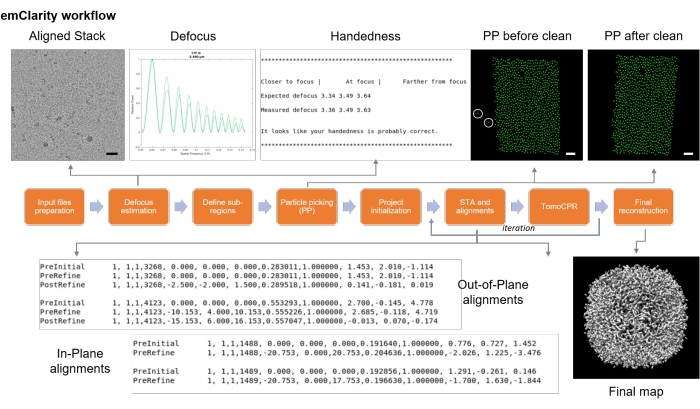
图 9:emClarity 流程图。流程图描述了冷冻断层扫描平均的各个步骤。比例尺 = 50 nm。请点击此处查看此图的大图。

图 10:使用 emClarity 进行模板匹配 。 (A)石墨烯涂层网格上载铁蛋白的典型显微照片。散焦:−3.430 μm。 (B) 模板搜索后与模型点叠加的断层图切片。(C)模型点的顶部和(D)侧面投影视图,表示单分散的载铁蛋白颗粒的单个平坦层。比例尺 = 50 nm。 请点击此处查看此图的大图。
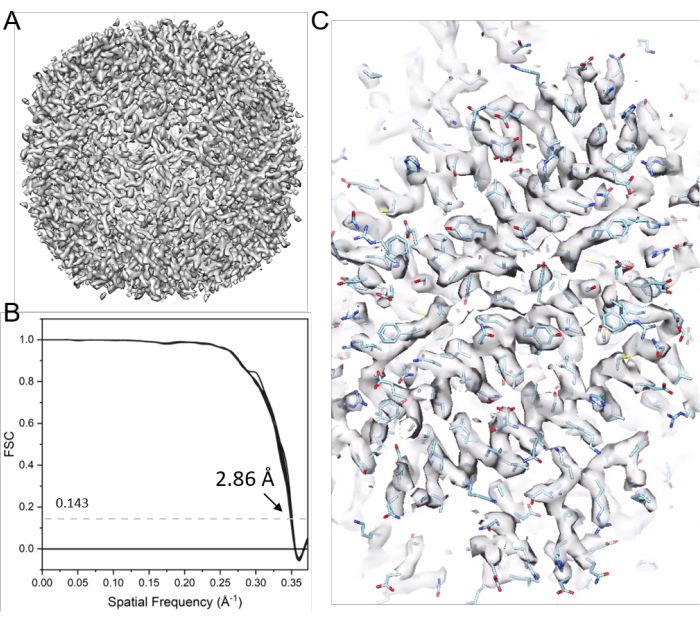
图 11:载铁蛋白的冷冻电子断层扫描 STA。 (A)21个周期的亚断层图对齐后的最终地图。(B)最终地图的傅里叶壳相关(FSC)图,报告的分辨率为2.86 Å,包含38个圆锥FSC。 (C)代表性密度图(拟合PDB模型6s6147)。请点击此处查看此图的大图。
| 样本 | 冷冻电子断层扫描型 | Å/px | 倾斜范围 (+/-) | 倾斜步长(°) | 散焦范围(μm) | 总剂量 (e-/Å2) | 分辨率 | 原始数据 | 参考 |
| 阿扑铁蛋白 | 纯化/分子 (STA) | 1.34 | 60 | 3 | 1.5 – 3.5 | 102 | 2.86 | EMPIAR-10787 | 17 & 本文 |
| HIV-1 堵嘴 | 纯化/分子 (STA) | 1.35 | 60 | 3 | 1.5 – 3.96 | 120 | 3.1 | EMPIAR-10164 | 17 |
| 核糖体 | 纯化/分子 (STA) | 2.1 | 60 | 3 | 2.2 – 4.3 | 120 | 7 | EMPIAR-10304 | 42 |
| SARS-CoV-2 峰值 | 薄片/分子 (STA) | 2.13 | 54 | 3 | 2 – 7 | 120 | 16 | EMPIAR-10753 | 45 |
| 神经元轴突结构式 | 蜂窝(超微结构) | 5.46 | 60 | 2 | 3.5 – 5 | 90 | 北达科他州 | EMPIAR-10922 | 47 |
表 1:几项冷冻电子断层扫描研究的收集参数。 针对从纯化或 原位 蛋白重建分子细节的研究与旨在解析和分割超微结构细胞特征的研究进行比较。
视频 1:在正常 EM 网格上对阿扑铁蛋白样品进行断层扫描,然后使用配备带有兼容滤光片的超高分辨率相机的冷冻透射电镜成像。 在电子断层扫描软件中,倾斜系列采用剂量对称方案获得,倾斜跨度为54°,总剂量为134 e-/A2 。比例尺 = 50 nm。 请点击这里下载此影片。
视频2:在EM网格上生长的初级神经元的断层图,然后用配备带有兼容过滤器的超高分辨率相机的冷冻透射电镜直接成像。倾斜系列采用剂量对称方案获得,在电子断层扫描软件中,倾斜跨度为60°,总剂量为120 e-/A2。比例尺 = 100 nm。请点击这里下载此影片。
视频3: EM网格上的蓝藻断层图,进行FIB铣削,然后用配备高速相机和兼容过滤器的冷冻透射电镜成像。倾斜系列采用剂量对称方案获得,在电子断层扫描软件中,倾斜跨度为50°,总剂量为120 e-/A2 。比例尺 = 87.2 nm。 请点击这里下载此影片。
补充文件 1:用于估计散焦的参数文件模板。请点击此处下载此文件。
补充表1:数据收集和显微镜设置细节。请按此下载此表格。
补充表 2:按执行顺序排列的命令列表。请按此下载此表格。
讨论
托莫5
断层扫描软件的工作流程描述重点介绍了(远程)批量断层扫描会话设置的一种潜在且最简化的方法。虽然该软件对初学者来说很容易,但一些初步的冷冻电镜经验和基本的断层扫描理解可以帮助进行设置。协议中突出显示了关键步骤,即使使用了不同的设置方法,也应有助于排除故障。该软件的进步将简化(远程)数据收集,并使冷冻电子断层扫描更容易被广泛的用户群使用。下面介绍了一些有助于解决常见问题的提示和技巧。
要讨论的一个重要点是网格的选择,因为当将试样倾斜到±60°时,高倾斜的网格条会遮挡视线(图8)。在TEM网格上,网格大小是指网格每单位长度的网格正方形数。网格数越大,每单位长度的网格正方形越多,网格正方形的密度越高,网格正方形越小,即 400 目网格的正方形比 200 目网格小。断层扫描网格的不错选择是 200 目或 300 目网格。 如图 8 所示,随着网格的倾斜,要收集的可用区域会减少。在±60°倾斜时,300目网格将具有一个小视野,可以在其上获取完整的断层扫描。200目网格的优点是较大的网格正方形使分子断层扫描设置更快,并且随着网格平方面积的增加,一个网格可能足以过夜收集。缺点是 200 目网格更脆弱,因此处理和裁剪需要更多的技巧。
此外,如果在EM网格上使用多孔支撑膜(见 材料表),则必须考虑孔间距,以设置相对于曝光区域的焦点和跟踪区域。理想情况下,所需放大倍率下的光束直径应足够小,以覆盖沿倾斜轴与曝光区域相邻的碳区域,以实现最佳和快速设置。这样,就可以获取每个孔中潜在的感兴趣区域。
由于软件的真心高度例程目前不那么强大,例如 serialEM 例程,以下提示可以解决此问题。如果使用真心高度预设确定真心高度失败,则可以改用概览预设并重新运行"通过载物台倾斜自动同心";如果以心为中心的高度远离 0,这可以解决问题。如果成功,则可以使用"真心高度"预设重新运行"通过载物台倾斜自动真心"以提高精度。如果失败,可以使用预设的真心高度运行"通过光束倾斜自动同心",然后重新运行"通过载物台倾斜自动同心",或者在TEM用户界面的"载物台"设置下手动设置通过"通过光束倾斜自动同心"合并的z高度。如果使用具有重复孔模式的网格,它们可能会阻止识别单个互相关峰。可以尝试将真心高度预设更改为较低的离焦偏移,例如-25μm和/或更短的曝光时间,以减少空穴图案的互相关。另一方面,使用蕾丝网格/薄片可能无法为强互相关峰提供足够的信号。可以尝试将真心高度预设更改为更大的离焦偏移,例如-75μm和/或延长曝光时间,以增强互相关峰。另一种选择是调整图像过滤器设置;它们可以在"准备"选项卡中找到。可以针对低(概览/网格平方)、中(共中心高度)和高放大倍率(跟踪/聚焦)设置调整滤镜设置的选项,以找到每个预设的最佳互相关峰值。所需的输入是一个图像,即 0° 和 5° 处,然后单击 比较 以比较两个图像。最长波长的推荐起始值为影像中比例尺的四分之一,最短波长的起始值为比例尺的四十分之一。如果峰不能可靠地确定,则可以优化设置,直到找到令人信服的峰。无需每次都重新获取图像;只需按"比较"就足够了。如果TOMO仍然无法自动找到真心高度,则可以使用手动共心高度校准。应该在"准备"选项卡中以概览放大倍率居中相当大的冰晶,然后转到TEM用户界面的"载物台控制",将alpha设置为-30°,并调整载物台z值以使用荧光屏图像重新居中晶体。在TEM用户界面中选择"高分辨率"和"高对比度"设置将使这变得简单(荧光屏幕窗口底部的按钮)。或者,如果可以访问具有实时模式的相机,则可用于确定真心高度;这将比荧光屏上更容易。
5.8 之前的 Tomo5 版本的最大限制是缺少中等放大倍率蒙太奇、缺少剂量对称方案以及与真心高度查找相关的问题。这些存在于 serialEM 中,这是一个具有快速开发和社区支持的免费软件,一个强大的以心为中心的高度程序,以及脚本选项,即定制的剂量对称方案。从 Tomo5 的 5.8 版本开始,通过实现设置共心高度接受标准的选项,解决了查找真心高度时最常遇到的问题,即围绕目标 z 值循环失败。但是,对于不同的网格和样品类型,强烈建议调整图像过滤器设置,以反映各个会话的独特成像条件,并提供最佳的互相关峰,以找到真心高度,并使焦点和跟踪区域在断层扫描采集期间可靠地工作。
总体而言,许多设施在大流行期间已迅速适应远程操作。Tomo5软件提供了一种易于访问和用户友好的断层扫描途径,非常适合远程操作。软件的进步无疑将继续使远程数据收集和断层扫描收集在社区中更加主流。
清晰
由于emClarity使用基于模板的粒子拾取方法,因此它需要感兴趣对象的模板。颗粒拾取(步骤2.6)非常敏感,是最终结构的关键。在平均和对齐(步骤 2.9)之前,必须确保仔细检查并手动删除误报。当模板不可用时,emClarity 可能不容易使用,但可以使用其他软件(例如 Dynamo37 和 PEET48)来创建初始模型。
对于异构样品,emClarity 配备了一种分类方法,使用户能够专注于不同尺度的特定特征。在分类之前运行几个对齐周期并在更高的分箱(例如箱 4 或箱 3)下运行它会很有帮助。
与第一个版本(V1.0)相比,该软件的最新版本(V1.5.3.11)进行了重大更新17。这些包括但不限于CTF估计期间的偏手性检查(步骤2.3);对齐对称性(CX、I、I2、O);计算每个粒子的3D采样函数(3DSF);切换到 MATLAB 2019a 以实现兼容性和稳定性;以及使用原始投影图像 (CISTEM) 进行重建。该软件将继续针对各种样品进行改进,最新的公告可以在线找到(见 材料表)。
披露声明
作者没有利益冲突。
致谢
我们感谢钻石光源访问和支持英国国家电子生物成像中心(eBIC)的冷冻电镜设施,该中心由威康信托基金会,MRC和BBRSC资助。我们还要感谢Andrew Howe收购了Apoferritin断层扫描(电影1),Ishika Kumar准备和获取了神经元断层扫描(电影2),以及Craig MacGregor-Chatwin的蓝藻层断层扫描(电影3)。
材料
| Name | Company | Catalog Number | Comments |
| Software | |||
| Tomography | Thermo Fisher Scientific | 5.9.0 | Internal terminology: Tomo5 in document |
| TEM server | Thermo Fisher Scientific | 7.10.1 | |
| TIA | Thermo Fisher Scientific | 5.10.1 | |
| DigitalMicrograph | Gatan | 3.44 | |
| emClarity | Open-Source software | 1.5.3.11 | Software for high-resolution cryo-electron tomography and subtomogram averaging |
| IMOD | Open-Source software | 4.11 | Modeling, display and image processing programs used for 3D reconstruction and modeling of microscopy images with a special emphasis on electron microscopy data |
| MotionCor2 | Free for academic use | 1.1.0 | A multi-GPU program that corrects beam-induced sample motion recorded on dose fractionated movie stacks |
| ETomo | Open-Source software | 4.11 | ETomo is an interface for running a subset of IMOD and PEET commands. |
| NoMachine | NoMachine, freeware | 7.9.2 | Remote desktop software |
| TeamViewer | TeamViewer AG | - | Remote access and remote control computer software |
| Materials | |||
| Quantifoil (holey support film) EM grids | Quantifoil | - | A flat film of carbon with pre-defined hole size, shape and arrangement |
| Instrumentation | |||
| Titan Krios microscope | Thermo Fisher Scientific | Titan Krios G2 | |
| K3 camera and GIB energy filter | Gatan | - | |
| Falcon 4 camera and Selectris X energy filter | Thermo Fisher Scientific | - | |
| Website | |||
| Website 1: https://github.com/bHimes/emClarity/ | - | - | Link to download the emClarity software package |
| Website 2: https://bio3d.colorado.edu/imod/ | - | - | Link to download IMOD |
| Website 3: https://github.com/ffyr2w/emClarity-tutorial | - | - | Link to the emClarity online tutorial |
| Website 4: https://emcore.ucsf.edu/ucsf-software | - | - | Link to download MotionCor2 |
| Website 5: https://github-wiki-see.page/m/bHimes/emClarity/wiki | - | - | Link to the newest announcements including updates and bug fixs for emClarity |
参考文献
- Bai, X. -. C., Fernandez, I. S., McMullan, G., Scheres, S. H. W. Ribosome structures to near-atomic resolution from thirty thousand cryo-EM particles. eLife. 2, 00461 (2013).
- Li, X., et al. Electron counting and beam-induced motion correction enable near-atomic-resolution single-particle cryo-EM. Nature Methods. 10 (6), 584-590 (2013).
- Nakane, T., et al. Single-particle cryo-EM at atomic resolution. Nature. 587 (7832), 152-156 (2020).
- Kato, T., et al. CryoTEM with a cold field emission gun that moves structural biology into a new stage. Microscopy and Microanalysis. 25, 998-999 (2019).
- Yip, K. M., Fischer, N., Paknia, E., Chari, A., Stark, H. Atomic-resolution protein structure determination by cryo-EM. Nature. 587 (7832), 157-161 (2020).
- Mastronarde, D. N. SerialEM: A program for automated tilt series acquisition on Tecnai microscopes using prediction of specimen position. Microscopy and Microanalysis. 9, 1182-1183 (2003).
- Schorb, M., Haberbosch, I., Hagen, W. J. H., Schwab, Y., Mastronarde, D. N. Software tools for automated transmission electron microscopy. Nature Methods. 16 (6), 471-477 (2019).
- Deng, Y., et al. Smart EPU: SPA getting intelligent. Microscopy and Microanalysis. 27 (1), 454-455 (2021).
- Carragher, B., et al. Leginon: An automated system for acquisition of images from vitreous ice specimens. Journal of Structural Biology. 132 (1), 33-45 (2000).
- Bell, J. M., Chen, M., Baldwin, P. R., Ludtke, S. J. High resolution single particle refinement in EMAN2.1. Methods. 100, 25-34 (2016).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. cryoSPARC: Algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods. 14 (3), 290-296 (2017).
- Grant, T., Rohou, A., Grigorieff, N. cisTEM, user-friendly software for single-particle image processing. eLife. 7, 35383 (2018).
- Zivanov, J., et al. New tools for automated high-resolution cryo-EM structure determination in RELION-3. eLife. 7, 42166 (2018).
- Kimanius, D., Dong, L., Sharov, G., Nakane, T., Scheres, S. H. W. New tools for automated cryo-EM single-particle analysis in RELION-4.0. Biochemical Journal. 478 (24), 4169-4185 (2021).
- Galaz-Montoya, J. G., Flanagan, J., Schmid, M. F., Ludtke, S. J. Single particle tomography in EMAN2. Journal of Structural Biology. 190 (3), 279-290 (2015).
- Bharat, T. A. M., Scheres, S. H. W. Resolving macromolecular structures from electron cryo-tomography data using subtomogram averaging in RELION. Nature protocols. 11 (11), 2054-2065 (2016).
- Himes, B. A., Zhang, P. emClarity: Software for high-resolution cryo-electron tomography and subtomogram averaging. Nature Methods. 15 (11), 955-961 (2018).
- Weissenberger, G., Henderikx, R. J. M., Peters, P. J. Understanding the invisible hands of sample preparation for cryo-EM. Nature Methods. 18 (5), 463-471 (2021).
- White, J. B. R., et al. Single particle cryo-electron microscopy: From sample to structure. Journal of Visualized Experiments. (171), e62415 (2021).
- Orlova, E. V., Saibil, H. R. Structural analysis of macromolecular assemblies by electron microscopy. Chemical Reviews. 111 (12), 7710-7748 (2011).
- Turk, M., Baumeister, W. The promise and the challenges of cryo-electron tomography. FEBS Letters. 594 (20), 3243-3261 (2020).
- Liu, Y. -. T., et al. Isotropic reconstruction of electron tomograms with deep learning. bioRxiv. , (2021).
- Briggs, J. A. G. Structural biology in situ-The potential of subtomogram averaging. Current Opinion in Structural Biology. 23 (2), 261-267 (2013).
- Grünewald, K., et al. Three-dimensional structure of herpes simplex virus from cryo-electron tomography. Science. 302 (5649), 1396-1398 (2003).
- Allegretti, M., et al. In-cell architecture of the nuclear pore and snapshots of its turnover. Nature. 586 (7831), 796-800 (2020).
- Wang, Z., et al. The molecular basis for sarcomere organization in vertebrate skeletal muscle. Cell. 184 (8), 2135-2150 (2021).
- Winey, M., Meehl, J. B., O'Toole, E. T., Giddings, T. H. Conventional transmission electron microscopy. Molecular Biology of the Cell. 25 (3), 319-323 (2014).
- Davies, K. M., et al. Macromolecular organization of ATP synthase and complex I in whole mitochondria. Proceedings of the National Academy of Sciences. 108 (34), 14121 (2011).
- Wagner, J., Schaffer, M., Fernández-Busnadiego, R. Cryo-electron tomography-The cell biology that came in from the cold. FEBS Letters. 591 (17), 2520-2533 (2017).
- Mahamid, J., et al. Visualizing the molecular sociology at the HeLa cell nuclear periphery. Science. 351 (6276), 969-972 (2016).
- Erdmann, P. S., et al. In situ cryo-electron tomography reveals gradient organization of ribosome biogenesis in intact nucleoli. Nature Communications. 12 (1), 5364 (2021).
- Tegunov, D., Xue, L., Dienemann, C., Cramer, P., Mahamid, J. Multi-particle cryo-EM refinement with M visualizes ribosome-antibiotic complex at 3.5 Å in cells. Nature Methods. 18 (2), 186-193 (2021).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152 (1), 36-51 (2005).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Heymann, J. B., Belnap, D. M. Bsoft: Image processing and molecular modeling for electron microscopy. Journal of Structural Biology. 157 (1), 3-18 (2007).
- Tang, G., et al. EMAN2: An extensible image processing suite for electron microscopy. Journal of Structural Biology. 157 (1), 38-46 (2007).
- Castaño-Díez, D., Kudryashev, M., Arheit, M., Stahlberg, H. Dynamo: A flexible, user-friendly development tool for subtomogram averaging of cryo-EM data in high-performance computing environments. Journal of Structural Biology. 178 (2), 139-151 (2012).
- Hrabe, T., et al. PyTom: A python-based toolbox for localization of macromolecules in cryo-electron tomograms and subtomogram analysis. Journal of Structural Biology. 178 (2), 177-188 (2012).
- Himes, B. A. emClarity. GitHub. , (2021).
- . The IMOD Home Page Available from: https://bio3d.colorado.edu/imod/ (2020)
- . GitHub: emClarity-tutorial Available from: https://github.com/ffyr2w/emClarity-tutorial (2021)
- Ni, T., et al. High-resolution in situ structure determination by cryo-electron tomography and subtomogram averaging using emClarity. Nature Protocols. 17 (2), 421-444 (2022).
- . MotionCor2 Available from: https://emcore.ucsf.edu/ucsf-software (2016)
- . Apoferritin from mouse at 1.84 angstrom resolution Available from: https://www.emdataresource.org/EMD-10101 (2022)
- Nedozralova, H., et al. In situ cryo-electron tomography reveals local cellular machineries for axon branch development. Journal of Cell Biology. 221 (4), 202106086 (2022).
- Mendonça, L., et al. Correlative multi-scale cryo-imaging unveils SARS-CoV-2 assembly and egress. Nature Communications. 12 (1), 4629 (2021).
- . Apoferritin from mouse at 1.84 angstrom resolution Available from: https://www.rcsb.org/structure/6s61 (2019)
- Nicastro, D., et al. The molecular architecture of axonemes revealed by cryoelectron tomography. Science. 313 (5789), 944-948 (2006).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。