Method Article
クライオ電子線トモグラフィー遠隔データ収集とサブトモグラム平均化
要約
本プロトコルは、Tomo5を用いた高分解能クライオ電子線トモグラフィー遠隔データ収集と、その後のデータ処理およびemClarityを用いたサブトモグラム平均化について記述する。アポフェリチンは、2.86 Åの分解能でクライオET構造を達成するための詳細なステップバイステップのプロセスを説明するための例として使用されています。
要約
クライオ電子線トモグラフィー(クライオET)は、特に直接電子検出器の導入、自動取得戦略の改善、クライオETを使用して電子顕微鏡が高解像度で画像化できるものの可能性を広げる分取技術、および新しいサブトモグラム平均化ソフトウェア。さらに、データ取得はますます合理化され、多くのユーザーにとってよりアクセスしやすくなっています。SARS-CoV-2のパンデミックは、世界中の多くの施設で、特に単粒子クライオEMのリモートクライオ電子顕微鏡(クライオEM)データ収集をさらに加速し、パンデミックの間、最先端の機器への中断のないユーザーアクセスを提供しました。Tomo5(3D電子線トモグラフィー用ソフトウェア)の最近の進歩により、リモートクライオETデータ収集は堅牢になり、世界中のどこからでも簡単に処理できるようになりました。この記事は、詳細なトラブルシューティングを伴う(リモート)クライオETデータ収集セッションのプロセスのための断層撮影ソフトウェアのデータ収集セットアップから始めて、詳細なウォークスルーを提供することを目的としています。(リモート)データ収集プロトコルは、例としてアポフェリチンを使用して、emClarityによるサブトモグラム平均化による原子分解能に近い構造決定のワークフローでさらに補完されます。
概要
極低温電子顕微鏡(クライオEM)は、ルネッサンス期を経験したことが広く知られており、構造生物学の中核的かつ中心的に有用なツールになるのを加速しています。直接電子検出器の開発と利用 1,2,3, 改良型顕微鏡・電子源 3,4,5, 自動化・スループットの向上 6,7,8,9, 単粒子解析における計算の進歩10,11,12,13、14および断層撮影15、16、17はすべて、部分的には、この技術の最近の成功に責任があります。これらの技術的推進力は、極低温およびネイティブ条件下で生物学的高分子構造を解明するクライオEMの能力を開発しました。容易に得られる解像度は、原子レベルで正確なモデリングに十分であり、この技術を構造生物学の分野の最前線にもたらしました。目的の生物学的標的を発現および精製するための還元主義的アプローチは、基礎生物学研究、創薬、およびトランスレーショナルサイエンスのための高分子結晶学(MX)で長い間成功していることが証明されています。同じアプローチで、クライオEMは高解像度MX研究と同等の結果を提供できるようになりました。構造生物学のクライオ電子顕微鏡分野における現在の大きな成功は、単粒子分析(SPA)と呼ばれ、精製されたタンパク質標本18の典型的な2D投影画像を取得して、生物学的高分子19の数千ビューを取得します。これらの画像には、(1)3D空間でのターゲットの向きを完全に表すさまざまなビューからの情報が含まれており、(2)オブジェクトのコンフォメーションの不均一性をキャプチャし、後で分離して調査できます。
生物学的サンプルのこれらの2D投影画像を取得するための代替アプローチは、その 場 で精製することなく、クライオ電子線トモグラフィー(cryo-ET)です。クライオETは、試料を機械的に回転させることにより、同じ物体の一連の画像を傾斜角度で撮影します。したがって、SPAで収集された2D突起は、関心のある分子の角度姿勢を表し、クライオETイメージング実験20の一部として本質的に収集される。次に、トモグラフィーチルトシリーズは、画像化された高分子複合体の3D表現を含む断層撮影に再構築されます。断層撮影データ収集の性質は、2D画像のコレクションから分子の完全な3D表現を達成するために、平均化への依存度をある程度減らします。ただし、現在のステージ設計により、標本は通常-60°から+60°に傾いており、断層撮影3D再構成で欠落したウェッジ21 の情報が残ります。
単一の断層撮影での3D再構成では、情報のくさびが欠落し、信号対雑音比が低くなります。個々の高分子をサブトモグラムとして抽出し、一緒に平均してこれに取り組むことができます。サブトモグラム内の各高分子が異なる方向で見つかる場合、欠落したウェッジはターゲットオブジェクトの各サブトモグラムで異なる方向を向いているため、多くのコピーにわたって平均化すると、欠落したウェッジによる情報が埋められます。画像処理における最近の開発はまた、意味のあるデータ22で欠けているくさびを埋めるために人工知能ニューラルネットワークを訓練することを試みた。この平均化プロセスは、単粒子分析での平均化の目標と同様に、信号対ノイズも増加するため、再構成の品質と分解能が向上します。目的の分子が対称性を有する場合、それも平均化中に定義および採用され、再構成分解能がさらに向上する。トモグラムからサブトモグラムのセットへの高分子の3Dボリュームの抽出とその後の処理は、サブトモグラム平均化(STA)23として知られています。各サブトモグラムが研究対象の分子の固有のコピーを表す場合、STAワークフローを使用して構造的不均一性を調べることができます。SPAワークフローで一般的に利用されるように、分類技術は、目的の複合体の立体配座状態を解剖するためにSTA中に採用され得る。クライオETでの高分解能再構成を可能にするSTAに加えて、このアプローチにより、この手法は、天然の細胞環境における高分子またはSPA24,25,26に従わないことが多い標的の構造メカニズムを調べるための強力なツールになります。
電子線トモグラフィーは、室温で細胞標本の3D微細構造を決定してきた長い歴史があります27。標本の物理的な傾きによるビューの取得は、細胞長スケールでの物体の3D再構成に十分な情報を提供し、細胞構造が平均化のための規則性を欠いている場合に特に重要です。細胞は、試料が電子透過性であるのに十分薄い細胞端でクライオETイメージングのために基板上に凍結することもできます。これらの条件下では、STAは、標本が電子透過性であるのに十分薄い場合でも、細胞環境における高分子構造を決定するために使用され得る28。ただし、クライオ相関光および電子顕微鏡(クライオCLEM)および集束イオンビームミリング(クライオ-FIB)などの追加の分取技術と組み合わせると、クライオETを使用して極低温条件下で細胞全体の内部をイメージングできます29。これは、細胞の微細構造を研究するクライオETの力とSTAの力を組み合わせて、高分子複合体の構造をその場で決定し、それらの細胞位置を特定し30、動的プロセスに関与する複合体のスナップショットを提供する31。細胞標本を画像化し、いくつかの研究でSTAを採用する技術の能力は、SPA32に匹敵する解像度であっても、その場で高分子構造を解決する技術の力を浮き彫りにしました。さらなる利点は、断層図30における最終的に分類された3D再構成によって表される巨大分子の元の位置の知識に見出される。したがって、高分子構造は細胞の超微細構造と相関させることができる。長さスケールにわたるこれらの観察は、おそらく、機能研究の文脈における構造メカニズムが細胞の変化と相関している可能性がある重要な発見につながるでしょう。
Cryo-ETおよびSTAは、分子、細胞、ラメラ断層撮影の3つの主要なワークフローでデータ収集を可能にします。精製高分子複合体の構造は、分子断層撮影法によるクライオETにより決定することができる。細胞が十分に薄い細胞環境におけるタンパク質構造を決定することは、細胞断層撮影として説明され得る。最近では、極低温ターゲティングとミリングの開発に伴い、これらの同じ技術をラメラ断層撮影ワークフローに適用して、天然環境の細胞深部にあるタンパク質構造を決定し、それらのタンパク質が観察される細胞状況を明らかにすることができます。利用可能なソフトウェアパッケージに応じて、そして最も重要なことに、試料の要件に応じて、異なるデータ収集戦略を使用することができます。精製タンパク質の銅TEMグリッド上の分子または非付着性サンプルは、通常、取り扱いが少なくて済むため、理想的な場合には平坦で損傷を受けません。電子断層撮影は、ホールカーボングリッドを横切って簡単に直列に設定でき、数十から数百のトモグラムを体系的に迅速に取得できます。タンパク質がグリッド上に豊富に存在する分子トモグラフィーサンプルをセットアップする最も簡単な方法は、Tomo5を使用することです(本研究で使用される3D電子線トモグラフィー用のソフトウェア、材料表を参照)。レギノン9やシリアルEM6などの他の断層撮影ソフトウェアも利用できます。これらは、データ収集のためのよりパーソナライズされたアプローチのためのより多くのセットアップオプションを提供しますが、より複雑であり、その結果、特にトモグラフィーに不慣れなユーザーやセッションにリモートでアクセスするユーザーにとっては、ナビゲートが難しくなる可能性があります。大規模で多様なユーザーベースを持つ施設の場合、Tomo5はリモート環境での操作とユーザーのトレーニングが簡単です。接着細胞の場合、グリッドは通常、より多くの取り扱いステップを必要とし、壊れやすい金グリッドを使用する必要があるため、取り扱いおよびデータ収集戦略におけるケアを改善する必要性が高まります。細胞の関心領域を見つけやすくし、高い傾斜角でグリッド自体からの閉塞を回避するために、より大きなメッシュサイズを使用することも有益ですが、本質的に壊れやすいという犠牲を払っています。ラメラサンプルの場合、サンプルの脆弱性はラメラの品質によって決定され、これは変動する可能性があります。これらの要因により、セットアップ時間と考慮事項が増加しますが、適応性と堅牢性の向上により、Tomo5はこのタイプのデータ収集に適しています。ただし、ワークフローごとに特殊なデータ収集シナリオが存在します。BISECTとPACE-tomo(どちらもSerialEMで実行)は、特に分子断層撮影において、断層撮影における断層撮影速度28を向上させるために、断層撮影中にスクリプト化されたビーム画像シフトの可能性を導入します。SerialEM 6,7,33の中倍率モンタージュ(MMM)は、すべてのワークフローで分子の特徴をよりよく識別し、正確にターゲットにすることができますが、執筆時点では、これらの特徴はTomo5で実装され始めています。
SPAと同様に、クライオETとSTAは、取得ソフトウェアの改善と、平均16、17、32、34、35、36、37、38のサブトモグラム用の豊富な利用可能なパッケージを通じて、ますますアクセスしやすくなっています。さらに、パンデミックの間、クライオEM装置へのリモートアクセスを可能にすることは、英国のダイヤモンド光源(DLS)にある電子バイオイメージングセンター(eBIC)などの国家施設の継続的な運用に不可欠になりました。これらの開発により、クライオETは、この技術を利用したい研究者にとって、よりアクセスしやすく堅牢になりました。データが取得されると、STAは、最大の分解能再構成を取得し、高分子の不均一性の分類を可能にするために、再発オブジェクトを分析するための不可欠なツールです。現在のプロトコルは、クライオETデータ収集用のクライオTEM顕微鏡の準備と、例としてアポフェリチンの分子断層撮影データセットでemClarityを使用してサブトモグラム平均化を実行する方法の詳細なウォークスルーを提供することを目的としています。emClarity(高分解能クライオ電子線トモグラフィーおよびサブトモグラム平均化のためのソフトウェア、資料表を参照)を使用するには、コマンドラインからスクリプトを実行する必要があるため、Linux/UNIXシステムに精通していることを前提としています。
リモート接続は、各機関/施設のネットワーク環境によって異なります。eBIC では、リモートシステムは、Diamond で使用される特定のネットワーク設定でリモートデータ収集を可能にするプログラムを使用します。顕微鏡へのリモート接続は、NoMachineとTeamViewerの2つのプラットフォームによって容易になります( 材料表を参照)。プログラムNoMachineを使用して、ユーザーはリモートのWindowsデスクトップにログオンできます。NoMachineが提供するリモートWindowsデスクトップは、顕微鏡と同じネットワーク上にあるため、顕微鏡の仮想サポートPCとして機能します。仮想サポートPCから、ユーザーはTeamViewerを介して顕微鏡に接続し、TUIおよびTomoを実行している顕微鏡PCに直接アクセスして制御します。
本プロトコルは、2つの部分(ステップ1およびステップ2)からなる。ステップ1では、Tomo5(3D電子線トモグラフィー用ソフトウェア)を使用したリモートクライオETデータ取得に焦点を当てています。(リモート)セッションのウォークスルーは、ますます高い倍率で画像をキャプチャし、最終的にユーザーが断層撮影データ収集のために標本領域をターゲットにするように断層撮影ソフトウェアに指示できるようにします。 図 1 は、このプロセスをまとめたものです。ステップ2では、emClarity(高分解能クライオ電子線トモグラフィーおよびサブトモグラム平均化のためのソフトウェア)を使用したクライオET STAデータ処理について詳しく説明します。 図 9 に、このプロセスをまとめます。
このプロトコルは、リモートの視聴者を対象としています。これは、顕微鏡で物理的にサンプルをロードしている人が直接位置合わせを行い、カメラのチューニングとゲインリファレンスの取得を処理したことを前提としています。このプロトコルでは、オートローダーを備えた3コンデンサーレンズシステムが想定されています。断層撮影ソフトウェアの詳細なガイドラインについては、製造元による詳細なマニュアルが、ソフトウェアがロードされたWindowsの[スタート]ボタンで入手できます。
プロトコル
この研究で使用されたソフトウェアパッケージは、部分的に無料で入手できます( 材料表を参照)。
1. Tomo5を用いたリモートクライオETデータ取得
- ソフトウェアがロードされていない場合は、TEMサーバーPCからこのソフトウェアを起動することから始めます(材料表を参照)。
- 初期設定を行い、 プリセットを選択して撮影条件を設定します。
- プリセットの[準備]タブで画像取得パラメータを調整して、セッションのセットアップを開始します(図2A、B)。
- 「概観倍率」をグリッドスクエアのサイズと線量に合わせて調整し、適切なカウントを取得します。
注: グリッド タイプに適した倍率が不明な場合は、グリッドを読み込んだ後にこの手順に戻ります。 - 「ユーセントリック高さ」と「検索倍率」は同じにすることができます。「トモグラフィー」 タブの検索をクリックして 位置を設定し、倍率が関心のある特徴を表示し、「露出」と「トラッキング/フォーカス」領域を視野に収めるのに十分であることを確認します(図2C)。
- 「露出倍率」を希望のターゲットピクセルサイズに設定します。これが不明な場合は、関心領域に合わせて倍率を目的の視野に調整して確立します。
- 「概観倍率」をグリッドスクエアのサイズと線量に合わせて調整し、適切なカウントを取得します。
- 画像取得(露出)のパラメーターを他のすべての高倍率プリセット(トラッキング、ドリフト、フォーカス、トーンリング、ゼロロス)にコピーします。これを行うには、[設定]を押して顕微鏡に[露出]を設定し、[取得]を押して、個別に選択した後、他のすべての高倍率に「露出設定」を取得します。
- 特に「フォーカス」と「トラッキング」の露光時間を調整して、60°の厚さ、つまり0°と60°の両方を考慮して、サンプルを通過して相互相関の強力な信号を提供します。
注:推定として、「露出」プリセットに等しい「トラッキング」と「フォーカス」の露出時間は、最初の測定値として適しています。「曝露」プリセットの線量計算例については、図3を参照してください。 「準備」タブで「露出」プリセットの計算された露出時間を設定します。
- 特に「フォーカス」と「トラッキング」の露光時間を調整して、60°の厚さ、つまり0°と60°の両方を考慮して、サンプルを通過して相互相関の強力な信号を提供します。
- 「ゼロロスプリセット」を調整して、より高い線量が必要なため、より明るく大きなスポットサイズを使用します(スポットサイズが大きいほど、数値は小さくなります)。
注意: このプリセットは、顕微鏡上にあるエネルギーフィルターのスリットを合わせるのに最適です。
- プリセットの[準備]タブで画像取得パラメータを調整して、セッションのセットアップを開始します(図2A、B)。
- 以下の手順に従ってアトラス収集を実行します。
- アトラスを収集するには、[アトラス]タブで [新しい セッション]をクリックし、セッションの設定を行い、ストレージパスと出力形式を入力して、[ 適用]を押します。 スクリーニング を選択し(図4)、取得するすべてのアトラスにチェックマークを付けます。 コルバルブを閉じる を選択して、顕微鏡を教師なしのままにします。これにより、選択したすべてのアトラスが収集された後にカラムバルブが閉じます。スクリーニングを開始するには、 スタート ボタンを押します。
- 「スクリーニング」(図4)の下の左側のパネルのグリッドをクリックして、単一または複数のアトラスでターゲットを調べ、マウスを左クリックしてドラッグして移動し、中央スクロールしてズームインおよびズームアウトします。ターゲット設定のグリッドを選択して選択し、ソフトウェア内から[サンプルのロード]をクリックします。
- 画像シフトキャリブレーションでは、引き続きアトラススクリーニングタブを使用して、ロードされたグリッド内を移動します。すべての倍率プリセットで認識できる識別可能な特徴、つまり、穴のエッジまたは別の認識可能な特徴と重なる氷晶を見つけます(図5)。マウスを右クリックしてその正方形に移動し、「ステージをグリッド正方形に移動」します。
注:画像シフトキャリブレーションを正しく実装するには、ステージがユーセントリックの高さにある必要があります。
- 画像シフトキャリブレーションを実行します。
- 「自動機能」タブ(図6)で、プリセットを「ユーセントリック高さ」に設定し、「ステージチルトによる自動ユーセントリック」に移動して 、[開始]を押します。ステータスウィンドウを監視して、ユーセントリックの高さが成功したことを確認し、正と負の傾き画像を監視します。それらは相関しなければなりません。
注:Tomoソフトウェアのバージョン5.8以降、ユーセントリック高度許容の基準を変更できます。デフォルトは0.25μmで、0.5〜0.8μmに設定すると余裕があります。値は顕微鏡の性能によって異なりますが、できるだけ小さくすることをお勧めします。- ユーセントリックの高さにあり、焦点が合っているときは、ステージをフィーチャの中央に配置します。「アトラス」プリセットを使用してプレビューを収集します。「準備」タブからすべての「プリセット」を設定します。その後、倍率をプレビュー >[概要>中央の特徴]に上げてから、「検索倍率」を繰り返し、最後に「露出倍率」を繰り返します。
- ターゲティング中にフィーチャが中央に配置されたままの場合は、画像シフトの調整をスキップします。それ以外の場合、画像シフトを調整するには、[準備]タブに移動し、[ 画像シフトの調整]を選択して、[ 開始 ]を押します(図5)。これにより、露出倍率を中心としたフィーチャに低倍率が繰り返し位置合わせされます。
注意: 最初のプリセット「露出」がこの段階で中央に配置されていない場合、再センタリングのために、ソフトウェアはこのステップにのみステージシフトを使用します。- 「露出」プリセットの [続行 ]を押します。次に表示される画像は「検索プリセット」になります。プリセット間の画像シフトを調整するには、「検索」プレビューで「露出」プレビューの中心がある場所を左ダブルクリックし、[ 再取得 ]を押します(図5)。[ 続行 ]をクリックして、次のプリセットのペアに移動します。
注意: このキャリブレーション中にアトラス倍率での画像シフトが適用された場合は、画像シフトキャリブレーションが完了した後にアトラスを再取得してください。目的のアトラスを選択し、トップパネルの[アトラス]タブで [選択をリセット ]を押します。確認して、そのアトラスの取得を再開します。
- 「露出」プリセットの [続行 ]を押します。次に表示される画像は「検索プリセット」になります。プリセット間の画像シフトを調整するには、「検索」プレビューで「露出」プレビューの中心がある場所を左ダブルクリックし、[ 再取得 ]を押します(図5)。[ 続行 ]をクリックして、次のプリセットのペアに移動します。
- 「自動機能」タブ(図6)で、プリセットを「ユーセントリック高さ」に設定し、「ステージチルトによる自動ユーセントリック」に移動して 、[開始]を押します。ステータスウィンドウを監視して、ユーセントリックの高さが成功したことを確認し、正と負の傾き画像を監視します。それらは相関しなければなりません。
- トモグラフィーのセットアップを実行します。
- 「トモグラフィー」タブでトモグラフィーデータ収集セットアップを作成します。
注意: 特に明記されていない限り、セットアップは完全に[断層撮影]タブで行われます。 - 新しいセッションを開始します。生物学的サンプルの「セッション設定」で、サンプルタイプとしてスラブライクを選択し、バッチと低用量を選択し、出力形式と保存フォルダーを選択し、オプションで電子メール受信者を追加して、[適用]を押します。
注意: 「バッチ位置」メニュー項目が使用可能になります(図7A)。現在ロードされているアトラスは自動的にインポートされます。「概要」と「検索画像」は、新しい画像を取得するまで取得して再訪することができます。また、バージョン5.8からは、「検索マップ取得」で3×3、4×4、5×5タイルの検索マップを取得できるようになり、ターゲットの検索や設定が容易になりました。それらは中倍率のモンタージュのバージョンに対応しています。 - ターゲットを設定するには、「アトラス矢印」に移動し、関心のある領域を見つけて、マウスの右クリックでポップアップするオプションを選択してそこに移動します。概観画像を撮影してユーセントリックの高さ調整に適した位置を確認し、 自動ユーセントリックを押します。これにより、ユーセントリックバイステージチルトルーチンが実行されます。次に、新しい概観図画像を再取得して、ユーセントリック高度を更新します。
注意: 位置がユーセントリックの高さにある場合、「オートフォーカス」ボタンは必要ありません。ユーセントリック高さの画像が焦点から大きく外れているように見える場合は、ユーセントリックの高さが正しくない可能性があり、やり直す必要があります(ユーセントリック高さのトラブルシューティングについては、説明のセクションを参照してください)。 - 最初の位置を設定する前に、「概要」プリセットを使用してターゲットを確認します。「概要」プリセットを選択し、ステージを±60°に傾けて、位置を設定できるグリッドバーからの距離を確認します(図8)。これを実行するには、目的の傾斜角度の値を「傾斜の設定」ウィンドウに入力します(図8A)。
- 四角い「概要」または取得した「地図検索」を調べ、関心領域に移動して、 検索を取得を押します。「検索」画像を調べます。関心領域が中央に配置されていない場合は、目的の位置を右クリックし、[ ここにステージを移動 して 画像を取得]を繰り返します。[傾斜 (°)] を 0.00 (またはステージが処理できるその他の開始角度) に設定して、断層図の開始角度を定義します。
- 最初の断層撮影では、目的の断層像命名 規則の名前を入力し 、各断層図に必要な焦点ぼけ値を設定するための焦点ぼ け を入力します。
- オプションで、Tomoバージョン5.8以降では、デフォーカス値を一度に体系的に変更できます。そのためには、[位置の選択]をクリックし、変更する位置を選択するか、チェックマークにチェックマークを付けてすべての 位置を選択し、[ デフォーカスの更新 ]をクリックしてパラメーターを調整します(図7A)。
- 「フォーカス」と「トラッキング」領域を調整します(図2C)。左クリックして、トラッキング領域とフォーカス領域(黄色と青色の円)をドラッグします。トラッキング/フォーカスエリアが(ほとんど)カーボンまたは別の適切なトラッキング機能、つまり関心領域と同様の機能上にあることを確認します。亀裂、氷の汚染、厚すぎる領域、空の穴を避けてください。
- 多くのトラッキング機能のないブランクカーボンを選択する場合は、焦点を合わせるのに十分な高さの線量を選択し、より高い傾斜でサンプルをゆっくりと燃焼するようにトラッキングすることにより、トモグラムの途中で領域が燃焼し始めることを確認してください。これは、追跡精度を維持するのに役立ちます。トラッキング/フォーカス領域が後の取得領域を公開していないことを確認します。
注意: 何が近くにあり、何がビームに入るかに注意してください。グリッド バーなど、トラッキングや露出に干渉するフィーチャを含む領域は避けてください。
- 多くのトラッキング機能のないブランクカーボンを選択する場合は、焦点を合わせるのに十分な高さの線量を選択し、より高い傾斜でサンプルをゆっくりと燃焼するようにトラッキングすることにより、トモグラムの途中で領域が燃焼し始めることを確認してください。これは、追跡精度を維持するのに役立ちます。トラッキング/フォーカス領域が後の取得領域を公開していないことを確認します。
- すべてのパラメータが設定され、目的の関心領域と「フォーカス」と「トラッキング」が定義されたら、[ 位置の追加 ]を押します。新しいターゲットについても繰り返します。
注:ユーセントリック高さが設定されたバッチ位置に対して正しく校正されていることを確認するには、いくつかの利用可能な方法があります。実行される断層撮影セッションのタイプ(分子、細胞、ラメラ)に基づいて1つを選択することをお勧めします。 - 分子断層撮影の場合、グリッドがかなり平坦で、ターゲット位置を含む各正方形の中心でユーセントリック高さが実行されていると仮定すると、「すべて絞り込み」(または位置が選択されている場合は選択を絞り込む、 図7A)手順をスキップします。Tomoがステージチルトによる自動ユーセントリックルーチンに苦労している場合は、おそらく スキップユーセントリックをクリックします。
- 細胞サンプルまたはラメラサンプルの場合、各バッチ位置は異なるz高さにある場合があります。「すべて絞り込み」を使用して注意するか、「ユーセントリックをスキップ」オプションにチェックを入れないでください。
注意: 「すべて絞り込み」は、「位置の選択」で設定または選択されたすべての断層撮影を繰り返します。ユーセントリック高さを調整し、データ収集の前に追跡とフォーカスを実行します。この手順では、次のトモグラムフォーカス/トラッキング領域(図7B)からのオーバーラップ露出を露出させる可能性があり、先に進む前に考慮する必要があります。 - 絞り込みに失敗した正方形を確認して再訪します。マウスの右ボタンで、 失敗した位置 をクリックしてオプションを表示します。ユーセントリックの高さが失敗した場合は、「ステージチルトによる自動ユーセントリック」(ステップ1.4.1)を見つけ、新しい「検索」画像を取得してz高さを更新し、「位置を追加」します。失敗した/以前に初期化された位置を削除します。クリックしてください ユーセントリックをスキップ 「すべて絞り込む」で行われたオプション。
注: [すべて絞り込み] が実行されていない場合は、[ユーセントリックをスキップ] をオンにしないでください。Tomo5は、各トモグラムが取得される前にユーセントリックリファインを実行します。このようにして、ユーセントリックリファインに失敗したポジションはスキップされます。
- 細胞サンプルまたはラメラサンプルの場合、各バッチ位置は異なるz高さにある場合があります。「すべて絞り込み」を使用して注意するか、「ユーセントリックをスキップ」オプションにチェックを入れないでください。
- 「トモグラフィー」タブでトモグラフィーデータ収集セットアップを作成します。
- 以下の手順に従って自動機能を実行します。
- 設定を確認して、[自動機能]タブでアトラスをカーボンの領域に移動して、アライメントを実行します。その領域をユーセントリックの高さにし、以下に示すように配置の順序に従います。
- 「ユーセントリック高さ」プリセットを使用して、ステージチルトによって 自動ユーセントリック を実行します。
- 「フォーカス」プリセットを使用して オートフォーカスルーチン を実行します。
- 「トーンリング」プリセットを使用して オートスティグメイト ルーチンを実行します。
- 「トーンリング」プリセットを使用して オートコマ ルーチンを実行します。
- 目的の対物レンズアパーチャ(図6B)、おそらく100μmアパーチャを挿入します。
- 絞り挿入後に オートスティグメイト ルーチンを繰り返します。
注:ピクセルサイズが約3 Å以下の解像度の倍率では、オートコマルーチンが失敗する可能性があります。この場合、TEMユーザーインターフェイスの直接位置合わせの下にあるTomo回転中心を確認して位置合わせします。
- 自動ゼロロスセンタリングルーチンがサンプルで機能することを確認してください。適度に高用量でプリセットされた「ゼロロス」(ステップ1.2.3)を使用すると、自動機能のルーチンが成功する可能性が高くなります。
- 設定を確認して、[自動機能]タブでアトラスをカーボンの領域に移動して、アライメントを実行します。その領域をユーセントリックの高さにし、以下に示すように配置の順序に従います。
- データ収集を実行します。
- 「トモグラフィー」タブで自動取得を開始するには、 データ収集 スラブアウトを選択し、目的のパラメータを設定します(図7C)。データ取得パラメータの設定:チルトステップ(°)、最大正の角度(°)、最大負の角度(°)、トラッキングスキーム(推奨: 取得前にトラック/フォーカスを選択)。ユーセントリック高さスキームの クローズコラムバルブ を選択します。
注意: めったに使用されないパラメータは、「露出時間の調整」、「ZLPの調整」、「位相板の使用」、「傾斜後に一時停止」です。- STAを対象としていない断層撮影の 露出時間の調整 を使用して、傾斜が大きいときに指定した比率で露光時間を増やします。
- [ZLP の調整] オプションは、周期性の設定に関係なく、各トモグラムの後に ZLP リファインメントを実行します。それは遅く、明白な理由なしに失敗することがあり、そのポジション取得をスキップします。ただし、非常に狭いスリット幅、つまりSelectris(X)フィルターで3〜5 eVの場合に役立ちます。
- フェーズプレートの使用は 、エネルギーフィルター付きのDEDの導入以来、めったに使用されていません。
- [傾いた後に一時停止] は集録を一時停止しますが、ソフトウェアの変更は許可しません。
- データ収集パラメータ(「準備」タブ)を再確認し、線量計算と希望の傾斜スキームに同意し、「曝露」プリセットで 分数(Nr.) を指定してから、取得を開始します。
注:分数の数は、サンプル、取得パラメータ、および計画された後処理ステップ、つまりSTAと形態素解析によって異なり、動き補正とCTF推定が十分な信号を得る値に保つ必要があります。4〜10フラクションの範囲は適切な推定値であり、4はより厚いサンプルに適しており、10はより薄い分子サンプルに適しています。- 次に、[ 開始 ]をクリックしてデータ収集を開始し、最初の断層撮影を監視して、断層撮影が意図したとおりに取得されていることを確認します。
注意: 最近のトモグラフィーソフトウェアバージョンには、取得したトモグラムを視聴できる[トモグラフィー]タブに追加のムービープレーヤースラブアウトがあります。ロードプロセス中はしばらくお待ちください。
- 次に、[ 開始 ]をクリックしてデータ収集を開始し、最初の断層撮影を監視して、断層撮影が意図したとおりに取得されていることを確認します。
- 「トモグラフィー」タブで自動取得を開始するには、 データ収集 スラブアウトを選択し、目的のパラメータを設定します(図7C)。データ取得パラメータの設定:チルトステップ(°)、最大正の角度(°)、最大負の角度(°)、トラッキングスキーム(推奨: 取得前にトラック/フォーカスを選択)。ユーセントリック高さスキームの クローズコラムバルブ を選択します。
2. エムクラリティを用いたアポフェリチンのクライオET STA
注:ここでは、EMクラリティソフトウェア17 を使用して、STAによるクライオET構造決定を説明する。 図 9 はこのプロセスをまとめたものです。例として、6つのチルトシリーズのアポフェリチン(EMPIAR-10787)を取り上げました。八面体対称性が適用され、最終的なマップの解像度は2.86 Åで、わずか4,800個の粒子から得られ、ナイキスト周波数(2.68 Å)に近くなりました。
- emClarityソフトウェアパッケージ39 がダウンロードされ、インストールされていることを確認します( 材料表を参照)。ソフトウェア処理用のIMODをインストールする40.
メモ: Linux コマンドの基本的な知識が必要です。より詳細なチュートリアルは参考文献41にあり、リボソームデータセット(EMPIAR-10304)を例として取り上げ、段階的な手順を説明しています。さまざまな種類のタンパク質サンプルについて、以前に公開されたレポートも利用できます42。 - 入力ファイルとディレクトリを準備します。
注:生のフレームは、MotionCor2(パッチ5 x 5)43 によってモーション補正されました( 材料の表を参照)。動き補正された画像を積み重ねて、newstack(IMOD)を使用してチルトシリーズを生成し、Etomo( 材料表を参照)を使用してパッチトラッキングと手動で位置合わせし、続いてemClarityを使用しました。より良い位置合わせを得るには、Etomoでオフセットと傾き補正を実行することをお勧めします。6つのチルトシリーズはTS1からTS6に改名されています。以下は、例としての最初のチルトシリーズ(TS1)の手順とコマンドです。- プロジェクト フォルダーを確立します。
注: ソフトウェアの最新バージョンは 1.5.3.11 です。関連するすべてのソフトウェア ログは、ローカル プロジェクト フォルダー (.../logFile/emClarity.logfile) にあります。 - プロジェクト フォルダーの下に、別の新しいフォルダー "fixedStacks" を作成し、TS1.fixed、TS1.xf、および TS1.tlt の入力ファイルを準備します。
注:TS1.fixed:オリジナルのチルトシリーズ、Etomoでは TS1.st としても知られています。TS1.xf:このファイルには、Etomoチルタライン後の変換座標が含まれています。TS1.tlt:このファイルには傾斜角が含まれています。
- プロジェクト フォルダーを確立します。
- 以下の手順に従って、焦点ぼけを推定します。
- 焦点ぼけを計算します。 補足表1に続く顕微鏡およびイメージングパラメータでパラメータファイルを更新します。パラメーター ファイルをプロジェクト フォルダーにコピーし、その名前を param_ctf.m に変更して、 emClarity ctf 推定値 param_ctf.m TS1 を実行します。
注: パラメータ ファイル テンプレートは、 補足ファイル 1 またはローカル ソフトウェア インストール ディレクトリ (.../emClarity_1.5.3.11/docs/exampleParametersAndRunScript/) にあります。
- 焦点ぼけを計算します。 補足表1に続く顕微鏡およびイメージングパラメータでパラメータファイルを更新します。パラメーター ファイルをプロジェクト フォルダーにコピーし、その名前を param_ctf.m に変更して、 emClarity ctf 推定値 param_ctf.m TS1 を実行します。
- 各スタックのCTF推定結果を確認します。
- 3dmodを使用して位置合わせされたスタック(aliStacks/TS1_ali1.fixedなど)をチェックし、基準ビーズが正しく消去されていることを確認します。
- ログファイル/emClarity.logファイル(図9)にある利き手が正しいことをログファイルで報告していることを確認します。
- デフォーカス値 (fixedStacks/ctf/TS_ali1_psRadial_1.pdf など) を確認し、理論上の CTF 推定値と一致することを確認します。
- サブ領域を定義します。
- ビン断層撮影を生成します。プロジェクト フォルダーで、 sh recScript2.sh -1 を実行します。
注:各スタックの小さい(サイズの)トモグラムは、新しいフォルダ「bin10」に保存されます。プロセスを高速化するために、1つまたは複数のサブ領域の座標をbin10断層画像で定義することができます。スクリプト ファイルは、ローカル ソフトウェア インストール ディレクトリ (.../emClarity_1.5.3.11/docs/) にあります。 - 境界を決定するには、x 最小値、x 最大値、y最小値、y最大値、Z最小値、Z最大値の 6 つのポイントを選択して、1 つのサブ領域を作成します。フォルダ "bin10"の下で、次のコマンドを実行します:3dmod TS1_bin10.rec。
注: 1 つの傾斜系列に 4 つのサブ領域を作成する場合は、6 × 4 = 24 ポイントを選択します。各スタックには、フォルダー "bin10" の下に 1 つのモデル ファイル (*.mod) が必要です。 - モデル ファイルを emClarity 形式に変換します。これにより、新しいフォルダーの偵察が作成されます。このフォルダーの下に各サブ領域のすべての座標を格納します。プロジェクト フォルダーで、 sh recScript2.sh TS1 を実行します。
- ビン断層撮影を生成します。プロジェクト フォルダーで、 sh recScript2.sh -1 を実行します。
- パーティクルを選択します。
- 粒子をピッキングするためのアポフェリチンテンプレートを見つけます。電子顕微鏡データバンク (EMD-10101)44 からテンプレートをダウンロードします。テンプレートのピクセル サイズが生データのピクセル サイズと一致していることを確認します。プロジェクト フォルダーで、 emClarity rescale ApoF.mrc ApoF_Template_rescale.mrc 3.60 1.34 cpu を実行します。
- 粒子ピッキング用のCTF補正断層撮影を生成します。プロジェクトフォルダで、 emClarity ctf 3d param_ts.m templateSearchを実行します。
- サブ領域ごとにパーティクル ピッキングを実行します。アポフェリチンデータセットについては、bin6でテンプレート検索を実行します。Tmp_angleSearchパラメータ(θアウト、Δアウト、θイン、Δイン)を変更して、面内または面外検索の角度範囲と間隔を度単位で決定します。プロジェクト フォルダーで、emClarity templateSearch param_ts.m TS1 1 ApoF_Template_rescale.mrc O 1 を実行します。
注: この手順では、フォルダー "convmap_wedge_Type2_bin6" が生成されます。このフォルダの下にあるcsvファイル(TS1_1_bin6.csvなど)には、選択されたパーティクルに関するすべての情報が含まれています。 - 3dmodを使用して誤ったパーティクルを削除します。フォルダ "convmap_wedge_Type2_bin6"の下で、次のコマンドを実行します: 3dmod ../cache/TS1_1_bin6.rec TS1_1_bin6.mod.
注意: これらの領域のカーボンエッジや氷の汚染の隣に間違った粒子を見つけるのはよくあることです。間違ったポイントを右クリックし、キーボードの バックスペース を押して削除します。この手順では、ほとんどの誤検知ポイントが削除されていることを確認します(図10)。 - emClarityが次の手順でconvmapの下のサブリージョン情報を見つけるので、フォルダ名「convmap_wedge_Type2_bin6」を「convmap」に変更します。
- プロジェクトを初期化します。プロジェクトフォルダで、 emClarity init param0.mを実行して、 emClarity、ApoF.mat用のデータベースを作成します。
- STAとアライメントの前に断層撮影の再構成を行います。bin4でCTF補正されたサブ領域断層撮影を生成するには、 emClarity ctf 3d param0.mを実行します。
注:CTF補正された断層撮影(たとえば、cache/TS1_1_bin4.rec) は、新しいフォルダ「キャッシュ」に生成されます。 - STA とアライメントを実行します。
- bin4から始まるCTF補正サブトモグラムを使用して平均化を実行します。プロジェクト フォルダーで、 emClarity avg param0.m 0 RawAlignment を実行します。
- アライメントを続けて実行します: emClarity alignRaw param0.m 0。
注: 平均化ステップでは、このステップで emClarity がパーティクルを整列させるために使用する参照が生成されます。Raw_angleSearchパラメータ(θout、Δout、θin、Δin)を変更して、各サイクルの角度範囲とステップサイズを設定できます。ほとんどの不正なパーティクルがデータベースから削除されるため(ステップ 2.6.4)、アライメントのこれらの角度設定は比較的小さな角度から開始できます。 - Raw_angleSearchパラメーターを更新し、さらにいくつかのサイクルを実行します (ステップ 2.9.1 および 2.9.2)。プロセスを高速化するには、面内アライメントと面外アライメントを別々に実行します。
注意: ビニングごとに、複数のサイクルを実行し、角度設定を徐々に減らすことをお勧めします。ApoFデータセットについては、ビン4でさらに5サイクルが実行され、詳細は補足表2に記載されています。cycle001 の場合は、プロジェクト フォルダーで、emClarity avg param1.m 1 RawAlignment を実行し、続いて emClarity alignRaw param1.m 1 を実行します。 - 重なった粒子をきれいにします。プロジェクト フォルダーで、 emClarity removeDuplicates param5.m 5 を実行します。
- トモCPRでチルトシリーズリファインを実行します。
注: この手順はオプションです。- tomoCPR を実行して、スタック ジオメトリを調整します。プロジェクトフォルダで、 emClarity tomoCPR param5.m 5を実行します。
- 新しく位置合わせされたスタックを生成し、ジオメトリ ファイルを更新します。プロジェクトフォルダで、 emClarity ctf update param6.mを実行します。
注: 新しいジオメトリ ファイル (fixedStacks/ctf/TS1_ali2_ctf.tlt など) と新しく位置合わせされたスタック (aliStacks/TS1_ali2.fixed など) が正しく生成されていることを確認します。 - bin2に新しいサブトモグラムを作成します。プロジェクト フォルダーで、 emClarity ctf 3d param6.m を実行します。
注意: これに続いて、bin2での平均化と位置合わせ(ステップ2.9.1、2.9.2、および2.9.3)、重複クリーニング(ステップ2.9.4)、およびオプションのtomoCPR(ステップ2.10)の数サイクルが続きます。高い対称性のために、bin2でさらに5サイクルをアポフェリチンについて行った。各サイクルのRaw_angleSearchパラメータを更新します。
- 最終的な再構築を実行します。
- bin1 および tomoCPR で数サイクル実行を続けます (ステップ 2.9 およびステップ 2.10)。アポフェリチンデータセットに対して、bin1でさらに10サイクルを実行しました。コマンドおよびパラメーターの詳細については、 表 2 を参照してください。
- 2 つのハーフ データセットを組み合わせて最終的な再構築を実行します。プロジェクト フォルダーで、emClarity avg param21.m 21 RawAlignment を実行し、続いて emClarity avg param21.m 21 FinalAlignment を実行します。
注: 最終的なマップ(たとえば、B 係数が 10、cycle021_ApoF_class0_final_bFact-10.mrc) が生成されます ( 図 11)。
結果
細胞サンプルとラメラサンプルの場合、データ収集戦略はサンプルとイメージング研究の目標に大きく依存します(図1)。ターゲティングアプローチは、分子ターゲットが in situ であるか、分子ターゲットを含む高分解能再構成標本用の精製高分子複合体から調製されているかによって異なります。ホール状(炭素)グリッド上にビトリファイされた精製複合体の場合、ターゲティングは、(炭素)支持フィルムの孔内のイメージングに基づいて簡単に行うことができます。 in situ 作業の場合、ターゲティングアプローチでは、相関データまたは既知の低倍率の細胞ランドマークに基づく分子実体の位置に関する知識が必要です。セルラーランドマークは、概観画像を撮影するときに理想的に識別可能であり、関心のある領域を大まかにローカライズするのに十分であれば、検索画像でターゲットの身元を確認する簡単な方法を提供できます。ただし、観察可能なイベントがまれである場合は、ターゲットが正しいと認定するために中倍率の検索画像が必要になる場合があります。検索マップは、検索画像の中倍率モンタージュであるため、ターゲットの検索をはるかに簡単にすることができ、関心のあるフィーチャが見える倍率で取得できます。その後、検索マップをスクリーニングして、ターゲットバッチ位置を見つけて設定できます。微細構造再構築のための細胞の特徴を含む標本の場合、ターゲティングアプローチは似ていますが、さまざまな倍率での細胞イベントの可視性とサンプル中のその有病率に等しく依存します。
データ取得戦略も考慮する必要があります。いずれの場合も、研究の目標は、データの収集方法を大きく左右します。細胞の超微細構造の再構築には、低倍率(20-5 Å/px)と広い視野が適切かもしれませんが、分子または高解像度の詳細(5-1 Å/px)を再構築するには高倍率が必要です。理想的な条件下で1.5 Å/pxで収集されたデータセットは、3.0 Å/pxのナイキスト周波数でのみ物理的に再構成を生成できます。しかし、実際には、試料の厚さ、サイズ、不均一性を含むがこれらに限定されない多くの要因がすべて、得られる再構成品質に影響を与えます。正確なイメージングパラメータは、研究の目標に基づく倍率と、十分な情報を含む視野とのバランスもとっています。この記事では、2.86 Åに達するサブトモグラム平均化症例を提示しますが、異なるアウトカム17、42、45、45、46を対象とした研究に関連する収集パラメータを説明するために、追加の研究17、42、43、44、45を表1に示します。
クライオETのターゲティングワークフローとデータ取得体制が確立されると、さまざまな種類のサンプルのデータ収集が可能になります。ここでは、アポフェリチンなどの分子サンプル(動画1)、薄い細胞突起(動画2)、厚い細胞検体のFIBミルドラメラ(動画3)など、さまざまな標本の代表的な断層撮影を紹介します。
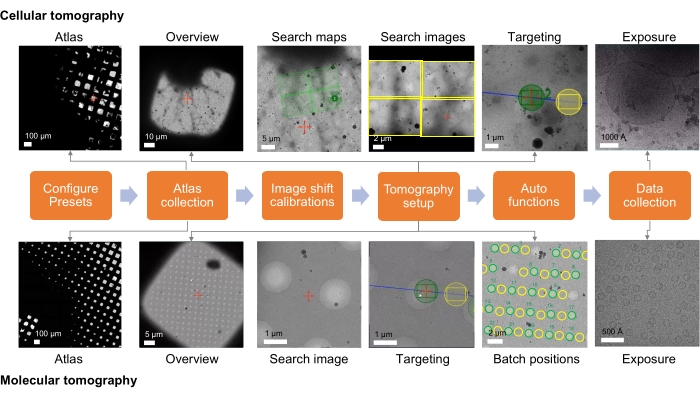
図1:トモグラフィーワークフロー設定の概要。 プロトコルに記載されているクライオETイメージングワークフローをフローチャートとして示します。取得が期待される画像は、細胞および分子ワークフローのために示されています。提示された命名はTomo5規則に従いますが、ほとんどの断層撮影取得ソフトウェアはこれらの画像を収集するための共通の原則を共有しています。 この図の拡大版を表示するには、ここをクリックしてください。
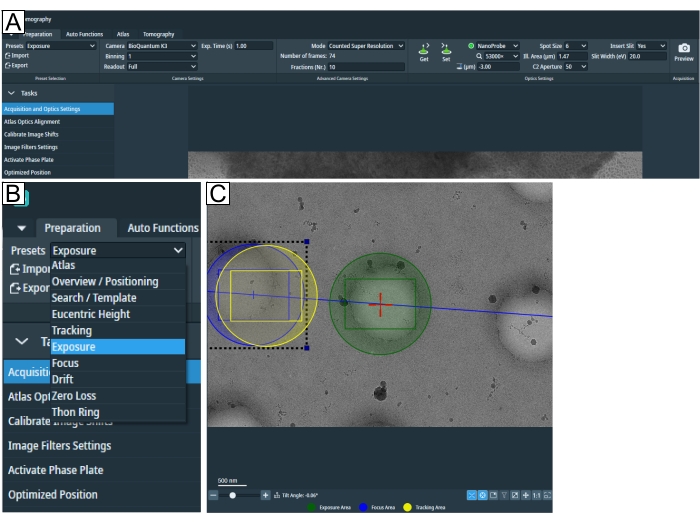
図2:準備タブと検索プリセット条件 。 (A) タブ全体の概要図。このタブでは撮影条件のプリセットが設定され、「タスク」ドロップダウンには「画像シフトのキャリブレーション」と「画像フィルター設定」があります。(B)「プリセット」ドロップダウンを拡大し、個々の撮影条件設定に合わせて各プリセットを選択できます。(C)露出と「フォーカスとトラッキング」領域の両方を視野に収めるのに十分な倍率を描いた画像。 この図の拡大版を表示するには、ここをクリックしてください。
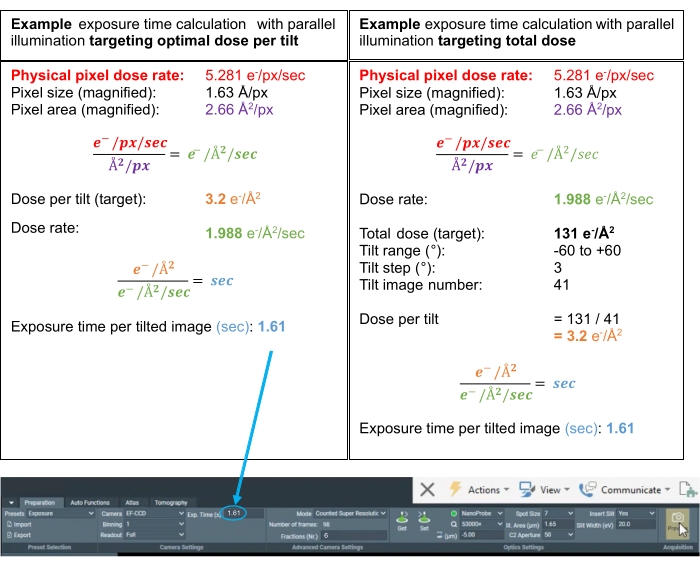
図3:線量計算。 線量率が真空上で測定された可能な断層図取得スキームの線量計算の例。2つの計算は、傾斜ごとの最適線量またはフル傾斜シリーズの最適な総線量を対象とするかどうかにかかわらず、各傾斜の曝露時間(秒)を決定する。サブトモグラム平均化では、3.0〜3.5 e-/Å2の範囲の傾斜画像あたりの最適線量をターゲットにするのが一般的な方法です。どちらの場合も、「分数(Nr.)」は6に設定され、モーション補正を実行するのに十分な信号を得るために、チルトのムービーフレームあたり~0.5 e-/Å2 の線量を達成します。 この図の拡大版を表示するには、ここをクリックしてください。
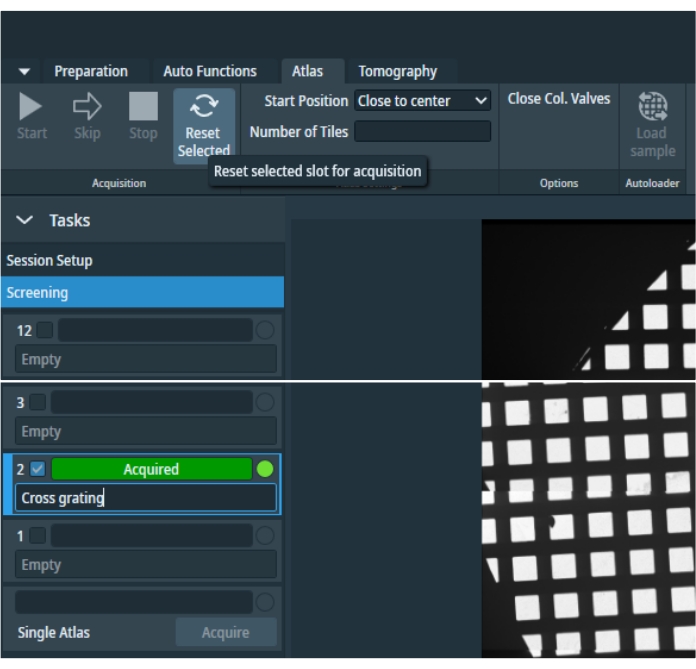
図4:アトラスタブ。 「アトラス」タブの概要画像。画像は、空のグリッド位置スペースを避けるためにトリミングされています。「タスク」メニューには、インベントリされたすべてのグリッドを個別に選択するためのセッション設定設定とグリッド選択スペース、およびカセットがオートローダーから取り外された後に単一のアトラスを取得するオプションがあります。選択したグリッドをリセットしてから再取得できます。 この図の拡大版を表示するには、ここをクリックしてください。
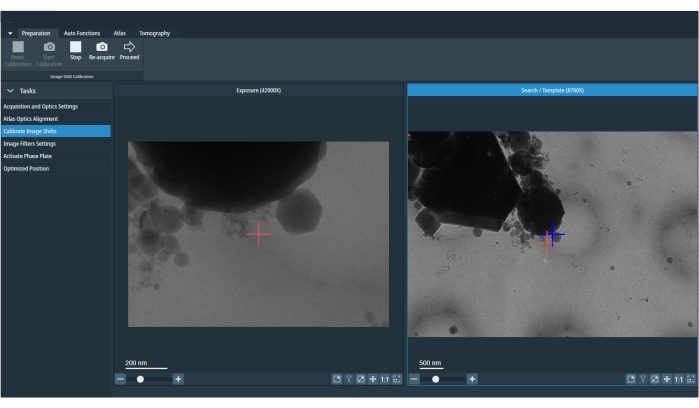
図5:キャリブレーションされた画像シフト。 画像は露出と検索の画像を表しています。検索画像の赤い十字は、露出と検索の間のオフセットを補正するためのシフトマーカーです。セッション開始時または撮像条件プリセットの変更後に、画像シフトキャリブレーションをやり直す必要があります。 この図の拡大版を表示するには、ここをクリックしてください。
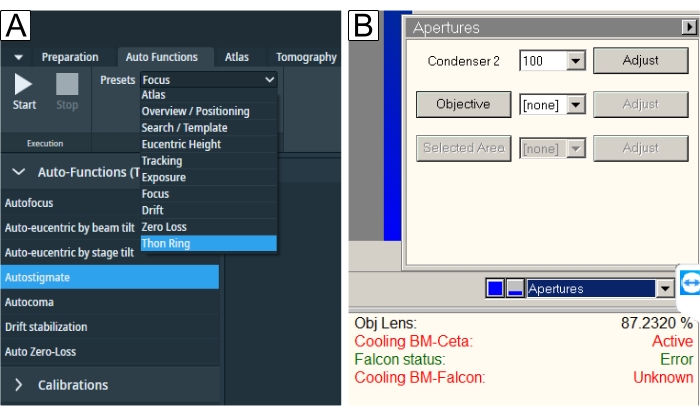
図6:自動機能と絞り 。 (A)「自動機能」タブには、「プリセット」ドロップダウンと「タスク」の選択が表示されます。青で下線が引かれているのは、「オートスティグメイト」(これも青で下線が引かれている)と「オートコマ」に必要な「トーンリング」プリセットです。各タスクのそれぞれのプリセットを選択してから、[ スタート ]ボタンを押す必要があります。(B)開口部はTEMユーザーインターフェイスにあります。自動機能を実行した後、目的の「対物レンズ絞り」を選択し、絞りを入れて「自動スティグメイト」を実行します。 この図の拡大版を表示するには、ここをクリックしてください。
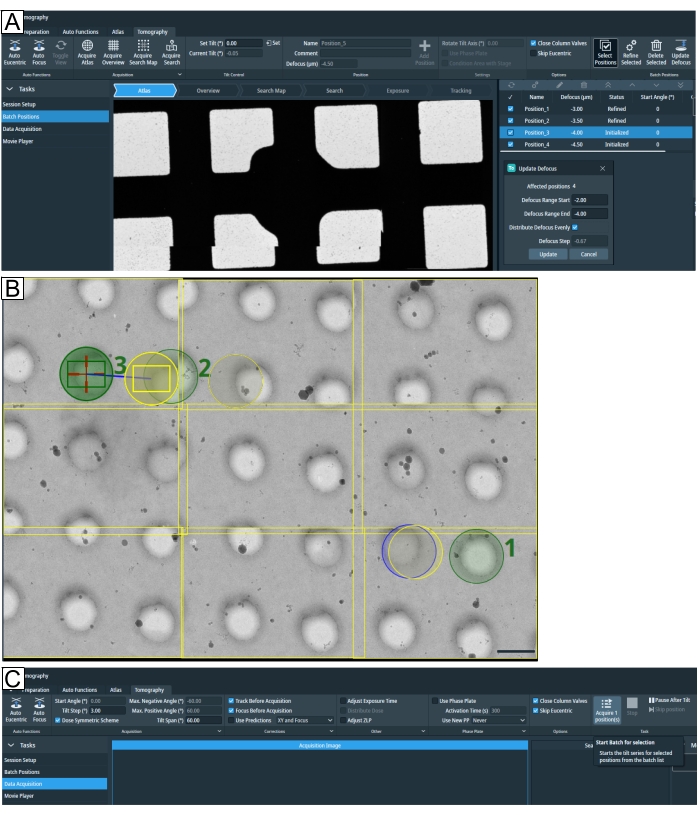
図7:トモグラフィータブの概要。 画像はTomo 5.8のユーザーインターフェースです。(A) バッチ位置。画像に示されている最新の機能:「検索マップの取得」オプション。トモグラムの位置は、ズームインオプション付きのアトラスビューに表示されます。右上で強調表示されているのは「ポジションの選択」です。以下の4つの位置すべてが「デフォーカスの更新」パラメータに選択されています。(B)検索マップ上で、1、2、3というラベルの付いた3つの位置が選択されます。位置 1 は、「すべて絞り込み」を実行しても問題になりません。ただし、位置2と位置3に示されているように、ラメラ位置が選択されている場合など、ターゲット密度が高い設定の場合、「すべて絞り込み」は位置2の「露出」領域で「トラッキング」および「フォーカス」ルーチンを実行し、トモグラムが取得される前にターゲットを露光します。グリッドタイプはR1.2 / 1.3です。スケールバー = 1.2 μm. (C)データ収集。「バッチポジション」で設定した選択ポジションを個別に取得できるようになりました。 この図の拡大版を表示するには、ここをクリックしてください。
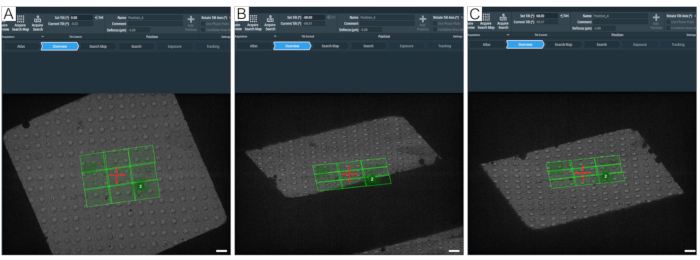
図8:最大傾斜角の定義。 図は、断層撮影の最大傾斜範囲を決定するための段階的なアプローチを示しています。(A)0°の概観図とグリッド正方形の中央にある検索マップ。傾斜範囲をテストするには、トモグラフィーソフトウェアで「傾斜(°)の設定」を使用して、目的の値を入力して [設定]を押すことで角度を設定できます。(B)ステージは-60°に傾いており、検索マップのコーナーが-60°で完全に取得されないことを示しています。(C)ステージが60°傾いています。±60°で消えた穴を数えることで、グリッド正方形の傾斜範囲についてのアイデアを得ることができます。スケールバー = 2.5 μm。 この図の拡大版を表示するには、ここをクリックしてください。
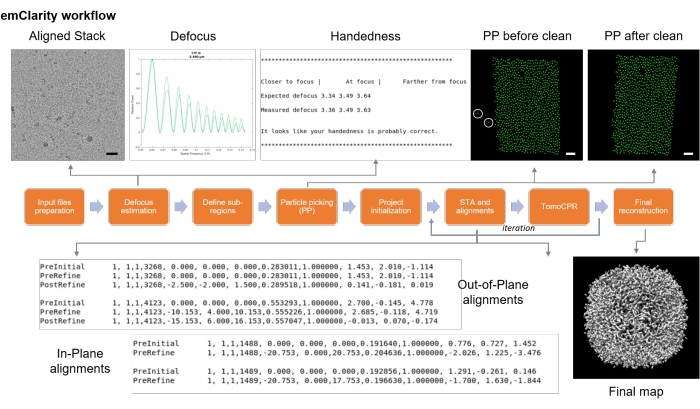
図9:emClarityフローチャート。 フローチャートには、クライオサブトモグラム平均化のための様々なステップが記載された。スケールバー= 50 nm。 この図の拡大版を表示するには、ここをクリックしてください。

図10:emClarityを使用したテンプレートマッチング 。 (A)グラフェン被覆グリッド上のアポフェリチンの典型的な顕微鏡写真。焦点ぼけ: −3.430 μm。 (B)テンプレート検索後にモデルポイントを重ねたトモグラムのスライス。(C)モデル点の上部および(D)側面投影図は、単分散アポフェリチン粒子の単一の平坦層を示す。スケールバー= 50 nm。 この図の拡大版を表示するには、ここをクリックしてください。
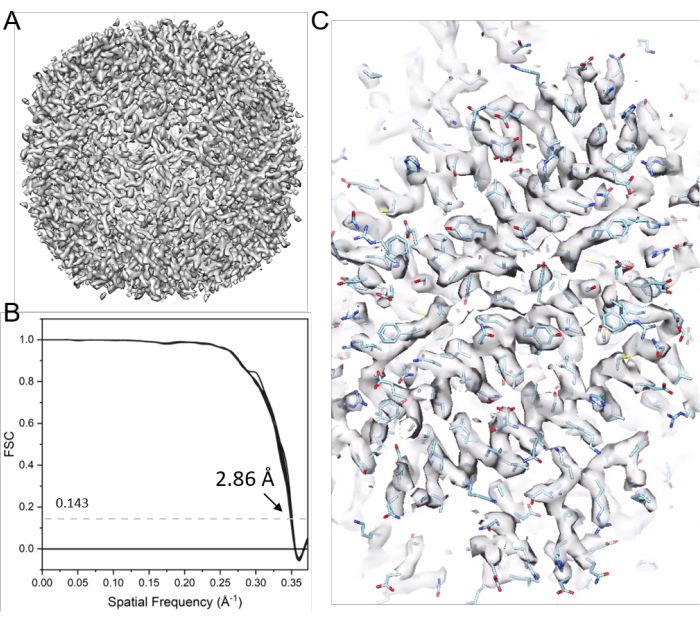
図11:アポフェリチンのクライオET STA 。 (a)サブトモグラムアライメントの21サイクル後の最終マップ。(B)38個のコーンFSCを含む、報告された解像度2.86 Åの最終マップのフーリエシェル相関(FSC)プロット。 (C)代表的な密度マップ(PDBモデル6s6147に適合)。 この図の拡大版を表示するには、ここをクリックしてください。
| 見本 | クライオETタイプ | Å/px | 傾斜範囲 (+/-) | チルトステップ(°) | デフォーカス範囲(μm) | 総線量(e-/Å2) | 解決 | ローデータ | 参考 |
| アポフェリチン | 精製/分子(STA) | 1.34 | 60 | 3 | 1.5 – 3.5 | 102 | 2.86 | EMPIAR-10787 | 17 & この論文 |
| HIV-1ギャグ | 精製/分子(STA) | 1.35 | 60 | 3 | 1.5 – 3.96 | 120 | 3.1 | EMPIAR-10164 | 17 |
| リボソーム | 精製/分子(STA) | 2.1 | 60 | 3 | 2.2 – 4.3 | 120 | 7 | EMPIAR-10304 | 42 |
| SARS-CoV-2スパイク | ラメラ/モレキュラー(STA) | 2.13 | 54 | 3 | 2 – 7 | 120 | 16 | EMPIAR-10753 | 45 |
| ニューロンの軸索構造 | セルラー(微細構造) | 5.46 | 60 | 2 | 3.5 – 5 | 90 | N.D. | EMPIAR-10922 | 47 |
表1:いくつかのクライオET研究のための収集パラメータ。 精製タンパク質または in situ タンパク質からの分子詳細の再構築を標的とした研究と、微細構造細胞の特徴の解明とセグメント化を目的とした研究。
動画1:アポフェリチンサンプルを通常のEMグリッド上にトモグラムし、互換性のあるフィルターを備えた超高解像度カメラを備えたクライオTEMで画像化します。 チルトシリーズは、電子線トモグラフィーソフトウェアで54°のチルトスパンと134 e-/A2 の総線量で、線量対称スキームで取得されました。スケールバー= 50 nm。 この動画をダウンロードするには、ここをクリックしてください。
動画2: EMグリッド上で成長させた初代ニューロンを、互換性のあるフィルターを備えた超高解像度カメラを搭載したクライオTEMで直接イメージングしたトモグラム。チルトシリーズは、電子線トモグラフィーソフトウェアで60°のチルトスパンと120 e-/A2 の総線量で、線量対称スキームで取得されました。スケールバー= 100 nm。 この動画をダウンロードするには、ここをクリックしてください。
動画3: EMグリッド上のシアノバクテリアのトモグラムをFIB粉砕し、高速カメラと互換性のあるフィルターを備えたクライオTEMで画像化。チルトシリーズは、電子線トモグラフィーソフトウェアで50°のチルトスパンと120 e-/A2 の総線量で、線量対称スキームで取得されました。スケールバー= 87.2 nm。 この動画をダウンロードするには、ここをクリックしてください。
補足ファイル 1: 焦点ぼけを推定するためのパラメーター ファイル テンプレート。このファイルをダウンロードするには、ここをクリックしてください。
補足表1:データ収集と顕微鏡セットアップの詳細。この表をダウンロードするには、ここをクリックしてください。
補足表2:実行順のコマンドのリスト。この表をダウンロードするには、ここをクリックしてください。
ディスカッション
トモ5
トモグラフィーソフトウェアのワークフローの説明は、(リモート)バッチ断層撮影セッションのセットアップのための1つの潜在的かつ最も合理化された方法を強調しています。このソフトウェアは初心者にとって簡単ですが、クライオEMの最初の経験と基本的な断層撮影の理解がセットアップに役立ちます。重要な手順はプロトコルで強調表示されており、別のセットアップアプローチが使用されている場合でもトラブルシューティングに役立ちます。ソフトウェアの進歩により、(リモート)データ収集が容易になり、幅広いユーザーベースがcryo-ETにアクセスしやすくなります。一般的に発生する問題のトラブルシューティングに役立ついくつかのヒントとコツを以下に説明します。
試験片を±60°に傾けると、高い傾斜のグリッドバーが視界を覆い隠す可能性があるため、考慮すべき重要なポイントの1つはグリッドの選択です(図8)。TEMグリッドでは、メッシュサイズはグリッドの単位長さあたりのグリッド正方形の数を指します。メッシュ数が大きいほど、単位長さあたりのグリッド正方形が多くなり、グリッド正方形の密度が高くなり、グリッド正方形が小さい、つまり、400メッシュグリッドは200メッシュグリッドよりも正方形が小さくなります。断層撮影用のグリッドの適切な選択は、200メッシュまたは300メッシュグリッドです。 図 8 に示すように、グリッドが傾くと、収集できる領域が減少します。±60°の傾斜では、300メッシュのグリッドは、完全な断層撮影を取得できる小さな視野を持ちます。200メッシュグリッドの利点は、グリッド正方形が大きいほど分子断層撮影のセットアップが速くなり、グリッド正方形の面積が増えると、一晩の収集には1つの正方形で十分になることです。欠点は、200メッシュグリッドはより壊れやすいため、取り扱いとクリッピングにはより多くのフィネスが必要になることです。
さらに、EMグリッドに穴のあるサポートフィルム( 材料表を参照)を使用する場合は、露光領域に対するフォーカス領域とトラッキング領域の設定に穴の間隔を考慮する必要があります。理想的には、最適な迅速なセットアップのために、所望の倍率でのビーム直径は、傾斜軸に沿って露光領域に隣接する炭素領域をカバーするのに十分小さくなければなりません。このようにして、各穴の潜在的な関心領域を取得できます。
ソフトウェアのユーセントリック高さルーチンは現在、serialEMルーチンほど堅牢ではないため、次のヒントでこの問題を回避できます。ユーセントリック高さプリセットを使用してユーセントリック高さの決定が失敗した場合は、代わりに概要プリセットを使用して、「ステージチルトによる自動ユーセントリック」を再実行できます。これにより、ユーセントリックの高さが 0 から遠く離れている場合の問題を解決できます。これが成功した場合は、「ユーセントリック高さ」プリセットを使用して「ステージチルトによる自動ユーセントリック」を再実行して、精度を向上させることができます。失敗した場合は、ユーセントリック高さプリセットを使用して「ビームチルトによる自動ユーセントリック」を実行してから、「ステージチルトによる自動ユーセントリック」を再実行するか、「ステージ」設定のTEMユーザーインターフェイスで「ビームチルトによる自動ユーセントリック」によって統合されたz高さを手動で設定できます。ホールの繰り返しパターンを持つグリッドが使用される場合、それらは単一の相互相関ピークの識別を妨げる可能性があります。ユーセントリック高さプリセットを-25μmなどの低いデフォーカスオフセットに変更したり、露光時間を短くしたりして、ホールパターンからの相互相関を減らすことができます。一方、レーシーグリッド/ラメラを使用すると、強い相互相関ピークに対して十分な信号が得られない場合があります。ユーセントリック高さプリセットを変更して、-75μmなどのより大きなデフォーカスオフセットや長時間の露光時間を変更して、相互相関ピークを強化することができます。別のオプションは、画像フィルター設定を調整することです。それらは「準備」タブにあります。フィルター設定を調整するオプションは、低(概観/グリッドスクエア)、中(ユーセントリック高さ)、高倍率(トラッキング/フォーカス)に設定して、各プリセットに最適な相互相関ピークを見つけることができます。必要な入力は、0°と5°の1つの画像で、その後に比較をクリックして両方の画像を 比較 します。最長波長の推奨開始値は画像内の縮尺記号の 4 分の 1 で、最短波長の推奨開始値は縮尺記号の 40 分の 1 です。ピークが確実に特定されない場合は、説得力のあるピークが見つかるまで設定を最適化できます。毎回画像を再取得する必要はありません。「比較」を押すだけで十分です。それでもTOMOがユーセントリック高さを自動的に見つけられない場合は、手動のユーセントリック高さキャリブレーションを使用できます。「準備」タブの概観倍率で適度に大きな氷の結晶を中央に配置し、TEMユーザーインターフェイスの「ステージコントロール」に移動し、アルファを-30°に設定し、ステージのz値を調整して、蛍光スクリーン画像を使用して結晶を再中心化します。TEMユーザーインターフェースで「高解像度」および「高コントラスト」設定を選択すると、これが簡単になります(蛍光スクリーンウィンドウの下部にあるボタン)。オプションで、ライブモードのカメラにアクセスできる場合は、これを使用してユーセントリックの高さを決定できます。蛍光スクリーンよりも簡単になります。
5.8より前のTomo5バージョンの最大の制限は、中倍率モンタージュの欠落、線量対称スキームの欠落、およびユーセントリック高さ検出に関連する問題です。これらは、迅速な開発とコミュニティサポートを備えたフリーウェアであるSerialEM、堅牢なユーセントリック高さルーチン、およびスクリプトのオプション、つまりカスタムビルドの線量対称スキームに存在します。Tomo5 のバージョン 5.8 以降では、ユーセントリック高さを見つける際に最もよく発生する問題、つまり目標 Z 値のループが失敗する問題は、ユーセントリック高さの許容基準を設定するオプションを実装することで解決されました。ただし、グリッドとサンプルの種類が異なる場合は、画像フィルター設定を調整して、個々のセッション固有のイメージング条件を反映し、ユーセントリックの高さを見つけるために可能な限り最良の相互相関ピークを提供し、トモグラム取得中にフォーカス領域とトラッキング領域が確実に機能することを強くお勧めします。
全体として、多くの施設はパンデミックの際にリモート操作に急速に適応しました。Tomo5ソフトウェアは、リモート操作に適したトモグラフィーへの簡単なアクセスとユーザーフレンドリーなルートを提供します。ソフトウェアの進歩により、リモートデータ収集とトモグラフィー収集が一般的にコミュニティでより主流になることは間違いありません。
エムクラリティ
emClarityはテンプレートベースのパーティクルピッキング方法を使用するため、目的のオブジェクトのテンプレートが必要です。粒子ピッキング(ステップ2.6)は非常に敏感であり、最終的な構造の鍵となります。平均化とアライメント(ステップ2.9)の前に、誤検知を注意深くチェックし、手動で削除する必要があります。テンプレートが利用できない場合、emClarity は使い勝手が悪いかもしれませんが、Dynamo37 や PEET48 などの他のソフトウェアを使用して初期モデルを作成することは可能です。
異種サンプルの場合、emClarityには、ユーザーがさまざまなスケールの特定の特徴に集中できる分類方法が装備されています。分類の前に数サイクルの線形を実行し、より高いビン分割 (ビン 4 やビン 3 など) で実行すると便利です。
ソフトウェアの最新バージョン(V1.5.3.11)は、最初のリリース(V1.0)17と比較して大幅に更新されています。これらには、CTF推定中の利き手チェックが含まれますが、これらに限定されません(ステップ2.3)。アライメントの対称性(CX、I、I2、O)。粒子ごとの3Dサンプリング関数(3DSF)の計算。互換性と安定性のためのMATLAB 2019aへの切り替え。生の投影画像(cisTEM)を使用した再構成。ソフトウェアはさまざまなサンプルで改善を続けており、最新の発表はオンラインで見つけることができます(材料表を参照)。
開示事項
著者には利益相反はありません。
謝辞
私たちは、ウェルカムトラスト、MRC、およびBBRSCが資金提供する英国の国立電子バイオイメージングセンター(eBIC)のクライオEM施設へのアクセスとサポートについて、ダイヤモンド光源に感謝しています。また、アポフェリチン断層撮影(動画1)の取得についてアンドリュー・ハウ氏、ニューロン断層撮影(動画2)の作成と取得をしてくれたイシカ・クマール氏、シアノバクテリアラメラ断層撮影法(動画3)を撮影してくれたクレイグ・マクレガー・チャトウィン氏にも感謝します。
資料
| Name | Company | Catalog Number | Comments |
| Software | |||
| Tomography | Thermo Fisher Scientific | 5.9.0 | Internal terminology: Tomo5 in document |
| TEM server | Thermo Fisher Scientific | 7.10.1 | |
| TIA | Thermo Fisher Scientific | 5.10.1 | |
| DigitalMicrograph | Gatan | 3.44 | |
| emClarity | Open-Source software | 1.5.3.11 | Software for high-resolution cryo-electron tomography and subtomogram averaging |
| IMOD | Open-Source software | 4.11 | Modeling, display and image processing programs used for 3D reconstruction and modeling of microscopy images with a special emphasis on electron microscopy data |
| MotionCor2 | Free for academic use | 1.1.0 | A multi-GPU program that corrects beam-induced sample motion recorded on dose fractionated movie stacks |
| ETomo | Open-Source software | 4.11 | ETomo is an interface for running a subset of IMOD and PEET commands. |
| NoMachine | NoMachine, freeware | 7.9.2 | Remote desktop software |
| TeamViewer | TeamViewer AG | - | Remote access and remote control computer software |
| Materials | |||
| Quantifoil (holey support film) EM grids | Quantifoil | - | A flat film of carbon with pre-defined hole size, shape and arrangement |
| Instrumentation | |||
| Titan Krios microscope | Thermo Fisher Scientific | Titan Krios G2 | |
| K3 camera and GIB energy filter | Gatan | - | |
| Falcon 4 camera and Selectris X energy filter | Thermo Fisher Scientific | - | |
| Website | |||
| Website 1: https://github.com/bHimes/emClarity/ | - | - | Link to download the emClarity software package |
| Website 2: https://bio3d.colorado.edu/imod/ | - | - | Link to download IMOD |
| Website 3: https://github.com/ffyr2w/emClarity-tutorial | - | - | Link to the emClarity online tutorial |
| Website 4: https://emcore.ucsf.edu/ucsf-software | - | - | Link to download MotionCor2 |
| Website 5: https://github-wiki-see.page/m/bHimes/emClarity/wiki | - | - | Link to the newest announcements including updates and bug fixs for emClarity |
参考文献
- Bai, X. -. C., Fernandez, I. S., McMullan, G., Scheres, S. H. W. Ribosome structures to near-atomic resolution from thirty thousand cryo-EM particles. eLife. 2, 00461 (2013).
- Li, X., et al. Electron counting and beam-induced motion correction enable near-atomic-resolution single-particle cryo-EM. Nature Methods. 10 (6), 584-590 (2013).
- Nakane, T., et al. Single-particle cryo-EM at atomic resolution. Nature. 587 (7832), 152-156 (2020).
- Kato, T., et al. CryoTEM with a cold field emission gun that moves structural biology into a new stage. Microscopy and Microanalysis. 25, 998-999 (2019).
- Yip, K. M., Fischer, N., Paknia, E., Chari, A., Stark, H. Atomic-resolution protein structure determination by cryo-EM. Nature. 587 (7832), 157-161 (2020).
- Mastronarde, D. N. SerialEM: A program for automated tilt series acquisition on Tecnai microscopes using prediction of specimen position. Microscopy and Microanalysis. 9, 1182-1183 (2003).
- Schorb, M., Haberbosch, I., Hagen, W. J. H., Schwab, Y., Mastronarde, D. N. Software tools for automated transmission electron microscopy. Nature Methods. 16 (6), 471-477 (2019).
- Deng, Y., et al. Smart EPU: SPA getting intelligent. Microscopy and Microanalysis. 27 (1), 454-455 (2021).
- Carragher, B., et al. Leginon: An automated system for acquisition of images from vitreous ice specimens. Journal of Structural Biology. 132 (1), 33-45 (2000).
- Bell, J. M., Chen, M., Baldwin, P. R., Ludtke, S. J. High resolution single particle refinement in EMAN2.1. Methods. 100, 25-34 (2016).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. cryoSPARC: Algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods. 14 (3), 290-296 (2017).
- Grant, T., Rohou, A., Grigorieff, N. cisTEM, user-friendly software for single-particle image processing. eLife. 7, 35383 (2018).
- Zivanov, J., et al. New tools for automated high-resolution cryo-EM structure determination in RELION-3. eLife. 7, 42166 (2018).
- Kimanius, D., Dong, L., Sharov, G., Nakane, T., Scheres, S. H. W. New tools for automated cryo-EM single-particle analysis in RELION-4.0. Biochemical Journal. 478 (24), 4169-4185 (2021).
- Galaz-Montoya, J. G., Flanagan, J., Schmid, M. F., Ludtke, S. J. Single particle tomography in EMAN2. Journal of Structural Biology. 190 (3), 279-290 (2015).
- Bharat, T. A. M., Scheres, S. H. W. Resolving macromolecular structures from electron cryo-tomography data using subtomogram averaging in RELION. Nature protocols. 11 (11), 2054-2065 (2016).
- Himes, B. A., Zhang, P. emClarity: Software for high-resolution cryo-electron tomography and subtomogram averaging. Nature Methods. 15 (11), 955-961 (2018).
- Weissenberger, G., Henderikx, R. J. M., Peters, P. J. Understanding the invisible hands of sample preparation for cryo-EM. Nature Methods. 18 (5), 463-471 (2021).
- White, J. B. R., et al. Single particle cryo-electron microscopy: From sample to structure. Journal of Visualized Experiments. (171), e62415 (2021).
- Orlova, E. V., Saibil, H. R. Structural analysis of macromolecular assemblies by electron microscopy. Chemical Reviews. 111 (12), 7710-7748 (2011).
- Turk, M., Baumeister, W. The promise and the challenges of cryo-electron tomography. FEBS Letters. 594 (20), 3243-3261 (2020).
- Liu, Y. -. T., et al. Isotropic reconstruction of electron tomograms with deep learning. bioRxiv. , (2021).
- Briggs, J. A. G. Structural biology in situ-The potential of subtomogram averaging. Current Opinion in Structural Biology. 23 (2), 261-267 (2013).
- Grünewald, K., et al. Three-dimensional structure of herpes simplex virus from cryo-electron tomography. Science. 302 (5649), 1396-1398 (2003).
- Allegretti, M., et al. In-cell architecture of the nuclear pore and snapshots of its turnover. Nature. 586 (7831), 796-800 (2020).
- Wang, Z., et al. The molecular basis for sarcomere organization in vertebrate skeletal muscle. Cell. 184 (8), 2135-2150 (2021).
- Winey, M., Meehl, J. B., O'Toole, E. T., Giddings, T. H. Conventional transmission electron microscopy. Molecular Biology of the Cell. 25 (3), 319-323 (2014).
- Davies, K. M., et al. Macromolecular organization of ATP synthase and complex I in whole mitochondria. Proceedings of the National Academy of Sciences. 108 (34), 14121 (2011).
- Wagner, J., Schaffer, M., Fernández-Busnadiego, R. Cryo-electron tomography-The cell biology that came in from the cold. FEBS Letters. 591 (17), 2520-2533 (2017).
- Mahamid, J., et al. Visualizing the molecular sociology at the HeLa cell nuclear periphery. Science. 351 (6276), 969-972 (2016).
- Erdmann, P. S., et al. In situ cryo-electron tomography reveals gradient organization of ribosome biogenesis in intact nucleoli. Nature Communications. 12 (1), 5364 (2021).
- Tegunov, D., Xue, L., Dienemann, C., Cramer, P., Mahamid, J. Multi-particle cryo-EM refinement with M visualizes ribosome-antibiotic complex at 3.5 Å in cells. Nature Methods. 18 (2), 186-193 (2021).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152 (1), 36-51 (2005).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Heymann, J. B., Belnap, D. M. Bsoft: Image processing and molecular modeling for electron microscopy. Journal of Structural Biology. 157 (1), 3-18 (2007).
- Tang, G., et al. EMAN2: An extensible image processing suite for electron microscopy. Journal of Structural Biology. 157 (1), 38-46 (2007).
- Castaño-Díez, D., Kudryashev, M., Arheit, M., Stahlberg, H. Dynamo: A flexible, user-friendly development tool for subtomogram averaging of cryo-EM data in high-performance computing environments. Journal of Structural Biology. 178 (2), 139-151 (2012).
- Hrabe, T., et al. PyTom: A python-based toolbox for localization of macromolecules in cryo-electron tomograms and subtomogram analysis. Journal of Structural Biology. 178 (2), 177-188 (2012).
- Himes, B. A. emClarity. GitHub. , (2021).
- . The IMOD Home Page Available from: https://bio3d.colorado.edu/imod/ (2020)
- . GitHub: emClarity-tutorial Available from: https://github.com/ffyr2w/emClarity-tutorial (2021)
- Ni, T., et al. High-resolution in situ structure determination by cryo-electron tomography and subtomogram averaging using emClarity. Nature Protocols. 17 (2), 421-444 (2022).
- . MotionCor2 Available from: https://emcore.ucsf.edu/ucsf-software (2016)
- . Apoferritin from mouse at 1.84 angstrom resolution Available from: https://www.emdataresource.org/EMD-10101 (2022)
- Nedozralova, H., et al. In situ cryo-electron tomography reveals local cellular machineries for axon branch development. Journal of Cell Biology. 221 (4), 202106086 (2022).
- Mendonça, L., et al. Correlative multi-scale cryo-imaging unveils SARS-CoV-2 assembly and egress. Nature Communications. 12 (1), 4629 (2021).
- . Apoferritin from mouse at 1.84 angstrom resolution Available from: https://www.rcsb.org/structure/6s61 (2019)
- Nicastro, D., et al. The molecular architecture of axonemes revealed by cryoelectron tomography. Science. 313 (5789), 944-948 (2006).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved