Method Article
Kryo-Elektronen-Tomographie Ferndatenerfassung und Subtomogramm-Mittelwertbildung
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Das vorliegende Protokoll beschreibt die hochauflösende Kryo-Elektronentomographie-Ferndatenerfassung mit Tomo5 und die anschließende Datenverarbeitung und Subtomogramm-Mittelung mittels emClarity. Apoferritin wird als Beispiel verwendet, um detaillierte Schritt-für-Schritt-Prozesse zu veranschaulichen, um eine Kryo-ET-Struktur bei 2,86 Å Auflösung zu erreichen.
Zusammenfassung
Die Kryo-Elektronentomographie (Kryo-ET) hat in den letzten Jahren an Dynamik gewonnen, insbesondere seit der Einführung von Direktelektronendetektoren, verbesserten automatisierten Erfassungsstrategien, präparativen Techniken, die die Möglichkeiten erweitern, was das Elektronenmikroskop mit Kryo-ET hochauflösend abbilden kann, und neuer Subtomogramm-Mittelungssoftware. Darüber hinaus wurde die Datenerfassung zunehmend rationalisiert, wodurch sie für viele Benutzer zugänglicher wird. Die SARS-CoV-2-Pandemie hat die Datenerfassung der Kryo-Elektronenmikroskopie (Kryo-EM), insbesondere für Einzelpartikel-Kryo-EM, in vielen Einrichtungen weltweit weiter beschleunigt und bietet während der Pandemie ununterbrochenen Benutzerzugriff auf modernste Instrumente. Mit den jüngsten Fortschritten bei Tomo5 (Software für die 3D-Elektronentomographie) ist die Ferndatenerfassung von Kryo-ET robust und einfach von überall auf der Welt zu handhaben. Dieser Artikel zielt darauf ab, eine detaillierte Anleitung zu bieten, beginnend mit der Einrichtung der Datenerfassung in der Tomographie-Software für den Prozess einer (Remote-) Kryo-ET-Datenerfassungssitzung mit detaillierter Fehlerbehebung. Das (Fern-)Datenerfassungsprotokoll wird durch den Workflow zur Strukturbestimmung bei nahezu atomarer Auflösung durch Subtomogrammmittelung mit emClarity am Beispiel von Apoferritin ergänzt.
Einleitung
Es ist allgemein bekannt, dass die kryogene Elektronenmikroskopie (Kryo-EM) eine Renaissance erlebt hat und sich zu einem zentralen und zentral nützlichen Werkzeug in der Strukturbiologie entwickelt hat. Die Entwicklung und Nutzung von Direktelektronendetektoren 1,2,3, verbesserte Mikroskope und Elektronenquellen3,4,5, Verbesserungen in der Automatisierung/Durchsatz6,7,8,9 und rechnerische Fortschritte in der Einzelteilchenanalyse 10,11,12,13 ,14 und Tomographie15,16,17 sind alle zum Teil für den jüngsten Erfolg der Technik verantwortlich. Diese technologischen Treiber haben die Fähigkeit von Kryo-EM entwickelt, biologische makromolekulare Strukturen unter kryogenen und nativen Bedingungen zu lösen. Die Auflösungen, die leicht erhältlich sind, reichen für eine atomar genaue Modellierung aus und haben die Technik an die Spitze der strukturbiologischen Arena gebracht. Ein reduktionistischer Ansatz zur Expression und Reinigung eines biologischen Ziels von Interesse hat sich in der makromolekularen Kristallographie (MX) für die biologische Grundlagenforschung, die Wirkstoffforschung und die translationale Wissenschaft seit langem als erfolgreich erwiesen. Mit dem gleichen Ansatz kann Kryo-EM nun Ergebnisse liefern, die parallel zu hochauflösenden MX-Studien verlaufen. Der derzeitige große Erfolg im Kryo-EM-Zweig der Strukturbiologie wird als Einzelpartikelanalyse (SPA) bezeichnet, die 2D-Projektionsbilder typischerweise einer gereinigten Proteinprobe18 aufnimmt, um Tausende von Ansichten eines biologischen Makromoleküls19 zu erhalten. Diese Bilder (1) enthalten Informationen aus einer Reihe von Ansichten, die die Orientierungen des Ziels im 3D-Raum vollständig darstellen und (2) erfassen die Objektkonformationsheterogenität, die später getrennt und untersucht werden kann.
Ein alternativer Ansatz zur Aufnahme dieser 2D-Projektionsbilder biologischer Proben, auch in situ und ohne Reinigung, ist die Kryo-Elektronentomographie (Kryo-ET). Cryo-ET nimmt eine Reihe von Bildern desselben Objekts in geneigten Winkeln auf, indem die Probe mechanisch gedreht wird. Daher werden die in SPA gesammelten 2D-Projektionen, die die Winkelposen des interessierenden Moleküls darstellen, inhärent als Teil des Kryo-ET-Bildgebungsexperiments20 gesammelt. Tomographische Neigungsreihen werden dann in ein Tomogramm rekonstruiert, das 3D-Darstellungen der abgebildeten makromolekularen Komplexe enthält. Die Art der tomographischen Datenerfassung verringert bis zu einem gewissen Grad die Abhängigkeit von der Mittelwertbildung, um eine vollständige 3D-Darstellung eines Moleküls aus einer Sammlung von 2D-Bildern zu erhalten. Aufgrund aktueller Bühnendesigns ist die Probe jedoch typischerweise von −60° bis +60° geneigt, so dass in der tomographischen 3D-Rekonstruktion ein Keil21 an Informationen fehlt.
Die 3D-Rekonstruktionen in einem einzigen Tomogramm haben dann einen fehlenden Informationskeil und ein geringes Signal-Rauschen. Einzelne Makromoleküle können als Subtomogramme extrahiert und zusammen gemittelt werden, um dies anzugehen. Wenn jedes Makromolekül in einem Subtomogramm in einer anderen Orientierung gefunden wird, ist der fehlende Keil in jedem Subtomogramm des Zielobjekts anders ausgerichtet, so dass die Mittelung über viele Kopien Informationen aufgrund des fehlenden Keils ausfüllt. Jüngste Entwicklungen in der Bildverarbeitung haben auch versucht, neuronale Netze der künstlichen Intelligenz zu trainieren, um den fehlenden Keil mit aussagekräftigen Daten zu füllen22. Dieser Mittelungsprozess erhöht auch das Signal zu Rauschen, ähnlich dem Ziel der Mittelung in der Einzelpartikelanalyse, so dass sich die Qualität und Auflösung der Rekonstruktion verbessert. Wenn das interessierende Molekül eine Symmetrie besitzt, kann auch diese definiert und während der Mittelung verwendet werden, wodurch die Rekonstruktionsauflösung weiter verbessert wird. Die Extraktion von 3D-Volumina eines Makromoleküls aus einem Tomogramm in eine Reihe von Subtomogrammen und deren anschließende Verarbeitung wird als Subtomogrammmittelung (STA)23 bezeichnet. Wenn jedes Subtomogramm eine eindeutige Kopie des untersuchten Moleküls darstellt, kann jede strukturelle Heterogenität mit dem STA-Workflow abgefragt werden. Wie im SPA-Workflow üblich, können Klassifizierungstechniken während der STA eingesetzt werden, um die Konformationszustände des interessierenden Komplexes zu analysieren. Neben der STA, die eine hochauflösende Rekonstruktion in Kryo-ET ermöglicht, macht dieser Ansatz die Technik zu einem leistungsfähigen Werkzeug, um die strukturellen Mechanismen von Makromolekülen in ihrer nativen zellulären Umgebung oder von Zielen zu untersuchen, die SPA24,25,26 oft nicht zugänglich sind.
Die Elektronentomographie hat eine lange Geschichte der Bestimmung der 3D-Ultrastruktur von Zellproben bei Raumtemperatur27. Die Aufnahme von Ansichten durch physikalisches Kippen der Probe liefert genügend Informationen für die 3D-Rekonstruktion eines Objekts auf zellulären Längenskalen und ist besonders wichtig, wenn zellulären Strukturen die Regelmäßigkeit für die Mittelung fehlt. Zellen können auch auf Substraten für die Kryo-ET-Bildgebung an den Zellrändern eingefroren werden, wo die Probe dünn genug ist, um elektronentransparent zu sein. Unter diesen Bedingungen kann STA verwendet werden, um makromolekulare Strukturen in einer zellulären Umgebung zu bestimmen, wenn die Probe dünn genug ist, um elektronentransparent zu sein28. In Kombination mit zusätzlichen präparativen Techniken, einschließlich kryokorrelativer Licht- und Elektronenmikroskopie (Kryo-CLEM) und fokussiertem Ionenstrahlfräsen (Kryo-FIB), kann Kryo-ET jedoch verwendet werden, um ganze Zellen unter kryogenen Bedingungen abzubilden29. Dies vereint die Kraft der Kryo-ET zur Untersuchung der zellulären Ultrastruktur mit der Kraft von STA, um die Strukturen makromolekularer Komplexe in situ zu bestimmen, während sie ihre zelluläre Position30 identifizieren und Momentaufnahmen von Komplexen liefern, die an dynamischen Prozessen beteiligt sind31. Die Fähigkeit der Technik, zelluläre Proben abzubilden und STA in mehreren Studien einzusetzen, hat die Leistungsfähigkeit der Technik zur Lösung makromolekularer Strukturen in situ unterstrichen, selbst bei Auflösungen, die mit SPA32 vergleichbar sind. Ein weiterer Vorteil liegt in der Kenntnis der ursprünglichen Position des Makromoleküls, dargestellt durch die abschließende klassifizierte 3D-Rekonstruktion im Tomogramm30. Daher kann die makromolekulare Struktur mit der zellulären Ultrastruktur korreliert werden. Diese Beobachtungen über Längenskalen hinweg werden vermutlich zu wichtigen Erkenntnissen führen, bei denen strukturelle Mechanismen im Rahmen funktioneller Studien mit zellulären Veränderungen korreliert werden können.
Kryo-ET und STA ermöglichen die Datenerfassung in drei Hauptarbeitsabläufen: molekulare, zelluläre und Lamellentomographie. Die Strukturen gereinigter makromolekularer Komplexe können mittels Kryo-ET durch molekulare Tomographie bestimmt werden. Die Bestimmung von Proteinstrukturen in ihrer zellulären Umgebung, in der die Zelle dünn genug ist, kann als zelluläre Tomographie bezeichnet werden. In jüngerer Zeit, mit der Entwicklung von kryogenem Targeting und Mahlen, können dieselben Techniken in Lamellentomographie-Workflows angewendet werden, um die Proteinstrukturen tief in der Zelle in ihrer natürlichen Umgebung zu bestimmen und gleichzeitig den zellulären Kontext aufzudecken, in dem diese Proteine beobachtet werden. Je nach verfügbarem Softwarepaket und vor allem je nach Anforderung der Probe können unterschiedliche Datenerfassungsstrategien eingesetzt werden. Molekulare oder nicht haftende Proben auf einem Kupfer-TEM-Gitter eines gereinigten Proteins erfordern typischerweise weniger Handhabung und bleiben daher im Idealfall flach und unbeschädigt. Elektronentomogramme können leicht in Reihe über ein löchriges Kohlenstoffgitter aufgestellt werden, um schnell Dutzende bis Hunderte von Tomogrammen systematisch zu erfassen. Der einfachste Weg für Benutzer, molekulare Tomographieproben einzurichten, in denen Proteine reichlich im Gitter vorhanden sind, wäre die Verwendung von Tomo5 (Software für die 3D-Elektronentomographie, die in der vorliegenden Studie verwendet wird, siehe Tabelle der Materialien). Andere Tomographie-Software wie Leginon9 und serialEM6 sind ebenfalls verfügbar; Sie bieten mehr Setup-Optionen für personalisiertere Ansätze zur Datenerfassung, sind jedoch komplexer und können daher schwieriger zu navigieren sein, insbesondere für Benutzer, die neu in der Tomographie sind, und für Benutzer, die auf ihre Sitzung aus der Ferne zugreifen. Für eine Einrichtung mit einer großen und vielfältigen Benutzerbasis ist Tomo5 einfach in einer entfernten Umgebung zu bedienen und Benutzer darin zu schulen. Für adhärente Zellen erfordern Gitter typischerweise mehr Handhabungsschritte, und die Notwendigkeit, zerbrechliche Goldgitter zu verwenden, erhöht den Bedarf an verbesserter Sorgfalt bei der Handhabung und Datenerfassungsstrategien. Um das Auffinden einer Zellregion von Interesse zu erleichtern und eine Okklusion aus dem Gitter selbst bei hohen Neigungswinkeln zu vermeiden, ist es auch vorteilhaft, größere Maschenweite zu verwenden, aber auf Kosten dessen, dass sie von Natur aus zerbrechlicher sind. Bei Lamellenproben wird die Zerbrechlichkeit der Probe durch die Qualität der Lamelle bestimmt, die variabel sein kann. Diese Faktoren erhöhen die Einrichtungszeit und Überlegungen, aber die erhöhte Anpassungsfähigkeit und Robustheit machen Tomo5 wieder für diese Art der Datenerfassung geeignet. Für jeden Workflow gibt es jedoch spezielle Datenerfassungsszenarien. BISECT und PACE-tomo (beide laufen in SerialEM) führen die Möglichkeit der skriptgesteuerten Strahlbildverschiebung während der Tomographieaufnahme ein, um die Tomogramm-Erfassungsgeschwindigkeit zu erhöhen28, insbesondere in der molekularen Tomographie. Montagen mit mittlerer Vergrößerung (MMM) in SerialEM 6,7,33 können molekulare Merkmale in allen Arbeitsabläufen besser identifizieren und präzise anvisieren, obwohl diese Funktionen zum Zeitpunkt des Schreibens in Tomo5 implementiert werden.
Wie SPA werden Kryo-ET und STA durch die Verbesserungen der Erfassungssoftware und eine Fülle verfügbarer Pakete für Subtomogramme von durchschnittlich 16,17,32,34,35,36,37,38 immer zugänglicher. Darüber hinaus wurde während der Pandemie der Fernzugriff auf Kryo-EM-Instrumente für den weiteren Betrieb nationaler Einrichtungen wie dem Electron Bio-Imaging Centre (eBIC) an der Diamond Light Source (DLS) in Großbritannien unerlässlich. Diese Entwicklungen haben die Kryo-ET für Forscher, die die Technik einsetzen möchten, zugänglicher und robuster gemacht. Sobald die Daten erfasst wurden, ist STA ein wesentliches Werkzeug für die Analyse wiederkehrender Objekte, um eine Rekonstruktion mit maximaler Auflösung zu erhalten und die Klassifizierung der makromolekularen Heterogenität zu ermöglichen. Das aktuelle Protokoll zielt darauf ab, eine detaillierte Anleitung zur Vorbereitung eines Kryo-TEM-Mikroskops für die Kryo-ET-Datenerfassung und zur Durchführung einer Subtomogramm-Mittelung mit emClarity an einem molekularen Tomographie-Datensatz von Apoferritin als Beispiel zu bieten. Die Verwendung von emClarity (Software für hochauflösende Kryo-Elektronentomographie und Subtomogramm-Mittelwertbildung, siehe Materialtabelle) erfordert das Ausführen von Skripten über die Befehlszeile, so dass ein gewisses Maß an Vertrautheit mit Linux/UNIX-Systemen vorausgesetzt wird.
Die Remote-Verbindung hängt von der Netzwerkumgebung in jedem Institut/jeder Einrichtung ab. Bei eBIC verwendet das Remote-System Programme, die eine Remote-Datenerfassung über die bei Diamond verwendete spezifische Netzwerkkonfiguration ermöglichen. Die Fernverbindung zum Mikroskop wird durch zwei Plattformen erleichtert: NoMachine und TeamViewer (siehe Materialtabelle). Mit dem Programm NoMachine kann sich der Benutzer an einem entfernten Windows-Desktop anmelden. Der von NoMachine bereitgestellte Remote-Windows-Desktop befindet sich im selben Netzwerk wie das Mikroskop und fungiert somit als virtueller Support-PC für das Mikroskop. Vom virtuellen Support-PC aus verbindet sich der Benutzer über TeamViewer mit dem Mikroskop und bietet direkten Zugriff und Steuerung zum Mikroskop-PC, auf dem TUI und Tomo ausgeführt werden.
Das vorliegende Protokoll besteht aus zwei Teilen (Schritt 1 und Schritt 2). Schritt 1 konzentriert sich auf die Kryo-ET-Ferndatenerfassung mit Tomo5 (Software für 3D-Elektronentomographie). Der Rundgang für eine (Remote-)Sitzung erfasst Bilder mit immer höheren Vergrößerungen, um es dem Benutzer letztendlich zu ermöglichen, die Tomographie-Software auf Zielprobenbereiche für die tomographische Datenerfassung zu lenken. Abbildung 1 fasst diesen Prozess zusammen. Schritt 2 beschreibt die Kryo-ET STA-Datenverarbeitung mit emClarity (Software für hochauflösende Kryo-Elektronentomographie und Subtomogramm-Mittelung). Abbildung 9 fasst diesen Prozess zusammen.
Das Protokoll ist für ein Remote-Publikum gedacht. Es geht davon aus, dass die Person physisch am Mikroskop und beim Laden der Proben die direkte Ausrichtung vorgenommen und sich um die Kameraabstimmung und die Referenzerfassung gekümmert hat. Für dieses Protokoll wird ein Drei-Kondensator-Linsensystem mit einem Autoloader angenommen. Weitere detaillierte Anleitungen zur Tomographie-Software finden Sie in der Windows-Starttaste, von der aus die Software geladen wurde, ein ausführliches Handbuch des Herstellers.
Protokoll
Die in dieser Studie verwendeten Softwarepakete sind teilweise frei verfügbar (siehe Materialtabelle).
1. Kryo-ET-Ferndatenerfassung mit Tomo5
- Wenn die Software nicht geladen ist, starten Sie diese Software zunächst vom TEM-Server-PC (siehe Materialtabelle).
- Führen Sie erste Prüfungen durch und konfigurieren Sie die Imaging-Bedingung, indem Sie Voreinstellungen auswählen.
- Starten Sie die Einrichtung der Sitzung, indem Sie die Bildaufnahmeparameter auf der Registerkarte "Vorbereitung" unter Voreinstellungen anpassen (Abbildung 2A,B).
- Passen Sie die "Übersichtsvergrößerung" an die Rasterquadratgröße und Dosis an, um angemessene Zählungen zu erhalten.
HINWEIS: Wenn die geeignete Vergrößerung für den Rastertyp unbekannt ist, kehren Sie nach dem Laden eines Rasters zu diesem Schritt zurück. - "Euzentrische Höhe" und "Suchvergrößerung" können identisch sein. Klicken Sie auf der Registerkarte "Tomographie" auf Suchen , um Positionen einzurichten und sicherzustellen, dass die Vergrößerung ausreicht, um die gewünschte Kennzahl anzuzeigen und den Bereich "Belichtung" und "Tracking/Fokus" im Sichtfeld unterzubringen (Abbildung 2C).
- Stellen Sie "Belichtungsvergrößerung" auf die gewünschte Zielpixelgröße ein. Wenn dies unbekannt ist, stellen Sie es fest, indem Sie die Vergrößerung an das gewünschte Sichtfeld anpassen, um es an den interessierenden Bereich anzupassen.
- Passen Sie die "Übersichtsvergrößerung" an die Rasterquadratgröße und Dosis an, um angemessene Zählungen zu erhalten.
- Kopieren Sie die Parameter der Bildaufnahme (Belichtung) in alle anderen Voreinstellungen mit hoher Vergrößerung (Tracking, Drift, Fokus, Thon Ring, Zero Loss,). Drücken Sie dazu Einstellen , um "Belichtung" auf das Mikroskop einzustellen, und auf Abrufen, um die "Belichtungseinstellungen" auf alle anderen hohen Vergrößerungen zu erhalten, nachdem Sie sie einzeln ausgewählt haben.
- Passen Sie die Belichtungszeiten an, insbesondere für "Fokus" und "Tracking", um eine angemessene Anzahl von Zählungen zu erhalten, auch unter Berücksichtigung der Dicke bei 60°, dh sowohl bei 0° als auch bei 60°, damit genügend Elektronen die Probe passieren können, um ein starkes Signal für die Kreuzkorrelation zu liefern.
HINWEIS: Als Schätzung sind die Belichtungszeiten für "Tracking" und "Fokus", die der Voreinstellung "Belichtung" entsprechen, eine gute erste Maßnahme. Ein Beispiel für die Dosisberechnung für die Voreinstellung "Exposition" finden Sie in Abbildung 3. Stellen Sie die berechnete Belichtungszeit für die Voreinstellung "Belichtung" in der Registerkarte "Präparation" ein.
- Passen Sie die Belichtungszeiten an, insbesondere für "Fokus" und "Tracking", um eine angemessene Anzahl von Zählungen zu erhalten, auch unter Berücksichtigung der Dicke bei 60°, dh sowohl bei 0° als auch bei 60°, damit genügend Elektronen die Probe passieren können, um ein starkes Signal für die Kreuzkorrelation zu liefern.
- Stellen Sie das "Zero Loss Preset" so ein, dass eine hellere und größere Spotgröße verwendet wird (eine größere Spotgröße entspricht einer kleineren Zahl), da dies eine höhere Dosis erfordert.
HINWEIS: Diese Voreinstellung wird am besten verwendet, um den Schlitz des Energiefilters auszurichten, wenn er am Mikroskop vorhanden ist.
- Starten Sie die Einrichtung der Sitzung, indem Sie die Bildaufnahmeparameter auf der Registerkarte "Vorbereitung" unter Voreinstellungen anpassen (Abbildung 2A,B).
- Führen Sie die Atlaserfassung mit den folgenden Schritten durch.
- Um einen Atlas zu sammeln, klicken Sie auf der Registerkarte "Atlas" auf Neue Sitzung , legen Sie die Sitzungseinstellungen fest, geben Sie den Speicherpfad und das Ausgabeformat ein und drücken Sie Übernehmen. Wählen Sie Screening (Abbildung 4), und kreuzen Sie dann alle zu erfassenden Atlanten an. Wählen Sie Ventile schließen , um das Mikroskop unbeaufsichtigt zu lassen. Dadurch werden die Säulenventile geschlossen, nachdem alle ausgewählten Atlanten gesammelt wurden. Um die Vorführung zu starten, drücken Sie die Taste Start .
- Untersuchen Sie die einzelnen oder mehrere Atlanten auf Ziele, indem Sie auf das Raster im linken Bereich unter "Screening" (Abbildung 4) klicken, dann mit der linken Maustaste klicken und die Maus ziehen, um sich zu bewegen, und die mittlere Bildlauftaste, um sie zu vergrößern und zu verkleinern. Wählen Sie ein Raster für das Ziel-Setup, wählen Sie es aus und klicken Sie in der Software auf Load Sample .
- Verwenden Sie für die Bildverschiebungskalibrierungen weiterhin die Registerkarte Atlas-Screening, um innerhalb des geladenen Rasters zu navigieren. Finden Sie ein identifizierbares Merkmal, das bei allen Vergrößerungsvoreinstellungen erkennbar ist, d. h. ein Eiskristall, der mit einer Kante eines Lochs oder einem anderen erkennbaren Merkmal überlappt (Abbildung 5). Bewegen Sie sich mit der rechten Maustaste zu diesem Quadrat und dann zu "Bühne in Rasterquadrat verschieben".
HINWEIS: Der Tisch muss sich auf euzentrischer Höhe befinden, um Bildverschiebungskalibrierungen korrekt durchzuführen.
- Führen Sie eine Bildverschiebungskalibrierung durch.
- Stellen Sie auf der Registerkarte "Auto Functions" (Abbildung 6) die Voreinstellung auf "Eucentric Height" (Euzentrische Höhe) ein, navigieren Sie zu "Auto-Eucentric by Stage Tilt" (Auto-Euzentrisch durch Bühnenneigung), und drücken Sie Start. Überwachen Sie das Statusfenster, um sicherzustellen, dass die euzentrische Höhe erfolgreich ermittelt wird, und behalten Sie die positiven und negativen Neigungsbilder im Auge. Sie müssen korrelieren.
HINWEIS: Ab Version 5.8 der Tomo-Software kann das Kriterium für die euzentrische Höhenakzeptanz geändert werden; Der Standardwert ist 0,25 μm, und die Einstellung auf 0,5-0,8 μm gibt mehr Spielraum. Die Werte hängen von der Leistung des Mikroskops ab, es wird jedoch empfohlen, sie so klein wie möglich zu halten.- Wenn Sie sich in euzentrischer Höhe befinden und scharf sind, zentrieren Sie die Bühne auf ein Feature. Sammeln Sie eine Vorschau mit der Vorgabe "Atlas". Stellen Sie alle "Presets" über den Reiter "Vorbereitung" ein. Erhöhen Sie anschließend die Vergrößerung auf Übersicht > mittlere Funktion > Vorschau, wiederholen Sie dann "Suchvergrößerung" und schließlich "Belichtungsvergrößerung".
- Wenn das Feature während des Targetings zentriert blieb, überspringen Sie die Kalibrierung der Bildverschiebung. Um die Bildverschiebung zu kalibrieren, wechseln Sie zur Registerkarte "Preparation", wählen Sie Calibrate Image Shift (Bildverschiebung kalibrieren) aus, und drücken Sie Start (Abbildung 5). Dadurch werden die niedrigeren Vergrößerungen iterativ an dem bei der Belichtungsvergrößerung zentrierten Feature ausgerichtet.
HINWEIS: Wenn die ursprüngliche Voreinstellung "Belichtung" zu diesem Zeitpunkt nicht zentriert ist, verwendet die Software für die Neuzentrierung nur für diesen Schritt die Stufenverschiebung.- Drücken Sie Fortfahren , um die Voreinstellung "Belichtung" anzuzeigen. Das nächste gezeigte Bild ist die "Suchvoreinstellung". Um die Bildverschiebung zwischen den Voreinstellungen zu kalibrieren, doppelklicken Sie mit der linken Maustaste in die Vorschau "Suchen" auf die Stelle, an der sich die Mitte der Vorschau "Belichtung" befindet, und drücken Sie dann Erneut aufnehmen (Abbildung 5). Klicken Sie auf Fortfahren , um zum nächsten Preset-Paar zu wechseln.
HINWEIS: Wenn während dieser Kalibrierung eine Bildverschiebung bei Atlasvergrößerung angewendet wurde, stellen Sie sicher, dass Sie den Atlas nach Abschluss der Bildverschiebungskalibrierung erneut erfassen. Wählen Sie den gewünschten Atlas aus und drücken Sie Reset Selected im oberen Bereich auf der Registerkarte "Atlas". Bestätigen Sie und beginnen Sie erneut, diesen Atlas zu erwerben.
- Drücken Sie Fortfahren , um die Voreinstellung "Belichtung" anzuzeigen. Das nächste gezeigte Bild ist die "Suchvoreinstellung". Um die Bildverschiebung zwischen den Voreinstellungen zu kalibrieren, doppelklicken Sie mit der linken Maustaste in die Vorschau "Suchen" auf die Stelle, an der sich die Mitte der Vorschau "Belichtung" befindet, und drücken Sie dann Erneut aufnehmen (Abbildung 5). Klicken Sie auf Fortfahren , um zum nächsten Preset-Paar zu wechseln.
- Stellen Sie auf der Registerkarte "Auto Functions" (Abbildung 6) die Voreinstellung auf "Eucentric Height" (Euzentrische Höhe) ein, navigieren Sie zu "Auto-Eucentric by Stage Tilt" (Auto-Euzentrisch durch Bühnenneigung), und drücken Sie Start. Überwachen Sie das Statusfenster, um sicherzustellen, dass die euzentrische Höhe erfolgreich ermittelt wird, und behalten Sie die positiven und negativen Neigungsbilder im Auge. Sie müssen korrelieren.
- Führen Sie die Tomographie-Einrichtung durch.
- Erstellen Sie die Einrichtung der Tomographie-Datenerfassung auf der Registerkarte "Tomographie".
HINWEIS: Sofern nicht ausdrücklich angegeben, erfolgt die Einrichtung vollständig auf der Registerkarte "Tomographie". - Starten Sie eine neue Sitzung. Wählen Sie unter "Session Setup" für biologische Proben Slab-like als Probentyp und wählen Sie Batch und Low Dose, wählen Sie das Ausgabeformat und den Speicherordner, fügen Sie optional einen E-Mail-Empfänger hinzu und drücken Sie dann Übernehmen.
HINWEIS: Ein Menüpunkt "Batch Positions" wird jetzt verfügbar (Abbildung 7A). Der aktuell geladene Atlas wird automatisch importiert. "Übersicht" und "Bilder suchen" können erfasst und erneut aufgerufen werden, bis ein neues Bild aufgenommen wird. Darüber hinaus können ab Version 5.8 Suchkarten mit 3 x 3, 4 x 4 und 5 x 5 Kacheln mit "Suchkarte erfassen" erfasst werden, um die Zielsuche und -einrichtung zu erleichtern. Sie entsprechen einer Version von Montagen mit mittlerer Vergrößerung. - Um Ziele einzurichten, gehen Sie zum "Atlaspfeil", suchen Sie eine Region von Interesse und bewegen Sie sich dorthin, indem Sie die Optionen auswählen, die mit einem rechten Mausklick angezeigt werden. Machen Sie ein Übersichtsbild, um eine gute Position für die euzentrische Höhenverstellung zu bestätigen, und drücken Sie dann auf Auto Eucentric; Dadurch wird die euzentrische By-Stage-Tilt-Routine ausgeführt. Erfassen Sie dann erneut ein neues Übersichtsbild, um die euzentrische Höhe zu aktualisieren.
HINWEIS: Die Taste "Autofokus" sollte nicht erforderlich sein, wenn sich die Position auf euzentrischer Höhe befindet. Wenn das euzentrische Höhenbild weit unscharf erscheint, ist die euzentrische Höhe wahrscheinlich nicht korrekt und muss neu erstellt werden (zur Fehlerbehebung bei euzentrischer Höhe siehe den Diskussionsabschnitt). - Überprüfen Sie das Ziel mit der Voreinstellung "Übersicht", bevor Sie die erste Position einrichten. Wählen Sie die Voreinstellung "Übersicht" und neigen Sie den Tisch um ±60°, um zu überprüfen, welchen Abstand von den Rasterbalken die Positionen einstellen können (Abbildung 8). Geben Sie dazu den gewünschten Neigungswinkelwert in das Fenster "Set Tilt" ein (Abbildung 8A).
- Überprüfen Sie das Quadrat "Übersicht" oder die erworbene "Suchkarte", wechseln Sie zu einem Bereich von Interesse und drücken Sie Suche erfassen. Überprüfen Sie das Bild "Suchen". Wenn der interessierende Bereich nicht zentriert ist, klicken Sie mit der rechten Maustaste auf die gewünschte Position, und wiederholen Sie dann Stufe hier verschieben und Bild erfassen. Legen Sie die Neigung (°) auf 0,00 (oder einen anderen Startwinkel, den die Bühne verarbeiten kann) fest, um den Startwinkel des Tomogramms zu definieren.
- Geben Sie für das erste Tomogramm die Namenskonvention Name für das gewünschte Tomogramm und die Defokussierung ein, um den gewünschten Unschärfewert für jedes Tomogramm festzulegen.
- Optional können ab Tomo Version 5.8 die Defokussierungswerte systematisch auf einmal geändert werden; Klicken Sie dazu auf Select Positions (Positionen auswählen), wählen Sie die zu ändernden Positionen aus oder aktivieren Sie das Häkchen, um alle Positionen auszuwählen, und klicken Sie dann auf Defocus aktualisieren und passen Sie die Parameter an (Abbildung 7A).
- Passen Sie die Bereiche "Fokus" und "Tracking" an (Abbildung 2C). Klicken Sie mit der linken Maustaste, um die Tracking- und Fokusbereiche (gelbe und blaue Kreise) zu ziehen. Stellen Sie sicher, dass der Tracking-/Fokusbereich (meistens) auf Kohlenstoff oder einem anderen geeigneten Tracking-Merkmal liegt, d. H. Ein ähnliches Merkmal wie die Region von Interesse; Vermeiden Sie Risse, Eiskontamination, zu dicke Bereiche und leere Löcher.
- Wenn Sie leeren Kohlenstoff ohne viele Tracking-Funktionen wählen, stellen Sie sicher, dass der Bereich auf halbem Weg durch das Tomogramm zu brennen beginnt, indem Sie eine ausreichend hohe Dosis für den Fokus wählen und die Probe bei höheren Neigungen langsam verbrennen. Dies kann vorteilhaft sein, um die Tracking-Genauigkeit zu erhalten. Stellen Sie sicher, dass der Tracking-/Fokusbereich keinen späteren Erfassungsbereich freilegt.
HINWEIS: Seien Sie vorsichtig, was in der Nähe ist und was in den Strahl kommt. Vermeiden Sie Bereiche mit Merkmalen, die in die Verfolgung und/oder Belichtung eingreifen, einschließlich Rasterbalken.
- Wenn Sie leeren Kohlenstoff ohne viele Tracking-Funktionen wählen, stellen Sie sicher, dass der Bereich auf halbem Weg durch das Tomogramm zu brennen beginnt, indem Sie eine ausreichend hohe Dosis für den Fokus wählen und die Probe bei höheren Neigungen langsam verbrennen. Dies kann vorteilhaft sein, um die Tracking-Genauigkeit zu erhalten. Stellen Sie sicher, dass der Tracking-/Fokusbereich keinen späteren Erfassungsbereich freilegt.
- Klicken Sie auf Position hinzufügen , sobald alle Parameter eingestellt und der gewünschte Interessenbereich sowie "Fokus" und "Tracking" definiert sind. Wiederholen Sie diesen Vorgang für neue Ziele.
HINWEIS: Um zu überprüfen, ob die euzentrische Höhe für die eingestellten Chargenpositionen korrekt kalibriert ist, stehen mehrere Strategien zur Verfügung. Es wird empfohlen, eine Tomographie-Sitzung basierend auf der Art der durchgeführten Tomographie-Sitzung (molekular, zellulär, Lamellen) zu wählen. - Für die molekulare Tomographie, vorausgesetzt, die Gitter sind ziemlich flach und euzentrische Höhe wurde in der Mitte jedes Quadrats durchgeführt, das Zielpositionen enthält, überspringen Sie das Verfahren "Alle verfeinern" (oder Auswahl verfeinern, wenn Positionen ausgewählt wurden, Abbildung 7A). Wenn Tomo mit der auto-euzentrischen Stufenneigungsroutine zu kämpfen hat, klicken Sie möglicherweise auf Euzentrisch überspringen.
- Bei Zell- oder Lamellenproben kann jede Chargenposition auf einer anderen z-Höhe liegen. Verwenden Sie entweder "Alle verfeinern", um vorsichtig zu sein, oder aktivieren Sie nicht die Option "Euzentrisch überspringen".
HINWEIS: "Alle verfeinern" durchläuft alle Tomogramme, die über "Positionen auswählen" eingerichtet oder ausgewählt wurden. Verfeinern Sie die euzentrische Höhe und führen Sie vor der Datenerfassung Tracking und Fokussierung durch. Dieses Verfahren kann jede überlappende Exposition aus dem nächsten Tomogrammfokus-/Tracking-Bereich (Abbildung 7B) aufdecken, die vor dem Fortfahren berücksichtigt werden sollte. - Überprüfen und besuchen Sie die Quadrate, bei denen die Verfeinerung fehlgeschlagen ist. Klicken Sie mit der rechten Maustaste auf die fehlerhafte Position , um Optionen anzuzeigen. Wenn die euzentrische Höhe fehlschlägt, suchen Sie nach "Auto-euzentrisch durch Bühnenneigung" (Schritt 1.4.1), erfassen Sie ein neues "Suchen"-Bild, um die z-Höhe zu aktualisieren, und "Position hinzufügen". Löschen Sie die fehlgeschlagene/zuvor initialisierte Position. Klicken Sie auf die Option Euzentrisch überspringen , was mit "Alle verfeinern" geschehen ist.
HINWEIS: Wenn "Alle verfeinern" nicht ausgeführt wird, darf "Euzentrisch überspringen" nicht aktiviert sein. Tomo5 führt eine euzentrische Verfeinerung durch, bevor jedes Tomogramm erfasst wird. Auf diese Weise werden Positionen, die an der euzentrischen Verfeinerung scheitern, übersprungen.
- Bei Zell- oder Lamellenproben kann jede Chargenposition auf einer anderen z-Höhe liegen. Verwenden Sie entweder "Alle verfeinern", um vorsichtig zu sein, oder aktivieren Sie nicht die Option "Euzentrisch überspringen".
- Erstellen Sie die Einrichtung der Tomographie-Datenerfassung auf der Registerkarte "Tomographie".
- Führen Sie automatische Funktionen aus, indem Sie die folgenden Schritte ausführen.
- Überprüfen Sie die Einstellungen, um die Ausrichtung über die Registerkarte "Autofunktionen" durchzuführen, indem Sie im Atlas zu einem Bereich aus Kohlenstoff navigieren. Bringen Sie diesen Bereich auf euzentrische Höhe und folgen Sie der Reihenfolge der Ausrichtungen, wie unten angegeben.
- Führen Sie Auto-euzentrisch durch Stufenneigung mit der Voreinstellung "Euzentrische Höhe" aus.
- Führen Sie die Autofokus-Routine mit der Voreinstellung "Fokus" aus.
- Führen Sie die Autostigmate-Routine mit der Voreinstellung "Thon Ring" aus.
- Führen Sie die Autocoma-Routine mit der Voreinstellung "Thon Ring" aus.
- Fügen Sie die gewünschte Objektivblende (Abbildung 6B), wahrscheinlich die 100-μm-Blende, als guten Kompromiss für einen erhöhten Abtastkontrast und nur einen kleinen Signalschnitt bei sehr hoher Auflösung ein.
- Wiederholen Sie die Autostigmate-Routine nach dem Einsetzen der Blende.
HINWEIS: Bei Vergrößerungen mit Pixelgrößen um 3 Å und niedrigeren Auflösungen schlägt die Autocoma-Routine möglicherweise fehl. Aktivieren und richten Sie in diesem Fall Tomo Rotation Center unter Direkte Ausrichtung in der TEM-Benutzeroberfläche aus.
- Überprüfen Sie, ob die Auto-Zero-Loss-Zentrierroutine an Ihrer Probe funktioniert. Mit der Voreinstellung "Zero Loss" bei einer relativ hohen Dosis (Schritt 1.2.3) wird die Routine in Auto Functions eher erfolgreich sein.
- Überprüfen Sie die Einstellungen, um die Ausrichtung über die Registerkarte "Autofunktionen" durchzuführen, indem Sie im Atlas zu einem Bereich aus Kohlenstoff navigieren. Bringen Sie diesen Bereich auf euzentrische Höhe und folgen Sie der Reihenfolge der Ausrichtungen, wie unten angegeben.
- Führen Sie die Datenerfassung durch.
- Um die automatisierte Erfassung auf der Registerkarte "Tomographie" zu starten, wählen Sie die Datenerfassungsbramme aus und stellen Sie die gewünschten Parameter ein (Abbildung 7C). Richten Sie die Datenerfassungsparameter ein: Neigungsschritt (°), Max. Positiver Winkel (°), Max. negativer Winkel (°), Tracking-Schema (empfohlen: Wählen Sie Spur/Fokus vor Aufnahme). Wählen Sie Säulenventile schließen für euzentrisches Höhenschema.
HINWEIS: Selten verwendete Parameter sind "Belichtungszeit anpassen", "ZLP anpassen", "Phasenplatte verwenden" und "Pause After Tilt".- Verwenden Sie die Option Belichtungszeit anpassen für Tomogramme, die nicht für STA vorgesehen sind, um die Belichtungszeit bei einem bestimmten Verhältnis bei höheren Neigungen zu erhöhen.
- Die Option ZLP anpassen führt nach jedem Tomogramm eine ZLP-Verfeinerung durch, unabhängig von der eingestellten Periodizität. Es ist langsam, scheitert gelegentlich ohne offensichtliche Gründe und wird diese Positionsübernahme überspringen. Es kann jedoch für sehr schmale Spaltbreiten nützlich sein, d.h. 3-5 eV auf einem Selectris(X)-Filter.
- Use Phase Plate wird seit der Einführung von DEDs mit Energiefiltern selten verwendet.
- Pause After Tilt pausiert zwar die Erfassung, erlaubt aber keine Änderungen an der Software.
- Überprüfen Sie die Datenerfassungsparameter (auf der Registerkarte "Vorbereitung"), stimmen Sie der Dosisberechnung und dem gewünschten Neigungsschema zu, geben Sie die Anzahl der Fraktionen (Nr.) in der Voreinstellung "Belichtung" an und beginnen Sie dann mit der Erfassung.
HINWEIS: Die Anzahl der Fraktionen hängt von der Probe, den Erfassungsparametern und den geplanten Nachbearbeitungsschritten ab, d. H. STA vs. morphologische Analyse, und muss auf Werte gehalten werden, bei denen Bewegungskorrektur und CTF-Schätzung genügend Signal erhalten. Ein Bereich von 4-10 Fraktionen ist eine gute Schätzung, wobei 4 für dickere Proben und 10 für dünnere molekulare Proben besser geeignet ist.- Klicken Sie dann auf Start , um mit der Datenerfassung zu beginnen und die erste Tomogrammerfassung zu überwachen, um sicherzustellen, dass die Tomogramme wie vorgesehen erfasst werden.
HINWEIS: Neuere Tomographie-Softwareversionen verfügen über eine zusätzliche Movie Player-Slab-Out in der Registerkarte "Tomographie", wo die erfassten Tomogramme angesehen werden können. Seien Sie geduldig während des Ladevorgangs.
- Klicken Sie dann auf Start , um mit der Datenerfassung zu beginnen und die erste Tomogrammerfassung zu überwachen, um sicherzustellen, dass die Tomogramme wie vorgesehen erfasst werden.
- Um die automatisierte Erfassung auf der Registerkarte "Tomographie" zu starten, wählen Sie die Datenerfassungsbramme aus und stellen Sie die gewünschten Parameter ein (Abbildung 7C). Richten Sie die Datenerfassungsparameter ein: Neigungsschritt (°), Max. Positiver Winkel (°), Max. negativer Winkel (°), Tracking-Schema (empfohlen: Wählen Sie Spur/Fokus vor Aufnahme). Wählen Sie Säulenventile schließen für euzentrisches Höhenschema.
2. Kryo-ET STA von Apoferritin unter Verwendung von emClarity
HINWEIS: Hier wird die emClarity-Software17 verwendet, um die Kryo-ET-Strukturbestimmung durch STA zu veranschaulichen. Abbildung 9 fasst diesen Prozess zusammen. Als Beispiel wurden sechs Kippserien von Apoferritin (EMPIAR-10787) genommen. Oktaedersymmetrie wurde angewendet, und die endgültige Karte hatte eine Auflösung von 2,86 Å, die aus nur 4.800 Teilchen gewonnen wurde und nahe der Nyquist-Frequenz (2,68 Å) lag.
- Stellen Sie sicher, dass das emClarity-Softwarepaket39 heruntergeladen und installiert ist (siehe Materialtabelle). Installieren Sie IMOD für die Softwareverarbeitung40.
HINWEIS: Grundkenntnisse der Linux-Befehle sind erforderlich. Ein detaillierteres Tutorial finden Sie in Referenz41, das am Beispiel eines Ribosomendatensatzes (EMPIAR-10304) die Schritt-für-Schritt-Vorgehensweise beschreibt. Für verschiedene Arten von Proteinproben ist auch ein zuvor veröffentlichter Bericht verfügbar42. - Bereiten Sie die Eingabedateien und -verzeichnisse vor.
HINWEIS: Die Rohbilder wurden von MotionCor2 (Patch 5 x 5)43 bewegungskorrigiert (siehe Materialtabelle). Die bewegungskorrigierten Bilder wurden gestapelt, um die Tilt-Serie mit Newstack (IMOD) zu generieren, und manuell mit Patch-Tracking mit Etomo ausgerichtet (siehe Materialtabelle), gefolgt von emClarity. Um eine bessere Ausrichtung zu erhalten, wird empfohlen, Offsets und Neigungskompensationen in Etomo durchzuführen. Die sechs Neigungsserien werden in TS1 in TS6 umbenannt. Im Folgenden finden Sie die Anweisungen und Befehle für die erste Neigungsserie (TS1) als Beispiel.- Richten Sie einen Projektordner ein.
HINWEIS: Die neueste Version der Software ist 1.5.3.11. Alle zugehörigen Softwareprotokolle befinden sich im lokalen Projektordner (.../logFile/emClarity.logfile). - Erstellen Sie unter dem Projektordner einen weiteren neuen Ordner, "fixedStacks", und bereiten Sie die Eingabedateien vor: TS1.fixed, TS1.xf und TS1.tlt.
HINWEIS: TS1.fixed: die ursprüngliche Tilt-Serie, in Etomo auch TS1.st genannt. TS1.xf: Diese Datei enthält die Transformationskoordinaten nach Etomo tiltalign. TS1.tlt: Diese Datei enthält Neigungswinkel.
- Richten Sie einen Projektordner ein.
- Schätzen Sie die Unschärfe anhand der folgenden Schritte.
- Berechnen Sie die Unschärfe. Aktualisieren Sie die Parameterdatei mit den Mikroskop- und Bildgebungsparametern gemäß Zusatztabelle 1. Kopieren Sie eine Parameterdatei in den Projektordner, ändern Sie ihren Namen in param_ctf.m, und führen Sie Folgendes aus: emClarity ctf estimate param_ctf.m TS1.
HINWEIS: Die Parameterdateivorlage befindet sich in der Zusatzdatei 1 oder im lokalen Softwareinstallationsverzeichnis (.../emClarity_1.5.3.11/docs/exampleParametersAndRunScript/).
- Berechnen Sie die Unschärfe. Aktualisieren Sie die Parameterdatei mit den Mikroskop- und Bildgebungsparametern gemäß Zusatztabelle 1. Kopieren Sie eine Parameterdatei in den Projektordner, ändern Sie ihren Namen in param_ctf.m, und führen Sie Folgendes aus: emClarity ctf estimate param_ctf.m TS1.
- Überprüfen Sie die CTF-Schätzergebnisse für jeden Stack.
- Überprüfen Sie die ausgerichteten Stapel (z. B. aliStacks/TS1_ali1.fixed) mit 3dmod und stellen Sie sicher, dass die Passerperlen korrekt gelöscht werden.
- Stellen Sie sicher, dass die Protokolldatei die Händigkeit meldet, die in logfile/emClarity.logfile zu finden ist (Abbildung 9).
- Überprüfen Sie den Defokussierungswert (z. B. fixedStacks/ctf/TS_ali1_psRadial_1.pdf), und stellen Sie sicher, dass er mit der theoretischen CTF-Schätzung übereinstimmt.
- Definieren Sie die Unterbereiche.
- Generieren Sie ein Behälter-Tomogramm. Führen Sie im Projektordner Folgendes aus: sh recScript2.sh -1.
HINWEIS: Ein kleineres (in der Größe) Tomogramm für jeden Stapel wird in einem neuen Ordner "bin10" gespeichert. Um den Prozess zu beschleunigen, können die Koordinaten eines oder mehrerer Teilbereiche in einem bin10-Tomogramm definiert werden. Die Skriptdatei befindet sich im lokalen Softwareinstallationsverzeichnis (.../emClarity_1.5.3.11/docs/). - Bestimmen Sie die Grenzen, indem Sie sechs Punkte auswählen: x min, x max, ymin, y max, zmin und z max, um einen Unterbereich zu erstellen. Führen Sie unter dem Ordner "bin10" Folgendes aus: 3dmod TS1_bin10.rec.
HINWEIS: Wenn vier Teilbereiche in einer Neigungsreihe erstellt werden sollen, wählen Sie 6 × 4 = 24 Punkte. Jeder Stack muss eine Modelldatei (*.mod) im Ordner "bin10" haben. - Konvertieren Sie die Modelldateien in das emClarity-Format. Dadurch wird eine neue Ordneraufklärung erstellt. Speichern Sie alle Koordinaten jeder Unterregion in diesem Ordner. Führen Sie im Projektordner Folgendes aus: sh recScript2.sh TS1.
- Generieren Sie ein Behälter-Tomogramm. Führen Sie im Projektordner Folgendes aus: sh recScript2.sh -1.
- Partikel auswählen.
- Finden Sie eine Apoferritin-Vorlage zum Sammeln von Partikeln. Laden Sie eine Vorlage aus der Elektronenmikroskopie-Datenbank (EMD-10101)44 herunter. Stellen Sie sicher, dass die Pixelgröße der Vorlage mit der der Rohdaten übereinstimmt. Führen Sie im Projektordner Folgendes aus: emClarity rescale ApoF.mrc ApoF_Template_rescale.mrc 3.60 1.34 cpu.
- Generieren Sie CTF-korrigierte Tomogramme für die Partikelkommissionierung. Führen Sie im Projektordner Folgendes aus: emClarity ctf 3d param_ts.m templateSearch.
- Führen Sie die Partikelauswahl für jeden Unterbereich durch. Führen Sie für den Apoferritin-Datensatz eine Vorlagensuche bei bin6 durch. Ändern Sie die Tmp_angleSearch Parameter (θ out, Δout, θ in, Δin), um den Winkelbereich und die Intervalle für die Suche in Grad innerhalb oder außerhalb der Ebene zu bestimmen. Führen Sie im Projektordner Folgendes aus: emClarity templateSearch param_ts.m TS1 1 ApoF_Template_rescale.mrc O 1.
HINWEIS: In diesem Schritt wird ein Ordner "convmap_wedge_Type2_bin6" generiert. Die CSV-Datei unter diesem Ordner (z. B. TS1_1_bin6.csv) enthält alle Informationen über die ausgewählten Partikel. - Entfernen Sie die falschen Partikel mit 3dmod. Führen Sie unter dem Ordner "convmap_wedge_Type2_bin6" aus: 3dmod .. /cache/TS1_1_bin6.rec TS1_1_bin6.mod.
HINWEIS: Es ist üblich, falsche Partikel neben Kohlenstoffkanten oder Eisverunreinigungen in diesen Bereichen zu finden. Führen Sie einen rechten Mausklick auf die falschen Punkte durch und drücken Sie die Rücktaste auf der Tastatur, um sie zu löschen. Stellen Sie in diesem Schritt sicher, dass die meisten falsch positiven Punkte entfernt werden (Abbildung 10). - Ändern Sie den Ordnernamen "convmap_wedge_Type2_bin6" in "convmap", da emClarity in den folgenden Schritten Informationen zu Unterregionen unter convmap findet.
- Initialisieren Sie das Projekt. Führen Sie im Projektordner emClarity init param0.m aus, um eine Datenbank für emClarity, ApoF.mat, zu erstellen.
- Führen Sie Tomogramme vor STA und Ausrichtung durch. Um CTF-korrigierte Subregion-Tomogramme in bin4 zu generieren, führen Sie Folgendes aus: emClarity ctf 3d param0.m.
HINWEIS: CTF-korrigierte Tomogramme (z.B. cache/TS1_1_bin4.rec) werden in einem neuen Ordner "cache" generiert. - Führen Sie STA und Ausrichtung durch.
- Führen Sie die Mittelwertbildung mit den CTF-korrigierten Subtomogrammen ab bin4 durch. Führen Sie im Projektordner Folgendes aus: emClarity avg param0.m 0 RawAlignment.
- Führen Sie die Ausrichtung fort und führen Sie Folgendes aus: emClarity alignRaw param0.m 0.
HINWEIS: Der Mittelwertbildungsschritt generiert eine Referenz, die von emClarity in diesem Schritt verwendet wird, um die Partikel auszurichten. Die Raw_angleSearch Parameter (θ out, Δout, θ in, Δin) können geändert werden, um den Winkelbereich und die Schrittweiten für jeden Zyklus einzustellen. Da die meisten falschen Partikel aus der Datenbank entfernt werden (Schritt 2.6.4), können diese Winkeleinstellungen für die Ausrichtung mit einem relativ kleinen Grad beginnen. - Aktualisieren Sie die Raw_angleSearch Parameter und führen Sie einige weitere Zyklen durch (Schritte 2.9.1 und 2.9.2). Um den Prozess zu beschleunigen, führen Sie In-Plane- und Out-of-Plane-Ausrichtungen separat durch.
HINWEIS: Für jedes Binning wird empfohlen, mehrere Zyklen auszuführen und die Winkeleinstellungen schrittweise zu reduzieren. Für den ApoF-Datensatz wurden fünf weitere Zyklen in der Ablage 4 durchgeführt, weitere Details sind in der Zusatztabelle 2 verfügbar. Führen Sie für cycle001 im Projektordner Folgendes aus: emClarity avg param1.m 1 RawAlignment, gefolgt von emClarity alignRaw param1.m 1. - Reinigen Sie die überlappenden Partikel. Führen Sie im Projektordner Folgendes aus: emClarity removeDuplicates param5.m 5.
- Führen Sie eine Verfeinerung der Tilt-Serie mit tomoCPR durch.
HINWEIS: Dieser Schritt ist optional.- Führen Sie tomoCPR aus, um die Stapelgeometrie zu verfeinern. Führen Sie im Projektordner Folgendes aus: emClarity tomoCPR param5.m 5.
- Generieren Sie neu ausgerichtete Stapel und aktualisieren Sie die Geometriedateien. Führen Sie im Projektordner Folgendes aus: emClarity ctf update param6.m.
HINWEIS: Stellen Sie sicher, dass die neuen Geometriedateien (z. B. fixedStacks/ctf/TS1_ali2_ctf.tlt) und die neu ausgerichteten Stapel (z. B. aliStacks/TS1_ali2.fixed) korrekt generiert werden. - Erstellen Sie neue Sub-Tomogramme in bin2. Führen Sie im Projektordner Folgendes aus: emClarity ctf 3d param6.m.
HINWEIS: Es folgen einige Zyklen für die Mittelwertbildung und Ausrichtung in bin2 (Schritte 2.9.1, 2.9.2 und 2.9.3), die Duplikatreinigung (Schritt 2.9.4) und die optionale tomoCPR (Schritt 2.10). Aufgrund der hohen Symmetrie wurden weitere fünf Zyklen an bin2 für Apoferritin durchgeführt. Aktualisieren Sie die Raw_angleSearch Parameter für jeden Zyklus.
- Führen Sie die endgültige Rekonstruktion durch.
- Führen Sie einige Zyklen bei bin1 und tomoCPR fort (Schritt 2.9 und Schritt 2.10). Weitere 10 Zyklen bei bin1 wurden für den Apoferritin-Datensatz durchgeführt. Weitere Details zu den Befehlen und Parametern finden Sie in Tabelle 2.
- Führen Sie die endgültige Rekonstruktion durch, indem Sie die beiden halben Datensätze kombinieren. Führen Sie im Projektordner Folgendes aus: emClarity avg param21.m 21 RawAlignment, gefolgt von emClarity avg param21.m 21 FinalAlignment.
HINWEIS: Die endgültige Karte (z. B. mit einem B-Faktor von 10, cycle021_ApoF_class0_final_bFact-10.mrc) wird generiert, Abbildung 11.
Ergebnisse
Bei Zell- und Lamellenproben hängt die Datenerhebungsstrategie weitgehend von der Probe und dem Ziel der Bildgebungsstudie ab (Abbildung 1). Der Targeting-Ansatz hängt davon ab, ob das molekulare Target in situ ist oder aus dem gereinigten makromolekularen Komplex für hochauflösende Rekonstruktionsproben mit molekularen Targets hergestellt wird. Für gereinigte Komplexe, die auf löchrigen (Kohlenstoff-) Gittern verglast sind, kann das Targeting einfach auf der Bildgebung in den Löchern der (Kohlenstoff-) Stützfolie basieren. Für In-situ-Arbeiten erfordert der Targeting-Ansatz die Kenntnis der Position der molekularen Entität basierend auf korrelativen Daten oder bekannten zellulären Landmarken mit geringer Vergrößerung. Zelluläre Landmarken wären idealerweise bei der Aufnahme von Übersichtsbildern identifizierbar und können, wenn sie ausreichen, um Regionen von Interesse grob zu lokalisieren, eine schnelle Möglichkeit bieten, die Zielidentität mit einem Suchbild zu bestätigen. Wenn die beobachtbaren Ereignisse jedoch selten sind, können Suchbilder mit mittlerer Vergrößerung erforderlich sein, um zu qualifizieren, dass das Ziel korrekt ist. Suchkarten sind Montagen mit mittlerer Vergrößerung von Suchbildern und können somit die Zielsuche erheblich erleichtern, wo sie bei einer Vergrößerung erfasst werden können, bei der das interessierende Merkmal sichtbar ist. Suchkarten können dann durchleuchtet werden, um Zielstapelpositionen zu finden und einzurichten. Für Proben, die zelluläre Merkmale für die ultrastrukturelle Rekonstruktion enthalten, ist der Targeting-Ansatz ähnlich, hängt jedoch gleichermaßen von der Sichtbarkeit des zellulären Ereignisses bei verschiedenen Vergrößerungen und seiner Prävalenz in der Probe ab.
Die Datenerfassungsstrategie muss ebenfalls berücksichtigt werden; In allen Fällen bestimmt das Ziel der Studie maßgeblich, wie die Daten erhoben werden. Für die Rekonstruktion der zellulären Ultrastruktur kann eine geringe Vergrößerung (20-5 Å / px) und ein großes Sichtfeld angebracht sein, aber hohe Vergrößerungen sind erforderlich, um molekulare oder hochauflösende Details (5-1 Å / px) zu rekonstruieren. Ein Datensatz, der bei 1,5 Å/px unter idealen Bedingungen gesammelt wurde, wird physikalisch nur in der Lage sein, eine Rekonstruktion bei der Nyquist-Frequenz von 3,0 Å/px zu erzeugen; In Wirklichkeit beeinflussen jedoch viele Faktoren, einschließlich, aber nicht beschränkt auf Probendicke, Größe und Heterogenität, die erhaltene Rekonstruktionsqualität. Die genauen Bildgebungsparameter gleichen auch die Vergrößerung basierend auf dem Ziel der Studie aus, wobei das Sichtfeld genügend Informationen enthält. Dieser Artikel stellt ein Subtomogramm vor, das einen durchschnittlichen Fall von 2,86 Å erreicht, aber zusätzliche Studien 17,42,43,44,45 sind in Tabelle 1 dargestellt, um die Sammlungsparameter zu veranschaulichen, die mit Studien verbunden sind, die auf verschiedene Endpunkteabzielen 17,42,45,46.
Sobald ein Targeting-Workflow und ein Datenerfassungsregime für Kryo-ET etabliert sind, ist die Datenerfassung vieler verschiedener Probentypen möglich. Repräsentative Tomogramme einer Vielzahl von Proben werden hier vorgestellt: molekulare Proben wie Apoferritin (Film 1), dünnzellige Prozesse (Film 2) und FIB gefräste Lamellen der dickzelligen Probe (Film 3).
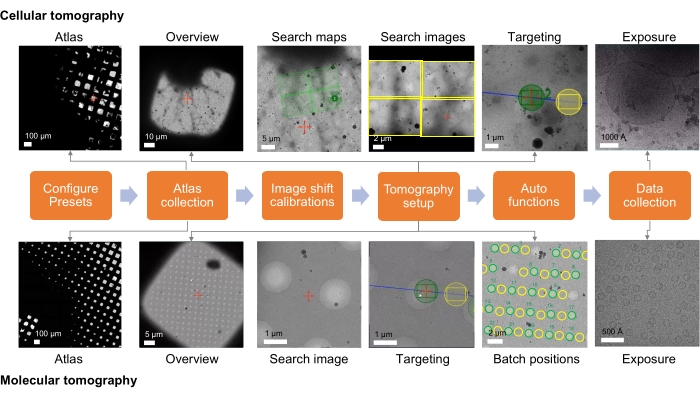
Abbildung 1: Überblick über den Aufbau des Tomographie-Workflows. Der im Protokoll beschriebene Kryo-ET-Imaging-Workflow wird als Flussdiagramm dargestellt. Die zu erwartenden Bilder werden für den zellulären und molekularen Workflow gezeigt. Die vorgestellte Benennung folgt der Tomo5-Konvention, obwohl die meisten Tomographie-Erfassungssoftware gemeinsame Prinzipien für die Erfassung dieser Bilder hat. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
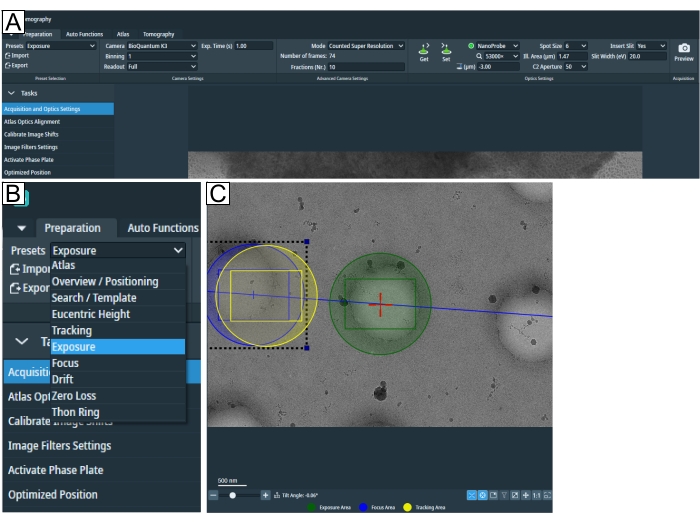
Abbildung 2: Registerkarte "Vorbereitung" und voreingestellte Suchbedingungen . (A) Übersichtsbild der gesamten Registerkarte. In dieser Registerkarte werden die Voreinstellungen für Bildbedingungen festgelegt, und die Einstellungen "Bildverschiebung kalibrieren" und "Bildfilter" finden Sie im Dropdown-Menü "Aufgaben". (B) Vergrößern des Dropdown-Menüs "Presets", in dem jedes Preset für die Einrichtung individueller Bildbedingungen ausgewählt werden kann. (C) Bild mit einer ausreichenden Vergrößerung, um sowohl die Belichtung als auch den Bereich "Fokus und Tracking" in das Sichtfeld zu passen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
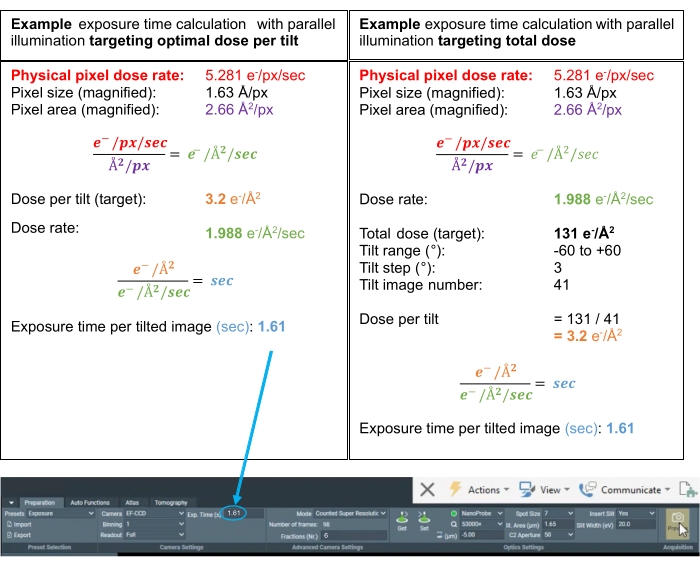
Abbildung 3: Dosisberechnungen. Beispieldosisberechnungen für mögliche Tomographie-Erfassungsschemata, bei denen die Dosisleistung über Vakuum gemessen wurde. Die beiden Berechnungen bestimmen die Belichtungszeit(en) für jede Neigung, unabhängig davon, ob sie auf eine optimale Dosis pro Neigung oder eine optimale Gesamtdosis für die Full Tilt-Serie abzielen. Bei der Subtomogramm-Mittelwertbildung ist es üblich, eine optimale Dosis pro gekipptem Bild im Bereich von 3,0-3,5 e-/Å2 anzustreben. In beiden Fällen werden die "Fraktionen (Nr.)" auf 6 gesetzt, um ~0,5 e-/Å2 Dosis pro Filmbild der Neigung für ein ausreichendes Signal zur Bewegungskorrektur zu erreichen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
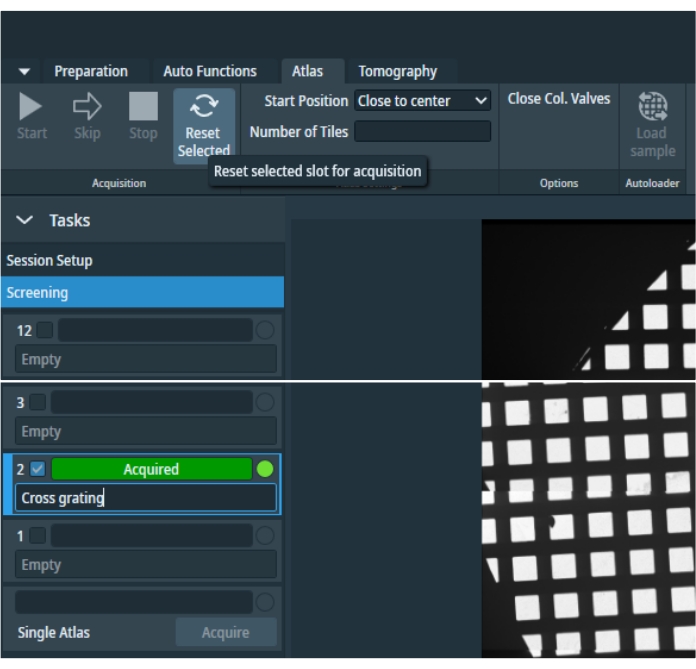
Abbildung 4: Registerkarte Atlas. Übersichtsbild der Registerkarte "Atlas". Das Bild wurde zugeschnitten, um leere Rasterpositionsräume zu vermeiden. Das Menü "Aufgaben" enthält Einstellungen für die Sitzungseinrichtung und Rasterauswahlbereiche für die individuelle Auswahl aller inventarisierten Raster und eine Option zum Abrufen eines einzelnen Atlas, nachdem die Kassette aus dem Autoloader entfernt wurde. Ein ausgewähltes Raster kann zurückgesetzt und dann wieder erfasst werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
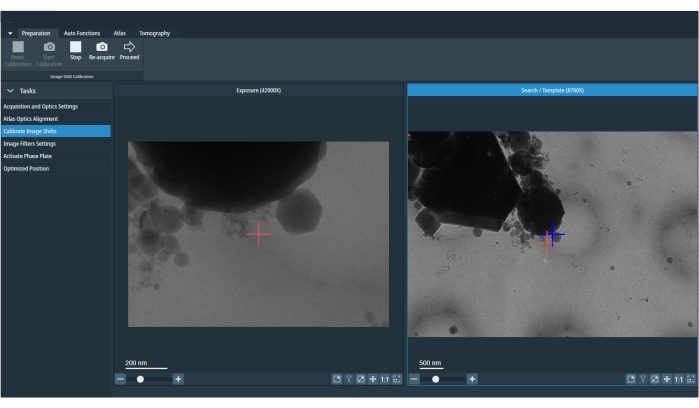
Abbildung 5: Kalibrierte Bildverschiebungen. Das Bild zeigt eine Belichtung und ein Suchbild. Das rote Kreuz im Suchbild ist die verschobene Markierung, um den Versatz zwischen Belichtung und Suche zu korrigieren. Man sollte die Kalibrierung der Bildverschiebung zu Beginn der Sitzung oder nach dem Ändern der Voreinstellungen für die Bildgebungsbedingungen wiederholen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
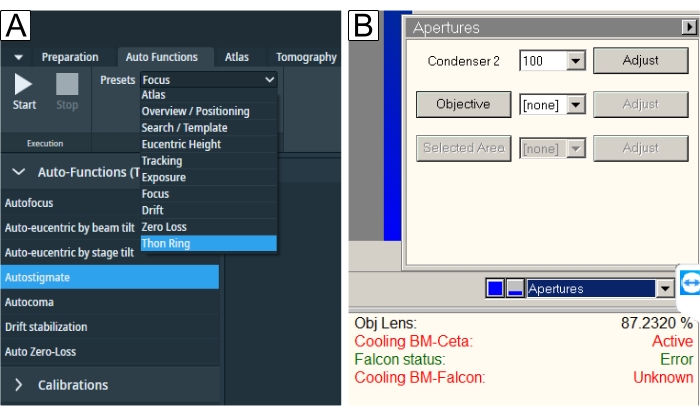
Abbildung 6: Autofunktionen und Blenden. (A) Die Registerkarte "Auto-Funktionen" zeigt das Dropdown-Menü "Presets" und die Auswahl "Task". Blau unterstrichen ist die Voreinstellung "Thon Ring", die für "Autostigmate" (auch blau unterstrichen) und "Autocoma" erforderlich ist. Man sollte für jede Aufgabe entsprechende Voreinstellungen auswählen und dann die Starttaste drücken. (B) Blenden befinden sich in der TEM-Benutzeroberfläche. Wählen Sie die gewünschte "Objektivblende", nachdem die Autofunktionen ausgeführt wurden, und führen Sie "Autostigmate" mit der Blende aus. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
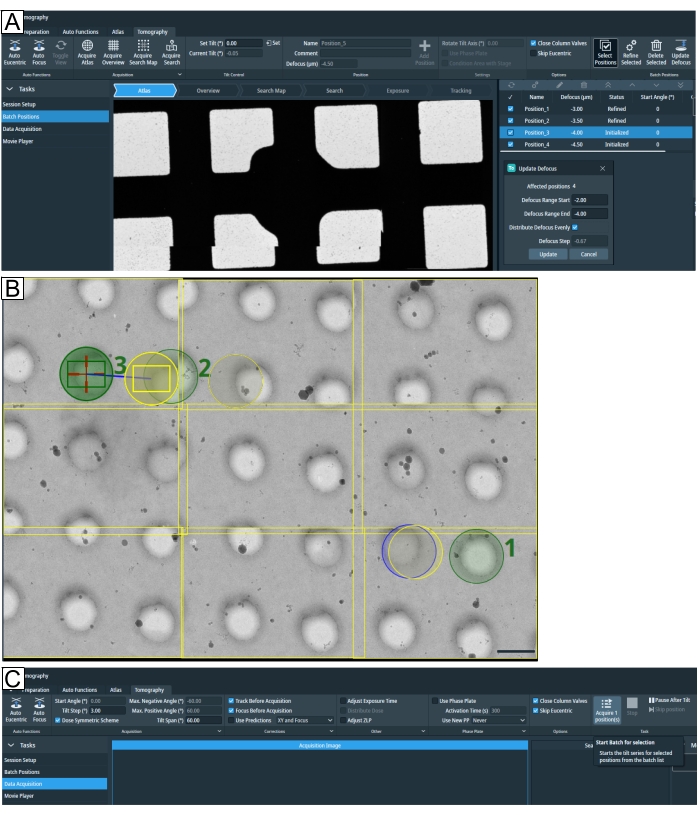
Abbildung 7: Übersicht über die Registerkarte Tomografie. Bilder zeigen die Benutzeroberfläche von Tomo 5.8. (A) Chargenpositionen. Die neuesten Funktionen, die im Bild dargestellt sind: die Option "Suchkarte erfassen"; Tomogrammpositionen werden in der Atlasansicht mit der Zoom-in-Option angezeigt. oben rechts hervorgehoben ist "Positionen auswählen"; unten wurden alle vier Positionen zu den Parametern "Defokussierung aktualisieren" ausgewählt. (B) Auf der Suchkarte werden drei Positionen mit den Bezeichnungen 1, 2 und 3 ausgewählt. Position 1 stellt kein Problem dar, wenn "Alle verfeinern" ausgeführt wird. Wenn jedoch in einer Einstellung mit hoher Zieldichte, z. B. wenn Lamellenpositionen ausgewählt werden, wie für Position 2 und Position 3 dargestellt, führt "Alle verfeinern" die Routine "Tracking" und "Fokus" für den Bereich "Belichtung" von Position 2 aus und legt das Ziel frei, bevor das Tomogramm erfasst wird. Der Rastertyp ist R1.2/1.3. Maßstabsbalken = 1,2 μm. (C) Datenerfassung. Ausgewählte Positionen, die in "Chargenpositionen" eingerichtet sind, können nun individuell erfasst werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
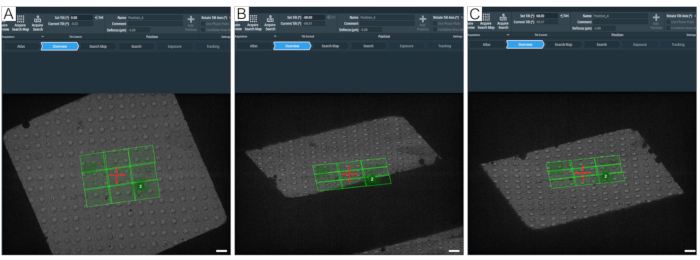
Abbildung 8: Definition des maximalen Neigungswinkels. Die Abbildung zeigt einen schrittweisen Ansatz zur Bestimmung des maximalen Neigungsbereichs für Tomogrammaufnahmen. (A) 0° Übersicht und eine Suchkarte in der Mitte des Rasterquadrats. Um den Neigungsbereich zu testen, kann der Winkel in der Tomographie-Software mit "Neigung einstellen (°)" eingestellt werden, indem der gewünschte Wert eingegeben und anschließend auf Einstellen gedrückt wird. (B) Die Bühne wurde auf −60° geneigt, was zeigt, dass eine Ecke der Suchkarte bei −60° nicht vollständig erfasst wird. (C) Die Bühne wurde um 60° geneigt. Durch das Zählen der verschwundenen Löcher bei ±60° kann man sich ein Bild vom Neigungsbereich eines Rasterquadrats machen. Maßstabsbalken = 2,5 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
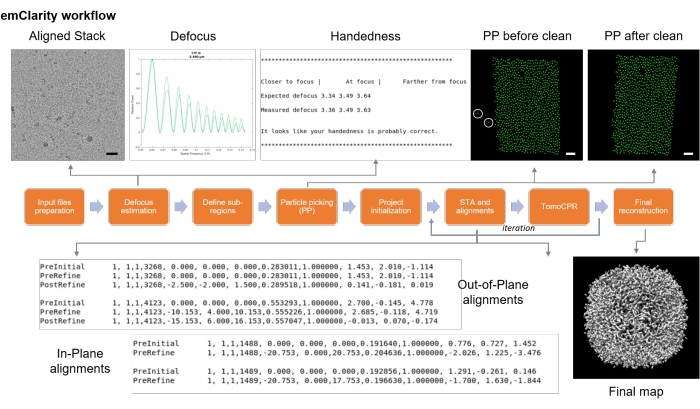
Abbildung 9: emClarity-Flussdiagramm. Das Flussdiagramm beschrieb die verschiedenen Schritte für die Kryo-Sub-Tomogramm-Mittelung. Maßstabsbalken = 50 nm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 10: Vorlagenabgleich mit emClarity . (A) Eine typische Mikroaufnahme von Apoferritin auf einem graphenbeschichteten Gitter. Defokussierung: −3,430 μm. (B) Ein Ausschnitt des Tomogramms, der nach der Vorlagensuche mit Modellpunkten überlagert wird. (C) Eine obere und (D) eine Seitenprojektionsansicht von Modellpunkten, die eine einzelne flache Schicht monodisperser Apoferritinpartikel anzeigen. Maßstabsbalken = 50 nm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
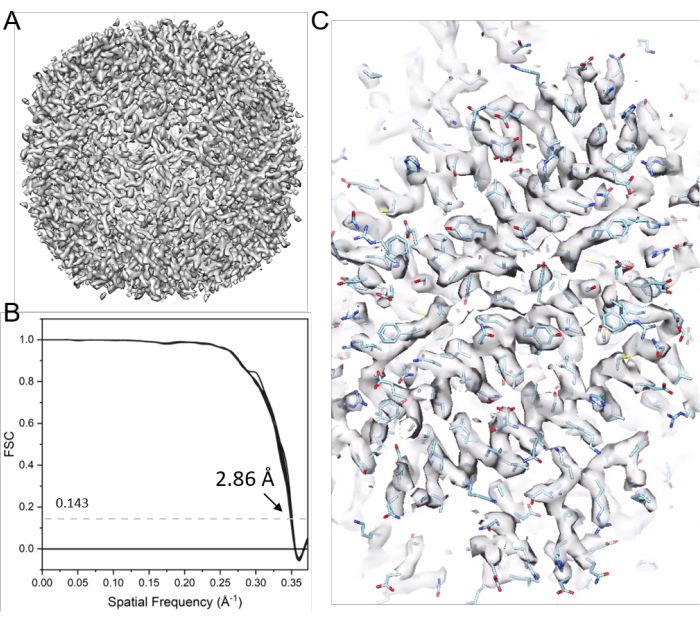
Abbildung 11: Kryo-ET-STA von Apoferritin . (A) Die endgültige Karte nach 21 Zyklen der Sub-Tomogramm-Ausrichtung. (B) Das Fourier-Shell-Korrelationsdiagramm (FSC) der endgültigen Karte mit einer gemeldeten Auflösung von 2,86 Å, das 38 Kegel-FSCs enthält. (C) Repräsentative Dichtekarten (ausgestattet mit PDB-Modell 6s6147). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
| Probe | Kryo-ET-Typ | Å/px | Neigungsbereich (+/-) | Kippstufe (°) | Unschärfebereich (μm) | Gesamtdosis (e-/Å2) | Auflösung | Rohdaten | Referenz |
| Apoferritin | Gereinigt/Molekular (STA) | 1.34 | 60 | 3 | 1.5 – 3.5 | 102 | 2.86 | EMPIAR-10787 | 17 & dieses Papier |
| HIV-1-Knebel | Gereinigt/Molekular (STA) | 1.35 | 60 | 3 | 1.5 – 3.96 | 120 | 3.1 | EMPIAR-10164 | 17 |
| Ribosom | Gereinigt/Molekular (STA) | 2.1 | 60 | 3 | 2.2 – 4.3 | 120 | 7 | EMPIAR-10304 | 42 |
| SARS-CoV-2-Spitze | Lamellen/Moleküle (STA) | 2.13 | 54 | 3 | 2 – 7 | 120 | 16 | EMPIAR-10753 | 45 |
| Neuronenaxonstruktur | Zellulär (ultrastrukturell) | 5.46 | 60 | 2 | 3.5 – 5 | 90 | N.D. | EMPIAR-10922 | 47 |
Tabelle 1: Sammelparameter für mehrere Kryo-ET-Studien. Studien, die auf die Rekonstruktion molekularer Details aus gereinigten oder in situ Proteinen abzielen, verglichen mit einer Studie, die darauf abzielt, die ultrastrukturellen zellulären Merkmale aufzulösen und zu segmentieren.
Film 1: Ein Tomogramm von Apoferritin-Proben auf einem normalen EM-Gitter und dann mit einem Kryo-TEM, das mit einer ultrahochauflösenden Kamera mit einem kompatiblen Filter ausgestattet ist. Die Tilt-Serien wurden mit einem dosissymmetrischen Schema mit einer Neigungsspanne von 54° und einer Gesamtdosis von 134 e-/A2in der Elektronentomographie-Software erfasst. Maßstabsbalken = 50 nm. Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 2: Ein Tomogramm eines primären Neurons, das auf einem EM-Gitter gezüchtet und dann direkt mit einem Kryo-TEM abgebildet wurde, das mit einer ultrahochauflösenden Kamera mit einem kompatiblen Filter ausgestattet ist. Die Neigungsserien wurden dosissymmetrisch mit einer Neigungsspanne von 60° und einer Gesamtdosis von 120 e-/A2in der Elektronentomographie-Software erfasst. Maßstabsbalken = 100 nm. Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 3: Ein Tomogramm eines Cyanobakteriums auf einem EM-Gitter, das einer FIB-Fräsung unterzogen und dann mit einem Kryo-TEM abgebildet wurde, das mit einer Hochgeschwindigkeitskamera und einem kompatiblen Filter ausgestattet ist. Die Neigungsserien wurden mit einem dosissymmetrischen Schema mit einer Neigungsspanne von 50° und einer Gesamtdosis von 120 e-/A2in der Elektronentomographie-Software erfasst. Maßstabsbalken = 87,2 nm. Bitte klicken Sie hier, um diesen Film herunterzuladen.
Zusatzdatei 1: Die Parameterdateivorlage zur Schätzung der Unschärfe. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle 1: Details zur Datenerhebung und zum Aufbau des Mikroskops. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Zusatztabelle 2: Liste der Befehle in der Reihenfolge ihrer Ausführung. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Tomo5
Die Workflow-Beschreibung der Tomographie-Software zeigt einen möglichen und optimierten Weg für eine (Remote-) Batch-Tomographie-Sitzung auf. Während die Software für Anfänger einfach ist, können einige erste Kryo-EM-Erfahrungen und grundlegendes Tomographie-Verständnis bei der Einrichtung helfen. Kritische Schritte werden im Protokoll hervorgehoben und sollten bei der Fehlerbehebung helfen, auch wenn ein anderer Setup-Ansatz verwendet wurde. Die Weiterentwicklung der Software wird die (Fern-)Datenerfassung erleichtern und die Kryo-ET für eine breite Benutzerbasis zugänglicher machen. Im Folgenden werden einige Tipps und Tricks zur Behebung häufig auftretender Probleme beschrieben.
Ein wichtiger Punkt ist die Wahl der Raster, da bei einer Neigung der Probe auf ±60° Rasterbalken bei hohen Neigungen die Sicht verdecken können (Abbildung 8). In einem TEM-Gitter bezieht sich die Maschenweite auf die Anzahl der Gitterquadrate pro Längeneinheit des Gitters. Größere Netznummern haben mehr Rasterquadrate pro Längeneinheit, eine höhere Dichte von Gitterquadraten und kleinere Rasterquadrate, d. h. ein 400-Netzraster hat kleinere Quadrate als ein 200-Netzraster. Eine gute Wahl der Gitter für die Tomographie sind 200-Mesh- oder 300-Mesh-Gitter. Wie in Abbildung 8 dargestellt, verringert sich die verfügbare zu erfassende Fläche, wenn das Raster geneigt wird. Bei einer Neigung von ±60° hat ein 300-Maschen-Raster ein kleines Sichtfeld, auf dem ein vollständiges Tomogramm erfasst werden kann. Die Vorteile von 200-Mesh-Gittern sind, dass die größeren Gitterquadrate den molekularen Tomographieaufbau beschleunigen, und mit der vergrößerten Raster-Quadrat-Fläche wird ein Quadrat wahrscheinlich für eine Übernachtsammlung ausreichen. Der Nachteil ist, dass 200-Mesh-Gitter zerbrechlicher sind, so dass Handhabung und Clipping mehr Finesse erfordern.
Darüber hinaus muss bei Verwendung von löchriger Stützfolie (siehe Materialtabelle) auf EM-Gittern der Lochabstand für die Einrichtung des Fokus- und Trackingbereichs in Bezug auf den Belichtungsbereich berücksichtigt werden. Idealerweise sollte der Strahldurchmesser bei der gewünschten Vergrößerung klein genug sein, um den Kohlenstoffbereich neben dem Belichtungsbereich entlang der Neigungsachse abzudecken, um eine optimale und schnelle Einrichtung zu ermöglichen. Auf diese Weise können potenzielle Interessengebiete in jedem Bohrloch erfasst werden.
Da die euzentrische Höhenroutine der Software derzeit nicht so robust ist, wie die serialEM-Routine, können die folgenden Tipps dieses Problem umgehen. Wenn die euzentrische Höhenbestimmung mit der euzentrischen Höhenvoreinstellung fehlschlägt, kann man stattdessen die Übersichtsvoreinstellung verwenden und "Auto-euzentrisch durch Bühnenneigung" erneut ausführen; Dies kann Probleme lösen, wenn die euzentrische Höhe weit von 0 entfernt ist. Wenn dies gelingt, kann man "Auto-eucentric by stage tilt" mit "Eucentric Height" -Presets erneut ausführen, um die Präzision zu verbessern. Wenn dies fehlschlägt, kann man "Auto-Eucentric by beam tilt" mit der euzentrischen Höhenvoreinstellung ausführen und dann "Auto-Eucentric by stage tilt" erneut ausführen oder die durch "Auto-Eucentric by beam tilt" konsolidierte z-Höhe manuell in der TEM-Benutzeroberfläche unter "Stage" -Einstellungen einstellen. Wenn Gitter mit einem sich wiederholenden Muster von Löchern verwendet werden, können sie die Identifizierung eines einzelnen Kreuzkorrelationspeaks verhindern. Man kann versuchen, die euzentrische Höhenvoreinstellung auf einen niedrigeren Defokussierungsversatz wie −25 μm und/oder eine kürzere Belichtungszeit zu ändern, um die Kreuzkorrelation von den Lochmustern zu reduzieren. Auf der anderen Seite liefert die Verwendung von Spitzengittern / Lamellen möglicherweise kein ausreichendes Signal für eine starke Kreuzkorrelationsspitze. Man kann versuchen, die euzentrische Höhenvoreinstellung auf einen größeren Defokussierungsversatz wie −75 μm und/oder eine verlängerte Belichtungszeit zu ändern, um den Kreuzkorrelationspeak zu erhöhen. Eine weitere Möglichkeit besteht darin, die Bildfiltereinstellungen anzupassen. Sie finden sie im Reiter "Vorbereitung". Optionen zum Anpassen der Filtereinstellungen können für niedrige (Übersicht/Rasterquadrat), mittlere (euzentrische Höhe) und hohe Vergrößerung (Tracking/Fokus) eingestellt werden, um den optimalen Kreuzkorrelationspeak für jede Voreinstellung zu finden. Die erforderliche Eingabe ist ein Bild, d. h. bei 0° und eines bei 5°, gefolgt von einem Klick auf Vergleichen , um beide Bilder zu vergleichen. Der empfohlene Startwert für die längste Wellenlänge beträgt ein Viertel des Maßstabsbalkens im Bild und für die kürzeste Wellenlänge ein Vierzigstel des Maßstabsbalkens. Wenn der Peak nicht robust identifiziert wird, kann man die Einstellungen optimieren, bis ein überzeugender Peak gefunden werden kann. Es ist nicht notwendig, jedes Mal Bilder neu zu erfassen. Ein einfaches Drücken von "Vergleichen" genügt. Sollte TOMO die euzentrische Höhe immer noch nicht automatisch finden, kann die manuelle euzentrische Höhenkalibrierung verwendet werden. Man sollte über einem einigermaßen großen Eiskristall in Übersichtsvergrößerung in der Registerkarte "Vorbereitung" zentrieren, dann zur "Bühnensteuerung" der TEM-Benutzeroberfläche gehen, Alpha auf −30° einstellen und den Stufen-Z-Wert anpassen, um den Kristall mit dem fluoreszierenden Bildschirmbild neu zu zentrieren. Die Auswahl der Einstellungen "Hohe Auflösung" und "Hoher Kontrast" in der TEM-Benutzeroberfläche erleichtert dies (Schaltflächen am unteren Rand des fluoreszierenden Bildschirmfensters). Optional, wenn es Zugriff auf eine Kamera mit Live-Modus gibt, kann dies verwendet werden, um die euzentrische Höhe zu bestimmen; Es wird einfacher sein als auf dem fluoreszierenden Bildschirm.
Die größten Einschränkungen in Tomo5-Versionen vor 5.8 sind die fehlenden Montagen mit mittlerer Vergrößerung, das fehlende dosissymmetrische Schema und Probleme im Zusammenhang mit der euzentrischen Höhenfindung. Diese existieren in serialEM, einer Freeware mit schneller Entwicklung und Community-Unterstützung, einer robusten euzentrischen Höhenroutine und der Option zu skripten, d.h. einem maßgeschneiderten dosissymmetrischen Schema. Ab Version 5.8 in Tomo5 wurde das am häufigsten auftretende Problem beim Finden der euzentrischen Höhe, d.h. eine erfolglose Schleife um den Ziel-Z-Wert, durch die Implementierung der Option zum Festlegen eines euzentrischen Höhenakzeptanzkriteriums gelöst. Bei verschiedenen Raster- und Probentypen wird jedoch dringend empfohlen, die Bildfiltereinstellungen anzupassen, um die einzigartigen Bildbedingungen einzelner Sitzungen widerzuspiegeln und den bestmöglichen Kreuzkorrelationspeak zu erzielen, um die euzentrische Höhe zu finden und den Fokus und den Tracking-Bereich während der Tomogrammaufnahme zuverlässig zu arbeiten.
Insgesamt haben sich viele Einrichtungen während der Pandemie schnell an den Fernbetrieb angepasst. Die Tomo5-Software bietet einen einfachen Zugang und eine benutzerfreundliche Route zur Tomographie, die sich gut für die Fernbedienung eignet. Die Fortschritte in der Software werden zweifellos die Ferndatenerfassung und die Tomographieerfassung im Allgemeinen in der Community weiter zum Mainstream machen.
emClarity
Da emClarity eine schablonenbasierte Partikelpicking-Methode verwendet, benötigt es eine Vorlage für das Objekt von Interesse. Die Partikelpickung (Schritt 2.6) ist sehr empfindlich und entscheidend für die endgültige Struktur. Vor der Mittelwertbildung und Ausrichtung (Schritt 2.9) muss sichergestellt werden, dass die Fehlalarme sorgfältig überprüft und manuell entfernt werden. Wenn eine Vorlage nicht verfügbar ist, ist emClarity möglicherweise nicht einfach zu verwenden, aber es ist möglich, andere Software, z. B. Dynamo37 und PEET48, zu verwenden, um ein erstes Modell zu erstellen.
Für heterogene Stichproben ist emClarity mit einer Klassifizierungsmethode ausgestattet, die es Anwendern ermöglicht, sich auf bestimmte Merkmale mit unterschiedlichen Skalen zu konzentrieren. Es ist hilfreich, vor der Klassifizierung einige Ausrichtungszyklen auszuführen und sie in einem höheren Binning auszuführen (z. B. Bin 4 oder Bin 3).
Die aktuelle Version der Software (V1.5.3.11) weist im Vergleich zum ersten Release (V1.0)17 erhebliche Updates auf. Dazu gehören unter anderem eine Händigkeitsprüfung während der CTF-Schätzung (Schritt 2.3); Symmetrie für Ausrichtungen (CX, I, I2, O); Berechnung von Pro-Partikel-3D-Probenahmefunktionen (3DSF); ein Wechsel zu MATLAB 2019a für Kompatibilität und Stabilität; und Rekonstruktion mit Hilfe der Rohprojektionsbilder (cisTEM). Die Software wird für verschiedene Muster weiter verbessert, und die neuesten Ankündigungen finden Sie online (siehe Materialtabelle).
Offenlegungen
Die Autoren haben keine Interessenkonflikte.
Danksagungen
Wir danken Diamond Light Source für den Zugang zu und die Unterstützung der Kryo-EM-Einrichtungen im nationalen Electron Bio-Imaging Centre (eBIC) des Vereinigten Königreichs, das vom Wellcome Trust, MRC und BBRSC finanziert wird. Wir danken auch Andrew Howe für den Erwerb des Apoferritin-Tomogramms (Film 1), Ishika Kumar für die Vorbereitung und den Erwerb des Neuronen-Tomogramms (Film 2) und Craig MacGregor-Chatwin für das Cyanobakterien-Lamella-Tomogramm (Film 3).
Materialien
| Name | Company | Catalog Number | Comments |
| Software | |||
| Tomography | Thermo Fisher Scientific | 5.9.0 | Internal terminology: Tomo5 in document |
| TEM server | Thermo Fisher Scientific | 7.10.1 | |
| TIA | Thermo Fisher Scientific | 5.10.1 | |
| DigitalMicrograph | Gatan | 3.44 | |
| emClarity | Open-Source software | 1.5.3.11 | Software for high-resolution cryo-electron tomography and subtomogram averaging |
| IMOD | Open-Source software | 4.11 | Modeling, display and image processing programs used for 3D reconstruction and modeling of microscopy images with a special emphasis on electron microscopy data |
| MotionCor2 | Free for academic use | 1.1.0 | A multi-GPU program that corrects beam-induced sample motion recorded on dose fractionated movie stacks |
| ETomo | Open-Source software | 4.11 | ETomo is an interface for running a subset of IMOD and PEET commands. |
| NoMachine | NoMachine, freeware | 7.9.2 | Remote desktop software |
| TeamViewer | TeamViewer AG | - | Remote access and remote control computer software |
| Materials | |||
| Quantifoil (holey support film) EM grids | Quantifoil | - | A flat film of carbon with pre-defined hole size, shape and arrangement |
| Instrumentation | |||
| Titan Krios microscope | Thermo Fisher Scientific | Titan Krios G2 | |
| K3 camera and GIB energy filter | Gatan | - | |
| Falcon 4 camera and Selectris X energy filter | Thermo Fisher Scientific | - | |
| Website | |||
| Website 1: https://github.com/bHimes/emClarity/ | - | - | Link to download the emClarity software package |
| Website 2: https://bio3d.colorado.edu/imod/ | - | - | Link to download IMOD |
| Website 3: https://github.com/ffyr2w/emClarity-tutorial | - | - | Link to the emClarity online tutorial |
| Website 4: https://emcore.ucsf.edu/ucsf-software | - | - | Link to download MotionCor2 |
| Website 5: https://github-wiki-see.page/m/bHimes/emClarity/wiki | - | - | Link to the newest announcements including updates and bug fixs for emClarity |
Referenzen
- Bai, X. -. C., Fernandez, I. S., McMullan, G., Scheres, S. H. W. Ribosome structures to near-atomic resolution from thirty thousand cryo-EM particles. eLife. 2, 00461 (2013).
- Li, X., et al. Electron counting and beam-induced motion correction enable near-atomic-resolution single-particle cryo-EM. Nature Methods. 10 (6), 584-590 (2013).
- Nakane, T., et al. Single-particle cryo-EM at atomic resolution. Nature. 587 (7832), 152-156 (2020).
- Kato, T., et al. CryoTEM with a cold field emission gun that moves structural biology into a new stage. Microscopy and Microanalysis. 25, 998-999 (2019).
- Yip, K. M., Fischer, N., Paknia, E., Chari, A., Stark, H. Atomic-resolution protein structure determination by cryo-EM. Nature. 587 (7832), 157-161 (2020).
- Mastronarde, D. N. SerialEM: A program for automated tilt series acquisition on Tecnai microscopes using prediction of specimen position. Microscopy and Microanalysis. 9, 1182-1183 (2003).
- Schorb, M., Haberbosch, I., Hagen, W. J. H., Schwab, Y., Mastronarde, D. N. Software tools for automated transmission electron microscopy. Nature Methods. 16 (6), 471-477 (2019).
- Deng, Y., et al. Smart EPU: SPA getting intelligent. Microscopy and Microanalysis. 27 (1), 454-455 (2021).
- Carragher, B., et al. Leginon: An automated system for acquisition of images from vitreous ice specimens. Journal of Structural Biology. 132 (1), 33-45 (2000).
- Bell, J. M., Chen, M., Baldwin, P. R., Ludtke, S. J. High resolution single particle refinement in EMAN2.1. Methods. 100, 25-34 (2016).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. cryoSPARC: Algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods. 14 (3), 290-296 (2017).
- Grant, T., Rohou, A., Grigorieff, N. cisTEM, user-friendly software for single-particle image processing. eLife. 7, 35383 (2018).
- Zivanov, J., et al. New tools for automated high-resolution cryo-EM structure determination in RELION-3. eLife. 7, 42166 (2018).
- Kimanius, D., Dong, L., Sharov, G., Nakane, T., Scheres, S. H. W. New tools for automated cryo-EM single-particle analysis in RELION-4.0. Biochemical Journal. 478 (24), 4169-4185 (2021).
- Galaz-Montoya, J. G., Flanagan, J., Schmid, M. F., Ludtke, S. J. Single particle tomography in EMAN2. Journal of Structural Biology. 190 (3), 279-290 (2015).
- Bharat, T. A. M., Scheres, S. H. W. Resolving macromolecular structures from electron cryo-tomography data using subtomogram averaging in RELION. Nature protocols. 11 (11), 2054-2065 (2016).
- Himes, B. A., Zhang, P. emClarity: Software for high-resolution cryo-electron tomography and subtomogram averaging. Nature Methods. 15 (11), 955-961 (2018).
- Weissenberger, G., Henderikx, R. J. M., Peters, P. J. Understanding the invisible hands of sample preparation for cryo-EM. Nature Methods. 18 (5), 463-471 (2021).
- White, J. B. R., et al. Single particle cryo-electron microscopy: From sample to structure. Journal of Visualized Experiments. (171), e62415 (2021).
- Orlova, E. V., Saibil, H. R. Structural analysis of macromolecular assemblies by electron microscopy. Chemical Reviews. 111 (12), 7710-7748 (2011).
- Turk, M., Baumeister, W. The promise and the challenges of cryo-electron tomography. FEBS Letters. 594 (20), 3243-3261 (2020).
- Liu, Y. -. T., et al. Isotropic reconstruction of electron tomograms with deep learning. bioRxiv. , (2021).
- Briggs, J. A. G. Structural biology in situ-The potential of subtomogram averaging. Current Opinion in Structural Biology. 23 (2), 261-267 (2013).
- Grünewald, K., et al. Three-dimensional structure of herpes simplex virus from cryo-electron tomography. Science. 302 (5649), 1396-1398 (2003).
- Allegretti, M., et al. In-cell architecture of the nuclear pore and snapshots of its turnover. Nature. 586 (7831), 796-800 (2020).
- Wang, Z., et al. The molecular basis for sarcomere organization in vertebrate skeletal muscle. Cell. 184 (8), 2135-2150 (2021).
- Winey, M., Meehl, J. B., O'Toole, E. T., Giddings, T. H. Conventional transmission electron microscopy. Molecular Biology of the Cell. 25 (3), 319-323 (2014).
- Davies, K. M., et al. Macromolecular organization of ATP synthase and complex I in whole mitochondria. Proceedings of the National Academy of Sciences. 108 (34), 14121 (2011).
- Wagner, J., Schaffer, M., Fernández-Busnadiego, R. Cryo-electron tomography-The cell biology that came in from the cold. FEBS Letters. 591 (17), 2520-2533 (2017).
- Mahamid, J., et al. Visualizing the molecular sociology at the HeLa cell nuclear periphery. Science. 351 (6276), 969-972 (2016).
- Erdmann, P. S., et al. In situ cryo-electron tomography reveals gradient organization of ribosome biogenesis in intact nucleoli. Nature Communications. 12 (1), 5364 (2021).
- Tegunov, D., Xue, L., Dienemann, C., Cramer, P., Mahamid, J. Multi-particle cryo-EM refinement with M visualizes ribosome-antibiotic complex at 3.5 Å in cells. Nature Methods. 18 (2), 186-193 (2021).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152 (1), 36-51 (2005).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Heymann, J. B., Belnap, D. M. Bsoft: Image processing and molecular modeling for electron microscopy. Journal of Structural Biology. 157 (1), 3-18 (2007).
- Tang, G., et al. EMAN2: An extensible image processing suite for electron microscopy. Journal of Structural Biology. 157 (1), 38-46 (2007).
- Castaño-Díez, D., Kudryashev, M., Arheit, M., Stahlberg, H. Dynamo: A flexible, user-friendly development tool for subtomogram averaging of cryo-EM data in high-performance computing environments. Journal of Structural Biology. 178 (2), 139-151 (2012).
- Hrabe, T., et al. PyTom: A python-based toolbox for localization of macromolecules in cryo-electron tomograms and subtomogram analysis. Journal of Structural Biology. 178 (2), 177-188 (2012).
- Himes, B. A. emClarity. GitHub. , (2021).
- . The IMOD Home Page Available from: https://bio3d.colorado.edu/imod/ (2020)
- . GitHub: emClarity-tutorial Available from: https://github.com/ffyr2w/emClarity-tutorial (2021)
- Ni, T., et al. High-resolution in situ structure determination by cryo-electron tomography and subtomogram averaging using emClarity. Nature Protocols. 17 (2), 421-444 (2022).
- . MotionCor2 Available from: https://emcore.ucsf.edu/ucsf-software (2016)
- . Apoferritin from mouse at 1.84 angstrom resolution Available from: https://www.emdataresource.org/EMD-10101 (2022)
- Nedozralova, H., et al. In situ cryo-electron tomography reveals local cellular machineries for axon branch development. Journal of Cell Biology. 221 (4), 202106086 (2022).
- Mendonça, L., et al. Correlative multi-scale cryo-imaging unveils SARS-CoV-2 assembly and egress. Nature Communications. 12 (1), 4629 (2021).
- . Apoferritin from mouse at 1.84 angstrom resolution Available from: https://www.rcsb.org/structure/6s61 (2019)
- Nicastro, D., et al. The molecular architecture of axonemes revealed by cryoelectron tomography. Science. 313 (5789), 944-948 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten