Method Article
分离和加工小鼠白脂肪细胞用于转录组和表观基因组分析
摘要
本方案总结了一种用于分离、纯化和上游加工小鼠白脂肪细胞的通用方法,该方法针对下游总 RNA 测序、SONication 细胞核提取 (NEXSON) 和 ChIP-seq 进行了优化。
摘要
肥胖是一种复杂的疾病,受遗传学、表观遗传学、环境及其相互作用的影响。成熟的脂肪细胞是白色脂肪组织中的主要细胞类型。了解脂肪细胞的功能和对(表观)遗传和环境信号的反应对于确定肥胖的原因至关重要。RNA 和染色质以前使用酶消化从脂肪细胞中分离出来。此外,已经开发了用于核分离的方案,其中通过脂肪细胞特异性转基因报告基因的荧光激活细胞分选 (FACS) 实现纯化。在此类方案中实现高产量和质量的最大挑战之一是脂肪组织中含有大量的脂质。本方案描述了一种用于分离成熟脂肪细胞的优化程序,该程序利用庚烷将脂质与感兴趣的靶标(RNA/染色质)分离。所得 RNA 具有高完整性,可产生高质量的 RNA-seq 结果。同样,该程序提高了细胞核得率,并在样本中产生可重复的 ChIP-seq 结果。因此,目前的研究提供了一种适用于全基因组转录组和表观基因组研究的可靠且通用的小鼠脂肪细胞分离方案。
引言
肥胖通常被理解为一种脂肪堆积过多的疾病,导致 2 型糖尿病、心脏代谢疾病和几种癌症的风险增加 1,2,3。虽然目前对肥胖的理解在很大程度上植根于遗传学(来自人类和啮齿动物研究),但大约 30%-70% 的代谢疾病易感性是非遗传起源的 4,5,6,7,8,并且仍然定义不明确。
脂肪组织在肥胖和其他代谢疾病中起着关键作用 9,10。脂肪组织包括成熟的脂肪细胞和基质血管部分,包括前脂肪细胞、内皮细胞和免疫细胞。目前尚不清楚每种细胞类型如何导致肥胖,以及脂肪细胞失调如何导致肥胖。用于成熟脂肪细胞表观基因组研究的可重复且有效的分离和纯化方案是该领域的兴趣所在。
长期以来,成熟脂肪细胞已被分离用于基因表达分析11 和表观基因组研究12,13。分离脂肪细胞有两种主要策略。第一种是使用酶消化将成熟的脂肪细胞与基质血管部分11,14 中的其他细胞类型分离。第二种是剥离脂肪组织以释放完整的细胞核,然后通过荧光激活细胞分选 (FACS) 12,13 基于荧光报告基因恢复细胞核,这需要专门的转基因报告基因模型。每种情况下的技术挑战是成熟脂肪细胞含有高浓度的脂质(图 1),这会降低总 RNA15,16 和细胞核17 的质量和/或产量。这里描述了一种用于分离成熟脂肪细胞的优化酶消化程序,其中庚烷的优势是在 RNA 提取或通过 SONication 细胞核提取 (NEXSON) 进行细胞核分离步骤之前快速有效地溶解和去除脂质 1819。该方案可确保全基因组研究中总 RNA 的出色回收率和质量,并显著提高完整细胞核的产量,以实现可重复的 ChIP-seq。
研究方案
所有动物实验均已获得机构动物护理和使用委员会 (IACUC) 的批准,方案编号:18-10-028。用 CO2 对 12 周龄雄性 C57BL/6J 小鼠实施安乐死并解剖,从附睾白色脂肪组织 (eWAT) 中收集脂肪垫。
1. 脂肪细胞分离
- 准备消化缓冲液(表 1)并在水浴中加热至 37 °C。
- 将附睾白色脂肪组织 (eWAT) 的脂肪垫放入培养皿中。使用镊子和手术刀在 5 mL DMEM 中将组织切成小块 (5 mm x 5 mm)。
注:首选不含细胞粘附涂层的培养皿。轻轻握住脂肪垫以避免损坏细胞也很重要。脂肪组织处理得越多,产量受到的影响就越大。手术刀/镊子方法比单独用剪刀或镊子切割脂肪垫更可取,因为它们会损害脂肪细胞。 - 在不干扰组织碎片的情况下排出多余的 DMEM。
注意:不要从培养皿表面捡起组织碎屑。 - 向培养皿中加入 5 mL 消化缓冲液,旋转以从培养皿中释放组织碎片,然后将全部内容物倒入 50 mL 离心管中。重复该过程,直到所有组织碎片都转移到离心管中。
- 将剩余的消化缓冲液添加到离心管中,使最终体积为 ~20 mL。
- 将组织碎片在 37 °C 振荡水浴中以 100 rpm 孵育 20-30 分钟,或直到组织碎片小于 1 mm x 1 mm。
- 向离心管中加入 0.4 mL 的 0.5 M EDTA 和 0.2 mL 的 0.5 M EGTA ,并在 37 °C 下继续振荡 10 分钟。
- 用 10 mL 移液器将细胞悬液通过另一个 50 mL 离心管顶部的细基质过滤器(420 μm,参见 材料表)转移,以去除未消化的组织。
- 在室温下以 200 x g 的速度离心 50 mL 离心管 5 分钟,以分离纯脂肪细胞(漂浮在顶部)与基质血管部分(底部沉淀)(图 2A)。
- 将漂浮的脂肪细胞层倒入新的 50 mL 试管中,注意不要破坏沉淀。
注意:避免使用移液器吸头转移漂浮的脂肪细胞,因为它们会粘在移液器吸头壁上(因此会降低产量)。一些消化缓冲液将带入新管中;使用注射器和针头从脂肪细胞层下去除残留的消化缓冲液。 - 立即使用纯化的脂肪细胞进行总 RNA 提取(步骤 2)和/或 ChIP-seq(步骤 3. 和步骤 4.)。
注意:用户可以决定在第 2 步中使用全脂肪细胞提取物。或第 3 步,具体取决于研究目的。用户还可以将整个脂肪细胞提取物分成两部分,然后继续第 2 步。和步骤 3.同时从生物相同的样品中获得分析结果。
2. 总 RNA 提取
注意:在化学罩中执行此步骤。
- 将 RNA 分离试剂 (1 mL)(参见 材料表)添加到 50 mL 离心管中纯化的脂肪细胞中(步骤 1.11)。
- 使用 1 mL 移液器吸头上下移液,将脂肪细胞彻底溶解在 RNA 分离试剂中,然后转移到 2 mL 微量离心管中。
- 在室温下孵育试管 15 分钟。
- 将 0.5 mL 庚烷加入离心管中,全速涡旋 30 秒。
注意:脂质将溶解在庚烷中(图 2B)。 - 将试管在 4 °C 下以 1,000 x g 离心 10 分钟。
- 用注射器和 30 G 针头获取 RNA 分离试剂的底层,然后转移到新的 1.5 mL 试管中。
- 向 RNA 分离试剂提取物中加入 0.1 mL 1溴-3氯丙烷(参见 材料表),并全速涡旋 30 秒。在室温下孵育试管 15 分钟。
- 将试管在 4 °C 下以 12,000 x g 离心 15 分钟。 使用移液管收集上层水相并将其转移到新试管中。
- 加入 0.5 mL 异丙醇,并在 4 °C 下孵育 10 分钟。 将试管在 4 °C 下以 12,000 x g 离心 10 分钟。
注意:RNA 将在试管底部沉淀为沉淀。 - 使用移液管去除上清液,小心避免接触沉淀。
- 加入 1 mL 75% 乙醇并短暂全速涡旋。
- 将试管在 4 °C 下以 7,500 x g 离心 5 分钟。 用移液器吸头小心去除上清液。不要打扰颗粒。
- 在室温下风干沉淀 10 分钟或直到沉淀变得透明/透明。
- 将沉淀溶解在 50 μL 无核酸酶水中。
3. 用于 ChIP-seq 的脂肪细胞固定
注意:执行步骤 3。在化学罩中。
- 将 0.3 mL 的 0.5 M EDTA、0.2 mL 的 0.5 M EGTA、14.8 mL 的 DMEM 和 10 mL 的庚烷加入含有分离脂肪细胞的 50 mL 试管中(步骤 1.11)。
- 加入 0.7 mL 的 16% 甲醛(最终 0.7%),并在管旋转器(参见 材料表)上以 10 rpm 的速度在室温下旋转 5 分钟。
注意: 设置计时器并计数 5 分钟。如果处理多个样品,则每隔 30 秒依次添加甲醛,以实现所有样品的精确和可比较的固定时间。 - 加入 1.78 mL 的 1.25 M 甘氨酸(最终 0.125 M),并在室温下以 10 rpm 的速度在旋转器上混合不超过 5 分钟。
注意:即使在添加甘氨酸后,甲醛仍保持活性。因此,重要的是在 5 分钟孵育步骤后立即通过离心(步骤 3.4.)分离各层。如果有多个样品,则向每个样品中加入甘氨酸,以在 30 s 的时间间隔内依次终止反应。 - 接下来,在室温下以 200 x g 离心 5 分钟。
注意:必须有三个明显且离散的液体层(图 2C)。中间的白色层包含固定的脂肪细胞。 - 使用 1 mL 移液器吸头将脂肪细胞(白色)层转移到新的 15 mL 离心管中,最大限度地减少带入新管的固定剂和庚烷的量。
- 用 10 mL(室温)DMEM 填充试管,并补充有蛋白酶抑制剂混合物(PIC,参见 材料表)片剂。倒置充分混合。
- 在室温下以 ≤200 x g 离心管 5 分钟。将离心力保持在 100 x g 以尽量减少细胞爆裂。
- 重复步骤 3.5.-3.7。1 倍,然后前进到步骤 3.9。
- 使用 1 mL 移液器吸头将脂肪细胞(白色)层转移到 1.5 mL 或 2 mL 微量离心管中。
- 在室温下以 100-200 x g 离心管 5 分钟,并使用 1 ml 移液器吸头去除庚烷(顶层)和 DMEM(底层)。
- 将固定的脂肪细胞在 -80 °C 下储存长达 6 个月直至使用。
4. 细胞核提取和染色质剪切
注意:该程序改编自 Arrigoni 等人 19。
- 打开超声仪(参见 材料表),将峰值功率设置为 75 W,占空比为 2%,每次循环 200 次,水浴冷却器设置为 20 °C。
- 将蛋白酶抑制剂混合物 (PIC) 添加到 Farnham 实验室缓冲液和剪切缓冲液中(表 1)。
注意:请查看 Arrigoni 等人 19 中更多建议的超声仪和参数。 - 在 1 mL 超声处理管中用 750-800 μL 冰冷的 Farnham 实验室缓冲液重悬固定的脂肪细胞(来自步骤 3.)(参见 材料表)。
注意: 建议不要将试管装满。否则,脂肪细胞将漂浮到试管的最顶部,从而降低超声处理效率。 - 对样品进行超声处理 2.5 分钟。
注意:在相差显微镜上检查细胞核提取,以确定是否需要进一步超声处理。细胞核必须是圆形且完整的,如图 2D 所示。 - 在进一步作之前,将超声仪峰值功率设置为 140 W,占空比为 5%,每次循环 200 次,水浴冷却器设置为 4 °C。
- 使用移液管将上清液转移到 1.5 mL 微量离心管中。将试管在 4 °C 下以 1,000 x g 离心 5 分钟。
- 小心地从沉淀(细胞核)中取出上清液并保留沉淀。
- 在冰上用 1 mL Farnham 实验室缓冲液洗涤沉淀。
- 将试管在 4 °C 下以 1,000 x g 离心 5 分钟,使用移液管除去上清液,并保留沉淀。
- 重复步骤 4.8.-4.9。1 次,然后继续执行步骤 4.11。
注:分离和洗涤的细胞核可在 -80 °C 下储存 3 个月,直至进一步使用。 - 将分离的细胞核重悬于 1 mL 剪切缓冲液中,并将它们转移到新的 1 mL 超声管中。确保管中没有气泡。
- 以 140 W 的峰值功率、5% 的占空比和 200 次循环/脉冲 12 分钟在超声仪中对细胞核进行超声处理,以在 4 °C 下剪切染色质。
- 将裂解物转移到 1.5 mL 微量离心管中。
注:剪切的染色质从细胞核中释放出来,并包含在缓冲液(裂解物)中。 - 将试管在 4 °C 下以 10,000 x g 离心 5 分钟以沉淀不溶性碎片。将上清液(染色质)转移到新的 1.5 mL 微量离心管中。
- 剪切的染色质可在 4 °C 下储存 10 天或在 -80 °C 下储存 3 个月,直至进一步使用。
注:额外的质量检查:按照先前发布的报告19,将 25 μL 剪切的染色质分装以去交联并用不含 DNase 的 RNase 去除 RNA。对于本研究,按照制造商的说明用 DNA 纯化试剂盒纯化所得 DNA,用 DNA 定量仪上的 DNA 定量试剂盒定量 DNA,并通过自动电泳仪评估片段大小(参见 材料表)。剪切的染色质大小应在 100-800 bp 的范围内。其余的剪切染色质可以继续进行下游 ChIP-Seq 应用。
结果
从 6 个脂肪垫中分离脂肪细胞,进行基于庚烷的 RNA 提取(步骤 2.),并在自动电泳仪上分析所得 RNA。所有样品的计算 RNA 完整性值 (RIN) 为 >8(图 3A),表明 RNA 制备质量高且可重复。然后使用总 RNA 制备试剂盒制备 RNA 文库,并在下一代测序仪上对每个样品进行测序,以达到 ≥4000 万个读数的读取深度。所有样品(图 3B)和每个序列(图 3C)的 Phred 评分为 ≥30,表明高质量 DNA 序列20。因此,庚烷去除支持分离适合 RNA 测序的高产量、高质量 RNA。
在甲醛固定步骤中,还进行了 3 种含庚烷的核分离制备和 1 种不含庚烷的对照制备。然后在自动电泳仪上分析剪切的染色质。在这两种情况下,染色质都被剪切成 100-800 bp 的大小范围(图 4A),非常适合下游 ChIP-seq 程序19。重要的是,与未处理的样品 (2.2 ng/μL) 相比,庚烷处理的样品 (11.5 ng/μL、9.42 ng/μL 和 9.6 ng/μL) 获得的染色质多 ~5 倍。H3K4me3 和 H3K27me3 ChIP-seq 在 Arrigoni 等人之后进行19。如图 4B 所示,来自三个独立庚烷处理样品的信号轨迹与两个组蛋白标记物的未经庚烷处理样品的轨迹相当。庚烷处理不会干扰 ChIP-seq 的质量,但会显著提高细胞核(和剪切染色质)的产量。
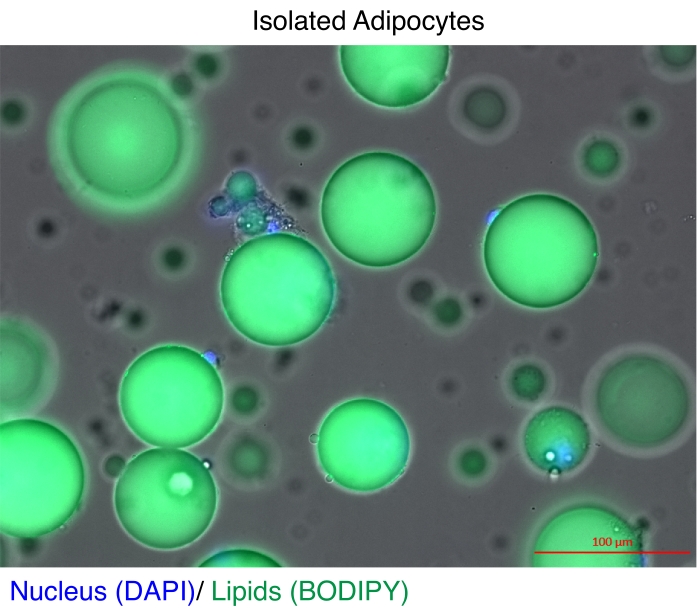
图 1:分离的脂肪细胞。 分离的脂肪细胞用 DAPI(蓝色)染色以对细胞核进行染色,用 BODIPY (绿色)对脂质进行染色。胞质脂滴占脂肪细胞体积的 95%,对在 RNA 和染色质提取过程中实现高产量构成了技术挑战。比例尺 = 100 μm。 请点击此处查看此图的较大版本。
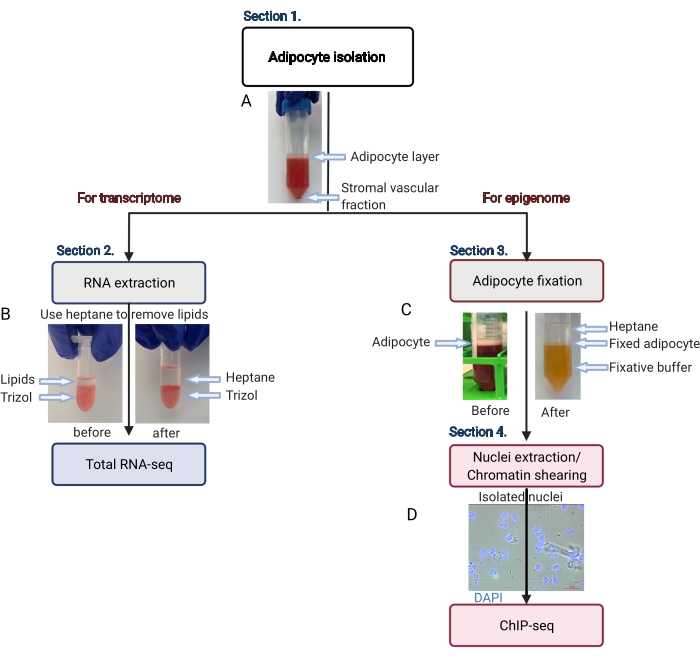
图 2:用于转录组和表观基因组分析的脂肪细胞分离示意图。 描述了从脂肪细胞分离到转录组或表观基因组应用的整个工作流程。显示了关键步骤和代表性结果。(A) 分离的脂肪细胞漂浮在顶层,在管底部以颗粒形式与基质血管部分分离。(B) 在用 RNA 分离试剂提取 RNA 之前使用庚烷去除脂质。(C) 在脂肪细胞固定过程中使用庚烷去除脂质。(D) 分离的脂肪细胞核的代表性图像必须完整且圆润。比例尺 = 20 μm。该计划是使用 BioRender.com 创建的。 请单击此处查看此图的较大版本。
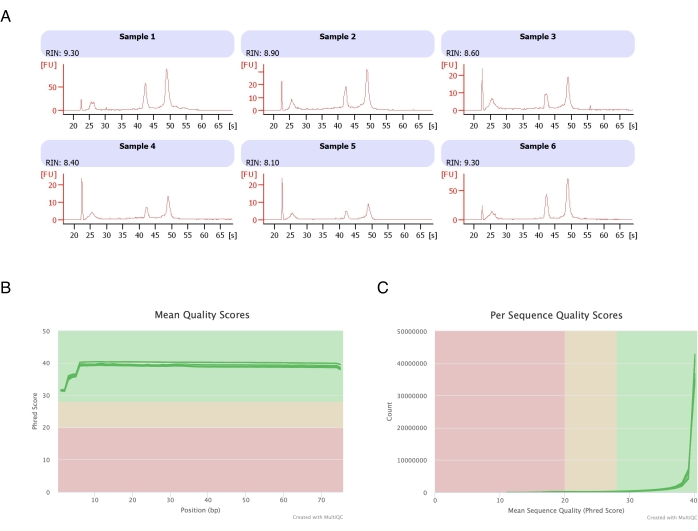
图 3:RNA-seq 后 RNA 完整性和质量评分的代表性电泳图。 (A) 样品 1-6 代表庚烷处理的脂肪细胞的 6 个重复,它们的 RNA 完整性分析在自动电泳仪上运行。RNA 完整性值 (RIN) 基于 18S 和 28S 核糖体 RNA 比率,代表 RNA 质量。从 1(严重降级)到 10(最低降级)进行缩放。(B,C)通过多重 QC 分析 (B) 确定碱基平均质量评分 (C) 和每个序列的质量评分。 请单击此处查看此图的较大版本。
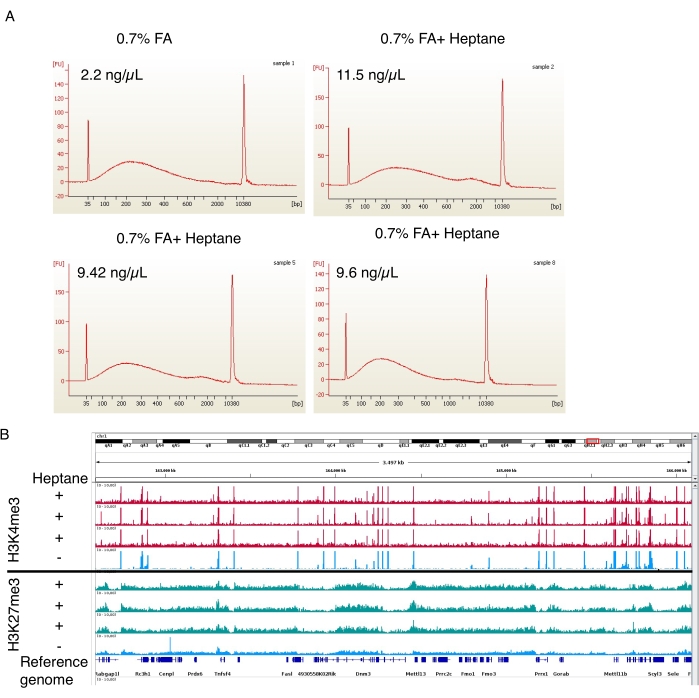
图 4:来自 ChIP-seq 的剪切染色质和富集峰的代表性电泳图。 (A) 来自自动电泳仪的代表性图谱,显示了对照(样品 1,左上)和庚烷处理的脂肪细胞染色质制备物(样品 2、5 和 8,右上和下 2)的染色质大小分布。最终制备物中的染色质浓度显示在每张图像的左上角。(B) 使用 Integrative Genomics Viewer (IGV) 制作的基因组浏览器屏幕截图。图的上半部分显示了三个(红色)庚烷处理样品和一个(蓝色)对照的 H3K4me3 ChIP-seq 图谱。下图显示 H3K27me3 的 ChIP-seq 也是如此。 请单击此处查看此图的较大版本。
| 缓冲区 | 组成 |
| 消化缓冲液(3000 毫克或更少的脂肪垫/20 mL) | 20 mL Dulbecco 改良 Eagle 培养基 (DMEM) |
| 0.3 克不含脂肪酸的 BSA | |
| 0.1 克 2 型胶原酶 | |
| Farnham 实验室缓冲液 | 5 毫米管道 (pH 8) |
| 85 毫米氯化钾 | |
| 0.5% IGEPAL | |
| 剪切缓冲器 | 10 毫米 Tris-HCl pH 值 8 |
| 0.1% 安全数据表 | |
| 1 毫米 EDTA |
表 1:本研究中使用的不同缓冲液的组成。
讨论
此处介绍的脂肪细胞分离方案基于公认的酶消化方法11,14,用于将成熟的脂肪细胞(漂浮的)与白色脂肪组织剩余的基质血管部分分离。它提供了一种简单而通用的方法,用于纯化任何小鼠模型中的成熟脂肪细胞。如上所述,该方案适用于全转录组和 ChIP-seq 分析的下游使用。它提供足够的产量,可从同一个单独的脂肪垫生成多个表观基因组图谱(例如,多个组蛋白修饰加 RNA-seq)。
为了确保能够制备此类匹配表观基因组数据的高产量,一个关键步骤是使用庚烷溶解完整脂肪细胞和在此过程中裂解的脂肪细胞中的脂质。根据我们的经验,此步骤通过防止 RNA 和细胞核丢失到脂质层,大大提高了可重复性和产量。包含正在进行固定的脂肪细胞的管的连续旋转同样重要,因为它增强了从脂肪细胞中去除脂质的均匀性。与许多 ChIP-seq 文献中报道的 1% 甲醛固定缓冲液21,22 不同,发现有必要将浓度降低到 0.7% 甲醛以避免过度固定。染色质剪切时间也优化为 12 分钟,以实现 ChIP-seq 的最佳染色质大小范围 (100-800 bp)。
该方案针对大量转录组和表观基因组研究进行了优化。目前的方案对于单细胞转录组和表观基因组应用仍有其自身的局限性。考虑到脂肪领域中报道的细胞异质性23,24,这种分离方法可以进一步发展以适应单细胞测定。在这种情况下,脂肪细胞限制性标志物,如硼-二吡咯甲烯 (BODIPY)25 和 perilipin-1 (PLIN1)26、ASC-127 或脂肪细胞特异性细胞核标志物,在定量额外的、功能性的、相关的细胞参数方面可能很有价值28。除了应用于基因组研究外,该方案还可能改进脂肪细胞蛋白质组学研究,由于脂肪细胞中的脂质含量高,这有一个额外的障碍29。
该方案还可用于成功提取人类脂肪细胞核(数据未显示),将其应用扩展到人类肥胖的研究。
披露声明
作者没有什么可披露的。
致谢
我们非常感谢 MPI-IE 光学成像、测序、生物信息学核心和人员。这项工作得到了 MPG、Van Andel 研究所、欧盟地平线 2020 研究、NIH(R01HG012444 和 R21HG011964)、Marie Skłodowska-Curie 赠款协议第 675610 号和联邦教育和研究部的项目编号 01KU1216(德国表观基因组计划,DEEP)的资助。
材料
| Name | Company | Catalog Number | Comments |
| 16% Formaldehyde Solution (w/v). Methanol-free | ThermoFisher SCIENTIFIC | 28908 | |
| 1-bromo-3-chloropropane | SIGMA | B9673 | |
| 5 M NaCl | invitrogen | AM9759 | |
| Automated electrophoresis instrument (2100 Bioanalyzer Instrument) | Agilent Technologies | G2939BA | |
| BODIPY | ThermoFisher SCIENTIFIC | D3922 | |
| Collagenase Type 2 | Worthington Biochemical Corp. | 43D14184B | |
| Complete, EDTA-free, Protease inhibitor cocktail (PIC) | Roche | 4693159001 | EDTA free |
| DAPI | ThermoFisher SCIENTIFIC | D1306 | |
| DMEM (+ GlutaMAX) | life technologies | 61965-026 | |
| DNA purification kit (PCR purification kit) | Qiagen | 28104 | |
| DNA quantification instrument (Qubit 4 Fluorometer) | Fisher SCIENTIFIC | Q33238 | |
| DNA quantification kit (DNA high sensitivity assay) | Agilent Technologies | 5067-4626 | |
| EDTA | Fisher chimical | E478-500 | |
| EGTA | Fisher chimical | O2783-100 | |
| EtOH | PHARMCO | 111000200 | |
| Fatty acid-free BSA | SERVA | 11932.01 | bovine albumin fraction V, fatty acid-free lyophilized |
| Glycine | SIGMA | G7126-100G | |
| Heptane | SIGMA | H2198-1L | |
| High Sensitivity RNA Analysis | Agilent Technologies | 5067-1513 | |
| Igepal | SIGMA | 18896-50ML | |
| KCl | SIGMA | P3911-25G | |
| Matrix filter (420um) | Tisch Scientific | ME17238 | |
| Next-Generation Sequencer (HiSeq 3000 Sequencer) | Illumina | ||
| Nuclease-free water | Invitrogen | 4387936 | |
| PIPES | ACROS organics | 5625-37-6 | |
| Proteinase K | ThermoFisher SCIENTIFIC | EO0491 | |
| RNA isolation reagent (Trizol) | ThermoFisher SCIENTIFIC | 15596026 | |
| RNase A, DNase-free | ThermoFisher SCIENTIFIC | EN0531 | |
| Rotator (Thermo Scientific Tube Revolver Rotator) | ThermoFisher SCIENTIFIC | 88881001 | |
| SDS, Sodium dodecyl sulfate | SIGMA | 8170341000 | |
| Sonication tube (1 mL milliTUBE with AFA Fiber) | Covaris | 520135 | |
| Sonicator (Evolution Focused-ultra-sonicator (S220 or E220)) | Covaris | 500217 or 500239 | |
| Total RNA Prep kit ( Illumina Stranded Total RNA Prep kit) | Illumina | 20040525 | |
| Tris-HCl | Fisher bioreagents | BP153-500 |
参考文献
- Albuquerque, D., Stice, E., Rodríguez-López, R., Manco, L., Nóbrega, C. Current review of genetics of human obesity: From molecular mechanisms to an evolutionary perspective. Molecular Genetics and Genomics. 290 (4), 1191-1221 (2015).
- Weihrauch-Blüher, S., Schwarz, P., Klusmann, J. H. Childhood obesity: Increased risk for cardiometabolic disease and cancer in adulthood. Metabolism. 92, 147-152 (2019).
- Weihrauch-Blüher, S., Wiegand, S. Risk factors and implications of childhood obesity. Current Obesity Reports. 7 (4), 254-259 (2018).
- Gluckman, P. D. Epigenetics, the life-course and metabolic disease. Nature Reviews Endocrinology. 8 (2), 74-76 (2012).
- Loh, M., Zhou, L., Ng, H. K., Chambers, J. C. Epigenetic disturbances in obesity and diabetes: Epidemiological and functional insights. Molecular Metabolism. 27, 33-41 (2019).
- Sayols-Baixeras, S., et al. DNA methylation and obesity traits: An epigenome-wide association study. The REGICOR study. Epigenetics. 12 (10), 909-916 (2017).
- Xia, Q., Grant, S. F. A. The genetics of human obesity. Annals of the New York Academy of Sciences. 1281 (1), 178-190 (2013).
- Yokoi, N. Epigenetic dysregulation in pancreatic islets and pathogenesis of type 2 diabetes. Journal of Diabetes Investigation. 9 (3), 475-477 (2018).
- Frayn, K. N. Obesity and metabolic disease: Is adipose tissue the culprit. Proceedings of the Nutrition Society. 64 (1), 7-13 (2005).
- Kawai, T., Autieri, M. V., Scalia, R. Adipose tissue inflammation and metabolic dysfunction in obesity. American Journal of Physiology-Cell Physiology. 320 (3), 375-391 (2021).
- Cat, A. N. D., Briones, A. M., Touyz, R. M., Schiffrin, E. L. . Hypertension: Methods and Protocols. , 283-295 (2017).
- Ambati, S., et al. Adipocyte nuclei captured from VAT and SAT. BMC Obesity. 3, 35 (2016).
- Roh, H. C., et al. Simultaneous transcriptional and epigenomic profiling from specific cell types within heterogeneous tissues in vivo. Cell Reports. 18 (4), 1048-1061 (2017).
- Jacobsen, M. J., et al. Epigenetic and transcriptomic characterization of pure adipocyte fractions from obese pigs identifies candidate pathways controlling metabolism. Frontiers in Genetics. 10, 1268 (2019).
- Janke, J., Engeli, S., Gorzelniak, K., Sharma, A. M. Extraction of total RNA from adipocytes. Hormone and Metabolic Research. 33 (4), 213-215 (2001).
- Cirera, S. Highly efficient method for isolation of total RNA from adipose tissue. BMC Research Notes. 6 (1), 472 (2013).
- Rajbhandari, P., et al. Single cell analysis reveals immune cell-adipocyte crosstalk regulating the transcription of thermogenic adipocytes. eLife. 8, 49501 (2019).
- Buddrick, O., Jones, O. A. H., Morrison, P. D., Small, D. M. Heptane as a less toxic option than hexane for the separation of vitamin E from food products using normal phase HPLC. RSC Advances. 3 (46), 24063-24068 (2013).
- Arrigoni, L., et al. Standardizing chromatin research: A simple and universal method for ChIP-seq. Nucleic Acids Research. 44 (7), 67 (2016).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Schmidt, D., et al. ChIP-seq: Using high-throughput sequencing to discover protein-DNA interactions. Methods. 48 (3), 240-248 (2009).
- Hoffman, E. A., Frey, B. L., Smith, L. M., Auble, D. T. Formaldehyde crosslinking: A tool for the study of chromatin complexes. Journal of Biological Chemistry. 290 (44), 26404-26411 (2015).
- Rondini, E. A., Granneman, J. G. Single cell approaches to address adipose tissue stromal cell heterogeneity. Biochemical Journal. 477 (3), 583-600 (2020).
- Corvera, S. Cellular heterogeneity in adipose tissues. Annu Review of Physiology. 83, 257-278 (2021).
- Katz, L. S., Geras-Raaka, E., Gershengorn, M. C. Heritability of fat accumulation in white adipocytes. American Journal of Physiology-Endocrinology and Metabolism. 307 (3), 335-344 (2014).
- Hansen, J. S., de Maré, S., Jones, H. A., Göransson, O., Lindkvist-Petersson, K. Visualization of lipid directed dynamics of perilipin 1 in human primary adipocytes. Scientific Reports. 7 (1), 15011 (2017).
- Ussar, S., et al. ASC-1, PAT2, and P2RX5 are cell surface markers for white, beige, and brown adipocytes. Science Translational Medicine. 6 (247), (2014).
- Onogi, Y., Khalil, A. E. M. M., Ussar, S. Identification and characterization of adipose surface epitopes. Biochemical Journal. 477 (13), 2509-2541 (2020).
- Kim, E. Y., et al. Recent advances in proteomic studies of adipose tissues and adipocytes. International Journal of Molecular Sciences. 16 (3), 4581-4599 (2015).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。