Method Article
Isolamento ed elaborazione di adipociti bianchi murini per analisi di trascrittoma ed epigenoma
In questo articolo
Riepilogo
Il presente protocollo riassume un metodo universale per l'isolamento, la purificazione e l'elaborazione a monte di adipociti bianchi murini ottimizzato per il sequenziamento dell'RNA totale a valle, l'estrazione di nuclei mediante SONication (NEXSON) e ChIP-seq.
Abstract
L'obesità è una malattia complessa influenzata dalla genetica, dall'epigenetica, dall'ambiente e dalle loro interazioni. Gli adipociti maturi rappresentano il principale tipo di cellula nel tessuto adiposo bianco. Comprendere come funzionano gli adipociti e rispondono ai segnali (epi)genetici e ambientali è essenziale per identificare le cause dell'obesità. L'RNA e la cromatina sono stati precedentemente isolati dagli adipociti utilizzando la digestione enzimatica. Inoltre, sono stati sviluppati protocolli per l'isolamento nucleare, in cui la purificazione si ottiene mediante smistamento cellulare attivato dalla fluorescenza (FACS) di reporter transgenici specifici per adipociti. Una delle maggiori sfide per ottenere un'elevata resa e qualità durante tali protocolli è la notevole quantità di lipidi contenuti nel tessuto adiposo. Il presente protocollo descrive una procedura ottimizzata per l'isolamento degli adipociti maturi che sfrutta l'eptano per separare i lipidi dai bersagli di interesse (RNA/cromatina). L'RNA risultante ha un'elevata integrità e genera risultati RNA-seq di alta qualità. Allo stesso modo, la procedura migliora il tasso di resa dei nuclei e genera risultati ChIP-seq riproducibili tra i campioni. Pertanto, il presente studio fornisce un protocollo di isolamento degli adipociti murini affidabile e universale adatto per studi di trascrittoma ed epigenoma dell'intero genoma.
Introduzione
L'obesità è tipicamente intesa come una malattia di accumulo di grasso in eccesso che contribuisce ad un aumento del rischio di diabete di tipo 2, malattie cardiometaboliche e diverse forme di cancro 1,2,3. Mentre l'attuale comprensione dell'obesità è fortemente radicata nella genetica (sia da studi sull'uomo che sui roditori), circa il 30%-70% della predisposizione alle malattie metaboliche è di origine non genetica 4,5,6,7,8 e rimane mal definita.
Il tessuto adiposo svolge un ruolo fondamentale nell'obesità e in altre malattie metaboliche 9,10. Il tessuto adiposo comprende gli adipociti maturi e la frazione vascolare stromale, compresi i preadipociti, le cellule endoteliali e le cellule immunitarie. Non è ancora chiaro come ogni tipo di cellula contribuisca all'obesità e come la disregolazione degli adipociti contribuisca all'obesità. Protocolli di isolamento e purificazione riproducibili ed efficaci per studi sull'epigenoma degli adipociti maturi sono di interesse per il campo.
Gli adipociti maturi sono stati a lungo isolati per analisi di espressione genica11 e studi sull'epigenoma 12,13. Esistono due strategie principali per isolare gli adipociti. Il primo consiste nell'utilizzare la digestione enzimatica per separare gli adipociti maturi dal resto dei tipi di cellule nella frazione vascolare stromale11,14. Il secondo consiste nel far sì che il tessuto adiposo rilasci nuclei intatti e quindi recuperare i nuclei sulla base di un reporter fluorescente mediante smistamento cellulare attivato dalla fluorescenza (FACS)12,13, che richiede modelli reporter transgenici specializzati. La sfida tecnica in ogni caso è che gli adipociti maturi contengono alte concentrazioni di lipidi (Figura 1), il che riduce la qualità e/o la resa dell'RNAtotale 15,16 e dei nuclei17. Qui, viene descritta una procedura di digestione enzimatica ottimizzata per isolare gli adipociti maturi, in cui il vantaggio dell'eptano è quello di dissolvere e rimuovere rapidamente ed efficacemente i lipidi18 prima dell'estrazione dell'RNA o le fasi di isolamento dei nuclei mediante estrazione dei nuclei mediante SONication (NEXSON)19. Il protocollo garantisce un eccellente recupero e qualità dell'RNA totale per gli studi sull'intero genoma e migliora significativamente la resa dei nuclei intatti per ChIP-seq riproducibile.
Protocollo
Tutti gli esperimenti sugli animali sono stati approvati dall'Institutional Animal Care and Use Committee (IACUC), numero di protocollo: 18-10-028. Topi maschi C57BL/6J di 12 settimane sono stati soppressi con CO2 e sezionati per raccogliere i cuscinetti adiposi dal tessuto adiposo bianco dell'epididimo (eWAT).
1. Isolamento degli adipociti
- Preparare il tampone di digestione (Tabella 1) e scaldare a bagnomaria a 37 °C.
- Metti il cuscinetto adiposo bianco dell'epididimo (eWAT) in una capsula di Petri. Tagliare il tessuto in piccoli pezzi (5 mm x 5 mm) utilizzando una pinza e un bisturi in 5 ml di DMEM.
NOTA: È preferibile una piastra di Petri che non contenga un rivestimento di adesione cellulare. È anche importante tenere delicatamente il cuscinetto adiposo per evitare di danneggiare le cellule. Più il tessuto adiposo viene maneggiato, più la resa è compromessa. Un metodo di bisturi/pinza è preferibile al taglio dei cuscinetti adiposi con le forbici o le pinze da soli in quanto possono danneggiare gli adipociti. - Drenare il DMEM in eccesso senza disturbare i detriti tissutali.
NOTA: Non raccogliere i detriti di tessuto dalla superficie della capsula di Petri. - Aggiungere 5 ml di tampone di digestione alla piastra di Petri, agitare per rilasciare i frammenti di tessuto dalla piastra e versare l'intero contenuto in una provetta da centrifuga da 50 ml. Ripetere il processo fino a quando tutti i frammenti di tessuto non sono stati trasferiti nella provetta da centrifuga.
- Aggiungere il tampone di digestione rimanente nella provetta da centrifuga in modo che il volume finale sia ~20 ml.
- Incubare i frammenti di tessuto in un bagno d'acqua agitato a 37 °C a 100 giri/min per 20-30 minuti, o fino a quando i frammenti di tessuto sono più piccoli di 1 mm x 1 mm.
- Aggiungere 0,4 mL di EDTA 0,5 M e 0,2 mL di EGTA 0,5 M alla provetta da centrifuga e continuare ad agitare a 37 °C per 10 min.
- Trasferire la sospensione cellulare con una pipetta da 10 mL attraverso un filtro a matrice fine (420 μm, vedere la Tabella dei materiali) sopra un'altra provetta da centrifuga da 50 mL per rimuovere il tessuto non digerito.
- Centrifugare la provetta da centrifuga da 50 mL a 200 x g per 5 minuti a temperatura ambiente per separare gli adipociti puri (galleggianti sulla parte superiore) dalla frazione vascolare stromale (pellet sul fondo) (Figura 2A).
- Versare lo strato di adipociti galleggianti in una nuova provetta da 50 mL, facendo attenzione a non rompere il pellet.
NOTA: Evitare di utilizzare i puntali delle pipette per trasferire gli adipociti galleggianti perché possono attaccarsi alla parete del puntale della pipetta (riducendo quindi la resa). Un po' di tampone per la digestione verrà trasferito nel nuovo tubo; Utilizzare una siringa e un ago per rimuovere il tampone digestivo residuo da sotto lo strato di adipociti. - Utilizzare immediatamente gli adipociti purificati per l'estrazione dell'RNA totale (fase 2) e/o ChIP-seq (fase 3 e fase 4).
NOTA: L'utente può decidere di utilizzare l'intero estratto di adipociti per il passaggio 2. o passaggio 3., a seconda dello scopo della ricerca. L'utente può anche dividere l'intero estratto di adipociti in due porzioni e continuare il passaggio 2. e il passaggio 3. in parallelo per ottenere i risultati dell'analisi dai campioni biologicamente identici.
2. Estrazione dell'RNA totale
NOTA: Eseguire questo passaggio in una cappa chimica.
- Aggiungere il reagente di isolamento dell'RNA (1 mL) (vedere la Tabella dei materiali) agli adipociti purificati nella provetta da centrifuga da 50 mL (passaggio 1.11.).
- Pipettare su e giù utilizzando un puntale da 1 ml per sciogliere completamente gli adipociti nel reagente di isolamento dell'RNA e trasferirli in una provetta per microcentrifuga da 2 ml.
- Incubare la provetta per 15 minuti a temperatura ambiente.
- Aggiungere 0,5 mL di eptano nella provetta da centrifuga e agitare alla massima velocità per 30 s.
NOTA: I lipidi si dissolveranno nell'eptano (Figura 2B). - Centrifugare la provetta a 1.000 x g per 10 minuti a 4 °C.
- Acquisire lo strato inferiore del reagente di isolamento dell'RNA con una siringa e un ago da 30 G e trasferirlo in una nuova provetta da 1,5 mL.
- Aggiungere 0,1 mL di 1bromo-3cloropropano (vedere la Tabella dei materiali) all'estratto del reagente di isolamento dell'RNA e agitare a piena velocità per 30 s. Incubare la provetta a temperatura ambiente per 15 minuti.
- Centrifugare la provetta a 12.000 x g per 15 minuti a 4 °C. Utilizzare una pipetta per raccogliere la fase acquosa superiore e trasferirla in una nuova provetta.
- Aggiungere 0,5 mL di isopropanolo e incubare per 10 minuti a 4 °C. Centrifugare la provetta a 12.000 x g per 10 minuti a 4 °C.
NOTA: L'RNA verrà precipitato sotto forma di pellet sul fondo del tubo. - Utilizzare una pipetta per rimuovere il surnatante, facendo attenzione a non toccare il pellet.
- Aggiungere 1 mL di EtOH al 75% e agitare brevemente alla massima velocità.
- Centrifugare la provetta a 7.500 x g per 5 minuti a 4 °C. Rimuovere con cautela il surnatante con la punta di una pipetta. Non disturbare il pellet.
- Asciugare il pellet all'aria a temperatura ambiente per 10 minuti o fino a quando il pellet non diventa chiaro/trasparente.
- Sciogliere il pellet in 50 μL di acqua priva di nucleasi.
3. Fissazione degli adipociti per ChIP-seq
NOTA: Eseguire il passaggio 3. in una cappa chimica.
- Aggiungere 0,3 mL di EDTA 0,5 M, 0,2 mL di EGTA 0,5 M, 14,8 mL di DMEM e 10 mL di eptano nella provetta da 50 mL contenente adipociti isolati (passaggio 1.11.).
- Aggiungere 0,7 mL di formaldeide al 16% (0,7% finale) e ruotare su un rotatore di provette (vedi Tabella dei materiali) a 10 giri/min per 5 minuti a temperatura ambiente.
NOTA: Imposta un timer e conta per 5 minuti. Se si elaborano più campioni, aggiungere formaldeide in sequenza a intervalli di 30 s per consentire una tempistica di fissazione precisa e comparabile su tutti i campioni. - Aggiungere 1,78 mL di glicina 1,25 M (0,125 M finale) e mescolare sul rotatore a 10 giri/min per non più di 5 minuti a temperatura ambiente.
NOTA: La formaldeide rimane attiva anche dopo l'aggiunta di glicina. Pertanto, è importante separare gli strati mediante centrifugazione (passaggio 3.4.) immediatamente dopo la fase di incubazione di 5 minuti. Se c'è più di un campione, aggiungere glicina a ciascun campione per fermare la reazione in sequenza in intervalli di tempo di 30 secondi. - Successivamente, centrifugare a 200 x g per 5 minuti a temperatura ambiente.
NOTA: Devono esserci tre strati di liquido evidenti e discreti (Figura 2C). Lo strato bianco centrale contiene gli adipociti fissi. - Utilizzare un puntale per pipetta da 1 ml per trasferire lo strato di adipociti (bianco) in una nuova provetta da centrifuga da 15 ml, riducendo al minimo la quantità di fissativo ed eptano trasportati nella nuova provetta.
- Riempire la provetta con 10 mL (temperatura ambiente) di DMEM integrato con la compressa del cocktail di inibitori della proteasi (PIC, vedere Tabella dei materiali). Mescolare accuratamente per inversione.
- Centrifugare la provetta a ≤200 x g per 5 minuti a temperatura ambiente. Mantenere la forza di centrifugazione a 100 x g per ridurre al minimo lo scoppio delle celle.
- Ripetere i passaggi 3.5.-3.7. 1 volta prima di andare al passaggio 3.9.
- Utilizzare un puntale per pipetta da 1 ml per trasferire lo strato di adipociti (bianco) in una provetta per microcentrifuga da 1,5 ml o 2 ml.
- Centrifugare la provetta a 100-200 x g per 5 minuti a temperatura ambiente e utilizzare un puntale per pipetta da 1 ml per rimuovere l'eptano (strato superiore) e il DMEM (strato inferiore).
- Conservare gli adipociti fissati a -80 °C per un massimo di 6 mesi fino all'uso.
4. Estrazione dei nuclei e taglio della cromatina
NOTA: Questa procedura è adattata da Arrigoni et al.19.
- Accendere il sonicatore (vedere la tabella dei materiali) e impostare la potenza di picco a 75 W, il fattore di lavoro al 2% e 200 cicli/scoppio, con il refrigeratore a bagno d'acqua impostato a 20 °C.
- Aggiungere il cocktail di inibitori della proteasi (PIC) al tampone di laboratorio Farnham e al tampone di taglio (Tabella 1).
NOTA: Si prega di controllare alcuni altri sonicatori e parametri suggeriti in Arrigoni et al.19. - Risospendere gli adipociti fissati (dal passaggio 3) con 750-800 μL di tampone Farnham lab ghiacciato in una provetta di sonicazione da 1 mL (vedere la Tabella dei materiali).
NOTA: Si consiglia di non riempire il tubo fino alla sua piena capacità. In caso contrario, gli adipociti galleggeranno nella parte più alta del tubo, riducendo l'efficienza della sonicazione. - Sonicare il campione per 2,5 minuti.
NOTA: Controllare l'estrazione dei nuclei su un microscopio a contrasto di fase per determinare se è necessaria un'ulteriore sonicazione. I nuclei devono essere rotondi e integri, come mostrato nella Figura 2D. - Prima di procedere, impostare la potenza di picco del sonicatore a 140 W, il fattore di lavoro al 5% e 200 cicli/scoppio, con il refrigeratore a bagno d'acqua impostato a 4 °C.
- Utilizzare una pipetta per trasferire il surnatante in una provetta da microcentrifuga da 1,5 mL. Centrifugare la provetta a 1.000 x g per 5 minuti a 4 °C.
- Rimuovere con cautela il surnatante dal pellet (nuclei) e conservare il pellet.
- Lavare il pellet con 1 mL di tampone da laboratorio Farnham con ghiaccio.
- Centrifugare la provetta a 1.000 x g per 5 minuti a 4 °C, utilizzare una pipetta per rimuovere il surnatante e conservare il pellet.
- Ripetere i passaggi 4.8.-4.9. 1 volta prima di andare avanti al passaggio 4.11.
NOTA: I nuclei isolati e lavati possono essere conservati a -80 °C per 3 mesi fino a un nuovo utilizzo. - Risospendere i nuclei isolati in 1 mL di tampone di taglio e trasferirli in una nuova provetta di sonicazione da 1 mL. Assicurarsi che non ci siano bolle nel tubo.
- Sonicare i nuclei nel sonicatore con una potenza di picco di 140 W, un fattore di lavoro del 5% e 200 cicli/burst per 12 minuti per tagliare la cromatina a 4 °C.
- Trasferire il lisato in una provetta microfuge da 1,5 mL.
NOTA: La cromatina tranciata viene rilasciata dai nuclei ed è contenuta nel tampone (lisato). - Centrifugare la provetta a 10.000 x g per 5 minuti a 4 °C per pellettare i detriti insolubili. Trasferire il surnatante (cromatina) in una nuova provetta da microcentrifuga da 1,5 mL.
- La cromatina tranciata può essere conservata a 4 °C per 10 giorni o a -80 °C per 3 mesi fino a nuovo utilizzo.
NOTA: Ulteriore controllo di qualità: Aliquotare 25 μL della cromatina tranciata per de-reticolare e rimuovere l'RNA con una RNasi priva di DNasi seguendo il rapporto precedentemente pubblicato19. Per il presente studio, il DNA risultante è stato purificato con un kit di purificazione del DNA secondo le istruzioni del produttore, il DNA è stato quantificato con un kit di quantificazione del DNA sullo strumento di quantificazione del DNA e la dimensione del frammento è stata valutata dallo strumento di elettroforesi automatizzato (vedi Tabella dei materiali). La dimensione della cromatina tranciata dovrebbe essere compresa tra 100 e 800 bp. Il resto della cromatina di taglio può procedere per l'applicazione ChIP-Seq a valle.
Risultati
Gli adipociti sono stati isolati da sei cuscinetti adiposi, è stata eseguita l'estrazione dell'RNA a base di eptano (fase 2) e l'RNA risultante è stato analizzato sullo strumento di elettroforesi automatizzato. Il numero di integrità dell'RNA (RIN) calcolato per tutti i campioni era >8 (Figura 3A), indicando una preparazione di RNA di alta qualità e riproducibile. Il kit di preparazione dell'RNA totale è stato quindi utilizzato per preparare le librerie di RNA e ogni campione è stato sequenziato sul sequenziatore di nuova generazione per raggiungere una profondità di lettura di ≥40 milioni di letture. Il punteggio Phred per tutti i campioni (Figura 3B) e per sequenza (Figura 3C) è stato di ≥30, indicativo di sequenze di DNA di alta qualità20. Pertanto, la rimozione dell'eptano supporta l'isolamento di RNA ad alto rendimento e di alta qualità adatti al sequenziamento dell'RNA.
Nella fase di fissazione della formaldeide, sono state eseguite anche tre preparazioni di isolamento nucleare contenenti eptano e una preparazione di controllo senza eptano. La cromatina tranciata è stata quindi analizzata sullo strumento di elettroforesi automatizzato. In entrambi i casi, la cromatina è stata tranciata a un intervallo di dimensioni 100-800 bp (Figura 4A), ideale per le procedure ChIP-seq a valle19. È importante sottolineare che ~5 volte più cromatina è stata ottenuta dai campioni trattati con eptano (11,5 ng/μL, 9,42 ng/μL e 9,6 ng/μL) rispetto ai campioni non trattati (2,2 ng/μL). H3K4me3 e H3K27me3 ChIP-seq sono stati eseguiti seguendo Arrigoni et al.19. Come mostrato nella Figura 4B, le tracce del segnale provenienti da tre campioni indipendenti trattati con eptano erano paragonabili alla traccia del campione non trattato con eptano per entrambi i segni istonici. Il trattamento con eptano non interferisce con la qualità del ChIP-seq, ma migliora significativamente la resa dei nuclei (e della cromatina tranciata).
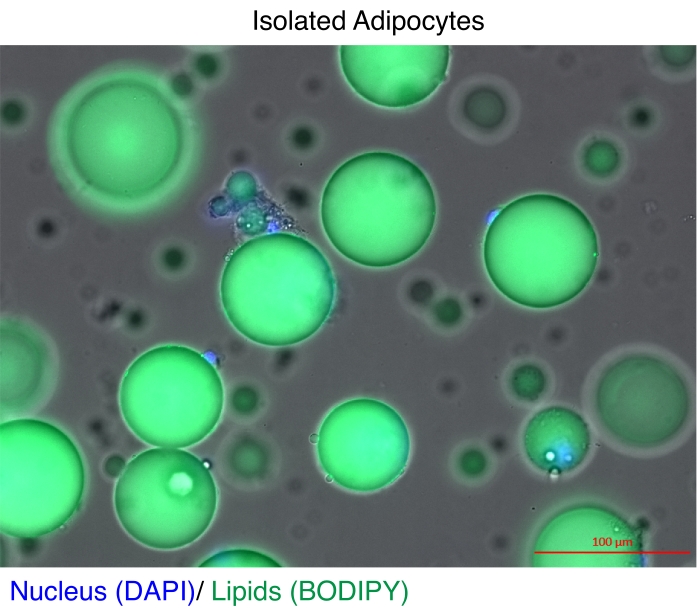
Figura 1: Adipociti isolati. Gli adipociti isolati sono stati colorati con DAPI (blu) per colorare il nucleo e BODIPY (verde) per i lipidi. Le goccioline lipidiche citosoliche comprendono fino al 95% del volume degli adipociti e rappresentano una sfida tecnica per ottenere rese elevate durante le estrazioni di RNA e cromatina. Barra della scala = 100 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
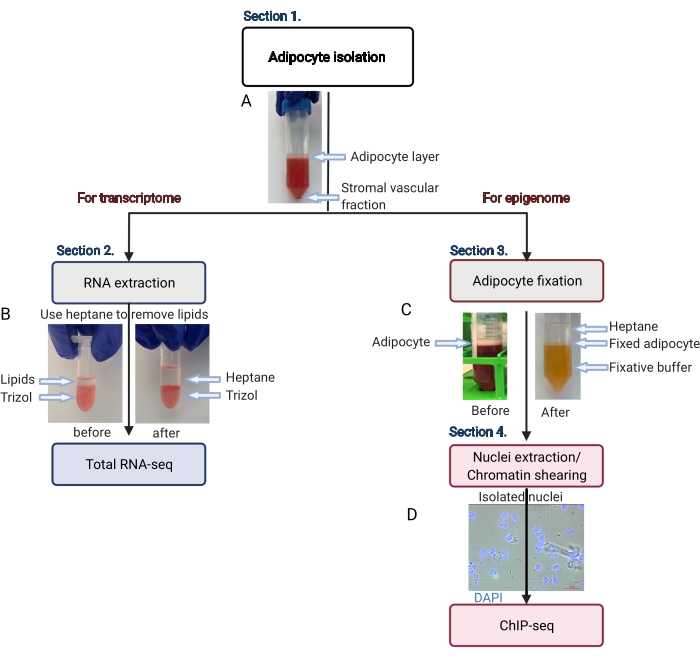
Figura 2: Flusso schematico dell'isolamento degli adipociti per l'analisi del trascrittoma e dell'epigenoma. Viene illustrato l'intero flusso di lavoro, dall'isolamento degli adipociti all'applicazione del trascrittoma o dell'epigenoma. Vengono mostrati i passaggi chiave e i risultati rappresentativi. (A) Gli adipociti isolati galleggiano sullo strato superiore, separandosi dalla frazione vascolare stromale come un pellet sul fondo del tubo. (B) L'uso dell'eptano per rimuovere i lipidi prima dell'estrazione dell'RNA con il reagente di isolamento dell'RNA. (C) L'uso dell'eptano per rimuovere i lipidi durante la fissazione degli adipociti. (D) L'immagine rappresentativa dei nuclei degli adipociti isolati deve essere intatta e rotonda. Barra della scala = 20 μm. Il programma è stato creato con BioRender.com. Clicca qui per visualizzare una versione più grande di questa figura.
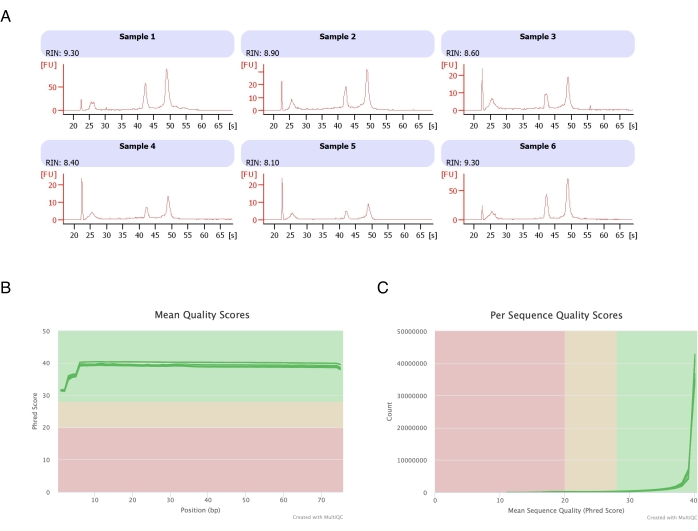
Figura 3: Elettroferogramma rappresentativo dei punteggi di integrità e qualità dell'RNA dopo RNA-seq. (A) I campioni 1-6 rappresentano sei repliche di adipociti trattati con eptano e la loro analisi dell'integrità dell'RNA è stata eseguita sullo strumento automatizzato per elettroforesi. Il numero di integrità dell'RNA (RIN) si basava sui rapporti dell'RNA ribosomiale 18S e 28S e rappresenta la qualità dell'RNA. Scala da 1 (molto degradato) a 10 (il meno degradato). (B,C) Il punteggio di qualità della lettura del sequenziamento è stato determinato mediante analisi multiQC (B) per il punteggio medio di qualità sulle basi (C) e per il punteggio di qualità per sequenza. Clicca qui per visualizzare una versione più grande di questa figura.
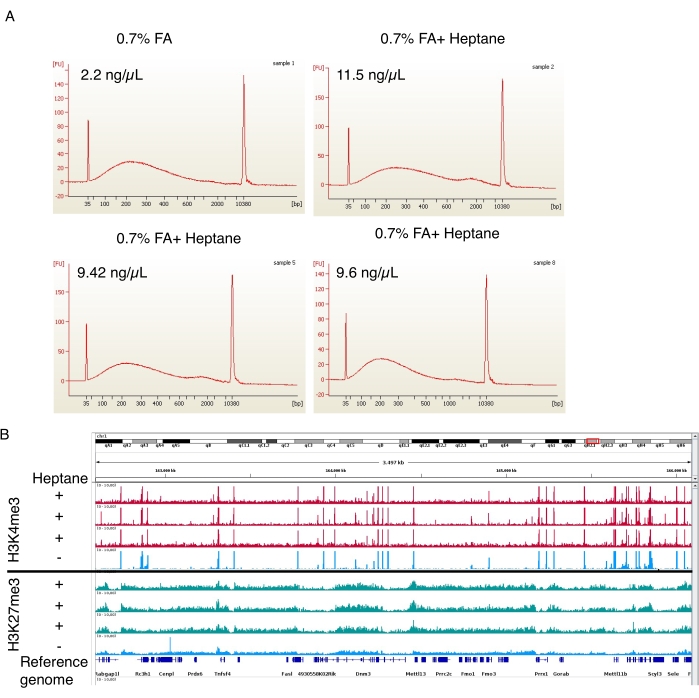
Figura 4: Elettroferogramma rappresentativo della cromatina tranciata e dei picchi di arricchimento da ChIP-seq. (A) Profili rappresentativi dallo strumento di elettroforesi automatizzato che mostrano le distribuzioni dimensionali della cromatina del controllo (campione 1, in alto a sinistra) e delle preparazioni di cromatina adipocitaria trattate con eptano (campioni 2, 5 e 8, in alto a destra e due in basso). Le concentrazioni di cromatina nelle preparazioni finali sono indicate in alto a sinistra di ogni immagine. (B) Screenshot del browser del genoma realizzato utilizzando l'Integrative Genomics Viewer (IGV). La parte superiore del grafico mostra i profili ChIP-seq H3K4me3 per tre campioni trattati con eptano (rosso) e un controllo (blu). Il pannello inferiore mostra lo stesso per il ChIP-seq di H3K27me3. Clicca qui per visualizzare una versione più grande di questa figura.
| Buffer | Composizione |
| Tampone per la digestione (per 3000 mg o meno di cuscinetto adiposo/20 ml) | 20 ml di Dulbecco's Modified Eagle Medium (DMEM) |
| 0,3 g di BSA senza acidi grassi | |
| 0,1 g di collagenasi di tipo 2 | |
| Tampone di laboratorio Farnham | TUBI 5 mM (pH 8) |
| 85 mM KCl | |
| 0,5% IGEPAL | |
| Buffer di tranciatura | 10 mM Tris-HCl pH 8 |
| 0,1% SDS | |
| 1 mM EDTA |
Tabella 1: Composizione dei diversi tamponi utilizzati nel presente studio.
Discussione
Il protocollo di isolamento degli adipociti qui presentato si basa su metodi di digestione enzimatica ben accettati11,14 per separare gli adipociti maturi (galleggianti) dalla frazione vascolare stromale rimanente del tessuto adiposo bianco. Fornisce un approccio semplice e universale alla purificazione degli adipociti maturi in qualsiasi modello murino. Come dimostrato in precedenza, il protocollo è adatto per l'uso a valle per l'analisi dell'intero trascrittoma e del ChIP-seq. Fornisce una resa sufficiente per generare più profili epigenomici (ad esempio, diverse modifiche istoniche più RNA-seq) dallo stesso cuscinetto adiposo individuale.
Per garantire le rese elevate che consentono la preparazione di tali dati epigenomici corrispondenti, un passaggio critico consiste nell'utilizzare l'eptano per dissolvere i lipidi dagli adipociti intatti e dagli adipociti che si lisano durante il processo. Nella nostra esperienza, questo passaggio aumenta sostanzialmente la riproducibilità e la resa impedendo che l'RNA e i nuclei vengano persi nello strato lipidico. La rotazione continua delle tube contenenti gli adipociti sottoposti a fissazione è altrettanto importante in quanto migliora l'omogeneità con cui i lipidi vengono rimossi dagli adipociti. Invece del tampone fissativo di formaldeide all'1% riportato in gran parte della letteratura ChIP-seq21,22, è stato riscontrato che è necessario ridurre la concentrazione allo 0,7% di formaldeide per evitare l'eccessiva fissazione. Anche il tempo di taglio della cromatina è stato ottimizzato a 12 minuti per ottenere un intervallo di dimensioni ottimali della cromatina (100-800 bp) per ChIP-seq.
Il protocollo è stato ottimizzato per studi di trascrittoma di massa ed epigenoma. L'attuale protocollo ha ancora i suoi limiti per le applicazioni di trascrittoma ed epigenoma a singola cellula. Considerando l'eterogeneità cellulare riportata nel campo di adiposità 23,24, questo metodo di isolamento potrebbe essere ulteriormente sviluppato per essere adattato per saggi su singola cellula. In tali contesti, marcatori adipocitari limitati come il boro-dipirrometene (BODIPY)25 e la perilipina-1 (PLIN1)26, ASC-127 o marcatori nucleici adipociti-specifici potrebbero essere preziosi per consentire la quantificazione di parametri cellulari aggiuntivi, funzionali e rilevanti28. Oltre all'applicazione agli studi genomici, questo protocollo potrebbe anche migliorare gli studi di proteomica degli adipociti, che hanno un ulteriore ostacolo a causa dell'elevato contenuto di lipidi negli adipociti29.
Questo protocollo può essere utilizzato anche per estrarre con successo nuclei di adipociti umani (dati non mostrati), estendendo la sua applicazione alla ricerca sull'obesità umana.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Siamo in debito con l'imaging ottico MPI-IE, il sequenziamento, il nucleo bioinformatico e il personale. Questo lavoro è stato sostenuto da finanziamenti dell'MPG, dell'Istituto di ricerca Van Andel, della ricerca Horizon 2020 dell'Unione Europea, degli NIH (R01HG012444 e R21HG011964), dell'accordo di sovvenzione Marie Skłodowska-Curie n. 675610 e del Ministero federale dell'Istruzione e della Ricerca nell'ambito del progetto numero 01KU1216 (Deutsches Epigenom Programm, DEEP).
Materiali
| Name | Company | Catalog Number | Comments |
| 16% Formaldehyde Solution (w/v). Methanol-free | ThermoFisher SCIENTIFIC | 28908 | |
| 1-bromo-3-chloropropane | SIGMA | B9673 | |
| 5 M NaCl | invitrogen | AM9759 | |
| Automated electrophoresis instrument (2100 Bioanalyzer Instrument) | Agilent Technologies | G2939BA | |
| BODIPY | ThermoFisher SCIENTIFIC | D3922 | |
| Collagenase Type 2 | Worthington Biochemical Corp. | 43D14184B | |
| Complete, EDTA-free, Protease inhibitor cocktail (PIC) | Roche | 4693159001 | EDTA free |
| DAPI | ThermoFisher SCIENTIFIC | D1306 | |
| DMEM (+ GlutaMAX) | life technologies | 61965-026 | |
| DNA purification kit (PCR purification kit) | Qiagen | 28104 | |
| DNA quantification instrument (Qubit 4 Fluorometer) | Fisher SCIENTIFIC | Q33238 | |
| DNA quantification kit (DNA high sensitivity assay) | Agilent Technologies | 5067-4626 | |
| EDTA | Fisher chimical | E478-500 | |
| EGTA | Fisher chimical | O2783-100 | |
| EtOH | PHARMCO | 111000200 | |
| Fatty acid-free BSA | SERVA | 11932.01 | bovine albumin fraction V, fatty acid-free lyophilized |
| Glycine | SIGMA | G7126-100G | |
| Heptane | SIGMA | H2198-1L | |
| High Sensitivity RNA Analysis | Agilent Technologies | 5067-1513 | |
| Igepal | SIGMA | 18896-50ML | |
| KCl | SIGMA | P3911-25G | |
| Matrix filter (420um) | Tisch Scientific | ME17238 | |
| Next-Generation Sequencer (HiSeq 3000 Sequencer) | Illumina | ||
| Nuclease-free water | Invitrogen | 4387936 | |
| PIPES | ACROS organics | 5625-37-6 | |
| Proteinase K | ThermoFisher SCIENTIFIC | EO0491 | |
| RNA isolation reagent (Trizol) | ThermoFisher SCIENTIFIC | 15596026 | |
| RNase A, DNase-free | ThermoFisher SCIENTIFIC | EN0531 | |
| Rotator (Thermo Scientific Tube Revolver Rotator) | ThermoFisher SCIENTIFIC | 88881001 | |
| SDS, Sodium dodecyl sulfate | SIGMA | 8170341000 | |
| Sonication tube (1 mL milliTUBE with AFA Fiber) | Covaris | 520135 | |
| Sonicator (Evolution Focused-ultra-sonicator (S220 or E220)) | Covaris | 500217 or 500239 | |
| Total RNA Prep kit ( Illumina Stranded Total RNA Prep kit) | Illumina | 20040525 | |
| Tris-HCl | Fisher bioreagents | BP153-500 |
Riferimenti
- Albuquerque, D., Stice, E., Rodríguez-López, R., Manco, L., Nóbrega, C. Current review of genetics of human obesity: From molecular mechanisms to an evolutionary perspective. Molecular Genetics and Genomics. 290 (4), 1191-1221 (2015).
- Weihrauch-Blüher, S., Schwarz, P., Klusmann, J. H. Childhood obesity: Increased risk for cardiometabolic disease and cancer in adulthood. Metabolism. 92, 147-152 (2019).
- Weihrauch-Blüher, S., Wiegand, S. Risk factors and implications of childhood obesity. Current Obesity Reports. 7 (4), 254-259 (2018).
- Gluckman, P. D. Epigenetics, the life-course and metabolic disease. Nature Reviews Endocrinology. 8 (2), 74-76 (2012).
- Loh, M., Zhou, L., Ng, H. K., Chambers, J. C. Epigenetic disturbances in obesity and diabetes: Epidemiological and functional insights. Molecular Metabolism. 27, 33-41 (2019).
- Sayols-Baixeras, S., et al. DNA methylation and obesity traits: An epigenome-wide association study. The REGICOR study. Epigenetics. 12 (10), 909-916 (2017).
- Xia, Q., Grant, S. F. A. The genetics of human obesity. Annals of the New York Academy of Sciences. 1281 (1), 178-190 (2013).
- Yokoi, N. Epigenetic dysregulation in pancreatic islets and pathogenesis of type 2 diabetes. Journal of Diabetes Investigation. 9 (3), 475-477 (2018).
- Frayn, K. N. Obesity and metabolic disease: Is adipose tissue the culprit. Proceedings of the Nutrition Society. 64 (1), 7-13 (2005).
- Kawai, T., Autieri, M. V., Scalia, R. Adipose tissue inflammation and metabolic dysfunction in obesity. American Journal of Physiology-Cell Physiology. 320 (3), 375-391 (2021).
- Cat, A. N. D., Briones, A. M., Touyz, R. M., Schiffrin, E. L. . Hypertension: Methods and Protocols. , 283-295 (2017).
- Ambati, S., et al. Adipocyte nuclei captured from VAT and SAT. BMC Obesity. 3, 35 (2016).
- Roh, H. C., et al. Simultaneous transcriptional and epigenomic profiling from specific cell types within heterogeneous tissues in vivo. Cell Reports. 18 (4), 1048-1061 (2017).
- Jacobsen, M. J., et al. Epigenetic and transcriptomic characterization of pure adipocyte fractions from obese pigs identifies candidate pathways controlling metabolism. Frontiers in Genetics. 10, 1268 (2019).
- Janke, J., Engeli, S., Gorzelniak, K., Sharma, A. M. Extraction of total RNA from adipocytes. Hormone and Metabolic Research. 33 (4), 213-215 (2001).
- Cirera, S. Highly efficient method for isolation of total RNA from adipose tissue. BMC Research Notes. 6 (1), 472 (2013).
- Rajbhandari, P., et al. Single cell analysis reveals immune cell-adipocyte crosstalk regulating the transcription of thermogenic adipocytes. eLife. 8, 49501 (2019).
- Buddrick, O., Jones, O. A. H., Morrison, P. D., Small, D. M. Heptane as a less toxic option than hexane for the separation of vitamin E from food products using normal phase HPLC. RSC Advances. 3 (46), 24063-24068 (2013).
- Arrigoni, L., et al. Standardizing chromatin research: A simple and universal method for ChIP-seq. Nucleic Acids Research. 44 (7), 67 (2016).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Schmidt, D., et al. ChIP-seq: Using high-throughput sequencing to discover protein-DNA interactions. Methods. 48 (3), 240-248 (2009).
- Hoffman, E. A., Frey, B. L., Smith, L. M., Auble, D. T. Formaldehyde crosslinking: A tool for the study of chromatin complexes. Journal of Biological Chemistry. 290 (44), 26404-26411 (2015).
- Rondini, E. A., Granneman, J. G. Single cell approaches to address adipose tissue stromal cell heterogeneity. Biochemical Journal. 477 (3), 583-600 (2020).
- Corvera, S. Cellular heterogeneity in adipose tissues. Annu Review of Physiology. 83, 257-278 (2021).
- Katz, L. S., Geras-Raaka, E., Gershengorn, M. C. Heritability of fat accumulation in white adipocytes. American Journal of Physiology-Endocrinology and Metabolism. 307 (3), 335-344 (2014).
- Hansen, J. S., de Maré, S., Jones, H. A., Göransson, O., Lindkvist-Petersson, K. Visualization of lipid directed dynamics of perilipin 1 in human primary adipocytes. Scientific Reports. 7 (1), 15011 (2017).
- Ussar, S., et al. ASC-1, PAT2, and P2RX5 are cell surface markers for white, beige, and brown adipocytes. Science Translational Medicine. 6 (247), (2014).
- Onogi, Y., Khalil, A. E. M. M., Ussar, S. Identification and characterization of adipose surface epitopes. Biochemical Journal. 477 (13), 2509-2541 (2020).
- Kim, E. Y., et al. Recent advances in proteomic studies of adipose tissues and adipocytes. International Journal of Molecular Sciences. 16 (3), 4581-4599 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon