Method Article
Transcriptome 및 Epigenome 분석을 위한 Murine White adipocytes의 분리 및 처리
요약
본 프로토콜은 다운스트림 전체 RNA 염기서열분석, NEXSON(Nuclei Extraction by SONication) 및 ChIP-seq에 최적화된 쥐 백색 지방세포의 분리, 정제 및 업스트림 처리를 위한 보편적인 방법을 요약합니다.
초록
비만은 유전학, 후성유전학, 환경 및 이들의 상호 작용에 의해 영향을 받는 복잡한 질병입니다. 성숙한 지방세포(adipocyte)는 백색 지방 조직의 주요 세포 유형을 나타냅니다. 지방세포가 어떻게 기능하고 (후성)유전적, 환경적 신호에 반응하는지 이해하는 것은 비만의 원인을 규명하는 데 필수적입니다. RNA와 염색질은 이전에 효소 분해를 사용하여 지방 세포에서 분리되었습니다. 또한, 핵 분리를 위한 프로토콜이 개발되었으며, 여기서 정제는 지방 세포 특이적 형질전환 리포터의 형광 활성화 세포 분류(FACS)를 통해 이루어집니다. 이러한 프로토콜 동안 높은 수율과 품질을 달성하기 위한 가장 큰 과제 중 하나는 지방 조직에 포함된 상당한 양의 지질입니다. 본 프로토콜은 헵탄을 활용하여 관심 대상(RNA/염색질)에서 지질을 분리하는 성숙한 지방 세포를 분리하기 위한 최적화된 절차를 설명합니다. 그 결과 생성된 RNA는 무결성이 높고 고품질 RNA-seq 결과를 생성합니다. 마찬가지로, 이 절차는 핵 수율을 개선하고 시료 전반에 걸쳐 재현 가능한 ChIP-seq 결과를 생성합니다. 따라서 현재 연구는 전체 게놈 전사체 및 후성유전체 연구에 적합한 신뢰할 수 있고 보편적인 쥐 지방 세포 분리 프로토콜을 제공합니다.
서문
비만은 일반적으로 제2형 당뇨병, 심장대사질환 및 여러 형태의 암의 위험을 높이는 데 기여하는 과도한 지방 축적 질환으로 이해됩니다 1,2,3. 비만에 대한 현재의 이해는 유전학(인간 및 설치류 연구 모두)에 크게 뿌리를 두고 있지만, 대사 질환 소인의 약 30%-70%는 기원이 비유전적이며 4,5,6,7,8 여전히 잘못 정의되어 있습니다.
지방 조직은 비만 및 기타 대사 질환에서 중요한 역할을 합니다 9,10. 지방 조직은 성숙한 지방세포와 전지방세포, 내피 세포 및 면역 세포를 포함한 기질 혈관 분획으로 구성됩니다. 각 세포 유형이 비만에 어떻게 기여하는지, 지방 세포 조절 장애가 비만에 어떻게 기여하는지는 아직 불분명합니다. 성숙한 지방 세포 후성유전체 연구를 위한 재현 가능하고 효과적인 분리 및 정제 프로토콜은 이 분야의 관심사입니다.
성숙한 지방세포는 유전자 발현 분석(11)과 후성유전체(epigenome) 연구12,13를 위해 오랫동안 분리되어 왔다. 지방 세포를 분리하는 두 가지 주요 전략이 있습니다. 첫 번째는 효소 소화를 사용하여 기질 혈관 분획11,14의 나머지 세포 유형에서 성숙한 지방 세포를 분리하는 것입니다. 두 번째는 지방 조직을 떨어뜨려 온전한 핵을 방출한 다음 형광 활성화 세포 분류(FACS)12,13에 의해 형광 리포터를 기반으로 핵을 회수하는 것인데, 이를 위해서는 특수 형질전환 리포터 모델이 필요합니다. 각 사례의 기술적 과제는 성숙한 지방세포가 고농도의 지질을 함유하고 있으며(그림 1), 이는 총 RNA15,16 및 핵17의 품질 및/또는 수율을 감소시킨다는 것입니다. 여기에서는 성숙한 지방 세포를 분리하기 위한 최적화된 효소 분해 절차가 설명되며, 여기서 헵탄의 장점은 RNA 추출 또는 SONication(NEXSON)에 의한 핵 분리 단계 전에 지질(18)을 빠르고 효율적으로 용해 및 제거하는 것입니다(19). 이 프로토콜은 게놈 전체 연구를 위해 전체 RNA의 우수한 회수율과 품질을 보장하고 재현성 있는 ChIP-seq를 위해 온전한 핵의 수율을 크게 향상시킵니다.
프로토콜
모든 동물 실험은 IACUC(Institutional Animal Care and Use Committee)의 승인을 받았으며 프로토콜 번호: 18-10-028입니다. 생후 12주 된 수컷 C57BL/6J 마우스를 CO2 로 안락사시키고 부고환 백색 지방 조직(eWAT)에서 지방 패드를 채취하기 위해 해부했습니다.
1. 지방세포 격리
- 소화 완충액(표 1)을 준비하고 수조에서 37°C로 예열합니다.
- 부고환 백색 지방 조직(eWAT)의 지방 패드를 페트리 접시에 넣습니다. 겸자와 메스를 사용하여 조직을 5mL의 DMEM에 넣고 작은 조각(5mm x 5mm)으로 자릅니다.
참고: 세포 접착 코팅이 포함되지 않은 페트리 접시가 선호됩니다. 세포가 손상되지 않도록 지방 패드를 부드럽게 잡는 것도 중요합니다. 지방 조직을 더 많이 처리할수록 수율이 더 많이 손상됩니다. 메스/겸자 방법은 지방 세포를 손상시킬 수 있으므로 가위나 집게만으로 지방 패드를 절단하는 것보다 선호됩니다. - 조직 파편을 방해하지 않고 과도한 DMEM을 배출합니다.
알림: 페트리 접시 표면에서 조직 찌꺼기를 제거하지 마십시오. - 페트리 접시에 5mL의 소화 완충액을 넣고 소용돌이쳐 접시에서 조직 조각을 분리한 다음 전체 내용물을 50mL 원심분리 튜브에 붓습니다. 모든 조직 조각이 원심분리기 튜브로 옮겨질 때까지 이 과정을 반복합니다.
- 최종 부피가 ~20mL가 되도록 원심분리 튜브에 남은 분해 버퍼를 추가합니다.
- 조직 조각을 37°C 진탕 수조에서 100rpm으로 20-30분 동안 또는 조직 조각이 1mm x 1mm보다 작아질 때까지 배양합니다.
- 원심분리 튜브에 0.4mL의 0.5M EDTA와 0.2mL의 0.5M EGTA를 넣고 37°C에서 10분 동안 계속 흔듭니다.
- 10mL 피펫으로 세포 현탁액을 다른 50mL 원심분리 튜브 위에 미세 매트릭스 필터(420μm, 재료 표 참조)를 통해 옮기고 분해되지 않은 조직을 제거합니다.
- 50mL 원심분리 튜브를 실온에서 200 x g 으로 5분 동안 원심분리하여 기질 혈관 분획(아래쪽 펠릿)에서 순수한 지방 세포(위쪽에 떠 있음)를 분리합니다(그림 2A).
- 떠다니는 지방 세포층을 새로운 50mL 튜브에 붓고 펠릿이 방해받지 않도록 주의합니다.
참고: 부유 지방 세포를 전달하기 위해 피펫 팁을 사용하지 마십시오. 이는 피펫 팁 벽에 달라붙을 수 있으므로(따라서 수율을 감소시킬 수 있음). 일부 소화 버퍼는 새 튜브로 전달됩니다. 주사기와 바늘을 사용하여 지방 세포층 아래에 남아 있는 소화 완충액을 제거합니다. - 총 RNA 추출(2단계) 및/또는 ChIP-seq(3단계 및 4단계)를 위해 정제된 지방세포를 즉시 사용합니다.
참고: 사용자는 2단계에 전체 지방 세포 추출물을 사용하기로 결정할 수 있습니다. 또는 3단계, 연구 목적에 따라. 사용자는 또한 전체 지방 세포 추출물을 두 부분으로 나누고 2단계를 계속할 수 있습니다. 및 3단계. 생물학적으로 동일한 샘플에서 분석 결과를 얻기 위해 병행합니다.
2. 총 RNA 추출
알림: 화학 물질 후드에서 이 단계를 수행하십시오.
- RNA 분리 시약(1mL)( 재료 표 참조)을 50mL 원심분리 튜브의 정제된 지방 세포에 추가합니다(1.11단계).
- 1mL 피펫 팁을 사용하여 위아래로 피펫팅하여 RNA 분리 시약의 지방 세포를 완전히 용해하고 2mL 마이크로 원심분리 튜브로 옮깁니다.
- 실온에서 15분 동안 튜브를 배양합니다.
- 0.5mL의 헵탄을 원심분리기 튜브에 넣고 30초 동안 최고 속도로 와류에 넣습니다.
참고: 지질은 헵탄에 용해됩니다(그림 2B). - 튜브를 1,000 x g 에서 4°C에서 10분 동안 원심분리합니다.
- 주사기와 30G 바늘로 RNA 분리 시약의 맨 아래층을 확보하고 새로운 1.5mL 튜브로 옮깁니다.
- 0.1mL의 1브로모-3클로로프로판( 재료 표 참조)을 RNA 분리 시약 추출물에 추가하고 30초 동안 최고 속도로 와류를 일으킵니다. 튜브를 실온에서 15분 동안 배양합니다.
- 튜브를 12,000 x g 에서 4°C에서 15분 동안 원심분리합니다. 피펫을 사용하여 상부 수성상을 수집하여 새 튜브로 옮깁니다.
- 0.5mL의 이소프로판올을 넣고 4°C에서 10분 동안 배양합니다. 튜브를 12,000 x g 에서 4°C에서 10분 동안 원심분리합니다.
참고: RNA는 튜브 바닥에 펠릿으로 침전됩니다. - 피펫을 사용하여 상층액을 제거하고 펠릿을 만지지 않도록 주의하십시오.
- 75% EtOH 1mL를 넣고 최대 속도로 와류를 짧게 합니다.
- 튜브를 7,500 x g 에서 4°C에서 5분 동안 원심분리합니다. 피펫 팁으로 상층액을 조심스럽게 제거합니다. 펠릿을 방해하지 마십시오.
- 펠릿을 실온에서 10분 동안 또는 펠릿이 투명해지고 투명해질 때까지 자연 건조합니다.
- 펠릿을 뉴클레아제가 없는 물 50μL에 용해시킵니다.
3. ChIP-seq를 위한 지방세포 정착
참고: 3단계를 수행합니다. 화학 후드에.
- 0.3mL의 0.5M EDTA, 0.2mL의 0.5M EGTA, 14.8mL의 DMEM 및 10mL의 헵탄을 분리된 지방세포가 포함된 50mL 튜브에 추가합니다(1.11단계).
- 0.7mL의 16% 포름알데히드(최종 0.7%)를 넣고 실온에서 10rpm으로 5분 동안 튜브 회전기( 재료 표 참조)에서 회전합니다.
알림: 타이머를 설정하고 5분 동안 계산합니다. 여러 샘플을 처리하는 경우 30초 간격으로 포름알데히드를 순차적으로 첨가하여 모든 샘플에 걸쳐 정확하고 비교 가능한 고정 타이밍을 가능하게 합니다. - 1.78mL의 1.25M 글리신(최종 0.125M)을 추가하고 실온에서 10rpm으로 5분 이상 회전하지 않고 혼합합니다.
참고: 포름알데히드는 글리신을 첨가한 후에도 활성 상태를 유지합니다. 따라서 5분 배양 단계 직후 원심분리(3.4단계)로 층을 분리하는 것이 중요합니다. 샘플이 두 개 이상인 경우 각 샘플에 글리신을 추가하여 30초의 시간 간격으로 순차적으로 반응을 중지합니다. - 다음으로, 실온에서 200 x g 에서 5분 동안 원심분리합니다.
참고: 세 개의 명확하고 분리된 액체층이 있어야 합니다(그림 2C). 중간 흰색 층에는 고정 된 지방 세포가 포함되어 있습니다. - 1mL 피펫 팁을 사용하여 지방 세포(흰색) 층을 새로운 15mL 원심분리 튜브로 이동시켜 새 튜브로 운반되는 고정액 및 헵탄의 양을 최소화합니다.
- 프로테아제 억제제 칵테일(PIC, 재료 표 참조) 정제가 보충된 10mL(실온) DMEM으로 튜브를 채웁니다. 반전으로 철저히 혼합하십시오.
- 실온에서 ≤200 x g 의 튜브를 5분 동안 원심분리합니다. 세포 파열을 최소화하기 위해 원심분리력을 100 x g 로 유지하십시오.
- 3.5.-3.7단계를 반복합니다. 3.9단계로 진행하기 전에 1배.
- 1mL 피펫 팁을 사용하여 지방세포(흰색) 층을 1.5mL 또는 2mL 마이크로 원심분리 튜브로 옮깁니다.
- 실온에서 100-200 x g 의 튜브를 5분 동안 원심분리하고 1ml 피펫 팁을 사용하여 헵탄(맨 위 층)과 DMEM(맨 아래 층)을 제거합니다.
- 고정 지방 세포는 사용할 때까지 최대 6개월 동안 -80°C에서 보관하십시오.
4. 핵 적출과 염색질 깎는
참고: 이 절차는 Arrigoni et al.19에서 발췌한 것입니다.
- 초음파 처리기를 켜고( 재료 표 참조) 수조 냉각기를 200°C로 설정하고 피크 전력을 75W로, 듀티 팩터를 2%로, 200사이클/버스트로 설정합니다.
- 프로테아제 억제제 칵테일(PIC)을 Farnham 실험실 완충액과 전단 완충액에 추가합니다(표 1).
참고: Arrigoni et al.19에서 제안된 몇 가지 초음파 발생기 및 매개변수를 확인하십시오. - 1mL 초음파 처리 튜브에 750-800 μL의 얼음처럼 차가운 Farnham 실험실 완충액으로 고정 된 지방 세포를 재현 탁 (3 단계에서)합니다 ( 재료 표 참조).
알림: 튜브를 최대 용량으로 채우지 않는 것이 좋습니다. 그렇지 않으면 지방 세포가 튜브의 가장 높은 부분으로 떠올라 초음파 처리 효율이 감소합니다. - 샘플을 2.5분 동안 초음파 처리합니다.
참고: 위상차 현미경에서 핵 추출을 확인하여 추가 초음파 처리가 필요한지 확인하십시오. 핵은 그림 2D와 같이 둥글고 손상되지 않아야 합니다. - 계속 진행하기 전에 초음파 발생기 피크 전력을 140W로, 듀티 팩터를 5 %로, 수조 냉각기를 200 ° C로 설정 한 상태에서 4 사이클 / 버스트로 설정하십시오.
- 피펫을 사용하여 상층액을 1.5mL 마이크로 원심분리 튜브로 옮깁니다. 튜브를 1,000 x g 에서 4°C에서 5분 동안 원심분리합니다.
- 펠릿(핵)에서 상등액을 조심스럽게 제거하고 펠릿을 보관하십시오.
- 얼음에 Farnham lab buffer 1mL로 펠릿을 씻습니다.
- 튜브를 1,000 x g 에서 4°C에서 5분 동안 원심분리하고 피펫을 사용하여 상층액을 제거한 다음 펠릿을 보관합니다.
- 4.8.-4.9단계를 반복합니다. 4.11단계로 진행하기 전에 1배.
참고: 분리 및 세척된 핵은 추가 사용까지 3개월 동안 -80°C에서 보관할 수 있습니다. - 분리된 핵을 1mL의 전단 완충액에 재현탁시키고 새로운 1mL 초음파 처리 튜브로 옮깁니다. 튜브에 기포가 없는지 확인하십시오.
- 140W의 피크 출력, 5%의 듀티 팩터 및 12분 동안 200 사이클/버스트로 초음파 처리하여 4°C에서 염색질을 전단합니다.
- 용해물을 1.5mL 마이크로분리 튜브로 옮깁니다.
참고: 전단된 염색질은 핵에서 방출되어 완충액(용해물)에 포함됩니다. - 튜브를 10,000 x g 에서 4°C에서 5분 동안 원심분리하여 불용성 파편을 펠릿으로 만듭니다. 상층액(크로마틴)을 새로운 1.5mL 마이크로 원심분리 튜브로 옮깁니다.
- 전단된 염색질은 4°C에서 10일 동안 보관하거나 -80°C에서 3개월 동안 사용할 때까지 보관할 수 있습니다.
참고: 추가 품질 검사: 이전에 발표된 보고서19에 따라 DNase-free RNase로 RNA를 탈가교결합하고 제거하기 위해 전단 염색질의 25μL를 분취합니다. 본 연구를 위해, 생성된 DNA는 제조사의 지침에 따라 DNA 정제 키트로 정제하고, DNA는 DNA 정량 기기에서 DNA 정량 키트로 정량화하고, 단편 크기는 자동 전기영동 기기로 평가했습니다( 재료 표 참조). 전단된 염색질 크기는 100-800bp 범위여야 합니다. 나머지 전단 염색질은 다운스트림 ChIP-Seq 응용 프로그램을 위해 진행할 수 있습니다.
결과
6개의 지방 패드에서 지방세포를 분리하고, 헵탄 기반 RNA 추출을 수행하고(2단계), 생성된 RNA를 자동 전기영동 기기에서 분석했습니다. 모든 샘플에 대해 계산된 RNA 무결성 수(RIN)는 >8(그림 3A)이었으며, 이는 고품질의 재현 가능한 RNA 준비를 나타냅니다. 그런 다음 전체 RNA 준비 키트를 사용하여 RNA 라이브러리를 준비하고, 각 샘플은 차세대 염기서열분석기에서 염기서열분석을 수행하여 4천만 판독 ≥ 판독 깊이에 도달했습니다. 모든 샘플(그림 3B) 및 염기서열별(그림 3C)에 대한 Phred 점수는 ≥30이었으며, 이는 고품질 DNA 염기서열20을 나타냅니다. 따라서 헵탄 제거는 RNA 염기서열분석에 적합한 고수율, 고품질 RNA의 분리를 지원합니다.
포름알데히드 고정 단계에서는 헵탄을 함유한 핵 분리 제제 3개와 헵탄이 없는 대조군 제제 1개도 수행했습니다. 그런 다음 전단된 염색질을 자동 전기영동 기기에서 분석했습니다. 두 경우 모두, 염색질은 100-800 bp 크기 범위(그림 4A)로 전단되었으며, 이는 다운스트림 ChIP-seq 절차19에 이상적입니다. 중요한 것은 헵탄 처리된 샘플(11.5ng/μL, 9.42ng/μL 및 9.6ng/μL)에서 처리되지 않은 샘플(2.2ng/μL)에 비해 ~5배 더 많은 염색질이 얻어졌다는 것입니다. H3K4me3 및 H3K27me3 ChIP-seq는 Arrigoni et al.19에 따라 수행되었습니다. 그림 4B에서 볼 수 있듯이, 3개의 독립적인 헵탄 처리된 샘플의 신호 트랙은 두 히스톤 마크에 대한 헵탄이 처리되지 않은 샘플의 트랙과 유사했습니다. 헵탄 처리는 ChIP-seq의 품질을 방해하지 않지만 핵(및 전단 염색질) 수율을 크게 향상시킵니다.
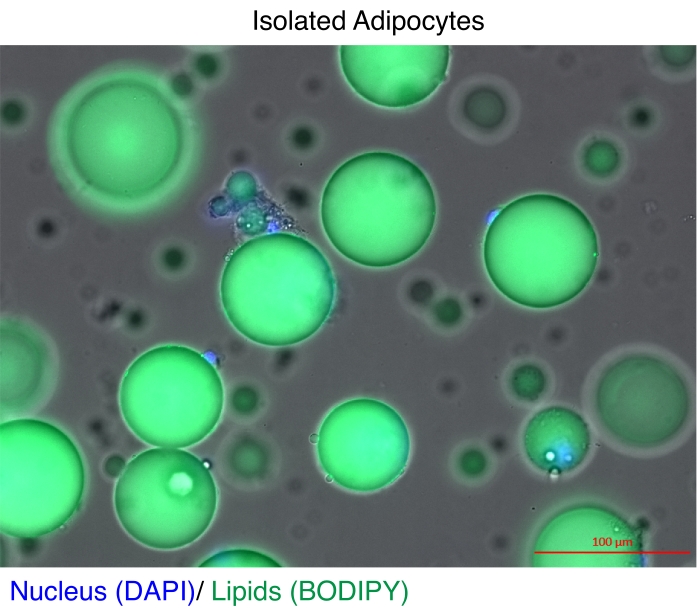
그림 1: 분리된 지방세포. 분리된 지방세포는 핵을 염색하기 위해 DAPI(파란색)로 염색하고 지질에 대해서는 BODIPY(녹색)로 염색했습니다. 세포질 지질 방울은 지방 세포 부피의 최대 95%를 구성하며 RNA 및 염색질 추출 중에 높은 수율을 달성하기 위한 기술적 과제를 제기합니다. 스케일 바 = 100μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
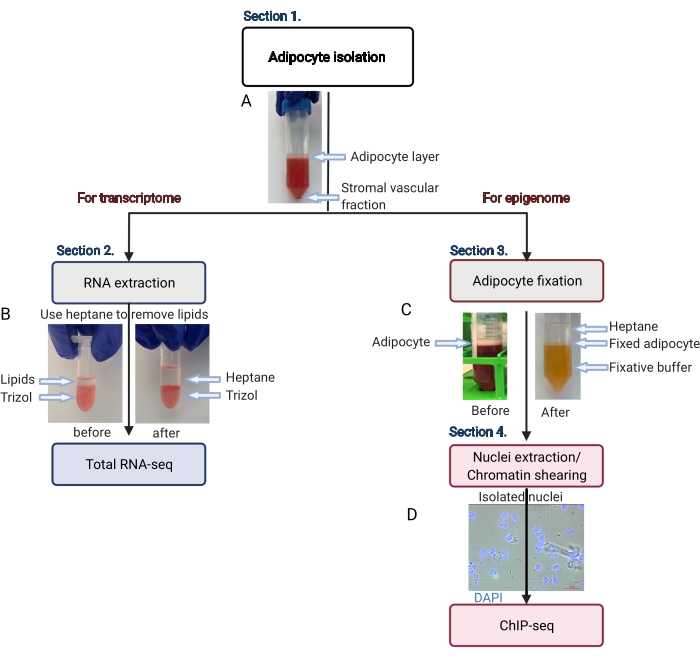
그림 2: 전사체 및 후성유전체 분석을 위한 지방세포 분리의 개략적 흐름. 지방 세포 분리에서 전사체 또는 후성유전체 적용에 이르기까지 전체 작업 흐름이 묘사되어 있습니다. 주요 단계와 대표적인 결과가 표시됩니다. (A) 분리된 지방세포는 최상층에 떠 있으며, 튜브 바닥에서 펠릿으로 기질 혈관 분획과 분리됩니다. (B) RNA 분리 시약으로 RNA를 추출하기 전에 지질을 제거하기 위해 헵탄을 사용하는 것. (C) 지방 세포 고정 중 지질을 제거하기 위해 헵탄을 사용하는 것. (D) 분리된 지방세포 핵의 대표 이미지는 온전하고 둥글어야 합니다. 스케일 바 = 20μm. 이 계획은 BioRender.com 함께 만들어졌습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
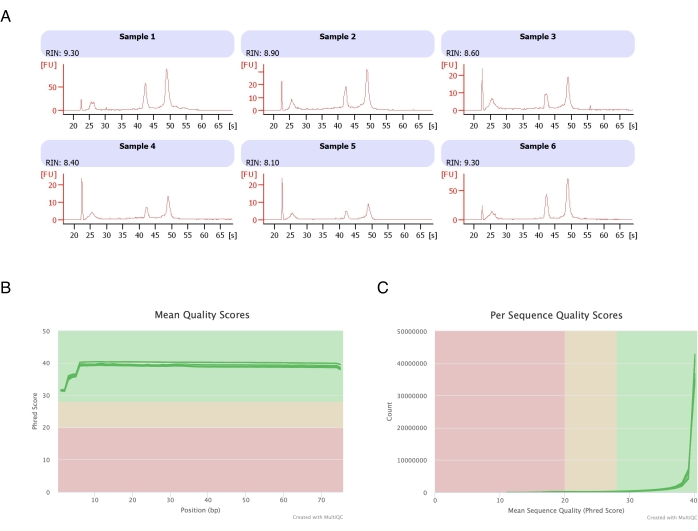
그림 3: RNA 염기서열 분석 후 RNA 무결성 및 품질 점수의 대표적인 전기표.(A) 샘플 1-6은 헵탄 처리된 지방세포의 6개 복제물을 나타내며, 이들의 RNA 무결성 분석은 자동 전기영동 기기에서 실행되었습니다. RNA 무결성 수(RIN)는 18S 및 28S 리보솜 RNA 비율을 기반으로 하며 RNA 품질을 나타냅니다. 1(많이 저하됨)에서 10(가장 성능이 낮음)까지 크기를 조정합니다. (B,C) 염기서열분석 판독 품질 점수는 염기(C)의 평균 품질 점수와 염기서열당 품질 점수에 대한 multiQC 분석(B)에 의해 결정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
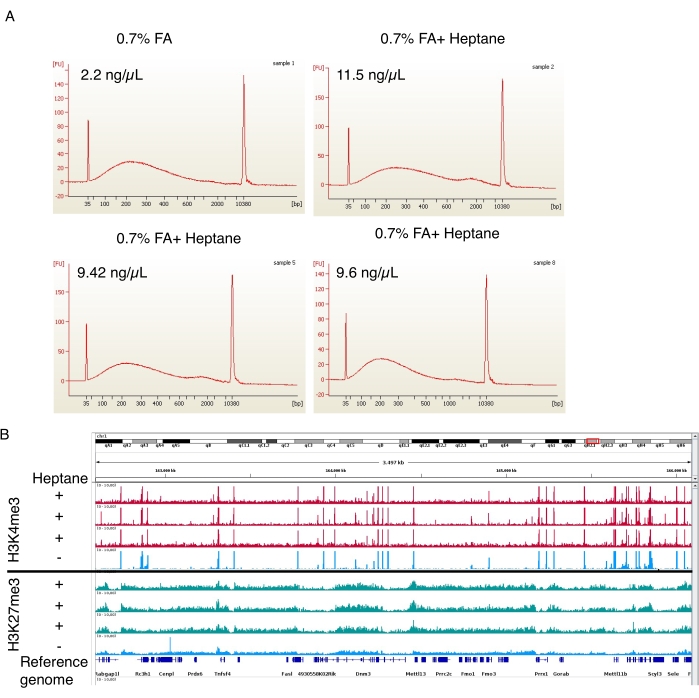
그림 4: ChIP-seq의 전단 염색질 및 농축 피크의 대표적인 전기영 동도.(A) 대조군(샘플 1, 왼쪽 상단)과 헵탄 처리된 지방 세포 염색질 제제(샘플 2, 5 및 8, 오른쪽 상단 및 하단 2)의 염색질 크기 분포를 보여주는 자동 전기영동 기기의 대표 프로파일. 최종 제제의 염색질 농도는 각 이미지의 왼쪽 상단에 표시되어 있습니다. (B) Integrative Genomics Viewer(IGV)를 사용하여 만든 게놈 브라우저 스크린샷. 플롯의 상단 부분은 3개의 (빨간색) 헵탄 처리 샘플과 1개의 (파란색) 대조군에 대한 H3K4me3 ChIP-seq 프로파일을 보여줍니다. 하단 패널은 H3K27me3의 ChIP-seq에 대해 동일하게 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 완충기 | 구성 |
| 소화 완충액 (지방 패드 3000mg 이하 / 20mL) | Dulbecco의 변형 이글 배지(DMEM) 20mL |
| 무 지방산 BSA 0.3 g | |
| 0.1g 콜라겐분해효소 2형 | |
| Farnham 실험실 버퍼 | 5mM 파이프(pH 8) |
| 85mM KCl | |
| 0.5% 이게팔 | |
| 전단 버퍼 | 10mM 트리스-HCl pH 8 |
| 0.1% 증권 시세 표시기 | |
| 1mM EDTA |
표 1: 본 연구에 사용된 다양한 완충액의 조성.
토론
여기에 제시된 지방세포 분리 프로토콜은 백색 지방 조직의 잔여 기질 혈관 분획에서 성숙한 지방세포(부유)를 분리하기 위해 잘 알려진 효소 분해 방법11,14를 기반으로 합니다. 이는 모든 마우스 모델에서 성숙한 지방 세포를 정제하기 위한 간단하고 보편적인 접근 방식을 제공합니다. 위에서 설명한 바와 같이, 이 프로토콜은 전체 전사체 및 ChIP-seq 분석을 위한 다운스트림 사용에 적합합니다. 동일한 개별 지방 패드에서 여러 후성유전체 프로파일(예: 여러 히스톤 변형 및 RNA-seq)을 생성하기에 충분한 수율을 제공합니다.
이러한 일치하는 후성유전체 데이터를 준비할 수 있는 높은 수율을 보장하기 위해 중요한 단계는 헵탄을 사용하여 온전한 지방세포와 공정 중에 용해되는 지방세포에서 지질을 용해하는 것입니다. 우리의 경험에 비추어 볼 때, 이 단계는 RNA와 핵이 지질층으로 손실되는 것을 방지함으로써 재현성과 수율을 크게 증가시킵니다. 고정을 받는 지방세포를 포함하는 관의 지속적인 회전은 지방세포에서 지질이 제거되는 균질성을 향상시키는 만큼 똑같이 중요합니다. ChIP-seq 문헌21,22의 대부분에 보고된 1% 포름알데히드 고착 완충액 대신, 과도한 고정을 피하기 위해 농도를 0.7% 포름알데히드로 줄이는 것이 필요하다는 것이 밝혀졌습니다. ChIP-seq에 대한 최적의 염색질 크기 범위(100-800bp)를 달성하기 위해 염색질 전단 시간도 12분으로 최적화되었습니다.
이 프로토콜은 벌크 전사체 및 후성유전체 연구에 최적화되었습니다. 현재 프로토콜은 여전히 단일 세포 전사체 및 후성유전체 응용 분야에 대한 자체 제한이 있습니다. 비뚱장23,24에서 보고된 세포 이질성을 고려할 때, 이 분리 방법은 단일 세포 분석에 적용하기 위해 추가로 개발될 수 있습니다. 이러한 맥락에서 붕소-디피로메텐(BODIPY)25 및 페리리핀-1(PLIN1)26, ASC-127과 같은 지방세포 제한 마커 또는 지방세포 특이적 핵 마커는 추가적이고 기능적이며 관련된 세포 매개변수의 정량화를 가능하게 하는 데 유용할 수 있습니다28. 게놈 연구에 대한 응용 외에도 이 프로토콜은 지방 세포의 높은 지질 함량으로 인해 추가 장애물이 있는 지방 세포 단백질체 연구를 개선할 수도 있습니다29.
이 프로토콜은 또한 인간 지방 세포 핵(데이터는 표시되지 않음)을 성공적으로 추출하는 데 사용할 수 있으며, 인간 비만에 대한 연구로 응용 프로그램을 확장할 수 있습니다.
공개
저자는 공개할 내용이 없습니다.
감사의 말
우리는 MPI-IE 광학 이미징, 염기서열 분석, 생물 정보학 핵심 및 인력에 빚을 지고 있습니다. 이 작업은 MPG, Van Andel Research Institute, European Union의 Horizon 2020 연구, NIH(R01HG012444 및 R21HG011964), Marie Skłodowska-Curie 보조금 계약 번호 675610 및 프로젝트 번호 01KU1216(Deutsches Epigenom Programm, DEEP)에 따라 연방 교육 연구부의 자금 지원을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| 16% Formaldehyde Solution (w/v). Methanol-free | ThermoFisher SCIENTIFIC | 28908 | |
| 1-bromo-3-chloropropane | SIGMA | B9673 | |
| 5 M NaCl | invitrogen | AM9759 | |
| Automated electrophoresis instrument (2100 Bioanalyzer Instrument) | Agilent Technologies | G2939BA | |
| BODIPY | ThermoFisher SCIENTIFIC | D3922 | |
| Collagenase Type 2 | Worthington Biochemical Corp. | 43D14184B | |
| Complete, EDTA-free, Protease inhibitor cocktail (PIC) | Roche | 4693159001 | EDTA free |
| DAPI | ThermoFisher SCIENTIFIC | D1306 | |
| DMEM (+ GlutaMAX) | life technologies | 61965-026 | |
| DNA purification kit (PCR purification kit) | Qiagen | 28104 | |
| DNA quantification instrument (Qubit 4 Fluorometer) | Fisher SCIENTIFIC | Q33238 | |
| DNA quantification kit (DNA high sensitivity assay) | Agilent Technologies | 5067-4626 | |
| EDTA | Fisher chimical | E478-500 | |
| EGTA | Fisher chimical | O2783-100 | |
| EtOH | PHARMCO | 111000200 | |
| Fatty acid-free BSA | SERVA | 11932.01 | bovine albumin fraction V, fatty acid-free lyophilized |
| Glycine | SIGMA | G7126-100G | |
| Heptane | SIGMA | H2198-1L | |
| High Sensitivity RNA Analysis | Agilent Technologies | 5067-1513 | |
| Igepal | SIGMA | 18896-50ML | |
| KCl | SIGMA | P3911-25G | |
| Matrix filter (420um) | Tisch Scientific | ME17238 | |
| Next-Generation Sequencer (HiSeq 3000 Sequencer) | Illumina | ||
| Nuclease-free water | Invitrogen | 4387936 | |
| PIPES | ACROS organics | 5625-37-6 | |
| Proteinase K | ThermoFisher SCIENTIFIC | EO0491 | |
| RNA isolation reagent (Trizol) | ThermoFisher SCIENTIFIC | 15596026 | |
| RNase A, DNase-free | ThermoFisher SCIENTIFIC | EN0531 | |
| Rotator (Thermo Scientific Tube Revolver Rotator) | ThermoFisher SCIENTIFIC | 88881001 | |
| SDS, Sodium dodecyl sulfate | SIGMA | 8170341000 | |
| Sonication tube (1 mL milliTUBE with AFA Fiber) | Covaris | 520135 | |
| Sonicator (Evolution Focused-ultra-sonicator (S220 or E220)) | Covaris | 500217 or 500239 | |
| Total RNA Prep kit ( Illumina Stranded Total RNA Prep kit) | Illumina | 20040525 | |
| Tris-HCl | Fisher bioreagents | BP153-500 |
참고문헌
- Albuquerque, D., Stice, E., Rodríguez-López, R., Manco, L., Nóbrega, C. Current review of genetics of human obesity: From molecular mechanisms to an evolutionary perspective. Molecular Genetics and Genomics. 290 (4), 1191-1221 (2015).
- Weihrauch-Blüher, S., Schwarz, P., Klusmann, J. H. Childhood obesity: Increased risk for cardiometabolic disease and cancer in adulthood. Metabolism. 92, 147-152 (2019).
- Weihrauch-Blüher, S., Wiegand, S. Risk factors and implications of childhood obesity. Current Obesity Reports. 7 (4), 254-259 (2018).
- Gluckman, P. D. Epigenetics, the life-course and metabolic disease. Nature Reviews Endocrinology. 8 (2), 74-76 (2012).
- Loh, M., Zhou, L., Ng, H. K., Chambers, J. C. Epigenetic disturbances in obesity and diabetes: Epidemiological and functional insights. Molecular Metabolism. 27, 33-41 (2019).
- Sayols-Baixeras, S., et al. DNA methylation and obesity traits: An epigenome-wide association study. The REGICOR study. Epigenetics. 12 (10), 909-916 (2017).
- Xia, Q., Grant, S. F. A. The genetics of human obesity. Annals of the New York Academy of Sciences. 1281 (1), 178-190 (2013).
- Yokoi, N. Epigenetic dysregulation in pancreatic islets and pathogenesis of type 2 diabetes. Journal of Diabetes Investigation. 9 (3), 475-477 (2018).
- Frayn, K. N. Obesity and metabolic disease: Is adipose tissue the culprit. Proceedings of the Nutrition Society. 64 (1), 7-13 (2005).
- Kawai, T., Autieri, M. V., Scalia, R. Adipose tissue inflammation and metabolic dysfunction in obesity. American Journal of Physiology-Cell Physiology. 320 (3), 375-391 (2021).
- Cat, A. N. D., Briones, A. M. Hypertension: Methods and Protocols. Touyz, R. M., Schiffrin, E. L. , Springer. New York. 283-295 (2017).
- Ambati, S., et al. Adipocyte nuclei captured from VAT and SAT. BMC Obesity. 3, 35(2016).
- Roh, H. C., et al. Simultaneous transcriptional and epigenomic profiling from specific cell types within heterogeneous tissues in vivo. Cell Reports. 18 (4), 1048-1061 (2017).
- Jacobsen, M. J., et al. Epigenetic and transcriptomic characterization of pure adipocyte fractions from obese pigs identifies candidate pathways controlling metabolism. Frontiers in Genetics. 10, 1268(2019).
- Janke, J., Engeli, S., Gorzelniak, K., Sharma, A. M. Extraction of total RNA from adipocytes. Hormone and Metabolic Research. 33 (4), 213-215 (2001).
- Cirera, S. Highly efficient method for isolation of total RNA from adipose tissue. BMC Research Notes. 6 (1), 472(2013).
- Rajbhandari, P., et al. Single cell analysis reveals immune cell-adipocyte crosstalk regulating the transcription of thermogenic adipocytes. eLife. 8, 49501(2019).
- Buddrick, O., Jones, O. A. H., Morrison, P. D., Small, D. M. Heptane as a less toxic option than hexane for the separation of vitamin E from food products using normal phase HPLC. RSC Advances. 3 (46), 24063-24068 (2013).
- Arrigoni, L., et al. Standardizing chromatin research: A simple and universal method for ChIP-seq. Nucleic Acids Research. 44 (7), 67(2016).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Schmidt, D., et al. ChIP-seq: Using high-throughput sequencing to discover protein-DNA interactions. Methods. 48 (3), 240-248 (2009).
- Hoffman, E. A., Frey, B. L., Smith, L. M., Auble, D. T. Formaldehyde crosslinking: A tool for the study of chromatin complexes. Journal of Biological Chemistry. 290 (44), 26404-26411 (2015).
- Rondini, E. A., Granneman, J. G. Single cell approaches to address adipose tissue stromal cell heterogeneity. Biochemical Journal. 477 (3), 583-600 (2020).
- Corvera, S. Cellular heterogeneity in adipose tissues. Annu Review of Physiology. 83, 257-278 (2021).
- Katz, L. S., Geras-Raaka, E., Gershengorn, M. C. Heritability of fat accumulation in white adipocytes. American Journal of Physiology-Endocrinology and Metabolism. 307 (3), 335-344 (2014).
- Hansen, J. S., de Maré, S., Jones, H. A., Göransson, O., Lindkvist-Petersson, K. Visualization of lipid directed dynamics of perilipin 1 in human primary adipocytes. Scientific Reports. 7 (1), 15011(2017).
- Ussar, S., et al. ASC-1, PAT2, and P2RX5 are cell surface markers for white, beige, and brown adipocytes. Science Translational Medicine. 6 (247), (2014).
- Onogi, Y., Khalil, A. E. M. M., Ussar, S. Identification and characterization of adipose surface epitopes. Biochemical Journal. 477 (13), 2509-2541 (2020).
- Kim, E. Y., et al. Recent advances in proteomic studies of adipose tissues and adipocytes. International Journal of Molecular Sciences. 16 (3), 4581-4599 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유