Method Article
トランスクリプトームおよびエピゲノム解析のためのマウス白色脂肪細胞の単離とプロセシング
要約
本プロトコールは、ダウンストリームの全RNAシーケンシング、SONicationによるNuclei Extraction (NEXSON)、およびChIP-seqに最適化されたマウス白色脂肪細胞の単離、精製、およびアップストリームプロセッシングのための普遍的な方法をまとめたものです。
要約
肥満は、遺伝学、エピジェネティクス、環境、およびそれらの相互作用の影響を受ける複雑な病気です。成熟脂肪細胞は、白色脂肪組織の主要な細胞型です。脂肪細胞がどのように機能し、(エピ)遺伝的および環境的シグナルに反応するかを理解することは、肥満の原因を特定するために不可欠です。RNAとクロマチンは、これまで酵素消化法を用いて脂肪細胞から単離されてきました。さらに、核単離のためのプロトコルが開発されており、脂肪細胞特異的トランスジェニックレポーターの蛍光活性化細胞選別(FACS)によって精製が達成されます。このようなプロトコールで高収率と高品質を達成するための最大の課題の1つは、脂肪組織に含まれる大量の脂質です。本プロトコールは、ヘプタンを活用して目的の標的(RNA/クロマチン)から脂質を分離する成熟脂肪細胞を単離するための最適化された手順を説明しています。得られたRNAは高い完全性を持ち、高品質なRNA-seq結果が得られます。同様に、この手順により核の収量が向上し、サンプル間で再現性のあるChIP-seq結果が得られます。したがって、現在の研究は、全ゲノムトランスクリプトームおよびエピゲノム研究に適した信頼性の高い普遍的なマウス脂肪細胞単離プロトコルを提供します。
概要
肥満は、通常、2型糖尿病、心血管代謝疾患、およびいくつかの形態の癌1,2,3のリスクを高める一因となる過剰な脂肪蓄積の病気として理解されています。肥満の現在の理解は遺伝学(ヒトとげっ歯類の両方の研究から)に大きく根ざしていますが、代謝性疾患の素因の約30%〜70%は非遺伝的起源であり、4,5,6,7,8は不明のままです。
脂肪組織は、肥満やその他の代謝性疾患において重要な役割を果たしています9,10。脂肪組織は、成熟脂肪細胞と、前駆脂肪細胞、内皮細胞、免疫細胞などの間質血管画分で構成されています。各細胞型がどのように肥満に寄与し、脂肪細胞の調節不全がどのように肥満に寄与するかは、まだ不明です。成熟脂肪細胞エピゲノム研究のための再現性のある効果的な単離および精製プロトコルは、この分野にとって興味深いものです。
成熟脂肪細胞は、遺伝子発現解析11およびエピゲノム研究12,13のために長い間単離されてきました。脂肪細胞を単離するための2つの主要な戦略があります。1つ目は、酵素消化を使用して、間質血管画分11,14の成熟脂肪細胞を残りの細胞型から分離することです。第2は、脂肪組織を腫瘤化して無傷の核を放出し、次いで蛍光活性化細胞選別(FACS)12,13による蛍光レポーターに基づいて核を回復させることであり、これには特殊なトランスジェニックレポーターモデルが必要である。いずれの場合も技術的な課題は、成熟脂肪細胞が高濃度の脂質を含んでおり(図1)、それが全RNA15,16および核17の品質および/または収量を低下させることです。ここでは、成熟脂肪細胞を単離するための最適化された酵素消化手順が記載されており、ヘプタンの利点は、RNA抽出またはSONication(NEXSON)19による核抽出19による核分離ステップに先立って、脂質18を迅速かつ効率的に溶解および除去することである。このプロトコールは、ゲノムワイド研究のためのトータルRNAの優れた回収率と品質を保証し、再現性のあるChIP-seqのためのインタクト核の収量を大幅に改善します。
プロトコル
すべての動物実験は、施設動物管理および使用委員会(IACUC、プロトコル番号:18-10-028)によって承認されました。12週齢の雄C57BL/6JマウスをCO2 で安楽死させ、精巣上体白脂肪組織(eWAT)から脂肪パッドを採取するために解剖した。
1.脂肪細胞の分離
- 消化バッファー(表1)を調製し、ウォーターバスで37°Cに温めます。
- 精巣上体白色脂肪組織(eWAT)の脂肪パッドをペトリ皿に入れます。5mLのDMEMに鉗子とメスを使用して、組織を小片(5 mm x 5 mm)に切断します。
注:細胞接着コーティングを含まないペトリ皿が好ましい。また、細胞を傷つけないように、脂肪パッドを優しく持つことも重要です。脂肪組織を取り扱うほど、歩留まりが損なわれます。メス/鉗子法は、脂肪細胞に損傷を与える可能性があるため、ハサミや鉗子だけで脂肪パッドを切断するよりも好ましい方法です。 - 組織の破片を乱さずに余分なDMEMを排出します。
注意: ペトリ皿の表面から組織の破片を拾わないでください。 - 5 mLの消化バッファーをシャーレに加え、渦巻き状に回転させてディッシュから組織片を放出し、内容物全体を50 mLの遠心チューブに注ぎます。すべての組織片が遠心分離チューブに移されるまで、このプロセスを繰り返します。
- 残りの消化バッファーを遠心チューブに加え、最終容量が~20 mLになるようにします。
- 組織片を37°Cの振とう水槽で100rpmで20〜30分間、または組織片が1 mm x 1 mmより小さくなるまでインキュベートします。
- 0.4 mLの0.5 M EDTAと0.2 mLの0.5 M EGTAを遠心チューブに加え、37°Cで10分間振とう続けます。
- 10 mLピペットで細胞懸濁液をファインマトリックスフィルター(420 μm、 材料表を参照)に通し、別の50 mL遠心チューブの上に移し、未消化組織を除去します。
- 50 mLの遠心チューブを200 x g で室温で5分間遠心分離し、純粋な脂肪細胞(上部に浮かんでいるもの)を間質血管画分(下部のペレット)から分離します(図2A)。
- 浮遊脂肪細胞層を新しい50mLチューブに注ぎ、ペレットを乱さないように注意します。
注:浮遊脂肪細胞はピペットチップ壁に付着する可能性があるため(したがって、歩留まりが低下する)、ピペットチップを使用して浮遊脂肪細胞を移すことは避けてください。一部の消化バッファーは新しいチューブに引き継がれます。シリンジと針を使用して、脂肪細胞層の下から残留消化バッファーを除去します。 - 精製した脂肪細胞を直ちに全RNA抽出(ステップ2)および/またはChIP-seq(ステップ3およびステップ4)に使用してください。
注:ユーザーは、ステップ2で全脂肪細胞抽出物を使用することを決定できます。またはステップ3.、研究目的に応じて。また、脂肪細胞抽出物全体を2つの部分に分割し、ステップ2を続行することもできます。そしてステップ3。並行して、生物学的に同一のサンプルから分析結果を取得します。
2. 全RNA抽出
注意: この手順は、化学薬品フードで実行します。
- RNA単離試薬(1 mL)( 材料表を参照)を50 mL遠心チューブ内の精製脂肪細胞に加えます(ステップ1.11)。
- 1 mLのピペットチップを使用してピペットを上下に動かし、脂肪細胞をRNA単離試薬に完全に溶解し、2 mLの微量遠心チューブに移します。
- チューブを室温で15分間インキュベートします。
- 0.5 mLのヘプタンを遠心分離管に加え、30秒間全速でボルテックスします。
注:脂質はヘプタンに溶解します(図2B)。 - チューブを1,000 x g で4°Cで10分間遠心分離します。
- シリンジと30 G針でRNA単離試薬の最下層を取得し、新しい1.5 mLチューブに移します。
- 0.1 mLの1ブロモ-3クロロプロパン( 材料表を参照)をRNA単離試薬抽出物に加え、30秒間全速でボルテックスします。チューブを室温で15分間インキュベートします。
- チューブを12,000 x g で4°Cで15分間遠心分離します。 ピペットを使用して上部水相を採取し、新しいチューブに移します。
- 0.5 mLのイソプロパノールを加え、4°Cで10分間インキュベートします。 チューブを12,000 x g で4°Cで10分間遠心分離します。
注:RNAはチューブの底にペレットとして沈殿します。 - ペレットに触れないように注意しながら、ピペットを使用して上清を取り除きます。
- 1mLの75%EtOHを加え、全速力で短時間ボルテックスします。
- チューブを7,500 x g で4°Cで5分間遠心分離します。 ピペットチップで上清を慎重に取り除きます。ペレットを邪魔しないでください。
- ペレットを室温で10分間、またはペレットが透明/透明になるまで風乾します。
- ペレットを50μLのヌクレアーゼフリー水に溶解します。
3. ChIP-seqのための脂肪細胞固定
メモ: 手順 3 を実行します。ケミカルフードで。
- 0.3 mLの0.5 M EDTA、0.2 mLの0.5 M EGTA、14.8 mLのDMEM、および10 mLのヘプタンを、単離された脂肪細胞を含む50 mLチューブに加えます(ステップ1.11)。
- 0.7 mLの16%ホルムアルデヒド(最終0.7%)を加え、チューブローテーター( 材料表を参照)で室温で10 rpmで5分間回転させます。
注意: タイマーを設定し、5分間カウントします。複数のサンプルを処理する場合は、ホルムアルデヒドを30秒間隔で順次添加し、すべてのサンプルで正確かつ比較可能な固定タイミングを可能にします。 - 1.78 mLの1.25 Mグリシン(最終0.125 M)を加え、ローテーターで10 rpmで室温で5分以内に混合します。
注:ホルムアルデヒドはグリシンを添加した後も活性を維持します。したがって、5分間のインキュベーションステップの直後に遠心分離(ステップ3.4.)により層を分離することが重要です。サンプルが複数ある場合は、各サンプルにグリシンを添加して、30秒の時間間隔で反応を順次停止させます。 - 次に、200 x g で室温で5分間遠心分離します。
注意: 3つの明白で離散的な液体層が必要です(図2C)。真ん中の白い層には、固定された脂肪細胞が含まれています。 - 1 mLピペットチップを使用して、脂肪細胞(白色)層を新しい15 mL遠心チューブに移し、新しいチューブに運ばれる固定剤とヘプタンの量を最小限に抑えます。
- チューブに10 mL(室温)DMEMを充填し、プロテアーゼ阻害剤カクテル(PIC、 材料表を参照)錠剤を補充します。反転させてよく混ぜます。
- チューブを≤200 x g で室温で5分間遠心分離します。遠心分離力を100 x g に保ち、細胞の破裂を最小限に抑えます。
- 手順3.5.-3.7を繰り返します。ステップ3.9に進む前に1倍。
- 1 mLピペットチップを使用して、脂肪細胞(白)層を1.5 mLまたは2 mLの微量遠心チューブに移します。
- チューブを100〜200 x g で室温で5分間遠心分離し、1 mLのピペットチップを使用してヘプタン(最上層)とDMEM(最下層)を除去します。
- 固定脂肪細胞は、使用まで-80°Cで最大6ヶ月間保存してください。
4. 核抽出とクロマチン剪断
注:この手順は、Arrigoni et al.19から採用されています。
- ソニケーターの電源を入れ( 材料の表を参照)、ピーク電力を75 W、デューティ係数を2%、200サイクル/バーストに設定し、ウォーターバスチラーを20°Cに設定します。
- プロテアーゼ阻害剤カクテル(PIC)をFarnhamラボバッファーおよびシャーリングバッファーに添加します(表1)。
注:Arrigoni et al.19でさらに推奨される超音波発生器とパラメータをいくつか確認してください。 - 固定脂肪細胞(ステップ3から)を750〜800μLの氷冷Farnhamラボバッファーで1mLの超音波処理チューブに再懸濁します( 材料の表を参照)。
注意: チューブを全容量まで充填しないことをお勧めします。さもなければ、脂肪細胞はチューブの最上部に浮遊し、超音波処理効率を低下させる。 - サンプルを2.5分間超音波処理します。
注:位相差顕微鏡で核抽出を確認し、さらなる超音波処理が必要かどうかを判断してください。原子核は、 図2Dに示すように、丸く無傷でなければなりません。 - 先に進む前に、ソニケーターのピーク電力を140W、デューティファクターを5%、ウォーターバスチラーを4°Cに設定して、200サイクル/バーストに設定します。
- ピペットを使用して、上清を1.5 mLの微量遠心チューブに移します。チューブを1,000 x g で4°Cで5分間遠心分離します。
- ペレット(核)から上清を慎重に取り除き、ペレットを保管します。
- ペレットを1 mLのFarnhamラボバッファーで氷上で洗浄します。
- チューブを1,000 x g で4°Cで5分間遠心分離し、ピペットを使用して上清を除去し、ペレットを保持します。
- 手順4.8.-4.9を繰り返します。1x を実行してから、ステップ 4.11 に進みます。
注:単離および洗浄された核は、さらに使用するまで-80°Cで3か月間保存できます。 - 単離された核を1mLのせん断緩衝液に再懸濁し、それらを新しい1mLの超音波処理チューブに移します。チューブ内に気泡がないことを確認してください。
- ソニケーターの核を、ピーク電力140 W、デューティファクター5%、および200サイクル/バーストで12分間超音波処理し、クロマチンを4°Cでせん断します。
- ライセートを1.5 mLのマイクロチューブに移します。
注:せん断されたクロマチンは核から放出され、バッファー(ライセート)に含まれています。 - チューブを10,000 x g で4°Cで5分間遠心分離し、不溶性の破片をペレット化します。上清(クロマチン)を新しい1.5 mL微量遠心チューブに移します。
- 剪断したクロマチンは、4°Cで10日間、または-80°Cで3ヶ月間保存することができます。
注:追加の品質チェック:以前に発表されたレポート19に従って、剪断されたクロマチンの25μLをアリコートして、DNaseフリーのRNaseでRNAを脱架橋および除去します。本研究では、得られたDNAを製造元の指示に従ってDNA精製キットで精製し、DNA定量装置上のDNA定量キットでDNAを定量し、フラグメントサイズを自動電気泳動装置で評価しました( 材料の表を参照)。せん断クロマチンサイズは100〜800 bpの範囲である必要があります。残りのシアークロマチンは、下流のChIP-Seqアプリケーションに進むことができます。
結果
脂肪細胞を6つの脂肪パッドから単離し、ヘプタンベースのRNA抽出を行い(ステップ2)、得られたRNAを自動電気泳動装置で分析しました。すべてのサンプルについて計算されたRNAインテグリティナンバー(RIN)は>8(図3A)であり、高品質で再現性の高いRNA調製物であることを示しています。次に、Total RNA Prep Kitを使用してRNAライブラリを調製し、各サンプルを次世代シーケンサーでシーケンシングして、≥4,000万リードのリード深度に到達しました。すべてのサンプル(図3B)および配列ごと(図3C)のPhredスコアは≥30であり、高品質のDNA配列20を示している。このように、ヘプタン除去は、RNAシーケンシングに適した高収量で高品質なRNAの単離をサポートします。
ホルムアルデヒド固定ステップでは、ヘプタン含有核分離調製物3種とヘプタンを含まないコントロール調製物1種も行いました。次に、せん断されたクロマチンを自動電気泳動装置で分析しました。いずれの場合も、クロマチンは100-800 bpのサイズ範囲まで剪断され(図4A)、下流のChIP-seq手順に理想的でした19。重要なことに、ヘプタン処理したサンプル(11.5 ng/μL、9.42 ng/μL、9.6 ng/μL)は、未処理のサンプル(2.2 ng/μL)と比較して、~5倍多くのクロマチンが得られました。H3K4me3およびH3K27me3 ChIP-seqは、Arrigoni et al.19に従って実施しました。 図4Bに示すように、3つの独立したヘプタン処理サンプルからのシグナルトラックは、両方のヒストンマークについて、ヘプタン未処理サンプルのトラックと同等でした。ヘプタン処理はChIP-seqの品質を妨げませんが、核(およびせん断クロマチン)の収量を大幅に改善します。
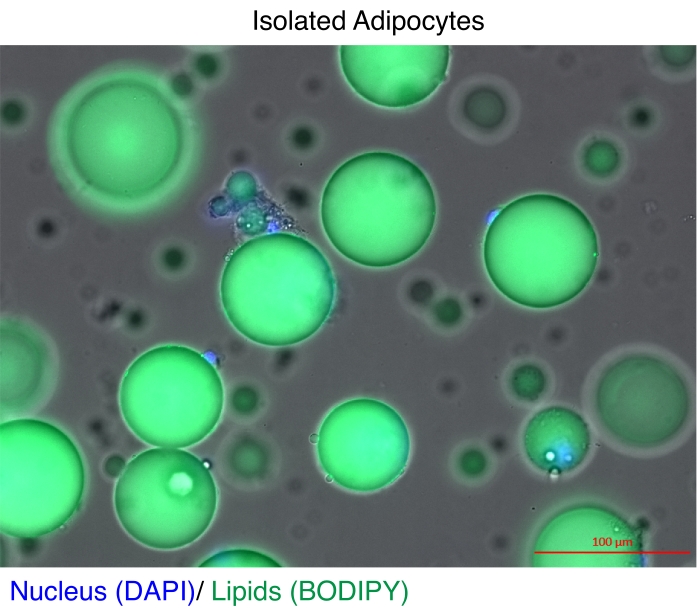
図1:単離された脂肪細胞。 単離した脂肪細胞をDAPI(青)で染色して核を染色し、BODIPY(緑)で脂質を染色しました。細胞質脂質液滴は、脂肪細胞の体積の最大95%を占めており、RNAおよびクロマチン抽出中に高い収量を達成するための技術的な課題を提起しています。スケールバー = 100 μm. この図の拡大版を表示するには、ここをクリックしてください。
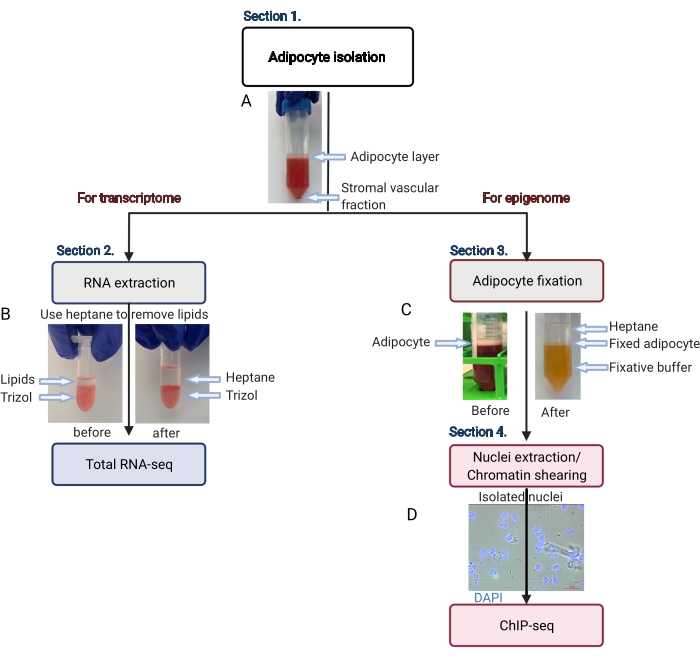
図2:トランスクリプトームおよびエピゲノム解析のための脂肪細胞単離の概略フロー。 脂肪細胞の単離からトランスクリプトームまたはエピゲノムの適用まで、ワークフロー全体が描かれています。主要な手順と代表的な結果を示します。(A)単離された脂肪細胞は最上層に浮かび、チューブの底でペレットとして間質血管画分から分離します。(B)RNA単離試薬によるRNA抽出前の脂質除去にヘプタンを使用する。(C)脂肪細胞固定中に脂質を除去するためのヘプタンの使用。(D)単離された脂肪細胞核の代表的な画像は、無傷で丸いものでなければなりません。スケールバー = 20 μm。このスキームは BioRender.com で作成されました。 この図の拡大版を表示するには、ここをクリックしてください。
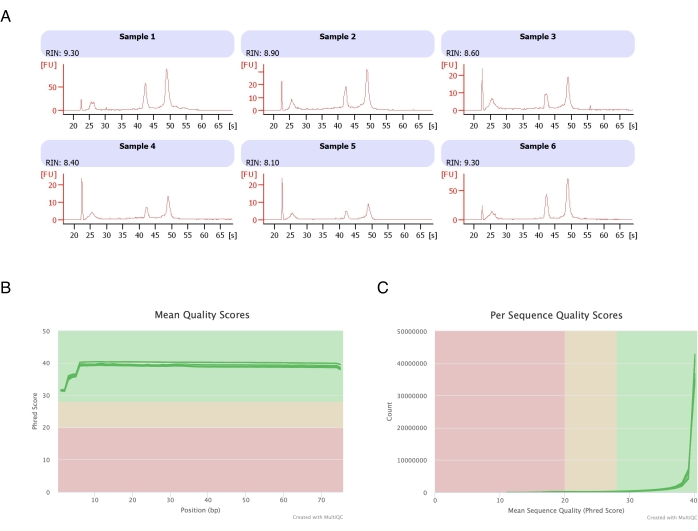
図3:RNAシーケンス後のRNA完全性と品質スコアの代表的なエレクトロフェログラム (A) サンプル1-6はヘプタン処理脂肪細胞の6回の複製を表し、それらのRNA完全性分析は自動電気泳動装置で実行されました。RNAインテグリティナンバー(RIN)は、18Sおよび28SリボソームRNA比に基づいており、RNAの品質を表しています。1 (大幅に低下している) から 10 (最も劣化が少ない) までスケーリングします。(B,C)シーケンシングリードの品質スコアは、塩基上の平均品質スコア(C)およびシーケンスごとの品質スコアについて、multiQC解析(B)によって決定されました。この図の拡大版を表示するには、ここをクリックしてください。
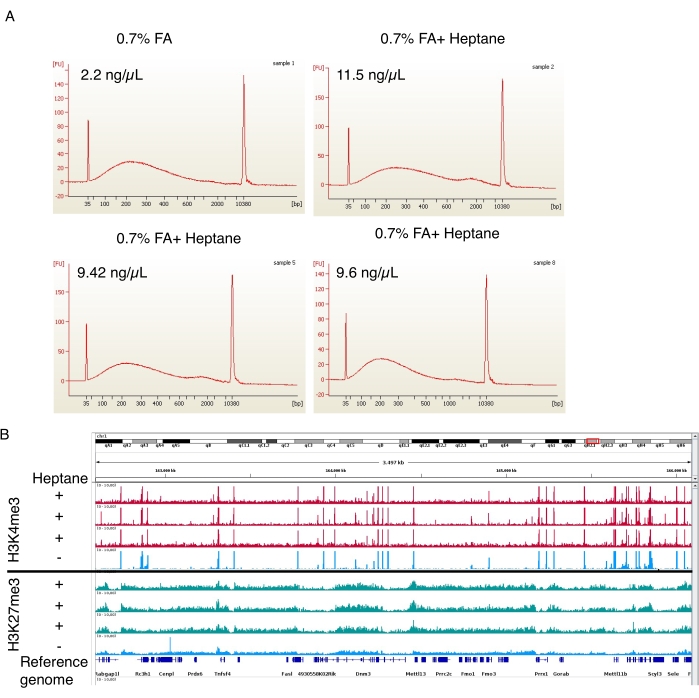
図4:ChIP-seq.のせん断されたクロマチンと濃縮ピークの代表的なエレクトロフェログラム (A) コントロール(サンプル1、左上)とヘプタン処理した脂肪細胞クロマチン調製物(サンプル2、5、8、右上と下2)のクロマチンサイズ分布を示す自動電気泳動装置の代表的なプロファイル。最終調製物中のクロマチン濃度は、各画像の左上に示されています。(B)Integrative Genomics Viewer(IGV)を使用して作成したゲノムブラウザのスクリーンショット。プロットの上部は、ヘプタン処理された3つのサンプル(赤)と1つのコントロール(青)のH3K4me3 ChIP-seqプロファイルを示しています。下のパネルは、H3K27me3のChIP-seqについても同じことを示しています。 この図の拡大版を表示するには、ここをクリックしてください。
| バッファ | 組成 |
| 消化緩衝液(脂肪パッド3000mg以下/ 20mL用) | 20 mL の Dulbecco's Modified Eagle Medium (DMEM) |
| 0.3gの脂肪酸を含まないBSA | |
| コラゲナーゼ2型0.1 g | |
| ファーナムラボバッファー | 5 mMパイプ(pH 8) |
| 85 mM KCl | |
| 0.5%のIGEPAL | |
| せん断バッファー | 10 mM Tris-HCl pH 8 |
| 0.1%のSDS | |
| 1 mM EDTAの |
表1:本研究で使用したさまざまなバッファーの組成。
ディスカッション
ここで提示する脂肪細胞単離プロトコルは、白色脂肪組織の残りの間質血管画分から成熟脂肪細胞(浮遊)を分離するための、広く受け入れられている酵素消化法11,14に基づいている。これは、あらゆるマウスモデルで成熟脂肪細胞を精製するための簡単で普遍的なアプローチを提供します。上述の通り、このプロトコールは、全トランスクリプトームおよびChIP-seq解析のためのダウンストリームでの使用に適しています。これは、同じ個々の脂肪パッドから複数のエピゲノムプロファイル(例えば、いくつかのヒストン修飾とRNA-seq)を生成するのに十分な収量を提供します。
このような一致したエピゲノムデータの調製を可能にする高収率を確保するためには、ヘプタンを使用して、無傷の脂肪細胞やプロセス中に溶解する脂肪細胞から脂質を溶解することが重要です。私たちの経験では、このステップにより、RNAと核が脂質層に失われるのを防ぐことにより、再現性と収量が大幅に向上します。固定中の脂肪細胞を含むチューブの連続回転は、脂肪細胞から脂質が除去される均質性を高めるため、同様に重要です。ChIP-seqの文献の多くで報告されている1%ホルムアルデヒド固定緩衝液の代わりに21,22、過剰固定を避けるためには、濃度を0.7%ホルムアルデヒドに下げる必要があることがわかりました。また、クロマチンの剪断時間も12分に最適化し、ChIP-seqの最適なクロマチンサイズ範囲(100-800 bp)を実現しました。
このプロトコールは、バルクトランスクリプトームおよびエピゲノム研究に最適化されています。現在のプロトコルには、シングルセルトランスクリプトームおよびエピゲノムアプリケーションに対してまだ独自の制限があります。脂肪分野23,24で報告された細胞の不均一性を考慮すると、この単離法は、シングルセルアッセイに適応するためにさらに開発することができる。このような状況では、ホウ素-ジピロメテン(BODIPY)25やペリリピン-1(PLIN1)26、ASC-127、または脂肪細胞特異的核マーカーなどの脂肪細胞制限マーカーは、追加の機能的で関連性のある細胞パラメータの定量を可能にする上で価値がある可能性がある28。ゲノム研究への応用とは別に、このプロトコルは、脂肪細胞の脂質含有量が高いために追加のハードルがある脂肪細胞プロテオミクス研究も改善する可能性があります29。
このプロトコルは、ヒト脂肪細胞の核(データは示されていません)の抽出にも使用でき、その適用をヒト肥満の研究に拡張します。
開示事項
著者は何も開示していません。
謝辞
MPI-IEの光学イメージング、シーケンシング、バイオインフォマティクスコア、そして人員の方々にお世話になっています。この研究は、MPG、Van Andel Research Institute、欧州連合のHorizon 2020研究、NIH(R01HG012444およびR21HG011964)、Marie Skłodowska-Curie助成金契約番号675610、およびプロジェクト番号01KU1216(Deutsches Epigenom Programm、DEEP)に基づく連邦教育研究省からの資金提供によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| 16% Formaldehyde Solution (w/v). Methanol-free | ThermoFisher SCIENTIFIC | 28908 | |
| 1-bromo-3-chloropropane | SIGMA | B9673 | |
| 5 M NaCl | invitrogen | AM9759 | |
| Automated electrophoresis instrument (2100 Bioanalyzer Instrument) | Agilent Technologies | G2939BA | |
| BODIPY | ThermoFisher SCIENTIFIC | D3922 | |
| Collagenase Type 2 | Worthington Biochemical Corp. | 43D14184B | |
| Complete, EDTA-free, Protease inhibitor cocktail (PIC) | Roche | 4693159001 | EDTA free |
| DAPI | ThermoFisher SCIENTIFIC | D1306 | |
| DMEM (+ GlutaMAX) | life technologies | 61965-026 | |
| DNA purification kit (PCR purification kit) | Qiagen | 28104 | |
| DNA quantification instrument (Qubit 4 Fluorometer) | Fisher SCIENTIFIC | Q33238 | |
| DNA quantification kit (DNA high sensitivity assay) | Agilent Technologies | 5067-4626 | |
| EDTA | Fisher chimical | E478-500 | |
| EGTA | Fisher chimical | O2783-100 | |
| EtOH | PHARMCO | 111000200 | |
| Fatty acid-free BSA | SERVA | 11932.01 | bovine albumin fraction V, fatty acid-free lyophilized |
| Glycine | SIGMA | G7126-100G | |
| Heptane | SIGMA | H2198-1L | |
| High Sensitivity RNA Analysis | Agilent Technologies | 5067-1513 | |
| Igepal | SIGMA | 18896-50ML | |
| KCl | SIGMA | P3911-25G | |
| Matrix filter (420um) | Tisch Scientific | ME17238 | |
| Next-Generation Sequencer (HiSeq 3000 Sequencer) | Illumina | ||
| Nuclease-free water | Invitrogen | 4387936 | |
| PIPES | ACROS organics | 5625-37-6 | |
| Proteinase K | ThermoFisher SCIENTIFIC | EO0491 | |
| RNA isolation reagent (Trizol) | ThermoFisher SCIENTIFIC | 15596026 | |
| RNase A, DNase-free | ThermoFisher SCIENTIFIC | EN0531 | |
| Rotator (Thermo Scientific Tube Revolver Rotator) | ThermoFisher SCIENTIFIC | 88881001 | |
| SDS, Sodium dodecyl sulfate | SIGMA | 8170341000 | |
| Sonication tube (1 mL milliTUBE with AFA Fiber) | Covaris | 520135 | |
| Sonicator (Evolution Focused-ultra-sonicator (S220 or E220)) | Covaris | 500217 or 500239 | |
| Total RNA Prep kit ( Illumina Stranded Total RNA Prep kit) | Illumina | 20040525 | |
| Tris-HCl | Fisher bioreagents | BP153-500 |
参考文献
- Albuquerque, D., Stice, E., Rodríguez-López, R., Manco, L., Nóbrega, C. Current review of genetics of human obesity: From molecular mechanisms to an evolutionary perspective. Molecular Genetics and Genomics. 290 (4), 1191-1221 (2015).
- Weihrauch-Blüher, S., Schwarz, P., Klusmann, J. H. Childhood obesity: Increased risk for cardiometabolic disease and cancer in adulthood. Metabolism. 92, 147-152 (2019).
- Weihrauch-Blüher, S., Wiegand, S. Risk factors and implications of childhood obesity. Current Obesity Reports. 7 (4), 254-259 (2018).
- Gluckman, P. D. Epigenetics, the life-course and metabolic disease. Nature Reviews Endocrinology. 8 (2), 74-76 (2012).
- Loh, M., Zhou, L., Ng, H. K., Chambers, J. C. Epigenetic disturbances in obesity and diabetes: Epidemiological and functional insights. Molecular Metabolism. 27, 33-41 (2019).
- Sayols-Baixeras, S., et al. DNA methylation and obesity traits: An epigenome-wide association study. The REGICOR study. Epigenetics. 12 (10), 909-916 (2017).
- Xia, Q., Grant, S. F. A. The genetics of human obesity. Annals of the New York Academy of Sciences. 1281 (1), 178-190 (2013).
- Yokoi, N. Epigenetic dysregulation in pancreatic islets and pathogenesis of type 2 diabetes. Journal of Diabetes Investigation. 9 (3), 475-477 (2018).
- Frayn, K. N. Obesity and metabolic disease: Is adipose tissue the culprit. Proceedings of the Nutrition Society. 64 (1), 7-13 (2005).
- Kawai, T., Autieri, M. V., Scalia, R. Adipose tissue inflammation and metabolic dysfunction in obesity. American Journal of Physiology-Cell Physiology. 320 (3), 375-391 (2021).
- Cat, A. N. D., Briones, A. M., Touyz, R. M., Schiffrin, E. L. . Hypertension: Methods and Protocols. , 283-295 (2017).
- Ambati, S., et al. Adipocyte nuclei captured from VAT and SAT. BMC Obesity. 3, 35 (2016).
- Roh, H. C., et al. Simultaneous transcriptional and epigenomic profiling from specific cell types within heterogeneous tissues in vivo. Cell Reports. 18 (4), 1048-1061 (2017).
- Jacobsen, M. J., et al. Epigenetic and transcriptomic characterization of pure adipocyte fractions from obese pigs identifies candidate pathways controlling metabolism. Frontiers in Genetics. 10, 1268 (2019).
- Janke, J., Engeli, S., Gorzelniak, K., Sharma, A. M. Extraction of total RNA from adipocytes. Hormone and Metabolic Research. 33 (4), 213-215 (2001).
- Cirera, S. Highly efficient method for isolation of total RNA from adipose tissue. BMC Research Notes. 6 (1), 472 (2013).
- Rajbhandari, P., et al. Single cell analysis reveals immune cell-adipocyte crosstalk regulating the transcription of thermogenic adipocytes. eLife. 8, 49501 (2019).
- Buddrick, O., Jones, O. A. H., Morrison, P. D., Small, D. M. Heptane as a less toxic option than hexane for the separation of vitamin E from food products using normal phase HPLC. RSC Advances. 3 (46), 24063-24068 (2013).
- Arrigoni, L., et al. Standardizing chromatin research: A simple and universal method for ChIP-seq. Nucleic Acids Research. 44 (7), 67 (2016).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Schmidt, D., et al. ChIP-seq: Using high-throughput sequencing to discover protein-DNA interactions. Methods. 48 (3), 240-248 (2009).
- Hoffman, E. A., Frey, B. L., Smith, L. M., Auble, D. T. Formaldehyde crosslinking: A tool for the study of chromatin complexes. Journal of Biological Chemistry. 290 (44), 26404-26411 (2015).
- Rondini, E. A., Granneman, J. G. Single cell approaches to address adipose tissue stromal cell heterogeneity. Biochemical Journal. 477 (3), 583-600 (2020).
- Corvera, S. Cellular heterogeneity in adipose tissues. Annu Review of Physiology. 83, 257-278 (2021).
- Katz, L. S., Geras-Raaka, E., Gershengorn, M. C. Heritability of fat accumulation in white adipocytes. American Journal of Physiology-Endocrinology and Metabolism. 307 (3), 335-344 (2014).
- Hansen, J. S., de Maré, S., Jones, H. A., Göransson, O., Lindkvist-Petersson, K. Visualization of lipid directed dynamics of perilipin 1 in human primary adipocytes. Scientific Reports. 7 (1), 15011 (2017).
- Ussar, S., et al. ASC-1, PAT2, and P2RX5 are cell surface markers for white, beige, and brown adipocytes. Science Translational Medicine. 6 (247), (2014).
- Onogi, Y., Khalil, A. E. M. M., Ussar, S. Identification and characterization of adipose surface epitopes. Biochemical Journal. 477 (13), 2509-2541 (2020).
- Kim, E. Y., et al. Recent advances in proteomic studies of adipose tissues and adipocytes. International Journal of Molecular Sciences. 16 (3), 4581-4599 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved