Method Article
Aislamiento y procesamiento de adipocitos blancos murinos para análisis de transcriptoma y epigenoma
En este artículo
Resumen
El presente protocolo resume un método universal para aislar, purificar y procesar aguas arriba de adipocitos blancos murinos optimizado para la secuenciación posterior de ARN total, extracción de núcleos por sonicación (NEXSON) y ChIP-seq.
Resumen
La obesidad es una enfermedad compleja influenciada por la genética, la epigenética, el medio ambiente y sus interacciones. Los adipocitos maduros representan el principal tipo de célula en el tejido adiposo blanco. Comprender cómo funcionan los adipocitos y cómo responden a las señales (epi)genéticas y ambientales es esencial para identificar las causas de la obesidad. El ARN y la cromatina se han aislado previamente de los adipocitos mediante digestión enzimática. Además, se han desarrollado protocolos para el aislamiento nuclear, en los que la purificación se logra mediante la clasificación de células activadas por fluorescencia (FACS) de reporteros transgénicos específicos de adipocitos. Uno de los mayores desafíos para lograr un alto rendimiento y calidad durante dichos protocolos es la cantidad sustancial de lípidos contenidos en el tejido adiposo. El presente protocolo describe un procedimiento optimizado para aislar adipocitos maduros que aprovecha el heptano para separar los lípidos de los objetivos de interés (ARN/cromatina). El ARN resultante tiene una alta integridad y genera resultados de secuenciación de ARN de alta calidad. Del mismo modo, el procedimiento mejora la tasa de rendimiento de los núcleos y genera resultados de ChIP-seq reproducibles en todas las muestras. Por lo tanto, el estudio actual proporciona un protocolo de aislamiento de adipocitos murinos fiable y universal adecuado para estudios de transcriptoma y epigenoma del genoma completo.
Introducción
La obesidad se entiende típicamente como una enfermedad de acumulación excesiva de grasa que contribuye a un mayor riesgo de diabetes tipo 2, enfermedad cardiometabólica y varias formas de cáncer 1,2,3. Si bien la comprensión actual de la obesidad está fuertemente arraigada en la genética (tanto en estudios en humanos como en roedores), entre el 30% y el 70% de la predisposición a las enfermedades metabólicas es de origen no genético 4,5,6,7,8 y sigue estando mal definida.
El tejido adiposo desempeña un papel fundamental en la obesidad y otras enfermedades metabólicas 9,10. El tejido adiposo comprende adipocitos maduros y la fracción vascular estromal, incluidos los preadipocitos, las células endoteliales y las células inmunitarias. Todavía no está claro cómo cada tipo de célula contribuye a la obesidad y cómo la desregulación de los adipocitos contribuye a la obesidad. Los protocolos de aislamiento y purificación reproducibles y eficaces para estudios de epigenomas de adipocitos maduros son de interés para este campo.
Los adipocitos maduros han sido aislados durante mucho tiempo para análisis de expresión génica11 y estudios de epigenoma12,13. Existen dos estrategias principales para aislar los adipocitos. La primera es utilizar la digestión enzimática para separar los adipocitos maduros del resto de los tipos celulares de la fracción vascular estromal11,14. La segunda consiste en remover el tejido adiposo para liberar núcleos intactos y luego recuperar los núcleos a partir de un localizador fluorescente mediante clasificación celular activada por fluorescencia (FACS)12,13, que requiere modelos reporteros transgénicos especializados. El reto técnico en cada caso es que los adipocitos maduros contienen altas concentraciones de lípidos (Figura 1), lo que reduce la calidad y/o el rendimiento del ARNtotal 15,16 y de los núcleos17. Aquí, se describe un procedimiento de digestión enzimática optimizado para aislar adipocitos maduros, en el que la ventaja del heptano es disolver y eliminar rápida y eficientemente los lípidos18 antes de la extracción de ARN o los pasos de aislamiento de núcleos por Extracción de Núcleos por SONicación (NEXSON)19. El protocolo garantiza una excelente recuperación y calidad del ARN total para estudios de todo el genoma y mejora significativamente el rendimiento de los núcleos intactos para la secuenciación reproducible de ChIP.
Protocolo
Todos los experimentos con animales fueron aprobados por el Comité Institucional de Cuidado y Uso de Animales (IACUC), número de protocolo: 18-10-028. Los ratones machos C57BL/6J de 12 semanas de edad fueron sacrificados con CO2 y diseccionados para recolectar las almohadillas de grasa del tejido adiposo blanco del epidídimo (eWAT).
1. Aislamiento de adipocitos
- Preparar el tampón de digestión (Tabla 1) y calentar a 37 °C en un baño de agua.
- Coloque la almohadilla de grasa del tejido adiposo blanco del epidídimo (eWAT) en una placa de Petri. Cortar el tejido en trozos pequeños (5 mm x 5 mm) con pinzas y un bisturí en 5 mL de DMEM.
NOTA: Se prefiere una placa de Petri que no contenga un recubrimiento de adhesión celular. También es importante sujetar la almohadilla de grasa suavemente para evitar dañar las células. Cuanto más se manipula el tejido adiposo, más se compromete el rendimiento. Es preferible un método de bisturí o pinza que cortar las almohadillas de grasa con tijeras o fórceps solos, ya que pueden dañar los adipocitos. - Drene el exceso de DMEM sin alterar los restos de tejido.
NOTA: No recoja los restos de tejido de la superficie de la placa de Petri. - Agregue 5 mL de tampón de digestión a la placa de Petri, agite para liberar los fragmentos de tejido de la placa y vierta todo el contenido en un tubo de centrífuga de 50 mL. Repita el proceso hasta que todos los fragmentos de tejido se transfieran al tubo de centrífuga.
- Agregue el tampón de digestión restante al tubo de centrífuga para que el volumen final sea de ~ 20 mL.
- Incubar los fragmentos de tejido en un baño de agua agitado a 37 °C a 100 rpm durante 20-30 min, o hasta que los fragmentos de tejido tengan un tamaño inferior a 1 mm x 1 mm.
- Añada 0,4 mL de EDTA 0,5 M y 0,2 mL de EGTA 0,5 M al tubo de centrífuga y continúe agitando a 37 °C durante 10 min.
- Transfiera la suspensión celular con una pipeta de 10 mL a través de un filtro de matriz fina (420 μm, consulte la tabla de materiales) encima de otro tubo de centrífuga de 50 mL para eliminar el tejido no digerido.
- Centrifugar el tubo de centrífuga de 50 mL a 200 x g durante 5 min a temperatura ambiente para separar los adipocitos puros (flotando en la parte superior) de la fracción vascular estromal (pellet en la parte inferior) (Figura 2A).
- Vierta la capa de adipocitos flotantes en un nuevo tubo de 50 ml, teniendo cuidado de no romper el pellet.
NOTA: Evite el uso de puntas de pipeta para transferir adipocitos flotantes porque pueden adherirse a la pared de la punta de la pipeta (lo que reduce el rendimiento). Parte del tampón de digestión se trasladará al nuevo tubo; Use una jeringa y una aguja para eliminar el tampón de digestión residual de debajo de la capa de adipocitos. - Utilice los adipocitos purificados para la extracción de ARN total (paso 2) y/o ChIP-seq (paso 3 y paso 4) inmediatamente.
NOTA: El usuario puede decidir utilizar todo el extracto de adipocitos para el paso 2. o el paso 3., dependiendo del propósito de la investigación. El usuario también puede dividir todo el extracto de adipocitos en dos porciones y continuar con el paso 2. y paso 3. en paralelo para obtener los resultados del análisis de las muestras biológicamente idénticas.
2. Extracción de ARN total
NOTA: Realice este paso en una campana química.
- Añada un reactivo de aislamiento de ARN (1 mL) (consulte la Tabla de materiales) a los adipocitos purificados en el tubo de centrífuga de 50 mL (paso 1.11.).
- Pipetear hacia arriba y hacia abajo con una punta de pipeta de 1 mL para disolver completamente los adipocitos en el reactivo de aislamiento de ARN y transferirlo a un tubo de microcentrífuga de 2 mL.
- Incubar el tubo durante 15 minutos a temperatura ambiente.
- Añada 0,5 mL de heptano en el tubo de centrífuga y en el vórtice a máxima velocidad durante 30 s.
NOTA: Los lípidos se disolverán en el heptano (Figura 2B). - Centrifugar el tubo a 1.000 x g durante 10 min a 4 °C.
- Adquiera la capa inferior del reactivo de aislamiento de ARN con una jeringa y una aguja de 30 G, y transfiérala a un nuevo tubo de 1,5 ml.
- Añada 0,1 mL de 1bromo-3cloropropano (ver Tabla de Materiales) al reactivo de aislamiento de ARN, extracto y vórtice a toda velocidad durante 30 s. Incuba el tubo a temperatura ambiente durante 15 min.
- Centrifugar el tubo a 12.000 x g durante 15 min a 4 °C. Utilice una pipeta para recoger la fase acuosa superior y transferirla a un nuevo tubo.
- Añadir 0,5 mL de isopropanol e incubar durante 10 min a 4 °C. Centrifugar el tubo a 12.000 x g durante 10 min a 4 °C.
NOTA: El ARN se precipitará como un gránulo en la parte inferior del tubo. - Utilice una pipeta para eliminar el sobrenadante, teniendo cuidado de evitar tocar el pellet.
- Añadir 1 mL de 75% de EtOH y vórtice a toda velocidad brevemente.
- Centrifugar el tubo a 7.500 x g durante 5 min a 4 °C. Retire con cuidado el sobrenadante con la punta de una pipeta. No moleste el pellet.
- Seque el pellet al aire a temperatura ambiente durante 10 minutos o hasta que el pellet se vuelva claro/transparente.
- Disuelva el pellet en 50 μL de agua libre de nucleasas.
3. Fijación de adipocitos para ChIP-seq
NOTA: Realice el paso 3. en una campana química.
- Añadir 0,3 mL de EDTA 0,5 M, 0,2 mL de EGTA 0,5 M, 14,8 mL de DMEM y 10 mL de heptano en el tubo de 50 mL que contiene adipocitos aislados (paso 1.11.).
- Agregue 0.7 mL de formaldehído al 16% (0.7% final) y gire sobre un rotador de tubo (ver Tabla de Materiales) a 10 rpm durante 5 min a temperatura ambiente.
NOTA: Configure un temporizador y cuente durante 5 minutos. Si procesa varias muestras, agregue formaldehído secuencialmente a intervalos de 30 s para permitir un tiempo preciso y comparable de fijación en todas las muestras. - Añadir 1,78 mL de glicina 1,25 M (0,125 M finales) y mezclar en el rotador a 10 rpm durante no más de 5 min a temperatura ambiente.
NOTA: El formaldehído permanece activo incluso después de agregar glicina. Por lo tanto, es importante separar las capas por centrifugación (paso 3.4.) inmediatamente después de la etapa de incubación de 5 minutos. Si hay más de una muestra, agregue glicina a cada muestra para detener la reacción secuencialmente en intervalos de tiempo de 30 s. - A continuación, centrifugar a 200 x g durante 5 min a temperatura ambiente.
NOTA: Debe haber tres capas líquidas obvias y discretas (Figura 2C). La capa blanca central contiene los adipocitos fijos. - Utilice una punta de pipeta de 1 mL para transferir la capa de adipocitos (blanca) a un tubo de centrífuga nuevo de 15 mL, minimizando la cantidad de fijador y heptano transportado al nuevo tubo.
- Llene el tubo con 10 mL (temperatura ambiente) de DMEM suplementado con el cóctel de inhibidores de la proteasa (PIC, ver Tabla de Materiales). Mezclar bien por inversión.
- Centrifugar el tubo a ≤200 x g durante 5 min a temperatura ambiente. Mantenga la fuerza de centrifugación en 100 x g para minimizar el estallido de la célula.
- Repita los pasos 3.5.-3.7. 1x antes de avanzar al paso 3.9.
- Utilice una punta de pipeta de 1 mL para transferir la capa de adipocitos (blancos) a un tubo de microcentrífuga de 1,5 mL o 2 mL.
- Centrifugar el tubo a 100-200 x g durante 5 min a temperatura ambiente y utilizar una punta de pipeta de 1 ml para eliminar el heptano (capa superior) y el DMEM (capa inferior).
- Almacene los adipocitos fijos a -80 °C durante un máximo de 6 meses hasta su uso.
4. Extracción de núcleos y cizallamiento de la cromatina
NOTA: Este procedimiento es una adaptación de Arrigoni et al.19.
- Encienda el sonicador (consulte la tabla de materiales) y ajuste la potencia máxima a 75 W, el factor de trabajo al 2% y 200 ciclos/ráfaga, con el enfriador del baño de agua ajustado a 20 °C.
- Agregue un cóctel de inhibidores de la proteasa (PIC) al tampón de laboratorio de Farnham y al tampón de cizallamiento (Tabla 1).
NOTA: Por favor, echa un vistazo a algunos sonicadores y parámetros más sugeridos en Arrigoni et al.19. - Vuelva a suspender los adipocitos fijos (desde el paso 3) con 750-800 μL de tampón de laboratorio Farnham helado en un tubo de sonicación de 1 mL (ver Tabla de Materiales).
NOTA: Se recomienda no llenar el tubo hasta su capacidad máxima. De lo contrario, los adipocitos flotarán hasta la parte superior del tubo, lo que reduce la eficacia de la sonicación. - Sonicar la muestra durante 2,5 min.
NOTA: Compruebe la extracción de los núcleos en un microscopio de contraste de fase para determinar si es necesaria una sonicación adicional. Los núcleos deben ser redondos e intactos, como se muestra en la Figura 2D. - Antes de continuar, ajuste la potencia máxima del sonicador a 140 W, el factor de trabajo al 5% y 200 ciclos/ráfaga, con el enfriador en baño de agua a 4 °C.
- Utilice una pipeta para transferir el sobrenadante a un tubo de microcentrífuga de 1,5 mL. Centrifugar el tubo a 1.000 x g durante 5 min a 4 °C.
- Retire con cuidado el sobrenadante del pellet (núcleos) y guarde el pellet.
- Lave el pellet con 1 mL de tampón de laboratorio Farnham con hielo.
- Centrifugar el tubo a 1.000 x g durante 5 min a 4 °C, utilizar una pipeta para eliminar el sobrenadante y conservar el pellet.
- Repita los pasos 4.8.-4.9. 1x antes de avanzar al paso 4.11.
NOTA: Los núcleos aislados y lavados pueden almacenarse a -80 °C durante 3 meses hasta su posterior uso. - Vuelva a suspender los núcleos aislados en 1 mL de tampón de cizallamiento y transfiéralos a un nuevo tubo de sonicación de 1 mL. Asegúrese de que no haya burbujas en el tubo.
- Sonicar los núcleos en el sonicador con una potencia máxima de 140 W, un factor de trabajo del 5% y 200 ciclos/ráfaga durante 12 minutos para cortar la cromatina a 4 °C.
- Transfiera el lisado a un tubo de microfuga de 1,5 mL.
NOTA: La cromatina cortada se libera de los núcleos y está contenida en el tampón (lisado). - Centrifugar el tubo a 10.000 x g durante 5 min a 4 °C para pellet los residuos insolubles. Transfiera el sobrenadante (cromatina) a un nuevo tubo de microcentrífuga de 1,5 mL.
- La cromatina cortada puede almacenarse a 4 °C durante 10 días o a -80 °C durante 3 meses hasta su uso posterior.
NOTA: Control de calidad adicional: Alícuota 25 μL de la cromatina cortada para desentrecruzarse y eliminar el ARN con una RNasa libre de DNasa siguiendo el informe publicado anteriormente19. Para el presente estudio, el ADN resultante se purificó con un kit de purificación de ADN según las instrucciones del fabricante, el ADN se cuantificó con un kit de cuantificación de ADN en el instrumento de cuantificación de ADN y el tamaño del fragmento se evaluó con el instrumento de electroforesis automatizado (ver Tabla de Materiales). El tamaño de la cromatina cortada debe estar en el rango de 100-800 pb. El resto de la cromatina de cizallamiento puede proceder a la aplicación posterior de ChIP-Seq.
Resultados
Los adipocitos se aislaron de seis almohadillas de grasa, se realizó la extracción de ARN a base de heptano (paso 2) y el ARN resultante se analizó en el instrumento de electroforesis automatizado. El número de integridad de ARN (RIN) calculado para todas las muestras fue de >8 (Figura 3A), lo que indica una preparación de ARN reproducible y de alta calidad. A continuación, el kit de preparación de ARN total se utilizó para preparar bibliotecas de ARN, y cada muestra se secuenció en el secuenciador de próxima generación para alcanzar una profundidad de lectura de ≥40 millones de lecturas. La puntuación Phred para todas las muestras (Figura 3B) y por secuencia (Figura 3C) fue de ≥30, indicativo de secuencias de ADN de alta calidad20. Por lo tanto, la eliminación de heptano apoya el aislamiento de ARN de alto rendimiento y alta calidad adecuado para la secuenciación de ARN.
En la etapa de fijación de formaldehído, también se realizaron tres preparaciones de aislamiento nuclear que contenían heptano y una preparación de control sin heptano. A continuación, la cromatina cortada se analizó en el instrumento de electroforesis automatizado. En ambos casos, la cromatina se cortó a un rango de tamaño de 100-800 pb (Figura 4A), ideal para los procedimientos posteriores de ChIP-seq19. Es importante destacar que se obtuvo ~5 veces más cromatina de las muestras tratadas con heptano (11,5 ng/μL, 9,42 ng/μL y 9,6 ng/μL) en relación con las muestras no tratadas (2,2 ng/μL). Se realizaron H3K4me3 y H3K27me3 ChIP-seq siguiendo a Arrigoni et al.19. Como se muestra en la Figura 4B, las trazas de señal de tres muestras independientes tratadas con heptano fueron comparables a la pista de la muestra sin tratar con heptano para ambas marcas de histonas. El tratamiento con heptano no interfiere con la calidad de ChIP-seq, pero mejora significativamente el rendimiento de los núcleos (y de la cromatina cortada).
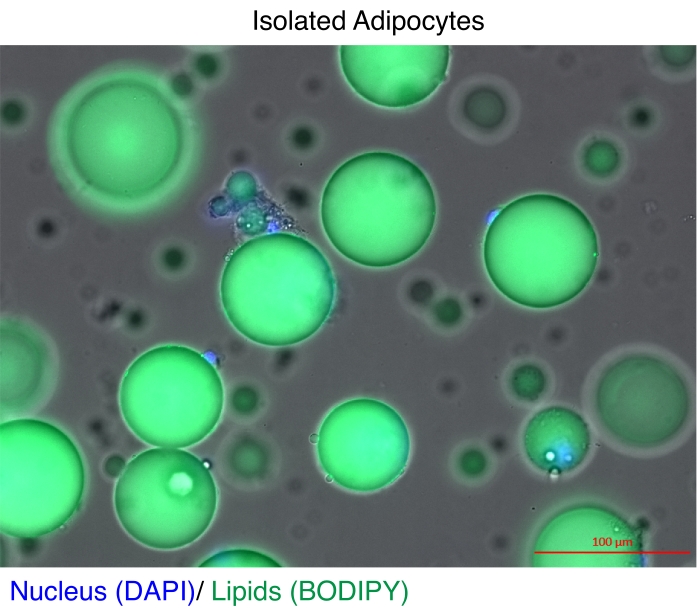
Figura 1: Adipocitos aislados. Los adipocitos aislados se tiñeron con DAPI (azul) para teñir el núcleo y BODIPY (verde) para los lípidos. Las gotas lipídicas citosólicas comprenden hasta el 95% del volumen de los adipocitos y plantean un desafío técnico para lograr altos rendimientos durante las extracciones de ARN y cromatina. Barra de escala = 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
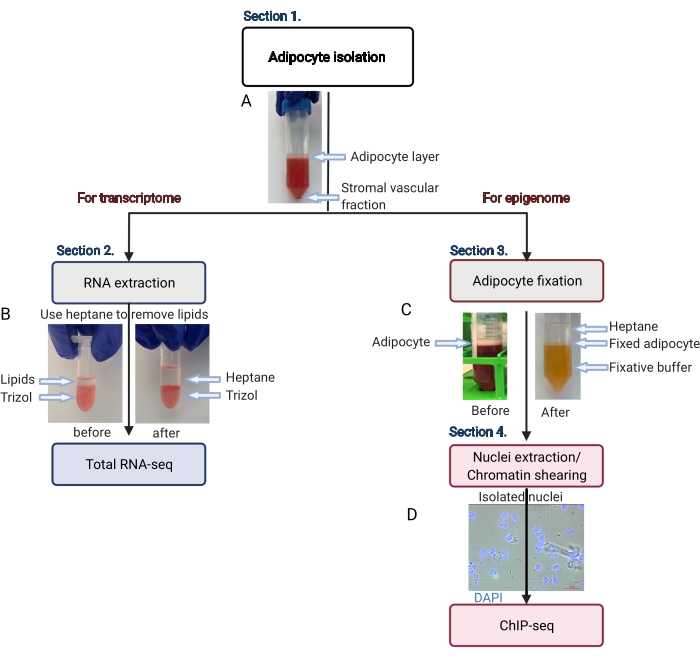
Figura 2: Flujo esquemático del aislamiento de adipocitos para el análisis de transcriptoma y epigenoma. Se representa todo el flujo de trabajo, desde el aislamiento de adipocitos hasta la aplicación del transcriptoma o el epigenoma. Se muestran los pasos clave y los resultados representativos. (A) Los adipocitos aislados flotan en la capa superior, separándose de la fracción vascular estromal como una bolita en la parte inferior del tubo. (B) El uso de heptano para eliminar lípidos antes de la extracción de ARN con reactivo de aislamiento de ARN. (C) El uso de heptano para eliminar los lípidos durante la fijación de los adipocitos. (D) La imagen representativa de los núcleos de los adipocitos aislados debe estar intacta y ser redonda. Barra de escala = 20 μm. El esquema fue creado con BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.
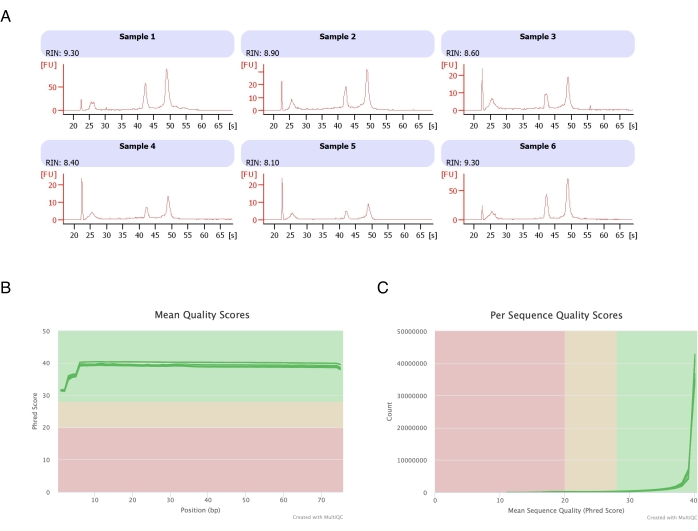
Figura 3: Electroferograma representativo de las puntuaciones de integridad y calidad del ARN después de la secuenciación de ARN. (A) Las muestras 1-6 representan seis réplicas de adipocitos tratados con heptano, y su análisis de integridad del ARN se ejecutó en el instrumento de electroforesis automatizado. El número de integridad del ARN (RIN) se basó en las proporciones de ARN ribosómico 18S y 28S y representa la calidad del ARN. Escale de 1 (muy degradado) a 10 (el menos degradado). (B,C) La puntuación de calidad de lectura de secuenciación se determinó mediante análisis multiQC (B) para la puntuación de calidad media en las bases (C) y para la puntuación de calidad por secuencia. Haga clic aquí para ver una versión más grande de esta figura.
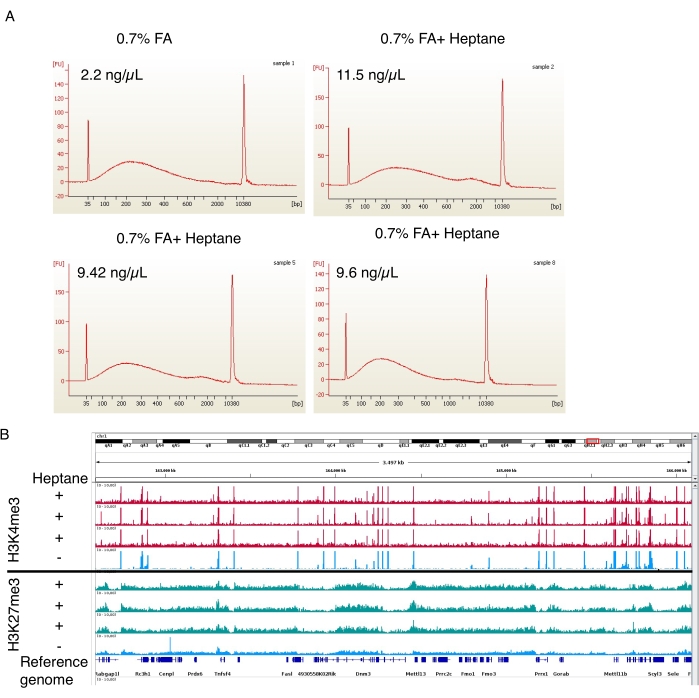
Figura 4: Electroferograma representativo de la cromatina cortada y los picos de enriquecimiento de ChIP-seq. (A) Perfiles representativos del instrumento de electroforesis automatizado que muestran las distribuciones de tamaño de cromatina del control (muestra 1, arriba a la izquierda) y las preparaciones de cromatina de adipocitos tratadas con heptano (muestras 2, 5 y 8, arriba a la derecha y dos abajo). Las concentraciones de cromatina en las preparaciones finales se indican en la parte superior izquierda de cada imagen. (B) Captura de pantalla del navegador del genoma realizada con el Visor de Genómica Integrativa (IGV). La parte superior del gráfico muestra los perfiles H3K4me3 ChIP-seq para tres muestras tratadas con heptano (rojo) y un control (azul). El panel inferior muestra lo mismo para el ChIP-seq de H3K27me3. Haga clic aquí para ver una versión más grande de esta figura.
| Búfer | Composición |
| Tampón de digestión (para 3000 mg o menos de almohadilla de grasa / 20 mL) | 20 mL de Medio Eagle Modificado de Dulbecco (DMEM) |
| 0,3 g de BSA sin ácidos grasos | |
| 0,1 g de colagenasa tipo 2 | |
| Tampón de laboratorio de Farnham | TUBOS DE 5 mM (pH 8) |
| 85 mM KCl | |
| 0.5% IGEPAL | |
| Tampón de cizallamiento | 10 mM Tris-HCl pH 8 |
| 0.1% SDS | |
| 1 mM EDTA |
Tabla 1: Composición de los diferentes tampones utilizados en el presente estudio.
Discusión
El protocolo de aislamiento de adipocitos que aquí se presenta se basa en métodos de digestión enzimática bien aceptados11,14 para separar los adipocitos maduros (flotantes) de la fracción vascular estromal restante del tejido adiposo blanco. Proporciona un enfoque sencillo y universal para purificar los adipocitos maduros en cualquier modelo de ratón. Como se ha demostrado anteriormente, el protocolo es adecuado para su uso posterior para análisis de transcriptoma completo y ChIP-seq. Proporciona suficiente rendimiento para generar múltiples perfiles epigenómicos (por ejemplo, varias modificaciones de histonas más RNA-seq) a partir de la misma almohadilla de grasa individual.
Para garantizar los altos rendimientos que permiten la preparación de estos datos de epigenoma coincidentes, un paso crítico es el uso de heptano para disolver los lípidos de los adipocitos intactos y de los adipocitos que se lisan durante el proceso. En nuestra experiencia, este paso aumenta sustancialmente la reproducibilidad y el rendimiento al evitar que el ARN y los núcleos se pierdan en la capa lipídica. La rotación continua de los tubos que contienen adipocitos en proceso de fijación es igualmente importante, ya que mejora la homogeneidad con la que se eliminan los lípidos de los adipocitos. En lugar del tampón fijador de formaldehído al 1% reportado en gran parte de la literatura ChIP-seq21,22, se encontró que es necesario reducir la concentración a 0,7% de formaldehído para evitar la sobrefijación. El tiempo de cizallamiento de la cromatina también se optimizó a 12 minutos para lograr un rango de tamaño de cromatina óptimo (100-800 pb) para ChIP-seq.
El protocolo se optimizó para estudios masivos de transcriptoma y epigenoma. El protocolo actual todavía tiene sus propias limitaciones para las aplicaciones de transcriptoma y epigenoma de una sola célula. Teniendo en cuenta la heterogeneidad celular reportada en el campo de adiposidad23,24, este método de aislamiento podría desarrollarse aún más para ser adaptado a ensayos de una sola célula. En tales contextos, los marcadores restringidos por adipocitos como el boro-dipirrometeno (BODIPY)25 y la perilipina-1 (PLIN1)26, ASC-127, o los marcadores de núcleos específicos de adipocitos podrían ser valiosos para permitir la cuantificación de parámetros celulares adicionales, funcionales y relevantes28. Además de la aplicación a los estudios genómicos, este protocolo también podría mejorar los estudios proteómicos de los adipocitos, que tienen un obstáculo adicional debido al alto contenido de lípidos en los adipocitos29.
Este protocolo también se puede utilizar para extraer con éxito núcleos de adipocitos humanos (datos no mostrados), extendiendo su aplicación a la investigación sobre la obesidad humana.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Estamos en deuda con el MPI-IE de imágenes ópticas, secuenciación, núcleo bioinformático y personal. Este trabajo contó con el apoyo financiero del MPG, el Instituto de Investigación Van Andel, la investigación Horizonte 2020 de la Unión Europea, los NIH (R01HG012444 y R21HG011964), el acuerdo de subvención Marie Skłodowska-Curie n.º 675610 y el Ministerio Federal de Educación e Investigación en el marco del proyecto número 01KU1216 (Deutsches Epigenom Programm, DEEP).
Materiales
| Name | Company | Catalog Number | Comments |
| 16% Formaldehyde Solution (w/v). Methanol-free | ThermoFisher SCIENTIFIC | 28908 | |
| 1-bromo-3-chloropropane | SIGMA | B9673 | |
| 5 M NaCl | invitrogen | AM9759 | |
| Automated electrophoresis instrument (2100 Bioanalyzer Instrument) | Agilent Technologies | G2939BA | |
| BODIPY | ThermoFisher SCIENTIFIC | D3922 | |
| Collagenase Type 2 | Worthington Biochemical Corp. | 43D14184B | |
| Complete, EDTA-free, Protease inhibitor cocktail (PIC) | Roche | 4693159001 | EDTA free |
| DAPI | ThermoFisher SCIENTIFIC | D1306 | |
| DMEM (+ GlutaMAX) | life technologies | 61965-026 | |
| DNA purification kit (PCR purification kit) | Qiagen | 28104 | |
| DNA quantification instrument (Qubit 4 Fluorometer) | Fisher SCIENTIFIC | Q33238 | |
| DNA quantification kit (DNA high sensitivity assay) | Agilent Technologies | 5067-4626 | |
| EDTA | Fisher chimical | E478-500 | |
| EGTA | Fisher chimical | O2783-100 | |
| EtOH | PHARMCO | 111000200 | |
| Fatty acid-free BSA | SERVA | 11932.01 | bovine albumin fraction V, fatty acid-free lyophilized |
| Glycine | SIGMA | G7126-100G | |
| Heptane | SIGMA | H2198-1L | |
| High Sensitivity RNA Analysis | Agilent Technologies | 5067-1513 | |
| Igepal | SIGMA | 18896-50ML | |
| KCl | SIGMA | P3911-25G | |
| Matrix filter (420um) | Tisch Scientific | ME17238 | |
| Next-Generation Sequencer (HiSeq 3000 Sequencer) | Illumina | ||
| Nuclease-free water | Invitrogen | 4387936 | |
| PIPES | ACROS organics | 5625-37-6 | |
| Proteinase K | ThermoFisher SCIENTIFIC | EO0491 | |
| RNA isolation reagent (Trizol) | ThermoFisher SCIENTIFIC | 15596026 | |
| RNase A, DNase-free | ThermoFisher SCIENTIFIC | EN0531 | |
| Rotator (Thermo Scientific Tube Revolver Rotator) | ThermoFisher SCIENTIFIC | 88881001 | |
| SDS, Sodium dodecyl sulfate | SIGMA | 8170341000 | |
| Sonication tube (1 mL milliTUBE with AFA Fiber) | Covaris | 520135 | |
| Sonicator (Evolution Focused-ultra-sonicator (S220 or E220)) | Covaris | 500217 or 500239 | |
| Total RNA Prep kit ( Illumina Stranded Total RNA Prep kit) | Illumina | 20040525 | |
| Tris-HCl | Fisher bioreagents | BP153-500 |
Referencias
- Albuquerque, D., Stice, E., Rodríguez-López, R., Manco, L., Nóbrega, C. Current review of genetics of human obesity: From molecular mechanisms to an evolutionary perspective. Molecular Genetics and Genomics. 290 (4), 1191-1221 (2015).
- Weihrauch-Blüher, S., Schwarz, P., Klusmann, J. H. Childhood obesity: Increased risk for cardiometabolic disease and cancer in adulthood. Metabolism. 92, 147-152 (2019).
- Weihrauch-Blüher, S., Wiegand, S. Risk factors and implications of childhood obesity. Current Obesity Reports. 7 (4), 254-259 (2018).
- Gluckman, P. D. Epigenetics, the life-course and metabolic disease. Nature Reviews Endocrinology. 8 (2), 74-76 (2012).
- Loh, M., Zhou, L., Ng, H. K., Chambers, J. C. Epigenetic disturbances in obesity and diabetes: Epidemiological and functional insights. Molecular Metabolism. 27, 33-41 (2019).
- Sayols-Baixeras, S., et al. DNA methylation and obesity traits: An epigenome-wide association study. The REGICOR study. Epigenetics. 12 (10), 909-916 (2017).
- Xia, Q., Grant, S. F. A. The genetics of human obesity. Annals of the New York Academy of Sciences. 1281 (1), 178-190 (2013).
- Yokoi, N. Epigenetic dysregulation in pancreatic islets and pathogenesis of type 2 diabetes. Journal of Diabetes Investigation. 9 (3), 475-477 (2018).
- Frayn, K. N. Obesity and metabolic disease: Is adipose tissue the culprit. Proceedings of the Nutrition Society. 64 (1), 7-13 (2005).
- Kawai, T., Autieri, M. V., Scalia, R. Adipose tissue inflammation and metabolic dysfunction in obesity. American Journal of Physiology-Cell Physiology. 320 (3), 375-391 (2021).
- Cat, A. N. D., Briones, A. M., Touyz, R. M., Schiffrin, E. L. . Hypertension: Methods and Protocols. , 283-295 (2017).
- Ambati, S., et al. Adipocyte nuclei captured from VAT and SAT. BMC Obesity. 3, 35 (2016).
- Roh, H. C., et al. Simultaneous transcriptional and epigenomic profiling from specific cell types within heterogeneous tissues in vivo. Cell Reports. 18 (4), 1048-1061 (2017).
- Jacobsen, M. J., et al. Epigenetic and transcriptomic characterization of pure adipocyte fractions from obese pigs identifies candidate pathways controlling metabolism. Frontiers in Genetics. 10, 1268 (2019).
- Janke, J., Engeli, S., Gorzelniak, K., Sharma, A. M. Extraction of total RNA from adipocytes. Hormone and Metabolic Research. 33 (4), 213-215 (2001).
- Cirera, S. Highly efficient method for isolation of total RNA from adipose tissue. BMC Research Notes. 6 (1), 472 (2013).
- Rajbhandari, P., et al. Single cell analysis reveals immune cell-adipocyte crosstalk regulating the transcription of thermogenic adipocytes. eLife. 8, 49501 (2019).
- Buddrick, O., Jones, O. A. H., Morrison, P. D., Small, D. M. Heptane as a less toxic option than hexane for the separation of vitamin E from food products using normal phase HPLC. RSC Advances. 3 (46), 24063-24068 (2013).
- Arrigoni, L., et al. Standardizing chromatin research: A simple and universal method for ChIP-seq. Nucleic Acids Research. 44 (7), 67 (2016).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Schmidt, D., et al. ChIP-seq: Using high-throughput sequencing to discover protein-DNA interactions. Methods. 48 (3), 240-248 (2009).
- Hoffman, E. A., Frey, B. L., Smith, L. M., Auble, D. T. Formaldehyde crosslinking: A tool for the study of chromatin complexes. Journal of Biological Chemistry. 290 (44), 26404-26411 (2015).
- Rondini, E. A., Granneman, J. G. Single cell approaches to address adipose tissue stromal cell heterogeneity. Biochemical Journal. 477 (3), 583-600 (2020).
- Corvera, S. Cellular heterogeneity in adipose tissues. Annu Review of Physiology. 83, 257-278 (2021).
- Katz, L. S., Geras-Raaka, E., Gershengorn, M. C. Heritability of fat accumulation in white adipocytes. American Journal of Physiology-Endocrinology and Metabolism. 307 (3), 335-344 (2014).
- Hansen, J. S., de Maré, S., Jones, H. A., Göransson, O., Lindkvist-Petersson, K. Visualization of lipid directed dynamics of perilipin 1 in human primary adipocytes. Scientific Reports. 7 (1), 15011 (2017).
- Ussar, S., et al. ASC-1, PAT2, and P2RX5 are cell surface markers for white, beige, and brown adipocytes. Science Translational Medicine. 6 (247), (2014).
- Onogi, Y., Khalil, A. E. M. M., Ussar, S. Identification and characterization of adipose surface epitopes. Biochemical Journal. 477 (13), 2509-2541 (2020).
- Kim, E. Y., et al. Recent advances in proteomic studies of adipose tissues and adipocytes. International Journal of Molecular Sciences. 16 (3), 4581-4599 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados