Method Article
解开聚糖-蛋白质相互作用:核磁共振 (NMR) 来拯救
摘要
在这里,我们提出了一个方案,详细介绍了一系列 NMR 实验的采集、处理和分析,旨在表征溶液中的蛋白质-聚糖相互作用。概述了最常见的基于配体和基于蛋白质的方法,这无疑有助于结构糖生物学和分子识别研究领域。
摘要
聚糖与蛋白质的相互作用调节许多与健康和疾病相关的事件。事实上,这些识别事件的建立及其生物学后果与双方的三维结构以及它们的动态特征和它们在相应细胞区室中的呈现密切相关。NMR 技术在解开这些特征方面是独一无二的,事实上,已经开发并应用了多种基于 NMR 的方法来监测游离寡糖与其相关受体的结合事件。该方案概述了获取、处理和分析 NMR 糖生物学领域采用的两种最强大的 NMR 方法的程序,即 1H-饱和转移差 (STD) 和 1 H,15N-异核单量子相干 (HSQC) 滴定实验,它们分别互补地提供了来自聚糖和蛋白质角度的信息。事实上,当它们结合在一起时,它们提供了一个强大的工具包,用于阐明分子识别过程的结构和动态方面。这种全面的方法增强了我们对聚糖-蛋白质相互作用的理解,并有助于推进化学糖生物学领域的研究。
引言
聚糖的分子识别对于与健康和疾病相关的许多过程至关重要。生物受体(凝集素、抗体、酶)对聚糖的特异性和选择性在很大程度上取决于调整焓(CH-π 和范德华、氢键、静电)和熵(疏水性、动力学、溶剂化-溶剂化)的不同成分之间的不稳定平衡1。
鉴于游离寡糖的巨大化学多样性和动态性质,NMR 方法被广泛用于剖析游离寡糖相互作用超过25 年2,因为这些方法以原子分辨率 3,4 提供了具有精确细节的分子识别事件的极好信息,即使采用其他方法无法检索到所需的相互作用证据也是如此。关键是,核磁共振是通用的,可以在不同时间尺度上研究原子水平上的动态事件,是迄今为止研究溶液中游离寡糖结构、构象和动力学的最佳技术。然而,解开这些信息可能是一个相当复杂的过程,需要采用定义明确的策略以及仔细的数据分析5。
核磁共振技术多种多样,事实上,有许多方法可以用来解开游离寡糖-蛋白质的相互作用6。我们在此描述了目前用于破译聚糖-受体相互作用的两种基本 NMR 方法 7,8,重点介绍了如何理清关键聚糖表位和蛋白质结合位点的呈递9。
在任何分子识别事件中,当受体与给定配体结合时,都会有一个化学交换过程,该过程会影响结合参与者的许多 NMR 参数10。因此,从 NMR 的角度来看,可以从聚糖配体或蛋白质受体11 的角度来监测相互作用。一般来说,蛋白质受体是一个大的生物分子(缓慢的旋转运动,速率在 ns 时间尺度上,因此,快速横向弛豫),而相互作用的聚糖可以被认为是一个小-中等大小的分子(快速旋转运动,速率在 ps 时间尺度上,横向弛豫缓慢)12。从标准角度来看,游离寡糖的 NMR 信号较窄,而受体的 NMR 信号较宽13。
基于配体的 NMR 方法依赖于许多游离 NMR 参数在从游离状态传递到结合态时所经历的巨大变化14。STD-NMR 是最常用的实验 NMR 技术,用于评估不同的聚糖结合特征15,从推断溶液状态下结合的存在到确定聚糖结合表位;即与蛋白质受体16 接触的配体原子。
或者,基于受体的 NMR 方法监测蛋白质受体在聚糖存在下信号相对于载脂蛋白状态记录的变化17。这些主要集中在筛选两种状态之间蛋白质信号的化学位移扰动。最常用的实验是 1 H-15N HSQC,或其 TROSY 替代品18。
这两种方法的结合允许将 NMR 应用于许多显示出广泛亲和力的不同系统。然而,对于基于受体的 NMR 方法,与基于配体的方法相比,必须有相对大量的可溶性、非聚集性、稳定的同位素标记 (15N) 蛋白质。
我们在这里描述了这两种方法,突出了它们的优点和缺点。请注意,协议中描述的基本步骤用作使用布鲁克光谱仪的示例。因此,命令和参数名称与 TopSpin(布鲁克的波谱仪控制软件)中使用的名称一致。
研究方案
1. 饱和转移差 NMR (STD-NMR)
注:后续几行概述了采集、处理和分析 STD-NMR 实验的基本程序。这些步骤用于举例说明该技术在检测配体结合和阐明配体结合表位方面的效用。要更深入地了解 NMR 实验的设计和采集,请参考 NMR 仪器随附的相应制造商手册。
- 收购
- 用蛋白质-配体复合物制备样品。聚糖:凝集素摩尔比在 10:1 和 100:1 之间,蛋白质浓度范围在 0.01 和 0.2 mM 之间。对于 h半乳糖凝集素-7 与 LacNAc 的相互作用,在 pH 7.4 的氘代磷酸盐缓冲盐水中使用 50:1 的蛋白质:配体比例。
注:蛋白质受体应该是纯的,并且可溶于所选缓冲液中(在 STD-NMR 实验的情况下,最好使用相应缓冲液的氘化版本,以减少可能的 1H NMR 信号干扰)。使用分光光度计预先检查蛋白质的浓度,以测量 280 nm 处的吸光度。 - 使用移液管从制备的溶液中,将总体积为 0.6 mL 的 NMR 管转移到 5 mm NMR 管中。
- 在所需温度下准备 NMR 仪器(常见实验温度在 10 °C 和 45 °C 之间)。使用 edte 命令打开温度控制监视器并设置所需的温度。对于 h半乳糖凝集素-7/LacNAc 研究,温度设置为 25 °C。
- 生成包含 zg 脉冲序列的新数据集。
- 对于简单的操作,请打开现有实验并键入 edc 命令。此时会出现一个对话框,定义实验的标题、特性(样品规格、溶剂)和一些参数。
- 如果需要更改原始脉冲序列,请在 ased(参数)和 AcquPars(采集参数)窗口中导航。此时,从波谱仪的库中选择所需的脉冲程序。
- 对于标准的 1H NMR 波谱,请从可用列表中选择 zg 脉冲序列。
注:在含水量增加的样品中,可能需要使用水抑制方案来提高信噪比。使用 zgesgp 等脉冲序列是可取的,它激发雕刻模块,提供出色的抑制效果,但控制剩余信号的相位。请参阅制造商的 NMR 教程,了解有关水抑制方案类型及其主要特性的更多信息。
- 通过激活样品提升空气,将 NMR 样品插入探头。使用 ej 命令,将样品放在磁力架顶部,然后使用 ij 命令停用样品提升。
注:要使用自动进样器将样品注入磁力架,请使用命令 sx ,后跟位置编号 n,对应于 NMR 管在自动进样器托盘中的位置。 - 通过键入命令 lock,然后从菜单中选择适当的溶剂来锁定溶剂信号。
- 将样品插入探头后,使用自动模块 atma 或手动模块 atmm 完成调谐和匹配过程。
- 通过 topshim gui 命令启动自动填充。这将打开一个图形界面,其中将选择并启动垫片维度 1D。
注意:为了最大限度地减少由于细微的磁场或温度变化而导致的匀场不稳定性,可以激活自动匀场进行实验采集。这可以通过访问 BSMS 控制窗口并单击 autoshim 来执行。变为绿色高亮显示表示 autoshim 已激活。请注意,使用自动填充物时,必须忽略潜在的样品不稳定问题。因此,建议在使用 autoshim 时谨慎。 - 确定 1小时 90° 脉冲。这可以通过 pulsecal 命令自动执行。
- 在 AcquPars 窗口中修改各种参数。对于常规的 1H NMR 波谱,将扫描次数 (NS) 设置为 32,将所需的波谱窗口 (SW) 设置为 ca。12 页/分钟。
注: zgesgp 脉冲序列包括一个用于溶剂抑制的模块,以消除残余 HDO 信号,该信号应位于光谱中间的中心。为此,必须在 AcquPars 中准确定义 O1。 - 设置接收器增益以避免使用自动命令 raga 溢出。
- 现在,使用 zg 命令获取标准的 1H NMR 波谱。
- 采集完成后,通过 efp 命令处理频谱。使用 TopSpin 菜单栏应用基线和相位校正。
注:观察到来自聚糖和蛋白质的 1H NMR 信号(图 1)。如第 1.1.14 节所述,建议对获得的 NMR 波谱进行详细分析以实施 STD NMR 实验。 - 创建一个新数据集并上传 STD NMR 脉冲序列,以与第 1.1.4 节中 1H NMR 实验相同的方式使用。在布鲁克仪器中,脉冲程序目录中提供了不同的脉冲序列,均命名为 stddiffXXX。最简单的一个 (stddiff) 不包括任何水抑制方案或蛋白质抑制过滤器。
- 对于具有显著 H2O 含量的样品,请选择 stddiffgp19 或 stddiffesgp 序列,其中包括水门或激发雕刻模块。对于以强蛋白质 NMR 信号为背景的谱图,请选择 stddiffXXX.3 序列。在每种情况下,优化每个水抑制模块的相应特定参数(即水门事件方案中的 d19)。
- 定义 STD NMR 实验的关断谐振频率和导通谐振频率。在 FQ2LIST 条目下的 ased 窗口的 AcquPars 参数中找到频率列表。以赫兹为单位定义的导on-resonance和off-resonance频率必须手动写入列表中,并以新名称保存。这个新列表将用于 STD-NMR 实验。
- 对于典型游离寡糖,选择无游离寡糖信号的光谱区域的导通谐振频率,通常约为 δ(1H) 0 或 6.6 ppm(图 1)。将非谐振频率设置在不显示任何配体或蛋白质质子的区域。它可以安全地设置为 +18000 或 -18000 Hz。
- 在 ased 窗口的 AcquPars 参数中定义要在饱和时间内使用的整形脉冲。
注意:有很多可能性。可以安全地采用 Gaussian 或 Eburp 形状,选择性脉冲的 90° 宽度为 50 ms。 - 在 AcquPars 部分设置相应的参数。
- 设置 1小时 90° 脉冲长度。
- 设置形状脉冲的功率值(通过形状工具估计)。
- 设置总饱和时间。可以定期使用 1 s 到 4 s 之间的值。
- 将 relaxation delay 设置为 3 s。
- 将扫描次数 (NS) 设置为 8 的倍数。通常,它设置为 256、512 或 1024,以便在每个频率上以 2 为一组获得适当的信噪比。
- 将虚拟扫描次数 (DS) 设置为 8。
- 将 F2 中的点数设置为 16k、32k 或 64k。
注意:F2 中的点数增加将导致分辨率和信噪比的提高。因此,强烈建议至少使用 16k 个数据点。 - 在 F1 中设置点数。这是要使用的频率数,在本例中为 2 (on-resonance 和 off-resonance)。
注:按照惯例,F2 是指直接维度,即直接对自由感应衰减 (FID) 进行采样的维度,而 F1 表示间接维度。 - 设置接收器增益 (RG) 以避免使用自动命令 raga 溢出。
- 使用命令 expt 计算总实验的时间。
- 通过 zg 命令发送 Experiment 进行采集。
- 请始终在几分钟后检查实验是否正常运行。
- 用蛋白质-配体复合物制备样品。聚糖:凝集素摩尔比在 10:1 和 100:1 之间,蛋白质浓度范围在 0.01 和 0.2 mM 之间。对于 h半乳糖凝集素-7 与 LacNAc 的相互作用,在 pH 7.4 的氘代磷酸盐缓冲盐水中使用 50:1 的蛋白质:配体比例。
- 加工
注意:应用上述协议后获得伪 2D 光谱。行数对应于使用的频率数,通常为两个: on-resonance 和 off-resonance。- 处理第一个实验的 fid。
- 对 fid 编号 1 进行傅里叶变换(通过 efp 命令)并选择已处理光谱的目标(选择 procno 编号)。或者,使用 rser 1 命令读取第一个 fid。
- 然后,通过 lb 命令(通常为 3-5 Hz)调整线路展宽因子和相位。要手动相位,请单击 Process 选项卡,然后单击 adjust phase 子菜单。通过单击并拖动相应的按钮来执行零阶和一阶校正。保存阶段划分结果。此外,通过命令 abs 执行基线校正。
- 读取第二个实验的 fid,并使用相同的线展宽因子进行傅里叶变换(通过 efp 命令)。使用相同的相位参数和基线校正调整相位,并使用不同的代码保存处理后的光谱。
- 使用多重可视化 ( Δ) 中的可用按钮读取两个处理过的光谱,并使用多重可视化 (Δ) 中的可用按钮减去它们(非谐振 - 谐振)。新频谱是 STD NMR 频谱,它以不同的代码保存。
- 将 STD NMR 波谱与非谐振波谱叠加。
- 打开非谐振频谱 (fid 1) 并键入 .md 命令以打开多显示窗口。然后,上传 STD 频谱。
- 比较 STD NMR 波谱中信号的频率和强度(自动显示在右上角)。这提供了有关靠近蛋白质的质子及其相对接近度的所需信息。相对强度越高,它们越接近蛋白质(图 2)。
- 使用相应的软件测量非共振实验中的强度(积分)。在 TopSpin 中,转到 Analyse > Integrate。定义区域并将积分写入文件 (I0)。
- 使用相同的参数测量 STD NMR 实验中的强度(积分),并将它们写入文件 (ISTD)。
- 使用以下公式计算每个质子信号的 STD 值:
STD = (ISTD)/I0.
注:注:使用信号积分计算 STD 值需要充分分离质子信号。当发生信号重叠时,例如寡糖重叠时,可以通过评估 STD 和非共振光谱之间的信号强度比来确定 STD 值。 - 以百分比形式计算相对 STD。为此,请给质子一个 100% 的值,该值在非共振和 STD NMR 波谱的强度之间表现出最大差异。相应地计算其他质子的相对 STD 强度。
注意:正确分析 STD 数据,特别是用于确定配体结合表位,需要完全分配配体的 1H 信号。因此,强烈建议在采集 STD 光谱之前完成此任务。
- 处理第一个实验的 fid。
2. 1个 H-15N HSQC 实验
注:以下几行详细介绍了采用 1 H-15N HSQC 实验来监测受体(凝集素)的 1H 和 15N NMR 共振的化学位移变化,以响应配体(寡糖)含量增加的存在19。基于提取数据的化学位移扰动 (CSP) 分析对于鉴定结合配偶体以及绘制蛋白质结合界面和确定结合亲和力非常有价值。要更深入地了解 NMR 实验的设计和采集,请参考 NMR 仪器随附的相应制造商手册。
- 收购和加工
- 用感兴趣的凝集素制备样品。确保受体在骨架和侧链中的每个氨基酸残基中都完全 15N 标记。通常,为了检测光谱中可水交换的 HN 交叉峰,采用 H2O:D2O 的 90:10 混合物来制备缓冲溶液。所需的凝集素浓度范围在 0.05 至 0.2 mM 之间,具体取决于 15N 标记受体的可用性和必要的信噪比。
注意:蛋白质在整个实验时间内应保持稳定,在 NMR 管中不得产生可见的沉淀物。此外,它应该是纯的并且可溶于所选缓冲液。HSQC 交叉峰的完整 1H 和 15N 分配应该事先进行,以便 HSQC 谱图中的每个交叉峰都使用与特定氨基酸残基相对应的标记来识别。 - 从该制备中,将总体积为 0.6 mL 转移到 5 mm NMR 管中。
- 将 NMR 仪器设置在所需的温度。请参阅步骤 1.1.3 并执行相同的操作。
- 创建新数据集。请参阅步骤 1.1.4 并重复这些操作。
- 按照步骤 1.1.5 中的说明将 NMR 样品插入探头。
- 锁定溶剂信号。要启动锁定程序,请使用命令 lock 并从菜单中选择适当的溶剂。可以在锁定窗口中跟踪锁定信号。设置锁定增益,以便在锁定窗口中显示锁定信号。
- 自动 (通过命令 atma) 或手动 ( atmm 命令将打开 ATM 控制窗口以调整摆动曲线) 完成调谐和匹配过程。
- 使用 TopShim 工具设置最佳垫片。使用命令 topshim gui。请参阅步骤 1.1.8 中的说明。
- 确定 1H 90° 脉冲长度(如步骤 1.1.9 中所述)和偏移频率(命令 o1calib 将运行交互式 O1 校准程序,检索偏移频率)。当采用溶剂抑制方案的实验时,后一个参数极为重要。
- 按照 1.1.4 节中的说明创建新数据集。要减少或消除 H2O 信号的干扰,请使用脉冲序列 zgesgp。
- 通过在 AcquPars 窗口中修改各种参数来设置实验。
- 如前所述,引入 1H 90° 脉冲长度和偏移量 (o1),并将扫描次数 (NS) 设置为 32,将光谱窗口 (SW) 设置为 12 ppm 左右。
- 使用 Topspin 菜单栏中的 Shape 工具确定整形脉冲的功率电平。
- 使用自动命令 rga 设置接收器增益。
- 使用 zg 命令获取实验并处理得到的 FID 以获得 1H NMR 波谱。
- 创建一个新数据集用于获取 1 H-15N HSQC NMR 实验。在 AcquPars 选项卡中,选择脉冲程序目录中可用的脉冲程序 hsqcetfpf3gp 。
- 设置实验。使用命令 getprosol 加载默认形状、幂和时间。然后,更新 1H 90° 脉冲长度和偏移的值。
- 定义以下参数。
- 将松弛延迟设置为 1-5 秒。
- 将扫描次数设置为 4 的倍数。通常,它设置为 8、16、32 或 64 以获得适当的信噪比。
- 将虚拟扫描次数设置为 128。
- 将 F2 中的点数设置为 1k、2k 或 4k。
- 设置 F1 中的点数:要使用的 t1 增量数。根据光谱窗口,该值介于 128 和 256 之间。
- 将 15N 维度的光谱窗口中心调整为 δ 117 ppm,并将相应的光谱宽度设置为 36 ppm。这些值需要针对每个特定系统进行优化。
- 设置接收器增益以避免溢出(通过使用 rga 命令)
- 计算总实验的时间。典型的实验时间约为 1 小时。
- 键入 zg 以发送实验进行采集。
注意:请始终在几分钟后检查实验是否正常运行。 - 使用命令 xfb 处理 FID。使用"处理"选项卡中的命令 abs2 和相位校正执行基线校正。要手动相位,请单击 adjust phase 子菜单,然后选择 2D 光谱的几个交叉峰。然后,通过单击并拖动相应的按钮,按顺序将零和一阶校正应用于行和列。保存阶段划分结果。
- 保存生成的 2D 光谱。
- 制备配体的高浓度储备溶液。典型值为 50-100 mM。
- 从高浓度的聚糖储备液中,将相应体积(几微升)转移到含有受体的 NMR 管中,以获得所需的蛋白质:配体摩尔比并记录光谱。
注:此步骤启动滴定系列,其中配体被滴定到蛋白质样品中。必须针对每种特定情况确定适当的蛋白质与配体比率。如果结合亲和力完全未知,建议在初始点中使用亚化学计量量的配体。 - 对新制备的样品执行步骤 2.1.1 至 2.1.19。
- 对蛋白质与配体比率增加的样品重复步骤 2.1.21 和 2.1.22。
注:滴定系列数据的准确拟合需要采集多个 1 H-15N-HSQC 实验,涵盖广泛的蛋白质与配体比率,包括实现蛋白质饱和所需的比率。
- 用感兴趣的凝集素制备样品。确保受体在骨架和侧链中的每个氨基酸残基中都完全 15N 标记。通常,为了检测光谱中可水交换的 HN 交叉峰,采用 H2O:D2O 的 90:10 混合物来制备缓冲溶液。所需的凝集素浓度范围在 0.05 至 0.2 mM 之间,具体取决于 15N 标记受体的可用性和必要的信噪比。
- 分析
- 使用适当的软件可视化载脂蛋白物种的处理后 2D HSQC 光谱:TopSpin、MestReNova 和 CCPNMR 都是处理 NMR 数据的合适程序。
注意:这是蛋白质的指纹图谱。观察到的 1H 和 15N 化学位移取决于每个氨基酸的相应化学环境,这在很大程度上取决于蛋白质的 3D 结构。这个谱图谱称为蛋白质指纹图谱。分散良好的 2D 1 H-15N HSQC 谱图,其中所有交叉峰都显示出均匀的强度,强烈表明存在充分折叠的蛋白质19。 - 生成所有交叉峰的 1H 和 15N 频率列表。使用补充软件,例如 CCPNMR 程序20,可以帮助完成该过程。
- 将第一滴定点或第二滴定点的谱图叠加到 apo 蛋白的谱图上。
- 为此,请打开与 apo 状态对应的 2D 光谱,单击 Multiple display(多重显示)选项卡,然后添加第二个 2D 光谱。两个光谱的目视检查提供了有关配体和蛋白质之间是否存在相互作用的信息。
注:从蛋白质的角度来看,结合的存在提供了直接参与识别事件的氨基酸化学环境的变化,以及随之而来的化学位移扰动 (CSP)。
- 为此,请打开与 apo 状态对应的 2D 光谱,单击 Multiple display(多重显示)选项卡,然后添加第二个 2D 光谱。两个光谱的目视检查提供了有关配体和蛋白质之间是否存在相互作用的信息。
- 对每个滴定点重复步骤 2.2.2 和 2.2.3,为不同光谱中的所有交叉峰生成 1H 和 15N 频率列表,对应于不同的蛋白质 - 配体摩尔比。
注:无需执行任何新的交叉峰分配即可测量每个滴定点的化学位移。在凝集素:聚糖相互作用中常见的快速交换机制中,可以简单地在整个滴定过程中跟踪峰的逐渐移动。 - 检查最后一个滴定点是否与前一次添加相比,基本上没有化学位移扰动。这一事实表明蛋白质结合位点已被配体饱和,配体高度过量。
- 使用以下公式计算最大化学位移扰动 (maxCSP):
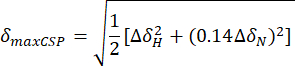
ΔH 和 ΔδN 分别是载脂蛋白状态和最后一个滴定点之间 1H 和 15N 频率的化学位移差异。 - 在 2D 图的垂直 y 轴上绘制最大化学位移扰动 (maxCSP) 与相应的氨基酸残基(在水平 x 轴上)的关系。
- 目视检查显示蛋白质结合状态和 apo 状态之间最大 CSP 的氨基酸残基。它们很可能属于结合位点或与其相邻。
- 如果蛋白质的 3D 结构可用,请使用适当的软件(如 PyMOL 或 BIOVIA Discovery studio)打开相应的 PDB。这些分子可视化程序广泛用于结构生物学应用。选择显示最高 maxCSP(高于标准偏差的两倍)的残基和特定颜色,以定位推定的结合位点。
- 在快速交换机制的情况下,从观察到的 CSP 在每个点 (Δδobs) 的 CSP 相对于该点的特定蛋白质 [P] 和配体 [L] 浓度的非线性最小二乘拟合中估计解离常数(KD):
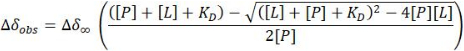
注:此公式可应用于显示清晰隔离信号的交叉峰。对获得的值进行平均,以提供 KD 的估计值。
- 使用适当的软件可视化载脂蛋白物种的处理后 2D HSQC 光谱:TopSpin、MestReNova 和 CCPNMR 都是处理 NMR 数据的合适程序。
结果
在此,我们提出了一种利用 1H-STD NMR 和 1 H-15N HSQC 实验的方案,以揭示凝集素和小寡糖之间结合相互作用的细节。包括在 hGalectin-7 (hGal-7) 对 LacNAc 的分子识别分析中获得的结果,作为成功实施该方案以及这些 NMR 方法研究分子识别过程的精细细节的有效性的说明性示例。图 3 显示了 LacNAc 与 hGal-7 相互作用的 1H-STD NMR 谱图。STD NMR 信号的存在表明结合(图 3A)。此外,只有那些属于与蛋白质紧密接触的质子的信号才会出现,从而可以描绘结合表位(图 3B)。图 4 突出显示了如何将蛋白质的 1 H-15N HSQC 谱图用作其指纹图谱,图 5 说明了 1 H-15N 异核单量子相干 (HSQC) 滴定实验的应用,以确定 LacNAc 结合后 h半乳糖凝集素-7 骨架酰胺基团的化学位移扰动。这些数据不仅揭示了相互作用的存在,还描绘了凝集素的结合界面。图 6 展示了滴定数据分析如何能够估计 h半乳糖凝集素-7 对 LacNAc 的结合亲和力,其位于高微摩尔范围内。这一发现与使用替代技术获得的结果一致。
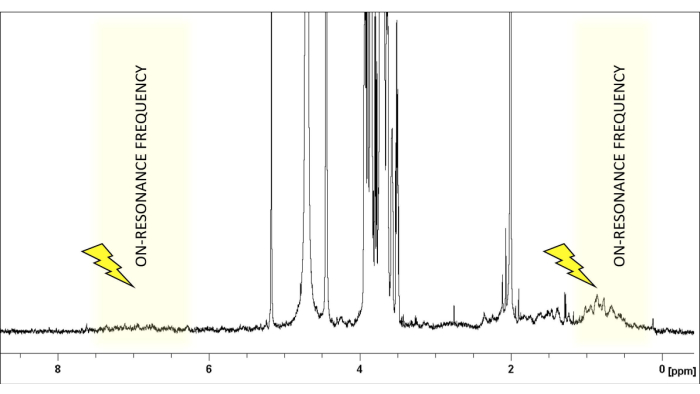
图 1:导通共振频率的选择。显示了 pH 值为 7.4 的氘代磷酸盐缓冲盐水中 LacNAc:hGal-7 的 50:1 比例的 1H-NMR 谱图。配体 (LacNAc) 的信号限制在 2.0-5.2 ppm 之间的区域内。饱和频率经过精心选择,以确保在 1-2 ppm 范围内不存在配体质子,从而允许对蛋白质的质子进行选择性照射。请单击此处查看此图的较大版本。
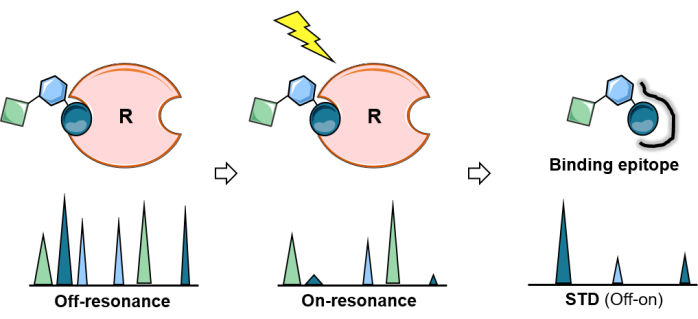
图 2:STD NMR 实验。 STD 实验的示意图:第一个光谱(非共振)用作参考,而在第二个光谱(on-resonance)中,进行蛋白质饱和。饱和度在整个蛋白质中有效传播,并转移到与蛋白质紧密接触的配体质子上。由此产生的差谱(STD 谱)仅产生那些经历过饱和的共振。STD 实验的分析允许结合糖的表位定位。 请单击此处查看此图的较大版本。
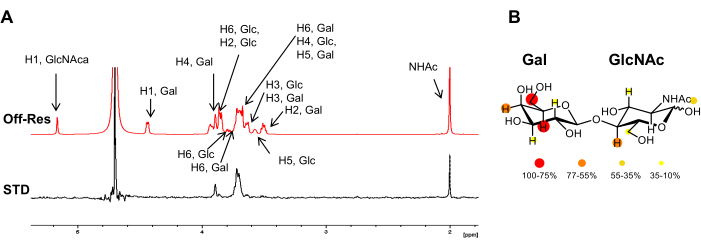
图 3:从配体角度进行的结合分析。(A) LacNAc 与 hGal-7 相互作用的非共振和 1H STD-NMR 谱图的叠加。在 STD 光谱中,只有那些属于与蛋白质紧密接触的质子的信号才会出现。配体的 1H 共振的注释在非共振光谱中报告。(B) 将相对 STD 强度着色映射到 LacNAc 的化学结构中。请单击此处查看此图的较大版本。
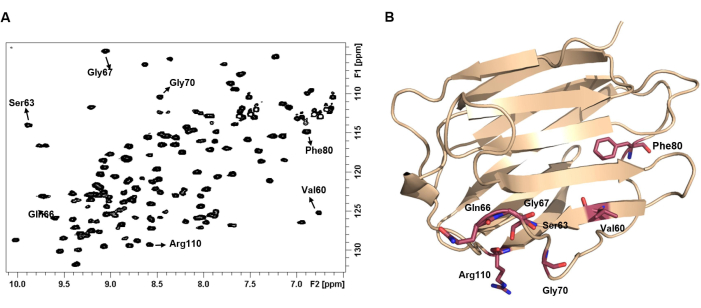
图 4:蛋白质的 1 H-15N HSQC 谱代表其指纹图谱。 (A) 载脂蛋白形式的 100 μM hGal-7 的 1 H-15N HSQC 光谱。在 25 °C 下记录光谱。 一些 NH 交叉峰用其相应氨基酸的标记进行注释。(B) 每个 NH 对都显示出独特的化学变化,这取决于化学环境,因此也取决于蛋白质的 3D 结构。请单击此处查看此图的较大版本。
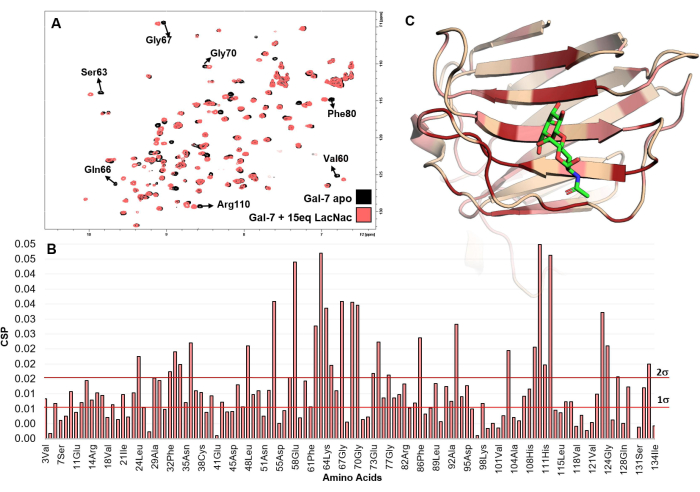
图 5:从蛋白质的角度进行结合分析。(A) 显示了将 LacNAc 滴定到 hGal-7 溶液中时记录的 1 H-15N HSQC 谱图的叠加。在光谱中,几个交叉峰经历化学位移变化,检查谱图清楚地表明了相互作用。(B) 从用 hGal-7 滴定 LacNAc(15 个当量)推导出主链酰胺信号的最大化学位移扰动 (maxCSP) 图。(C) 根据 CSP 分析,hGal-7 最受扰动的氨基酸被映射到 5gal PDB 结构中。在 3D 模型中,红色是指超过 2σ 的 CSP 值,而粉红色是指 1σ 到 2σ 之间的值。彩色区域可能代表结合位点。请单击此处查看此图的较大版本。
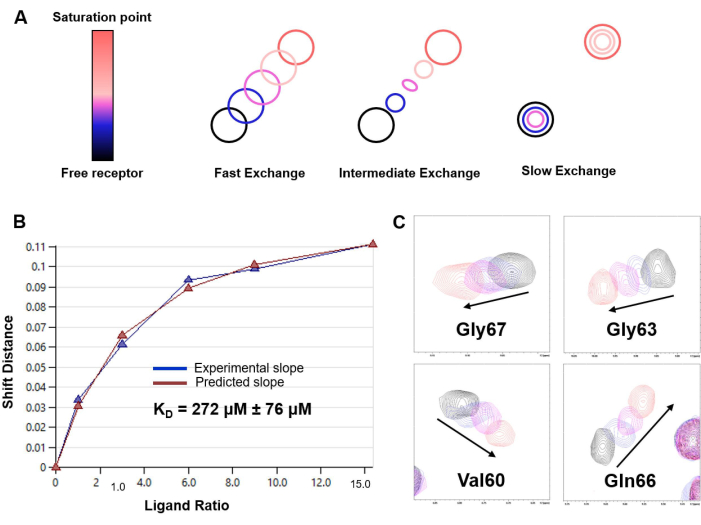
图 6:基于 1 H-15N HSQC 滴定实验的 KD 测定。(A) 基于 1 H-15N HSQC 的滴定模式的表示,具体取决于研究中系统 NMR 时间尺度中的化学交换速率(快速、中等或慢速)。在 LacNAc/hGal-7 相互作用的情况下观察到快速交换机制。(B) 从 hGal-7 和 LacNAc 二糖模型系统在不同配体浓度下的 CSP 分析中获得的拟合曲线和 KD 估计值。估计的 KD 报告为相应的误差,作为 20 种不同氨基酸数据的平均值;(C) 1 H,15N-HSQC 谱图的片段,显示了滴定过程中所选交叉峰的漂移。请单击此处查看此图的较大版本。
讨论
饱和转移差 NMR (STD-NMR) 已成为研究配体-蛋白质相互作用最常用和最通用的 NMR 方法。如上所示,它依赖于饱和转移现象,实验装置涉及两个一维 (1D) 1H 光谱的采集:on-resonance¬ 和 ¬off-resonance 光谱。在导通共振实验期间,通过在一定时间内(饱和时间通常为 1-3 秒)施加一系列低功率射频脉冲来实现蛋白质特定质子的饱和。为避免配体直接饱和,饱和脉冲的频率和长度经过优化,以选择性地照射蛋白质的特定质子;即,它们必须在任何配体信号的空位频率下应用,并具有适当的长度(图 1)。根据 50 ms 饱和脉冲的经验法则,从饱和区域到最近的配体信号应保持 1 ppm 的差异。通常,施加在蛋白质脂肪族区域的选择性饱和脉冲可提供增强的饱和效应。或者,如果配体分子不包含任何芳香族信号,也可以照射芳香族质子 (6-7 ppm)。这对于天然存在的聚糖非常有用,因为它们不带有芳香族基团。一旦蛋白质的某个区域被选择性照射,饱和度就会通过偶极 1 H-1H 交叉弛豫(自旋扩散)沿蛋白质传播。最终,饱和度到达结合位点的蛋白质质子,然后通过分子间 1 H-1H NOE 转移到与受体紧密接触 (r < 5 Å) 的糖质子。显然,饱和配体质子的信号强度降低。接收到饱和后,由于结合动力学,瞬时结合的配体(需要快速交换)解离,饱和信息在游离态下积累。由于这个过程,NMR 导通共振波谱呈现出减弱的信号(图 2)。
为了清楚地显示结合聚糖的 1H 原子核的这种强度扰动,获得了对照质子 NMR 谱(非共振),其中在相同条件下,饱和度远离任何受体或碳水化合物信号(通常在 40-100 ppm 之间)。磁共振和磁共振之间减去的 1D 谱图仅显示了强度改变的配体 1H 核的信号:那些离受体结合位点足够近以接收磁化的信号(图 2)。
然而,并非结合碳水化合物的所有 1H 细胞核都获得相同量的饱和度。理论上,从受体到结合配体的磁化转移是距离依赖性的 (1/r6)。这意味着游离寡糖 1H 核之间的转移饱和强度包含有关配体质子和受体质子之间空间接近度的信息,并且对于更靠近受体的质子,STD NMR 强度更大。因此,STD NMR 实验还允许确定碳水化合物的结合表位(图 2 和图 3),因为靠近蛋白质表面的配体质子比不直接参与结合的质子显示出更高的强度。
该实验可应用于弱中亲和力的系统,很少应用于低 μM 或 nM 范围内的强亲和力系统。事实上,它要求解离速率在弛豫时间尺度上要快。否则,饱和度转移信息会在配体解离之前通过弛豫丢失。
另一方面,基于蛋白质的 NMR 实验在揭示配体-蛋白质相互作用方面是独一无二的,无需解决原子分辨率结构即可以氨基酸水平的精度进行解析。它直接检查溶液中的分子识别现象,无需共结晶。CSP 分析映射对于发现配体和绘制蛋白质结合位点非常强大(图 4 和 图 5)。该方法适用于 mM 和 nM 范围之间的任何亲和力范围,甚至适用于化学变化时间尺度21 中汇率较慢的系统。
然而,由于松弛问题,这种方法可能不适用于分子量高于 30-40 kDa 的蛋白质。然后可以使用 TROSY 替代品18 ,当与蛋白质氘代偶联时,它特别强大。此外,应用 15N 均匀标记蛋白质(另一个样品用 13C 和 15N 双重标记,以便能够完成所需的骨架分配)。因此,应优化蛋白质表达条件,包括相应的表达系统,以便能够获得毫克级蛋白质。表现出寡聚化或聚集倾向的蛋白质也不适合此分析。本文用于记录 NMR 数据的仪器是配备 TCI 低温探头的 Bruker 800 MHz 波谱仪。使用低于 600 MHz 的仪器或没有低温探头使用这种方法将极具挑战性。
披露声明
作者没有什么可披露的。
致谢
我们感谢西班牙 Agencia Estatal de Investigación 的 Severo Ochoa 卓越中心认证 CEX2021-001136-S,由 MCIN/AEI/10.13039/ 501100011033 和 CIBERES,由 Instituto de Salud Carlos III(ISCIII,西班牙马德里)发起。我们还感谢欧盟委员会的 GLYCOTWINNING 项目。
材料
| Name | Company | Catalog Number | Comments |
| 5 mm Shigemi microtube set mat | CortecNet SAS | S30BMS-005B | |
| Alpha-Lactose-Agarose | Sigma-Aldrich Química S.L. | 7634-5ML | |
| Ammonium chloride (15 N, 99%) | LC-0179-N-50G | Tracer Tecnologías Analíticas S.L | |
| Ampicillin (Sodium Salt) | Melford Laboratories LTD | A40040 | |
| BIOVIA Discovery studio | BIOVIA, Dassault Systèmes | ||
| BL21(DE3) Chemically Competent Cells | Merck Life Science, S.L.U. | CMC0014-40X40UL | |
| Centrifuge | Beckman Coulter | Allegra X-22R | |
| D2O | Cambridge Isotope Laboratories, Inc. | DLM-4-1000 | |
| Incubator | Eppendorf | Innova 42 | |
| IPTG (Isopropyl ß-D-1-thiogalactopyranoside) | VWR International Eurolab S.L. | VW437144N | |
| LacNAc | Elicityl | GLY008 | |
| Luria Bertani (LB) Broth | Merck Life Science, S.L.U. | 3397-1KG | |
| Matraz Erlenmeyer B N 5000 CC | VWR International Eurolab S.L. | 214-1137 | |
| PBS 10x | Bio-Rad | 1610780 | |
| PyMOL | PyMOL Molecular Graphics System | Version 2.0 Schrödinger | |
| Sonicator | Sonics & Materials, Inc. | VC 505 | |
| Superconducting NMR magnet | Bruker | 600 MHz AVANCE III | |
| Superconducting NMR magnet | Bruker | 800 MHz AVANCE III |
参考文献
- Solís, D., et al. A guide into glycosciences: How chemistry, biochemistry and biology cooperate to crack the sugar code. Biochim Biophys Acta. 1850 (1), 186-235 (2015).
- Poveda, A., Jimenez-Barbero, J. NMR studies of carbohydrate-protein interactions in solution. Chem Soc Rev. 27, 133-144 (1998).
- Kogelberg, H., Solís, D., Jiménez-Barbero, J. New structural insights into carbohydrate-protein interactions from NMR spectroscopy. Curr Opin Struct Biol. 13 (5), 646-653 (2003).
- Widmalm, G. Glycan shape, motions, and interactions explored by NMR spectroscopy. JACS Au. 4 (1), 20-39 (2024).
- Marchetti, R., et al. 34;Rules of Engagement" of protein-glycoconjugate interactions: A molecular view achievable by using NMR spectroscopy and molecular modeling. ChemistryOpen. 5 (4), 274-296 (2016).
- Ardá, A., Jiménez-Barbero, J. The recognition of glycans by protein receptors. Insights from NMR spectroscopy. Chem Commun. 54 (38), 4761-4769 (2018).
- Gimeno, A., Valverde, P., Ardá, A., Jiménez-Barbero, J. Glycan structures and their interactions with proteins. A NMR view. Curr Opin Struct Biol. 62, 22-30 (2020).
- Cañada, F. J., et al. Conformational and structural characterization of carbohydrates and their interactions studied by NMR. Curr Med Chem. 29 (7), 1147-1172 (2022).
- Valverde, P., Quintana, J. I., Santos, J. I., Ardá, A., Jiménez-Barbero, J. Novel NMR avenues to explore the conformation and interactions of glycans. ACS Omega. 4, 13618-13630 (2019).
- Jiménez-Barbero, J., Asensio, J. L., Cañada, F. J., Poveda, A. Free and protein-bound carbohydrate structures. Curr Opin Struct Biol. 9 (5), 549-555 (1999).
- Quintana, J. I., Atxabal, U., Unione, L., Ardá, A., Jiménez-Barbero, J. Exploring multivalent carbohydrate-protein interactions by NMR. Chem Soc Rev. 52 (5), 1591-1613 (2023).
- Roldós, V., Cañada, F. J., Jiménez-Barbero, J. Carbohydrate-protein interactions: a 3D view by NMR. Chembiochem. 12 (7), 990-1005 (2011).
- Unione, L., Ardá, A., Jiménez-Barbero, J., Millet, O. NMR of glycoproteins: profiling, structure, conformation, and interactions. Curr Opin Struct Biol. 68, 9-17 (2021).
- del Carmen Fernández-Alonso, M., Díaz, D., Berbis, M. A., Marcelo, F., Cañada, J., Jiménez-Barbero, J. Protein-carbohydrate interactions studied by NMR: from molecular recognition to drug design. Curr Protein Pept Sci. 13 (8), 816-830 (2012).
- Angulo, J., Nieto, P. M. STD-NMR: application to transient interactions between biomolecules-a quantitative approach. Eur Biophys J. 40 (12), 1357-1369 (2012).
- Mayer, M., Meyer, B. Group epitope mapping by saturation transfer difference NMR to identify segments of a ligand in direct contact with a protein receptor. J Am Chem Soc. 123 (25), 6108-6117 (2001).
- Williamson, M. P. Using chemical shift perturbation to characterise ligand binding. Prog Nucl Magn Reson Spectrosc. 73, 1-16 (2013).
- Fernández, C., Adeishvili, K., Wüthrich, K. Transverse relaxation-optimized NMR spectroscopy with the outer membrane protein OmpX in dihexanoyl phosphatidylcholine micelles. Proc Natl Acad Sci U S A. 98 (5), 2358-2363 (2001).
- Yee, A., Gutmanas, A., Arrowsmith, C. H. Solution NMR in structural genomics. Curr Opin Struct Biol. 16 (5), 611-617 (2006).
- Skinner, S. P., Fogh, R. H., Boucher, W., Ragan, T. J., Mureddu, L. G., Vuister, G. W. CCPNMR AnalysisAssign: a flexible platform for integrated NMR analysis. J Biomol NMR. 66 (2), 111-124 (2016).
- Gimeno, A., et al. Minimizing the entropy penalty for ligand binding: Lessons from the molecular recognition of the histo blood-group antigens by human Galectin-3. Angew Chem Int Ed Engl. 58 (22), 7268-7272 (2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。