Method Article
Démêler les interactions glycanes-protéines : la résonance magnétique nucléaire (RMN) à la rescousse
Dans cet article
Résumé
Nous présentons ici un protocole détaillant l’acquisition, le traitement et l’analyse d’une série d’expériences RMN visant à caractériser les interactions protéine-glycane en solution. Les méthodologies les plus courantes basées sur les ligands et les protéines sont décrites, qui contribuent sans aucun doute aux domaines de la glycobiologie structurale et des études de reconnaissance moléculaire.
Résumé
Les interactions des glycanes avec les protéines modulent de nombreux événements liés à la santé et à la maladie. En fait, l’établissement de ces événements de reconnaissance et leurs conséquences biologiques sont intimement liés aux structures tridimensionnelles des deux partenaires, ainsi qu’à leurs caractéristiques dynamiques et à leur présentation sur les compartiments cellulaires correspondants. Les techniques de RMN sont uniques pour démêler ces caractéristiques et, en effet, diverses méthodologies basées sur la RMN ont été développées et appliquées pour surveiller les événements de liaison des glycanes avec leurs récepteurs associés. Ce protocole décrit les procédures d’acquisition, de traitement et d’analyse de deux des méthodologies de RMN les plus puissantes employées dans le domaine de la glycobiologie RMN, la différence de transfert de saturation 1H (STD) et lesexpériences de titrage de cohérence quantique unique (HSQC) 1 H,15 N-hétéronucléaire, qui offrent des informations complémentaires du point de vue des glycanes et des protéines, respectivement. En effet, lorsqu’ils sont combinés, ils offrent une boîte à outils puissante pour élucider à la fois les aspects structurels et dynamiques des processus de reconnaissance moléculaire. Cette approche globale améliore notre compréhension des interactions glycanes-protéines et contribue à faire progresser la recherche dans le domaine de la glycobiologie chimique.
Introduction
La reconnaissance moléculaire des glycanes est essentielle pour de nombreux processus liés à la santé et à la maladie. La spécificité et la sélectivité des récepteurs biologiques (lectines, anticorps, enzymes) pour les glycanes dépendent fortement de l’ajustement de l’équilibre précaire entre les divers composants de l’enthalpie (CH-π et van der Waals, liaisons hydrogène, électrostatique) et de l’entropie (hydrophobie, dynamique, solvatation-désolvatation)1.
Compte tenu de la grande diversité chimique et de la nature dynamique des glycanes, les méthodes RMN ont été largement utilisées pour disséquer les interactions des glycanes depuis plus de 25 ans2, car ces méthodologies fournissent d’excellentes informations sur les événements de reconnaissance moléculaire avec des détails précis, à une résolution atomiquede 3,4, même lorsque les preuves d’interaction requises ne peuvent pas être récupérées en utilisant d’autres méthodologies. En tant que point clé, la RMN est polyvalente et permet d’étudier des événements dynamiques, au niveau atomique, à différentes échelles de temps, constituant de loin la meilleure technique pour étudier la structure, la conformation et la dynamique des glycanes en solution. Néanmoins, démêler ces informations peut être un processus assez complexe qui nécessite l’utilisation de stratégies bien définies ainsi qu’une analyse minutieuse des données5.
Les techniques de RMN sont diverses et, en effet, il existe de nombreuses méthodologies qui peuvent être utilisées pour démêler les interactions glycanes-protéines6. Nous décrivons ici deux approches de base de RMN qui sont actuellement employées pour déchiffrer les interactions glycanes-récepteurs 7,8, en mettant l’accent sur la façon de démêler la présentation de l’épitope clé du glycane ainsi que du site de liaison aux protéines9.
Dans tout événement de reconnaissance moléculaire, lorsqu’un récepteur se lie à un ligand donné, il y a un processus d’échange chimique qui affecte de nombreux paramètres RMN des participants à la liaison10. Par conséquent, du point de vue de la RMN, l’interaction peut être suivie soit du point de vue du ligand du glycane, soit du point de vue du récepteur protéique11. D’une manière générale, le récepteur protéique est une grande biomolécule (mouvement de rotation lent, avec des vitesses dans l’échelle de temps ns, et donc, une relaxation transversale rapide), tandis que le glycane en interaction peut être considéré comme une molécule de taille petite à moyenne (mouvement de rotation rapide, avec des vitesses dans l’échelle de temps ps, et relaxation transversale lente)12. D’un point de vue standard, les signaux RMN du glycane sont étroits, tandis que ceux du récepteur sont larges13.
Les méthodes RMN basées sur les ligands reposent sur le changement spectaculaire que subissent de nombreux paramètres RMN des glycanes lors du passage de l’état libre à l’état lié14. La RMN-STD est la technique de RMN expérimentale la plus utilisée pour évaluer diverses caractéristiques de liaison des glycanes15, de la déduction de l’existence de la liaison à l’état de solution à la détermination de l’épitope de liaison des glycanes ; c’est-à-dire les atomes du ligand qui sont en contact avec le récepteur protéique16.
Alternativement, les méthodes RMN basées sur les récepteurs surveillent les changements qui se produisent dans les signaux du récepteur protéique en présence du glycane par rapport à ceux enregistrés pour l’état apo17. Ceux-ci sont principalement axés sur le criblage des perturbations chimiques des signaux protéiques entre les deux états. L’expérience la plus couramment utilisée est le HSQC 1 H-15N, ou ses alternatives TROSY18.
La combinaison des deux approches permet d’appliquer la RMN à de nombreux systèmes divers qui présentent un large éventail d’affinités. Cependant, pour les méthodes RMN basées sur les récepteurs, contrairement à celles basées sur le ligand, une quantité relativement importante de protéines solubles, non agrégées et marquées à des isotopes stables (15N) doit être disponible.
Nous décrivons ici les deux méthodes, en soulignant leurs forces et leurs faiblesses. Notez que les étapes de base décrites dans le protocole servent d’exemples pour l’utilisation des spectromètres Bruker. Par conséquent, les noms des commandes et des paramètres s’alignent sur ceux utilisés dans TopSpin (le logiciel de contrôle des spectromètres de Bruker).
Protocole
1. Différence de transfert de saturation RMN (STD-NMR)
REMARQUE : Les lignes suivantes décrivent les procédures fondamentales d’acquisition, de traitement et d’analyse des expériences de RMN-STD. Ces étapes servent à illustrer l’utilité de la technique pour détecter la liaison du ligand et pour élucider l’épitope de liaison du ligand. Pour une compréhension plus approfondie de la conception et de l’acquisition des expériences de RMN, veuillez vous référer au manuel du fabricant correspondant fourni avec l’instrument de RMN.
- Acquisition
- Préparez l’échantillon avec le complexe protéine-ligand. Utiliser des rapports molaires glycane :lectine compris entre 10:1 et 100:1 avec des concentrations en protéines comprises entre 0,01 et 0,2 mM. Pour l’interaction de la galectine-7 havec la LacNAc, utilisez le rapport protéine :ligand 50:1 dans une solution saline tamponnée au phosphate deutéré à un pH de 7,4.
REMARQUE : Le récepteur protéique doit être pur et soluble dans le tampon de son choix (dans le cas d’expériences de RMN-STD, des versions deutérées du tampon correspondant sont préférables pour réduire l’interférence possible du signal RMN 1H). La concentration de la protéine est vérifiée à l’avance à l’aide d’un spectrophotomètre pour mesurer l’absorbance à 280 nm. - À partir de la solution préparée, transférez un volume total de 0,6 mL dans un tube RMN de 5 mm à l’aide d’une pipette.
- Préparez l’instrument RMN à la température requise (les températures d’expérience courantes se situent entre 10 °C et 45 °C). Ouvrez le moniteur de contrôle de la température à l’aide de la commande edte et réglez la température souhaitée. Pour l’étude hGalectin-7/LacNAc, la température a été fixée à 25 °C.
- Générez un nouvel ensemble de données contenant la séquence d’impulsions zg.
- Pour une opération simple, ouvrez une expérience existante et tapez la commande edc . Une boîte de dialogue apparaît, définissant le titre, les caractéristiques (spécifications de l’échantillon, solvant) et certains paramètres de l’expérience.
- Si une modification de la séquence d’impulsions d’origine est nécessaire, naviguez dans les fenêtres ased (paramètres) et AcquPars (paramètres d’acquisition). À ce stade, choisissez le programme d’impulsions souhaité dans la bibliothèque du spectromètre.
- Pour un spectre RMN standard de 1H, sélectionnez la séquence d’impulsions zg dans la liste disponible.
REMARQUE : Dans le cas d’échantillons ayant une teneur en eau élevée, l’utilisation de dispositifs de suppression de l’eau peut être nécessaire pour augmenter le rapport signal/bruit. L’utilisation de séquences d’impulsions telles que le zgesgp, qui excitent des modules qui rendent une excellente suppression mais contrôlent la phase des signaux restants, sont souhaitables. Veuillez consulter le tutoriel RMN du fabricant pour obtenir de plus amples renseignements sur les types de dispositifs de suppression de l’eau et leurs principales caractéristiques.
- Insérez l’échantillon RMN dans la sonde en activant l’air de levage de l’échantillon. Utilisez la commande ej, positionnez l’échantillon sur le dessus de l’aimant et désactivez le soulèvement de l’échantillon à l’aide de la commande ij.
REMARQUE : Pour injecter l’échantillon dans l’aimant à l’aide d’un passeur d’échantillons, utilisez la commande sx suivie du numéro de position, n, correspondant à la position du tube RMN dans le plateau de l’échantillonneur automatique. - Verrouillez le signal du solvant en tapant la commande lock, puis en sélectionnant le solvant approprié dans le menu.
- Une fois l’échantillon inséré dans la sonde, terminez le processus de réglage et d’appariement à l’aide du module automatique atma ou du module manuel atmm.
- Démarrez le calage automatique à l’aide de la commande topshim gui. Cela ouvrira une interface graphique où la dimension de cale 1D sera sélectionnée et démarrée.
REMARQUE : Pour minimiser les instabilités de calage dues à de subtiles variations de champ ou de température, l’autoshim peut être activé pour l’acquisition de l’expérience. Cela peut être effectué en accédant à la fenêtre de contrôle BSMS et en cliquant sur autoshim. Le fait de se transformer en surbrillance verte indique que l’autoshim a été activé. Veuillez noter que lors de l’utilisation d’autoshim, les problèmes potentiels d’instabilité de l’échantillon doivent passer inaperçus. Par conséquent, la prudence est de mise lorsque l’autoshim est utilisé. - Déterminez l’impulsion de 1h à 90°. Cela peut être effectué automatiquement via la commande pulsecal .
- Modifiez divers paramètres dans la fenêtre AcquPars. Pour un spectre RMN régulier de 1H, réglez le nombre de balayages (NS) à 32 et la fenêtre spectrale souhaitée (SW) à env. 12 pages.
REMARQUE : La séquence d’impulsions zgesgp comprend un module de suppression de solvant pour éliminer le signal HDO résiduel, qui doit être centré au milieu du spectre. À cette fin, O1 doit être défini avec précision dans AcquPars. - Réglez le gain du récepteur pour éviter le débordement avec la commande automatique rga.
- Maintenant, acquérez le spectre RMN standard 1H à l’aide de la commande zg.
- Une fois l’acquisition terminée, traitez le spectre à l’aide de la commande efp. Appliquez les corrections de base et de phase à l’aide de la barre de menu TopSpin.
REMARQUE : Des signaux RMN 1H dérivant du glycane et de la protéine sont observés (Figure 1). L’analyse détaillée du spectre RMN acquis est recommandée pour la mise en œuvre de l’expérience de RMN STD, comme indiqué à la section 1.1.14. - Créez un nouvel ensemble de données et téléchargez la séquence d’impulsions RMN STD à utiliser de la même manière que celle décrite pour l’expérience RMN 1H à la section 1.1.4. Dans les instruments Bruker, différentes séquences d’impulsions sont disponibles dans le catalogue de programmes d’impulsions, toutes nommées stddiffXXX. Le plus simple (stddiff) n’inclut aucun schéma de suppression de l’eau ou filtre de suppression des protéines.
- Pour les échantillons avec une teneur significative en H2O, sélectionnez les séquences stddiffgp19 ou stddiffesgp, qui incluent un module de sculpture de porte d’eau ou d’excitation. Dans le cas d’un spectre avec des signaux RMN protéiques intenses comme arrière-plan, sélectionnez les séquences stddiffXXX.3. Dans chaque cas, optimisez les paramètres spécifiques correspondants pour chaque module de suppression d’eau (c’est-à-dire d19 dans les schémas de watergate).
- Définir les fréquences de résonance activée et désactivée pour l’expérience RMN STD. Vous trouverez la liste des fréquences dans les paramètres AcquPars de la fenêtre ased sous l’entrée FQ2LIST. Les fréquences de résonance activée et désactivée définies en Hertz doivent être écrites manuellement dans la liste et enregistrées sous un nouveau nom. Cette nouvelle liste sera utilisée dans l’expérience STD-NMR.
- Choisissez la fréquence de résonance à une région spectrale dépourvue de signaux de glycanes, généralement autour de δ(1H) 0 ou 6,6 ppm, pour les glycanes typiques (Figure 1). Réglez la fréquence de désactivation dans une région qui ne montre aucun ligand ou proton protéique. Il peut être réglé en toute sécurité à +18000 ou -18000 Hz.
- Définissez l’impulsion façonnée à utiliser pendant le temps de saturation dans les paramètres AcquPars de la fenêtre ased .
REMARQUE : Il existe de nombreuses possibilités. Les formes gaussiennes ou Eburp peuvent être utilisées en toute sécurité, avec une largeur de 90° de l’impulsion sélective de 50 ms. - Définissez les paramètres correspondants dans la section AcquPars.
- Réglez la longueur d’impulsion de 1H à 90°.
- Définissez la valeur de puissance de l’impulsion façonnée (estimée à l’aide de l’outil de forme).
- Réglez le temps de saturation total. Des valeurs comprises entre 1 s et 4 s peuvent être employées régulièrement.
- Réglez le délai de relaxation sur 3 s.
- Définissez le nombre de balayages (NS) sur un multiple de 8. Habituellement, il est réglé à 256, 512 ou 1024 pour obtenir le bon rapport signal/bruit par groupes de 2 à chaque fréquence.
- Réglez le nombre de balayages factices (DS) sur 8.
- Définissez le nombre de points dans F2 sur 16k, 32k ou 64k.
REMARQUE : Un nombre accru de points dans F2 se traduira par une résolution et un rapport signal/bruit améliorés. Pour cette raison, l’utilisation d’un minimum de 16 000 points de données est fortement conseillée. - Définissez le nombre de points en F1. Il s’agit du nombre de fréquences à utiliser, dans ce cas, 2 (la on-resonance et la off-resonance).
REMARQUE : Par convention, F2 fait référence à la dimension directe, la dimension le long de laquelle la décroissance à induction libre (FID) est échantillonnée directement, tandis que F1 désigne la dimension indirecte. - Réglez le gain du récepteur (RG) pour éviter le débordement avec la commande automatique rga.
- Calculez la durée de l’expérience totale à l’aide de la commande expt.
- Envoyez l’expérience pour acquisition via la commande zg.
- Vérifiez toujours que l’expérience se déroule correctement après quelques minutes.
- Préparez l’échantillon avec le complexe protéine-ligand. Utiliser des rapports molaires glycane :lectine compris entre 10:1 et 100:1 avec des concentrations en protéines comprises entre 0,01 et 0,2 mM. Pour l’interaction de la galectine-7 havec la LacNAc, utilisez le rapport protéine :ligand 50:1 dans une solution saline tamponnée au phosphate deutéré à un pH de 7,4.
- Traitement
REMARQUE : Un spectre pseudo-2D est obtenu après application du protocole décrit ci-dessus. Le nombre de rangées correspond au nombre de fréquences utilisées, généralement deux : la résonance activée et la fréquence désactivée.- Traitez le fid pour la première expérience.
- Effectuez la transformée de Fourier du nombre fid 1 (via la commande efp) et sélectionnez la destination des spectres traités (sélectionnez le numéro procno). Vous pouvez également utiliser la commande rser 1 pour lire le premier fid.
- Ensuite, ajustez le facteur d’élargissement de la ligne à l’aide de la commande lb (généralement 3-5 Hz) et de la phase. Pour mettre en phase manuellement, cliquez sur l’onglet Processus, puis sur le sous-menu Ajuster la phase. Effectuez des corrections du zéro et du premier ordre en cliquant et en faisant glisser le bouton correspondant. Enregistrez les résultats de la mise en phase. De plus, effectuez la correction de base à l’aide de la commande abs.
- Lisez le fid de la deuxième expérience, et faites la transformée de Fourier (via la commande efp) avec le même facteur d’élargissement de droite. Ajustez la phase avec les mêmes paramètres de phase et la même correction de base et enregistrez le spectre traité avec un code différent.
- Lisez les deux spectres traités avec la fonction multiple (commande : .md) et soustrayez-les (off-resonance - on-resonance) à l’aide du bouton disponible dans la visualisation multiple (Δ). Le nouveau spectre est le spectre RMN STD, qui est enregistré avec un code différent.
- Effectuez une superposition du spectre RMN STD avec le spectre hors résonance.
- Ouvrez le spectre hors résonance (fid 1) et tapez la commande .md pour ouvrir la fenêtre d’affichage multiple. Ensuite, téléchargez le spectre STD.
- Comparez les fréquences et les intensités (affichées automatiquement en haut à droite) des signaux dans le spectre RMN STD. Cela fournit les informations souhaitées sur les protons qui sont proches de la protéine et leur proximité relative. Plus l’intensité relative est élevée, plus ils sont proches de la protéine (Figure 2).
- Mesurez les intensités (intégrales) dans l’expérience hors résonance à l’aide du logiciel correspondant. Dans TopSpin, allez dans Analyser > Intégrer. Définissez les régions et écrivez les intégrales dans un fichier (I0).
- Mesurez les intensités (intégrales) dans l’expérience RMN STD en utilisant les mêmes paramètres et écrivez-les dans un fichier (ISTD).
- Calculer la valeur STD pour chaque signal de proton à l’aide de l’équation suivante :
STD = (ISTD)/I0.
REMARQUE : REMARQUE : L’utilisation de l’intégration du signal pour calculer les valeurs STD nécessite que les signaux de protons soient suffisamment séparés. Lorsqu’un chevauchement de signal se produit, comme dans le cas d’oligosaccharides, les valeurs STD peuvent être déterminées en évaluant le rapport d’intensité du signal entre les spectres STD et hors résonance. - Calculez la MST relative en pourcentage. Pour ce faire, donnez une valeur de 100% au proton qui présente la différence maximale entre les intensités dans le spectre de la résonance off et le spectre RMN STD. Calculez les intensités STD relatives pour les autres protons en conséquence.
REMARQUE : L’analyse correcte des données STD, en particulier pour déterminer l’épitope de liaison du ligand, nécessite l’attribution complète des signaux 1H du ligand. Par conséquent, il est fortement recommandé d’achever cette tâche avant l’acquisition des spectres STD.
- Traitez le fid pour la première expérience.
2. 1Expériences H-15N HSQC
REMARQUE : Les lignes suivantes détaillent l’utilisation d’expériences HSQC 1 H-15N pour surveiller les changements dans les changements chimiques des résonances RMN 1H et 15N du récepteur (lectine) en réponse à la présence de quantités croissantes du ligand (oligosaccharide)19. L’analyse CSP (Chemical Shift Perturbation) basée sur les données extraites est très précieuse pour l’identification des partenaires de liaison mais aussi pour cartographier l’interface de liaison des protéines et déterminer les affinités de liaison. Pour une compréhension plus approfondie de la conception et de l’acquisition des expériences de RMN, veuillez vous référer au manuel du fabricant correspondant fourni avec l’instrument de RMN.
- Acquisition et traitement
- Préparez l’échantillon avec la lectine qui vous intéresse. Assurez-vous que le récepteur est entièrement marqué au 15N dans chaque résidu d’acide aminé, à la fois dans le squelette et les chaînes latérales. En règle générale, pour détecter les pics croisés HN interchangeables dans l’eau dans le spectre, utilisez un mélange 90:10 de H2O : D2O pour préparer la solution tamponnée. Les concentrations de lectine requises varient entre 0,05 et 0,2 mM, en fonction de la disponibilité du récepteur marqué au 15N et du rapport signal/bruit nécessaire.
REMARQUE : La protéine doit être stable pendant toute la durée de l’expérience sans génération visible de précipité dans le tube RMN. De plus, il doit être pur et soluble dans le tampon sélectionné. L’attribution complète despics croisés HSQC 1 H et 15N doit avoir été effectuée au préalable de manière à ce que chaque pic croisé du spectre HSQC soit identifié par une étiquette correspondant au résidu d’acide aminé spécifique. - À partir de cette préparation, transférez un volume total de 0,6 mL dans un tube RMN de 5 mm.
- Réglez l’instrument RMN à la température requise. Voir l’étape 1.1.3 et suivez les mêmes opérations.
- Créez un nouveau jeu de données. Voir l’étape 1.1.4 et répéter les opérations.
- Insérez l’échantillon RMN dans la sonde comme décrit à l’étape 1.1.5.
- Verrouillez le signal du solvant. Pour démarrer la procédure de verrouillage, utilisez la commande verrouillage et sélectionnez le solvant approprié dans le menu. Le signal de verrouillage peut être tracé dans la fenêtre de verrouillage. Réglez le gain de verrouillage de manière à ce que le signal de verrouillage soit visible dans la fenêtre de verrouillage.
- Terminez le processus de réglage et de correspondance automatiquement (via la commande atma) ou manuellement (la commande atmm ouvrira la fenêtre de contrôle ATM pour ajuster la courbe d’oscillation).
- Définissez les cales optimales à l’aide de l’outil TopShim. Utilisez la commande topshim gui. Voir les instructions à l’étape 1.1.8.
- Déterminez la longueur d’impulsion de 1H à 90° (comme décrit à l’étape 1.1.9) et la fréquence de décalage (la commande o1calib exécutera une routine interactive d’étalonnage O1, récupérant la fréquence de décalage). Ce dernier paramètre est extrêmement important lorsque des expériences avec des schémas de suppression des solvants sont utilisées.
- Créez un nouvel ensemble de données comme décrit à la section 1.1.4. Pour réduire ou éliminer les interférences du signal H2O, utilisez la séquence d’impulsions zgesgp.
- Configurez l’expérience en modifiant divers paramètres dans la fenêtre AcquPars.
- Introduisez la longueur d’impulsion de 1H à 90° et le décalage (o1) comme déterminé précédemment, et réglez le nombre de balayages (NS) à 32 et la fenêtre spectrale (SW) à environ 12 ppm.
- Déterminez le niveau de puissance de l’impulsion façonnée à l’aide de l’outil Forme disponible dans la barre de menu Topspin.
- Réglez le gain du récepteur à l’aide de la commande automatique rga.
- Acquérez l’expérience à l’aide de la commande zg et traitez le FID résultant pour obtenir le spectre RMN 1H.
- Créez un nouvel ensemble de données à utiliser pour l’acquisition de l’expérience RMN 1 H-15N HSQC. Dans l’onglet AcquPars, sélectionnez le programme d’impulsions hsqcetfpf3gp disponible dans le catalogue des programmes d’impulsions.
- Configurez l’expérience. Chargez les formes, les puissances et les temps par défaut à l’aide de la commande getprosol. Ensuite, mettez à jour les valeurs de la longueur d’impulsion et du décalage de 1H à 90°.
- Définissez les paramètres suivants.
- Réglez le délai de relaxation sur 1-5 s.
- Définissez le nombre de numérisations sur un multiple de 4. Habituellement, il est réglé sur 8, 16, 32 ou 64 pour obtenir le bon rapport signal/bruit.
- Définissez le nombre de balayages factices sur 128.
- Définissez le nombre de points dans F2 sur 1k, 2k ou 4k.
- Définissez le nombre de points en F1 : le nombre d’incréments de t1 à utiliser. Selon la fenêtre spectrale, celle-ci se situe entre 128 et 256.
- Ajustez le centre de la fenêtre spectrale dans la dimension 15N sur δ 117 ppm et réglez la largeur spectrale correspondante sur 36 ppm. Ces valeurs doivent être optimisées pour chaque système particulier.
- Réglez le gain du récepteur pour éviter le débordement (à l’aide de la commande rga )
- Calculez la durée de l’expérience totale. Un temps expérimental typique est d’environ 1 h.
- Tapez zg pour envoyer l’expérience pour acquisition.
REMARQUE : vérifiez toujours que l’expérience se déroule correctement après quelques minutes. - Traitez le FID à l’aide de la commande xfb. Effectuez la correction de la ligne de base à l’aide de la commande abs2 et corrections de phase dans l’onglet Processus. Pour mettre en phase manuellement, cliquez sur le sous-menu ajuster la phase, puis sélectionnez plusieurs pics croisés des spectres 2D. Ensuite, appliquez séquentiellement des corrections de zéro et de premier ordre aux lignes et aux colonnes en cliquant et en faisant glisser le bouton correspondant. Enregistrez les résultats de la mise en phase.
- Enregistrez le spectre 2D résultant.
- Préparez une solution mère hautement concentrée du ligand. Les valeurs typiques sont de 50 à 100 mM.
- À partir de la solution mère hautement concentrée du glycane, transférez le volume correspondant (quelques microlitres) dans le tube RMN contenant le récepteur pour obtenir le rapport protéine/ligand molaire souhaité et enregistrer les spectres.
REMARQUE : Cette étape déclenche la série de titrage, où le ligand est titré dans l’échantillon de protéine. Les rapports protéines/ligands appropriés doivent être déterminés pour chaque cas particulier. Si l’affinité de liaison est complètement inconnue, il est recommandé d’utiliser des quantités substœchiométriques du ligand dans les points initiaux. - Effectuez les étapes 2.1.1 à 2.1.19 pour l’échantillon nouvellement préparé.
- Répéter les étapes 2.1.21 et 2.1.22 pour les échantillons dont le rapport protéine/ligand augmente.
REMARQUE : L’ajustement précis des données de la série de titrage nécessite l’acquisition de plusieurs expériences 1 H-15N-HSQC, couvrant une large gamme de rapports protéines/ligands, y compris ceux nécessaires pour atteindre la saturation en protéines.
- Préparez l’échantillon avec la lectine qui vous intéresse. Assurez-vous que le récepteur est entièrement marqué au 15N dans chaque résidu d’acide aminé, à la fois dans le squelette et les chaînes latérales. En règle générale, pour détecter les pics croisés HN interchangeables dans l’eau dans le spectre, utilisez un mélange 90:10 de H2O : D2O pour préparer la solution tamponnée. Les concentrations de lectine requises varient entre 0,05 et 0,2 mM, en fonction de la disponibilité du récepteur marqué au 15N et du rapport signal/bruit nécessaire.
- Analyse
- Visualisez le spectre HSQC 2D traité pour l’espèce apo à l’aide du logiciel approprié : TopSpin, MestReNova et CCPNMR sont tous des programmes adaptés au traitement des données RMN.
REMARQUE : Il s’agit du spectre d’empreintes digitales de la protéine. Les décalages chimiques 1 Het 15N observés dépendent de l’environnement chimique correspondant de chaque acide aminé, qui dépend fortement de la structure 3D de la protéine. Ce spectre est appelé spectre d’empreintes digitales de protéines. Un spectre HSQC 2D 1 H-15N bien dispersé dans lequel tous les pics croisés présentent des intensités uniformes suggère fortement la présence d’une protéine bien repliée19. - Générez la liste des fréquences 1H et 15N pour tous les pics croisés. L’utilisation de logiciels supplémentaires, tels que le programme CCPNMR20, peut faciliter le processus.
- Superposez le spectre du premier ou du second point de titrage à celui de la protéine apo.
- Pour ce faire, ouvrez le spectre 2D correspondant à l’état apo, cliquez sur l’onglet Affichage multiple puis ajoutez le deuxième spectre 2D. L’inspection visuelle des deux spectres fournit des informations sur l’existence d’une interaction entre le ligand et la protéine.
REMARQUE : Du point de vue de la protéine, l’existence de la liaison fournit des changements dans l’environnement chimique des acides aminés directement impliqués dans l’événement de reconnaissance, avec les perturbations concomitantes de déplacement chimique (CSP).
- Pour ce faire, ouvrez le spectre 2D correspondant à l’état apo, cliquez sur l’onglet Affichage multiple puis ajoutez le deuxième spectre 2D. L’inspection visuelle des deux spectres fournit des informations sur l’existence d’une interaction entre le ligand et la protéine.
- Répétez les étapes 2.2.2 et 2.2.3 pour chaque point de titrage, en générant des listes des fréquences 1H et 15N pour tous les pics croisés dans les différents spectres, correspondant à différents rapports molaires protéine-ligand.
REMARQUE : Les décalages chimiques à chaque point de titrage peuvent être mesurés sans qu’il soit nécessaire d’effectuer une nouvelle affectation de pics croisés. Dans le cas d’un régime d’échange rapide, couramment observé dans les interactions lectine :glycane, on peut simplement suivre le mouvement progressif des pics tout au long du titrage. - Vérifiez que, pour le dernier point de titrage, il n’y a pratiquement pas de perturbations de décalage chimique par rapport à l’ajout précédent. Ce fait indique que le site de liaison aux protéines a été saturé par le ligand, qui est en fort excès.
- Calculez les perturbations maximales par déplacement chimique (maxCSP) à l’aide de l’équation ci-dessous :
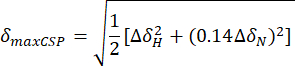
ΔH et ΔδN sont les différences de décalage chimique dans les fréquences 1H et 15N entre l’état apo et le dernier point de titrage, respectivement. - Tracez les perturbations maximales de décalage chimique (maxCSP) dans l’axe vertical des y d’un graphique 2D en fonction du résidu d’acide aminé correspondant (dans l’axe horizontal des x).
- Faites une inspection visuelle des résidus d’acides aminés qui présentent le CSP maximal entre les états lié et apo de la protéine. Il est fort probable qu’ils appartiennent au site de liaison ou qu’ils en soient voisins.
- Si la structure 3D de la protéine est disponible, ouvrez la PDB correspondante avec le logiciel approprié tel que PyMOL ou BIOVIA Discovery studio. Ces programmes de visualisation moléculaire sont largement utilisés dans les applications de biologie structurale. Sélectionnez les résidus qui présentent le maxCSP le plus élevé (au-dessus de deux fois l’écart-type) avec une couleur particulière pour localiser le site de liaison présumé.
- Dans le cas d’un régime d’échange rapide, estimer la constante de dissociation (KD) à partir d’un ajustement non linéaire des moindres carrés de la CSP observée pour les pics croisés HSQC 1 H-15N en chaque point (Δδobs) par rapport à la concentration particulière de protéine [P] et de ligand [L] à ce point :
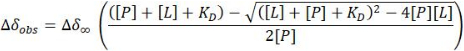
REMARQUE : Cette équation peut être appliquée aux pics croisés qui affichent des signaux isolés clairs. La moyenne des valeurs obtenues est calculée pour fournir l’estimation de KD.
- Visualisez le spectre HSQC 2D traité pour l’espèce apo à l’aide du logiciel approprié : TopSpin, MestReNova et CCPNMR sont tous des programmes adaptés au traitement des données RMN.
Résultats
Nous présentons ici un protocole pour l’exploitation d’expériences 1H-STD RMN et 1 H-15N HSQC afin de démêler les détails de l’interaction de liaison entre les lectines et les petits oligosaccharides. Les résultats obtenus dans l’analyse de la reconnaissance moléculaire de LacNAc par hGalectin-7 (hGal-7) sont inclus, servant d’exemple illustratif de la mise en œuvre réussie du protocole et de l’efficacité de ces méthodologies RMN pour étudier les détails fins du processus de reconnaissance moléculaire. La figure 3 montre le spectre RMN 1H-STD pour l’interaction de LacNAc avec hGal-7. L’existence de signaux RMN STD indique une liaison (Figure 3A). De plus, seuls les signaux appartenant aux protons en contact étroit avec la protéine apparaissent, ce qui permet de délimiter l’épitope de liaison (Figure 3B). La figure 4 met en évidence comment le spectreHSQC 1 H-15N d’une protéine peut être utilisé comme son empreinte digitale, et la figure 5 illustre l’applicationd’expériences de titrage en cohérence quantique unique (HSQC) hétéronucléaire 1 H-15N pour définir la perturbation par décalage chimique des groupes amides du squelette hGalectin-7 lors de la liaison LacNAc. Ces données révèlent non seulement l’existence d’une interaction, mais délimitent également l’interface de liaison de la lectine. La figure 6 montre comment l’analyse des données de titrage permet d’estimer l’affinité de liaison de LacNAc par hGalectine-7, qui se situe dans la gamme des micromolaires élevées. Cette constatation est cohérente avec les résultats obtenus à l’aide d’autres techniques.
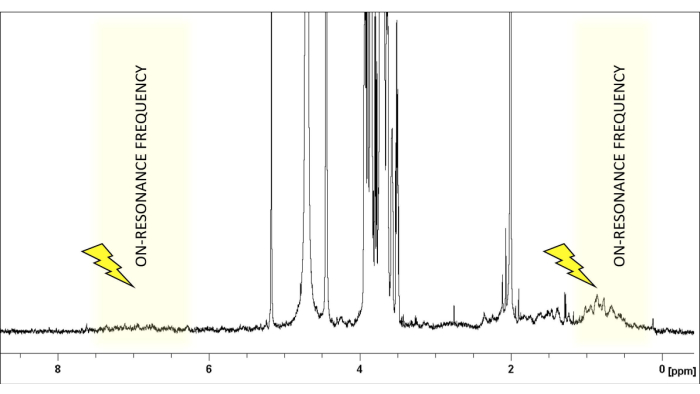
Figure 1 : La sélection de la fréquence de résonance. 1Le spectre H-RMN du rapport LacNAc :hGal-7 50:1 dans une solution saline tamponnée au phosphate deutéré à pH 7,4 est illustré. Les signaux du ligand (LacNAc) sont confinés dans la région entre 2,0 et 5,2 ppm. La fréquence de saturation est soigneusement sélectionnée pour garantir l’absence de protons ligands dans une plage de 1 à 2 ppm, permettant l’irradiation sélective des protons de la protéine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
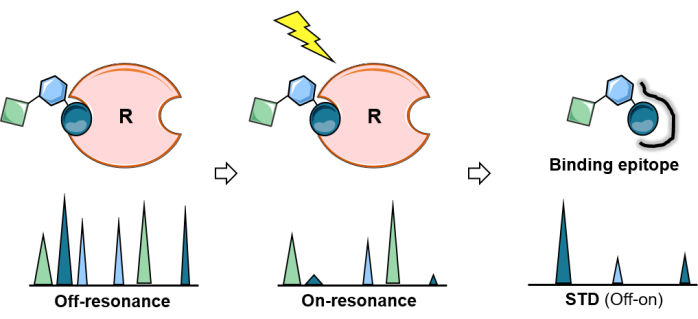
Figure 2 : L’expérience de RMN MST. Représentation schématique de l’expérience STD : le premier spectre (off-resonance) sert de référence tandis que dans le second (on-resonance), la saturation en protéines est effectuée. La saturation se propage efficacement sur l’ensemble de la protéine et est transférée aux protons du ligand en contact étroit avec la protéine. Le spectre de différence résultant (spectre STD) ne produit que les résonances qui ont connu la saturation. L’analyse de l’expérience MST permet la cartographie des épitopes du sucre de liaison. Veuillez cliquer ici pour voir une version agrandie de cette figure.
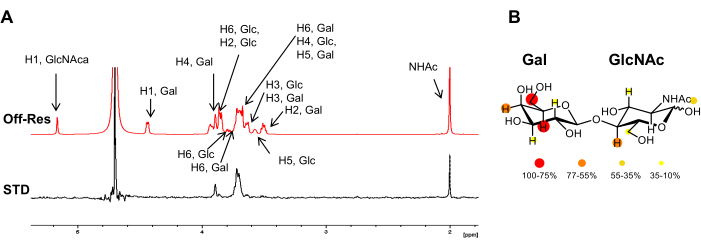
Figure 3 : Analyse de liaison du point de vue du ligand. (A) Superposition des spectres hors résonance et 1H STD-NMR pour l’interaction de LacNAc avec hGal-7. Dans le spectre STD, seuls les signaux appartenant aux protons en contact étroit avec la protéine apparaissent. L’annotation des résonances 1H du ligand est rapportée dans le spectre hors résonance. (B) Les intensités relatives des MST ont été cartographiées en couleur dans la structure chimique de LacNAc. Veuillez cliquer ici pour voir une version agrandie de cette figure.
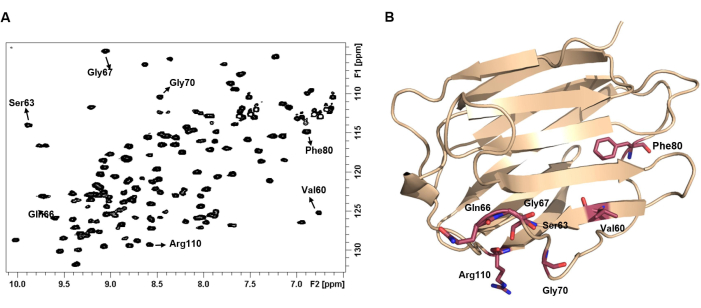
Figure 4 : Le spectre HSQC 1 H-15N d’une protéine représente son empreinte. (A) 1spectre HSQC H-15N de 100 μM de hGal-7 sous forme apo. Le spectre a été enregistré à 25 °C. Certains pics transversaux NH ont été annotés avec l’étiquette de leur acide aminé correspondant. (B) Chaque paire NH présente un décalage chimique unique qui dépend de l’environnement chimique et, par conséquent, de la structure 3D de la protéine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
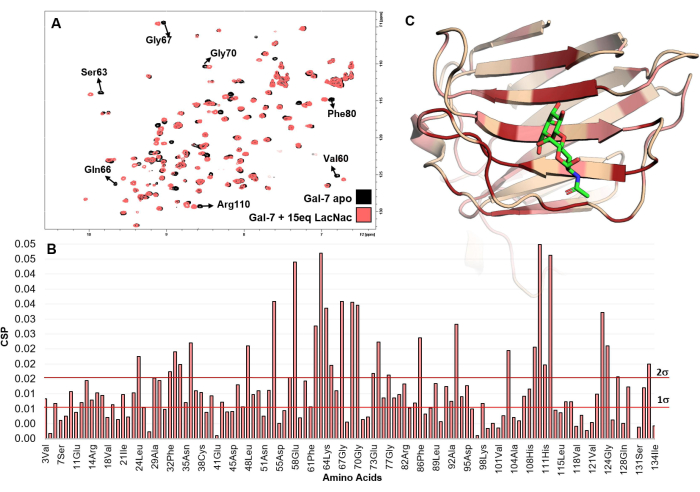
Figure 5 : Analyse de liaison du point de vue de la protéine. (A) La superposition des spectres HSQC1 H-15N enregistrés pour le titrage de LacNAc dans une solution hGal-7 est montrée. L’inspection des spectres, où plusieurs pics croisés subissent des changements de décalage chimique, indique clairement une interaction. (B) Le tracé des perturbations de décalage chimique maximal (maxCSP) des signaux amides de squelette déduits du titrage de LacNAc (15 équivalents) avec hGal-7. (C) Les acides aminés les plus perturbés de hGal-7, selon l’analyse CSP, sont cartographiés dans la structure PDB 5gal. Dans le modèle 3D, la coloration rouge fait référence à une valeur CSP supérieure à 2σ, tandis que les roses à des valeurs comprises entre 1σ et 2σ. La région colorée représente probablement le site de liaison. Veuillez cliquer ici pour voir une version agrandie de cette figure.
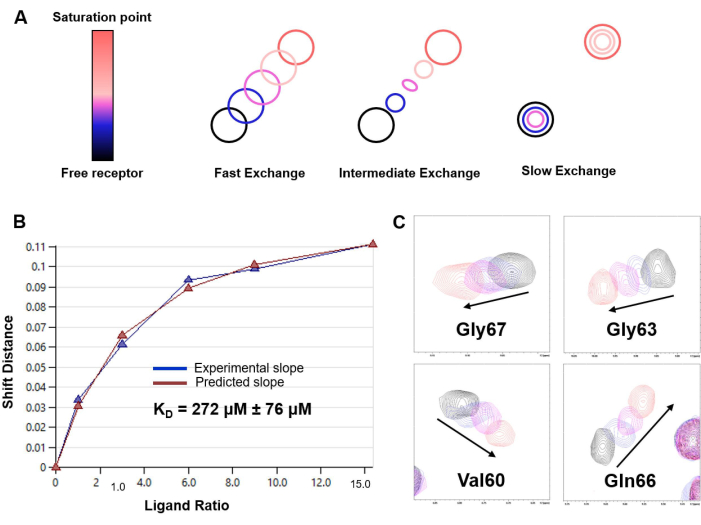
Figure 6 : Détermination de KD basée sur des expériences de titrage 1 H-15N HSQC. (A) Représentation du modèle de titrage 1 H-15N basé sur HSQC en fonction du taux d’échange chimique dans l’échelle de temps RMN du système dans l’étude (rapide, intermédiaire ou lent). Un régime d’échange rapide a été observé dans le cas de l’interaction LacNAc/hGal-7. (B) Courbe d’ajustement et estimationde K D obtenues à partir de l’analyse CSP à différentes concentrations de ligands pour le système modèle de hGal-7 et de disaccharide LacNAc. Le KD estimé est rapporté avec l’erreur correspondante comme une moyenne des données pour 20 acides aminés différents ; (C) Extraits des spectres 1 H,15N-HSQC affichant le décalage des pics croisés sélectionnés pendant le titrage. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
La RMN par différence de transfert de saturation (STD-NMR) est devenue la méthode RMN la plus utilisée et la plus polyvalente pour étudier les interactions ligand-protéine. Comme montré ci-dessus, il repose sur le phénomène de transfert de saturation, et le dispositif expérimental implique l’acquisition de deux spectres unidimensionnels (1D) 1H : le spectre on-resonance et le spectre off-resonance. Au cours de l’expérience sur résonance, la saturation de protons spécifiques de la protéine est obtenue en appliquant un train d’impulsions radiofréquences de faible puissance pendant une certaine période (le temps de saturation varie généralement de 1 à 3 s). Pour éviter la saturation directe du ligand, la fréquence et la longueur des impulsions de saturation sont optimisées pour irradier sélectivement des protons spécifiques de la protéine ; c’est-à-dire qu’ils doivent être appliqués à une fréquence dépourvue de tout signal ligand et avec une longueur appropriée (Figure 1). En règle générale, pour les impulsions de saturation de 50 ms, une différence de 1 ppm doit être maintenue entre la région de saturation et les signaux ligands les plus proches. Généralement, des impulsions de saturation sélectives appliquées sur la région aliphatique de la protéine fournissent des effets de saturation accrus. Alternativement, les protons aromatiques (6-7 ppm) peuvent également être irradiés si la molécule de ligand ne contient aucun signal aromatique. Ceci est très utile pour les glycanes naturels, car ils ne portent pas de groupes aromatiques. Une fois qu’une certaine région de la protéine est irradiée sélectivement, la saturation se propage le long de la protéine via une relaxation croisée dipolaire 1 H-1H (diffusion de spin). Finalement, la saturation atteint les protons protéiques au site de liaison, qui sont ensuite transférés aux protons de sucre qui sont en contact étroit (r < 5 Å) avec le récepteur via les NOE intermoléculaires 1 H-1H. De toute évidence, l’intensité des signaux des protons du ligand saturé diminue. Après avoir reçu la saturation, en raison de la cinétique de liaison, les ligands liés transitoirement (un échange rapide est nécessaire) se dissocient et l’information de saturation s’accumule à l’état libre. En raison de ce processus, les spectres RMN en résonance présentent des signaux diminués (Figure 2).
Pour mettre clairement en évidence cette perturbation d’intensité des noyaux 1H d’un glycane de liaison, un spectre RMN de proton de contrôle (off-resonance) est acquis dans lequel la saturation est appliquée loin de tout récepteur ou signal glucidique (généralement entre 40 et 100 ppm), dans les mêmes conditions. Le spectre 1D soustrait entre la résonance off et la résonance on montre exclusivement les signaux des noyaux 1H du ligand qui ont des intensités modifiées : ceux qui étaient suffisamment proches du site de liaison du récepteur pour recevoir l’aimantation (Figure 2).
Néanmoins, tous les noyaux 1H du glucide lié ne reçoivent pas la même quantité de saturation. Théoriquement, le transfert d’aimantation du récepteur au ligand lié dépend de la distance (1/r6). Cela signifie que les intensités de saturation transférée entre les noyaux de glycane 1H contiennent des informations sur les proximités spatiales entre les protons du ligand et ceux du récepteur, et que les intensités RMN STD sont plus grandes pour les protons qui sont plus proches du récepteur. En conséquence, l’expérience RMN STD permet également de déterminer l’épitope de liaison du glucide (Figure 2 et Figure 3) puisque les protons du ligand situés plus près de la surface de la protéine présentent des intensités plus élevées que ceux qui ne participent pas directement à la liaison.
L’expérience peut être appliquée à des systèmes d’affinité faible-moyenne, rarement à des systèmes à fortes affinités dans la gamme des μM ou nM. En effet, il faut que le taux de dissociation soit rapide dans l’échelle du temps de relaxation. Sinon, l’information de transfert de saturation est perdue par relaxation avant que le ligand ne se dissocie.
D’autre part, les expériences de RMN basées sur les protéines sont uniques pour démêler l’interaction ligand-protéine avec une précision au niveau des acides aminés sans résoudre les structures de résolution atomique. Il examine directement les phénomènes de reconnaissance moléculaire en solution sans avoir besoin de co-cristallisation. La cartographie d’analyse CSP est exceptionnellement puissante pour découvrir des ligands et cartographier le site de liaison aux protéines (Figure 4 et Figure 5). Cette méthode est applicable à toute gamme d’affinités entre les gammes mM et nM, même pour les systèmes où le taux de change est lent sur l’échelle de temps de décalage chimique21.
Néanmoins, cette approche ne fonctionnera probablement pas pour les protéines dont le poids moléculaire est supérieur à 30-40 kDa en raison de problèmes de relaxation. L’alternative TROSY18 peut alors être utilisée, étant particulièrement puissante lorsqu’elle est couplée à la deutération des protéines. De plus, la protéine doit être uniformément marquée avec 15N (et un autre échantillon marqué deux fois avec 13C et 15N pour pouvoir compléter l’affectation de base requise). Par conséquent, les conditions d’expression des protéines, y compris le système d’expression correspondant, doivent être optimisées pour pouvoir obtenir des quantités de milligrammes de protéines. Les protéines qui présentent une tendance à l’oligomérisation ou à l’agrégation ne conviennent pas non plus à cette analyse. L’instrument utilisé ici pour enregistrer les données RMN est un spectromètre Bruker 800 MHz équipé d’une cryosonde TCI. Il serait très difficile d’utiliser cette méthodologie avec des instruments inférieurs à 600 MHz ou sans sonde cryogénique.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions l’Agencia Estatal de Investigación d’Espagne pour l’accréditation du Centre d’excellence Severo Ochoa CEX2021-001136-S, financée par MCIN/AEI/10.13039/ 501100011033, et CIBERES, une initiative de l’Instituto de Salud Carlos III (ISCIII, Madrid, Espagne). Nous remercions également la Commission européenne pour le projet GLYCOTWINNING.
matériels
| Name | Company | Catalog Number | Comments |
| 5 mm Shigemi microtube set mat | CortecNet SAS | S30BMS-005B | |
| Alpha-Lactose-Agarose | Sigma-Aldrich Química S.L. | 7634-5ML | |
| Ammonium chloride (15 N, 99%) | LC-0179-N-50G | Tracer Tecnologías Analíticas S.L | |
| Ampicillin (Sodium Salt) | Melford Laboratories LTD | A40040 | |
| BIOVIA Discovery studio | BIOVIA, Dassault Systèmes | ||
| BL21(DE3) Chemically Competent Cells | Merck Life Science, S.L.U. | CMC0014-40X40UL | |
| Centrifuge | Beckman Coulter | Allegra X-22R | |
| D2O | Cambridge Isotope Laboratories, Inc. | DLM-4-1000 | |
| Incubator | Eppendorf | Innova 42 | |
| IPTG (Isopropyl ß-D-1-thiogalactopyranoside) | VWR International Eurolab S.L. | VW437144N | |
| LacNAc | Elicityl | GLY008 | |
| Luria Bertani (LB) Broth | Merck Life Science, S.L.U. | 3397-1KG | |
| Matraz Erlenmeyer B N 5000 CC | VWR International Eurolab S.L. | 214-1137 | |
| PBS 10x | Bio-Rad | 1610780 | |
| PyMOL | PyMOL Molecular Graphics System | Version 2.0 Schrödinger | |
| Sonicator | Sonics & Materials, Inc. | VC 505 | |
| Superconducting NMR magnet | Bruker | 600 MHz AVANCE III | |
| Superconducting NMR magnet | Bruker | 800 MHz AVANCE III |
Références
- Solís, D., et al. A guide into glycosciences: How chemistry, biochemistry and biology cooperate to crack the sugar code. Biochim Biophys Acta. 1850 (1), 186-235 (2015).
- Poveda, A., Jimenez-Barbero, J. NMR studies of carbohydrate-protein interactions in solution. Chem Soc Rev. 27, 133-144 (1998).
- Kogelberg, H., Solís, D., Jiménez-Barbero, J. New structural insights into carbohydrate-protein interactions from NMR spectroscopy. Curr Opin Struct Biol. 13 (5), 646-653 (2003).
- Widmalm, G. Glycan shape, motions, and interactions explored by NMR spectroscopy. JACS Au. 4 (1), 20-39 (2024).
- Marchetti, R., et al. 34;Rules of Engagement" of protein-glycoconjugate interactions: A molecular view achievable by using NMR spectroscopy and molecular modeling. ChemistryOpen. 5 (4), 274-296 (2016).
- Ardá, A., Jiménez-Barbero, J. The recognition of glycans by protein receptors. Insights from NMR spectroscopy. Chem Commun. 54 (38), 4761-4769 (2018).
- Gimeno, A., Valverde, P., Ardá, A., Jiménez-Barbero, J. Glycan structures and their interactions with proteins. A NMR view. Curr Opin Struct Biol. 62, 22-30 (2020).
- Cañada, F. J., et al. Conformational and structural characterization of carbohydrates and their interactions studied by NMR. Curr Med Chem. 29 (7), 1147-1172 (2022).
- Valverde, P., Quintana, J. I., Santos, J. I., Ardá, A., Jiménez-Barbero, J. Novel NMR avenues to explore the conformation and interactions of glycans. ACS Omega. 4, 13618-13630 (2019).
- Jiménez-Barbero, J., Asensio, J. L., Cañada, F. J., Poveda, A. Free and protein-bound carbohydrate structures. Curr Opin Struct Biol. 9 (5), 549-555 (1999).
- Quintana, J. I., Atxabal, U., Unione, L., Ardá, A., Jiménez-Barbero, J. Exploring multivalent carbohydrate-protein interactions by NMR. Chem Soc Rev. 52 (5), 1591-1613 (2023).
- Roldós, V., Cañada, F. J., Jiménez-Barbero, J. Carbohydrate-protein interactions: a 3D view by NMR. Chembiochem. 12 (7), 990-1005 (2011).
- Unione, L., Ardá, A., Jiménez-Barbero, J., Millet, O. NMR of glycoproteins: profiling, structure, conformation, and interactions. Curr Opin Struct Biol. 68, 9-17 (2021).
- del Carmen Fernández-Alonso, M., Díaz, D., Berbis, M. A., Marcelo, F., Cañada, J., Jiménez-Barbero, J. Protein-carbohydrate interactions studied by NMR: from molecular recognition to drug design. Curr Protein Pept Sci. 13 (8), 816-830 (2012).
- Angulo, J., Nieto, P. M. STD-NMR: application to transient interactions between biomolecules-a quantitative approach. Eur Biophys J. 40 (12), 1357-1369 (2012).
- Mayer, M., Meyer, B. Group epitope mapping by saturation transfer difference NMR to identify segments of a ligand in direct contact with a protein receptor. J Am Chem Soc. 123 (25), 6108-6117 (2001).
- Williamson, M. P. Using chemical shift perturbation to characterise ligand binding. Prog Nucl Magn Reson Spectrosc. 73, 1-16 (2013).
- Fernández, C., Adeishvili, K., Wüthrich, K. Transverse relaxation-optimized NMR spectroscopy with the outer membrane protein OmpX in dihexanoyl phosphatidylcholine micelles. Proc Natl Acad Sci U S A. 98 (5), 2358-2363 (2001).
- Yee, A., Gutmanas, A., Arrowsmith, C. H. Solution NMR in structural genomics. Curr Opin Struct Biol. 16 (5), 611-617 (2006).
- Skinner, S. P., Fogh, R. H., Boucher, W., Ragan, T. J., Mureddu, L. G., Vuister, G. W. CCPNMR AnalysisAssign: a flexible platform for integrated NMR analysis. J Biomol NMR. 66 (2), 111-124 (2016).
- Gimeno, A., et al. Minimizing the entropy penalty for ligand binding: Lessons from the molecular recognition of the histo blood-group antigens by human Galectin-3. Angew Chem Int Ed Engl. 58 (22), 7268-7272 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon