Method Article
Districare le interazioni glicano-proteina: la risonanza magnetica nucleare (NMR) in soccorso
In questo articolo
Riepilogo
Qui, presentiamo un protocollo che descrive in dettaglio l'acquisizione, l'elaborazione e l'analisi di una serie di esperimenti NMR volti a caratterizzare le interazioni proteina-glicano in soluzione. Vengono descritte le metodologie più comuni basate su ligandi e proteine, che indubbiamente contribuiscono ai campi della glicobiologia strutturale e degli studi di riconoscimento molecolare.
Abstract
Le interazioni dei glicani con le proteine modulano molti eventi legati alla salute e alla malattia. Infatti, l'instaurarsi di questi eventi di riconoscimento e le loro conseguenze biologiche sono intimamente correlate alle strutture tridimensionali di entrambi i partner, nonché alle loro caratteristiche dinamiche e alla loro presentazione sui corrispondenti compartimenti cellulari. Le tecniche NMR sono uniche per districare queste caratteristiche e, in effetti, sono state sviluppate e applicate diverse metodologie basate sulla NMR per monitorare gli eventi di legame dei glicani con i loro recettori associati. Questo protocollo delinea le procedure per acquisire, elaborare e analizzare due delle più potenti metodologie NMR impiegate nel campo della glicobiologia NMR, gli esperimenti di titolazione 1H-Saturation transfer difference (STD) e 1 H,15 N,15N-Eteronuclear single quantum coherence (HSQC), che offrono informazioni complementari rispettivamente dal punto di vista del glicano e della proteina. Infatti, quando combinati offrono un potente kit di strumenti per chiarire sia gli aspetti strutturali che dinamici dei processi di riconoscimento molecolare. Questo approccio globale migliora la nostra comprensione delle interazioni glicano-proteina e contribuisce a far progredire la ricerca nel campo della glicobiologia chimica.
Introduzione
Il riconoscimento molecolare dei glicani è essenziale per molti processi legati alla salute e alla malattia. La specificità e la selettività dei recettori biologici (lectine, anticorpi, enzimi) per i glicani dipendono fortemente dalla regolazione del precario equilibrio tra i diversi componenti dell'entalpia (CH-π e van der Waals, legami idrogeno, elettrostatica) e dell'entropia (idrofobicità, dinamica, solvatazione-desolvatazione)1.
Data la grande diversità chimica e la natura dinamica dei glicani, i metodi NMR sono stati ampiamente impiegati per sezionare le interazioni dei glicani per più di 25 anni2, poiché queste metodologie forniscono informazioni eccellenti sugli eventi di riconoscimento molecolare con dettagli precisi, alla risoluzione atomica 3,4, anche quando l'evidenza di interazione richiesta non può essere recuperata impiegando altre metodologie. Come punto chiave, la risonanza magnetica nucleare è versatile e consente di studiare eventi dinamici, a livello atomico, a diverse scale temporali, costituendo di gran lunga la tecnica migliore per studiare la struttura, la conformazione e la dinamica dei glicani in soluzione. Tuttavia, districare queste informazioni può essere un processo piuttosto complesso che richiede l'impiego di strategie ben definite insieme a un'attenta analisi dei dati5.
Le tecniche NMR sono diverse e, in effetti, ci sono molte metodologie che possono essere impiegate per svelare le interazioni glicano-proteina6. Di seguito descriviamo due approcci NMR di base che sono attualmente impiegati per decifrare le interazioni glicano-recettore 7,8, ponendo l'accento su come districare la presentazione dell'epitopo chiave del glicano e del sito di legame della proteina9.
In qualsiasi evento di riconoscimento molecolare, quando un recettore si lega a un dato ligando, si verifica un processo di scambio chimico che influenza molti parametri NMR dei partecipanti al legame10. Pertanto, dal punto di vista della RMN, l'interazione può essere monitorata sia dal punto di vista del ligando del glicano che da quello del recettore proteico11. In generale, il recettore proteico è una biomolecola di grandi dimensioni (movimento rotatorio lento, con velocità nella scala temporale ns e, quindi, rilassamento trasversale rapido), mentre il glicano interagente può essere considerato come una molecola di dimensioni medio-piccole (movimento rotatorio veloce, con velocità nella scala temporale ps e rilassamento trasversale lento)12. Da una prospettiva standard, i segnali NMR del glicano sono stretti, mentre quelli del recettore sono ampi13.
I metodi NMR basati su ligandi si basano sul drastico cambiamento che molti parametri NMR dei glicani sperimentano quando passano dallo stato libero a quello legato14. STD-NMR è la tecnica NMR sperimentale più impiegata per valutare diverse caratteristiche di legame dei glicani15, dalla deduzione dell'esistenza di legame nello stato di soluzione alla determinazione dell'epitopo di legame dei glicani; cioè, gli atomi del ligando che sono in contatto con il recettore proteico16.
In alternativa, i metodi NMR basati su recettori monitorano i cambiamenti che avvengono nei segnali del recettore proteico in presenza del glicano rispetto a quelli registrati per lo stato apo17. Questi si concentrano principalmente sullo screening delle perturbazioni di spostamento chimico dei segnali proteici tra i due stati. L'esperimento più comunemente impiegato è 1 H-15N HSQC, o le sue alternative TROSY18.
La combinazione di entrambi gli approcci consente di applicare la risonanza magnetica mobile a molti sistemi diversi che mostrano un'ampia gamma di affinità. Tuttavia, per i metodi NMR basati su recettori, a differenza di quelli basati sul ligando, deve essere disponibile una quantità relativamente grande di proteina solubile, non aggregata, marcata con isotopi stabili (15N).
Descriviamo qui entrambi i metodi, evidenziandone i punti di forza e di debolezza. Si noti che i passaggi di base descritti nel protocollo servono come esempi per l'uso degli spettrometri Bruker. Di conseguenza, i nomi dei comandi e dei parametri sono in linea con quelli utilizzati in TopSpin (il software di controllo degli spettrometri di Bruker).
Protocollo
1. Differenza di trasferimento di saturazione NMR (STD-NMR)
NOTA: Le righe successive delineano le procedure fondamentali per acquisire, elaborare e analizzare gli esperimenti STD-NMR. Questi passaggi servono a esemplificare l'utilità della tecnica per rilevare il legame del ligando e per chiarire l'epitopo del legame del ligando. Per una comprensione più approfondita della progettazione e dell'acquisizione degli esperimenti NMR, fare riferimento al manuale del produttore corrispondente fornito con lo strumento NMR.
- Acquisizione
- Preparare il campione con il complesso proteina-ligando. Impiega rapporti molari glicano:lectina compresi tra 10:1 e 100:1 con concentrazioni proteiche comprese tra 0,01 e 0,2 mM. Per l'interazione di hGalectina-7 con LacNAc, utilizzare il rapporto proteina/ligando 50:1 in soluzione salina tamponata con fosfato deuterato a pH 7,4.
NOTA: Il recettore proteico deve essere puro ed essere solubile nel tampone prescelto (nel caso di esperimenti STD-NMR, sono preferibili versioni deuterate del tampone corrispondente per ridurre la possibile interferenza del segnale NMR di 1H). La concentrazione della proteina viene controllata in anticipo utilizzando uno spettrofotometro per misurare l'assorbanza a 280 nm. - Dalla soluzione preparata, trasferire un volume totale di 0,6 mL in una provetta NMR da 5 mm utilizzando una pipetta.
- Preparare lo strumento NMR alla temperatura richiesta (le temperature comuni degli esperimenti scendono tra 10 °C e 45 °C). Aprire il monitor di controllo della temperatura utilizzando il comando edte e impostare la temperatura desiderata. Per lo studio hGalectin-7/LacNAc, la temperatura è stata impostata a 25 °C.
- Generare un nuovo set di dati contenente la sequenza di impulsi zg.
- Per un'operazione semplice, aprire un esperimento esistente e digitare il comando edc . Viene visualizzata una finestra di dialogo che definisce il titolo, le caratteristiche (specifiche del campione, solvente) e alcuni parametri dell'esperimento.
- Se è necessaria una modifica rispetto alla sequenza di impulsi originale, navigare tra le finestre ased (parametri) e AcquPars (parametri di acquisizione). A questo punto, scegliere il programma di impulsi desiderato dalla libreria dello spettrometro.
- Per uno spettro NMR standard di 1H, selezionare la sequenza di impulsi zg dall'elenco disponibile.
NOTA: Nel caso di campioni con un elevato contenuto d'acqua, potrebbe essere necessario l'uso di schemi di soppressione dell'acqua per aumentare il rapporto segnale/rumore. L'uso di sequenze di impulsi come zgesgp, che scolpiscono l'eccitazione in moduli che rendono eccellente la soppressione ma controllano la fase dei segnali rimanenti, è auspicabile. Si prega di fare riferimento al tutorial NMR dei produttori per ulteriori informazioni sui tipi di schemi di soppressione dell'acqua e sulle loro caratteristiche principali.
- Inserire il campione NMR nella sonda attivando l'aria di sollevamento del campione. Utilizzare il comando ej, posizionare il campione sulla parte superiore del magnete e disattivare il sollevamento del campione utilizzando il comando ij.
NOTA: Per iniettare il campione nel magnete utilizzando un autocampionatore, utilizzare il comando sx seguito dal numero di posizione, n, corrispondente alla posizione del tubo NMR nel vassoio dell'autocampionatore. - Bloccare il segnale del solvente digitando il comando lock e quindi selezionando il solvente appropriato dal menu.
- Una volta inserito il campione nella sonda, completare il processo di sintonizzazione e abbinamento utilizzando il modulo automatico atma o il modulo manuale atmm.
- Avvia lo shimming automatico tramite il comando topshim gui. Si aprirà un'interfaccia grafica in cui verrà selezionata e avviata la quota di spessore 1D.
NOTA: Per ridurre al minimo le instabilità di spessoramento dovute a sottili variazioni di campo o di temperatura, l'autoshim può essere attivato per l'acquisizione dell'esperimento. Questa operazione può essere eseguita accedendo alla finestra di controllo BSMS e facendo clic su autoshim. La trasformazione in un'evidenziazione verde indica che lo spessore automatico è stato attivato. Si prega di notare che quando si utilizza l'autoshim, i potenziali problemi di instabilità del campione devono essere ignorati. Pertanto, si consiglia cautela quando si utilizza l'autoshim. - Determinare il polso 1H 90°. Questa operazione può essere eseguita automaticamente tramite il comando pulsato .
- Modificare vari parametri nella finestra AcquPars. Per uno spettro NMR regolare di 1H, impostare il numero di scansioni (NS) a 32 e la finestra spettrale (SW) desiderata a ca. 12 pagine al minuto.
NOTA: La sequenza di impulsi zgesgp include un modulo per la soppressione del solvente per eliminare il segnale HDO residuo, che deve essere centrato al centro dello spettro. A tale scopo, l'O1 deve essere definito con precisione in AcquPars. - Impostare il guadagno del ricevitore per evitare l'overflow con il comando automatico rga.
- A questo punto, acquisire lo spettro NMR standard 1H utilizzando il comando zg.
- Una volta terminata l'acquisizione, elaborare lo spettro tramite il comando efp. Applicate le correzioni di base e di fase utilizzando la barra dei menu TopSpin.
NOTA: Si osservano segnali NMR a 1ora derivanti dal glicano e dalla proteina (Figura 1). L'analisi dettagliata dello spettro NMR acquisito è raccomandata per l'implementazione dell'esperimento NMR STD, come descritto nella sezione 1.1.14. - Creare un nuovo set di dati e caricare la sequenza di impulsi STD NMR da utilizzare nello stesso modo descritto perl'esperimento 1 H NMR nella sezione 1.1.4. Negli strumenti Bruker, nel catalogo dei programmi a impulsi sono disponibili diverse sequenze di impulsi, tutte denominate stddiffXXX. Il più semplice (stddiff) non include alcuno schema di soppressione dell'acqua o filtro di soppressione delle proteine.
- Per i campioni con un contenuto significativo di H2O, selezionare le sequenze stddiffgp19 o stddiffesgp, che includono un modulo di scultura a watergate o ad eccitazione. Nel caso di uno spettro con intensi segnali NMR proteici come sfondo, selezionare le sequenze stddiffXXX.3. In ogni caso, ottimizzare i parametri specifici corrispondenti per ciascun modulo di soppressione dell'acqua (ad esempio, d19 negli schemi watergate).
- Definire le frequenze di risonanza off e on per l'esperimento STD NMR. Trovare l'elenco delle frequenze nei parametri AcquPars della finestra ased sotto la voce FQ2LIST. Le frequenze di risonanza on e off definite in Hertz devono essere scritte manualmente nell'elenco e salvate con un nuovo nome. Questo nuovo elenco sarà utilizzato nell'esperimento STD-NMR.
- Scegliere la frequenza di risonanza in una regione spettrale priva di segnali di glicani, di solito intorno a δ(1H) 0 o 6,6 ppm, per i glicani tipici (Figura 1). Impostare la frequenza di fuori risonanza in una regione che non mostra alcun ligando o protone proteico. Può essere impostato in sicurezza a +18000 o -18000 Hz.
- Definire l'impulso sagomato da utilizzare durante il tempo di saturazione nei parametri AcquPars della finestra ased .
NOTA: Ci sono molte possibilità. Le forme gaussiane o Eburp possono essere impiegate in sicurezza, con un'ampiezza di 90° dell'impulso selettivo di 50 ms. - Impostare i parametri corrispondenti nella sezione AcquPars.
- Impostare la lunghezza dell'impulso di 1ora e 90°.
- Impostare il valore di potenza per l'impulso sagomato (stimato tramite lo strumento forma).
- Impostare il tempo di saturazione totale. Valori compresi tra 1 s e 4 s possono essere utilizzati regolarmente.
- Impostare il ritardo di rilassamento su 3 s.
- Impostare il numero di scansioni (NS) su un multiplo di 8. Di solito, è impostato su 256, 512 o 1024 per ottenere il corretto rapporto segnale/rumore in set di 2 a ciascuna frequenza.
- Impostare il numero di scansioni fittizie (DS) su 8.
- Imposta il numero di punti in F2 su 16k, 32k o 64k.
NOTA: Un numero maggiore di punti in F2 comporterà un miglioramento della risoluzione e del rapporto segnale/rumore. Per questo motivo, è fortemente consigliabile utilizzare un minimo di 16k punti dati. - Imposta il numero di punti in F1. Questo è il numero di frequenze da utilizzare, in questo caso, 2 (la risonanza on e la off-resonanza).
NOTA: Per convenzione, F2 si riferisce alla dimensione diretta, la dimensione lungo la quale il decadimento a induzione libera (FID) viene campionato direttamente, mentre F1 denota la dimensione indiretta. - Impostare il guadagno del ricevitore (RG) per evitare l'overflow con il comando automatico rga.
- Calcola il tempo dell'esperimento totale utilizzando il comando expt.
- Inviare l'esperimento per l'acquisizione tramite il comando zg.
- Verificare sempre che l'esperimento venga eseguito correttamente dopo alcuni minuti.
- Preparare il campione con il complesso proteina-ligando. Impiega rapporti molari glicano:lectina compresi tra 10:1 e 100:1 con concentrazioni proteiche comprese tra 0,01 e 0,2 mM. Per l'interazione di hGalectina-7 con LacNAc, utilizzare il rapporto proteina/ligando 50:1 in soluzione salina tamponata con fosfato deuterato a pH 7,4.
- Elaborazione
NOTA: Uno spettro pseudo-2D si ottiene dopo aver applicato il protocollo sopra descritto. Il numero di file corrisponde al numero di frequenze impiegate, tipicamente due: la risonanza on e la off-resonanza.- Elaborare il fid per il primo esperimento.
- Effettuare la trasformata di Fourier del numero fid 1 (tramite il comando efp) e selezionare la destinazione degli spettri elaborati (selezionare il numero procno). In alternativa, utilizzare il comando rser 1 per leggere il primo fid.
- Quindi, regolare il fattore di allargamento della linea tramite il comando lb (di solito 3-5 Hz) e la fase. Per eseguire manualmente la fase, fare clic sulla scheda Processo e quindi regolare il sottomenu della fase . Esegui correzioni zero e del primo ordine facendo clic e trascinando sul pulsante corrispondente. Salvare i risultati della gradualità. Inoltre, eseguire la correzione della linea di base tramite il comando abs.
- Leggere il fid per il secondo esperimento, e fare la trasformata di Fourier (attraverso il comando efp) con lo stesso fattore di allargamento della linea. Regola la fase con gli stessi parametri di fase e la correzione della linea di base e salva lo spettro elaborato con un codice diverso.
- Leggere i due spettri elaborati con la funzione multipla (comando: .md) e sottrarli (off-risonanza - on-risonanza) utilizzando il pulsante disponibile nella visualizzazione multipla (Δ). Il nuovo spettro è lo spettro STD NMR, che viene salvato con un codice diverso.
- Sovrapporre lo spettro STD NMR con lo spettro fuori risonanza.
- Aprire lo spettro di off-resonance (fid 1) e digitare il comando .md per aprire la finestra di visualizzazione multipla. Quindi, carica lo spettro STD.
- Confronta le frequenze e le intensità (visualizzate automaticamente in alto a destra) dei segnali nello spettro STD NMR. Questo fornisce le informazioni desiderate su quei protoni che sono vicini alla proteina e la loro relativa vicinanza. Maggiore è l'intensità relativa, più sono vicini alla proteina (Figura 2).
- Misurare le intensità (integrali) nell'esperimento di off-risonanza utilizzando il software corrispondente. In TopSpin, vai su Analizza > Integra. Definisci le regioni e scrivi gli integrali in un file (I0).
- Misurare le intensità (integrali) nell'esperimento STD NMR utilizzando gli stessi parametri e scriverli in un file (ISTD).
- Calcola il valore STD per ogni segnale di protone utilizzando la seguente equazione:
STD = (ISTD)/I0.
NOTA: NOTA: L'uso dell'integrazione del segnale per il calcolo dei valori STD richiede che i segnali protonici siano sufficientemente separati. Quando si verifica una sovrapposizione del segnale, come nel caso degli oligosaccaridi, i valori STD possono essere determinati valutando il rapporto di intensità del segnale tra gli spettri STD e quelli fuori risonanza. - Calcola l'STD relativo in percentuale. Per fare ciò, dare un valore del 100% al protone che mostra la massima differenza tra le intensità nella fuori risonanza e lo spettro NMR STD. Calcola di conseguenza le intensità STD relative per gli altri protoni.
NOTA: Una corretta analisi dei dati delle malattie sessualmente trasmissibili, in particolare per determinare l'epitopo di legame del ligando, richiede l'assegnazione completa di segnali 1H del ligando. Pertanto, si raccomanda vivamente di completare questa attività prima dell'acquisizione degli spettri STD.
- Elaborare il fid per il primo esperimento.
2. 1Esperimenti H-15N HSQC
NOTA: Le righe seguenti descrivono in dettaglio l'impiego di esperimentiHSQC 1 H-15N per monitorare i cambiamenti negli spostamenti chimici delle risonanze NMR 1H e 15N del recettore (lectina) in risposta alla presenza di quantità crescenti del ligando (oligosaccaride)19. L'analisi Chemical Shift Perturbation (CSP) basata sui dati estratti è molto preziosa per l'identificazione dei partner di legame, ma anche per mappare l'interfaccia di legame della proteina e determinare le affinità di legame. Per una comprensione più approfondita della progettazione e dell'acquisizione degli esperimenti NMR, fare riferimento al manuale del produttore corrispondente fornito con lo strumento NMR.
- Acquisizione e lavorazione
- Preparare il campione con la lectina di interesse. Assicurarsi che il recettore sia completamente marcato con 15N in ogni residuo di aminoacidi, sia nella spina dorsale che nelle catene laterali. Tipicamente, per rilevare i picchi trasversali di HN scambiabili in acqua nello spettro, utilizzare una miscela 90:10 di H2O: D2O per preparare la soluzione tamponata. Le concentrazioni di lectina richieste variano tra 0,05 e 0,2 mM, a seconda della disponibilità del recettore marcato con 15N e del rapporto segnale/rumore necessario.
NOTA: La proteina dovrebbe essere stabile durante l'intero tempo sperimentale senza la generazione visibile di precipitato nella provetta NMR. Inoltre, dovrebbe essere puro e solubile nel tampone selezionato. L'assegnazione completa di 1H e 15N dei picchi trasversali HSQC deve essere stata precedentemente effettuata in modo che ogni picco trasversale nello spettro HSQC sia identificato con un'etichetta corrispondente allo specifico residuo amminoacidico. - Da questa preparazione, trasferire un volume totale di 0,6 mL in una provetta NMR da 5 mm.
- Impostare lo strumento NMR alla temperatura richiesta. Vedere il passaggio 1.1.3 e seguire le stesse operazioni.
- Creare un nuovo set di dati. Vedere il passaggio 1.1.4 e ripetere le operazioni.
- Inserire il campione NMR nella sonda come descritto al punto 1.1.5.
- Bloccare il segnale del solvente. Per avviare la procedura di blocco, utilizzare il comando blocco e selezionare il solvente appropriato dal menu. Il segnale di blocco può essere tracciato nella finestra della serratura. Impostare il guadagno di blocco in modo che il segnale di blocco sia visibile nella finestra di blocco.
- Completa il processo di sintonizzazione e abbinamento automaticamente (tramite il comando atma) o manualmente (il comando atmm aprirà la finestra di controllo ATM per regolare la curva di oscillazione).
- Impostare gli spessori ottimali utilizzando lo strumento Spessore superiore. Usa il comando topshim gui. Vedere le istruzioni al passaggio 1.1.8.
- Determinare la lunghezza dell'impulso di 1ora e 90° (come descritto al punto 1.1.9) e la frequenza di offset (il comando o1calib eseguirà una routine di calibrazione O1 interattiva, recuperando la frequenza di offset). Quest'ultimo parametro è estremamente importante quando vengono impiegati esperimenti con schemi di soppressione dei solventi.
- Creare un nuovo set di dati come descritto nella sezione 1.1.4. Per ridurre o eliminare l'interferenza del segnale H2O, utilizzare la sequenza di impulsi zgesgp.
- Impostare l'esperimento modificando vari parametri nella finestra AcquPars.
- Introdurre la lunghezza dell'impulso di 1ora e 90° e l'offset (o1) come precedentemente determinato e impostare il numero di scansioni (NS) a 32 e la finestra spettrale (SW) a circa 12 ppm.
- Determinare il livello di potenza dell'impulso sagomato utilizzando lo strumento Forma disponibile nella barra dei menu Topspin.
- Impostare il guadagno del ricevitore con il comando automatico rga.
- Acquisire l'esperimento utilizzando il comando zg ed elaborare il FID risultante per ottenere lo spettro NMR 1H.
- Creare un nuovo set di dati da utilizzare per l'acquisizione dell'esperimento 1 H-15N HSQC NMR. Nella scheda AcquPars, selezionare il programma a impulsi hsqcetfpf3gp disponibile nel catalogo dei programmi a impulsi.
- Configura l'esperimento. Carica le forme, i poteri e i tempi predefiniti utilizzando il comando getprosol. Quindi, aggiornare i valori della lunghezza dell'impulso di 1ora a 90° e dell'offset.
- Definire i parametri riportati di seguito.
- Impostare il ritardo di rilassamento su 1-5 s.
- Impostare il numero di scansioni su un multiplo di 4. Di solito, è impostato su 8, 16, 32 o 64 per ottenere il corretto rapporto segnale/rumore.
- Impostare il numero di scansioni fittizie su 128.
- Impostare il numero di punti in F2 su 1k, 2k o 4k.
- Imposta il numero di punti in F1: il numero di incrementi di t1 da utilizzare. A seconda della finestra spettrale, questo è compreso tra 128 e 256.
- Regolare il centro della finestra spettrale nella dimensione 15N su δ 117 ppm e impostare la larghezza spettrale corrispondente su 36 ppm. Questi valori devono essere ottimizzati per ogni particolare sistema.
- Impostare il guadagno del ricevitore per evitare l'overflow (utilizzando il comando rga )
- Calcola il tempo dell'esperimento totale. Un tempo tipico sperimentale è di circa 1 ora.
- Digitare zg per inviare l'esperimento per l'acquisizione.
NOTA: Verificare sempre che l'esperimento venga eseguito correttamente dopo alcuni minuti. - Elabora il FID utilizzando il comando xfb. Eseguire la correzione della linea di base utilizzando il comando abs2 e le correzioni di fase nella scheda Processo. Per eseguire manualmente la fase, fare clic sul sottomenu di regolazione della fase e quindi selezionare diversi picchi trasversali degli spettri 2D. Successivamente, applica in sequenza le correzioni zero e di primo ordine sia alle righe che alle colonne facendo clic e trascinando il pulsante corrispondente. Salvare i risultati della gradualità.
- Salvare lo spettro 2D risultante.
- Preparare una soluzione madre altamente concentrata del ligando. I valori tipici sono 50-100 mM.
- Dalla soluzione madre altamente concentrata del glicano, trasferire il volume corrispondente (pochi microlitri) alla provetta NMR contenente il recettore per ottenere il rapporto proteina/molare ligando desiderato e registrare gli spettri.
NOTA: Questa fase avvia la serie di titolazione, in cui il ligando viene titolato nel campione proteico. Per ogni caso particolare devono essere determinati i rapporti proteina/ligando appropriati. Se l'affinità di legame è completamente sconosciuta, si raccomanda di utilizzare quantità substechiometriche del ligando nei punti iniziali. - Eseguire i passaggi da 2.1.1 a 2.1.19 per il campione appena preparato.
- Ripetere i passaggi 2.1.21 e 2.1.22 per campioni con rapporto proteine/ligando crescenti.
NOTA: L'adattamento accurato dei dati della serie di titolazione richiede l'acquisizione di più esperimenti1 H-15N-HSQC, che coprono un'ampia gamma di rapporti proteina-ligando, compresi quelli necessari per raggiungere la saturazione proteica.
- Preparare il campione con la lectina di interesse. Assicurarsi che il recettore sia completamente marcato con 15N in ogni residuo di aminoacidi, sia nella spina dorsale che nelle catene laterali. Tipicamente, per rilevare i picchi trasversali di HN scambiabili in acqua nello spettro, utilizzare una miscela 90:10 di H2O: D2O per preparare la soluzione tamponata. Le concentrazioni di lectina richieste variano tra 0,05 e 0,2 mM, a seconda della disponibilità del recettore marcato con 15N e del rapporto segnale/rumore necessario.
- Analisi
- Visualizzare lo spettro HSQC 2D elaborato per le specie di apo utilizzando il software appropriato: TopSpin, MestReNova e CCPNMR sono tutti programmi adatti per la gestione dei dati NMR.
NOTA: Questo è lo spettro delle impronte digitali della proteina. Gli spostamenti chimici osservati di 1H e 15N dipendono dall'ambiente chimico corrispondente di ogni amminoacido, che dipende fortemente dalla struttura 3D della proteina. Questo spettro è chiamato spettro dell'impronta digitale della proteina. Uno spettro HSQC 2D 1 H-15N ben disperso, in cui tutti i picchi trasversali mostrano intensità uniformi, suggerisce fortemente la presenza di una proteina19 ben ripiegata. - Genera l'elenco delle frequenze 1H e 15N per tutti i picchi trasversali. L'uso di software supplementari, come il programma CCPNMR20, può aiutare nel processo.
- Sovrapporre lo spettro per il primo o il secondo punto di titolazione a quello per la proteina apo.
- Per fare ciò, apri lo spettro 2D corrispondente allo stato apo, fai clic sulla scheda Display multiplo e quindi aggiungi il secondo spettro 2D. L'ispezione visiva di entrambi gli spettri fornisce informazioni sull'esistenza di interazione tra il ligando e la proteina.
NOTA: Dal punto di vista della proteina, l'esistenza del legame fornisce cambiamenti nell'ambiente chimico degli amminoacidi direttamente coinvolti nell'evento di riconoscimento, con le concomitanti perturbazioni di spostamento chimico (CSP).
- Per fare ciò, apri lo spettro 2D corrispondente allo stato apo, fai clic sulla scheda Display multiplo e quindi aggiungi il secondo spettro 2D. L'ispezione visiva di entrambi gli spettri fornisce informazioni sull'esistenza di interazione tra il ligando e la proteina.
- Ripetere i passaggi 2.2.2 e 2.2.3 per ogni punto di titolazione, generando elenchi delle frequenze 1H e 15N per tutti i picchi trasversali nei diversi spettri, corrispondenti a diversi rapporti molari proteina-ligando.
NOTA: Gli spostamenti chimici in ogni punto di titolazione possono essere misurati senza la necessità di eseguire alcuna nuova assegnazione del picco trasversale. Nel caso di un regime di scambio rapido, comunemente osservato nelle interazioni lectina:glicano, si può semplicemente seguire il movimento progressivo dei picchi durante la titolazione. - Verificare che, per l'ultimo punto di titolazione, non vi siano sostanzialmente perturbazioni di spostamento chimico rispetto all'aggiunta precedente. Questo fatto è indicativo che il sito di legame della proteina è stato saturo con il ligando, che è in alto eccesso.
- Calcola le perturbazioni massime dello spostamento chimico (maxCSP) utilizzando l'equazione seguente:
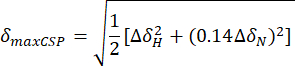
ΔH e ΔδN sono le differenze di spostamento chimico nelle frequenze 1H e 15N tra lo stato apo e l'ultimo punto di titolazione, rispettivamente. - Traccia le perturbazioni di spostamento chimico massimo (maxCSP) sull'asse y verticale di un grafico 2D rispetto al corrispondente residuo di amminoacidi (sull'asse x orizzontale).
- Effettuare un'ispezione visiva dei residui di amminoacidi che mostrano il CSP massimo tra lo stato legato e quello apo della proteina. È molto probabile che appartengano al sito di legame o siano vicini ad esso.
- Se la struttura 3D della proteina è disponibile, aprire il PDB corrispondente con il software appropriato come PyMOL o BIOVIA Discovery studio. Questi programmi di visualizzazione molecolare sono ampiamente utilizzati nelle applicazioni di biologia strutturale. Selezionare i residui che mostrano il maxCSP più alto (superiore al doppio della deviazione standard) con un colore particolare per localizzare il sito di legame putativo.
- Nel caso di un regime di scambio veloce, stimare la costante di dissociazione (KD) da un adattamento non lineare dei minimi quadrati del CSP osservato per i picchi incrociati HSQC 1 H-15N in ogni punto (Δδobs) rispetto alla particolare concentrazione di proteina [P] e ligando [L] in quel punto:
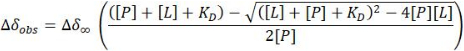
NOTA: Questa equazione può essere applicata a quei picchi trasversali che visualizzano chiari segnali isolati. I valori ottenuti vengono mediati per fornire la stima di KD.
- Visualizzare lo spettro HSQC 2D elaborato per le specie di apo utilizzando il software appropriato: TopSpin, MestReNova e CCPNMR sono tutti programmi adatti per la gestione dei dati NMR.
Risultati
In questo articolo, presentiamo un protocollo per lo sfruttamento di esperimenti 1H-STD NMR e 1 H-15N HSQC per svelare i dettagli dell'interazione di legame tra lectine e piccoli oligosaccaridi. Sono inclusi i risultati ottenuti nell'analisi del riconoscimento molecolare di LacNAc da parte di hGalectina-7 (hGal-7), che fungono da esempio illustrativo del successo dell'implementazione del protocollo e dell'efficacia di queste metodologie NMR per studiare i dettagli fini del processo di riconoscimento molecolare. La Figura 3 mostra lo spettro 1H-STD NMR per l'interazione di LacNAc con hGal-7. L'esistenza di segnali NMR STD indica il legame (Figura 3A). Inoltre, compaiono solo quei segnali appartenenti ai protoni a stretto contatto con la proteina, permettendo la delineazione dell'epitopo di legame (Figura 3B). La Figura 4 evidenzia come lo spettro HSQC 1 H-15N di una proteina possa essere utilizzato come impronta digitale, mentre la Figura 5 illustra l'applicazione di esperimenti di titolazione a coerenza quantistica singola eteronucleare (HSQC) 1 H-15N per definire la perturbazione dello spostamento chimico dei gruppi ammidici dellaspina dorsale h Galectina-7 al legame con LacNAc. Questi dati non solo rivelano l'esistenza di interazione, ma delineano anche l'interfaccia di legame della lectina. La Figura 6 mostra come l'analisi dei dati di titolazione consenta di stimare l'affinità di legame di LacNAc da parte della galectina-7, che rientra nell'intervallo micromolare alto. Questo risultato è coerente con i risultati ottenuti utilizzando tecniche alternative.
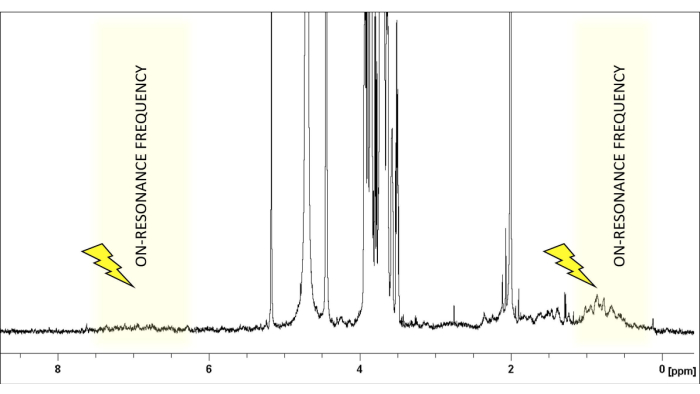
Figura 1: La selezione della frequenza di risonanza. 1Viene mostrato lo spettro H-NMR del rapporto LacNAc:hGal-7 50:1 in soluzione salina tamponata con fosfato deuterato a pH 7,4. I segnali del ligando (LacNAc) sono confinati nella regione tra 2,0-5,2 ppm. La frequenza di saturazione è accuratamente selezionata per garantire l'assenza di protoni ligando entro un intervallo di 1-2 ppm, consentendo l'irradiazione selettiva dei protoni della proteina. Clicca qui per visualizzare una versione più grande di questa figura.
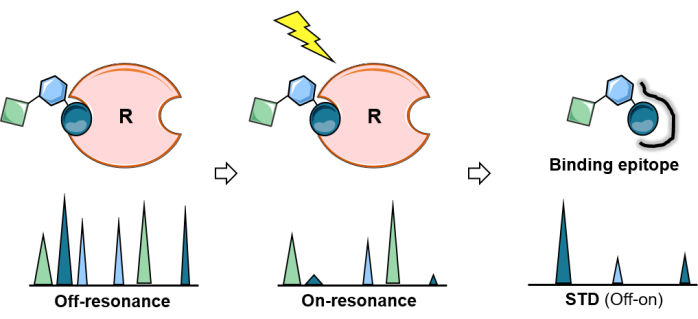
Figura 2: L'esperimento MRN STD. Rappresentazione schematica dell'esperimento STD: il primo spettro (off-resonanza) funge da riferimento mentre nel secondo (on-resonanza), viene eseguita la saturazione delle proteine. La saturazione viene propagata in modo efficiente attraverso l'intera proteina e trasferita ai protoni ligando a stretto contatto con la proteina. Lo spettro di differenza risultante (spettro STD) produce solo quelle risonanze che hanno subito la saturazione. L'analisi dell'esperimento STD consente la mappatura degli epitopi dello zucchero legante. Clicca qui per visualizzare una versione più grande di questa figura.
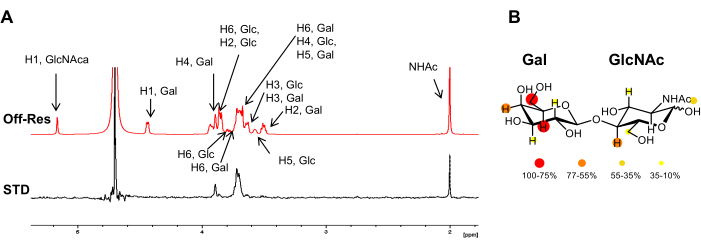
Figura 3: Analisi del legame dal punto di vista del ligando. (A) Sovrapposizione degli spettri off-risonanza e 1H STD-NMR per l'interazione di LacNAc con hGal-7. Nello spettro delle malattie sessualmente trasmissibili, compaiono solo i segnali appartenenti ai protoni a stretto contatto con la proteina. L'annotazione delle risonanze 1H del ligando è riportata nello spettro fuori risonanza. (B) Le intensità relative delle malattie sessualmente trasmissibili sono state mappate a colori nella struttura chimica del LacNAc. Clicca qui per visualizzare una versione più grande di questa figura.
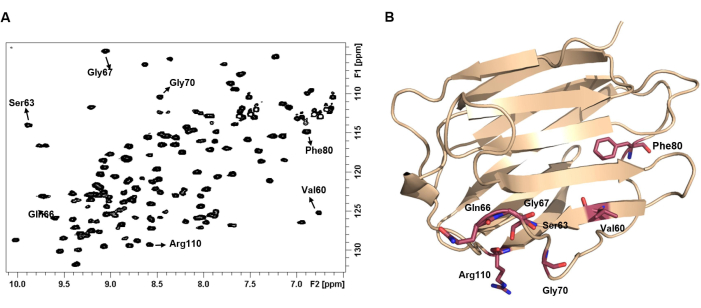
Figura 4: Lo spettro HSQC 1 H-15N di una proteina rappresenta la sua impronta digitale. (A) 1spettro HSQC H-15N di 100 μM di hGal-7 in forma apo. Lo spettro è stato registrato a 25 °C. Alcuni cross-peak NH sono stati annotati con l'etichetta del loro amminoacido corrispondente. (B) Ogni coppia NH mostra uno spostamento chimico unico che dipende dall'ambiente chimico e, di conseguenza, dalla struttura 3D della proteina. Clicca qui per visualizzare una versione più grande di questa figura.
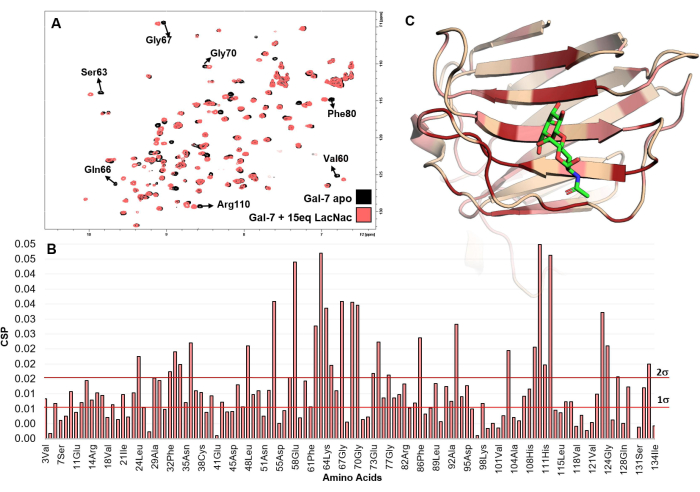
Figura 5: Analisi del legame dal punto di vista della proteina. (A) Viene mostrata la sovrapposizione degli spettri HSQC 1 H-15N registrati per la titolazione di LacNAc in soluzione di hGal-7. L'ispezione degli spettri, dove diversi picchi trasversali subiscono cambiamenti chimici di spostamento, indica chiaramente l'interazione. (B) Il grafico delle perturbazioni di spostamento chimico massimo (maxCSP) dei segnali dell'ammide dello scheletro dedotti dalla titolazione di LacNAc (15 equivalenti) con hGal-7. (C) Gli amminoacidi più perturbati di hGal-7, secondo l'analisi CSP, sono mappati nella struttura PDB 5gal. Nel modello 3D, la colorazione rossa si riferisce a valori CSP superiori a 2σ, mentre quelle rosa a valori compresi tra 1σ e 2σ. La regione colorata rappresenta probabilmente il sito di legame. Clicca qui per visualizzare una versione più grande di questa figura.
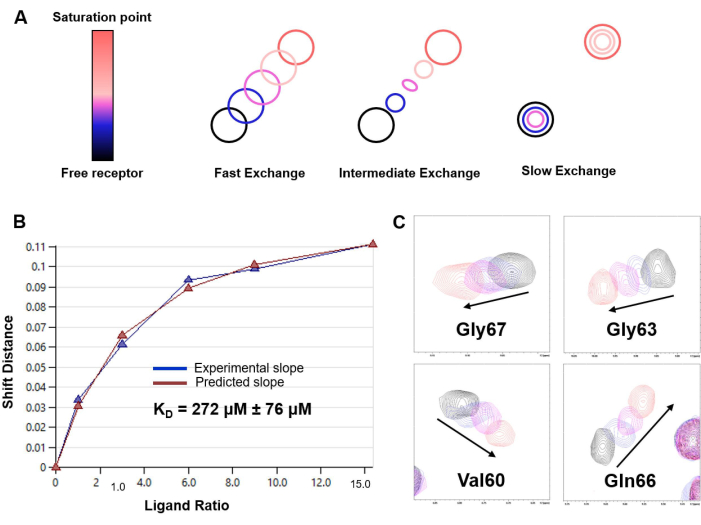
Figura 6: Determinazione KD basata su esperimenti di titolazione HSQC 1 H-15N. (A) Rappresentazione del modello di titolazione basata su HSQC 1 H-15N a seconda del tasso di scambio chimico nella scala temporale NMR del sistema nello studio (veloce, intermedio o lento). Un regime di scambio veloce è stato osservato nel caso dell'interazione LacNAc/hGal-7. (B) Curva di fitting e stima KD ottenute dall'analisi CSP a concentrazioni variabili di ligando per il sistema modello di hGal-7 e LacNAc disaccaride. Il KD stimato è riportato con l'errore corrispondente come media dei dati per 20 diversi amminoacidi; (C) Frammenti degli spettri 1 H,15N-HSQC che mostrano lo spostamento dei picchi trasversali selezionati durante la titolazione. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
La differenza di trasferimento di saturazione NMR (STD-NMR) è diventata il metodo NMR più utilizzato e versatile per studiare le interazioni ligando-proteina. Come mostrato sopra, si basa sul fenomeno del trasferimento di saturazione e la configurazione sperimentale prevede l'acquisizione di due spettri monodimensionali (1D) 1H: lo spettro in risonanza e lo spettro in fuori risonanza. Durante l'esperimento di risonanza, la saturazione di protoni specifici della proteina viene ottenuta applicando un treno di impulsi a radiofrequenza a bassa potenza durante un certo periodo (il tempo di saturazione varia tipicamente da 1 a 3 s). Per evitare la saturazione diretta del ligando, la frequenza e la lunghezza degli impulsi di saturazione sono ottimizzate per irradiare selettivamente protoni specifici della proteina; cioè, devono essere applicati a una frequenza libera da qualsiasi segnale del ligando e con una lunghezza appropriata (Figura 1). Come regola generale, per impulsi di saturazione di 50 ms, è necessario mantenere una differenza di 1 ppm tra la regione di saturazione e i segnali del ligando più vicini. Generalmente, gli impulsi di saturazione selettiva applicati sulla regione alifatica della proteina forniscono un aumento degli effetti di saturazione. In alternativa, i protoni aromatici (6-7 ppm) possono anche essere irradiati se la molecola del ligando non contiene alcun segnale aromatico. Questo è molto utile per i glicani presenti in natura, in quanto non portano gruppi aromatici. Una volta che una certa regione della proteina viene irradiata selettivamente, la saturazione si propaga lungo la proteina attraverso il cross-rilassamento dipolare 1 H-1H (diffusione dello spin). Alla fine, la saturazione raggiunge i protoni proteici nel sito di legame, che viene poi trasferito ai protoni dello zucchero che sono in stretto contatto (r < 5 Å) con il recettore tramite NOE intermolecolari 1 H-1H. Ovviamente, l'intensità dei segnali dei protoni ligando saturi diminuisce. Dopo aver ricevuto la saturazione, a causa della cinetica di legame, i ligandi legati transitoriamente (è richiesto uno scambio veloce) si dissociano e l'informazione di saturazione si accumula nello stato libero. A causa di questo processo, gli spettri di risonanza NMR presentano segnali ridotti (Figura 2).
Per mostrare chiaramente questa perturbazione dell'intensità dei nuclei 1H di un glicano legante, viene acquisito uno spettro NMR del protone di controllo (off-resonance) in cui la saturazione viene applicata lontano da qualsiasi segnale recettoriale o glucidico (di solito tra 40-100 ppm), nelle stesse condizioni. Lo spettro 1D sottratto tra la risonanza off e la risonanza on mostra esclusivamente i segnali dei nuclei 1H del ligando che hanno intensità modificate: quelli che erano abbastanza vicini al sito di legame del recettore per ricevere la magnetizzazione (Figura 2).
Tuttavia, non tutti i nuclei 1H del carboidrato legato ricevono la stessa quantità di saturazione. Teoricamente, il trasferimento di magnetizzazione dal recettore al ligando legato è dipendente dalla distanza (1/r6). Ciò significa che le intensità di saturazione trasferite tra i nuclei del glicano 1H contengono informazioni sulle prossimità spaziali tra i protoni del ligando e quelli del recettore, e le intensità STD NMR sono maggiori per quei protoni che sono più vicini al recettore. Di conseguenza, l'esperimento MRN STD consente anche di determinare l'epitopo di legame del carboidrato (Figura 2 e Figura 3) poiché i protoni del ligando che si trovano più vicino alla superficie proteica mostrano intensità più elevate rispetto a quelli che non partecipano direttamente al legame.
L'esperimento può essere applicato a sistemi con affinità medio-debole, raramente a sistemi con affinità forti nell'intervallo dei μM o nM. Infatti, richiede che il tasso di dissociazione sia veloce nella scala temporale del rilassamento. In caso contrario, l'informazione sul trasferimento della saturazione viene persa attraverso il rilassamento prima che il ligando si dissoci.
D'altra parte, gli esperimenti NMR basati sulle proteine sono unici per svelare l'interazione ligando-proteina con l'accuratezza del livello di amminoacido senza risolvere le strutture di risoluzione atomica. Esamina direttamente i fenomeni di riconoscimento molecolare in soluzione senza la necessità di co-cristallizzazione. La mappatura dell'analisi CSP è eccezionalmente potente per scoprire ligandi e mappare il sito di legame delle proteine (Figura 4 e Figura 5). Questo metodo è applicabile a qualsiasi intervallo di affinità tra l'intervallo mM e nM, anche per sistemi in cui il tasso di cambio è lento nella scala temporale di spostamento chimico21.
Tuttavia, questo approccio probabilmente non funzionerà per proteine con pesi molecolari superiori a 30-40 kDa a causa di problemi di rilassamento. L'alternativa TROSY18 può quindi essere utilizzata, particolarmente potente se accoppiata alla deuterazione delle proteine. Inoltre, la proteina deve essere marcata in modo uniforme con 15N (e un altro campione marcato due volte con 13C e 15N per essere in grado di completare l'assegnazione della spina dorsale richiesta). Pertanto, le condizioni di espressione proteica, incluso il corrispondente sistema di espressione, dovrebbero essere ottimizzate per essere in grado di ottenere quantità di milligrammi di proteine. Anche le proteine che mostrano una tendenza a oligomerizzarsi o aggregarsi non sono adatte per questa analisi. Lo strumento qui utilizzato per registrare i dati NMR è uno spettrometro Bruker da 800 MHz dotato di una criosonda TCI. Sarebbe molto impegnativo utilizzare questa metodologia utilizzando strumenti al di sotto di 600 MHz o senza una sonda criogenica.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo l'Agencia Estatal de Investigación della Spagna per l'accreditamento del Centro di Eccellenza Severo Ochoa CEX2021-001136-S, finanziato da MCIN/AEI/10.13039/ 501100011033, e CIBERES, un'iniziativa dell'Instituto de Salud Carlos III (ISCIII, Madrid, Spagna). Ringraziamo anche la Commissione Europea per il progetto GLYCOTWINNING.
Materiali
| Name | Company | Catalog Number | Comments |
| 5 mm Shigemi microtube set mat | CortecNet SAS | S30BMS-005B | |
| Alpha-Lactose-Agarose | Sigma-Aldrich Química S.L. | 7634-5ML | |
| Ammonium chloride (15 N, 99%) | LC-0179-N-50G | Tracer Tecnologías Analíticas S.L | |
| Ampicillin (Sodium Salt) | Melford Laboratories LTD | A40040 | |
| BIOVIA Discovery studio | BIOVIA, Dassault Systèmes | ||
| BL21(DE3) Chemically Competent Cells | Merck Life Science, S.L.U. | CMC0014-40X40UL | |
| Centrifuge | Beckman Coulter | Allegra X-22R | |
| D2O | Cambridge Isotope Laboratories, Inc. | DLM-4-1000 | |
| Incubator | Eppendorf | Innova 42 | |
| IPTG (Isopropyl ß-D-1-thiogalactopyranoside) | VWR International Eurolab S.L. | VW437144N | |
| LacNAc | Elicityl | GLY008 | |
| Luria Bertani (LB) Broth | Merck Life Science, S.L.U. | 3397-1KG | |
| Matraz Erlenmeyer B N 5000 CC | VWR International Eurolab S.L. | 214-1137 | |
| PBS 10x | Bio-Rad | 1610780 | |
| PyMOL | PyMOL Molecular Graphics System | Version 2.0 Schrödinger | |
| Sonicator | Sonics & Materials, Inc. | VC 505 | |
| Superconducting NMR magnet | Bruker | 600 MHz AVANCE III | |
| Superconducting NMR magnet | Bruker | 800 MHz AVANCE III |
Riferimenti
- Solís, D., et al. A guide into glycosciences: How chemistry, biochemistry and biology cooperate to crack the sugar code. Biochim Biophys Acta. 1850 (1), 186-235 (2015).
- Poveda, A., Jimenez-Barbero, J. NMR studies of carbohydrate-protein interactions in solution. Chem Soc Rev. 27, 133-144 (1998).
- Kogelberg, H., Solís, D., Jiménez-Barbero, J. New structural insights into carbohydrate-protein interactions from NMR spectroscopy. Curr Opin Struct Biol. 13 (5), 646-653 (2003).
- Widmalm, G. Glycan shape, motions, and interactions explored by NMR spectroscopy. JACS Au. 4 (1), 20-39 (2024).
- Marchetti, R., et al. 34;Rules of Engagement" of protein-glycoconjugate interactions: A molecular view achievable by using NMR spectroscopy and molecular modeling. ChemistryOpen. 5 (4), 274-296 (2016).
- Ardá, A., Jiménez-Barbero, J. The recognition of glycans by protein receptors. Insights from NMR spectroscopy. Chem Commun. 54 (38), 4761-4769 (2018).
- Gimeno, A., Valverde, P., Ardá, A., Jiménez-Barbero, J. Glycan structures and their interactions with proteins. A NMR view. Curr Opin Struct Biol. 62, 22-30 (2020).
- Cañada, F. J., et al. Conformational and structural characterization of carbohydrates and their interactions studied by NMR. Curr Med Chem. 29 (7), 1147-1172 (2022).
- Valverde, P., Quintana, J. I., Santos, J. I., Ardá, A., Jiménez-Barbero, J. Novel NMR avenues to explore the conformation and interactions of glycans. ACS Omega. 4, 13618-13630 (2019).
- Jiménez-Barbero, J., Asensio, J. L., Cañada, F. J., Poveda, A. Free and protein-bound carbohydrate structures. Curr Opin Struct Biol. 9 (5), 549-555 (1999).
- Quintana, J. I., Atxabal, U., Unione, L., Ardá, A., Jiménez-Barbero, J. Exploring multivalent carbohydrate-protein interactions by NMR. Chem Soc Rev. 52 (5), 1591-1613 (2023).
- Roldós, V., Cañada, F. J., Jiménez-Barbero, J. Carbohydrate-protein interactions: a 3D view by NMR. Chembiochem. 12 (7), 990-1005 (2011).
- Unione, L., Ardá, A., Jiménez-Barbero, J., Millet, O. NMR of glycoproteins: profiling, structure, conformation, and interactions. Curr Opin Struct Biol. 68, 9-17 (2021).
- del Carmen Fernández-Alonso, M., Díaz, D., Berbis, M. A., Marcelo, F., Cañada, J., Jiménez-Barbero, J. Protein-carbohydrate interactions studied by NMR: from molecular recognition to drug design. Curr Protein Pept Sci. 13 (8), 816-830 (2012).
- Angulo, J., Nieto, P. M. STD-NMR: application to transient interactions between biomolecules-a quantitative approach. Eur Biophys J. 40 (12), 1357-1369 (2012).
- Mayer, M., Meyer, B. Group epitope mapping by saturation transfer difference NMR to identify segments of a ligand in direct contact with a protein receptor. J Am Chem Soc. 123 (25), 6108-6117 (2001).
- Williamson, M. P. Using chemical shift perturbation to characterise ligand binding. Prog Nucl Magn Reson Spectrosc. 73, 1-16 (2013).
- Fernández, C., Adeishvili, K., Wüthrich, K. Transverse relaxation-optimized NMR spectroscopy with the outer membrane protein OmpX in dihexanoyl phosphatidylcholine micelles. Proc Natl Acad Sci U S A. 98 (5), 2358-2363 (2001).
- Yee, A., Gutmanas, A., Arrowsmith, C. H. Solution NMR in structural genomics. Curr Opin Struct Biol. 16 (5), 611-617 (2006).
- Skinner, S. P., Fogh, R. H., Boucher, W., Ragan, T. J., Mureddu, L. G., Vuister, G. W. CCPNMR AnalysisAssign: a flexible platform for integrated NMR analysis. J Biomol NMR. 66 (2), 111-124 (2016).
- Gimeno, A., et al. Minimizing the entropy penalty for ligand binding: Lessons from the molecular recognition of the histo blood-group antigens by human Galectin-3. Angew Chem Int Ed Engl. 58 (22), 7268-7272 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon