Method Article
글라이칸-단백질 상호 작용 해체: 핵 자기 공명(NMR)을 위한 구조
요약
여기에서는 용액에서 단백질-글라이칸 상호 작용을 특성화하는 것을 목표로 하는 일련의 NMR 실험의 획득, 처리 및 분석을 자세히 설명하는 프로토콜을 제시합니다. 가장 일반적인 리간드 기반 및 단백질 기반 방법론이 요약되어 있으며, 이는 의심할 여지 없이 구조 당생물학 및 분자 인식 연구 분야에 기여합니다.
초록
글라이칸과 단백질의 상호 작용은 건강 및 질병과 관련된 많은 이벤트를 조절합니다. 사실, 이러한 인식 사건의 확립과 그 생물학적 결과는 두 파트너의 3차원 구조뿐만 아니라 그들의 역동적인 특징 및 해당 세포 구획에 대한 표현과 밀접한 관련이 있습니다. NMR 기술은 이러한 특성을 풀기 위해 고유하며, 실제로 다양한 NMR 기반 방법론이 개발되어 관련 수용체와 글리칸의 결합 이벤트를 모니터링하는 데 적용되었습니다. 이 프로토콜은 NMR-glycobiology 분야에서 사용되는 가장 강력한 NMR 방법론 중 두 가지인 1H-포화 전달 차이(STD) 및 1 H,15N-Heteronuclear 단일 양자 일관성(HSQC) 적정 실험을 획득, 처리 및 분석하는 절차를 간략하게 설명하며, 각각 글라이칸 및 단백질 관점에서 정보를 보완적으로 제공합니다. 실제로, 이 둘을 결합하면 분자 인식 프로세스의 구조적 측면과 동적 측면을 모두 설명할 수 있는 강력한 툴킷을 제공합니다. 이 포괄적인 접근 방식은 글라이칸-단백질 상호 작용에 대한 이해를 높이고 화학 당생물학 분야의 연구를 발전시키는 데 기여합니다.
서문
글라이칸의 분자 인식은 건강 및 질병과 관련된 많은 과정에 필수적입니다. 글라이칸에 대한 생물학적 수용체(렉틴, 항체, 효소)의 특이성과 선택성은 엔탈피의 다양한 구성 요소(CH-π 및 van der Waals, 수소 결합, 정전기)와 엔트로피(소수성, 역학, 용매-탈용매화) 간의 불안정한 균형을 조정하는 데 크게 좌우됩니다1.
글라이칸의 큰 화학적 다양성과 동적 특성을 감안할 때, NMR 방법은 25년 이상 글라이칸 상호 작용을 해부하는 데 널리 사용되어 왔습니다2, 이러한 방법론은 다른 방법론을 사용하여 필요한 상호 작용 증거를 검색할 수 없는 경우에도 원자 분해능 3,4에서 정확한 세부 정보와 함께 분자 인식 이벤트에 대한 탁월한 정보를 제공하기 때문입니다. 요점으로, NMR은 다재다능하며 원자 수준에서, 다양한 시간 규모에서 동적 이벤트를 연구할 수 있게 해주어 용액 내 글라이칸의 구조, 형태 및 역학을 연구하기 위한 최고의 기술을 구성합니다. 그럼에도 불구하고, 이러한 정보를 분리하는 것은 신중한 데이터 분석과 함께 잘 정의된 전략을 필요로 하는 다소 복잡한 과정일 수 있다5.
NMR 기술은 다양하며 실제로 글리칸-단백질 상호 작용을 밝히기 위해 사용할 수 있는 많은 방법론이 있습니다6. 여기에서는 현재 글리칸-수용체 상호 작용을 해독하기 위해 사용되는 두 가지 기본 NMR 접근법을 설명하며7,8, 주요 글라이칸 에피토프와 단백질 결합 부위9의 제시를 푸는 방법에 중점을 둡니다.
임의의 분자 인식 이벤트에서, 수용체가 주어진 리간드에 결합할 때, 결합(10)에 있는 참가자의 많은 NMR 파라미터에 영향을 미치는 화학적 교환 과정이 있다. 따라서, NMR 관점에서, 상호 작용은 글라이칸 리간드의 관점에서 또는 단백질 수용체11의 관점에서 모니터링될 수 있다. 일반적으로 단백질 수용체는 큰 생체 분자(느린 회전 운동, ns 시간 척도의 속도, 따라서 빠른 횡방향 이완)인 반면, 상호 작용하는 글라이칸은 중소형 분자(빠른 회전 운동, ps 시간 척도의 속도 및 느린 횡방향 이완)로 간주될 수 있습니다12. 표준 관점에서 볼 때, 글리칸의 NMR 신호는 좁고 수용체의 NMR 신호는 넓습니다13.
리간드 기반 NMR 방법은 자유 상태에서 결합 상태로 전달할 때 많은 글라이칸 NMR 매개변수가 경험하는 극적인 변화에 의존합니다(14). STD-NMR은 용액 상태에서의 결합의 존재를 추론하는 것부터 글리칸 결합 에피토프의 결정에 이르기까지 다양한 글리칸 결합 특징(15)을 평가하기 위해 가장 많이 사용되는 실험적 NMR 기술이다. 즉, 단백질 수용체(16)와 접촉하는 리간드의 원자.
대안적으로, 수용체-기반 NMR 방법은 apo 상태(17)에 대해 기록된 것들에 대해 글라이칸의 존재 하에 단백질 수용체의 신호에서 발생하는 변화를 모니터링한다. 이들은 주로 두 상태 사이의 단백질 신호의 화학적 이동 섭동을 스크리닝하는 데 중점을 둡니다. 가장 일반적으로 사용되는 실험은 1 H-15N HSQC 또는 TROSY 대안18입니다.
두 접근 방식의 조합을 통해 광범위한 친화력을 나타내는 많은 다양한 시스템에 NMR을 적용할 수 있습니다. 그러나 수용체 기반 NMR 방법의 경우 리간드를 기반으로 하는 방법과 달리 상대적으로 많은 양의 용해성, 비응집성, 안정 동위원소 표지(15N) 단백질을 사용할 수 있어야 합니다.
여기에서는 두 가지 방법을 모두 설명하고 강점과 약점을 강조합니다. 프로토콜에 설명된 기본 단계는 Bruker 분광계 사용에 대한 예제로 사용됩니다. 결과적으로 명령 및 매개 변수 이름은 TopSpin(Bruker의 분광계 제어 소프트웨어)에서 사용되는 것과 일치합니다.
프로토콜
1. 포화 전달 차이 NMR (STD-NMR)
참고: 다음 줄에서는 STD-NMR 실험을 획득, 처리 및 분석하기 위한 기본 절차를 간략하게 설명합니다. 이러한 단계는 리간드 결합을 검출하고 리간드 결합 에피토프를 규명하기 위한 기술의 유용성을 예시하는 역할을 합니다. NMR 실험의 설계 및 획득에 대한 보다 심층적인 이해는 NMR 기기와 함께 제공된 해당 제조업체의 설명서를 참조하십시오.
- 획득
- 단백질-리간드 복합체로 시료를 준비합니다. 단백질 농도가 0.01에서 0.2 mM 사이인 10:1에서 100:1 사이의 글라이칸:렉틴 몰 비율을 사용합니다. hGalectin-7과 LacNAc의 상호 작용을 위해 pH 7.4에서 중수소화 인산염 완충 식염수에서 50:1 단백질: 리간드 비율을 사용합니다.
참고: 단백질 수용체는 순수해야 하며 선택한 완충액에 용해되어야 합니다(STD-NMR 실험의 경우 가능한 1HNMR 신호 간섭을 줄이기 위해 해당 완충액의 중수소화 버전이 바람직함). 단백질의 농도는 280nm에서 흡광도를 측정하기 위해 분광 광도계를 사용하여 미리 확인합니다. - 준비된 용액에서 피펫을 사용하여 총 부피 0.6mL를 5mm NMR 튜브로 옮깁니다.
- 필요한 온도에서 NMR 기기를 준비합니다(일반적인 실험 온도는 10°C에서 45°C 사이입니다). edte 명령을 사용하여 온도 제어 모니터를 열고 원하는 온도를 설정합니다. hGalectin-7/LacNAc 연구를 위해 온도를 25°C로 설정했습니다.
- zg 펄스 시퀀스를 포함하는 새 데이터셋을 생성합니다.
- 간단한 작업을 위해 기존 실험을 열고 edc 명령을 입력합니다. 대화 상자가 나타나면 제목, 특성(샘플 사양, 용매) 및 실험의 일부 매개변수를 정의합니다.
- 원래 펄스 시퀀스에서 변경이 필요한 경우 ased(파라미터) 및 AcquPars(획득 파라미터) 창을 탐색합니다. 이 시점에서 분광계의 라이브러리에서 원하는 펄스 프로그램을 선택합니다.
- 표준 1HNMR 스펙트럼의 경우 사용 가능한 목록에서 zg 펄스 시퀀스를 선택합니다.
알림: 수분 함량이 증가한 샘플의 경우 신호 대 잡음비를 높이기 위해 수분 억제 방식을 사용해야 할 수 있습니다. zgesgp와 같은 펄스 시퀀스를 사용하는 것이 바람직하며, 이는 우수한 억제를 제공하지만 나머지 신호의 위상을 제어하는 여기 조각 모듈입니다. 수분 억제 체계의 유형과 주요 특성에 대한 추가 정보는 제조업체의 NMR 자습서를 참조하십시오.
- NMR s를 삽입하십시오.ample를 활성화하여 프로브에 sample 리프트 공기를 사용합니다. ej 명령을 사용하고, 샘플을 자석 위에 배치하고, ij 명령을 사용하여 샘플 리프트를 비활성화합니다.
알림: 샘플을 오토를 사용하여 자석에 주입하려면ampler, sx 명령을 사용한 다음 autosampler 트레이의 NMR 튜브 위치에 해당하는 위치 번호 n을 사용합니다. - lock 명령을 입력한 다음 메뉴에서 적절한 용매를 선택하여 용매 신호를 잠급니다.
- 샘플이 프로브에 삽입되면 자동 모듈 atma 또는 수동 모듈 atmm을 사용하여 튜닝 및 매칭 프로세스를 완료합니다.
- topshim gui 명령을 통해 자동 shim을 시작합니다. 그러면 shim dimension 1D가 선택되고 시작되는 그래픽 인터페이스가 열립니다.
참고: 미묘한 자기장 또는 온도 변화로 인한 shimming 불안정성을 최소화하기 위해 실험 획득을 위해 autoshim을 활성화할 수 있습니다. 이 작업은 BSMS 제어 창에 액세스하고 autoshim을 클릭하여 수행할 수 있습니다. 녹색 강조 표시로 바뀌면 자동 심이 활성화되었음을 나타냅니다. autoshim을 사용할 때 잠재적인 시료 불안정성 문제를 눈에 띄지 않아야 합니다. 따라서 autoshim을 사용할 때 주의가 권장됩니다. - 1H90° 펄스를 결정합니다. 이것은 pulsecal 명령을 통해 자동으로 수행될 수 있습니다.
- AcquPars 창에서 다양한 매개변수를 수정합니다. 일반 1HNMR 스펙트럼의 경우 스캔 수(NS)를 32로 설정하고 원하는 스펙트럼 창(SW)을 ca로 설정합니다. 12 페이지/분
참고: zgesgp 펄스 시퀀스에는 스펙트럼 중앙에 위치해야 하는 잔류 HDO 신호를 제거하기 위한 용매 억제용 모듈이 포함되어 있습니다. 이를 위해 AcquPars에서 O1을 정확하게 정의해야 합니다. - 자동 명령 rga로 오버플로를 피하기 위해 수신기 게인을 설정하십시오.
- 이제 zg 명령을 사용하여 표준 1HNMR 스펙트럼을 획득합니다.
- 수집이 완료되면 efp 명령을 통해 스펙트럼을 처리합니다. TopSpin 메뉴 모음을 사용하여 기준선 및 위상 보정을 적용합니다.
참고: 글라이칸과 단백질에서 파생된 1HNMR 신호가 관찰됩니다(그림 1). 획득한 NMR 스펙트럼의 자세한 분석은 섹션 1.1.14에 설명된 대로 STD NMR 실험의 구현에 권장됩니다. - 새 데이터 세트를 생성하고 섹션 1.1.4에서 1HNMR 실험에 대해 설명한 것과 동일한 방식으로 사용할 STD NMR 펄스 시퀀스를 업로드합니다. Bruker 기기에서는 펄스 프로그램 카탈로그에서 다양한 펄스 시퀀스를 사용할 수 있으며, 모두 stddiffXXX라는 이름이 지정되어 있습니다. 가장 간단한 것(stddiff)에는 수분 억제 체계나 단백질 억제 필터가 포함되어 있지 않습니다.
- H2O 함량이 높은 샘플의 경우 워터게이트 또는 여기 조각 모듈을 포함하는 stddiffgp19 또는 stddiffesgp 시퀀스를 선택합니다. 강렬한 단백질 NMR 신호를 배경으로 하는 스펙트럼의 경우 stddiffXXX.3 서열을 선택합니다. 각각의 경우, 각 수분 억제 모듈(즉, 워터게이트 체계의 d19)에 해당하는 특정 매개변수를 최적화합니다.
- STD NMR 실험에 대한 오프 및 온 공진 주파수를 정의합니다. FQ2LIST 항목 아래에 있는 ased 창의 AcquPars 매개변수에서 주파수 목록을 찾습니다. Hertz에서 정의된 on-resonance 및 off-resonance 주파수는 수동으로 목록에 기록하고 새 이름으로 저장해야 합니다. 이 새로운 목록은 STD-NMR 실험에 사용됩니다.
- 일반적인 글라이칸의 경우 일반적으로 약 δ(1H) 0 또는 6.6ppm의 글라이칸 신호가 없는 스펙트럼 영역에서 온 공명 주파수를 선택합니다(그림 1). 리간드 또는 단백질 양성자를 보여주지 않는 영역에서 오프 공명 주파수를 설정합니다. +18000 또는 -18000Hz로 안전하게 설정할 수 있습니다.
- ased 창의 AcquPars 매개변수에서 포화 시간 동안 사용할 모양의 펄스를 정의합니다.
참고: 많은 가능성이 있습니다. 가우시안 또는 에버트 모양은 50ms의 선택적 펄스의 90° 너비로 안전하게 사용할 수 있습니다. - AcquPars 섹션에서 해당 매개 변수를 설정합니다.
- 1H90° 펄스 길이를 설정합니다.
- 형상 펄스의 전력 값을 설정합니다( 형상 도구를 통해 추정).
- 총 포화 시간을 설정합니다. 1초에서 4초 사이의 값을 정기적으로 사용할 수 있습니다.
- 이완 지연을 3초로 설정합니다.
- 스캔 횟수(NS)를 8의 배수로 설정합니다. 일반적으로 256, 512 또는 1024로 설정되어 각 주파수에서 2 세트로 적절한 신호 대 잡음 비율을 얻습니다.
- 더미 스캔(DS) 수를 8로 설정합니다.
- F2의 점 수를 16k, 32k 또는 64k로 설정합니다.
참고: F2에서 포인트 수를 늘리면 해상도와 신호 대 잡음비가 향상됩니다. 따라서 최소 16k 데이터 포인트를 사용하는 것이 좋습니다. - F1에서 포인트 수를 설정합니다. 이것은 사용할 주파수의 수이며, 이 경우 2(온 공명 및 오프 공진)입니다.
참고: 규칙에 따라 F2는 FID(Free Induction Decay)가 직접 샘플링되는 차원인 직접 차원을 나타내고 F1은 간접 차원을 나타냅니다. - 자동 명령 rga로 오버플로를 방지하도록 수신기 게인(RG)을 설정합니다.
- expt 명령을 사용하여 전체 실험의 시간을 계산합니다.
- zg 명령을 통해 수집을 위해 실험을 보냅니다.
- 항상 몇 분 후에 실험이 제대로 실행되고 있는지 확인하세요.
- 단백질-리간드 복합체로 시료를 준비합니다. 단백질 농도가 0.01에서 0.2 mM 사이인 10:1에서 100:1 사이의 글라이칸:렉틴 몰 비율을 사용합니다. hGalectin-7과 LacNAc의 상호 작용을 위해 pH 7.4에서 중수소화 인산염 완충 식염수에서 50:1 단백질: 리간드 비율을 사용합니다.
- 가공
참고: 의사 2D 스펙트럼은 위에서 설명한 프로토콜을 적용한 후에 얻어집니다. 행의 수는 사용된 주파수의 수에 해당하며, 일반적으로 두 가지, 즉 온 공명과 오프 공진에 해당합니다.- 첫 번째 실험에 대한 fid를 처리합니다.
- fid 번호 1의 푸리에 변환을 수행하고( efp 명령을 통해) 처리된 스펙트럼의 대상을 선택합니다(procno 수 선택). 또는 rser 1 명령을 사용하여 첫 번째 fid를 읽습니다.
- 그런 다음 lb 명령(일반적으로 3-5Hz)을 통해 라인 확장 계수와 위상을 조정합니다. 수동으로 단계를 지정하려면 프로세스 탭을 클릭한 다음 단계 하위 메뉴를 조정합니다 . 0 및 1차 보정을 수행하려면 해당 버튼을 클릭하고 드래그합니다. 위상 조정 결과를 저장합니다. 또한 abs 명령을 통해 기준선 수정을 수행합니다.
- 두 번째 실험에 대한 fid를 읽고, 동일한 선 확장 인자를 사용하여 푸리에 변환을 ( efp 명령을 통해) 수행합니다. 동일한 위상 파라미터와 기준선 보정으로 위상을 조정하고 처리된 스펙트럼을 다른 코드로 저장합니다.
- multiple 함수(명령: .md)를 사용하여 처리된 두 스펙트럼을 읽고 다중 시각화(Δ)에서 사용할 수 있는 버튼을 사용하여 뺍니다(off-resonance - on-resonance). 새로운 스펙트럼은 다른 코드로 저장되는 STD NMR 스펙트럼입니다.
- STD NMR 스펙트럼을 off-resonance 스펙트럼과 중첩합니다.
- 오프 공명 스펙트럼(fid 1)을 열고 .md 명령을 입력하여 다중 디스플레이 창을 엽니다. 그런 다음 STD 스펙트럼을 업로드합니다.
- STD NMR 스펙트럼에 있는 신호의 주파수와 강도(오른쪽 상단에 자동으로 표시됨)를 비교합니다. 이것은 단백질에 가까운 양성자와 그들의 상대적 근접성에 대한 원하는 정보를 제공합니다. 상대 강도가 높을수록 단백질에 더 가깝습니다(그림 2).
- 해당 소프트웨어를 사용하여 off-resonance 실험에서 강도(적분)를 측정합니다. TopSpin에서 분석 > 통합으로 이동합니다. 영역을 정의하고 파일(I0)에 적분을 씁니다.
- 동일한 매개변수를 사용하여 STD NMR 실험에서 강도(적분)를 측정하고 파일(ISTD)에 씁니다.
- 다음 방정식을 사용하여 각 양성자 신호에 대한 STD 값을 계산합니다.
성병 = (I성병)/나0.
참고: 참고: STD 값을 계산하기 위해 신호 적분을 사용하려면 양성자 신호가 충분히 분리되어야 합니다. 올리고당과 같이 신호 중복이 발생하는 경우, STD와 오프 공진 스펙트럼 사이의 신호 강도 비율을 평가하여 STD 값을 결정할 수 있습니다. - 상대 STD를 백분율로 계산합니다. 이렇게하려면 오프 공진과 STD NMR 스펙트럼의 강도 사이의 최대 차이를 나타내는 양성자에 100 % 값을 부여하십시오. 그에 따라 다른 양성자에 대한 상대적 STD 강도를 계산합니다.
참고: 특히 리간드 결합 에피토프를 결정하기 위한 STD 데이터의 적절한 분석은 리간드의 1H신호를 완전히 할당해야 합니다. 따라서 STD 스펙트럼을 획득하기 전에 이 작업을 완료하는 것이 강력히 권장됩니다.
- 첫 번째 실험에 대한 fid를 처리합니다.
2. 1 H-15N HSQC 실험
참고: 다음 줄은 리간드(올리고당)19의 증가량에 대한 반응으로 수용체(렉틴)의 1H및 15NNMR 공명의 화학적 이동 변화를 모니터링하기 위해 1 H-15N HSQC 실험의 사용을 자세히 설명합니다. 추출된 데이터를 기반으로 한 CSP(Chemical Shift Perturbation) 분석은 결합 파트너를 식별할 뿐만 아니라 단백질 결합 인터페이스를 매핑하고 결합 친화도를 결정하는 데에도 매우 중요합니다. NMR 실험의 설계 및 획득에 대한 보다 심층적인 이해는 NMR 기기와 함께 제공된 해당 제조업체의 설명서를 참조하십시오.
- 취득 및 처리
- 관심 있는 렉틴으로 샘플을 준비합니다. 수용체가 백본과 곁사슬 모두의 모든 아미노산 잔류물에서 완전히 15N-라벨링되어 있는지 확인합니다. 일반적으로 스펙트럼에서 물 교환 가능한 HN 교차 피크를 검출하려면 H2O : D2O의 90 : 10 혼합물을 사용하여 완충 용액을 준비합니다. 필요한 렉틴 농도는 0.05mM에서 0.2mM 사이이며, 이는 15N-표지 수용체의 가용성과 필요한 신호 대 잡음비에 따라 다릅니다.
참고: 단백질은 NMR 튜브에서 눈에 띄는 침전물 생성 없이 전체 실험 시간 동안 안정적이어야 합니다. 또한, 선택한 완충액에 순수하고 용해되어야 합니다. HSQC 교차 피크의 전체 1H및 15N할당은 HSQC 스펙트럼의 모든 교차 피크가 특정 아미노산 잔기에 해당하는 레이블로 식별되도록 사전에 수행되어야 합니다. - 이 준비에서 총 0.6mL의 부피를 5mm NMR 튜브로 옮깁니다.
- NMR 기기를 필요한 온도로 설정합니다. 1.1.3 단계를 참조하고 동일한 작업을 수행하십시오.
- 새 데이터 세트를 만듭니다. 1.1.4단계를 참조하고 작업을 반복합니다.
- NMR s를 삽입하십시오.amp1.1.5단계에서 설명한 대로 프로브에 넣습니다.
- 용매 신호를 잠급니다. 잠금 절차를 시작하려면 lock 명령을 사용하고 메뉴에서 적절한 용매를 선택합니다. 잠금 신호는 잠금 창에서 추적할 수 있습니다. 잠금 창에서 잠금 신호가 보이도록 잠금 게인을 설정합니다.
- 조정 및 일치 프로세스를 자동으로( atma 명령을 통해) 또는 수동으로( atmm 명령은 워블 곡선을 조정하기 위해 ATM 제어 창을 엽니다) 완료합니다.
- TopShim 도구를 사용하여 최적의 shim을 설정합니다. topshim gui 명령을 사용합니다. 1.1.8단계의 지침을 참조하십시오.
- 1H90° 펄스 길이(1.1.9단계에서 설명한 대로)와 오프셋 주파수를 결정합니다(o1calib 명령은 대화형 O1 보정 루틴을 실행하여 오프셋 주파수를 검색함). 이 나중 파라미터는 용매 억제 체계를 사용한 실험을 사용할 때 매우 중요합니다.
- 섹션 1.1.4에 설명된 대로 새 데이터 세트를 만듭니다. H2O 신호의 간섭을 줄이거나 제거하려면 펄스 시퀀스 zgesgp를 사용하십시오.
- AcquPars 창에서 다양한 매개변수를 수정하여 실험을 설정합니다.
- 이전에 결정한 대로 1H90° 펄스 거리와 오프셋(o1)을 도입하고 스캔 수(NS)를 32로, 스펙트럼 창(SW)을 약 12ppm으로 설정합니다.
- Topspin 메뉴 모음에서 사용할 수 있는 Shape 도구를 사용하여 모양 펄스의 전력 수준을 결정합니다.
- 자동 명령 rga로 수신기 게인을 설정합니다.
- zg 명령을 사용하여 실험을 획득하고 결과 FID를 처리하여 1HNMR 스펙트럼을 얻습니다.
- 1 H-15N HSQC NMR 실험을 획득하는 데 사용할 새 데이터 세트를 만듭니다. AcquPars 탭에서 펄스 프로그램 카탈로그에서 사용 가능한 펄스 프로그램 hsqcetfpf3gp를 선택하십시오.
- 실험을 설정합니다. getprosol 명령을 사용하여 디폴트 shapes, powers, 시간을 불러옵니다. 그런 다음 1H90° 펄스 길이와 오프셋 값을 업데이트합니다.
- 다음 매개 변수를 정의합니다.
- 이완 지연을 1-5초로 설정합니다.
- 스캔 횟수를 4의 배수로 설정합니다. 일반적으로 적절한 신호 대 잡음비를 얻기 위해 8, 16, 32 또는 64로 설정됩니다.
- 더미 스캔 수를 128로 설정합니다.
- F2의 점 수를 1k, 2k 또는 4k로 설정합니다.
- F1의 포인트 수를 설정합니다: 사용할 t1 증분의 수입니다. 스펙트럼 창에 따라 이 값은 128에서 256 사이입니다.
- 15N차원에서 스펙트럼 창의 중심을 117ppm δ로 조정하고 해당 스펙트럼 폭을 36ppm으로 설정합니다. 이러한 값은 각 특정 시스템에 맞게 최적화해야 합니다.
- 오버플로를 방지하도록 수신기 게인을 설정합니다( rga 명령 사용).
- 전체 실험의 시간을 계산합니다. 일반적인 실험 시간은 약 1시간입니다.
- zg를 입력하여 수집을 위해 실험을 보냅니다.
참고: 몇 분 후에 실험이 제대로 실행되고 있는지 항상 확인하십시오. - xfb 명령을 사용하여 FID를 처리합니다. 프로세스 탭에서 abs2 및 phase corrections 명령을 사용하여 기준선 보정을 수행합니다. 수동으로 위상을 지정하려면 adjust phase 하위 메뉴를 클릭한 다음 2D 스펙트럼의 여러 교차 피크를 선택합니다. 그런 다음 해당 버튼을 클릭하고 드래그하여 행과 열 모두에 0 및 1차 수정을 순차적으로 적용합니다. 위상 조정 결과를 저장합니다.
- 결과로 생성되는 2D 스펙트럼을 저장합니다.
- 리간드의 고농축 스톡 용액을 준비합니다. 일반적인 값은 50-100mM입니다.
- 글라이칸의 고농축 원액에서 해당 부피(몇 마이크로리터)를 수용체가 포함된 NMR 튜브로 옮겨 원하는 단백질: 리간드 몰 비율을 얻고 스펙트럼을 기록합니다.
참고: 이 단계는 리간드가 단백질 샘플에 적정되는 적정 시리즈를 시작합니다. 각 특정 사례에 대해 적절한 단백질 대 리간드 비율을 결정해야 합니다. 결합 친화도를 완전히 알 수 없는 경우 초기 지점에서 리간드의 화학량론적 양을 사용하는 것이 좋습니다. - 새로 준비된 샘플에 대해 2.1.1-2.1.19단계를 수행합니다.
- 단백질 대 리간드 비율이 증가하는 샘플에 대해 2.1.21 및 2.1.22 단계를 반복합니다.
참고: 적정 시리즈 데이터를 정확하게 맞추기 위해서는 단백질 포화도를 달성하는 데 필요한 비율을 포함하여 광범위한 단백질 대 리간드 비율을 포괄 하는 여러 1 H-15N-HSQC 실험을 획득해야 합니다.
- 관심 있는 렉틴으로 샘플을 준비합니다. 수용체가 백본과 곁사슬 모두의 모든 아미노산 잔류물에서 완전히 15N-라벨링되어 있는지 확인합니다. 일반적으로 스펙트럼에서 물 교환 가능한 HN 교차 피크를 검출하려면 H2O : D2O의 90 : 10 혼합물을 사용하여 완충 용액을 준비합니다. 필요한 렉틴 농도는 0.05mM에서 0.2mM 사이이며, 이는 15N-표지 수용체의 가용성과 필요한 신호 대 잡음비에 따라 다릅니다.
- 분석
- 적절한 소프트웨어를 사용하여 apo 종에 대한 처리된 2D HSQC 스펙트럼 시각화: TopSpin, MestReNova 및 CCPNMR은 모두 NMR 데이터 처리에 적합한 프로그램입니다.
참고: 이것은 단백질의 지문 스펙트럼입니다. 관찰된 1H 및 15N의 화학적 변화는 모든 아미노산의 해당 화학적 환경에 따라 달라지며, 이는 단백질의 3D 구조에 크게 의존합니다. 이 스펙트럼을 단백질 지문 스펙트럼이라고 합니다. 모든 교차 피크가 균일한 강도를 나타내는 잘 분산된 2D 1 H-15N HSQC 스펙트럼은 잘 접힌 단백질(19)의 존재를 강력하게 시사한다. - 모든 교차 피크에 대해 1H 주파수와 15N 주파수의 목록을 생성합니다. CCPNMR 프로그램(20)과 같은 보조 소프트웨어의 사용이 프로세스에 도움이 될 수 있습니다.
- 첫 번째 또는 두 번째 적정 지점에 대한 스펙트럼을 apo 단백질에 대한 스펙트럼에 겹쳐 놓습니다.
- 그렇게 하려면 apo 상태에 해당하는 2D 스펙트럼을 열고 다중 디스플레이 탭을 클릭한 다음 두 번째 2D 스펙트럼을 추가합니다. 두 스펙트럼의 육안 검사는 리간드와 단백질 사이의 상호 작용 존재에 대한 정보를 제공합니다.
참고: 단백질의 관점에서 볼 때, 결합의 존재는 인식 이벤트에 직접 관여하는 아미노산의 화학적 환경 변화와 그에 수반되는 화학적 이동 섭동(CSP)을 제공합니다.
- 그렇게 하려면 apo 상태에 해당하는 2D 스펙트럼을 열고 다중 디스플레이 탭을 클릭한 다음 두 번째 2D 스펙트럼을 추가합니다. 두 스펙트럼의 육안 검사는 리간드와 단백질 사이의 상호 작용 존재에 대한 정보를 제공합니다.
- 모든 적정점에 대해 2.2.2 및 2.2.3 단계를 반복하여 서로 다른 단백질-리간드 몰 비율에 해당하는 서로 다른 스펙트럼의 모든 교차 피크에 대한 1H 및 15N 주파수 목록을 생성합니다.
참고: 각 적정 지점에서의 화학적 변화는 새로운 교차 피크 할당을 수행할 필요 없이 측정할 수 있습니다. lectin:glycan 상호 작용에서 일반적으로 관찰되는 빠른 교환 체제의 경우, 적정 전반에 걸쳐 피크의 점진적인 움직임을 간단히 추적할 수 있습니다. - 마지막 적정 지점에 대해 이전 첨가에 대해 기본적으로 화학적 이동 섭동이 없는지 확인하십시오. 이 사실은 단백질 결합 부위가 리간드로 포화되어 과도하게 존재한다는 것을 나타냅니다.
- 아래 방정식을 사용하여 최대 화학적 이동 섭동(maxCSP)을 계산합니다.
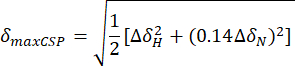
ΔH 및 ΔδN 은 각각 apo 상태와 마지막 적정점 사이의 1H 및 15N 주파수의 화학적 이동 차이입니다. - 2D 플롯의 세로 y축에 최대 화학적 이동 섭동(maxCSP)을 해당 아미노산 잔류물(가로 x축)에 플롯합니다.
- 단백질의 결합 상태와 apo 상태 사이의 최대 CSP를 표시하는 아미노산 잔기를 육안으로 검사합니다. 그들은 바인딩 사이트에 속하거나 이웃일 가능성이 높습니다.
- 단백질의 3D 구조를 사용할 수 있는 경우 PyMOL 또는 BIOVIA Discovery studio와 같은 적절한 소프트웨어를 사용하여 해당 PDB를 엽니다. 이러한 분자 시각화 프로그램은 구조 생물학 응용 분야에서 널리 사용됩니다. 특정 색상으로 가장 높은 maxCSP(표준 편차의 두 배 이상)를 표시하는 잔기를 선택하여 추정 결합 부위의 위치를 파악합니다.
- 빠른 교환 체제의 경우, 각 지점(Δδobs)에서 1 H-15N HSQC 교차 피크에 대해 관찰된 CSP의 비선형 최소 제곱 적합치에서 해리 상수(KD)를 해당 지점의 특정 단백질[P] 및 리간드[L] 농도와 비교하여 추정합니다.
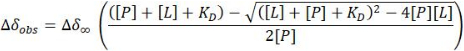
참고: 이 방정식은 명확한 절연 신호를 표시하는 교차 피크에 적용할 수 있습니다. 얻어진 값들은 KD의 추정치를 제공하기 위해 평균화된다.
- 적절한 소프트웨어를 사용하여 apo 종에 대한 처리된 2D HSQC 스펙트럼 시각화: TopSpin, MestReNova 및 CCPNMR은 모두 NMR 데이터 처리에 적합한 프로그램입니다.
결과
여기에서는 렉틴과 작은 올리고당 간의 결합 상호 작용에 대한 세부 사항을 밝히기 위해 1H-STD NMR 및 1 H-15N HSQC 실험을 활용하기 위한 프로토콜을 제시합니다. hGalectin-7(hGal-7)에 의한 LacNAc의 분자 인식 분석에서 얻은 결과가 포함되어 있으며, 이는 프로토콜의 성공적인 구현과 분자 인식 프로세스의 세부 사항을 연구하기 위한 이러한 NMR 방법론의 효과에 대한 예시적인 예시 역할을 합니다. 그림 3은 LacNAc와 hGal-7의 상호 작용에 대한 1H-STD NMR 스펙트럼을 보여줍니다. STD NMR 신호의 존재는 결합을 나타냅니다(그림 3A). 더욱이, 단백질과 밀접하게 접촉하는 양성자에 속하는 신호만 나타나 결합 항원결정기(binding epitope)를 묘사할 수 있습니다(그림 3B). 그림 4는 단백질의 1 H-15N HSQC 스펙트럼을 지문으로 사용할 수 있는 방법을 보여주며, 그림 5는 LacNAc 결합 시 hGalectin-7 백본 아미드 그룹의 화학적 이동 섭동을 정의하기 위해 1 H-15N 이종핵 단일 양자 일관성(HSQC) 적정 실험을 적용하는 방법을 보여줍니다. 이러한 데이터는 상호 작용의 존재를 나타낼 뿐만 아니라 렉틴의 결합 인터페이스를 설명합니다. 그림 6은 적정 데이터 분석을 통해 높은 마이크로몰 범위에 속하는 hGalectin-7에 의한 LacNAc의 결합 친화도를 추정할 수 있는 방법을 보여줍니다. 이 결과는 대체 기술을 사용하여 얻은 결과와 일치합니다.
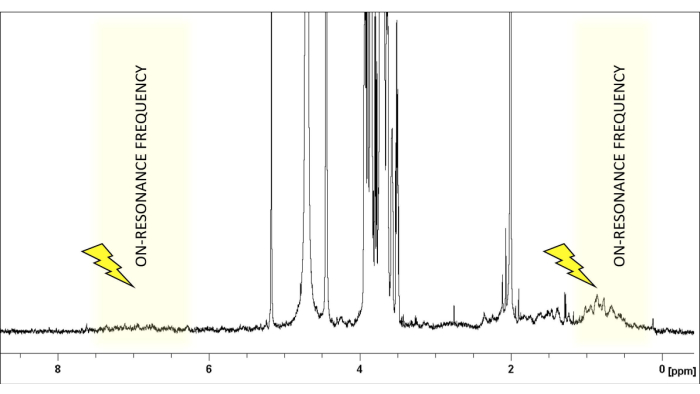
그림 1: 온 공진 주파수의 선택. pH 7.4에서 중수소화 인산염 완충 식염수에서 LacNAc:hGal-7 50:1 비율의 1H-NMR 스펙트럼이 표시됩니다. 리간드(LacNAc)의 신호는 2.0-5.2ppm 사이의 영역에 국한됩니다. 포화 주파수는 1-2ppm 범위 내에서 리간드 양성자가 없는지 확인하기 위해 신중하게 선택되어 단백질의 양성자를 선택적으로 조사할 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
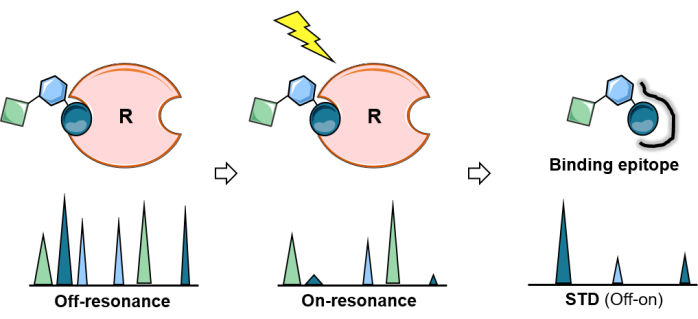
그림 2: STD NMR 실험. STD 실험의 개략도: 첫 번째 스펙트럼(off-resonance)은 참조 역할을 하고 두 번째 스펙트럼(on-resonance)에서는 단백질 포화가 수행됩니다. 포화도는 전체 단백질에 걸쳐 효율적으로 전파되고 단백질과 밀접하게 접촉하는 리간드 양성자로 전달됩니다. 결과 차이 스펙트럼(STD 스펙트럼)은 포화를 경험한 공명만 생성합니다. STD 실험의 분석은 결합 설탕의 epitope 매핑을 허용합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
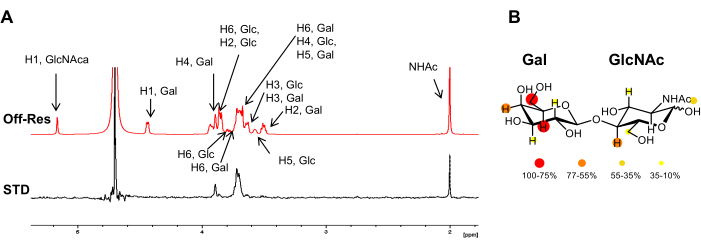
그림 3: 리간드의 관점에서 본 결합 분석. (A) LacNAc와 hGal-7의 상호 작용에 대한 오프 공진 및 1H STD-NMR 스펙트럼의 중첩. STD 스펙트럼에서는 단백질과 밀접하게 접촉하는 양성자에 속하는 신호만 나타납니다. 리간드의 1H공명에 대한 주석은 오프 공명 스펙트럼에서 보고됩니다. (B) 상대적인 STD 강도는 LacNAc의 화학 구조에 컬러 매핑되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
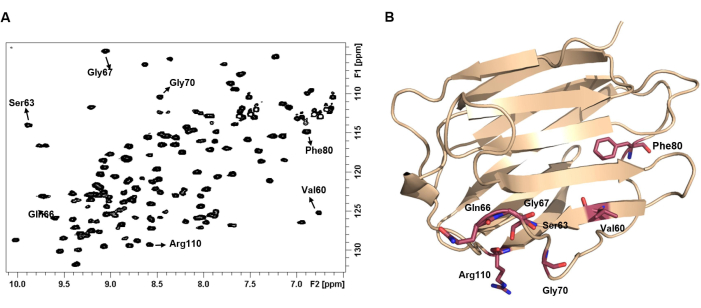
그림 4: 단백질의 1 H-15N HSQC 스펙트럼은 지문을 나타냅니다. (A) apo 형태로 100μM의 hGal-7의 1 H-15N HSQC 스펙트럼. 스펙트럼은 25°C에서 기록되었습니다. 일부 NH cross-peaks는 해당 아미노산의 라벨로 주석을 달았습니다. (B) 각 NH 쌍은 화학적 환경과 결과적으로 단백질의 3D 구조에 따라 달라지는 고유한 화학적 변화를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
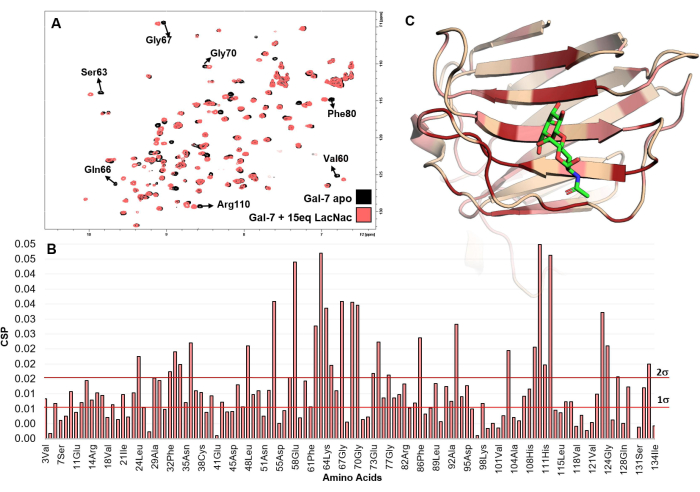
그림 5: 단백질의 관점에서 본 결합 분석. (A) LacNAc를 hGal-7 용액에 적정하기 위해 기록된 1 H-15N HSQC 스펙트럼의 중첩이 표시됩니다. 여러 교차 피크가 화학적 이동 변화를 경험하는 스펙트럼을 검사하면 상호 작용이 명확하게 나타납니다. (B) hGal-7을 사용한 LacNAc(15개 등가물)의 적정에서 추론된 백본 아미드 신호의 최대 화학적 이동 섭동(maxCSP)의 플롯. (C) CSP 분석에 따르면 hGal-7의 가장 교란된 아미노산은 5gal PDB 구조에 매핑됩니다. 3D 모델에서 빨간색은 2σ 이상의 CSP 값을 나타내고 분홍색은 1σ에서 2σ 사이의 값을 나타냅니다. 색상이 지정된 영역은 결합 부위를 나타낼 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
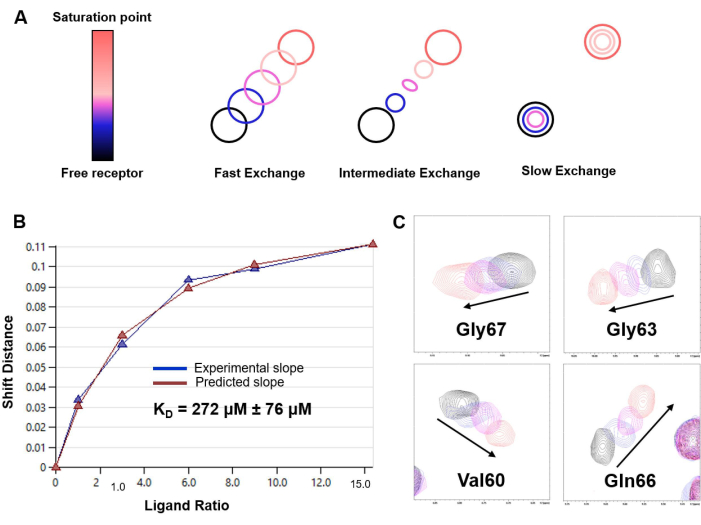
그림 6: 1 H-15N HSQC 적정 실험을 기반으로 한 KD 측정. (A) 연구 시스템의 NMR 시간 척도에서 화학적 교환율에 따른 1 H-15N HSQC 기반 적정의 패턴 표현(빠름, 중간 또는 느림). LacNAc/hGal-7 상호작용의 경우 빠른 교환 체제가 관찰되었습니다. (B) hGal-7 및 LacNAc 이당류의 모델 시스템에 대한 다양한 리간드 농도에서 CSP 분석에서 얻은 피팅 곡선및 KD 추정. 추정된 KD는 20개의 서로 다른 아미노산에 대한 데이터의 평균으로서 해당 오차와 함께 보고됩니다. (C) 적정 중 선택된 교차 피크의 이동을 표시하는 1 H,15N-HSQC 스펙트럼의 스니펫. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
포화 전달 차이 NMR(STD-NMR)은 리간드-단백질 상호 작용을 연구하기 위해 가장 많이 사용되는 다재다능한 NMR 방법이 되었습니다. 위에서 볼 수 있듯이 포화 전달 현상에 의존하며, 실험 설정에는 두 개의 1차원(1D) 1H스펙트럼, 즉 on-resonance¬ 및 ¬off-resonance 스펙트럼의 획득이 포함됩니다. 온 공명 실험 중에 단백질의 특정 양성자의 포화는 특정 기간(포화 시간 범위는 일반적으로 1-3초 범위) 동안 저전력 무선 주파수 펄스 트레인을 적용하여 달성됩니다. 리간드의 직접적인 포화를 피하기 위해, 포화 펄스의 주파수와 길이는 단백질의 특정 양성자를 선택적으로 조사하도록 최적화됩니다. 즉, 모든 리간드 신호의 빈 주파수에 적절한 길이로 적용되어야 합니다(그림 1). 일반적으로 50ms 포화 펄스의 경우 포화 영역에서 가장 가까운 리간드 신호까지 1ppm의 차이를 유지해야 합니다. 일반적으로, 단백질의 지방족 영역에 적용되는 선택적 포화 펄스는 증가된 포화 효과를 제공합니다. 대안적으로, 리간드 분자가 어떠한 방향족 신호도 포함하지 않는 경우 방향족 양성자(6-7 ppm)를 조사할 수 있다. 이것은 방향족 그룹을 가지고 있지 않기 때문에 자연적으로 발생하는 글라이칸에 매우 유용합니다. 단백질의 특정 영역에 선택적으로 조사되면 포화도는 쌍극자 1 H-1H 교차 이완(스핀 확산)을 통해 단백질을 따라 전파됩니다. 결국, 포화도는 결합 부위의 단백질 양성자에 도달하고, 이는 분자간 1 H-1H NOE를 통해 수용체와 밀접하게 접촉(r < 5 Å)되는 당 양성자로 전달됩니다. 분명히, 포화 리간드 양성자의 신호 강도는 감소합니다. 포화를 받은 후 결합 역학으로 인해 일시적으로 결합된 리간드(빠른 교환이 필요함)가 해리되고 포화 정보가 자유 상태로 축적됩니다. 이 과정으로 인해 NMR 온 공명 스펙트럼은 감소된 신호를 나타냅니다(그림 2).
결합 글라이칸의 1H핵의 이러한 강도 섭동을 명확하게 나타내기 위해, 포화가 동일한 조건에서 수용체 또는 탄수화물 신호(보통 40-100ppm 사이)에서 멀리 떨어진 곳에 적용되는 제어 양성자 NMR 스펙트럼(오프 공명)을 획득합니다. off-resonance와 on-resonance 사이의 빼진 1D 스펙트럼은 강도가 수정된 리간드의 1H핵의 신호, 즉 자화를 받을 수 있을 만큼 수용체 결합 부위에 충분히 가까운 신호를 독점적으로 보여줍니다(그림 2).
그럼에도 불구하고 결합 된 탄수화물의 모든 1H핵이 동일한 양의 포화도를받는 것은 아닙니다. 이론적으로, 수용체에서 결합된 리간드로의 자화 전달은 거리에 따라 달라집니다(1/r6). 이는 글리칸 1H핵 사이의 전이 포화 강도가 리간드의 양성자와 수용체의 양성자 사이의 공간적 근접성에 대한 정보를 포함하고 STD NMR 강도가 수용체에 더 가까운 양성자에 대해 더 크다는 것을 의미합니다. 따라서 STD NMR 실험은 단백질 표면에 더 가깝게 위치한 리간드의 양성자가 결합에 직접 참여하지 않는 것보다 더 높은 강도를 보여주기 때문에 탄수화물의 결합 에피토프를 결정할 수도 있습니다(그림 2 및 그림 3).
이 실험은 약한 친화도-중간 친화도를 가진 시스템에 적용할 수 있으며, 낮은 μM 또는 nM 범위에서 강한 친화도를 가진 시스템에는 드물게 적용할 수 있습니다. 실제로, 이완 시간 척도에서 해리 속도가 빨라야 합니다. 그렇지 않으면 리간드가 해리되기 전에 이완을 통해 포화 전달 정보가 손실됩니다.
반면에 단백질 기반 NMR 실험은 원자 분해능 구조를 해결하지 않고 아미노산 수준 정확도로 리간드-단백질 상호 작용을 밝히는 데 독특합니다. 공결정화(co-crystallization) 없이 용액 내 분자 인식 현상을 직접 검사합니다. CSP 분석 매핑은 리간드를 발견하고 단백질 결합 부위를 매핑하는 데 매우 강력합니다(그림 4 및 그림 5). 이 방법은 mM과 nM 범위 사이의 친화도의 모든 범위에 적용 가능하며, 화학적 이동 시간 척도(21)에서 환율이 느린 시스템의 경우에도 적용할 수 있다.
그럼에도 불구하고 이 접근법은 이완 문제로 인해 분자량이 30-40kDa 이상인 단백질에는 효과가 없을 것입니다. 그런 다음 TROSY 대안18 을 사용할 수 있으며, 단백질 중수소화와 결합할 때 특히 강력합니다. 또한, 단백질은 15N으로 균일하게 표지되어야 합니다(그리고 필요한 백본 할당을 완료할 수 있도록 13C 및 15N으로 이중 표지된 또 다른 샘플). 따라서, 해당 발현 시스템을 포함한 단백질 발현 조건은 밀리그램 양의 단백질을 얻을 수 있도록 최적화되어야 합니다. 올리고머화(oligomerize) 또는 응집(응집) 경향을 보이는 단백질도 이 분석에 적합하지 않습니다. NMR 데이터를 기록하기 위해 본 명세서에서 사용되는 기기는 TCI cryoprobe가 장착된 Bruker 800MHz 분광계이다. 이 방법론을 600MHz 미만의 기기를 사용하거나 극저온 프로브 없이 사용하는 것은 매우 어려울 수 있습니다.
공개
저자는 밝힐 것이 없습니다.
감사의 말
MCIN/AEI/10.13039/501100011033가 후원하는 Severo Ochoa Center of Excellence Accreditation CEX2021-001136-S와 Instituto de Salud Carlos III(ISCIII, Madrid, Spain)의 이니셔티브인 CIBERES에 대해 스페인의 Agencia Estatal de Investigación에 감사드립니다. 우리는 또한 GLYCOTWINNING 프로젝트를 위해 유럽연합 집행위원회(European Commission)에 감사를 표합니다.
자료
| Name | Company | Catalog Number | Comments |
| 5 mm Shigemi microtube set mat | CortecNet SAS | S30BMS-005B | |
| Alpha-Lactose-Agarose | Sigma-Aldrich Química S.L. | 7634-5ML | |
| Ammonium chloride (15 N, 99%) | LC-0179-N-50G | Tracer Tecnologías Analíticas S.L | |
| Ampicillin (Sodium Salt) | Melford Laboratories LTD | A40040 | |
| BIOVIA Discovery studio | BIOVIA, Dassault Systèmes | ||
| BL21(DE3) Chemically Competent Cells | Merck Life Science, S.L.U. | CMC0014-40X40UL | |
| Centrifuge | Beckman Coulter | Allegra X-22R | |
| D2O | Cambridge Isotope Laboratories, Inc. | DLM-4-1000 | |
| Incubator | Eppendorf | Innova 42 | |
| IPTG (Isopropyl ß-D-1-thiogalactopyranoside) | VWR International Eurolab S.L. | VW437144N | |
| LacNAc | Elicityl | GLY008 | |
| Luria Bertani (LB) Broth | Merck Life Science, S.L.U. | 3397-1KG | |
| Matraz Erlenmeyer B N 5000 CC | VWR International Eurolab S.L. | 214-1137 | |
| PBS 10x | Bio-Rad | 1610780 | |
| PyMOL | PyMOL Molecular Graphics System | Version 2.0 Schrödinger | |
| Sonicator | Sonics & Materials, Inc. | VC 505 | |
| Superconducting NMR magnet | Bruker | 600 MHz AVANCE III | |
| Superconducting NMR magnet | Bruker | 800 MHz AVANCE III |
참고문헌
- Solís, D., et al. A guide into glycosciences: How chemistry, biochemistry and biology cooperate to crack the sugar code. Biochim Biophys Acta. 1850 (1), 186-235 (2015).
- Poveda, A., Jimenez-Barbero, J. NMR studies of carbohydrate-protein interactions in solution. Chem Soc Rev. 27, 133-144 (1998).
- Kogelberg, H., Solís, D., Jiménez-Barbero, J. New structural insights into carbohydrate-protein interactions from NMR spectroscopy. Curr Opin Struct Biol. 13 (5), 646-653 (2003).
- Widmalm, G. Glycan shape, motions, and interactions explored by NMR spectroscopy. JACS Au. 4 (1), 20-39 (2024).
- Marchetti, R., et al. 34;Rules of Engagement" of protein-glycoconjugate interactions: A molecular view achievable by using NMR spectroscopy and molecular modeling. ChemistryOpen. 5 (4), 274-296 (2016).
- Ardá, A., Jiménez-Barbero, J. The recognition of glycans by protein receptors. Insights from NMR spectroscopy. Chem Commun. 54 (38), 4761-4769 (2018).
- Gimeno, A., Valverde, P., Ardá, A., Jiménez-Barbero, J. Glycan structures and their interactions with proteins. A NMR view. Curr Opin Struct Biol. 62, 22-30 (2020).
- Cañada, F. J., et al. Conformational and structural characterization of carbohydrates and their interactions studied by NMR. Curr Med Chem. 29 (7), 1147-1172 (2022).
- Valverde, P., Quintana, J. I., Santos, J. I., Ardá, A., Jiménez-Barbero, J. Novel NMR avenues to explore the conformation and interactions of glycans. ACS Omega. 4, 13618-13630 (2019).
- Jiménez-Barbero, J., Asensio, J. L., Cañada, F. J., Poveda, A. Free and protein-bound carbohydrate structures. Curr Opin Struct Biol. 9 (5), 549-555 (1999).
- Quintana, J. I., Atxabal, U., Unione, L., Ardá, A., Jiménez-Barbero, J. Exploring multivalent carbohydrate-protein interactions by NMR. Chem Soc Rev. 52 (5), 1591-1613 (2023).
- Roldós, V., Cañada, F. J., Jiménez-Barbero, J. Carbohydrate-protein interactions: a 3D view by NMR. Chembiochem. 12 (7), 990-1005 (2011).
- Unione, L., Ardá, A., Jiménez-Barbero, J., Millet, O. NMR of glycoproteins: profiling, structure, conformation, and interactions. Curr Opin Struct Biol. 68, 9-17 (2021).
- del Carmen Fernández-Alonso, M., Díaz, D., Berbis, M. A., Marcelo, F., Cañada, J., Jiménez-Barbero, J. Protein-carbohydrate interactions studied by NMR: from molecular recognition to drug design. Curr Protein Pept Sci. 13 (8), 816-830 (2012).
- Angulo, J., Nieto, P. M. STD-NMR: application to transient interactions between biomolecules-a quantitative approach. Eur Biophys J. 40 (12), 1357-1369 (2012).
- Mayer, M., Meyer, B. Group epitope mapping by saturation transfer difference NMR to identify segments of a ligand in direct contact with a protein receptor. J Am Chem Soc. 123 (25), 6108-6117 (2001).
- Williamson, M. P. Using chemical shift perturbation to characterise ligand binding. Prog Nucl Magn Reson Spectrosc. 73, 1-16 (2013).
- Fernández, C., Adeishvili, K., Wüthrich, K. Transverse relaxation-optimized NMR spectroscopy with the outer membrane protein OmpX in dihexanoyl phosphatidylcholine micelles. Proc Natl Acad Sci U S A. 98 (5), 2358-2363 (2001).
- Yee, A., Gutmanas, A., Arrowsmith, C. H. Solution NMR in structural genomics. Curr Opin Struct Biol. 16 (5), 611-617 (2006).
- Skinner, S. P., Fogh, R. H., Boucher, W., Ragan, T. J., Mureddu, L. G., Vuister, G. W. CCPNMR AnalysisAssign: a flexible platform for integrated NMR analysis. J Biomol NMR. 66 (2), 111-124 (2016).
- Gimeno, A., et al. Minimizing the entropy penalty for ligand binding: Lessons from the molecular recognition of the histo blood-group antigens by human Galectin-3. Angew Chem Int Ed Engl. 58 (22), 7268-7272 (2019).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유