需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
应用无监督多组学因子分析来揭示与心血管疾病相关的变异模式和分子过程
摘要
我们提出了一种灵活、可扩展的基于 Jupyter 实验室的工作流程,用于复杂多组学数据集的无监督分析,该工作流程结合了不同的预处理步骤、多组学因子分析模型的估计和多种下游分析。
摘要
疾病机制通常很复杂,并受几个不同分子过程的相互作用控制。复杂的多维数据集是生成对这些过程的更多见解的宝贵资源,但此类数据集的分析可能具有挑战性,因为例如,来自不同疾病状况、时间点和组学以不同分辨率捕获该过程的高维结果。
在这里,我们展示了一种以无监督方式分析和探索如此复杂的多组学数据集的方法,方法是将多组学因子分析 (MOFA) 应用于从捕获急性和慢性冠状动脉综合征免疫反应的血液样本生成的数据集。该数据集由不同分辨率的几种分析组成,包括样本水平的细胞因子数据、血浆蛋白质组学和中性粒细胞引物 seq 以及单细胞 RNA-seq (scRNA-seq) 数据。通过测量每个患者和几个患者亚组的几个不同时间点,进一步增加了复杂性。
分析工作流程概述了如何分几个步骤整合和分析数据:(1) 数据预处理和协调,(2) MOFA 模型的估计,(3) 下游分析。步骤 1 概述了如何处理不同数据类型的特征,过滤掉低质量的特征,并对其进行归一化以协调其分布以供进一步分析。第 2 步展示了如何应用 MOFA 模型并探索数据集中所有组学和特征的主要方差来源。第 3 步提出了几种用于捕获模式下游分析的策略,将它们与疾病状况和控制这些条件的潜在分子过程联系起来。
总体而言,我们提出了一种对复杂的多组学数据集进行无监督数据探索的工作流程,以能够识别由不同分子特征组成的主要变异轴,这些变异轴也可以应用于其他环境和多组学数据集(包括示例性用例中介绍的其他分析)。
引言
疾病机制通常很复杂,并受几个不同分子过程的相互作用控制。破译导致特定疾病或控制疾病演变的复杂分子机制是一项具有高度医学相关性的任务,因为它可能会为理解和治疗疾病提供新的见解。
最近的技术进步能够同时在更高分辨率(例如,在单细胞水平上)和各种生物层(例如,DNA、mRNA、染色质可及性、DNA 甲基化、蛋白质组学)上测量这些过程。这导致大型多维生物数据集的生成越来越多,可以对其进行联合分析以生成对基本过程的更多见解。与此同时,以具有生物学意义的方式组合和分析不同的数据源仍然是一项具有挑战性的任务1。
不同组学之间的不同技术限制、噪音和变异范围构成了一个挑战。例如,单细胞 RNA 测序 (scRNA-seq) 数据非常稀疏,并且经常受到大型技术或批次效应的影响。此外,特征空间通常非常大,范围涵盖数千个测量基因或蛋白质,而样本量有限。复杂的设计使情况更加复杂,其中可能包括多种疾病状况、混杂因素、时间点和分辨率。例如,在所展示的用例中,在单单元或样本(批量)级别上提供了不同的数据类型。除此之外,数据可能不完整,并且并非所有测量结果都适用于所有分析对象。
由于这些挑战,不同的组学和包含的特征通常仍然只单独分析2,即使执行综合分析不仅可以提供完整的过程图,而且来自一个组学的生物学和技术噪音也可能被其他组学补偿 3,4。已经提出了几种不同的方法来对多组学数据进行综合分析,包括贝叶斯方法、基于网络的方法 5,6、多模态深度学习7 和通过矩阵分解的降维方法 8,9。对于后者,一项大型基准研究10 的结果表明,当数据应与临床注释相关联时,MOFA9(多组学因子分析)方法是更适合的工具之一。
特别是在复杂的环境中,无监督矩阵分解方法是降低复杂性并从不同数据源和特征中提取共享和互补信号的有用方法。通过将复杂空间分解为较低等级的潜在表示,可以快速探索数据中方差的主要来源并将其与已知的协变量相关联。如果多个特征(例如,基因或蛋白质)共享相同的变异模式,则可能会将其聚合为几个因子,同时降低噪声。正则化可用于增加模型系数的稀疏性,这使得该方法非常适合于特征空间较大而样本数量有限9 的设置。
该协议提供了一个灵活的分析工作流程,该工作流程使用 MOFA 模型来展示如何快速探索复杂的多组学数据集并提取表征该数据集的主要变化模式。该工作流包括三个主要步骤。在第一步,数据预处理和协调中,提出了基于不同输入数据类型(scRNA-seq、蛋白质组学、细胞因子、临床数据)的数据预处理的不同策略。该协议详细说明了如何处理不同输入数据集的特征,过滤掉低质量的特征,并对其进行归一化以协调它们的分布。我们还展示了这些预处理决策如何影响下游结果。在第二步中,将 MOFA 模型应用于数据,所得的方差分解可用于评估不同数据集的整合。第三步演示如何将捕获的因子与协变量联系起来,并揭示定义这些因子的分子程序。通过提出的工作流程,我们能够在冠状动脉综合征患者数据集中提取与临床协变量相关的几个潜在因素,并从以前的项目11 中确定潜在的潜在多细胞免疫程序。我们将在这里使用这个数据集,但该协议可以很容易地应用于其他环境,包括其他组学。
该数据集包括来自稳定期慢性冠脉综合征 (CCS)、急性冠脉综合征 (ACS) 和冠状动脉健康对照组(非 CCS)的样本(图 1)。ACS 是由先前存在的 CCS 中的斑块破裂引起的,导致流向心肌的血流急性中断,随后导致心脏缺血性损伤。这种损伤会引起免疫系统的炎症反应,然后是修复期,一直持续到急性事件发生后的几天12。为了能够描述 ACS 患者的这种免疫反应,在四个不同的时间点采集了血样:急性 (TP1);再通后 (14 [± 8] h) (TP2);60 [± 12] 小时后 (TP3);出院前(6.5 [±1.5] 天)(TP4)(图 1A)。对于 CCS 和健康冠状动脉患者,只有一个时间点可用 - (TP0)。对于所有患者和时间点,根据血样测量不同的检测方法:炎症的临床标志物 (肌酸激酶 (CK)、CK-MB、肌钙蛋白、C 反应蛋白 (CRP))、外周血单核细胞 (PBMC) 的 scRNA-seq、细胞因子分析、血浆蛋白质组学和中性粒细胞的 prime-seq13 数据。
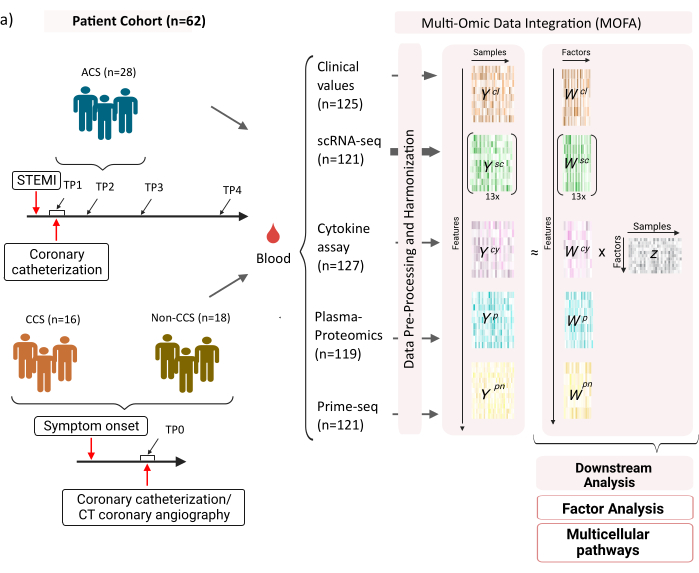
图 1:心肌梗死多组学输入数据集。 输入数据集:分析的数据包括急性冠脉综合征 (ACS)、慢性冠脉综合征 (CCS) 患者 (n = 62) 和健康冠状动脉 (non-CCS) 患者的血液样本。对于 ACS 患者,包括 4 个不同时间点 (TP1-4) 的血样,CCS 和非 CCS 患者的血样在单个时间点 (TP0)。在分析中,每个患者和时间点组合都被视为单独的样本。对样本测量不同的组学检测:临床血液检测 (n = 125)、scRNA-seq (n = 121)、血浆蛋白质组学 (n = 119)、细胞因子检测 (n = 127) 和中性粒细胞引物测序 (n = 121)。随后,应用所描述的方案来整合所有组学的数据,并使用 MOFA 模型和进一步的下游分析 (因子分析、途径富集) 对其进行探索。 请单击此处查看此图的较大版本。
作为此处介绍的工作流程的输入,我们在使用 cellranger 和质量控制 (QC) 处理后从 scRNA-seq 数据中提取原始计数,例如,如 scanpy14 预处理教程中所述。对于单元类型注释,我们使用了自动化的 Azimuth15 管道。然后,通过对每个样本和细胞类型的所有细胞进行平均值(伪批量聚合),在样本水平上聚合每种细胞类型的计数。血浆蛋白质组学包括归一化和以中位数为中心的强度,对于中性粒细胞,我们从 prime-seq 中获取 umi 唯一分子标识符 (UMI) 外显子计数。关于细胞因子和临床值,之前没有应用任何预处理。有关(实验)数据生成的更多细节,请参见相应的手稿11。由于此处提供的结果基于对 scRNA-seq 数据中的细胞类型使用自动 Azimuth 注释,与参考出版物中使用的基于标记的策略相比,此处提供的结果与出版物中提供的结果相似但不完全相同。在手稿中可以表明,细胞类型注释策略不会改变分析的主要模式和生物学解释,但模型导致的确切值的微小变化可能会有所不同。总体而言,输入数据是一个复杂的多维数据集,包括 10,000 多个不同特征(基因、蛋白质、临床值)的不同时间点和测量水平(单细胞与大量)。严格的预处理和数据协调策略,然后进行 MOFA 分析已被证明是探索数据和提取相关免疫程序的有用且快速的工具。在 MOFA 分析中,每个时间点和患者组合都被视为独立样本。在 MOFA 分析中,每种数据类型和单元格类型都被视为一个单独的视图。
此协议提供了为工作流准备输入数据、执行不同的工作流步骤、自定义配置、解释结果数字以及根据解释迭代调整配置的说明。技术工作流程概述概述了协议的不同步骤、每步所需的输入数据集以及生成的图形和数据集(图 2)。
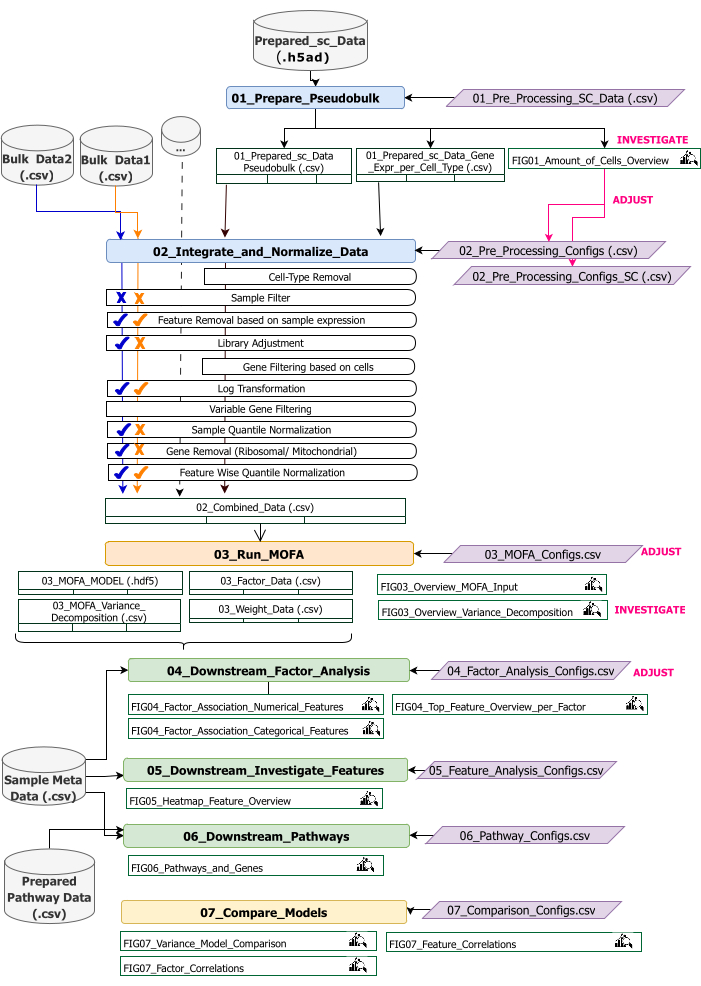
图 2:技术工作流程概述。 多组学数据集分析的工作流程概述。不同的元素由不同的颜色和符号突出显示。属于 Data Preprocessing and Harmonization (1) 步骤的 Jupyter Notebook 以蓝色显示。属于“MOFA 模型”(2) 步骤的 Jupyter Notebook 以橙色显示。属于“Downstream Analysis”(下游分析) (3) 步骤的 Jupyter Notebook 以绿色显示。一个用于比较结果的 Jupyter Notebook 为黄色。可以修改工作流执行参数的配置文件以紫色突出显示。运行工作流所需的输入数据集由数据集符号指示,并以灰色突出显示。在工作流执行期间生成的所有图窗输出都由放大镜符号指示。工作流程执行期间生成的数据集表示为表。通常,工作流程是按顺序执行的:(1) 数据预处理和协调包括两个步骤:首先基于 scRNA-seq 输入数据 (01_Prepare_Pseudobulk) 生成伪批量表,随后将此数据与所有其他样本级(批量)输入 (02_Integrate_and_Normalize_Data) 一起集成和标准化。在此步骤中,通过配置文件,可以为每个数据集单独配置应应用哪些指示的预处理和规范化步骤(例如,样本过滤器)。(2) “MOFA 模型”:使用配置文件中指定的配置在第一步生成的输入上运行 MOFA 模型 (03_MOFA_configs.csv) (3) “下游分析”:由三个不同的笔记本组成,它们可以彼此独立运行,以生成对生成的 MOFA 结果的见解,并将它们与通过“样本元Data.csv”文件作为输入提供的样本元数据(协变量)相关联。(4) “模型比较”:是一个单独的小步骤,可用于比较步骤 2 中生成的不同模型。 请单击此处查看此图的较大版本。
该工作流由多个用 R 和 Python 编写的 Jupyter Notebook 组成(运行工作流不需要 R 和 Python 语言知识,但在出现错误时可能会有所帮助)。在协议的各个步骤中,参数通过配置文件(名称中包含后缀 '_Configs' 的 '.csv' 文件)进行更改。在协议中,我们只概述了从默认配置开始需要更改的参数。
还可以更改其他几个参数,例如自定义预处理。这些文件 'Documentation_Config_Parameter' 中提供了这些参数和解释的文档,该文件包含在下载的存储库中。
研究方案
1. 准备工作:技术设置和安装
注意:要运行此程序,请在设备上预装 wget、git 和 Apptainer。在不同系统(Linux、Windows、Mac)上安装 Apptainer 的指南如下:https://apptainer.org/docs/admin/main/installation.html。有关 git 的安装信息,请访问:https://git-scm.com/book/en/v2/Getting-Started-Installing-Git。根据不同输入数据集的大小,建议在合适的计算机(16 个 CPU,64GB 内存)上运行工作流。可以使用提供的示例数据在本地计算机上执行冒烟测试。补充文件 1 中给出了对示例数据运行协议的说明和预期输出。有关在上述数据集上执行的协议的重要步骤,请参阅 补充视频文件 1 。
- 打开控制台,然后选择或创建一个文件夹,用于存储所有分析代码和输出。通过键入命令导航到文件夹:在终端中 cd path_to_folder。
- 从 Github (https://github.com/heiniglab/mofa_workflow) 下载或克隆代码存储库,或者在终端窗口中键入 git clone https://github.com/heiniglab/mofa_workflow.git 。
- 通过在终端窗口中键入 wget https://zenodo.org/records/11192947/files/mofa_image.sif,从 Zenodo 下载包含所有必需安装的映像。
- 通过在终端窗口中键入 mkdir results 生成一个文件夹,所有结果数据都将存储在其中。
- 通过在终端窗口中键入 mkdir input_data 生成一个文件夹,其中将添加要在分析中使用的所有输入数据。
- 通过在终端中键入以下命令,执行将启动 JupyterLab 会话的容器: apptainer run mofa_image.sif。将命令返回的 URL 复制到浏览器,这将打开 Jupyter-lab 会话(有关 Jupyter-lab 的更多信息,请参阅软件文档16)。
注意:在笔记本电脑上本地执行工作流时,建议使用命令 apptainer exec mofa_image.sif jupyter-lab ,该命令将直接返回本地主机地址。如果容器在集群计算环境中执行,则可能需要设置端口转发,这可以通过 ssh 完成。
2. 初始化和数据准备
- 在 Jupyter-Lab 会话中,使用左侧的导航菜单。双击 input_data 文件夹 input_data 导航到该文件夹。
- 使用 Drag&Drop 将所有将用作分析输入的数据集复制到 input_data 目录。将文件从当前所在的文件夹中拖放到 Jupyter-lab 会话中 input_data 文件夹下方的区域。
注意:所有数据集都需要采用 .csv 或 .h5ad (对于单单元格数据)格式。所有 .csv 文件都必须包含匹配的 sample_id 列(必须在数据集中使用相同的 ID)。所有其他列将用作特征。在 h5ad- 文件中,单元格注释必须包含两个标识符,分别指定 sample_id 和 cluster_id。这些将用于聚合和匹配。使用前需要将其他格式的 Omic 数据集转换为指定的 .csv 格式(图 3)。以 .h5seurat 格式给出的 scRNA-seq 数据集可以转换为 .h5ad 执行 Jupyter-notebook: 00_Data_Conversion.ipynb。 - 通过单击文件夹符号,然后双击文件夹 mofa_workflow、 scripts 和 configurations 导航到 configurations 文件夹。在文件夹中,双击打开文件Data_configs.csv。
- 在 value 列中,将路径添加到 input_data (data_path) 和 results (result_path) 文件夹。在configuration_name的值列中添加一个名称,该名称将作为文件扩展名添加到所有保存的文件(该协议MI_v1 [Myocardial Infarction version1])(图 4)。
- 通过单击顶部菜单中的 文件 > 保存 CSV 文件 来保存更改。
- 使用左侧的导航菜单,通过单击 scripts 导航到 scripts 文件夹。双击 00_Configuration_Update.ipynb 打开初始化笔记本。通过单击顶部的 Restart kernel and run all cells 按钮,然后单击弹出窗口中的 Restart 来执行脚本(图 5)。
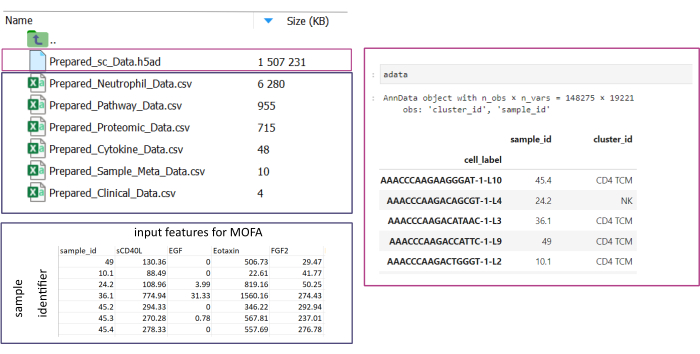
图 3:数据输入和设置。 为了执行工作流,所有数据都需要存储在指定的 input_data 文件夹中。对于每个输入数据集,应提供一个单独的文件。单细胞数据应以 .h5ad 的形式给出,其中包含cluster_id上的细胞注释(例如,来自先前的细胞类型注释步骤的结果)和sample_id列(唯一标识应分析的每个单独样本)。所有其他输入数据集应以 “.csv” 格式给出,包括一列指定sample_id(与单细胞数据的相应列匹配)和所有其他列中要用于 MOFA 分析的特征。 请单击此处查看此图的较大版本。
图 4:Jupyter-lab 配置文件。 在工作流执行期间,参数的更改(例如,调整过滤选项等)是通过“.csv”配置文件指定的。在克隆的存储库中,包含每个步骤的默认配置文件。可以直接在 jupyter-lab 控制台中编辑它们,就像在电子表格中一样。 请单击此处查看此图的较大版本。
图 5:Jupyter-notebooks 脚本。 完整的工作流由一系列 Jupyter 笔记本组成,这些笔记本将在修改相应的配置文件后按顺序执行。双击左侧的 Jupyter 笔记本,相应的文件将在右侧打开。文件的完整执行可以通过顶部突出显示的按钮开始。 请单击此处查看此图的较大版本。
3. 数据预处理和协调
- 预处理 - 将 sc 数据转换为 pseudobulk。
注意:仅当分析中使用单单元格数据时,才需要执行此步骤。- 使用左侧的导航菜单,双击 configuration 导航到 configurations 文件夹。双击打开文件 01_Preprocessing_SC_Data.csv 。检查文件中自动填充的值,如有必要,调整 data_name 列中的值,以对应于将用于分析的 input_data 文件夹中单单元数据集的文件名。
注意:默认情况下,input data 文件夹中 .h5ad 文件的所有名称都将添加到初始化脚本的配置文件中。如果某些数据集不应用于分析,则可以在此处删除它们。 - 通过单击顶部菜单中的 文件 > 保存 CSV 文件 保存所做的更改。
- 使用左侧的导航菜单,通过单击 scripts 导航到 scripts 文件夹。双击 notebook 01_Prepare_Pseudobulk.ipynb 打开它。通过单击顶部的 Restart kernel and run all cells 按钮,然后单击弹出窗口中的 Restart 来执行脚本。
- 使用左侧的导航菜单导航到 figures 文件夹,方法是先双击 figures,然后双击 01_figures。双击新生成的绘图FIG01_Amount_of_Cells_overview
注意:执行笔记本可能需要几分钟时间。 成功执行 notebook 后,将出现一个弹出窗口,文件 FIG01_Amount_of_Cells_Overview 将由 notebook 更新或重新生成。Last Modified (上次修改时间 ) 列可以指示文件的生成时间,以评估它是新文件还是旧文件。 - 研究该图以识别每个样品细胞数量非常少的细胞型簇。记下这些cluster_ids的名称,以便在后续步骤中将其排除(图 6)。
- 使用左侧的导航菜单,通过单击 ...,然后双击 Configurations。双击打开文件 02_Preprocessing_Configs_SC.csv 。
- 检查 configuration_name 和 data_name 列中的值,并根据需要进行调整。
注意:在初始化脚本中,这些值预先填充了输入数据文件夹中 .h5ad 文件的所有名称以及之前在 Data_Configs.csv 文件中设置的 configuration_name 值。如果应从分析中排除文件或应使用其他文件名扩展名,则可以在此处进行调整。 - 调整 cell_type_exclusion 列中的值,并添加在上一步中标识要排除的所有cluster_id,以 ',' 分隔。
- 通过单击顶部导航栏中的 File > Save CSV File 来保存更改。
- 使用左侧的导航菜单,双击 configuration 导航到 configurations 文件夹。双击打开文件 01_Preprocessing_SC_Data.csv 。检查文件中自动填充的值,如有必要,调整 data_name 列中的值,以对应于将用于分析的 input_data 文件夹中单单元数据集的文件名。
- 预处理 - 协调和整合其他组学数据源。
- 双击打开文件 02_Preprocessing_Configs.csv ,并调整将包含并存储在 data_input 文件夹中的每个数据集的预处理配置(每个数据集一行)。
- 检查 configuration_name 和 data_name 列中的值,并根据需要进行调整。
- 根据应应用的预处理步骤,相应地调整列中的其他参数。
注意:将为 input_dataset 文件夹中找到的每个数据集添加默认值,但不特定于数据的单个数据类型。因此,调整将是必要的。 Documentation_Config_Parameter.doc 文件中给出了参数的详细文档。 - 通过单击 File > Save CSV File.
- 使用左侧的导航菜单,通过单击 scripts 导航到 scripts 文件夹。双击 notebook 02_Integrate_and_Normalized_Data_Sources.ipynb 打开它。通过单击顶部的 Restart kernel and run all cells 按钮,然后单击弹出窗口中的 Restart 来执行脚本。
- 使用左侧的导航菜单导航到生成的 02_results 文件夹,方法是单击 文件夹 符号,然后双击 results 和 02_results。检查是否包含包含组合预处理数据输入文件的 02_Combined_data_'configuration_name'_Integrated.csv 文件。
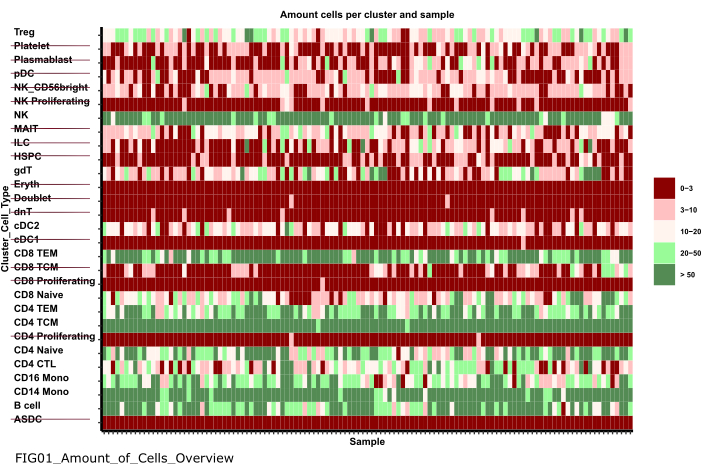
图 6:数据预处理和协调。 '01_Prepare_Pseudobulk' 步骤的一个输出是图 'Fig01_Amount_of_Cells_Overview'。在这里,对于每个cluster_id(y 轴表示先前细胞类型注释步骤中的细胞类型),给出了每个样本的细胞数 ('sample_id')。在呈现的结果中,每个样品中细胞数量较少的细胞类型被排除在后续分析之外(由删除线表示)。 请单击此处查看此图的较大版本。
4. 运行 MOFA
- 在 Jupyter-Lab 中,使用左侧的导航菜单导航到配置文件夹,方法是单击 文件夹 符号,然后双击 mofa_workflow,然后双击 脚本 和 配置。双击打开文件 03_MOFA_Configs.csv 。
- 检查 configuration_name 和 mofa_result_name 列的条目,并调整应使用替代名称的条目。
注意: mofa_result_name 将作为文件扩展名附加到基于 MOFA 生成的所有结果文件中。这可能与 configuration_name 值不同,因为不同的 MOFA 设置可能使用相同的 input 数据运行(此协议使用 MI_v1_MOFA)。 - 输入应在 MOFA 模型(amount_of_factors 列)中估计的因子数量,并通过调整文件中的值来定义是否应应用加权和缩放(weighting_of_views 列和 scale_views 列)。
- 通过单击 File > Save CSV File.
- 使用左侧的导航菜单,通过单击“scripts”导航到 scripts 文件夹。双击 notebook 03_Run_MOFA.ipynb 打开该文件。通过单击顶部的 Restart kernel and run all cells 按钮并单击弹出窗口中的 Restart 来执行脚本。
- 通过双击图窗,然后03_figures导航到 03_figures 文件夹。打开生成的图FIG03_Overview_Variance_Decomposition_mofa_result_name并研究模型结果(图 7A)。
- 使用左侧的导航菜单导航到生成的 03_results 文件夹,方法是单击 文件夹 符号,然后双击 results 并 03_results。检查是否包含样本因子值文件 03_Factor_Data_'mofa_result_name'.csv 和特征因子权重文件 03_Weight_Data_'mofa_result_name'.csv。
5. 下游分析
- 因素解释。
- 使用左侧的 导航 菜单,通过单击文件夹符号,然后双击 input_data 导航到 input_data 文件夹。
- 准备一个.csv文件 (Prepared_Sample_Meta_Data.csv),其中包含将与生成的因子相关联的样本的所有元数据(协变量)。使用 Drag&Drop 将文件拖放到 input_data 文件夹的文件夹概览中,将文件复制到 input_data 文件夹。
注意:它需要包含 sample_id 列,以便将其与以前使用的数据进行匹配,并包含应分析的每个特征的更多列。 - 在 Jupyter-Lab 中,使用左侧的导航菜单导航回配置文件夹,方法是单击 文件夹 符号,然后双击 mofa_workflow,然后双击 脚本 和 配置。双击打开文件 04_Factor_Analysis.csv 。
- 检查 configuration_name 和 mofa_result_name 的条目是否包含将在脚本中分析的配置和 MOFA 结果的名称,并在必要时进行调整。
- 在 numeric_covariates 列中,添加 Prepared_Sample_Meta_Data.csv 文件中所有数字列的名称,这些列将与逗号分隔的 MOFA 因子进行调查(此协议使用 CRP,CK)。
- 在 categorical_covariates' 列中,添加 Prepared_Sample_Meta_Data.csv 文件中所有分类列的名称,这些列将与逗号分隔的 MOFA 因子进行调查(此协议使用 测量)。
- 通过单击 File > Save CSV File.
- 使用左侧的导航菜单,通过单击 脚本导航到 'scripts' 文件夹。双击笔记本 04_Downstream_Factor_Analysis.ipynb 打开它。通过单击顶部的 Restart kernel and run all cells 按钮并单击弹出窗口中的 restart 来执行脚本。
- 使用左侧的导航菜单,双击 figures 然后 04_figures 导航到 04_figures 文件夹。双击生成的绘图打开它们,并调查感兴趣的模式和关联的因素:FIG04_Factor_Association_with_numeric_features_
'mofa_result_name.pdf(图 7B)。FIG04_Factor_Association_
with_categorical_features_ mofa_result_name.pdf(图 7C)。FIG04_Top_Feature_Overview_per_Factor _'mofa_result_name.pdf(图 8A)。
- 特征分析
- 使用左侧的导航菜单,单击 ... ,然后双击 Configurations。双击打开文件 05_Feature_Analysis_Configs.csv 。
- 检查 configuration_name 和 mofa_result_name 列的条目是否与配置和生成的 MOFA 结果的名称相对应,该结果将用于下游分析,并根据需要进行调整。
- 在 因子 列中,添加将在下一个脚本中绘制主要特征的因子。
- 在列 faceting_variable中,添加 Prepared_Sample_Meta_Data中分类列的列名.csv该列将用于对图中的样本进行分组(此协议使用 测量)
- 通过单击 File > Save CSV File.
- 使用左侧的导航菜单,通过单击 scripts 导航到 scripts 文件夹。双击 notebook 05_Downstream_Investigate_Features_Heatmap.ipynb 打开它。通过单击顶部的 Restart kernel and run all cells 按钮,然后单击弹出窗口中的 Restart 来执行脚本。
- 使用左侧的导航菜单导航到 05_figures 文件夹,方法是先双击图窗,然后双击 05_figures。打开并调查生成的绘图 FIG05_Heatmap_Feature_Overview__ 'mofa_result_name' 。pdf 通过双击文件(图 8B)。
注意:根据图中显示的特征数量,可能需要调整05_Feature_Analysis_Configs.csv中的参数 plot_width 和 plot_height 并重新运行脚本以确保所有内容都适合图。
- 通路分析
- 使用左侧的 导航 菜单,通过单击文件夹符号,然后双击 input_data 导航到 input_data 文件夹。
- 准备一个 .csv 文件 (Prepared_Pathway_Data.csv),其中包含将进行富集测试的通路列表。使用 Drag&Drop 将文件拖放到 input_data 文件夹的文件夹概览中,将文件复制到 input_data 文件夹。
注意:它需要包含三列: ID (通路的唯一标识符)、 gene (属于通路的基因名称 (SYMBOL) 给出的基因,每个基因一行) pathway_name (通路的名称/文本描述)。 - 在 Jupyter-Lab 会话中,使用左侧的导航菜单通过单击 ...,然后双击 Configurations。双击打开文件 06_Pathway_Configs.csv 。
- 检查 mofa_result_name 列的条目,并确保它与生成的 MOFA 结果的名称相对应,该结果将用于通路富集计算。
- 检查 types 列中的条目,并删除 types 列中不包含与 Prepared_Pathway_Data.csv 文件中的 gene 列匹配的特征的条目。
注意:默认情况下,在工作流执行期间,MOFA 模型中使用的所有不同视图都会添加到此文件中。如果存在不包含与至少一条路径匹配的要素的视图,则需要将其删除;否则,执行将失败。一个例子是通路文件仅包含基因的通路注释,但有一个包含蛋白质名称的视图。 - 通过单击 File > Save CSV File.
- 使用导航菜单通过单击 scripts 导航到 scripts 文件夹。双击 notebook 06_Downstream_Pathways.ipynb 打开它。通过单击顶部的 Restart kernel and run all cells 按钮,然后单击弹出窗口中的 Restart 来执行脚本。
- 使用左侧的导航菜单导航到 06_figures 文件夹,方法是先双击 图 窗,然后 双击06_figures。双击生成的图 FIG06_Pathways_and_Genes_ ' mofa_result_name 打开它,并研究可视化的途径(图 8C)。
注意:可以通过配置文件配置如何选择可视化路径。有关更多详细信息,请参阅参数的文档。 - 使用左侧的导航菜单导航到生成的 06_results 文件夹,方法是单击 文件夹 符号,然后双击 results 和 06_results。检查它是否包含包含丰富通路 06_Pathway_enrichment__'mofa_result_name' 的文件。
6. 比较不同的配置和版本(补充图 1、补充图 2、补充图 3、补充图 4)
- 要比较在整个工作流中使用不同参数/配置的效果,请重新运行第 3-5 节,修改配置文件中的参数并使用不同的 configuration_name 和 mofa_result_name 标识符。
注意:新结果将与这些名称一起存储,以用于比较不同的运行。 - 在 Jupyter-Lab 中,使用左侧的导航菜单导航到 configurations 文件夹。双击打开文件 07_Comparison_Configs.csv 。
- 在 mofa_result_name 列中,添加将要比较的所有先前 MOFA 运行的名称(每个名称/配置一行,例如 MI_v1_MOFA、MI_v2_MOFA)。
- 在 compare_factors 列中,添加将在模型之间进行比较的因子。默认情况下,它是 Factor1,Factor2,Factor3。(补充图 2A)。
注意:在此脚本中,将通过关联不同模型的特征值和因子值来比较它们。这仅适用于基于相同样本(由 sample_id 表示)和相同特征集的模型。如果样本或特征在比较版本之间不匹配,它们将被排除在比较之外。 - 通过单击 File > Save CSV File.
- 使用左侧的菜单通过单击 scripts 导航到 scripts 文件夹。双击 notebook 07_Compare_Models.ipynb 打开它。通过单击 Restart kernel and run all cells 按钮并单击弹出窗口中的 Restart 来执行脚本。
- 使用左侧的菜单导航到 06_figures 文件夹,方法是先双击 图 窗,然后 双击06_figures。双击文件打开生成的绘图,以分析不同版本的相似性:
FIG07_Variance_Model_Comparison.pdf(补充图 2B)
FIG07_Factor_Correlations.pdf
(补充图 2C)
FIG07_Feature_Correlations.pdf
(补充图 3C)
7. 扩展工作流:添加其他参数和配置
注意:除了当前可在配置文件中配置的参数外,还可能包括代码中的其他调整或其他参数。例如,MOFA 模型本身提供了几个其他训练参数17 ,这些参数可以直接在代码中修改,也可以通过配置文件进行调整。该协议的下一部分将概述如何为其他 MOFA 模型训练参数执行此操作的示例。对于这部分,需要 R 编程知识。
- 在 Jupyter-Lab 中,使用左侧的导航菜单导航到 scripts 文件夹。双击 notebook 03_Run_MOFA.ipynb 打开它。
- 单击左侧的 Table of Contents 选项卡,然后导航到子部分 4.3 设置 MOFA 训练选项,然后单击它运行模型训练 。向下滚动以查看笔记本中可配置参数的 MOFA 模型的打印输出。
- 在标题下方代码的 R for 循环中,设置了所有 MOFA 数据、模型和训练选项。在 model_opts$num_factors = mofa_configs$amount_of_factors[i] 行下方,添加另一行代码
model_opts$likelihoods['data_type'] = '泊松'。
注意:这将更改模型作为所有 MOFA 运行的名称 data_type 所指定视图的输入的分布。为数据类型指定泊松时,仅当此数据类型的特征为整数(例如,从 RNA-seq 读取计数)时,模型才会运行。要获取有关 MOFA 数据、训练和模型选项的更多信息,还可以参考 MOFA 教程和文档17。 - 通过单击顶部的 Save (保存 ) 按钮,将更改保存在笔记本中。
- 要通过 .csv 配置文件移交新参数,请使用左侧的导航通过双击 configurations 导航到 configurations 文件夹,然后通过双击打开文件 03_MOFA_Configs.csv 。
- 添加一个指定参数名称的新列(例如 number_iterations 并输入一个值,例如 1000。通过单击 File > Save CSV File.
- 使用导航菜单通过单击 scripts 来导航 scripts 文件夹。双击 notebook 03_Run_Mofa.ipynb 打开它。单击左侧的 Table of Contents 选项卡,然后导航到子部分 4.3 设置 MOFA 训练选项,然后单击它运行模型训练 。
- 将行 train_opts$maxiter = 50000 替换为 train_opts$maxiter = mofa_configs$column_name[i] (当添加的列的名称 number_of_iterations 时,它是 train_opts$maxiter = mofa_configs$number_of_iterations[i])。
注意:此笔记本中的配置文件 03_MOFA_Configs.csv在笔记本的开头(子部分:先决条件配置和参数)作为会话中的 mofa_config data.frame 读取,因此,在这行代码中,此对象和相应的新生成列被引用。由于可以同时运行多个配置, 因此 i 标识 data.frame 的行,因为模型估计在 for 循环中跨 .csv 文件中的所有不同行运行。在笔记本开头的“先决条件、配置和参数'部分读取配置文件的原则对所有笔记本都是相同的,可以像这样进行进一步的修改。 - 单击 Save (保存) 按钮,将更改保存在笔记本中。
结果
成功执行工作流后,将生成多个表格和数字,如图 2 所示。图表放置在 /figures 文件夹中(图 6、 图 7、 图 8、 补充图 1、 补充图 2、 补充图 3、 补充图 4),表格将放置在指定的 /results 文件夹中。
讨论
通过概述的协议,提出了一个模块化且可扩展的基于 Jupyter 笔记本的工作流程,可用于快速探索复杂的多组学数据集。工作流程的主要部分包括预处理和数据协调部分(为数据过滤和标准化提供不同的标准步骤)、MOFA9 模型的估计和一些示例性的下游分析。主要的关键步骤之一是预处理、整合和协调不同的组学数据集。在这里,我们提出了一种数据集策...
披露声明
作者声明没有利益冲突。
致谢
C.L. 得到了亥姆霍兹协会在联合研究学院“慕尼黑数据科学学院 - MUDS”下的支持。
材料
| Name | Company | Catalog Number | Comments |
| Apptainer | NA | NA | https://apptainer.org/docs/admin/main/installation.html |
| Compute server or workstation or cloud (Linux, Mac or Windows environment). Depending on the size of the different input datasets we recommend running the workflow on a suitable machine (in our setting we use: 16 CPU, 64GB Memory) | Any manufacturer | 16 CPU, 64GB Memory | Large Memory is only required for the processing of the raw single cell data. After preprocessing the later analysis steps can also be performed on regular desktop or laptop computers |
| git | NA | NA | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git |
| GitHub | GitHub | NA | https://github.com/heiniglab/mofa_workflow |
参考文献
- Lähnemann, D., et al. Eleven grand challenges in single-cell data science. Genome Biol. 21 (1), 31 (2020).
- Colomé-Tatché, M., Theis, F. J. Statistical single cell multi-omics integration. Curr Opin Syst Biol. 7, 54-59 (2018).
- Hawe, J., Theis, F., Heinig, M. Inferring interaction networks from multi-omics data. Front Genet. 10, 535 (2019).
- Hawe, J. S., et al. Network reconstruction for trans acting genetic loci using multi-omics data and prior information. Genome Med. 14 (1), 125 (2022).
- Koh, H. W. L., Fermin, D., Vogel, C., Choi, K. P., Ewing, R. M., Choi, H. iOmicsPASS: network-based integration of multiomics data for predictive subnetwork discovery. NPJ Syst Biol Appl. 5, 22 (2019).
- Ogris, C., Hu, Y., Arloth, J., Müller, N. S. Versatile knowledge guided network inference method for prioritizing key regulatory factors in multi-omics data. Sci Rep. 11, 6806 (2021).
- Lee, C., vander Schaar, M. A variational information bottleneck approach to multi-omics data integration. Proceedings of The 24th International Conference on Artificial Intelligence and Statistics. 130, 1513-1521 (2021).
- Singh, A., et al. DIABLO: an integrative approach for identifying key molecular drivers from multi-omics assays. Bioinformatics. 35 (17), 3055-3062 (2019).
- Argelaguet, R., et al. Multi-omics factor analysis-a framework for unsupervised integration of multi-omics data sets. Mol Syst Biol. 14 (6), e8124 (2018).
- Cantini, L., et al. Benchmarking joint multi-omics dimensionality reduction approaches for the study of cancer. Nature Commun. 12 (1), 124 (2021).
- Pekayvaz, K., et al. Multiomic analyses uncover immunological signatures in acute and chronic coronary syndromes. Nature Medicine. 30 (6), 1696-1710 (2024).
- Swirski, F. K., Nahrendorf, M. Cardioimmunology: the immune system in cardiac homeostasis and disease. Nat Rev Immunol. 18 (12), 733-744 (2018).
- Janjic, A., et al. Prime-seq, efficient and powerful bulk RNA sequencing. Genome Biol. 23 (1), 88 (2022).
- Wolf, F. A., Angerer, P., Theis, F. J. SCANPY: large-scale single-cell gene expression data analysis. Genome Biol. 19 (1), 15 (2018).
- Cao, Y., et al. Integrated analysis of multimodal single-cell data with structural similarity. Nucleic Acids Res. 50 (21), e121 (2022).
- . Get Started - JupyterLab 4.1.0a4 documentation Available from: https://jupyterlab.readthedocs.io/en/latest/getting_started/overview.html (2024)
- . MOFA2: training a model in R Available from: https://raw.githack.com/bioFAM/MOFA2_tutorials/master/R_tutorials/getting_started_R.html (2020)
- De Silva, D., et al. Robust T cell activation requires an eIF3-driven burst in T cell receptor translation. eLife. 10, e74272 (2021).
- Li, G., Liang, X., Lotze, M. HMGB1: The central cytokine for all lymphoid cells. Front Immunol. 4, 68 (2013).
- Jassal, B., et al. The reactome pathway knowledgebase. Nucleic Acids Res. 48 (D1), D498-D503 (2020).
- Argelaguet, R., et al. MOFA+: a statistical framework for comprehensive integration of multimodal single-cell data. Genome Biol. 21 (1), 111 (2020).
- Velten, B., et al. Identifying temporal and spatial patterns of variation from multimodal data using MEFISTO. Nat Methods. 19 (2), 179-186 (2022).
- Qoku, A., Buettner, F. Encoding domain knowledge in multi-view latent variable models: A Bayesian approach with structured sparsity. Proceedings of The 26th International Conference on Artificial Intelligence and Statistics. 206, 11545-11562 (2022).
- Multi-Omics Factor Analysis. MOFA Available from: https://biofam.github.io/MOFA2/ (2024)
- Mitchel, J., et al. Tensor decomposition reveals coordinated multicellular patterns of transcriptional variation that distinguish and stratify disease individuals. bioRxiv. , (2023).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。