JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
심혈관 질환과 관련된 변동 패턴 및 분자 과정을 밝히기 위한 비지도 다중 오믹 요인 분석의 적용
요약
다양한 전처리 단계, 다중 오믹스 요인 분석 모델 추정 및 여러 다운스트림 분석을 결합한 복잡한 다중 오믹스 데이터 세트의 비감독 분석을 위한 유연하고 확장 가능한 Jupyter-lab 기반 워크플로를 제공합니다.
초록
질병 메커니즘은 일반적으로 복잡하며 몇 가지 뚜렷한 분자 과정의 상호 작용에 의해 제어됩니다. 복잡한 다차원 데이터 세트는 이러한 프로세스에 대한 더 많은 통찰력을 얻을 수 있는 귀중한 리소스이지만, 이러한 데이터 세트의 분석은 예를 들어 다양한 질병 상태, 시점 및 다양한 해상도에서 프로세스를 캡처하는 오믹스에서 발생하는 높은 차원성으로 인해 어려울 수 있습니다.
여기에서는 급성 및 만성 관상동맥 증후군의 면역 반응을 포착하는 혈액 샘플에서 생성된 데이터 세트에 다중 오믹스 요인 분석(MOFA)을 적용하여 이러한 복잡한 다중 오믹스 데이터 세트를 비지도 방식으로 분석하고 탐색하는 접근 방식을 보여줍니다. 데이터 세트는 시료 수준 사이토카인 데이터, 혈장 단백질체학 및 호중구 프라임 염기서열분석, 단일 세포 RNA-염기서열분석(scRNA-seq) 데이터를 포함하여 다양한 해상도의 여러 분석으로 구성됩니다. 환자당 여러 다른 시점을 측정하고 여러 환자 하위 그룹을 측정함으로써 복잡성이 더욱 가중됩니다.
분석 워크플로우는 (1) 데이터 전처리 및 조화, (2) MOFA 모델 추정, (3) 다운스트림 분석의 여러 단계로 데이터를 통합하고 분석하는 방법을 간략하게 설명합니다. 1단계에서는 다양한 데이터 유형의 특징을 처리하고, 품질이 낮은 특징을 필터링하고, 추가 분석을 위해 분포를 일치시키기 위해 정규화하는 방법을 간략하게 설명합니다. 2단계에서는 MOFA 모델을 적용하고 모든 오믹스 및 기능에 걸쳐 데이터 세트 내 분산의 주요 원인을 탐색하는 방법을 보여줍니다. 3단계에서는 캡처된 패턴의 다운스트림 분석을 위한 몇 가지 전략을 제시하여 이를 질병 상태 및 이러한 상태를 지배하는 잠재적인 분자 과정과 연결합니다.
전반적으로, 복잡한 다중 오믹스 데이터 세트의 비지도 데이터 탐색을 위한 워크플로우를 제시하여 다른 컨텍스트 및 다중 오믹스 데이터 세트(예시적인 사용 사례에 제시된 다른 분석 포함)에도 적용할 수 있는 다양한 분자 특징으로 구성된 주요 변동 축을 식별할 수 있도록 합니다.
서문
질병 메커니즘은 일반적으로 복잡하며 몇 가지 뚜렷한 분자 과정의 상호 작용에 의해 제어됩니다. 특정 질병을 유발하거나 질병의 진행을 관장하는 복잡한 분자 메커니즘을 해독하는 것은 질병을 이해하고 치료하기 위한 새로운 통찰력을 제공할 수 있기 때문에 의학적 관련성이 높은 작업입니다.
최근의 기술 발전으로 더 높은 해상도(예: 단일 세포 수준)와 다양한 생물학적 층(예: DNA, mRNA, 염색질 접근성, DNA 메틸화, 단백질체학)에서 이러한 과정을 동시에 측정할 수 있습니다. 이로 인해 대규모 다차원 생물학적 데이터 세트의 생성이 증가하고 있으며, 이를 공동으로 분석하여 기본 프로세스에 대한 더 많은 통찰력을 생성할 수 있습니다. 동시에 생물학적으로 의미 있는 방식으로 다양한 데이터 소스를 결합하고 분석하는 것은 여전히 어려운 작업입니다1.
서로 다른 기술적 한계, 소음 및 서로 다른 오믹스 간의 변동성 범위는 한 가지 문제를 제기합니다. 예를 들어, 단일 세포 RNA 염기서열분석(scRNA-seq) 데이터는 매우 희박하며 종종 대규모 기술 또는 배치 효과의 영향을 받습니다. 또한 특징 공간은 수천 개의 측정된 유전자 또는 단백질에 걸쳐 매우 큰 경우가 많으며 샘플 크기는 제한적입니다. 이는 여러 질병 상태, 교란 요인, 시점 및 해결 방법을 포함할 수 있는 복잡한 설계로 인해 더욱 복잡해집니다. 예를 들어, 제시된 사용 사례에서는 단일 셀 또는 샘플(대량) 수준에서 다양한 데이터 유형을 사용할 수 있었습니다. 그 외에도 데이터가 불완전할 수 있으며 분석된 모든 피험자에 대해 모든 측정이 가능하지 않을 수 있습니다.
이러한 문제로 인해, 통합 분석을 수행하면 공정에 대한 완전한 그림을 제공할 수 있을 뿐만 아니라 한 오믹스의 생물학적 및 기술적 노이즈가 다른 오믹스에 의해 보상될 수 있음에도 불구하고 서로 다른 오믹스 및 포함된 특징이 여전히 개별적으로 분석되는 경우가 많습니다 3,4. 베이지안 방법(Bayesian methods), 네트워크 기반 방법(network-based methods) 5,6, 멀티모달 딥러닝(multimodal deep learning)7, 행렬 분해를 통한 차원 축소(dimensionality reduction methods) 8,9 등 멀티오믹스 데이터의 통합 분석을 수행하기 위해 여러 가지 방법이 제안되었다. 후자의 경우, 대규모 벤치마킹 연구10의 결과에 따르면 MOFA9(다중 오믹 요인 분석) 방법은 데이터를 임상 주석에 연결해야 할 때 더 적합한 도구 중 하나임이 밝혀졌습니다.
특히 복잡한 환경에서 비지도 행렬 분해 방법은 복잡성을 줄이고 다양한 데이터 소스와 특징에서 공유 및 보완 신호를 추출하는 데 유용한 접근 방식입니다. 복잡한 공간을 더 낮은 순위의 잠재 표현으로 분해하면 데이터 내 분산의 주요 원인을 빠르게 탐색하고 알려진 공변량에 연결할 수 있습니다. 동일한 변이 패턴이 여러 특징(예: 유전자 또는 단백질)에 걸쳐 공유되는 경우, 이는 노이즈가 감소하면서 몇 가지 요인으로 집계될 수 있습니다. 정규화는 모델 계수의 희소성을 증가시키는 데 사용될 수 있으며, 이는 특징 공간이 크고 샘플 수가 제한되는 설정에서 이 접근 방식을 매우 적합하게 만듭니다9.
이 프로토콜은 MOFA 모델을 사용하여 복잡한 다중 오믹스 데이터 세트를 빠르게 탐색하고 이 데이터 세트를 특징짓는 주요 변동 패턴을 추출하는 방법을 보여주는 유연한 분석 워크플로우를 제공합니다. 워크플로는 세 가지 주요 단계로 구성됩니다. 첫 번째 단계인 Data pre-processing and harmonization에서는 다양한 입력 데이터 유형(scRNA-seq, proteomics, cytokine, clinical data)을 기반으로 한 데이터 전처리를 위한 다양한 전략을 제시합니다. 이 프로토콜은 서로 다른 입력 데이터 세트의 기능을 처리하고, 품질이 낮은 기능을 필터링하고, 분포를 조화시키기 위해 정규화하는 방법을 자세히 설명합니다. 또한 이러한 사전 처리 결정이 다운스트림 결과에 어떤 영향을 미칠 수 있는지도 보여줍니다. 두 번째 단계에서는 MOFA 모델을 데이터에 적용하고 결과 분산 분해를 사용하여 서로 다른 데이터 세트의 통합을 평가할 수 있습니다. 세 번째 단계에서는 캡처된 요인을 공변량에 연결하고 이러한 요인을 정의하는 분자 프로그램을 발견하는 방법을 보여줍니다. 제시된 워크플로우를 통해 관상 동맥 증후군을 앓고 있는 환자의 데이터 세트에서 임상 공변량과 관련된 여러 잠재 요인을 추출하고 이전 프로젝트11에서 잠재적인 기저 다세포 면역 프로그램을 식별할 수 있었습니다. 여기서는 이 데이터 세트를 사용하지만 프로토콜은 다른 오믹스를 포함한 다른 컨텍스트에 쉽게 적용할 수 있습니다.
이 데이터 세트는 안정적인 만성 관상동맥 증후군(CCS), 급성 관상동맥 증후군(ACS) 및 건강한 관상동맥(비CCS)이 있는 대조군 환자의 샘플로 구성됩니다(그림 1). ACS는 기존 CCS의 플라크 파열로 인해 발생하며, 심근으로 가는 혈류의 급성 중단과 그에 따른 심장의 허혈성 손상으로 이어집니다. 이 손상은 면역 체계에 의한 염증 반응을 일으킨 후 회복 단계를 일으키며, 이는 급성 사건12 후 몇 일까지 지속됩니다. ACS 환자에 대한 이러한 면역 반응을 특성화할 수 있도록 혈액 샘플을 4가지 다른 시점에서 채취했습니다: 급성(TP1); 재개통 후 (14 [± 8] h) (TP2); 60 [± 12] 시간 후 (TP3); 방전 전(6.5 [±1.5]일)(TP4)(그림 1A). CCS 및 건강한 관상동맥 환자의 경우 하나의 시점(TP0)만 사용할 수 있었습니다. 염증의 임상 지표(크레아틴-키나아제(CK), CK-MB, 트로포닌, C-반응성 단백질(CRP)), 말초 혈액 단핵 세포(PBMC)의 scRNA-seq, 사이토카인 분석, 혈장 단백질체학 및 호중구의prime-seq 13 데이터 등 혈액 샘플을 기반으로 한 다양한 분석을 측정했습니다.
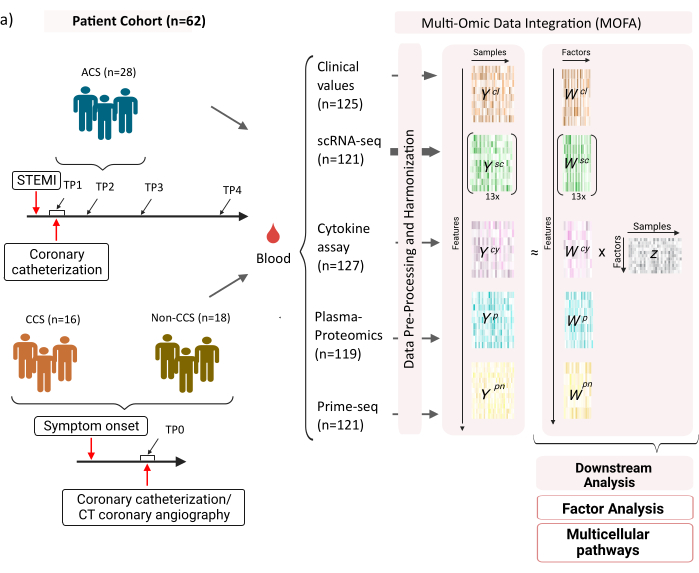
그림 1: 심근 경색 다중 OMIC 입력 데이터 세트. 입력 데이터 세트: 분석된 데이터에는 급성 관상동맥 증후군(ACS), 만성 관상동맥 증후군(CCS) 및 건강한 관상동맥(비CCS) 환자(n = 62)의 혈액 샘플이 포함됩니다. ACS 환자의 경우 4개의 다른 시점(TP1-4)에서 혈액 샘플을 포함시켰고, CCS 환자와 비CCS 환자의 경우 단일 시점(TP0)에서 혈액 샘플을 포함했습니다. 각 환자 및 시점 조합은 분석에서 별도의 샘플로 취급됩니다. 샘플에서 임상 혈액 검사(n=125), scRNA-seq(n=121), 혈장-단백질체학(n=119), 사이토카인 분석(n=127) 및 호중구 프라임-seq(n=121)와 같은 다양한 omic 분석을 측정했습니다. 그 후, 설명된 프로토콜을 적용하여 모든 오믹스에서 데이터를 통합하고 MOFA 모델 및 추가 다운스트림 분석(요인 분석, 경로 농축)을 사용하여 데이터를 탐색했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
여기에 제시된 워크플로우에 대한 입력으로, 예를 들어 scanpy14 전처리 튜토리얼에 설명된 대로 cellranger 및 품질 관리(QC)로 처리한 후 scRNA-seq 데이터에서 원시 계수를 가져옵니다. 셀 유형 주석의 경우 자동화된 Azimuth15 파이프라인을 사용했습니다. 그런 다음 각 샘플 및 셀 유형에 대한 모든 셀의 평균을 취하여 각 셀 유형에 대한 샘플 수준에서 카운트를 집계합니다(pseudobulk 집계). 혈장-단백질체학(Plasma-proteomics)은 정규화 강도(normalized tensity) 및 중앙-중심 강도(median-centered intensities)로 포함되며, 호중구의 경우 prime-seq에서 umi 고유 분자 식별자(UMI) 엑손 수를 취합니다. 사이토카인(cytokine) 및 임상적 가치에 대해서는 이전의 전처리가 적용되지 않았습니다. (실험적) 데이터 생성에 대한 자세한 내용은 해당 원고11에 요약되어 있다. 여기에 제시된 결과는 참조된 간행물에서 사용된 마커 기반 전략과 비교하여 scRNA-seq 데이터의 세포 유형에 대한 자동화된 방위각 주석을 사용한 것을 기반으로 하므로 여기에 제시된 결과는 유사하지만 간행물에 제시된 것과 정확히 동일하지는 않습니다. 원고에서 세포 유형 주석 전략이 분석의 주요 패턴과 생물학적 해석을 변경하지 않지만 모델로 인한 정확한 값의 작은 변화는 다를 수 있음을 보여줄 수 있습니다. 전반적으로 입력 데이터는 10,000개 이상의 서로 다른 특징(유전자, 단백질, 임상 값)의 다양한 시점과 측정 수준(단일 세포 대 벌크)을 포함하는 복잡한 다차원 데이터 세트였습니다. MOFA 분석에 따른 엄격한 전처리 및 데이터 조화 전략은 데이터를 탐색하고 관련 면역 프로그램을 추출하는 데 유용하고 빠른 도구인 것으로 나타났습니다. 각 시점 및 환자 조합은 MOFA 분석에서 독립적인 샘플로 취급됩니다. 각 데이터 유형 및 셀 유형은 MOFA 분석에서 별도의 뷰로 간주됩니다.
이 프로토콜은 워크플로우에 대한 입력 데이터를 준비하고, 다양한 워크플로우 단계를 실행하고, 구성을 사용자 정의하고, 결과 그림을 해석하고, 해석을 기반으로 구성을 반복적으로 조정하기 위한 지침을 제공합니다. 프로토콜의 여러 단계, 각 단계에서 필요한 입력 데이터 세트, 결과 수치 및 데이터 세트에 대한 개요는 기술 워크플로우 개요에 나와 있습니다(그림 2).
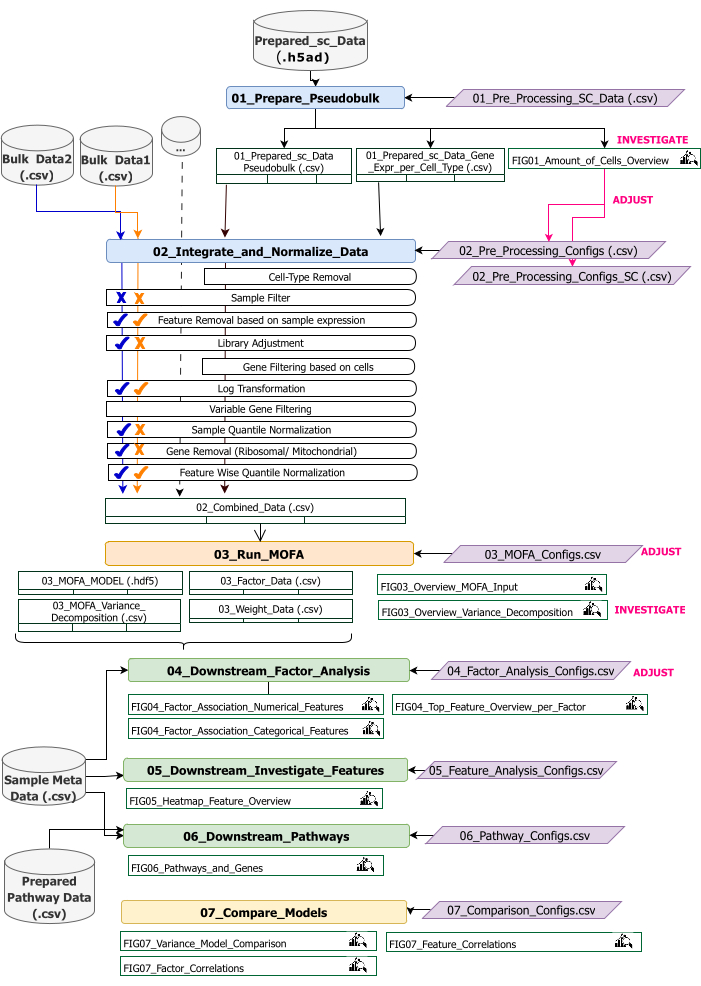
그림 2: 기술 워크플로우 개요. multi-omics 데이터 세트 분석을 위한 워크플로우 개요입니다. 다른 요소는 다른 색상과 기호로 강조 표시됩니다. 데이터 전처리 및 조화(Data Preprocessing and Harmonization)(1) 단계에 속하는 Jupyter Notebook은 파란색으로 표시됩니다. 'MOFA 모델'(2) 단계에 속하는 Jupyter Notebook은 주황색으로 표시됩니다. '다운스트림 분석'(3) 단계에 속하는 Jupyter Notebook은 녹색으로 표시됩니다. 결과 비교에 사용할 Jupyter Notebook 하나는 노란색으로 표시됩니다. 워크플로우 실행을 위한 매개 변수를 수정할 수 있는 구성 파일은 자주색으로 강조 표시됩니다. 워크플로를 실행하는 데 필요한 입력 데이터셋은 데이터셋 기호로 표시되며 회색으로 강조 표시됩니다. 워크플로 실행 중에 생성되는 모든 Figure 출력값은 돋보기 기호로 표시됩니다. 워크플로우 실행 중에 생성된 데이터셋은 테이블로 표시됩니다. 일반적으로 워크플로우는 순차적으로 실행됩니다: (1) 데이터 전처리 및 조화는 scRNA-seq 입력 데이터(01_Prepare_Pseudobulk)를 기반으로 하는 pseudobulk 테이블의 첫 번째 생성과 다른 모든 샘플 레벨(벌크) 입력(02_Integrate_and_Normalize_Data)과 함께 이 데이터의 후속 통합 및 정규화의 두 단계로 구성됩니다. 구성 파일을 통해 이 단계 내에서 각 데이터 세트에 대해 표시된 전처리 및 정규화 단계(예: Sample Filter) 중 어느 것을 적용해야 하는지 개별적으로 구성할 수 있습니다. (2) 'MOFA 모델': 구성 파일(03_MOFA_configs.csv)에 지정된 구성을 사용하여 첫 번째 단계에서 생성된 입력에 대해 MOFA 모델을 실행합니다. (3) '다운스트림 분석': 생성된 MOFA 결과에 대한 통찰력을 생성하고 이를 'Sample Meta Data.csv' 파일을 통해 입력으로 제공되는 샘플 메타 데이터(공변량)와 연결하기 위해 서로 독립적으로 실행할 수 있는 3개의 서로 다른 노트북으로 구성됩니다. (4) '모델 비교': 2단계에서 생성된 다른 모델을 비교하는 데 사용할 수 있는 작은 별도 단계입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
워크플로는 R 및 Python으로 작성된 여러 Jupyter Notebook으로 구성됩니다(워크플로를 실행하는 데 R 및 Python 언어에 대한 지식이 필요하지 않지만 오류가 표시되는 경우 도움이 될 수 있음). 프로토콜의 다양한 단계에서 매개변수는 구성 파일(이름에 접미사 '_Configs'를 포함하는 '.csv' 파일)을 통해 변경됩니다. 프로토콜 내에서는 기본 구성부터 변경해야 하는 매개변수만 간략하게 설명합니다.
예를 들어, 전처리를 사용자 정의하기 위해 몇 가지 다른 매개변수도 변경될 수 있습니다. 이러한 매개 변수 및 설명에 대한 설명서는 다운로드한 저장소에 포함된 'Documentation_Config_Parameter' 파일에 제공됩니다.
프로토콜
1. 준비: 기술 설정 및 설치
참고: 이 프로그램을 실행하려면 wget, git 및 Apptainer가 장치에 미리 설치되어 있어야 합니다. 다른 시스템(Linux, Windows, Mac)에 Apptainer를 설치하는 방법에 대한 안내는 https://apptainer.org/docs/admin/main/installation.html 여기에서 제공됩니다. git에 대한 설치 정보는 여기에서 찾을 수 있습니다. https://git-scm.com/book/en/v2/Getting-Started-Installing-Git. 다양한 입력 데이터 세트의 크기에 따라 적절한 컴퓨터(CPU 16개, 64GB 메모리)에서 워크플로를 실행하는 것이 좋습니다. 제공된 예제 데이터를 사용한 스모크 테스트를 로컬 컴퓨터에서 실행할 수 있습니다. 예제 데이터에서 프로토콜을 실행한 지침 및 예상 출력은 보충 파일 1에 나와 있습니다. 위에서 설명한 데이터 세트에서 실행되는 프로토콜의 중요한 단계에 대해서는 보충 비디오 파일 1 을 참조하십시오.
- 콘솔을 열고 모든 분석 코드와 출력이 저장될 폴더를 선택하거나 생성합니다. 터미널에 cd path_to_folder 명령을 입력하여 폴더로 이동합니다.
- Github(https://github.com/heiniglab/mofa_workflow)에서 또는 터미널 창에 git clone https://github.com/heiniglab/mofa_workflow.git 를 입력하여 코드 리포지토리를 다운로드하거나 복제합니다.
- 터미널 창에 wget https://zenodo.org/records/11192947/files/mofa_image.sif 를 입력하여 Zenodo에서 필요한 모든 설치가 포함된 이미지를 다운로드합니다.
- 터미널 창에 mkdir results 를 입력하여 모든 결과 데이터가 저장될 폴더를 생성합니다.
- 터미널 창에 mkdir input_data 를 입력하여 분석에 사용할 모든 입력 데이터가 추가될 폴더를 생성합니다.
- 터미널에 apptainer run mofa_image.sif 명령을 입력하여 JupyterLab 세션을 시작할 컨테이너를 실행합니다. 명령에 의해 반환된 URL을 브라우저에 복사하면 Jupyter-lab 세션이 열립니다(Jupyter-lab에 대한 자세한 내용은 소프트웨어 설명서16에서 찾을 수 있음).
참고: 워크플로우가 랩톱에서 로컬로 실행되는 경우 로컬 호스트 주소를 직접 반환하는 apptainer exec mofa_image.sif jupyter-lab 명령을 대신 사용하는 것이 좋습니다. 컨테이너가 클러스터 컴퓨팅 환경 내에서 실행되는 경우 ssh를 통해 수행할 수 있는 포트 전달을 설정해야 할 수 있습니다.
2. 초기화 및 데이터 준비
- Jupyter-Lab 세션에서 왼쪽의 탐색 메뉴를 사용합니다. input_data를 두 번 클릭하여 input_data 폴더로 이동합니다.
- Drag&Drop을 사용하여 분석의 입력으로 사용할 모든 데이터셋을 input_data 디렉토리에 복사합니다. 현재 파일이 있는 폴더에서 파일을 끌어 input_data 폴더 아래 영역의 Jupyter-lab 세션에 놓습니다.
참고: 모든 데이터 세트는 .csv 또는 .h5ad(단일 셀 데이터의 경우) 형식이어야 합니다. 모든 .csv 파일에는 일치하는 sample_id 열이 포함되어야 합니다(데이터 세트 간에 동일한 ID를 사용해야 함). 다른 모든 열은 피처로 사용됩니다. h5ad- 파일 내에서 셀 주석에는 sample_id와 cluster_id를 지정하는 두 개의 식별자가 포함되어야 합니다. 이는 집계 및 일치에 사용됩니다. 다른 형식의 Omic 데이터 세트는 사용하기 전에 지정된 .csv 형식으로 변환해야 합니다(그림 3). .h5seurat 형식으로 제공된 scRNA-seq 데이터셋은 Jupyter-notebook: 00_Data_Conversion.ipynb를 실행하여 .h5ad로 변환될 수 있습니다. - 폴더 기호를 클릭하여 configurations 폴더로 이동한 다음 mofa_workflow, scripts 및 configurations 폴더를 두 번 클릭합니다. 폴더 내에서 파일 Data_configs.csv 두 번 클릭하여 엽니다.
- 값 열에서 input_data(data_path) 및 결과(result_path) 폴더의 폴더 경로를 추가합니다. configuration_name의 값 열에 파일 확장명으로 추가될 이름을 추가합니다(이 프로토콜은 [Myocardial Infarction version1]MI_v1 사용됨)(그림 4).
- 상단 메뉴에서 파일 > CSV 파일 저장 을 클릭하여 변경 사항을 저장합니다.
- 왼쪽의 탐색 메뉴를 사용하여 스크립트를 클릭하여 스크립트 폴더로 이동합니다. 00_Configuration_Update.ipynb를 두 번 클릭하여 초기화 Notebook을 엽니다. 맨 위에 있는 Restart kernel and run all cells 버튼을 클릭하고 팝업에서 Restart를 클릭하여 스크립트를 실행합니다(그림 5).
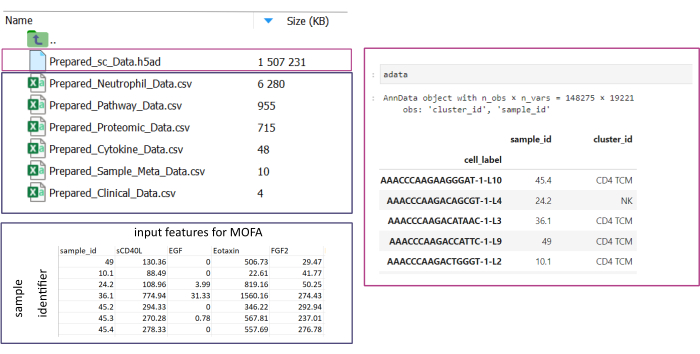
그림 3: 데이터 입력 및 설정. 워크플로를 실행하려면 모든 데이터를 지정된 input_data 폴더에 저장해야 합니다. 각 입력 데이터 세트에 대해 별도의 파일을 제공해야 합니다. 단일 셀 데이터는 cluster_id의 셀 주석(예: 이전 셀 유형 주석 단계의 결과) 및 sample_id 열(분석해야 하는 각 개별 샘플을 고유하게 식별)을 포함하는 .h5ad로 제공해야 합니다. 다른 모든 입력 데이터 세트는 '.csv' 형식으로 제공되어야 하며, 여기에는 sample_id(단일 셀 데이터의 해당 열과 일치)을 지정하는 한 열과 다른 모든 열에서 MOFA 분석에 사용할 기능이 포함됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
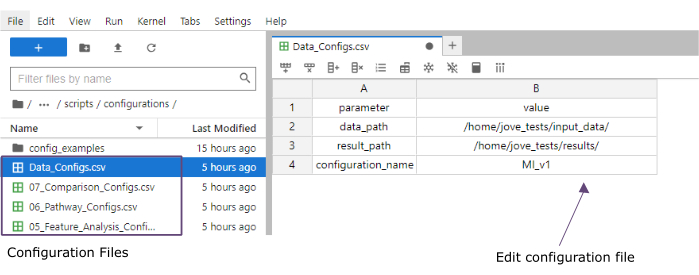
그림 4: Jupyter-lab 구성 파일. 워크플로우를 실행하는 동안 매개변수의 변경(예: 필터링 옵션 조정 등)은 '.csv' 구성 파일을 통해 지정됩니다. 복제된 리포지토리 내에는 각 단계에 대한 기본 구성 파일이 포함됩니다. 스프레드시트와 마찬가지로 jupyter-lab 콘솔에서 직접 편집할 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
그림 5: Jupyter-notebooks 스크립트. 전체 워크플로는 해당 구성 파일이 수정된 후 순차적으로 실행되는 일련의 Jupyter Notebook으로 구성됩니다. 왼쪽의 Jupyter 노트북을 두 번 클릭하면 해당 파일이 오른쪽에 열립니다. 파일의 전체 실행은 상단에 강조 표시된 버튼으로 시작할 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
3. 데이터 전처리 및 조화
- 전처리 - sc 데이터를 pseudobulk로 변환합니다.
참고: 이 단계는 단일 셀 데이터가 분석에 사용되는 경우에만 실행해야 합니다.- 왼쪽의 탐색 메뉴를 사용하여 configuration을 두 번 클릭하여 configurations 폴더로 이동합니다. 파일 01_Preprocessing_SC_Data.csv 두 번 클릭하여 엽니다. 파일에서 자동으로 입력된 값을 확인하고 필요한 경우 data_name 열의 값을 분석에 사용할 input_data 폴더에 있는 단일 셀 데이터셋의 파일 이름에 맞게 조정합니다.
참고: 기본적으로 입력 데이터 폴더에 있는 .h5ad 파일의 모든 이름은 초기화 스크립트의 구성 파일에 추가됩니다. 일부 데이터셋을 분석에 사용하지 않아야 하는 경우 여기에서 제거할 수 있습니다. - 상단 메뉴에서 파일 > CSV 파일 저장 을 클릭하여 변경 사항을 저장합니다.
- 왼쪽의 탐색 메뉴를 사용하여 스크립트를 클릭하여 스크립트 폴더로 이동합니다. Notebook 01_Prepare_Pseudobulk.ipynb를 두 번 클릭하여 엽니다. 상단의 Restart kernel and run all cells 버튼을 클릭하고 팝업에서 Restart를 클릭하여 스크립트를 실행합니다.
- 왼쪽의 탐색 메뉴를 사용하여 먼저 그림을 두 번 클릭한 다음 01_figures 두 번 클릭하여 그림 폴더로 이동합니다. 새로 생성된 플롯 FIG01_Amount_of_Cells_overview 두 번 클릭하여 엽니다.
참고: Notebook을 실행하는 데 몇 분 정도 걸릴 수 있습니다. 노트북이 성공적으로 실행되면 팝업이 나타나고 파일 FIG01_Amount_of_Cells_Overview 노트북에 의해 업데이트되거나 새로 생성됩니다. 마지막으로 수정한 날짜 열은 파일이 새 파일인지 이전 파일인지 평가하기 위해 파일이 생성된 시기를 나타낼 수 있습니다. - 표본당 세포 수가 매우 적은 세포 유형 군집을 식별하기 위해 플롯을 조사합니다. 이러한 cluster_ids의 이름을 기록해 두어 다음 단계에서 제외합니다(그림 6).
- 왼쪽의 탐색 메뉴를 사용하여 을 클릭하여 configurations 폴더로 다시 이동합니다. 그런 다음 구성을 두 번 클릭합니다. 파일 02_Preprocessing_Configs_SC.csv 두 번 클릭하여 엽니다.
- configuration_name 및 data_name 열의 값을 확인하고 필요한 경우 조정합니다.
참고: 초기화 스크립트 내에서 이러한 값은 입력 데이터 폴더에 있는 .h5ad 파일의 모든 이름과 이전에 Data_Configs.csv 파일 내에 설정된 configuration_name 값으로 미리 채워져 있습니다. 파일을 분석에서 제외해야 하거나 파일 이름에 대한 다른 확장자를 사용해야 하는 경우 여기에서 조정할 수 있습니다. - cell_type_exclusion 열의 값을 조정하고 이전 단계에서 제외하도록 식별된 모든 cluster_id ','로 구분하여 추가합니다.
- 상단의 탐색 모음에서 파일 > CSV 파일 저장 을 클릭하여 변경 사항을 저장합니다.
- 왼쪽의 탐색 메뉴를 사용하여 configuration을 두 번 클릭하여 configurations 폴더로 이동합니다. 파일 01_Preprocessing_SC_Data.csv 두 번 클릭하여 엽니다. 파일에서 자동으로 입력된 값을 확인하고 필요한 경우 data_name 열의 값을 분석에 사용할 input_data 폴더에 있는 단일 셀 데이터셋의 파일 이름에 맞게 조정합니다.
- 전처리 - 다른 오믹스 데이터 소스를 조화시키고 통합합니다.
- 파일 02_Preprocessing_Configs.csv 두 번 클릭하여 열고 data_input 폴더(데이터 세트당 한 행)에 포함되고 저장될 각 데이터 세트에 대한 전처리 구성을 조정합니다.
- configuration_name 및 data_name 열의 값을 확인하고 필요한 경우 조정합니다.
- 어떤 전처리 단계를 적용해야 하는지에 따라 열의 다른 매개 변수를 적절하게 조정합니다.
참고: 기본값은 input_dataset 폴더 내에 있는 각 데이터셋에 대해 추가되지만 데이터의 단일 데이터 유형에만 국한되지 않습니다. 따라서 조정이 필요합니다. 매개변수에 대한 자세한 설명서는 Documentation_Config_Parameter.doc 파일에 나와 있습니다. - 파일 > CSV 파일 저장을 클릭하여 변경 사항을 저장합니다.
- 왼쪽의 탐색 메뉴를 사용하여 스크립트를 클릭하여 스크립트 폴더로 이동합니다. Notebook 02_Integrate_and_Normalized_Data_Sources.ipynb를 두 번 클릭하여 엽니다. 상단의 Restart kernel and run all cells 버튼을 클릭하고 팝업에서 Restart를 클릭하여 스크립트를 실행합니다.
- 왼쪽의 탐색 메뉴를 사용하여 폴더 기호를 클릭하여 생성된 02_results 폴더로 이동한 다음 결과 및 02_results 두 번 클릭합니다. 결합된 사전 처리된 데이터 입력 파일이 포함된 파일02_Combined_data_'configuration_name'_Integrated.csv이 포함되어 있는지 확인합니다.
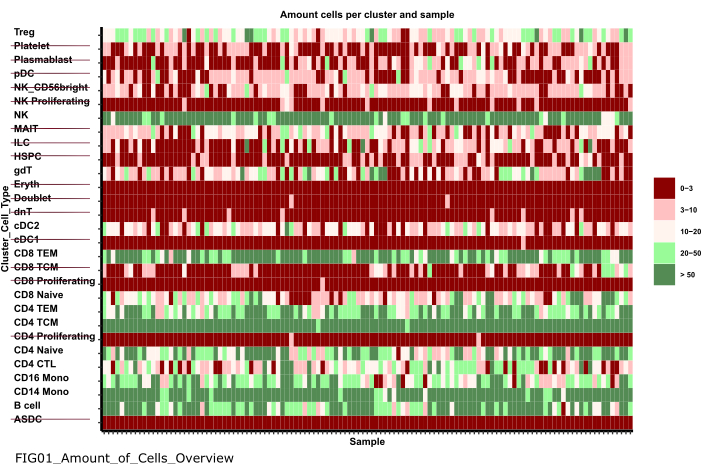
그림 6: 데이터 전처리 및 조화. '01_Prepare_Pseudobulk' 스텝의 출력값 중 하나는 플롯 'Fig01_Amount_of_Cells_Overview'입니다. 여기서 각 cluster_id(이전 셀 유형 주석 단계의 셀 유형을 나타내는 y축)에 대해 샘플당 셀 수('sample_id')가 제공됩니다. 제시된 결과 내에서 샘플당 세포 수가 적은 세포 유형은 후속 분석에서 제외됩니다(취소선으로 표시됨). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
4. MOFA 실행
- Jupyter-Lab에서 왼쪽의 탐색 메뉴를 사용하여 폴더 기호를 클릭한 다음 mofa_workflow를 두 번 클릭하고 스크립트 및 구성을 두 번 클릭하여 구성 폴더 로 이동합니다. 파일 03_MOFA_Configs.csv 두 번 클릭하여 엽니다.
- configuration_name 및 mofa_result_name 열에 대한 항목을 확인하고 대체 이름을 사용해야 하는 경우 항목을 조정합니다.
참고: mofa_result_name 는 MOFA를 기반으로 생성된 모든 결과 파일에 파일 확장자로 추가됩니다. 이는 다른 MOFA 설정이 동일한 입력 데이터로 실행될 수 있으므로 configuration_name 값과 다를 수 있습니다(이 프로토콜은 MI_v1_MOFA 사용). - MOFA 모델에서 추정해야 하는 요인의 양을 입력하고(amount_of_factors 열) 파일의 값을 조정하여 가중치 및 스케일링을 적용해야 하는지(weighting_of_views 및 scale_views 열)를 정의합니다.
- 파일 > CSV 파일 저장을 클릭하여 변경 사항을 저장합니다.
- 왼쪽의 탐색 메뉴를 사용하여 '스크립트'를 클릭하여 스크립트 폴더로 이동합니다. 파일을 두 번 클릭하여 Notebook 03_Run_MOFA.ipynb를 엽니다. 상단의 Restart kernel and run all cells 버튼을 클릭하고 팝업에서 Restart를 클릭하여 스크립트를 실행합니다.
- 그림을 두 번 클릭하여 03_figures 폴더로 이동한 다음 03_figures. 생성된 플롯 FIG03_Overview_Variance_Decomposition_ mofa_result_name 열고 모델 결과를 조사합니다(그림 7A).
- 왼쪽의 탐색 메뉴를 사용하여 폴더 기호를 클릭하고 결과 및 03_results 두 번 클릭하여 생성된 03_results 폴더로 이동합니다. 표본 요인 값 파일03_Factor_Data_'mofa_result_name'.csv 및 형상 요인 가중치 파일03_Weight_Data_'mofa_result_name'.csv이 포함되어 있는지 확인합니다.
5. 다운스트림 분석
- 요인 해석.
- 왼쪽의 탐색 메뉴를 사용하여 폴더 기호를 클릭하여 input_data 폴더로 이동한 다음 input_data 두 번 클릭합니다.
- 생성된 요인과 관련하여 분석할 표본의 모든 메타데이터(공변량)가 포함된 .csv 파일(Prepared_Sample_Meta_Data.csv)을 준비합니다. 드래그 앤 드롭을 사용하여 파일을 input_data 폴더에 복사한 다음 input_data 폴더의 폴더 개요에 파일을 놓습니다.
참고: 이전에 사용한 데이터와 일치시키기 위한 sample_id 열과 분석해야 하는 각 기능에 대한 추가 열을 포함해야 합니다. - Jupyter-Lab에서 왼쪽의 탐색 메뉴를 사용하여 폴더 기호를 클릭한 다음 mofa_workflow 두 번 클릭한 다음 스크립트 및 구성을 클릭하여 구성 폴더로 다시 이동합니다. 파일 04_Factor_Analysis.csv 두 번 클릭하여 엽니다.
- configuration_name 및 mofa_result_name에 대한 항목에 스크립트에서 분석할 구성 및 MOFA 결과의 이름이 포함되어 있는지 확인하고 필요한 경우 조정합니다.
- numeric_covariates 열에서 쉼표로 구분된 MOFA 요소와 관련하여 조사할 Prepared_Sample_Meta_Data.csv 파일의 모든 숫자 열 이름을 추가합니다(이 프로토콜은 CRP,CK를 사용함).
- categorical_covariates' 열에 MOFA 계수와 관련하여 조사할 Prepared_Sample_Meta_Data.csv 파일의 모든 범주형 열 이름을 쉼표로 구분하여 추가합니다(이 프로토콜은 측정 사용).
- 파일 > CSV 파일 저장을 클릭하여 변경 사항을 저장합니다.
- 왼쪽의 탐색 메뉴를 사용하여 스크립트를 클릭하여 'scripts' 폴더로 이동합니다. Notebook 04_Downstream_Factor_Analysis.ipynb 를 두 번 클릭하여 엽니다. 상단의 Restart kernel and run all cells 버튼을 클릭하고 팝업에서 restart를 클릭하여 스크립트를 실행합니다.
- 왼쪽의 탐색 메뉴를 사용하여 그림을 두 번 클릭한 다음 04_figures 클릭하여 04_figures 폴더로 이동합니다. 생성된 플롯을 두 번 클릭하여 열고 흥미로운 패턴과 연관성에 대한 요인을 조사합니다.FIG04_Factor_Association_with_numeric_features_
'mofa_result_name.pdf(그림 7B). FIG04_Factor_Association_
with_categorical_features_'mofa_result_name.pdf(그림 7C). FIG04_Top_Feature_Overview_per_Factor _'mofa_result_name.pdf(그림 8A).
- 피처 분석
- 왼쪽의 탐색 메뉴를 사용하여 을 클릭하여 configurations 폴더로 다시 이동합니다. 그런 다음 구성을 두 번 클릭합니다. 파일 05_Feature_Analysis_Configs.csv 두 번 클릭하여 엽니다.
- configuration_name 및 mofa_result_name 열의 항목이 다운스트림 분석에 사용될 구성 및 생성된 MOFA 결과의 이름과 일치하는지 확인하고 필요한 경우 조정합니다.
- factor 열에서, 다음 스크립트 내에서 상위 피처가 그려질 팩터를 추가합니다.
- 열 faceting_variable에서 Prepared_Sample_Meta_Data에 범주형 열의 열 이름을 추가하고.csv 이 열 이름은 그림의 표본을 그룹화하는 데 사용됩니다(이 프로토콜은 측정을 사용합니다).
- 파일 > CSV 파일 저장을 클릭하여 변경 사항을 저장합니다.
- 왼쪽의 탐색 메뉴를 사용하여 스크립트를 클릭하여 스크립트 폴더로 이동합니다. Notebook 05_Downstream_Investigate_Features_Heatmap.ipynb를 두 번 클릭하여 엽니다. 상단의 Restart kernel and run all cells 버튼을 클릭하고 팝업에서 Restart를 클릭하여 스크립트를 실행합니다.
- 왼쪽의 탐색 메뉴를 사용하여 먼저 그림을 두 번 클릭한 다음 05_figures를 두 번 클릭하여 05_figures 폴더로 이동합니다. 생성된 플롯 FIG05_Heatmap_Feature_Overview__ 'mofa_result_name'를 열고 조사합니다.파일을 두 번 클릭하여 pdf를 선택합니다(그림 8B).
참고: 플롯에 표시될 기능의 양에 따라 05_Feature_Analysis_Configs.csv 내에서 plot_width 및 plot_height 매개변수를 조정하고 스크립트를 다시 실행하여 모든 것이 플롯에 맞는지 확인해야 할 수 있습니다.
- 경로 분석
- 왼쪽의 탐색 메뉴를 사용하여 폴더 기호를 클릭하여 input_data 폴더로 이동한 다음 input_data 두 번 클릭합니다.
- 보강을 테스트할 경로 목록이 포함된 .csv 파일(Prepared_Pathway_Data.csv)을 준비합니다. 드래그 앤 드롭을 사용하여 파일을 input_data 폴더에 복사한 다음 input_data 폴더의 폴더 개요에 파일을 놓습니다.
참고: ID (경로의 고유 식별자), gene (경로에 속하는 유전자 이름(SYMBOL)로 지정된 유전자, 유전자당 한 행), pathway_name (경로의 이름/텍스트 설명)의 세 열을 포함해야 합니다. - Jupyter-Lab 세션에서 왼쪽의 탐색 메뉴를 사용하여 을 클릭하여 configurations 폴더로 이동합니다. 그런 다음 구성을 두 번 클릭합니다. 파일 06_Pathway_Configs.csv 두 번 클릭하여 엽니다.
- mofa_result_name 열의 항목을 확인하고 경로 보강 계산에 사용할 생성된 MOFA 결과의 이름과 일치하는지 확인합니다.
- types 열의 항목을 확인하고 Prepared_Pathway_Data.csv 파일의 gene 열과 일치하는 특징이 포함되지 않은 types 열 내의 항목을 제거합니다.
참고: 기본적으로 MOFA 모델 내에서 사용된 모든 다른 보기는 워크플로우 실행 중에 이 파일에 추가됩니다. 하나 이상의 경로와 일치하는 피처를 포함하지 않는 뷰가 있는 경우 제거해야 합니다. 그렇지 않으면 실행이 실패합니다. 예를 들어 경로 파일에는 유전자에 대한 경로 주석만 포함되어 있지만 단백질 이름이 포함된 보기가 있습니다. - 파일 > CSV 파일 저장을 클릭하여 변경 사항을 저장합니다.
- 탐색 메뉴를 사용하여 스크립트를 클릭하여 scripts 폴더로 이동합니다. Notebook 06_Downstream_Pathways.ipynb를 두 번 클릭하여 엽니다. 상단의 Restart kernel and run all cells 버튼을 클릭하고 팝업에서 Restart를 클릭하여 스크립트를 실행합니다.
- 왼쪽의 탐색 메뉴를 사용하여 먼저 그림을 두 번 클릭한 다음 06_figures 클릭하여 06_figures 폴더로 이동합니다. 생성된 플롯 FIG06_Pathways_and_Genes_ 'mofa_result_name'를 두 번 클릭하여 열고 시각화된 경로를 조사합니다(그림 8C).
참고: 시각화된 경로를 선택하는 방법은 구성 파일을 통해 구성할 수 있습니다. 자세한 내용은 매개 변수 설명서를 참조하세요. - 왼쪽의 탐색 메뉴를 사용하여 폴더 기호를 클릭하고 결과 및 06_results 두 번 클릭하여 생성된 06_results 폴더로 이동합니다. 보강된 경로 06_Pathway_enrichment__'mofa_result_name'를 포함하는 파일이 포함되어 있는지 확인합니다.
6. 다양한 구성 및 버전 비교(보충 그림 1, 보충 그림 2, 보충 그림 3, 보충 그림 4)
- 워크플로우 전체에서 다른 매개 변수/구성을 사용하는 효과를 비교하려면 섹션 3-5를 다시 실행하여 구성 파일의 매개 변수를 수정하고 다른 configuration_name 및 mofa_result_name 식별자를 사용합니다.
참고: 새 결과는 다른 실행을 비교하는 데 사용할 수 있도록 이러한 이름으로 저장됩니다. - Jupyter-Lab에서 왼쪽의 탐색 메뉴를 사용하여 구성 폴더로 이동합니다. 파일 07_Comparison_Configs.csv 두 번 클릭하여 엽니다.
- mofa_result_name 열에 비교할 모든 이전 MOFA 실행의 이름을 추가합니다(이름/구성당 하나의 행, 예: MI_v1_MOFA, MI_v2_MOFA).
- compare_factors 열에서 모델 간에 비교할 요인을 추가합니다. 기본적으로 Factor1,Factor2,Factor3입니다. (보충 그림 2A).
참고: 이 스크립트에서는 서로 다른 모델의 기능 및 요인 값을 상호 연관시켜 비교합니다. 이는 동일한 샘플( sample_id로 표시)과 동일한 기능 세트를 기반으로 하는 모델에만 적용됩니다. 비교된 버전 간에 샘플 또는 기능이 일치하지 않는 경우 비교에서 제외됩니다. - 파일 > CSV 파일 저장을 클릭하여 변경 사항을 저장합니다.
- 왼쪽의 메뉴를 사용하여 스크립트를 클릭하여 스크립트 폴더로 이동합니다. Notebook 07_Compare_Models.ipynb를 두 번 클릭하여 엽니다. Restart kernel and run all cells 버튼을 클릭하고 팝업에서 Restart를 클릭하여 스크립트를 실행합니다.
- 왼쪽의 메뉴를 사용하여 먼저 그림을 두 번 클릭한 다음 06_figures 클릭하여 06_figures 폴더로 이동합니다. 파일을 두 번 클릭하여 생성된 플롯을 열고 서로 다른 버전의 유사성을 분석합니다.
FIG07_Variance_Model_Comparison.pdf(보충 그림 2B)
FIG07_Factor_Correlations.pdf
(보충 그림 2C)
FIG07_Feature_Correlations.pdf
(보충 그림 3C)
7. 워크플로우 확장: 다른 매개 변수 및 구성 추가
참고: 구성 파일에서 현재 구성 가능한 매개 변수 외에도 코드 또는 다른 매개 변수의 다른 조정 사항이 포함될 수 있습니다. 예를 들어, MOFA 모델 자체는 코드에서 직접 수정하거나 구성 파일을 통해 조정할 수 있는 몇 가지 다른 훈련 매개변수(17 )를 제공한다. 프로토콜의 다음 섹션에서는 추가 MOFA 모델 학습 매개변수에 대해 이 작업을 수행하는 방법의 예를 간략하게 설명합니다. 이 부분에서는 R 프로그래밍 지식이 필요합니다.
- Jupyter-Lab에서 왼쪽의 탐색 메뉴를 사용하여 scripts 폴더로 이동합니다. Notebook 03_Run_MOFA.ipynb 를 두 번 클릭하여 엽니다.
- 왼쪽의 Table of Contents(목차 ) 탭을 클릭한 다음 4.3 Set MOFA Training Options(MOFA 교육 옵션 설정) 하위 섹션으로 이동하고 이를 클릭하여 Model Training(모델 교육)을 실행합니다 . 노트북에서 구성 가능한 매개변수의 MOFA 모델의 인쇄된 출력을 보려면 아래로 스크롤합니다.
- 제목 아래 코드의 R for 루프 내에서 모든 MOFA 데이터, 모델 및 교육 옵션이 설정됩니다. model_opts$num_factors = mofa_configs$amount_of_factors[i]줄 아래에 아래 코드가 있는 다른 줄을 추가합니다.
model_opts$likelihoods['data_type'] = '푸아송'.
참고: 이렇게 하면 모델이 모든 MOFA 실행에 대해 data_type 라는 이름으로 지정된 뷰에 대한 입력으로 사용하는 분포가 변경됩니다. 데이터 유형에 대해 poisson을 지정할 때 이 데이터 유형의 기능이 정수인 경우(예: RNA-seq에서 카운트 읽기)만 모델이 실행됩니다. MOFA 데이터, 교육 및 모델 옵션에 대한 자세한 내용은 MOFA 자습서 및 문서17을 참조할 수도 있습니다. - 맨 위에 있는 저장 버튼을 클릭하여 Notebook에 변경 사항을 저장합니다.
- .csv 구성 파일을 통해 새 매개 변수를 전달하려면 왼쪽의 탐색을 사용하여 구성을 두 번 클릭하여 구성 폴더로 이동하고 03_MOFA_Configs.csv 두 번 클릭하여 파일을 엽니다.
- 매개 변수 이름(예: number_iterations )을 지정하는 새 열을 추가하고 값(예: 1000)을 입력합니다. 파일 > CSV 파일 저장을 클릭하여 변경 사항을 저장합니다.
- 탐색 메뉴를 사용하여 스크립트를 클릭하여 스크립트 폴더를 탐색 합니다. Notebook 03_Run_Mofa.ipynb 를 두 번 클릭하여 엽니다. 왼쪽의 Table of Contents(목차 ) 탭을 클릭한 다음 4.3 Set MOFA Training Options(MOFA 교육 옵션 설정) 하위 섹션으로 이동하고 이를 클릭하여 Model Training(모델 교육)을 실행합니다 .
- train_opts$maxiter = 50000 줄을 train_opts$maxiter = mofa_configs$column_name[i]로 바꿉니다(추가된 열의 이름이 number_of_iterations인 경우 train_opts$maxiter = mofa_configs$number_of_iterations[i])).
참고: 이 노트북에 03_MOFA_Configs.csv 구성 파일은 노트북의 시작 부분(하위 섹션: 필수 구성 및 매개변수)에서 세션의 data.frame mofa_config 읽히므로 이 코드 줄에서 이 개체와 새로 생성된 해당 열을 참조합니다. 여러 구성을 동시에 실행할 수 있으므로 i 는 모델 추정이 .csv 파일의 모든 다른 행에서 for 루프로 실행될 때 data.frame의 행을 식별합니다. '필수 구성 구성 및 매개 변수' 섹션의 Notebook 시작 부분에 있는 구성 파일을 읽는 원칙은 모든 Notebook에 대해 동일하며 이와 같이 추가 수정을 수행할 수 있습니다. - 저장 버튼을 클릭하여 Notebook에 변경 사항을 저장합니다.
결과
워크플로우가 성공적으로 실행되면 그림 2와 같이 여러 테이블과 그림이 생성됩니다. 그림은 /figures 폴더(그림 6, 그림 7, 그림 8, 보충 그림 1, 보충 그림 2, 추가 그림 3, 보충 그림 4)에 배치되고 테이블은 지정된 /r...
토론
설명된 프로토콜을 사용하면 복잡한 다중 오믹스 데이터 세트를 빠르게 탐색하는 데 사용할 수 있는 확장 가능한 모듈식 Jupyter 노트북 기반 워크플로가 제공됩니다. 워크플로우의 주요 부분은 전처리 및 데이터 조화 부분(데이터 필터링 및 정규화를 위한 다양한 표준 단계 제공), MOFA9 모델 추정 및 몇 가지 예시적인 다운스트림 분석으로 구성됩니다. 가?...
공개
저자는 이해 상충이 없음을 선언합니다.
감사의 말
CL은 공동 연구 학교인 "Munich School for Data Science - MUDS"에서 Helmholtz Association의 지원을 받고 있습니다.
자료
| Name | Company | Catalog Number | Comments |
| Apptainer | NA | NA | https://apptainer.org/docs/admin/main/installation.html |
| Compute server or workstation or cloud (Linux, Mac or Windows environment). Depending on the size of the different input datasets we recommend running the workflow on a suitable machine (in our setting we use: 16 CPU, 64GB Memory) | Any manufacturer | 16 CPU, 64GB Memory | Large Memory is only required for the processing of the raw single cell data. After preprocessing the later analysis steps can also be performed on regular desktop or laptop computers |
| git | NA | NA | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git |
| GitHub | GitHub | NA | https://github.com/heiniglab/mofa_workflow |
참고문헌
- Lähnemann, D., et al. Eleven grand challenges in single-cell data science. Genome Biol. 21 (1), 31 (2020).
- Colomé-Tatché, M., Theis, F. J. Statistical single cell multi-omics integration. Curr Opin Syst Biol. 7, 54-59 (2018).
- Hawe, J., Theis, F., Heinig, M. Inferring interaction networks from multi-omics data. Front Genet. 10, 535 (2019).
- Hawe, J. S., et al. Network reconstruction for trans acting genetic loci using multi-omics data and prior information. Genome Med. 14 (1), 125 (2022).
- Koh, H. W. L., Fermin, D., Vogel, C., Choi, K. P., Ewing, R. M., Choi, H. iOmicsPASS: network-based integration of multiomics data for predictive subnetwork discovery. NPJ Syst Biol Appl. 5, 22 (2019).
- Ogris, C., Hu, Y., Arloth, J., Müller, N. S. Versatile knowledge guided network inference method for prioritizing key regulatory factors in multi-omics data. Sci Rep. 11, 6806 (2021).
- Lee, C., vander Schaar, M. A variational information bottleneck approach to multi-omics data integration. Proceedings of The 24th International Conference on Artificial Intelligence and Statistics. 130, 1513-1521 (2021).
- Singh, A., et al. DIABLO: an integrative approach for identifying key molecular drivers from multi-omics assays. Bioinformatics. 35 (17), 3055-3062 (2019).
- Argelaguet, R., et al. Multi-omics factor analysis-a framework for unsupervised integration of multi-omics data sets. Mol Syst Biol. 14 (6), e8124 (2018).
- Cantini, L., et al. Benchmarking joint multi-omics dimensionality reduction approaches for the study of cancer. Nature Commun. 12 (1), 124 (2021).
- Pekayvaz, K., et al. Multiomic analyses uncover immunological signatures in acute and chronic coronary syndromes. Nature Medicine. 30 (6), 1696-1710 (2024).
- Swirski, F. K., Nahrendorf, M. Cardioimmunology: the immune system in cardiac homeostasis and disease. Nat Rev Immunol. 18 (12), 733-744 (2018).
- Janjic, A., et al. Prime-seq, efficient and powerful bulk RNA sequencing. Genome Biol. 23 (1), 88 (2022).
- Wolf, F. A., Angerer, P., Theis, F. J. SCANPY: large-scale single-cell gene expression data analysis. Genome Biol. 19 (1), 15 (2018).
- Cao, Y., et al. Integrated analysis of multimodal single-cell data with structural similarity. Nucleic Acids Res. 50 (21), e121 (2022).
- . Get Started - JupyterLab 4.1.0a4 documentation Available from: https://jupyterlab.readthedocs.io/en/latest/getting_started/overview.html (2024)
- . MOFA2: training a model in R Available from: https://raw.githack.com/bioFAM/MOFA2_tutorials/master/R_tutorials/getting_started_R.html (2020)
- De Silva, D., et al. Robust T cell activation requires an eIF3-driven burst in T cell receptor translation. eLife. 10, e74272 (2021).
- Li, G., Liang, X., Lotze, M. HMGB1: The central cytokine for all lymphoid cells. Front Immunol. 4, 68 (2013).
- Jassal, B., et al. The reactome pathway knowledgebase. Nucleic Acids Res. 48 (D1), D498-D503 (2020).
- Argelaguet, R., et al. MOFA+: a statistical framework for comprehensive integration of multimodal single-cell data. Genome Biol. 21 (1), 111 (2020).
- Velten, B., et al. Identifying temporal and spatial patterns of variation from multimodal data using MEFISTO. Nat Methods. 19 (2), 179-186 (2022).
- Qoku, A., Buettner, F. Encoding domain knowledge in multi-view latent variable models: A Bayesian approach with structured sparsity. Proceedings of The 26th International Conference on Artificial Intelligence and Statistics. 206, 11545-11562 (2022).
- Multi-Omics Factor Analysis. MOFA Available from: https://biofam.github.io/MOFA2/ (2024)
- Mitchel, J., et al. Tensor decomposition reveals coordinated multicellular patterns of transcriptional variation that distinguish and stratify disease individuals. bioRxiv. , (2023).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유