Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Kardiyovasküler Hastalıklarla Bağlantılı Varyasyon Modellerini ve Moleküler Süreçleri Ortaya Çıkarmak için Denetimsiz Çoklu Oksijen Faktör Analizinin Uygulanması
Bu Makalede
Özet
Farklı ön işleme adımlarını, çoklu omik faktör analizi modelinin tahminini ve çeşitli aşağı akış analizlerini birleştiren karmaşık multi-omik veri kümelerinin denetimsiz analizi için esnek, genişletilebilir Jupyter-lab tabanlı bir iş akışı sunuyoruz.
Özet
Hastalık mekanizmaları genellikle karmaşıktır ve birkaç farklı moleküler sürecin etkileşimi tarafından yönetilir. Karmaşık, çok boyutlu veri kümeleri, bu süreçler hakkında daha fazla içgörü oluşturmak için değerli bir kaynaktır, ancak bu tür veri kümelerinin analizi, örneğin farklı hastalık koşullarından, zaman noktalarından ve süreci farklı çözünürlüklerde yakalayan omiklerden kaynaklanan yüksek boyutluluk nedeniyle zor olabilir.
Burada, akut ve kronik koroner sendromlarda bağışıklık tepkisini yakalayan kan örneklerinden oluşturulan bir veri setine multi-omik faktör analizi (MOFA) uygulayarak böylesine karmaşık bir multiomik veri setini denetimsiz bir şekilde analiz etmek ve keşfetmek için bir yaklaşım sergiliyoruz. Veri seti, numune düzeyinde sitokin verileri, plazma-proteomik ve nötrofil prime-seq ve tek hücreli RNA-seq (scRNA-seq) verileri dahil olmak üzere farklı çözünürlüklerde birkaç testten oluşur. Hasta başına ölçülen birkaç farklı zaman noktasına ve birkaç hasta alt grubuna sahip olarak daha fazla karmaşıklık eklenir.
Analiz iş akışı, verilerin birkaç adımda nasıl entegre edileceğini ve analiz edileceğini ana hatlarıyla belirtir: (1) Veri ön işleme ve uyumlaştırma, (2) MOFA modelinin tahmini, (3) Aşağı akış analizi. 1. Adım, farklı veri türlerinin özelliklerinin nasıl işleneceğini, düşük kaliteli özelliklerin nasıl filtreleneceğini ve daha fazla analiz için dağılımlarını uyumlu hale getirmek üzere bunların nasıl normalleştirileceğini ana hatlarıyla belirtir. 2. Adım, MOFA modelinin nasıl uygulanacağını ve tüm omikler ve özelliklerde veri kümesi içindeki ana varyans kaynaklarının nasıl keşfedileceğini gösterir. Adım 3, yakalanan modellerin aşağı akış analizi için çeşitli stratejiler sunar ve bunları hastalık koşullarına ve bu koşulları yöneten potansiyel moleküler süreçlere bağlar.
Genel olarak, diğer bağlamlara ve çoklu omik veri kümelerine de uygulanabilen farklı moleküler özelliklerden oluşan ana varyasyon eksenlerinin tanımlanmasını sağlamak için karmaşık multi-omik veri kümelerinin denetimsiz veri keşfi için bir iş akışı sunuyoruz (örnek kullanım durumunda sunulan diğer tahliller dahil).
Giriş
Hastalık mekanizmaları genellikle karmaşıktır ve birkaç farklı moleküler sürecin etkileşimi tarafından yönetilir. Belirli hastalıklara yol açan veya bir hastalığın evrimini yöneten karmaşık moleküler mekanizmaları deşifre etmek, hastalıkları anlamak ve tedavi etmek için yeni içgörüler ortaya çıkarabileceğinden, tıbbi önemi yüksek bir görevdir.
Son teknolojik gelişmeler, bu süreçlerin aynı anda daha yüksek bir çözünürlükte (örneğin, tek hücre düzeyinde) ve çeşitli biyolojik katmanlarda (örneğin, DNA, mRNA, kromatin erişilebilirliği, DNA metilasyonu, proteomik) ölçülmesini sağlar. Bu, altta yatan süreçler hakkında daha fazla bilgi oluşturmak için ortaklaşa analiz edilebilen büyük çok boyutlu biyolojik veri kümelerinin artan üretimine yol açar. Aynı zamanda, farklı veri kaynaklarını biyolojik olarak anlamlı bir şekilde birleştirmek ve analiz etmek zorlu bir görev olmaya devam etmektedir1.
Farklı omikler arasındaki farklı teknolojik sınırlar, gürültüler ve değişkenlik aralıkları bir zorluk teşkil eder. Örneğin, tek hücreli RNA dizileme (scRNA-dizilimi) verileri çok seyrektir ve genellikle büyük teknik veya toplu etkilerden etkilenir. Ek olarak, özellik alanı genellikle çok büyüktür ve birkaç bin ölçülen gen veya protein arasında değişirken, örneklem boyutları sınırlıdır. Bu, çeşitli hastalık durumlarını, kafa karıştırıcı faktörleri, zaman noktalarını ve çözümleri içerebilen karmaşık tasarımlarla daha da karmaşıklaşır. Örneğin, sunulan kullanım örneğinde, tek hücreli veya örneklem (toplu) düzeyinde farklı veri türleri mevcuttu. Bunun yanı sıra, veriler eksik olabilir ve analiz edilen tüm denekler için tüm ölçümler mevcut olmayabilir.
Bu zorluklar nedeniyle, farklı omikler ve dahil edilen özellikler hala genellikle yalnızca ayrı ayrı analiz edilmektedir2 entegre bir analiz yapmak yalnızca sürecin tam bir resmini sağlamakla kalmasa da, bir omikten kaynaklanan biyolojik ve teknik gürültüler diğer omikler tarafından da telafi edilebilmektedir 3,4. Bayes yöntemleri, ağ tabanlı yöntemler5,6, çok modlu derin öğrenme7 ve matris çarpanlarına ayırma 8,9 yoluyla boyutsallık azaltma yöntemleri dahil olmak üzere çoklu omik verilerin entegre bir analizini gerçekleştirmek için birkaç farklı yöntem önerilmiştir. İkincisi için, büyük bir kıyaslama çalışmasının10 sonuçları, MOFA9 (multi-omik faktör analizi) yönteminin, verilerin klinik açıklamalara bağlanması gerektiğinde daha uygun araçlardan biri olduğunu göstermiştir.
Özellikle karmaşık ortamlarda, denetimsiz matris çarpanlarına ayırma yöntemleri, karmaşıklığı azaltmak ve farklı veri kaynaklarından ve özelliklerden paylaşılan ve tamamlayıcı sinyalleri çıkarmak için yararlı bir yaklaşımdır. Karmaşık alanı daha düşük dereceli gizli temsillere ayrıştırarak, verilerdeki ana varyans kaynakları hızlı bir şekilde keşfedilebilir ve bilinen ortak değişkenlerle ilişkilendirilebilir. Aynı varyasyon modelinin birden fazla özellik (örneğin, genler veya proteinler) arasında paylaşılması durumunda, gürültü azaltılırken bu birkaç faktöre toplanabilir. Düzenleme, model katsayılarının seyrekliğini artırmak için kullanılabilir, bu da yaklaşımı, özellik alanının büyük olduğu ve örnek sayısının sınırlı olduğu ortamlarda çok uygun hale getirir9.
Bu protokol, karmaşık bir multi-omik veri kümesinin nasıl hızlı bir şekilde keşfedileceğini ve bu veri kümesini karakterize eden ana varyasyon modellerinin nasıl damıtılacağını göstermek için MOFA modelini kullanan esnek bir analiz iş akışı sunar. İş akışı üç ana adımdan oluşur. İlk adımda, Veri ön işleme ve uyumlaştırma, farklı girdi veri türlerine (scRNA-seq, proteomik, sitokin, klinik veriler) dayalı veri ön işleme için farklı stratejiler sunulmaktadır. Protokol, farklı girdi veri kümelerinin özelliklerinin nasıl işleneceğini, düşük kaliteli özelliklerin nasıl filtreleneceğini ve dağıtımlarını uyumlu hale getirmek için bunların nasıl normalleştirileceğini ayrıntılı olarak açıklar. Ayrıca, bu ön işleme kararlarının aşağı akış sonuçlarını nasıl etkileyebileceğini de gösteriyoruz. İkinci adımda, verilere MOFA modeli uygulanır ve elde edilen varyans ayrıştırması, farklı veri kümelerinin entegrasyonunu değerlendirmek için kullanılabilir. Üçüncü adım, yakalanan faktörlerin ortak değişkenlere nasıl bağlanacağını ve bu faktörleri tanımlayan moleküler programların nasıl ortaya çıkarılacağını gösterir. Sunulan iş akışıyla, koroner sendromlardan muzdarip hastalardan oluşan bir veri setindeki klinik ortak değişkenlerle bağlantılı birkaç gizli faktörü çıkarabildik ve önceki bir projeden altta yatan potansiyel çok hücreli bağışıklık programlarını belirleyebildik11. Bu veri kümesini burada kullanacağız, ancak protokol diğer omikler de dahil olmak üzere diğer bağlamlara kolayca uygulanabilir.
Veri seti, stabil kronik koroner sendromlu (CCS), akut koroner sendromlu (ACS) hastalardan ve sağlıklı koronerleri olan bir kontrol grubundan (CCS olmayan) alınan örneklerden oluşmaktadır (Şekil 1). ACS, önceden var olan CCS'deki plak rüptüründen kaynaklanır ve miyokarda giden kan akışının akut olarak bozulmasına ve ardından kalbin iskemik yaralanmasına yol açar. Bu yaralanma, bağışıklık sistemi tarafından enflamatuar bir yanıta ve ardından akut olaydan birkaç gün sonrasına kadar süren onarıcı bir faza neden olur12. ACS hastaları için bu bağışıklık tepkisini karakterize edebilmek için, dört farklı zaman noktasında kan örnekleri alındı: akut (TP1); rekanalizasyondan sonra (14 [± 8] h) (TP2); 60 [± 12] h sonra (TP3); taburcu olmadan önce (6.5 [±1.5] gün) (TP4) (Şekil 1A). CCS ve sağlıklı koronerleri olan hastalar için sadece bir zaman noktası mevcuttu - (TP0). Tüm hastalar ve zaman noktaları için kan örneklerine dayalı farklı testler ölçüldü: inflamasyonun klinik belirteçleri (Kreatin-Kinaz (CK), CK-MB, Troponin, C-reaktif protein (CRP)), periferik kan mononükleer hücrelerinin (PBMC'ler) scRNA-seq'i, sitokin analizi, plazma proteomiği ve nötrofillerin prime-seq13 verileri.
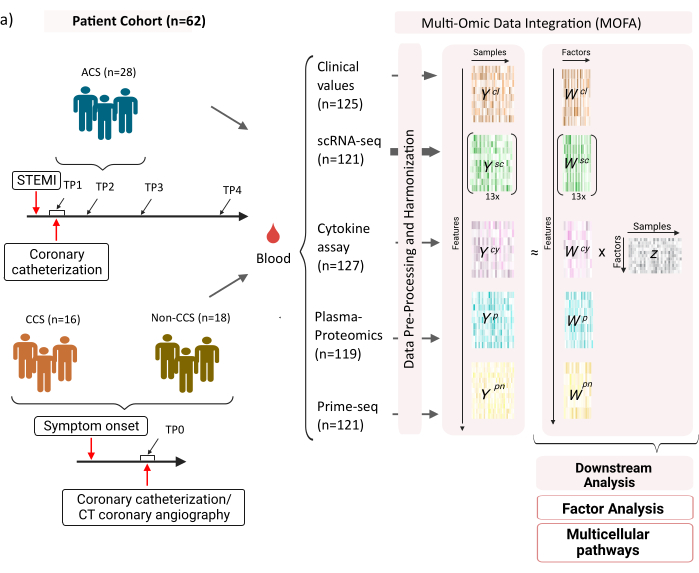
Şekil 1: Miyokard enfarktüsü multi-omik giriş veri seti. Giriş veri seti: Analiz edilen veriler, akut koroner sendromlu (ACS), kronik koroner sendromlu (CCS) ve sağlıklı koroner hastalı (CCS olmayan) hastalardan (n = 62) alınan kan örneklerini içerir. AKS hastaları için kan örnekleri dört farklı zaman noktasında (TP1-4), CCS ve CCS olmayan hastalar için tek bir zaman noktasında (TP0) dahil edildi. Her hasta ve zaman noktası kombinasyonu, analizde ayrı bir numune olarak ele alınır. Numuneler üzerinde farklı omik testler ölçüldü: klinik kan testleri (n = 125), scRNA-seq (n = 121), plazma-proteomik (n = 119), sitokin testi (n = 127) ve nötrofil prime-seq (n = 121). Daha sonra, açıklanan protokol, verileri tüm omiklere entegre etmek ve MOFA modelini ve daha fazla aşağı akış analizini (faktör analizi, yol zenginleştirme) kullanarak keşfetmek için uygulandı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Burada sunulduğu gibi iş akışı için girdi olarak, örneğinyetersiz 14 ön işleme eğitiminde belirtildiği gibi, cellranger ve kalite kontrol (QC) ile işlendikten sonra scRNA-seq verilerinden ham sayımlar alırız. Hücre tipi ek açıklama için otomatik Azimuth15 boru hattını kullandık. Sayımlar daha sonra, her örnek ve hücre tipi için tüm hücrelerdeki ortalama alınarak (psödobulk toplama) her hücre tipi için örnek düzeyinde toplanır. Plazma-proteomik, normalleştirilmiş ve medyan merkezli yoğunluklar olarak dahil edilir ve nötrofiller için, asal sıralamadan umi benzersiz moleküler tanımlayıcı (UMI) ekzon sayılarını alırız. Sitokin ve klinik değerlerde, daha önce herhangi bir ön işlem uygulanmamıştır. (Deneysel) veri üretimi ile ilgili daha fazla ayrıntı, ilgili el yazması11'de özetlenmiştir. Burada sunulan sonuçlar, atıfta bulunulan yayında kullanılan belirteç tabanlı stratejiye kıyasla scRNA-seq verilerindeki hücre tipleri için otomatik Azimut ek açıklamasının kullanılmasına dayandığından, burada sunulan sonuçlar benzerdir, ancak yayında sunulanla tam olarak aynı değildir. El yazmasında, hücre tipi açıklama stratejisinin analizin ana modellerini ve biyolojik yorumlarını değiştirmediği, ancak modelden kaynaklanan kesin değerlerdeki küçük değişikliklerin değişebileceği gösterilebilir. Genel olarak girdi verileri, 10.000'den fazla farklı özelliğin (genler, proteinler, klinik değerler) farklı zaman noktaları ve ölçüm seviyeleri (tek hücreler ve toplu) dahil olmak üzere karmaşık, çok boyutlu bir veri setiydi. MOFA analizinin izlediği sıkı bir ön işleme ve veri uyumlaştırma stratejisinin, verileri keşfetmek ve ilgili bağışıklık programını çıkarmak için yararlı ve hızlı bir araç olduğu gösterilmiştir. Her zaman noktası ve hasta kombinasyonu, MOFA analizinde bağımsız bir numune olarak ele alınır. Her veri türü ve hücre türü, MOFA analizinde ayrı bir görünüm olarak kabul edilir.
Bu protokol, iş akışı için giriş verilerinin hazırlanması, farklı iş akışı adımlarının yürütülmesi, konfigürasyonların özelleştirilmesi, elde edilen rakamların yorumlanması ve yorumlara dayalı olarak konfigürasyonların yinelemeli olarak ayarlanması için talimatlar sağlar. Protokolün farklı adımlarına, her adımda gerekli girdi veri kümelerine ve sonuçta elde edilen rakamlara ve veri kümelerine genel bir bakış, teknik iş akışına genel bakış tarafından verilmektedir (Şekil 2).
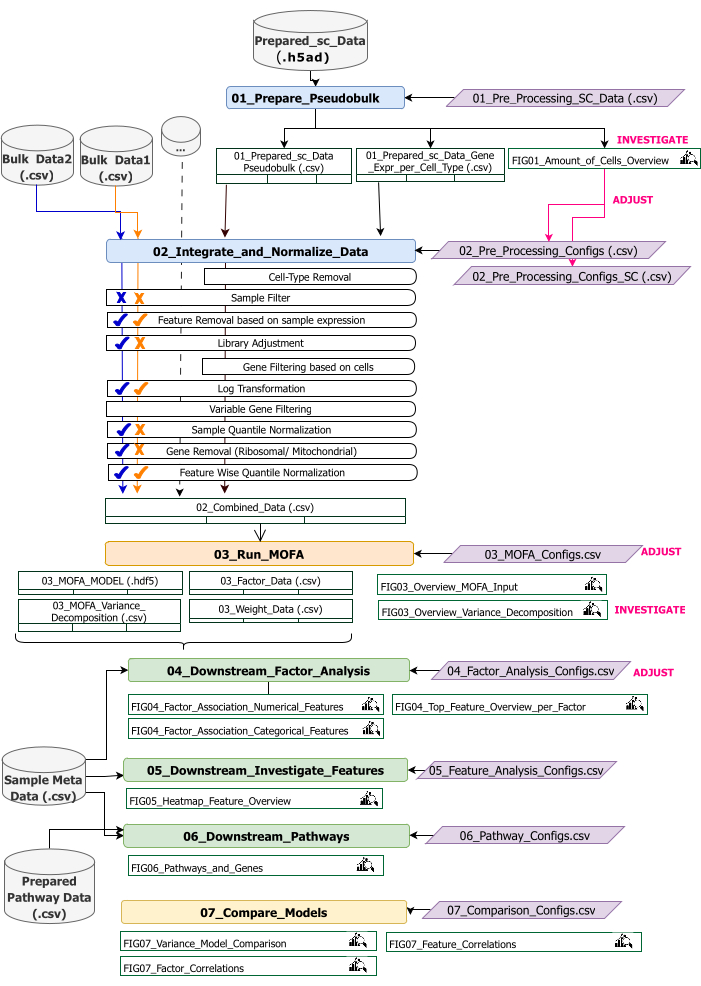
Şekil 2: Teknik iş akışına genel bakış. Multi-omics veri setinin analizi için iş akışının ana hatları. Farklı öğeler farklı renkler ve sembollerle vurgulanır. Veri Ön İşleme ve Uyumlaştırma (1) adımına ait Jupyter Notebook'lar mavi renktedir. 'MOFA Model' (2) basamağına ait Jupyter Notebook'lar turuncu renktedir. 'Aşağı Akış Analizi' (3) adımına ait Jupyter Notebook'lar yeşil renktedir. Sonuçların karşılaştırılması için kullanılacak bir Jupyter Notebook sarı renktedir. İş akışının yürütülmesi için parametrelerin değiştirilebildiği yapılandırma dosyaları mor renkle vurgulanır. İş akışını çalıştırmak için gereken giriş veri kümeleri, veri kümesi simgesiyle gösterilir ve gri renkle vurgulanır. İş akışının yürütülmesi sırasında oluşturulan tüm şekil çıktıları büyüteç sembolü ile gösterilir. İş akışı yürütme sırasında oluşturulan veri kümeleri tablo olarak gösterilir. Genel olarak, iş akışı sırayla yürütülür: (1) Veri Ön İşleme ve Uyumlaştırma iki adımdan oluşur: scRNA-seq giriş verilerine (01_Prepare_Pseudobulk) dayalı bir pseudobulk tablosunun ilk oluşturulması ve ardından bu verilerin diğer tüm örnek düzeyinde (toplu) girdilerle birlikte entegrasyonu ve normalleştirilmesi (02_Integrate_and_Normalize_Data). Bu adımda, konfigürasyon dosyaları aracılığıyla, her bir veri kümesi için belirtilen ön işleme ve normalleştirme adımlarından hangisinin (örn. Örnek Filtre) uygulanması gerektiğini ayrı ayrı yapılandırmak mümkündür. (2) 'MOFA Modeli': MOFA modelini, yapılandırma dosyasında belirtilen konfigürasyonlarla ilk adımın oluşturulan girdisi üzerinde çalıştırır (03_MOFA_configs.csv) (3) 'Aşağı Akış Analizi': oluşturulan MOFA sonuçlarına ilişkin içgörüler oluşturmak ve bunları 'Örnek Meta Data.csv' dosyası aracılığıyla girdi olarak sağlanan örnek meta verilerle (ortak değişkenler) ilişkilendirmek için birbirinden bağımsız olarak çalıştırılabilen üç farklı not defterinden oluşur. (4) 'Model Karşılaştırması': 2. adımda oluşturulan farklı modelleri karşılaştırmak için kullanılabilecek küçük ve ayrı bir adımdır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
İş akışı, R ve Python'da yazılmış birkaç Jupyter Notebook'tan oluşur (iş akışını çalıştırmak için R ve Python dili bilgisi gerekli değildir, ancak hataların ortaya çıkması durumunda yardımcı olabilir). Protokolün çeşitli adımlarında, parametreler konfigürasyon dosyaları (adında '_Configs' sonekini içeren '.csv' dosyaları) aracılığıyla değiştirilir. Protokol içinde, yalnızca varsayılan yapılandırmadan başlayarak değiştirilmesi gereken parametreleri ana hatlarıyla belirtiriz.
Örneğin ön işlemeyi özelleştirmek için başka parametreler de değiştirilebilir. Bu parametrelerin ve açıklamaların bir dokümantasyonu, indirilen depoda bulunan 'Documentation_Config_Parameter' dosyasında verilmiştir.
Protokol
1. Hazırlıklar: Teknik kurulum ve kurulum
NOT: Bu programı çalıştırmak için wget, git ve Apptainer'ın cihaza önceden yüklenmiş olmasını sağlayın. Apptainer'ı farklı sistemlere (Linux, Windows, Mac) kurmak için bir kılavuz burada verilmiştir: https://apptainer.org/docs/admin/main/installation.html. Git ile ilgili kurulum bilgileri burada bulunabilir: https://git-scm.com/book/en/v2/Getting-Started-Installing-Git. Farklı giriş veri kümelerinin boyutuna bağlı olarak, iş akışının uygun bir makinede (16 CPU, 64 GB Bellek) çalıştırılması önerilir. Sağlanan örnek verilerle bir duman testi yerel makinede gerçekleştirilebilir. Örnek veriler üzerinde protokolün çalıştırılmasından elde edilen talimatlar ve beklenen çıktılar Ek Dosya 1'de verilmiştir. Yukarıda özetlenen veri kümesinde yürütülen protokolün önemli adımları için Ek Video Dosyası 1'e bakın.
- Konsolu açın ve tüm analiz kodunun ve çıktılarının saklanacağı bir klasör seçin veya oluşturun. Terminalde şu komutu yazarak klasöre gidin: cd path_to_folder.
- Kod deposunu Github'dan (https://github.com/heiniglab/mofa_workflow) veya terminal penceresine git clone https://github.com/heiniglab/mofa_workflow.git yazarak indirin veya klonlayın.
- Terminal penceresine wget https://zenodo.org/records/11192947/files/mofa_image.sif yazarak gerekli tüm kurulumları içeren görüntüyü Zenodo'dan indirin.
- Terminal penceresine mkdir results yazarak tüm sonuç verilerinin saklanacağı bir klasör oluşturun.
- Terminal penceresine mkdir input_data yazarak analizde kullanılacak tüm giriş verilerinin ekleneceği bir klasör oluşturun.
- Terminalde aşağıdaki komutu yazarak bir JupyterLab oturumu başlatacak kapsayıcıyı yürütün: apptainer run mofa_image.sif. Komut tarafından döndürülen URL'yi tarayıcıya kopyalayın, böylece bir Jupyter-lab oturumu açılır (Jupyter-lab hakkında daha fazla bilgi yazılım belgelerinde16 bulunabilir).
NOT: İş akışı bir dizüstü bilgisayarda yerel olarak yürütüldüğünde, bunun yerine doğrudan yerel bir ana bilgisayar adresi döndürecek olan apptainer exec mofa_image.sif jupyter-lab komutunun kullanılması önerilir. Konteynerin kümelenmiş bir bilgi işlem ortamında yürütülmesi durumunda, ssh aracılığıyla yapılabilen port yönlendirmenin ayarlanması gerekebilir.
2. Başlatma ve veri hazırlama
- Jupyter-Lab oturumunda sol taraftaki gezinti menüsünü kullanın. input_data üzerine çift tıklayarak input_data klasörüne gidin.
- Analiz için girdi olarak kullanılacak tüm veri kümelerini Sürükle ve Bırak özelliğini kullanarak input_data dizinine kopyalayın. Dosyayı şu anda bulunduğu klasörden sürükleyin ve input_data klasörünün altındaki alanda Jupyter-lab oturumuna bırakın.
NOT: Tüm veri kümelerinin .csv veya .h5ad (tek hücreli veriler söz konusu olduğunda) biçiminde olması gerekir. Tüm .csv dosyalarının eşleşen bir sample_id sütunu içermesi gerekir (veri kümelerinde aynı kimliklerin kullanılması gerekir). Diğer tüm sütunlar özellik olarak kullanılacaktır. h5ad- dosyası içinde, hücre ek açıklaması, sample_id ve cluster_id belirten iki tanımlayıcı içermelidir. Bunlar toplama ve eşleştirme için kullanılacaktır. Diğer formatlardaki Omic veri kümelerinin kullanımdan önce belirtilen .csv formatına dönüştürülmesi gerekir (Şekil 3). .h5seurat biçiminde verilen scRNA-seq veri kümeleri, Jupyter-notebook: 00_Data_Conversion.ipynb yürütülerek .h5ad'ye dönüştürülebilir. - Klasör sembolüne tıklayarak ve ardından mofa_workflow, komut dosyaları ve yapılandırmalar klasörlerine çift tıklayarak yapılandırmalar klasörüne gidin. Klasörün içinde, üzerine çift tıklayarak dosyayı Data_configs.csv açın.
- Değer sütununda, input_data (data_path) ve sonuçlar (result_path) klasörlerinin yollarına ekleyin. configuration_name değer sütunundaki tüm kayıtlı dosyalara dosya uzantısı olarak eklenecek bir ad ekleyin (bu protokol [Miyokard Enfarktüsü sürüm 1] MI_v1 kullanılır) (Şekil 4).
- Üst kısımdaki menüden Dosya > CSV Dosyasını Kaydet'e tıklayarak değişiklikleri kaydedin.
- Komut dosyalarına tıklayarak komut dosyaları klasörüne gitmek için sol taraftaki gezinme menüsünü kullanın. 00_Configuration_Update.ipynb'ye çift tıklayarak başlatma not defterini açın. Üst kısımdaki Çekirdeği yeniden başlat ve tüm hücreleri çalıştır düğmesine tıklayarak ve açılır pencerede Yeniden Başlat'a tıklayarak komut dosyasını çalıştırın (Şekil 5).
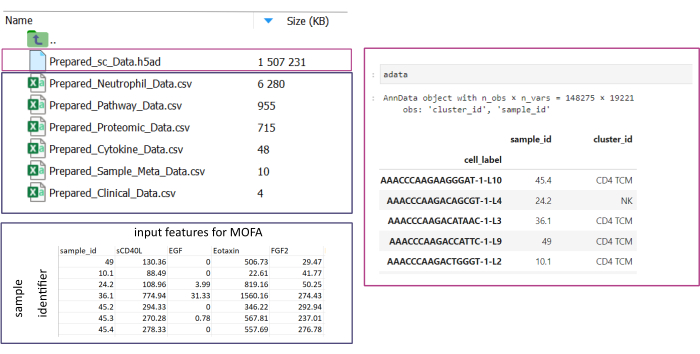
Şekil 3: Veri girişi ve kurulumu. İş akışının yürütülmesi için tüm verilerin belirli bir input_data klasöründe saklanması gerekir. Her giriş veri kümesi için ayrı bir dosya sağlanmalıdır. Tek hücreli veriler, cluster_id üzerinde hücre açıklaması (örneğin, önceki hücre tipi açıklama adımlarından elde edilen) ve bir sample_id sütunu (analiz edilmesi gereken her bir ayrı örneği benzersiz bir şekilde tanımlayan) içeren .h5ad olarak verilmelidir. Diğer tüm girdi veri kümeleri, diğer tüm sütunlarda MOFA analizinde kullanılacak sample_id (tek hücreli verinin karşılık gelen sütunuyla eşleşen) ve özellikleri belirten bir sütun da dahil olmak üzere '.csv' biçiminde verilmelidir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
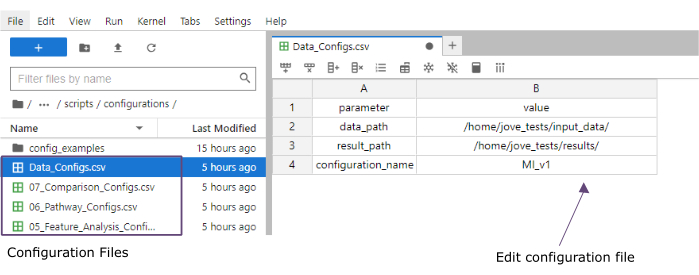
Şekil 4: Jupyter-lab yapılandırma dosyaları. İş akışının yürütülmesi sırasında, parametrelerdeki değişiklikler (örn. filtreleme seçeneklerinin ayarlanması vb.) '.csv' konfigürasyon dosyaları aracılığıyla belirtilir. Klonlanan depo içinde, her adım için varsayılan yapılandırma dosyaları dahil edilir. Bunlar, bir elektronik tabloda olduğu gibi doğrudan jupyter-lab konsolunda düzenlenebilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Şekil 5: Jupyter-notebooks betikleri. İş akışının tamamı, ilgili yapılandırma dosyaları değiştirildikten sonra sırayla yürütülecek bir dizi Jupyter not defterinden oluşur. Sol taraftaki Jupyter not defterine çift tıklandığında, ilgili dosya sağ tarafta açılacaktır. Dosyanın tam olarak yürütülmesi, üst kısımda vurgulanan düğme ile başlatılabilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
3. Veri ön işleme ve uyumlaştırma
- Ön işleme - sc verilerini pseudobulk'a dönüştürün.
NOT: Bu adımın yalnızca analizde tek hücreli veriler kullanılıyorsa yürütülmesi gerekir.- Yapılandırmaya çift tıklayarak yapılandırmalar klasörüne gitmek için sol taraftaki gezinme menüsünü kullanın. Çift tıklayarak 01_Preprocessing_SC_Data.csv dosyayı açın. Dosyadaki otomatik olarak doldurulan değerleri kontrol edin ve gerekirse, data_name sütunundaki değerleri, analiz için kullanılacak input_data klasöründeki tek hücreli veri kümelerinin dosya adlarına karşılık gelecek şekilde ayarlayın.
NOT: Varsayılan olarak, giriş verileri klasöründeki .h5ad dosyalarının tüm adları, başlatma komut dosyasındaki yapılandırma dosyasına eklenecektir. Bazı veri kümelerinin analiz için kullanılmaması gerekiyorsa, buradan kaldırılabilir. - Üstteki menüde Dosya > CSV Dosyasını Kaydet'e tıklayarak yapılan değişiklikleri kaydedin.
- Komut dosyalarına tıklayarak komut dosyaları klasörüne gitmek için sol taraftaki gezinme menüsünü kullanın. Not defterini 01_Prepare_Pseudobulk.ipynb üzerine çift tıklayarak açın. Üst kısımdaki Çekirdeği yeniden başlat ve tüm hücreleri çalıştır düğmesine tıklayarak ve açılır pencerede Yeniden Başlat'a tıklayarak komut dosyasını çalıştırın.
- Önce şekillere , ardından 01_figures üzerine çift tıklayarak şekiller klasörüne gitmek için sol taraftaki gezinme menüsünü kullanın. Yeni oluşturulan arsa FIG01_Amount_of_Cells_overview üzerine çift tıklayarak açın.
NOT: Dizüstü bilgisayarın çalıştırılması birkaç dakika sürebilir. Not defteri başarıyla yürütüldüğünde bir açılır pencere görüntülenir ve dosya FIG01_Amount_of_Cells_Overview not defteri tarafından güncelleştirilir veya yeni oluşturulur. Son Değiştirilme Yeri sütunu, dosyanın yeni mi yoksa eski mi olduğunu değerlendirmek için dosyanın ne zaman oluşturulduğunu gösterebilir. - Örnek başına çok az sayıda hücreye sahip hücre tipi kümeleri belirlemek için grafiği inceleyin. Sonraki adımlarda hariç tutmak için bu cluster_ids adlarını not edin (Şekil 6).
- Konfigürasyonlar klasörüne geri dönmek için sol taraftaki gezinme menüsünü kullanın... ve ardından yapılandırmalara çift tıklayın. Dosyayı 02_Preprocessing_Configs_SC.csv üzerine çift tıklayarak açın.
- configuration_name ve data_name sütunlarındaki değerleri kontrol edin ve gerekirse ayarlayın.
NOT: Başlatma komut dosyası içinde, bu değerler giriş verileri klasöründeki .h5ad dosyalarının tüm adları ve daha önce Data_Configs.csv dosyasında ayarlanan configuration_name değeri ile önceden doldurulur. Dosyaların analizden çıkarılması veya dosya adları için başka bir uzantı kullanılması gerektiği durumlarda, bu burada ayarlanabilir. - cell_type_exclusion sütunundaki değeri ayarlayın ve önceki adımda hariç tutulmak üzere tanımlanan tüm cluster_id'leri ',' ile ayırarak ekleyin.
- Üstteki gezinme çubuğunda Dosya > CSV Dosyasını Kaydet'e tıklayarak değişiklikleri kaydedin.
- Yapılandırmaya çift tıklayarak yapılandırmalar klasörüne gitmek için sol taraftaki gezinme menüsünü kullanın. Çift tıklayarak 01_Preprocessing_SC_Data.csv dosyayı açın. Dosyadaki otomatik olarak doldurulan değerleri kontrol edin ve gerekirse, data_name sütunundaki değerleri, analiz için kullanılacak input_data klasöründeki tek hücreli veri kümelerinin dosya adlarına karşılık gelecek şekilde ayarlayın.
- Ön işleme - Diğer omics veri kaynaklarını uyumlu hale getirin ve entegre edin.
- Üzerine çift tıklayarak 02_Preprocessing_Configs.csv dosyayı açın ve dahil edilecek ve data_input klasöründe (veri kümesi başına bir satır) depolanacak veri kümelerinin her biri için ön işleme yapılandırmasını ayarlayın.
- configuration_name ve data_name sütunlardaki değerleri kontrol edin ve gerekirse ayarlayın.
- Hangi ön işleme adımlarının uygulanması gerektiğine bağlı olarak sütunlardaki diğer parametreleri uygun şekilde ayarlayın.
NOT: input_dataset klasöründe bulunan ancak verilerin tek veri türlerine özgü olmayan her veri kümesi için varsayılan değerler eklenir. Bu nedenle, ayarlamalar gerekli olacaktır. Parametrelerin ayrıntılı bir dokümantasyonu Documentation_Config_Parameter.doc dosyasında verilmiştir. - Dosya > CSV Dosyasını Kaydet'e tıklayarak değişiklikleri kaydedin.
- Komut dosyalarına tıklayarak komut dosyaları klasörüne gitmek için sol taraftaki gezinme menüsünü kullanın. Not defterini 02_Integrate_and_Normalized_Data_Sources.ipynb üzerine çift tıklayarak açın. Üst kısımdaki Çekirdeği yeniden başlat ve tüm hücreleri çalıştır düğmesine tıklayarak ve açılır pencerede Yeniden Başlat'a tıklayarak komut dosyasını çalıştırın.
- Oluşturulan 02_results klasörüne gitmek için sol taraftaki gezinme menüsünü kullanın, klasör sembolüne tıklayın ve ardından sonuçlara çift tıklayın ve 02_results. Birleştirilmiş önceden işlenmiş veri giriş dosyasını içeren 02_Combined_data_'configuration_name'_Integrated.csv dosyasını içerip içermediğini kontrol edin.
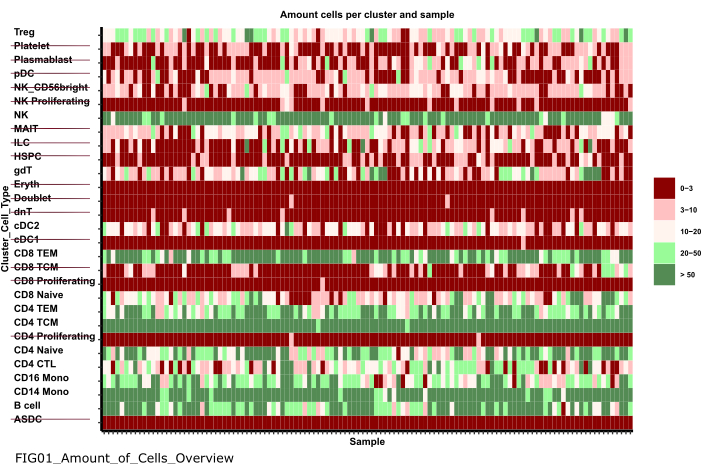
Şekil 6: Veri ön işleme ve uyumlaştırma. '01_Prepare_Pseudobulk' adımının bir çıktısı 'Fig01_Amount_of_Cells_Overview' grafiğidir. Burada, her cluster_id için (önceki hücre tipi açıklama adımlarından hücre tipini gösteren y ekseni), örnek başına hücre sayısı ('sample_id') verilir. Sunulan sonuçlar içinde, numune başına düşük miktarda hücreye sahip hücre tipleri, sonraki analizden hariç tutulur (üstü çizili ile gösterilir). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
4. MOFA'yı Çalıştırma
- Jupyter-Lab'da, klasör sembolüne tıklayarak ve ardından mofa_workflow'ye çift tıklayarak, ardından betiklere ve yapılandırmalara çift tıklayarak yapılandırmalar klasörüne gitmek için sol taraftaki gezinti menüsünü kullanın. Dosyayı 03_MOFA_Configs.csv üzerine çift tıklayarak açın.
- configuration_name ve mofa_result_name sütunlarının girişlerini kontrol edin ve alternatif adların kullanılması gerekiyorsa girişleri ayarlayın.
NOT: mofa_result_name , MOFA'ya dayalı olarak oluşturulan tüm sonuç dosyalarına bir dosya uzantısı olarak eklenecektir. Farklı MOFA kurulumları aynı giriş verileriyle çalıştırılabileceğinden , bu configuration_name değerinden farklı olabilir (bu protokol MI_v1_MOFA kullanır). - MOFA modelinde tahmin edilmesi gereken faktörlerin miktarını (amount_of_factors sütun) girin ve dosyadaki değerleri ayarlayarak ağırlıklandırma ve ölçeklendirmenin uygulanıp uygulanmayacağını tanımlayın (weighting_of_views ve scale_views sütunlar).
- Dosya > CSV Dosyasını Kaydet'e tıklayarak değişiklikleri kaydedin.
- 'Komut dosyaları'na tıklayarak komut dosyaları klasörüne gitmek için sol taraftaki gezinme menüsünü kullanın. Dosyaya çift tıklayarak 03_Run_MOFA.ipynb not defterini açın. Üst kısımdaki Çekirdeği yeniden başlat ve tüm hücreleri çalıştır düğmesine tıklayarak ve açılır pencerede Yeniden Başlat'a tıklayarak komut dosyasını çalıştırın.
- Şekillere çift tıklayarak 03_figures klasörüne gidin ve ardından 03_figures. Oluşturulan grafiği FIG03_Overview_Variance_Decomposition_'mofa_result_name açın ve model sonucunu inceleyin (Şekil 7A).
- Oluşturulan 03_results klasörüne gitmek için sol taraftaki gezinme menüsünü kullanın, klasör sembolüne tıklayın ve ardından sonuçlara çift tıklayın ve 03_results. 03_Factor_Data_'mofa_result_name'.csv örnek faktör değeri dosyasını ve 03_Weight_Data_'mofa_result_name'.csv özellik faktörü ağırlık dosyasını içerip içermediğini kontrol edin.
5. Aşağı akış analizi
- Faktör yorumlama.
- Klasör sembolüne tıklayarak ve ardından input_data çift tıklayarak input_data klasörüne gitmek için sol taraftaki Gezinme menüsünü kullanın.
- Oluşturulan faktörlerle ilişkili olarak analiz edilecek örneklerin tüm meta verilerini (ortak değişkenleri) içeren bir .csv dosyası (Prepared_Sample_Meta_Data.csv) hazırlayın. Sürükle ve Bırak özelliğini kullanarak dosyayı input_data klasörüne kopyalayın ve dosyayı input_data klasörünün klasör genel görünümüne bırakın.
NOT: Daha önce kullanılan verilerle eşleştirmek için sample_id sütununu ve analiz edilmesi gereken her özellik için daha fazla sütun içermesi gerekir. - Jupyter-Lab'da, klasör sembolüne tıklayarak ve ardından mofa_workflow'ye çift tıklayarak, ardından betikler ve yapılandırmayla yapılandırmalar klasörüne geri dönmek için soldaki gezinti menüsünü kullanın. Dosyayı 04_Factor_Analysis.csv üzerine çift tıklayarak açın.
- configuration_name ve mofa_result_name girişlerinin, kodda analiz edilecek yapılandırma ve MOFA sonuçlarının adlarını içerip içermediğini kontrol edin ve gerekirse bunları ayarlayın.
- numeric_covariates sütunda, MOFA faktörleriyle ilişkili olarak araştırılacak olan Prepared_Sample_Meta_Data.csv dosyasındaki tüm sayısal sütunların adını virgülle ayırarak ekleyin (Bu protokol CRP,CK kullanır).
- categorical_covariates' sütununda, MOFA faktörleriyle ilişkili olarak araştırılacak olan Prepared_Sample_Meta_Data.csv dosyasındaki tüm kategorik sütunların adını virgülle ayrılmış olarak ekleyin (Bu protokol ölçüm kullanır).
- Dosya > CSV Dosyasını Kaydet'e tıklayarak değişiklikleri kaydedin.
- Komut dosyalarına tıklayarak 'komut dosyaları' klasörüne gitmek için soldaki gezinme menüsünü kullanın. Not defterini 04_Downstream_Factor_Analysis.ipynb üzerine çift tıklayarak açın. Üst kısımdaki Çekirdeği yeniden başlat ve tüm hücreleri çalıştır düğmesine tıklayarak ve açılır pencerede yeniden başlat'a tıklayarak komut dosyasını çalıştırın.
- Şekillere çift tıklayarak 04_figures klasörüne gitmek için soldaki gezinme menüsünü kullanın ve ardından 04_figures. Oluşturulan grafikleri üzerlerine çift tıklayarak açın ve ilginç desenler ve ilişkilendirmeler için faktörleri araştırın:FIG04_Factor_Association_with_numeric_features_
'mofa_result_name.pdf (Şekil 7B). FIG04_Factor_Association_
with_categorical_features_'mofa_result_name.pdf (Şekil 7C). FIG04_Top_Feature_Overview_per_Factor _'mofa_result_name.pdf (Şekil 8A).
- Özellik analizi
- Konfigürasyonlar klasörüne geri dönmek için soldaki gezinme menüsünü kullanın... ve ardından yapılandırmalara çift tıklayın. Dosyayı 05_Feature_Analysis_Configs.csv üzerine çift tıklayarak açın.
- configuration_name ve mofa_result_name sütunlarının girişlerinin, aşağı akış analizi için kullanılacak konfigürasyonun ve oluşturulan MOFA sonucunun adlarına karşılık gelip gelmediğini kontrol edin ve gerekirse bunları ayarlayın.
- Faktör sütununa, bir sonraki komut dosyasında en üst özelliklerin çizileceği faktörü ekleyin.
- Sütun faceting_variable, çizimdeki örnekleri gruplandırmak için kullanılacak Prepared_Sample_Meta_Data kategorik bir sütunun sütun adını ekleyin.csv (Bu protokol ölçüm kullanır)
- Dosya > CSV Dosyasını Kaydet'e tıklayarak değişiklikleri kaydedin.
- Komut dosyalarına tıklayarak komut dosyaları klasörüne gitmek için soldaki gezinme menüsünü kullanın. Not defterini 05_Downstream_Investigate_Features_Heatmap.ipynb üzerine çift tıklayarak açın. Üst kısımdaki Çekirdeği yeniden başlat ve tüm hücreleri çalıştır düğmesine tıklayarak ve açılır pencerede Yeniden Başlat'a tıklayarak komut dosyasını çalıştırın.
- Önce şekillere, ardından 05_figures üzerine çift tıklayarak 05_figures klasörüne gitmek için soldaki gezinme menüsünü kullanın. Oluşturulan grafiği 'mofa_result_name' FIG05_Heatmap_Feature_Overview__ açın ve araştırın.pdf'ye çift tıklayarak (Şekil 8B).
NOT: Çizimde gösterilecek özelliklerin miktarına bağlı olarak, 05_Feature_Analysis_Configs.csv içinde plot_width ve plot_height parametrelerini ayarlamak ve her şeyin çizime uyduğundan emin olmak için komut dosyasını yeniden çalıştırmak gerekebilir.
- Yol analizi
- Klasör sembolüne tıklayarak ve ardından input_data çift tıklayarak input_data klasörüne gitmek için sol taraftaki Gezinme menüsünü kullanın.
- Zenginleştirme için test edilecek yolların listesini içeren bir .csv dosyası (Prepared_Pathway_Data.csv) hazırlayın. Sürükle ve Bırak özelliğini kullanarak dosyayı input_data klasörünün klasör genel görünümüne bırakarak dosyayı input_data klasörüne kopyalayın.
NOT: Üç sütun içermesi gerekir: ID (yolun benzersiz bir tanımlayıcısı), gen (yola ait gen adları (SEMBOL) tarafından verilen genler, gen başına bir satır), pathway_name (yolların adı/metinsel açıklaması). - Jupyter-Lab oturumunda, üzerine tıklayarak yapılandırmalar klasörüne gitmek için soldaki gezinme menüsünü kullanın... ve ardından yapılandırmalara çift tıklayın. Dosyayı 06_Pathway_Configs.csv üzerine çift tıklayarak açın.
- mofa_result_name sütununun girişini kontrol edin ve yol zenginleştirme hesaplaması için kullanılacak oluşturulan MOFA sonucunun adına karşılık geldiğinden emin olun.
- Türler sütunundaki girişi kontrol edin ve türler sütunundaki Prepared_Pathway_Data.csv dosyasındaki gen sütunuyla eşleşen özellikler içermeyen girişleri kaldırın.
NOT: Varsayılan olarak, MOFA modelinde kullanılmış olan tüm farklı görünümler, iş akışının yürütülmesi sırasında bu dosyaya eklenir. En az bir yolla eşleşen özellikler içermeyen görünümler olması durumunda, bunların kaldırılması gerekir; Aksi takdirde, yürütme başarısız olur. Bir örnek, yol dosyasının yalnızca genler için yol açıklamalarını içermesi olabilir, ancak protein adlarını içeren bir görünüm vardır. - Dosya > CSV Dosyasını Kaydet'e tıklayarak değişiklikleri kaydedin.
- Komut dosyalarına tıklayarak komut dosyaları klasörüne gitmek için gezinme menüsünü kullanın. Not defterini 06_Downstream_Pathways.ipynb üzerine çift tıklayarak açın. Üst kısımdaki Çekirdeği yeniden başlat ve tüm hücreleri çalıştır düğmesine tıklayarak ve açılır pencerede Yeniden Başlat'a tıklayarak komut dosyasını çalıştırın.
- Önce şekillere çift tıklayarak ve ardından 06_figures ederek 06_figures klasörüne gitmek için soldaki gezinme menüsünü kullanın. Oluşturulan grafiği FIG06_Pathways_and_Genes_ 'mofa_result_name üzerine çift tıklayarak açın ve görselleştirilmiş yolları inceleyin (Şekil 8C).
NOT: Görselleştirilen yolların nasıl seçileceği, yapılandırma dosyası aracılığıyla yapılandırılabilir. Daha fazla ayrıntı için parametrelerin belgelerine bakın. - Oluşturulan 06_results klasörüne gitmek için sol taraftaki gezinme menüsünü kullanın, böylece klasör sembolüne tıklayın ve ardından sonuçlara çift tıklayın ve 06_results. 'mofa_result_name' 06_Pathway_enrichment__ zenginleştirilmiş yolları içeren dosyayı içerip içermediğini kontrol edin.
6. Farklı konfigürasyonların ve sürümlerin karşılaştırılması (Ek Şekil 1, Ek Şekil 2, Ek Şekil 3, Ek Şekil 4)
- İş akışı boyunca farklı parametreler/konfigürasyonlar kullanmanın etkisini karşılaştırmak için, konfigürasyon dosyalarındaki parametreleri değiştirerek ve farklı configuration_name ve mofa_result_name tanımlayıcılar kullanarak 3-5 arasındaki bölümleri yeniden çalıştırın.
NOT: Yeni sonuçlar, farklı çalıştırmaları karşılaştırmak için kullanılmak üzere bu adlarla saklanacaktır. - Jupyter-Lab'da, yapılandırmalar klasörüne gitmek için soldaki gezinti menüsünü kullanın. Dosyayı 07_Comparison_Configs.csv üzerine çift tıklayarak açın.
- mofa_result_name sütununa, karşılaştırılacak önceki tüm MOFA çalıştırmalarının adlarını ekleyin (ad/yapılandırma başına bir satır, ör. MI_v1_MOFA, MI_v2_MOFA).
- compare_factors sütununda, modeller arasında karşılaştırılacak faktörleri ekleyin. Varsayılan olarak, Faktör1,Faktör2,Faktör3'tür. (Ek Şekil 2A).
NOT: Bu komut dosyasında, farklı modellerin özellik ve faktör değerleri ilişkilendirilerek karşılaştırılacaktır. Bu, yalnızca aynı örneklere ( sample_id ile gösterilir) ve aynı özellik kümesine dayanan modeller için geçerlidir. Karşılaştırılan sürümler arasında örnekler veya özellikler eşleşmezse, bunlar karşılaştırmadan hariç tutulacaktır. - Dosya > CSV Dosyasını Kaydet'e tıklayarak değişiklikleri kaydedin.
- Komut dosyalarına tıklayarak komut dosyaları klasörüne gitmek için soldaki menüyü kullanın. Not defterini 07_Compare_Models.ipynb üzerine çift tıklayarak açın. Çekirdeği yeniden başlat ve tüm hücreleri çalıştır düğmesine tıklayarak ve açılır pencerede Yeniden Başlat'a tıklayarak komut dosyasını çalıştırın.
- Önce şekillere çift tıklayarak ve ardından 06_figures ederek 06_figures klasörüne gitmek için soldaki menüyü kullanın. Farklı sürümlerin benzerliğini analiz etmek için dosyalara çift tıklayarak oluşturulan grafikleri açın:
FIG07_Variance_Model_Comparison.pdf (Ek Şekil 2B)
FIG07_Factor_Correlations.pdf
(Ek Şekil 2C)
FIG07_Feature_Correlations.pdf
(Ek Şekil 3C)
7. İş akışının genişletilmesi: Diğer parametrelerin ve konfigürasyonların eklenmesi
NOT: Yapılandırma dosyalarında şu anda yapılandırılabilir olan parametrelerin yanı sıra, koddaki veya diğer parametrelerdeki diğer ayarlamalar da dahil edilebilir. Örneğin, MOFA modelinin kendisi, doğrudan kodda değiştirilebilen veya konfigürasyon dosyaları aracılığıyla ayarlanabilir hale getirilebilen birkaç başka eğitim parametresi17 sunar. Protokolün bir sonraki bölümünde, ek MOFA model eğitim parametreleri için bunun nasıl yapılacağına dair bir örnek özetlenecektir. Bu kısım için R programlama bilgisi gereklidir.
- Jupyter-Lab'da, betikler klasörüne gitmek için soldaki gezinti menüsünü kullanın. Üzerine çift tıklayarak 03_Run_MOFA.ipynb not defterini açın.
- Sol taraftaki İçindekiler sekmesine tıklayın ve ardından 4.3 MOFA Eğitim Seçeneklerini Ayarlama alt bölümüne gidin ve üzerine tıklayarak Model Eğitimini çalıştırın. Not defterindeki yapılandırılabilir parametrelerin MOFA modelinin basılı çıktısını görmek için aşağı kaydırın.
- Başlığın altındaki kodda yer alan R for döngüsü içinde tüm MOFA verileri, modeli ve eğitim seçenekleri ayarlanır. model_opts$num_factors = mofa_configs$amount_of_factors[i] satırının altına aşağıdaki kodu içeren başka bir satır ekleyin
model_opts$olasılıklar['data_type'] = 'poisson'.
NOT: Bu, tüm MOFA çalıştırmaları için data_type adıyla belirtilen görünüm için modelin girdi olarak aldığı dağılımı değiştirecektir. Bir veri türü için poisson belirtilirken, model yalnızca bu veri türü için özellikler tamsayı olduğunda (örneğin, RNA-seq'den okuma sayıları) çalışacaktır. MOFA verileri, eğitimi ve model seçenekleri hakkında daha fazla bilgi edinmek için MOFA eğitimlerine ve belgelerinede başvurulabilir 17. - Üst kısımdaki Kaydet düğmesine tıklayarak not defterindeki değişiklikleri kaydedin.
- .csv konfigürasyon dosyaları aracılığıyla yeni parametreleri teslim etmek için, konfigürasyonlara çift tıklayarak konfigürasyonlar klasörüne gitmek için sol taraftaki gezinmeyi kullanın ve dosyayı çift tıklayarak 03_MOFA_Configs.csv açın.
- Parametre adını belirten yeni bir sütun ekleyin, örneğin number_iterations ve bir değer girin, örneğin 1000. Dosya > CSV Dosyasını Kaydet'e tıklayarak değişiklikleri kaydedin.
- Komut dosyalarına tıklayarak komut dosyaları klasöründe gezinmek için gezinme menüsünü kullanın. Not defterini 03_Run_Mofa.ipynb üzerine çift tıklayarak açın. Sol taraftaki İçindekiler sekmesine tıklayın ve ardından 4.3 MOFA Eğitim Seçeneklerini Ayarlama alt bölümüne gidin ve üzerine tıklayarak Model Eğitimini çalıştırın.
- train_opts$maxiter = 50000 satırını train_opts$maxiter = mofa_configs$column_name[i] ile değiştirin (eklenen sütunun adı number_of_iterations olduğunda train_opts$maxiter = mofa_configs$number_of_iterations[i]) olur.
NOT: Bu not defterinde 03_MOFA_Configs.csv yapılandırma dosyası, not defterinin başında (alt bölüm: Önkoşullar Yapılandırmalar ve Parametreler) oturumda mofa_config data.frame olarak okunur ve bu nedenle, bu kod satırında bu nesneye ve karşılık gelen yeni oluşturulan sütuna başvurulur. Aynı anda birden fazla konfigürasyon çalıştırılabildiğinden, model tahmini .csv dosyasındaki tüm farklı satırlar boyunca bir for döngüsünde çalıştırılırken i, data.frame satırını tanımlar. Not defterinin başındaki 'Ön Koşullar, Yapılandırmalar ve Parametreler' bölümündeki yapılandırma dosyasında okuma prensibi tüm dizüstü bilgisayarlar için aynıdır ve bu şekilde daha fazla değişiklik yapılabilir. - Kaydet düğmesine tıklayarak not defterindeki değişiklikleri kaydedin.
Sonuçlar
İş akışının başarılı bir şekilde yürütülmesinin ardından, Şekil 2'de gösterildiği gibi birkaç tablo ve şekil oluşturulur. Şekiller /figures klasörüne yerleştirilir (Şekil 6, Şekil 7, Şekil 8, Ek Şekil 1, Ek Şekil 2, Ek Şekil 3, Ek Şekil 4) ve tablolar beli...
Tartışmalar
Ana hatlarıyla belirtilen protokolle, karmaşık bir çoklu omik veri kümesini hızlı bir şekilde keşfetmek için kullanılabilecek modüler ve genişletilebilir Jupyter-notebook tabanlı bir iş akışı sunulmaktadır. İş akışının ana kısımları, ön işleme ve veri uyumlaştırma bölümünden (verilerin filtrelenmesi ve normalleştirilmesi için farklı standart adımlar sunar), MOFA9 modelinin tahmininden ve bazı örnek aşağı akış analizler...
Açıklamalar
Yazarlar herhangi bir çıkar çatışması beyan etmemektedir.
Teşekkürler
C.L., "Münih Veri Bilimi Okulu - MUDS" ortak araştırma okulu altındaki Helmholtz Derneği tarafından desteklenmektedir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Apptainer | NA | NA | https://apptainer.org/docs/admin/main/installation.html |
| Compute server or workstation or cloud (Linux, Mac or Windows environment). Depending on the size of the different input datasets we recommend running the workflow on a suitable machine (in our setting we use: 16 CPU, 64GB Memory) | Any manufacturer | 16 CPU, 64GB Memory | Large Memory is only required for the processing of the raw single cell data. After preprocessing the later analysis steps can also be performed on regular desktop or laptop computers |
| git | NA | NA | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git |
| GitHub | GitHub | NA | https://github.com/heiniglab/mofa_workflow |
Referanslar
- Lähnemann, D., et al. Eleven grand challenges in single-cell data science. Genome Biol. 21 (1), 31 (2020).
- Colomé-Tatché, M., Theis, F. J. Statistical single cell multi-omics integration. Curr Opin Syst Biol. 7, 54-59 (2018).
- Hawe, J., Theis, F., Heinig, M. Inferring interaction networks from multi-omics data. Front Genet. 10, 535 (2019).
- Hawe, J. S., et al. Network reconstruction for trans acting genetic loci using multi-omics data and prior information. Genome Med. 14 (1), 125 (2022).
- Koh, H. W. L., Fermin, D., Vogel, C., Choi, K. P., Ewing, R. M., Choi, H. iOmicsPASS: network-based integration of multiomics data for predictive subnetwork discovery. NPJ Syst Biol Appl. 5, 22 (2019).
- Ogris, C., Hu, Y., Arloth, J., Müller, N. S. Versatile knowledge guided network inference method for prioritizing key regulatory factors in multi-omics data. Sci Rep. 11, 6806 (2021).
- Lee, C., vander Schaar, M. A variational information bottleneck approach to multi-omics data integration. Proceedings of The 24th International Conference on Artificial Intelligence and Statistics. 130, 1513-1521 (2021).
- Singh, A., et al. DIABLO: an integrative approach for identifying key molecular drivers from multi-omics assays. Bioinformatics. 35 (17), 3055-3062 (2019).
- Argelaguet, R., et al. Multi-omics factor analysis-a framework for unsupervised integration of multi-omics data sets. Mol Syst Biol. 14 (6), e8124 (2018).
- Cantini, L., et al. Benchmarking joint multi-omics dimensionality reduction approaches for the study of cancer. Nature Commun. 12 (1), 124 (2021).
- Pekayvaz, K., et al. Multiomic analyses uncover immunological signatures in acute and chronic coronary syndromes. Nature Medicine. 30 (6), 1696-1710 (2024).
- Swirski, F. K., Nahrendorf, M. Cardioimmunology: the immune system in cardiac homeostasis and disease. Nat Rev Immunol. 18 (12), 733-744 (2018).
- Janjic, A., et al. Prime-seq, efficient and powerful bulk RNA sequencing. Genome Biol. 23 (1), 88 (2022).
- Wolf, F. A., Angerer, P., Theis, F. J. SCANPY: large-scale single-cell gene expression data analysis. Genome Biol. 19 (1), 15 (2018).
- Cao, Y., et al. Integrated analysis of multimodal single-cell data with structural similarity. Nucleic Acids Res. 50 (21), e121 (2022).
- . Get Started - JupyterLab 4.1.0a4 documentation Available from: https://jupyterlab.readthedocs.io/en/latest/getting_started/overview.html (2024)
- . MOFA2: training a model in R Available from: https://raw.githack.com/bioFAM/MOFA2_tutorials/master/R_tutorials/getting_started_R.html (2020)
- De Silva, D., et al. Robust T cell activation requires an eIF3-driven burst in T cell receptor translation. eLife. 10, e74272 (2021).
- Li, G., Liang, X., Lotze, M. HMGB1: The central cytokine for all lymphoid cells. Front Immunol. 4, 68 (2013).
- Jassal, B., et al. The reactome pathway knowledgebase. Nucleic Acids Res. 48 (D1), D498-D503 (2020).
- Argelaguet, R., et al. MOFA+: a statistical framework for comprehensive integration of multimodal single-cell data. Genome Biol. 21 (1), 111 (2020).
- Velten, B., et al. Identifying temporal and spatial patterns of variation from multimodal data using MEFISTO. Nat Methods. 19 (2), 179-186 (2022).
- Qoku, A., Buettner, F. Encoding domain knowledge in multi-view latent variable models: A Bayesian approach with structured sparsity. Proceedings of The 26th International Conference on Artificial Intelligence and Statistics. 206, 11545-11562 (2022).
- Multi-Omics Factor Analysis. MOFA Available from: https://biofam.github.io/MOFA2/ (2024)
- Mitchel, J., et al. Tensor decomposition reveals coordinated multicellular patterns of transcriptional variation that distinguish and stratify disease individuals. bioRxiv. , (2023).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır