Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Anwendung der unüberwachten Multi-Omik-Faktorenanalyse zur Aufdeckung von Variationsmustern und molekularen Prozessen im Zusammenhang mit Herz-Kreislauf-Erkrankungen
In diesem Artikel
Zusammenfassung
Wir präsentieren einen flexiblen, erweiterbaren Jupyter-Lab-basierten Workflow für die unüberwachte Analyse komplexer Multi-Omics-Datensätze, der verschiedene Vorverarbeitungsschritte, die Schätzung des Multi-Omics-Faktorenanalysemodells und mehrere nachgelagerte Analysen kombiniert.
Zusammenfassung
Krankheitsmechanismen sind in der Regel komplex und werden durch das Zusammenspiel mehrerer unterschiedlicher molekularer Prozesse bestimmt. Komplexe, mehrdimensionale Datensätze sind eine wertvolle Ressource, um mehr Einblicke in diese Prozesse zu gewinnen, aber die Analyse solcher Datensätze kann aufgrund der hohen Dimensionalität, die sich beispielsweise aus unterschiedlichen Krankheitszuständen, Zeitpunkten und Omics ergibt, die den Prozess mit unterschiedlichen Auflösungen erfassen, eine Herausforderung darstellen.
Hier stellen wir einen Ansatz vor, um einen solch komplexen Multiomics-Datensatz auf unüberwachte Weise zu analysieren und zu erforschen, indem wir die Multi-Omics-Faktorenanalyse (MOFA) auf einen Datensatz anwenden, der aus Blutproben generiert wurde, die die Immunantwort bei akuten und chronischen Koronarsyndromen erfassen. Der Datensatz besteht aus mehreren Assays mit unterschiedlichen Auflösungen, darunter Zytokindaten auf Probenebene, Plasma-Proteomics und Neutrophilen-Prime-Seq sowie Einzelzell-RNA-Seq-Daten (scRNA-seq). Weitere Komplexität wird durch die Messung mehrerer verschiedener Zeitpunkte pro Patient und mehreren Patientenuntergruppen erhöht.
Der Analyse-Workflow beschreibt, wie die Daten in mehreren Schritten integriert und analysiert werden können: (1) Datenvorverarbeitung und -harmonisierung, (2) Schätzung des MOFA-Modells, (3) Downstream-Analyse. In Schritt 1 wird beschrieben, wie die Features der verschiedenen Datentypen verarbeitet, Features mit geringer Qualität herausgefiltert und normalisiert werden, um ihre Verteilungen für die weitere Analyse zu harmonisieren. Schritt 2 zeigt, wie Sie das MOFA-Modell anwenden und die wichtigsten Varianzquellen innerhalb des Datasets über alle Omics und Features hinweg untersuchen. Schritt 3 stellt verschiedene Strategien für die nachgelagerte Analyse der erfassten Muster vor und verknüpft sie mit den Krankheitszuständen und potenziellen molekularen Prozessen, die diese Bedingungen bestimmen.
Insgesamt stellen wir einen Workflow für die unüberwachte Datenexploration komplexer Multi-Omics-Datensätze vor, um die Identifizierung von Hauptvariationsachsen zu ermöglichen, die sich aus unterschiedlichen molekularen Merkmalen zusammensetzen und auch auf andere Kontexte und Multi-Omics-Datensätze angewendet werden können (einschließlich anderer Assays, wie im beispielhaften Anwendungsfall vorgestellt).
Einleitung
Krankheitsmechanismen sind in der Regel komplex und werden durch das Zusammenspiel mehrerer unterschiedlicher molekularer Prozesse bestimmt. Die komplexen molekularen Mechanismen zu entschlüsseln, die zu bestimmten Krankheiten führen oder die Evolution einer Krankheit steuern, ist eine Aufgabe von hoher medizinischer Relevanz, da sie neue Erkenntnisse für das Verständnis und die Behandlung von Krankheiten liefern kann.
Jüngste technologische Fortschritte ermöglichen es, diese Prozesse mit höherer Auflösung (z.B. auf Einzelzellebene) und gleichzeitig auf verschiedenen biologischen Schichten (z.B. DNA, MRNA, Chromatinzugänglichkeit, DNA-Methylierung, Proteomik) zu messen. Dies führt dazu, dass zunehmend große mehrdimensionale biologische Datensätze generiert werden, die gemeinsam analysiert werden können, um mehr Einblicke in die zugrundeliegenden Prozesse zu gewinnen. Gleichzeitig bleibt die Kombination und Analyse der verschiedenen Datenquellen auf biologisch sinnvolle Weise eine herausfordernde Aufgabe1.
Unterschiedliche technologische Grenzen, Geräusche und Variabilitätsbereiche zwischen verschiedenen Omics stellen eine Herausforderung dar. Zum Beispiel sind die Daten zur Einzelzell-RNA-Sequenzierung (scRNA-seq) sehr spärlich und werden oft von großen technischen oder Batch-Effekten beeinflusst. Darüber hinaus ist der Merkmalsraum oft sehr groß und erstreckt sich über mehrere tausend gemessene Gene oder Proteine, während die Stichprobengrößen begrenzt sind. Dies wird durch komplexe Designs weiter erschwert, die mehrere Krankheitszustände, Störfaktoren, Zeitpunkte und Auflösungen umfassen können. So standen im vorgestellten Anwendungsfall unterschiedliche Datentypen entweder auf Einzelzell- oder Stichprobenebene (Bulk) zur Verfügung. Darüber hinaus können die Daten unvollständig sein und nicht alle Messungen für alle analysierten Probanden verfügbar sein.
Aufgrund dieser Herausforderungen werden verschiedene Omics und enthaltene Merkmale immer noch oft nur separat analysiert2, obwohl eine integrierte Analyse nicht nur ein vollständiges Bild des Prozesses liefern kann, sondern biologische und technische Geräusche von einem Omics auch durch andere Omics kompensiert werden können 3,4. Um eine integrierte Analyse von Multi-Omics-Daten durchzuführen, wurden verschiedene Methoden vorgeschlagen, darunter Bayes'sche Methoden, netzwerkbasierte Methoden 5,6, multimodales Deep Learning7 und Methoden zur Dimensionalitätsreduktion durch Matrixfaktorisierung 8,9. Für Letzteres haben die Ergebnisse einer großen Benchmarking-Studie10 gezeigt, dass die MOFA9-Methode (Multi-omic factor analysis) eines der besser geeigneten Instrumente ist, wenn Daten mit klinischen Annotationen verknüpft werden sollen.
Insbesondere in komplexen Umgebungen sind unüberwachte Matrixfaktorisierungsmethoden ein nützlicher Ansatz, um die Komplexität zu reduzieren und gemeinsame und komplementäre Signale aus verschiedenen Datenquellen und Merkmalen zu extrahieren. Durch die Zerlegung des komplexen Raums in latente Repräsentationen niedrigerer Ränge können die Hauptquellen der Varianz innerhalb der Daten schnell untersucht und mit bekannten Kovariaten verknüpft werden. Falls das gleiche Variationsmuster über mehrere Merkmale (z. B. Gene oder Proteine) hinweg auftritt, kann dies auf einige wenige Faktoren aggregiert werden, während das Rauschen reduziert wird. Die Regularisierung kann verwendet werden, um die Seltenheit der Modellkoeffizienten zu erhöhen, wodurch der Ansatz gut in Umgebungen geeignet ist, in denen der Merkmalsraum groß ist, während die Anzahl der Stichproben begrenzt ist9.
Dieses Protokoll stellt einen flexiblen Analyse-Workflow dar, der das MOFA-Modell verwendet, um zu zeigen, wie ein komplexer Multi-Omics-Datensatz schnell untersucht und die Hauptvariationsmuster herausgefiltert werden können, die diesen Datensatz charakterisieren. Der Workflow besteht aus drei Hauptschritten. Im ersten Schritt, Datenvorverarbeitung und -harmonisierung, werden verschiedene Strategien zur Datenvorverarbeitung auf Basis unterschiedlicher Eingabedatentypen (scRNA-seq, Proteomik, Zytokin, klinische Daten) vorgestellt. Das Protokoll erläutert, wie die Merkmale der verschiedenen Eingabedatensätze verarbeitet, Merkmale von geringer Qualität herausgefiltert und normalisiert werden, um ihre Verteilungen zu harmonisieren. Wir zeigen auch, wie sich diese Vorverarbeitungsentscheidungen auf die nachgelagerten Ergebnisse auswirken können. Im zweiten Schritt wird das MOFA-Modell auf die Daten angewendet, und die resultierende Varianzzerlegung kann verwendet werden, um die Integration der verschiedenen Datensätze zu bewerten. Der dritte Schritt zeigt, wie man die erfassten Faktoren mit Kovariaten verknüpft und die molekularen Programme aufdeckt, die diese Faktoren definieren. Mit dem vorgestellten Workflow konnten wir mehrere latente Faktoren, die mit klinischen Kovariaten verbunden sind, in einem Datensatz von Patienten mit Koronarsyndromen extrahieren und potenziell zugrunde liegende multizelluläre Immunprogramme aus einem früheren Projektidentifizieren 11. Wir werden diesen Datensatz hier verwenden, aber das Protokoll kann leicht auf andere Kontexte angewendet werden, einschließlich anderer Omics.
Der Datensatz besteht aus Proben von Patienten mit stabilen chronischen Koronarsyndromen (CCS), akuten Koronarsyndromen (ACS) und einer Kontrollgruppe mit gesunden Koronarsyndromen (ohne CCS) (Abbildung 1). ACS wird durch eine Plaqueruptur bei bereits bestehendem CCS verursacht, die zu einer akuten Störung des Blutflusses zum Myokard und einer anschließenden ischämischen Verletzung des Herzens führt. Diese Schädigung führt zu einer Entzündungsreaktion des Immunsystems, gefolgt von einer Reparaturphase, die bis mehrere Tage nach dem akuten Ereignis andauert12. Um diese Immunantwort bei ACS-Patienten charakterisieren zu können, wurden Blutproben zu vier verschiedenen Zeitpunkten entnommen: akut (TP1); nach Rekanalisation (14 [± 8] h) (TP2); 60 [± 12] h später (TP3); vor der Entlassung (6,5 [±1,5] Tage) (TP4) (Abbildung 1A). Für CCS und Patienten mit gesunden Herzkranzgefäßen stand nur ein Zeitpunkt zur Verfügung (TP0). Für alle Patienten und Zeitpunkte wurden verschiedene Assays auf Basis der Blutproben gemessen: klinische Marker für Entzündungen (Kreatinkinase (CK), CK-MB, Troponin, C-reaktives Protein (CRP)), scRNA-seq von mononukleären Zellen des peripheren Blutes (PBMCs), Zytokinanalyse, Plasmaproteomik und prime-seq13-Daten von Neutrophilen.
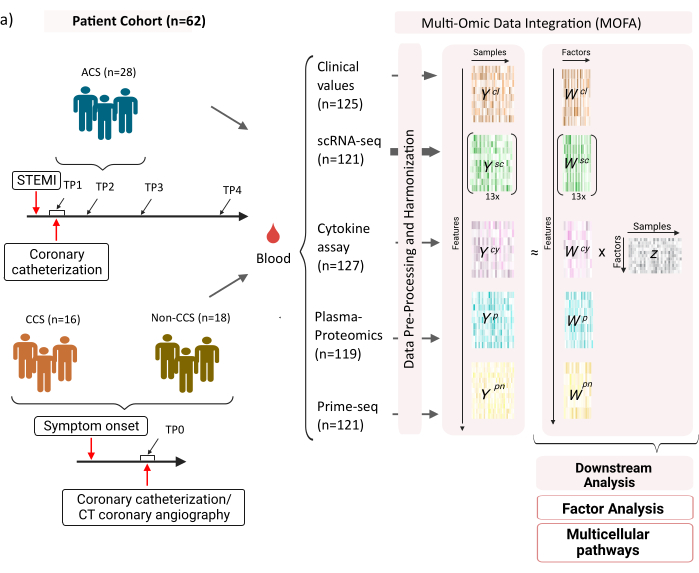
Abbildung 1: Multi-OMIC-Eingabedatensatz für Myokardinfarkt. Eingabedatensatz: Die analysierten Daten umfassen Blutproben von Patienten (n = 62) mit akutem Koronarsyndrom (ACS), chronischem Koronarsyndrom (CCS) und Patienten mit gesunden Koronargefäßen (nicht-CCS). Bei ACS-Patienten wurden Blutproben zu vier verschiedenen Zeitpunkten (TP1-4) eingeschlossen, bei CCS- und Nicht-CCS-Patienten zu einem einzigen Zeitpunkt (TP0). Jede Kombination aus Patient und Zeitpunkt wird in der Analyse als separate Probe behandelt. An den Proben wurden verschiedene omische Assays gemessen: klinische Bluttests (n = 125), scRNA-seq (n = 121), Plasma-Proteomics (n = 119), Zytokin-Assay (n = 127) und neutrophile prime-seq (n = 121). Anschließend wurde das beschriebene Protokoll angewendet, um die Daten über alle Omics hinweg zu integrieren und mit Hilfe des MOFA-Modells und weiterer nachgelagerter Analysen (Faktorenanalyse, Pathway Enrichment) zu untersuchen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Als Input für den hier vorgestellten Workflow nehmen wir Rohzählungen aus scRNA-seq-Daten nach der Verarbeitung mit cellranger und Qualitätskontrolle (QC), wie z.B. im scanpy14 Preprocessing Tutorial beschrieben. Für die Zelltyp-Annotation haben wir die automatisierte Azimuth15-Pipeline verwendet. Die Zählungen werden dann auf Stichprobenebene für jeden Zelltyp aggregiert, indem der Mittelwert für alle Zellen für jede Probe und jeden Zelltyp gemessen wird (Pseudobulk-Aggregation). Die Plasma-Proteomik wird als normalisierte und medianzentrierte Intensitäten eingeschlossen, und für Neutrophile nehmen wir die UMI-Exonzahlen (UMI) aus dem Prime-Seq. Bei den Zytokin- und klinischen Werten wurde keine vorherige Vorverarbeitung durchgeführt. Weitere Einzelheiten zur (experimentellen) Datengenerierung sind im entsprechenden Manuskript11 enthalten. Da die hier vorgestellten Ergebnisse auf der Verwendung der automatisierten Azimut-Annotation für Zelltypen in den scRNA-seq-Daten basieren, verglichen mit der markerbasierten Strategie, die in der referenzierten Publikation verwendet wurde, sind die hier vorgestellten Ergebnisse ähnlich, aber nicht genau die gleichen wie in der Publikation. Im Manuskript konnte gezeigt werden, dass die Zelltyp-Annotationsstrategie die Hauptmuster und biologischen Interpretationen der Analyse nicht verändert, aber kleine Änderungen in den genauen Werten, die sich aus dem Modell ergeben, variieren können. Insgesamt handelte es sich bei den Eingabedaten um einen komplexen, mehrdimensionalen Datensatz mit verschiedenen Zeitpunkten und Messniveaus (Einzelzellen vs. Volumen) von mehr als 10.000 verschiedenen Merkmalen (Gene, Proteine, klinische Werte). Es hat sich gezeigt, dass eine strenge Vorverarbeitungs- und Datenharmonisierungsstrategie, gefolgt von einer MOFA-Analyse, ein nützliches und schnelles Werkzeug ist, um die Daten zu untersuchen und relevante Immunprogramme zu extrahieren. Jeder Zeitpunkt und jede Patientenkombination wird in der MOFA-Analyse als eigenständige Probe behandelt. Jeder Datentyp und jeder Zellentyp wird in der MOFA-Analyse als separate Ansicht betrachtet.
Dieses Protokoll enthält Anweisungen zum Vorbereiten der Eingabedaten für den Workflow, zum Ausführen der verschiedenen Workflow-Schritte, zum Anpassen von Konfigurationen, zum Interpretieren der resultierenden Zahlen und zum iterativen Anpassen der Konfigurationen auf der Grundlage der Interpretationen. Einen Überblick über die verschiedenen Schritte des Protokolls, die erforderlichen Eingabedatensätze bei jedem Schritt und die daraus resultierenden Zahlen und Datensätze bietet die technische Workflow-Übersicht (Abbildung 2).
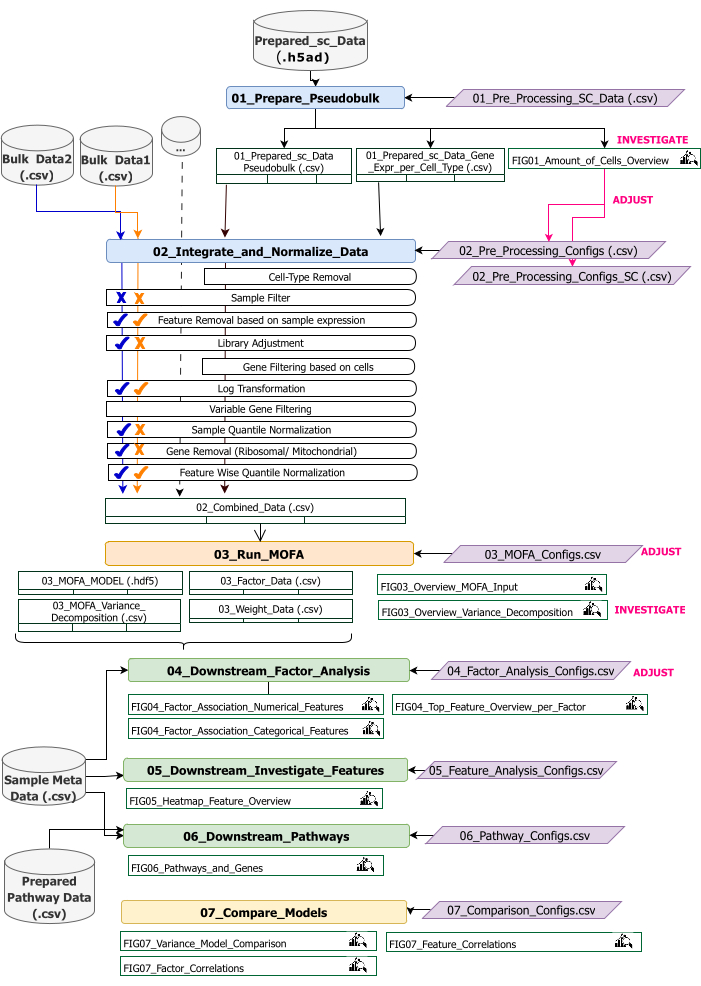
Abbildung 2: Überblick über den technischen Workflow. Skizzierung des Workflows für die Analyse des Multi-Omics-Datensatzes. Die verschiedenen Elemente werden durch unterschiedliche Farben und Symbole hervorgehoben. Jupyter Notebooks, die zum Schritt Datenvorverarbeitung und -harmonisierung (1) gehören, sind blau eingefärbt. Jupyter Notebooks, die zur Stufe 'MOFA Model' (2) gehören, sind orange eingefärbt. Jupyter Notebooks, die zum Schritt "Downstream-Analyse" (3) gehören, sind grün eingefärbt. Ein Jupyter Notebook, das zum Vergleich der Ergebnisse verwendet werden soll, ist gelb eingefärbt. Konfigurationsdateien, in denen Parameter für die Ausführung des Workflows geändert werden können, sind violett hervorgehoben. Eingabedatasets, die zum Ausführen des Workflows erforderlich sind, werden durch das Datensatzsymbol gekennzeichnet und grau hervorgehoben. Alle Abbildungsausgaben, die während der Workflow-Ausführung erzeugt werden, sind durch das Lupensymbol gekennzeichnet. Datensätze, die während der Workflow-Ausführung generiert werden, werden als Tabellen angezeigt. Im Allgemeinen wird der Workflow sequentiell ausgeführt: (1) Die Datenvorverarbeitung und -harmonisierung besteht aus zwei Schritten: der ersten Generierung einer Pseudobulk-Tabelle auf der Grundlage der scRNA-seq-Eingabedaten (01_Prepare_Pseudobulk) und der anschließenden Integration und Normalisierung dieser Daten zusammen mit allen anderen Eingaben auf Probenebene (Bulk) (02_Integrate_and_Normalize_Data). Innerhalb dieses Schritts ist es über die Konfigurationsdateien möglich, für jeden Datensatz separat zu konfigurieren, welcher der angegebenen Vorverarbeitungs- und Normalisierungsschritte (z.B. Sample Filter) angewendet werden soll. (2) "MOFA-Modell": führt das MOFA-Modell auf der generierten Eingabe des ersten Schritts mit den in der Konfigurationsdatei angegebenen Konfigurationen aus (03_MOFA_configs.csv). (3) "Downstream-Analyse": besteht aus drei verschiedenen Notebooks, die unabhängig voneinander ausgeführt werden können, um Einblicke in die generierten MOFA-Ergebnisse zu generieren und sie mit Beispiel-Metadaten (Kovariaten) zu verknüpfen, die als Eingabe über die Datei "Sample Meta Data.csv" bereitgestellt werden. (4) "Modellvergleich": ist ein kleiner separater Schritt, der zum Vergleich verschiedener in Schritt 2 erstellter Modelle verwendet werden kann. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Der Workflow besteht aus mehreren Jupyter Notebooks, die in R und Python geschrieben sind (Kenntnisse der Sprache R und Python sind zum Ausführen des Workflows nicht erforderlich, können aber hilfreich sein, falls Fehler auftreten). In verschiedenen Schritten des Protokolls werden Parameter über Konfigurationsdateien geändert ('.csv'-Dateien, die den Zusatz '_Configs' im Namen enthalten). Innerhalb des Protokolls skizzieren wir nur die Parameter, die ausgehend von der Standardkonfiguration geändert werden müssen.
Es können auch einige andere Parameter geändert werden, um beispielsweise die Vorverarbeitung anzupassen. Eine Dokumentation dieser Parameter und Erläuterungen finden Sie in der Datei 'Documentation_Config_Parameter', die im heruntergeladenen Repository enthalten ist.
Protokoll
1. Vorbereitungen: Technische Einrichtung und Installation
HINWEIS: Um dieses Programm auszuführen, müssen wget, git und Apptainer auf dem Gerät vorinstalliert sein. Eine Anleitung zur Installation von Apptainer auf verschiedenen Systemen (Linux, Windows, Mac) finden Sie hier: https://apptainer.org/docs/admin/main/installation.html. Informationen zur Installation von git finden Sie hier: https://git-scm.com/book/en/v2/Getting-Started-Installing-Git. Abhängig von der Größe der verschiedenen Eingabedatasets wird empfohlen, den Workflow auf einem geeigneten Computer (16 CPUs, 64 GB Arbeitsspeicher) auszuführen. Auf dem lokalen Rechner kann ein Smoke-Test mit den bereitgestellten Beispieldaten durchgeführt werden. Anweisungen und erwartete Ausgaben beim Ausführen des Protokolls für die Beispieldaten sind in der Ergänzungsdatei 1 enthalten. In der ergänzenden Videodatei 1 finden Sie die wichtigen Schritte des Protokolls, die auf dem oben beschriebenen Datensatz ausgeführt werden.
- Öffnen Sie die Konsole, und wählen Sie einen Ordner aus, in dem der gesamte Analysecode und die Ausgaben gespeichert werden, oder erstellen Sie einen Ordner. Navigieren Sie zu dem Ordner, indem Sie den Befehl: cd path_to_folder in das Terminal eingeben.
- Laden Sie das Code-Repository von Github herunter oder klonen Sie es von Github (https://github.com/heiniglab/mofa_workflow) oder geben Sie git clone https://github.com/heiniglab/mofa_workflow.git in das Terminalfenster ein.
- Laden Sie das Image herunter, das alle erforderlichen Installationen von Zenodo enthält, indem Sie wget https://zenodo.org/records/11192947/files/mofa_image.sif in das Terminalfenster eingeben.
- Generieren Sie einen Ordner, in dem alle Ergebnisdaten gespeichert werden, indem Sie mkdir results in das Terminalfenster eingeben.
- Generieren Sie einen Ordner, in dem alle Eingabedaten, die in der Analyse verwendet werden sollen, hinzugefügt werden, indem Sie mkdir input_data in das Terminalfenster eingeben.
- Führen Sie den Container aus, der eine JupyterLab-Sitzung startet, indem Sie den folgenden Befehl in das Terminal eingeben: apptainer run mofa_image.sif. Kopieren Sie die URL, die vom Befehl zurückgegeben wird, in den Browser, wodurch eine Jupyter-lab-Sitzung geöffnet wird (weitere Informationen zu Jupyter-lab finden Sie in der Softwaredokumentation16).
HINWEIS: Wenn der Workflow lokal auf einem Laptop ausgeführt wird, wird empfohlen, stattdessen den Befehl apptainer exec mofa_image.sif jupyter-lab zu verwenden, der direkt eine lokale Hostadresse zurückgibt. Falls der Container in einer Clustered Computing Umgebung ausgeführt wird, kann es notwendig sein, eine Portweiterleitung einzurichten, die über ssh erfolgen kann.
2. Initialisierung und Datenaufbereitung
- Verwenden Sie in der Jupyter-Lab-Sitzung das Navigationsmenü auf der linken Seite. Navigieren Sie mit einem Doppelklick auf input_data in den Ordner input_data.
- Kopieren Sie alle Datensätze, die als Eingabe für die Analyse verwendet werden, per Drag&Drop in das input_data Verzeichnis. Ziehen Sie die Datei aus dem Ordner, in dem sie sich gerade befindet, und legen Sie sie in der Jupyter-Lab-Sitzung im Bereich unterhalb des Ordners input_data ab.
HINWEIS: Alle Datensätze müssen entweder im .csv - oder im . h5ad-Format (im Falle von Einzelzellendaten) vorliegen. Alle .csv Dateien müssen eine passende sample_id Spalte enthalten (identische IDs müssen in allen Datensätzen verwendet werden). Alle anderen Spalten werden als Features verwendet. In der h5ad-Datei muss die Zellenanmerkung zwei Bezeichner enthalten, die den sample_id und den cluster_id angeben. Diese werden für die Aggregation und den Abgleich verwendet. Omic-Datasets in anderen Formaten müssen vor der Verwendung in das angegebene .csv-Format konvertiert werden (Abbildung 3). scRNA-seq-Datensätze, die im . h5seurat-Format vorliegen, können mit dem Jupyter-Notebook in .h5ad konvertiert werden: 00_Data_Conversion.ipynb. - Navigieren Sie in den Konfigurationsordner, indem Sie auf das Ordnersymbol klicken und dann auf die Ordner mofa_workflow, Skripte und Konfigurationen doppelklicken. Öffnen Sie innerhalb des Ordners die Datei Data_configs.csv indem Sie darauf doppelklicken.
- Fügen Sie in der Spalte Wert die Pfade zu den Ordnern der Ordner input_data (data_path) und Ergebnisse (result_path) hinzu. Fügen Sie einen Namen hinzu , der als Dateierweiterung zu allen gespeicherten Dateien in der Wertespalte für die configuration_name hinzugefügt wird (dieses Protokoll wurde MI_v1 [Myokardinfarkt Version 1] verwendet) (Abbildung 4).
- Speichern Sie die Änderungen, indem Sie im Menü oben auf Datei > CSV-Datei speichern klicken.
- Verwenden Sie das Navigationsmenü auf der linken Seite, um durch Klicken auf Skripte zum Skriptordner zu navigieren. Öffnen Sie das Initialisierungsnotebook mit einem Doppelklick auf 00_Configuration_Update.ipynb. Führen Sie das Skript aus, indem Sie oben auf die Schaltfläche Kernel neu starten und alle Zellen ausführen klicken und im Popup-Fenster auf Neustart klicken (Abbildung 5).
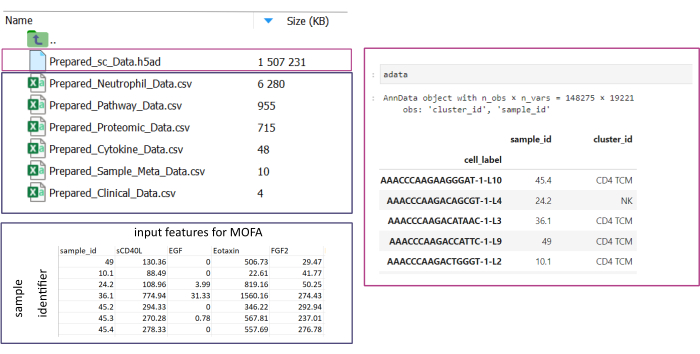
Abbildung 3: Dateneingabe und Einrichtung. Für die Ausführung des Workflows müssen alle Daten in einem bestimmten input_data Ordner gespeichert werden. Für jedes Eingabedataset sollte eine separate Datei bereitgestellt werden. Einzelzelldaten sollten als .h5ad angegeben werden, die eine Zellannotation auf der cluster_id enthalten (z. B. aus früheren Schritten zur Zelltyp-Annotation) und eine sample_id Spalte (die jede einzelne Probe, die analysiert werden soll, eindeutig identifiziert). Alle anderen Eingabe-Datasets sollten im Format ".csv" angegeben werden, einschließlich einer Spalte, in der die sample_id (die mit der entsprechenden Spalte der Einzelzellendaten übereinstimmt) und die in der MOFA-Analyse zu verwendenden Features in allen anderen Spalten angegeben sind. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
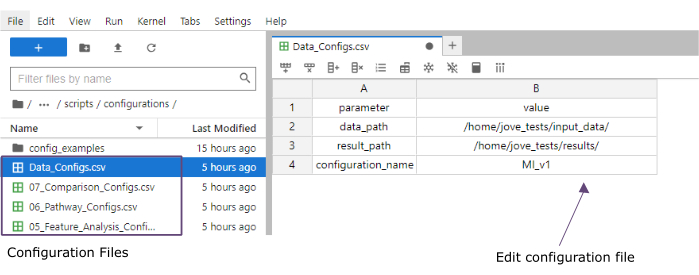
Abbildung 4: Konfigurationsdateien für Jupyter-lab. Während der Ausführung des Workflows werden Änderungen an Parametern (z.B. Anpassung der Filteroptionen etc.) über '.csv'-Konfigurationsdateien festgelegt. Im geklonten Repository sind Standardkonfigurationsdateien für jeden Schritt enthalten. Sie können direkt in der jupyter-lab-Konsole bearbeitet werden, ähnlich wie in einer Tabelle. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
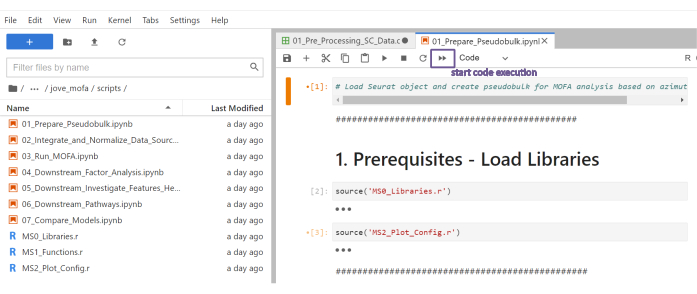
Abbildung 5: Jupyter-notebooks-Skripte. Der vollständige Workflow besteht aus einer Reihe von Jupyter-Notebooks, die nacheinander ausgeführt werden, nachdem die entsprechenden Konfigurationsdateien geändert wurden. Mit einem Doppelklick auf das Jupyter-Notebook auf der linken Seite wird die entsprechende Datei auf der rechten Seite geöffnet. Die vollständige Ausführung der Datei kann mit dem oben hervorgehobenen Button gestartet werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. Datenvorverarbeitung und -harmonisierung
- Vorverarbeitung - Konvertieren Sie sc-Daten in Pseudobulk.
HINWEIS: Dieser Schritt muss nur ausgeführt werden, wenn einzellige Daten in der Analyse verwendet werden.- Über das Navigationsmenü auf der linken Seite navigieren Sie mit einem Doppelklick auf Konfiguration zum Konfigurationsordner. Öffnen Sie die Datei 01_Preprocessing_SC_Data.csv mit einem Doppelklick. Überprüfen Sie die automatisch ausgefüllten Werte in der Datei und passen Sie gegebenenfalls die Werte in der Spalte data_name an die Dateinamen der einzelligen Datensätze im Ordner input_data an, der für die Analyse verwendet wird.
HINWEIS: Standardmäßig werden alle Namen von .h5ad-Dateien im Eingabedatenordner der Konfigurationsdatei im Initialisierungsskript hinzugefügt. Sollten einige der Datensätze nicht für die Analyse verwendet werden sollen, können diese hier entfernt werden. - Speichern Sie die vorgenommenen Änderungen, indem Sie im Menü oben auf Datei > CSV-Datei speichern klicken.
- Verwenden Sie das Navigationsmenü auf der linken Seite, um durch Klicken auf Skripte zum Skriptordner zu navigieren. Öffnen Sie das Notebook 01_Prepare_Pseudobulk.ipynb, indem Sie darauf doppelklicken. Führen Sie das Skript aus, indem Sie oben auf die Schaltfläche Kernel neu starten und alle Zellen ausführen klicken und im Popup-Fenster auf Neu starten klicken.
- Über das Navigationsmenü auf der linken Seite navigieren Sie zum Abbildungsordner, indem Sie zuerst auf Abbildungen und dann auf 01_figures doppelklicken. Öffnen Sie den neu generierten Plot FIG01_Amount_of_Cells_overview mit einem Doppelklick darauf.
HINWEIS: Die Ausführung des Notebooks kann einige Minuten dauern. Wenn das Notebook erfolgreich ausgeführt wurde, erscheint ein Popup und die FIG01_Amount_of_Cells_Overview Datei wird entweder vom Notebook aktualisiert oder neu generiert. In der Spalte "Letzte Änderung" kann angegeben werden, wann die Datei generiert wurde, um zu bewerten, ob es sich um eine neue oder eine alte Datei handelt. - Untersuchen Sie das Diagramm, um Zelltyp-Cluster mit einer sehr geringen Anzahl von Zellen pro Probe zu identifizieren. Notieren Sie sich die Namen dieser cluster_ids um sie in den nachfolgenden Schritten auszuschließen (Abbildung 6).
- Über das Navigationsmenü auf der linken Seite navigieren Sie mit einem Klick auf ... und doppelklicken Sie dann auf Konfigurationen. Öffnen Sie die Datei 02_Preprocessing_Configs_SC.csv mit einem Doppelklick darauf.
- Prüfen Sie die Werte in den Spalten configuration_name und data_name und passen Sie diese ggf. an.
HINWEIS: Innerhalb des Initialisierungsskripts werden diese Werte mit allen Namen der .h5ad-Dateien im Eingabedatenordner und dem zuvor in der Data_Configs.csv Datei festgelegten configuration_name Wert vorausgefüllt. Für den Fall, dass Dateien von der Analyse ausgeschlossen oder eine andere Erweiterung für Dateinamen verwendet werden soll, kann dies hier angepasst werden. - Passen Sie den Wert in der Spalte cell_type_exclusion an, und fügen Sie alle cluster_id hinzu, die im vorherigen Schritt als ausgeschlossen identifiziert wurden, getrennt durch ','.
- Speichern Sie die Änderungen, indem Sie oben in der Navigationsleiste auf Datei > CSV-Datei speichern klicken.
- Über das Navigationsmenü auf der linken Seite navigieren Sie mit einem Doppelklick auf Konfiguration zum Konfigurationsordner. Öffnen Sie die Datei 01_Preprocessing_SC_Data.csv mit einem Doppelklick. Überprüfen Sie die automatisch ausgefüllten Werte in der Datei und passen Sie gegebenenfalls die Werte in der Spalte data_name an die Dateinamen der einzelligen Datensätze im Ordner input_data an, der für die Analyse verwendet wird.
- Vorverarbeitung - Harmonisieren und integrieren Sie andere Omics-Datenquellen.
- Öffnen Sie die Datei 02_Preprocessing_Configs.csv , indem Sie darauf doppelklicken, und passen Sie die Vorverarbeitungskonfiguration für jeden Datensatz an, der einbezogen und im Ordner data_input gespeichert wird (eine Zeile pro Datensatz).
- Überprüfen Sie die Werte in den Spalten configuration_name und data_name und passen Sie diese ggf. an.
- Passen Sie die anderen Parameter in den Spalten entsprechend an, je nachdem, welche Vorverarbeitungsschritte angewendet werden sollen.
HINWEIS: Standardwerte werden für jedes Dataset hinzugefügt, das sich im Ordner input_dataset befindet, aber nicht spezifisch für die einzelnen Datentypen der Daten. Daher werden Anpassungen notwendig sein. Eine detaillierte Dokumentation der Parameter finden Sie in der Documentation_Config_Parameter.doc Datei. - Speichern Sie die Änderungen, indem Sie auf Datei > CSV-Datei speichern klicken.
- Verwenden Sie das Navigationsmenü auf der linken Seite, um durch Klicken auf Skripte zum Skriptordner zu navigieren. Öffnen Sie das Notebook 02_Integrate_and_Normalized_Data_Sources.ipynb mit einem Doppelklick. Führen Sie das Skript aus, indem Sie oben auf die Schaltfläche Kernel neu starten und alle Zellen ausführen klicken und im Popup-Fenster auf Neu starten klicken.
- Navigieren Sie über das Navigationsmenü auf der linken Seite zum generierten 02_results Ordner, indem Sie auf das Ordnersymbol klicken und anschließend auf Ergebnisse und 02_results doppelklicken. Prüfen Sie, ob die Datei 02_Combined_data_'configuration_name'_Integrated.csv die die kombinierte vorverarbeitete Dateneingabedatei enthält.
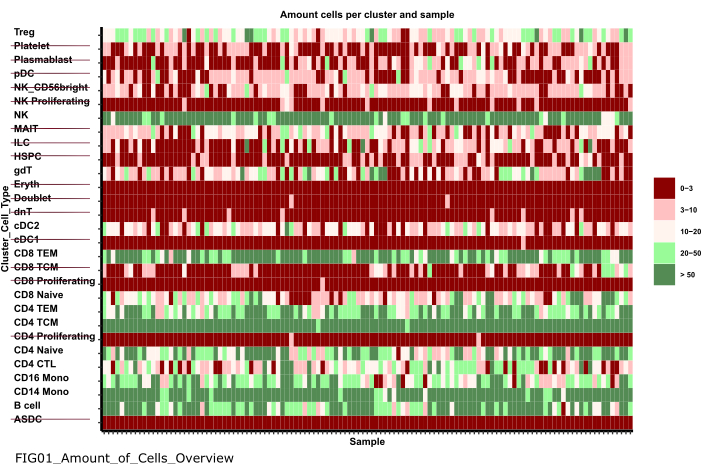
Abbildung 6: Datenvorverarbeitung und -harmonisierung. Eine Ausgabe des Schritts '01_Prepare_Pseudobulk' ist der Plot 'Fig01_Amount_of_Cells_Overview'. Hier wird für jede cluster_id (y-Achse, die den Zelltyp aus den vorherigen Annotationsschritten für den Zelltyp angibt) die Anzahl der Zellen pro Probe ('sample_id") angegeben. In den vorgestellten Ergebnissen werden Zelltypen mit einer geringen Anzahl von Zellen pro Probe von der anschließenden Analyse ausgeschlossen (gekennzeichnet durch das Durchstreichen). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. Ausführen von MOFA
- Navigieren Sie in Jupyter-Lab über das Navigationsmenü auf der linken Seite zum Konfigurationsordner, indem Sie auf das Ordnersymbol klicken und dann auf mofa_workflow doppelklicken, gefolgt von einem Doppelklick auf Skripte und Konfigurationen. Öffnen Sie die Datei 03_MOFA_Configs.csv mit einem Doppelklick darauf.
- Prüfen Sie die Einträge für die Spalten configuration_name und mofa_result_name und passen Sie die Einträge an, wenn alternative Namen verwendet werden sollen.
HINWEIS: Die mofa_result_name wird als Dateierweiterung an alle Ergebnisdateien angehängt, die auf der Grundlage des MOFA generiert wurden. Dieser kann vom configuration_name Wert abweichen, da verschiedene MOFA-Setups mit den gleichen Eingabedaten ausgeführt werden können (dieses Protokoll verwendet MI_v1_MOFA). - Geben Sie die Anzahl der Faktoren ein, die im MOFA-Modell geschätzt werden sollen (amount_of_factors Spalte), und legen Sie fest, ob Gewichtung und Skalierung angewendet werden sollen (weighting_of_views und scale_views Spalten), indem Sie die Werte in der Datei anpassen.
- Speichern Sie die Änderungen, indem Sie auf Datei > CSV-Datei speichern klicken.
- Verwenden Sie das Navigationsmenü auf der linken Seite, um zum Skriptordner zu navigieren, indem Sie auf "Skripte" klicken. Öffnen Sie das Notebook 03_Run_MOFA.ipynb , indem Sie auf die Datei doppelklicken. Führen Sie das Skript aus, indem Sie oben auf die Schaltfläche Kernel neu starten und alle Zellen ausführen klicken und im Popup-Fenster auf Neustart klicken.
- Navigieren Sie in den Ordner 03_figures , indem Sie auf die Abbildungen doppelklicken und dann 03_figures. Öffnen Sie das generierte Diagramm FIG03_Overview_Variance_Decomposition_ mofa_result_name , und untersuchen Sie das Modellergebnis (Abbildung 7A).
- Navigieren Sie über das Navigationsmenü auf der linken Seite zu dem generierten 03_results Ordner, indem Sie auf das Ordnersymbol klicken und dann auf Ergebnisse und 03_results doppelklicken. Überprüfen Sie, ob sie die Beispielfaktor-Wertdatei 03_Factor_Data_'mofa_result_name'.csv und die Feature-Faktor-Gewichtungsdatei 03_Weight_Data_'mofa_result_name'.csv enthält.
5. Nachgelagerte Analyse
- Interpretation des Faktors.
- Über das Navigationsmenü auf der linken Seite navigieren Sie mit einem Klick auf das Ordnersymbol und einem anschließenden Doppelklick auf input_data zum input_data Ordner.
- Bereiten Sie eine .csv Datei (Prepared_Sample_Meta_Data.csv) vor, die alle Metadaten (Kovariaten) der Stichproben enthält, die in Verbindung mit den generierten Faktoren analysiert werden. Kopieren Sie die Datei in den input_data Ordner, indem Sie die Datei per Drag&Drop in der Ordnerübersicht des input_data Ordners ablegen.
HINWEIS: Es muss die sample_id Spalte enthalten , um es mit den zuvor verwendeten Daten abzugleichen, sowie weitere Spalten für jedes Feature, das analysiert werden soll. - Navigieren Sie in Jupyter-Lab über das Navigationsmenü auf der linken Seite zurück in den Konfigurationsordner, indem Sie auf das Ordnersymbol klicken und dann auf mofa_workflow doppelklicken, gefolgt von Skripten und Konfiguration. Öffnen Sie die Datei 04_Factor_Analysis.csv mit einem Doppelklick darauf.
- Überprüfen Sie, ob die Einträge für die configuration_name und mofa_result_name die Namen der Konfigurations- und MOFA-Ergebnisse enthalten, die im Skript analysiert werden, und passen Sie diese gegebenenfalls an.
- Fügen Sie in der numeric_covariates Spalte die Namen aller numerischen Spalten in der Prepared_Sample_Meta_Data.csv Datei hinzu, die in Bezug auf die MOFA-Faktoren durch Komma getrennt untersucht werden (Dieses Protokoll verwendet CRP,CK).
- Fügen Sie in der Spalte categorical_covariates' die Namen aller kategorischen Spalten in der Prepared_Sample_Meta_Data.csv-Datei hinzu, die in Bezug auf die MOFA-Faktoren durch Komma getrennt untersucht werden (Dieses Protokoll verwendet eine Messung).
- Speichern Sie die Änderungen, indem Sie auf Datei > CSV-Datei speichern klicken.
- Verwenden Sie das Navigationsmenü auf der linken Seite, um zum Ordner "Skripte" zu navigieren, indem Sie auf Skripte klicken. Öffnen Sie das Notebook 04_Downstream_Factor_Analysis.ipynb , indem Sie darauf doppelklicken. Führen Sie das Skript aus, indem Sie oben auf die Schaltfläche Kernel neu starten und alle Zellen ausführen klicken und im Popup-Fenster auf Neustart klicken.
- Navigieren Sie über das Navigationsmenü auf der linken Seite mit einem Doppelklick auf die Abbildungen und dann 04_figures zum Ordner 04_figures. Öffnen Sie die generierten Plots mit einem Doppelklick darauf und untersuchen Sie die Faktoren auf interessante Muster und Assoziationen:FIG04_Factor_Association_with_numeric_features_
"mofa_result_name.pdf (Abbildung 7B). FIG04_Factor_Association_
with_categorical_features_'mofa_result_name.pdf (Abbildung 7C). FIG04_Top_Feature_Overview_per_Factor _'mofa_result_name.pdf (Abbildung 8A).
- Merkmalsanalyse
- Über das Navigationsmenü auf der linken Seite navigieren Sie mit einem Klick auf ... und doppelklicken Sie dann auf Konfigurationen. Öffnen Sie die Datei 05_Feature_Analysis_Configs.csv mit einem Doppelklick darauf.
- Prüfen Sie, ob die Einträge für die Spalten configuration_name und mofa_result_name mit den Namen der Konfiguration und des generierten MOFA-Ergebnisses übereinstimmen, die für die nachgelagerte Analyse verwendet werden, und passen Sie diese gegebenenfalls an.
- Fügen Sie in der Spalte Faktor den Faktor hinzu, für den die wichtigsten Features im nächsten Skript dargestellt werden.
- Fügen Sie in der Spalte faceting_variable den Spaltennamen einer kategorischen Spalte im Prepared_Sample_Meta_Data hinzu.csv die zur Gruppierung der Stichproben im Diagramm verwendet wird (Dieses Protokoll verwendet eine Messung)
- Speichern Sie die Änderungen, indem Sie auf Datei > CSV-Datei speichern klicken.
- Über das Navigationsmenü auf der linken Seite navigieren Sie mit einem Klick auf Skripte zum Skriptordner. Öffnen Sie das Notebook 05_Downstream_Investigate_Features_Heatmap.ipynb mit einem Doppelklick. Führen Sie das Skript aus, indem Sie oben auf die Schaltfläche Kernel neu starten und alle Zellen ausführen klicken und im Popup-Fenster auf Neu starten klicken.
- Navigieren Sie über das Navigationsmenü auf der linken Seite zum Ordner 05_figures , indem Sie zuerst auf die Abbildungen und dann auf 05_figures doppelklicken. Öffnen und untersuchen Sie den generierten Plot FIG05_Heatmap_Feature_Overview__ 'mofa_result_name'.pdf durch Doppelklick auf die Datei (Abbildung 8B).
HINWEIS: Abhängig von der Anzahl der Merkmale, die im Diagramm angezeigt werden, kann es erforderlich sein, die Parameter plot_width und plot_height innerhalb des 05_Feature_Analysis_Configs.csv anzupassen und das Skript erneut auszuführen, um sicherzustellen, dass alles in den Diagramm passt.
- Analyse des Signalwegs
- Über das Navigationsmenü auf der linken Seite navigieren Sie mit einem Klick auf das Ordnersymbol und einem anschließenden Doppelklick auf input_data zum input_data Ordner.
- Bereiten Sie eine .csv Datei (Prepared_Pathway_Data.csv) vor, die eine Liste von Pfaden enthält, die auf Anreicherung getestet werden. Kopieren Sie die Datei in den Ordner input_data , indem Sie die Datei per Drag&Drop in der Ordnerübersicht des input_data Ordners ablegen.
HINWEIS: Es muss drei Spalten enthalten: ID (eine eindeutige Kennung des Signalwegs), Gen (die Gene, die durch ihren Gennamen (SYMBOL) angegeben werden, der zum Signalweg gehört, eine Zeile pro Gen), pathway_name (ein Name/eine textuelle Beschreibung der Signalwege). - Navigieren Sie in der Jupyter-Lab-Sitzung über das Navigationsmenü auf der linken Seite zum Konfigurationsordner, indem Sie auf ... und doppelklicken Sie dann auf Konfigurationen. Öffnen Sie die Datei 06_Pathway_Configs.csv mit einem Doppelklick darauf.
- Überprüfen Sie den Eintrag für die Spalte mofa_result_name und stellen Sie sicher, dass er dem Namen des generierten MOFA-Ergebnisses entspricht, das für die Berechnung der Pfadanreicherung verwendet wird.
- Überprüfen Sie den Eintrag in der Spalte "Typen", und entfernen Sie die Einträge in der Spalte "Typen ", die keine Merkmale enthalten, die mit der Spalte "Gen " in der Prepared_Pathway_Data.csv Datei übereinstimmen.
HINWEIS: Standardmäßig werden alle verschiedenen Ansichten, die innerhalb des MOFA-Modells verwendet wurden, während der Ausführung des Workflows zu dieser Datei hinzugefügt. Falls Ansichten vorhanden sind, die keine Features enthalten, die mit mindestens einem Pfad übereinstimmen, müssen sie entfernt werden. Andernfalls schlägt die Ausführung fehl. Ein Beispiel wäre, dass die Pfaddatei nur Pfadanmerkungen für Gene enthält, aber es gibt eine Ansicht mit Proteinnamen. - Speichern Sie die Änderungen, indem Sie auf Datei > CSV-Datei speichern klicken.
- Verwenden Sie das Navigationsmenü, um zum Skriptordner zu navigieren , indem Sie auf Skripte klicken. Öffnen Sie das Notebook 06_Downstream_Pathways.ipynb , indem Sie darauf doppelklicken. Führen Sie das Skript aus, indem Sie oben auf die Schaltfläche Kernel neu starten und alle Zellen ausführen klicken und im Popup-Fenster auf Neu starten klicken.
- Über das Navigationsmenü auf der linken Seite navigieren Sie mit einem Doppelklick auf die Abbildungen und dann mit 06_figures in den Ordner 06_figures. Öffnen Sie den generierten Plot FIG06_Pathways_and_Genes_ 'mofa_result_name indem Sie darauf doppelklicken und untersuchen Sie die visualisierten Pfade (Abbildung 8C).
HINWEIS: Wie die visualisierten Pfade ausgewählt werden, kann über die Konfigurationsdatei konfiguriert werden. Weitere Informationen finden Sie in der Dokumentation der Parameter. - Über das Navigationsmenü auf der linken Seite navigieren Sie mit einem Klick auf das Ordnersymbol und einem Doppelklick auf Ergebnisse und 06_results zum generierten 06_results Ordner. Überprüfen Sie, ob die Datei mit den angereicherten Pfaden 06_Pathway_enrichment__'mofa_result_name" enthalten ist.
6. Vergleich verschiedener Konfigurationen und Versionen (Ergänzende Abbildung 1, Ergänzende Abbildung 2, Ergänzende Abbildung 3, Ergänzende Abbildung 4)
- Um die Auswirkungen der Verwendung verschiedener Parameter/Konfigurationen im gesamten Workflow zu vergleichen, führen Sie die Abschnitte 3 bis 5 erneut aus, ändern Sie die Parameter in den Konfigurationsdateien und verwenden Sie unterschiedliche configuration_name - und mofa_result_name-IDs .
HINWEIS: Neue Ergebnisse werden unter diesen Namen gespeichert, um sie für den Vergleich verschiedener Läufe zu verwenden. - Navigieren Sie in Jupyter-Lab über das Navigationsmenü auf der linken Seite zum Konfigurationsordner . Öffnen Sie die Datei 07_Comparison_Configs.csv , indem Sie darauf doppelklicken.
- Fügen Sie in der Spalte mofa_result_name die Namen aller vorherigen MOFA-Läufe hinzu, die verglichen werden sollen (eine Zeile pro Name/Konfiguration, z. B. MI_v1_MOFA MI_v2_MOFA).
- Fügen Sie in der Spalte compare_factors die Faktoren hinzu, die zwischen den Modellen verglichen werden. Standardmäßig ist es Factor1,Factor2,Factor3. (Ergänzende Abbildung 2A).
HINWEIS: In diesem Skript werden die Merkmals- und Faktorwerte der verschiedenen Modelle verglichen, indem sie miteinander korreliert werden. Dies funktioniert nur für Modelle, die auf denselben Stichproben (gekennzeichnet durch sample_id) und demselben Satz von Features basieren. Falls Muster oder Merkmale zwischen den verglichenen Versionen nicht übereinstimmen, werden sie vom Vergleich ausgeschlossen. - Speichern Sie die Änderungen, indem Sie auf Datei > CSV-Datei speichern klicken.
- Über das Menü auf der linken Seite navigieren Sie durch Klicken auf Skripte zum Skriptordner. Öffnen Sie das Notebook 07_Compare_Models.ipynb, indem Sie darauf doppelklicken. Führen Sie das Skript aus, indem Sie auf die Schaltfläche Kernel neu starten und alle Zellen ausführen klicken und im Popup-Fenster auf Neustart klicken.
- Über das Menü auf der linken Seite navigieren Sie zum Ordner 06_figures , indem Sie zuerst auf die Abbildungen und dann auf 06_figures doppelklicken. Öffnen Sie die generierten Diagramme mit einem Doppelklick auf die Dateien, um die Ähnlichkeit der verschiedenen Versionen zu analysieren:
FIG07_Variance_Model_Comparison.pdf (Ergänzende Abbildung 2B)
FIG07_Factor_Correlations.pdf
(Ergänzende Abbildung 2C)
FIG07_Feature_Correlations.pdf
(Ergänzende Abbildung 3C)
7. Erweitern des Workflows: Hinzufügen weiterer Parameter und Konfigurationen
HINWEIS: Neben den Parametern, die derzeit in den Konfigurationsdateien konfigurierbar sind, können andere Anpassungen im Code oder andere Parameter enthalten sein. So bietet das MOFA-Modell selbst etliche weitere Trainingsparameter17 an, die entweder direkt im Code modifiziert oder über die Konfigurationsdateien anpassbar gemacht werden können. Im nächsten Abschnitt des Protokolls wird ein Beispiel dafür skizziert, wie dies für zusätzliche MOFA-Modelltrainingsparameter zu tun ist. Für diesen Teil sind R-Programmierkenntnisse erforderlich.
- Verwenden Sie in Jupyter-Lab das Navigationsmenü auf der linken Seite, um zum Skriptordner zu navigieren. Öffnen Sie das Notebook 03_Run_MOFA.ipynb mit einem Doppelklick.
- Klicken Sie auf der linken Seite auf die Registerkarte Inhaltsverzeichnis und navigieren Sie dann zum Unterabschnitt 4.3 Festlegen von MOFA-Trainingsoptionen und führen Sie das Modelltraining aus , indem Sie darauf klicken. Scrollen Sie nach unten, um die gedruckte Ausgabe des MOFA-Modells mit konfigurierbaren Parametern im Notebook zu sehen.
- In der R for-Schleife im Code unterhalb der Überschrift werden alle MOFA-Daten, das Modell und alle Trainingsoptionen festgelegt. Fügen Sie unterhalb der Zeile model_opts$num_factors = mofa_configs$amount_of_factors[i]) eine weitere Zeile mit dem folgenden Code hinzu
model_opts$likelihoods['data_type'] = 'Poisson'.
HINWEIS: Dadurch wird die Verteilung geändert, die das Modell als Eingabe für die Ansicht verwendet, die durch den Namen data_type für alle MOFA-Durchläufe angegeben wird. Wenn Sie poisson für einen Datentyp angeben, wird das Modell nur ausgeführt, wenn Features für diesen Datentyp ganze Zahlen sind (z. B. Lesezählungen aus RNA-seq). Weitere Informationen zu den MOFA-Daten, dem Training und den Modelloptionen finden Sie in den MOFA-Tutorials und in der Dokumentation17. - Speichern Sie die Änderungen im Notizbuch, indem Sie oben auf die Schaltfläche Speichern klicken.
- Um neue Parameter über die .csv Konfigurationsdateien zu übergeben, navigieren Sie über die Navigation auf der linken Seite mit einem Doppelklick auf Konfigurationen in den Konfigurationsordner und öffnen Sie die Datei 03_MOFA_Configs.csv mit einem Doppelklick.
- Fügen Sie eine neue Spalte hinzu, die den Parameternamen angibt, z. B. number_iterations , und geben Sie einen Wert ein, z. B. 1000. Speichern Sie die Änderungen, indem Sie auf Datei > CSV-Datei speichern klicken.
- Verwenden Sie das Navigationsmenü, um durch den Skriptordner zu navigieren, indem Sie auf Skripte klicken. Öffnen Sie das Notebook 03_Run_Mofa.ipynb , indem Sie darauf doppelklicken. Klicken Sie auf der linken Seite auf die Registerkarte Inhaltsverzeichnis und navigieren Sie dann zum Unterabschnitt 4.3 Festlegen von MOFA-Trainingsoptionen und führen Sie das Modelltraining aus , indem Sie darauf klicken.
- Ersetzen Sie die Zeile train_opts$maxiter = 50000 durch train_opts$maxiter = mofa_configs$column_name[i] (wenn der Name der hinzugefügten Spalte number_of_iterations ist, lautet er train_opts$maxiter = mofa_configs$number_of_iterations[i])).
HINWEIS: Die Konfigurationsdatei, die in diesem Notebook 03_MOFA_Configs.csv, wird am Anfang des Notebooks (Unterabschnitt: Voraussetzungen Konfigurationen & Parameter) mofa_config als data.frame in der Sitzung gelesen und daher wird in dieser Codezeile auf dieses Objekt und die entsprechende neu generierte Spalte verwiesen. Da mehrere Konfigurationen gleichzeitig ausgeführt werden können, identifiziert i die Zeile des data.frame, während die Modellschätzung in einer for-Schleife über alle verschiedenen Zeilen in der .csv Datei ausgeführt wird. Das Prinzip des Einlesens der Konfigurationsdatei am Anfang des Notebooks im Abschnitt 'Voraussetzungen, Konfigurationen & Parameter' ist für alle Notebooks gleich, und weitere Modifikationen können so vorgenommen werden. - Speichern Sie die Änderungen im Notizbuch, indem Sie auf die Schaltfläche Speichern klicken.
Ergebnisse
Nach der erfolgreichen Ausführung des Workflows werden mehrere Tabellen und Abbildungen generiert, wie in Abbildung 2 dargestellt. Die Abbildungen werden im Ordner /figures abgelegt (Abbildung 6, Abbildung 7, Abbildung 8, Ergänzende Abbildung 1, Ergänzende Abbildung 2, Ergänzende Abbildung 3,
Diskussion
Mit dem skizzierten Protokoll wird ein modularer und erweiterbarer Jupyter-Notebook-basierter Workflow vorgestellt, mit dem ein komplexer Multi-Omics-Datensatz schnell untersucht werden kann. Die Hauptteile des Workflows bestehen aus dem Vorverarbeitungs- und Datenharmonisierungsteil (mit verschiedenen Standardschritten zur Filterung und Normalisierung der Daten), der Schätzung des MOFA9-Modells und einigen beispielhaften nachgelagerten Analysen. Einer der wichti...
Offenlegungen
Die Autoren erklären, dass kein Interessenkonflikt besteht.
Danksagungen
C.L. wird von der Helmholtz-Gemeinschaft im Rahmen der Joint Research School "Munich School for Data Science - MUDS" gefördert.
Materialien
| Name | Company | Catalog Number | Comments |
| Apptainer | NA | NA | https://apptainer.org/docs/admin/main/installation.html |
| Compute server or workstation or cloud (Linux, Mac or Windows environment). Depending on the size of the different input datasets we recommend running the workflow on a suitable machine (in our setting we use: 16 CPU, 64GB Memory) | Any manufacturer | 16 CPU, 64GB Memory | Large Memory is only required for the processing of the raw single cell data. After preprocessing the later analysis steps can also be performed on regular desktop or laptop computers |
| git | NA | NA | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git |
| GitHub | GitHub | NA | https://github.com/heiniglab/mofa_workflow |
Referenzen
- Lähnemann, D., et al. Eleven grand challenges in single-cell data science. Genome Biol. 21 (1), 31 (2020).
- Colomé-Tatché, M., Theis, F. J. Statistical single cell multi-omics integration. Curr Opin Syst Biol. 7, 54-59 (2018).
- Hawe, J., Theis, F., Heinig, M. Inferring interaction networks from multi-omics data. Front Genet. 10, 535 (2019).
- Hawe, J. S., et al. Network reconstruction for trans acting genetic loci using multi-omics data and prior information. Genome Med. 14 (1), 125 (2022).
- Koh, H. W. L., Fermin, D., Vogel, C., Choi, K. P., Ewing, R. M., Choi, H. iOmicsPASS: network-based integration of multiomics data for predictive subnetwork discovery. NPJ Syst Biol Appl. 5, 22 (2019).
- Ogris, C., Hu, Y., Arloth, J., Müller, N. S. Versatile knowledge guided network inference method for prioritizing key regulatory factors in multi-omics data. Sci Rep. 11, 6806 (2021).
- Lee, C., vander Schaar, M. A variational information bottleneck approach to multi-omics data integration. Proceedings of The 24th International Conference on Artificial Intelligence and Statistics. 130, 1513-1521 (2021).
- Singh, A., et al. DIABLO: an integrative approach for identifying key molecular drivers from multi-omics assays. Bioinformatics. 35 (17), 3055-3062 (2019).
- Argelaguet, R., et al. Multi-omics factor analysis-a framework for unsupervised integration of multi-omics data sets. Mol Syst Biol. 14 (6), e8124 (2018).
- Cantini, L., et al. Benchmarking joint multi-omics dimensionality reduction approaches for the study of cancer. Nature Commun. 12 (1), 124 (2021).
- Pekayvaz, K., et al. Multiomic analyses uncover immunological signatures in acute and chronic coronary syndromes. Nature Medicine. 30 (6), 1696-1710 (2024).
- Swirski, F. K., Nahrendorf, M. Cardioimmunology: the immune system in cardiac homeostasis and disease. Nat Rev Immunol. 18 (12), 733-744 (2018).
- Janjic, A., et al. Prime-seq, efficient and powerful bulk RNA sequencing. Genome Biol. 23 (1), 88 (2022).
- Wolf, F. A., Angerer, P., Theis, F. J. SCANPY: large-scale single-cell gene expression data analysis. Genome Biol. 19 (1), 15 (2018).
- Cao, Y., et al. Integrated analysis of multimodal single-cell data with structural similarity. Nucleic Acids Res. 50 (21), e121 (2022).
- . Get Started - JupyterLab 4.1.0a4 documentation Available from: https://jupyterlab.readthedocs.io/en/latest/getting_started/overview.html (2024)
- . MOFA2: training a model in R Available from: https://raw.githack.com/bioFAM/MOFA2_tutorials/master/R_tutorials/getting_started_R.html (2020)
- De Silva, D., et al. Robust T cell activation requires an eIF3-driven burst in T cell receptor translation. eLife. 10, e74272 (2021).
- Li, G., Liang, X., Lotze, M. HMGB1: The central cytokine for all lymphoid cells. Front Immunol. 4, 68 (2013).
- Jassal, B., et al. The reactome pathway knowledgebase. Nucleic Acids Res. 48 (D1), D498-D503 (2020).
- Argelaguet, R., et al. MOFA+: a statistical framework for comprehensive integration of multimodal single-cell data. Genome Biol. 21 (1), 111 (2020).
- Velten, B., et al. Identifying temporal and spatial patterns of variation from multimodal data using MEFISTO. Nat Methods. 19 (2), 179-186 (2022).
- Qoku, A., Buettner, F. Encoding domain knowledge in multi-view latent variable models: A Bayesian approach with structured sparsity. Proceedings of The 26th International Conference on Artificial Intelligence and Statistics. 206, 11545-11562 (2022).
- Multi-Omics Factor Analysis. MOFA Available from: https://biofam.github.io/MOFA2/ (2024)
- Mitchel, J., et al. Tensor decomposition reveals coordinated multicellular patterns of transcriptional variation that distinguish and stratify disease individuals. bioRxiv. , (2023).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten