É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Aplicação de análise fatorial multiômica não supervisionada para descobrir padrões de variação e processos moleculares ligados a doenças cardiovasculares
Neste Artigo
Resumo
Apresentamos um fluxo de trabalho flexível e extensível baseado em laboratório Jupyter para a análise não supervisionada de conjuntos de dados multiômicos complexos que combina diferentes etapas de pré-processamento, estimativa do modelo de análise fatorial multiômica e várias análises downstream.
Resumo
Os mecanismos da doença são geralmente complexos e governados pela interação de vários processos moleculares distintos. Conjuntos de dados complexos e multidimensionais são um recurso valioso para gerar mais insights sobre esses processos, mas a análise de tais conjuntos de dados pode ser desafiadora devido à alta dimensionalidade resultante, por exemplo, de diferentes condições de doença, pontos de tempo e ômicas capturando o processo em diferentes resoluções.
Aqui, mostramos uma abordagem para analisar e explorar um conjunto de dados multiômicos tão complexo de maneira não supervisionada, aplicando a análise fatorial multiômica (MOFA) a um conjunto de dados gerado a partir de amostras de sangue que capturam a resposta imune em síndromes coronarianas agudas e crônicas. O conjunto de dados consiste em vários ensaios em diferentes resoluções, incluindo dados de citocinas em nível de amostra, proteômica plasmática e prime-seq de neutrófilos e dados de RNA-seq de célula única (scRNA-seq). Mais complexidade é adicionada por ter vários pontos de tempo diferentes medidos por paciente e vários subgrupos de pacientes.
O fluxo de trabalho de análise descreve como integrar e analisar os dados em várias etapas: (1) Pré-processamento e harmonização de dados, (2) Estimativa do modelo MOFA, (3) Análise downstream. A Etapa 1 descreve como processar os recursos dos diferentes tipos de dados, filtrar recursos de baixa qualidade e normalizá-los para harmonizar suas distribuições para análise posterior. A etapa 2 mostra como aplicar o modelo MOFA e explorar as principais fontes de variação no conjunto de dados em todas as ômicas e recursos. A etapa 3 apresenta várias estratégias para a análise a jusante dos padrões capturados, vinculando-os às condições da doença e aos potenciais processos moleculares que governam essas condições.
No geral, apresentamos um fluxo de trabalho para exploração de dados não supervisionados de conjuntos de dados multiômicos complexos para permitir a identificação dos principais eixos de variação compostos por diferentes características moleculares que também podem ser aplicadas a outros contextos e conjuntos de dados multiômicos (incluindo outros ensaios, conforme apresentado no caso de uso exemplar).
Introdução
Os mecanismos da doença são geralmente complexos e governados pela interação de vários processos moleculares distintos. Decifrar os complexos mecanismos moleculares que levam a doenças específicas ou governam a evolução de uma doença é uma tarefa com alta relevância médica, pois pode revelar novos insights para a compreensão e o tratamento de doenças.
Avanços tecnológicos recentes permitem medir esses processos em uma resolução mais alta (por exemplo, no nível de uma única célula) e em várias camadas biológicas (por exemplo, DNA, mRNA, acessibilidade da cromatina, metilação do DNA, proteômica) ao mesmo tempo. Isso leva à geração crescente de grandes conjuntos de dados biológicos multidimensionais, que podem ser analisados em conjunto para gerar mais insights sobre os processos subjacentes. Ao mesmo tempo, combinar e analisar as diferentes fontes de dados de maneira biologicamente significativa continua sendo uma tarefa desafiadora1.
Diferentes limites tecnológicos, ruídos e faixas de variabilidade entre diferentes ômicas representam um desafio. Por exemplo, os dados de sequenciamento de RNA de célula única (scRNA-seq) são muito esparsos e muitas vezes influenciados por grandes efeitos técnicos ou de lote. Além disso, o espaço de recursos costuma ser muito grande, variando entre vários milhares de genes ou proteínas medidos, enquanto os tamanhos das amostras são limitados. Isso é ainda mais complicado por projetos complexos, que podem incluir várias condições de doença, fatores de confusão, pontos de tempo e resoluções. Por exemplo, no caso de uso apresentado, diferentes tipos de dados estavam disponíveis no nível de célula única ou de amostra (em massa). Além disso, os dados podem estar incompletos e nem todas as medidas podem estar disponíveis para todos os indivíduos analisados.
Devido a esses desafios, diferentes ômicas e características incluídas ainda são frequentemente analisadas apenas separadamente2, embora a realização de uma análise integrada não possa apenas fornecer uma imagem completa do processo, mas os ruídos biológicos e técnicos de uma ômica também possam ser compensados por outras ômicas 3,4. Vários métodos diferentes foram propostos para realizar uma análise integrada de dados multiômicos, incluindo métodos bayesianos, métodos baseados em rede 5,6, aprendizado profundo multimodal7 e métodos de redução de dimensionalidade via fatoração de matriz 8,9. Para este último, os resultados de um grande estudo de benchmarking10 mostraram que o método MOFA9 (análise fatorial multiômica) é uma das ferramentas mais adequadas quando os dados devem ser vinculados a anotações clínicas.
Especialmente em ambientes complexos, os métodos de fatoração de matriz não supervisionada são uma abordagem útil para reduzir a complexidade e extrair sinais compartilhados e complementares de diferentes fontes de dados e recursos. Ao decompor o espaço complexo em representações latentes de classificação inferior, as principais fontes de variação dentro dos dados podem ser rapidamente exploradas e vinculadas a covariáveis conhecidas. Caso o mesmo padrão de variação seja compartilhado entre vários recursos (por exemplo, genes ou proteínas), isso pode ser agregado a alguns fatores enquanto o ruído é reduzido. A regularização pode ser usada para aumentar a dispersão dos coeficientes do modelo, o que torna a abordagem adequada em configurações onde o espaço de recursos é grande enquanto o número de amostras é limitado9.
Este protocolo apresenta um fluxo de trabalho de análise flexível que usa o modelo MOFA para mostrar como explorar rapidamente um conjunto de dados multi-ômicos complexo e destilar os principais padrões de variação que caracterizam esse conjunto de dados. O fluxo de trabalho consiste em três etapas principais. Na primeira etapa, Pré-processamento e harmonização de dados, são apresentadas diferentes estratégias de pré-processamento de dados com base em diferentes tipos de dados de entrada (scRNA-seq, proteômica, citocina, dados clínicos). O protocolo elabora como processar os recursos dos diferentes conjuntos de dados de entrada, filtrar recursos de baixa qualidade e normalizá-los para harmonizar suas distribuições. Também mostramos como essas decisões de pré-processamento podem afetar os resultados downstream. Na segunda etapa, o modelo MOFA é aplicado aos dados, e a decomposição da variância resultante pode ser usada para avaliar a integração dos diferentes conjuntos de dados. A terceira etapa mostra como vincular os fatores capturados às covariáveis e descobrir os programas moleculares que definem esses fatores. Com o fluxo de trabalho apresentado, fomos capazes de extrair vários fatores latentes ligados a covariáveis clínicas em um conjunto de dados de pacientes que sofrem de síndromes coronarianas e identificar potenciais programas imunológicos multicelulares subjacentes de um projeto anterior11. Usaremos esse conjunto de dados aqui, mas o protocolo pode ser facilmente aplicado a outros contextos, incluindo outras ômicas.
O conjunto de dados consiste em amostras de pacientes com síndromes coronarianas crônicas estáveis (SCC), síndromes coronarianas agudas (SCA) e um grupo controle com coronárias saudáveis (não CCS) (Figura 1). A SCA é causada pela ruptura da placa na CCS preexistente, levando a uma interrupção aguda do fluxo sanguíneo para o miocárdio e uma subsequente lesão isquêmica do coração. Essa lesão provoca uma resposta inflamatória do sistema imunológico seguida de uma fase reparadora, que dura até vários dias após o evento agudo12. Para poder caracterizar essa resposta imune para pacientes com SCA, amostras de sangue foram coletadas em quatro momentos diferentes: aguda (TP1); após a recanalização (14 [± 8] h) (TP2); 60 [± 12] h mais tarde (TP3); antes da alta (6,5 [±1,5] dias) (TP4) (Figura 1A). Para CCS e pacientes com coronárias saudáveis, apenas um ponto de tempo estava disponível- (TP0). Para todos os pacientes e pontos de tempo diferentes ensaios baseados nas amostras de sangue foram medidos: marcadores clínicos de inflamação (creatina-quinase (CK), CK-MB, Troponina, proteína C reativa (PCR)), scRNA-seq de células mononucleares do sangue periférico (PBMCs), análise de citocinas, proteômica plasmática e dados prime-seq13 de neutrófilos.
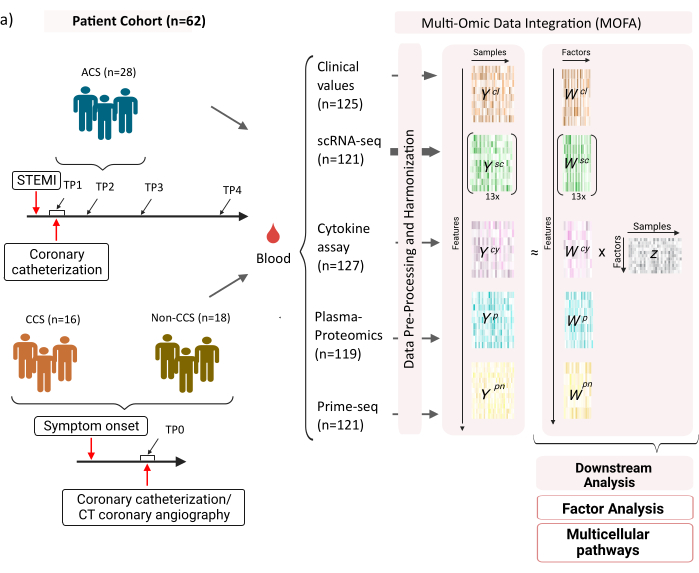
Figura 1: Conjunto de dados de entrada multiômica de infarto do miocárdio. Conjunto de dados de entrada: Os dados analisados incluem amostras de sangue de pacientes (n = 62) com síndrome coronariana aguda (SCA), síndromes coronarianas crônicas (SCC) e pacientes com coronárias saudáveis (não CCS). Para pacientes com SCA, amostras de sangue foram incluídas em quatro momentos diferentes (TP1-4), para pacientes com CCS e não CCS em um único momento (TP0). Cada combinação de paciente e ponto de tempo é tratada como uma amostra separada na análise. Diferentes ensaios ômicos foram medidos nas amostras: exames clínicos de sangue (n = 125), scRNA-seq (n = 121), plasma-proteômica (n = 119), ensaio de citocinas (n = 127) e neutrófilo prime-seq (n = 121). Posteriormente, o protocolo descrito foi aplicado para integrar os dados em todas as ômicas e explorá-los usando o modelo MOFA e posterior análise a jusante (análise fatorial, enriquecimento de via). Clique aqui para ver uma versão maior desta figura.
Como entrada para o fluxo de trabalho, conforme apresentado aqui, pegamos contagens brutas de dados scRNA-seq após o processamento com cellranger e controle de qualidade (QC), como, por exemplo, descrito no tutorial de pré-processamento scanpy14 . Para anotação do tipo de célula, usamos o pipeline automatizado Azimute15 . As contagens são então agregadas no nível da amostra para cada tipo de célula, tomando a média em todas as células para cada amostra e tipo de célula (agregação pseudobulk). A proteômica plasmática é incluída como intensidades normalizadas e centradas na mediana e, para neutrófilos, tomamos as contagens de éxons do identificador molecular único (UMI) do prime-seq. Sobre citocinas e valores clínicos, nenhum pré-processamento prévio foi aplicado. Mais detalhes sobre a geração de dados (experimentais) estão descritos no manuscrito correspondente11. Como os resultados apresentados aqui são baseados no uso da anotação automatizada de azimute para tipos de células nos dados scRNA-seq em comparação com a estratégia baseada em marcadores que foi usada na publicação referenciada, os resultados apresentados aqui são semelhantes, mas não exatamente os mesmos apresentados na publicação. No manuscrito, pode-se mostrar que a estratégia de anotação do tipo de célula não altera os principais padrões e interpretações biológicas da análise, mas pequenas mudanças nos valores exatos resultantes do modelo podem variar. No geral, os dados de entrada eram um conjunto de dados multidimensional complexo, incluindo diferentes pontos de tempo e níveis de medição (células únicas vs. bulk) de mais de 10.000 características diferentes (genes, proteínas, valores clínicos). Uma estratégia rigorosa de pré-processamento e harmonização de dados seguida pela análise MOFA demonstrou ser uma ferramenta útil e rápida para explorar os dados e extrair o programa imunológico relevante. Cada ponto de tempo e combinação de pacientes é tratado como uma amostra independente na análise MOFA. Cada tipo de dados e tipo de célula é considerado uma exibição separada na análise MOFA.
Este protocolo fornece instruções para preparar os dados de entrada para o fluxo de trabalho, executar as diferentes etapas do fluxo de trabalho, personalizar configurações, interpretar as figuras resultantes e ajustar iterativamente as configurações com base nas interpretações. Uma visão geral das diferentes etapas do protocolo, os conjuntos de dados de entrada necessários em cada etapa e as figuras e conjuntos de dados resultantes é fornecida pela visão geral do fluxo de trabalho técnico (Figura 2).
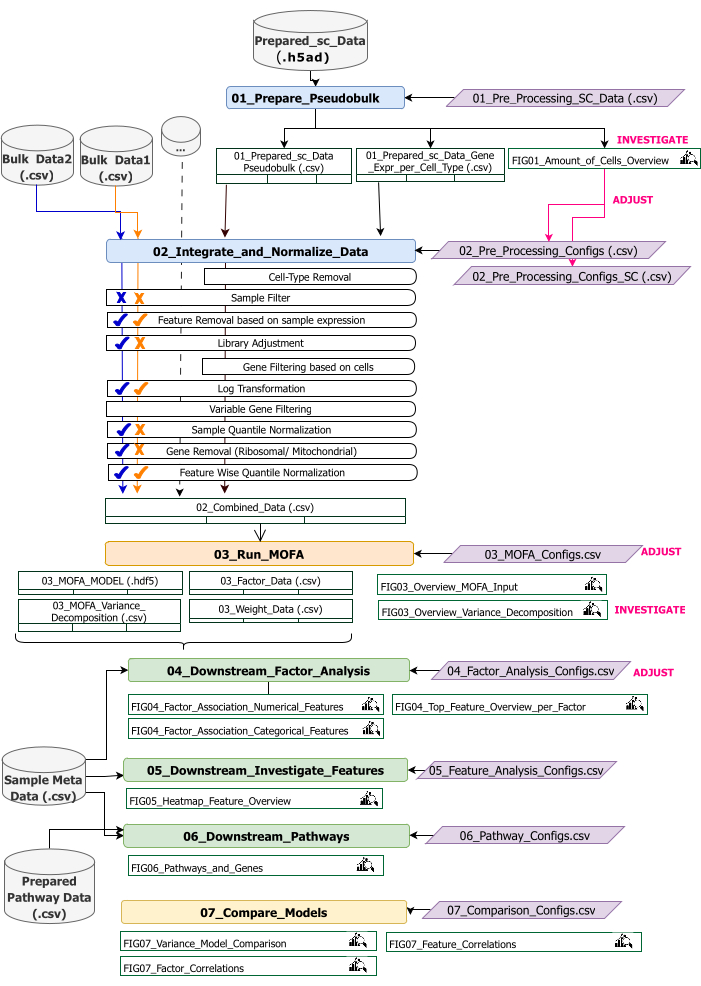
Figura 2: Visão geral do fluxo de trabalho técnico. Esboço do fluxo de trabalho para a análise do conjunto de dados multiômicos. Os diferentes elementos são destacados por diferentes cores e símbolos. Os Jupyter Notebooks pertencentes à etapa Pré-processamento e harmonização de dados (1) são coloridos em azul. Os Jupyter Notebooks pertencentes à etapa 'Modelo MOFA' (2) são coloridos em laranja. Os Jupyter Notebooks pertencentes à etapa "Análise downstream" (3) são coloridos em verde. Um Jupyter Notebook a ser usado para comparação dos resultados é colorido em amarelo. Os arquivos de configuração em que os parâmetros para a execução do fluxo de trabalho podem ser modificados são destacados em roxo. Os conjuntos de dados de entrada necessários para executar o fluxo de trabalho são indicados pelo símbolo do conjunto de dados e destacados em cinza. Todas as saídas de figura geradas durante a execução do fluxo de trabalho são indicadas pelo símbolo da lupa. Os conjuntos de dados gerados durante a execução do fluxo de trabalho são indicados como tabelas. Em geral, o fluxo de trabalho é executado sequencialmente: (1) O pré-processamento e a harmonização de dados consistem em duas etapas: primeira geração de uma tabela pseudobulk com base nos dados de entrada scRNA-seq (01_Prepare_Pseudobulk) e subsequente integração e normalização desses dados junto com todas as outras entradas (em massa) em nível de amostra (02_Integrate_and_Normalize_Data). Nesta etapa, por meio dos arquivos de configuração, é possível configurar para cada conjunto de dados separadamente quais das etapas de pré-processamento e normalização indicadas (por exemplo, Filtro de Amostra) devem ser aplicadas. (2) «Modelo MOFA»: executa o modelo MOFA na entrada gerada da primeira etapa com as configurações especificadas no ficheiro de configuração (03_MOFA_configs.csv) (3) «Análise a jusante»: consiste em três blocos de anotações diferentes que podem ser executados independentemente uns dos outros para gerar informações sobre os resultados MOFA gerados e associá-los a metadados de amostra (covariáveis) fornecidos como entrada através do ficheiro «Sample Meta Data.csv». (4) «Comparação de modelos»: é uma pequena etapa separada que pode ser utilizada para comparar diferentes modelos gerados na etapa 2. Clique aqui para ver uma versão maior desta figura.
O fluxo de trabalho consiste em vários Jupyter Notebooks escritos em R e Python (o conhecimento da linguagem R e Python não é necessário para executar o fluxo de trabalho, mas pode ser útil caso apareçam erros). Em várias etapas do protocolo, os parâmetros são alterados por meio de arquivos de configuração (arquivos '.csv' contendo o sufixo '_Configs' no nome). Dentro do protocolo, apenas descrevemos os parâmetros que precisam ser alterados a partir da configuração padrão.
Vários outros parâmetros também podem ser alterados, por exemplo, para personalizar o pré-processamento. Uma documentação desses parâmetros e explicações é fornecida no arquivo 'Documentation_Config_Parameter', que está incluído no repositório baixado.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Preparativos: Configuração técnica e instalação
NOTA: Para executar este programa, tenha wget, git e Apptainer pré-instalados no dispositivo. Um guia para instalar o Apptainer em diferentes sistemas (Linux, Windows, Mac) é fornecido aqui: https://apptainer.org/docs/admin/main/installation.html. As informações de instalação no git podem ser encontradas aqui: https://git-scm.com/book/en/v2/Getting-Started-Installing-Git. Dependendo do tamanho dos diferentes conjuntos de dados de entrada, é recomendável executar o fluxo de trabalho em um computador adequado (16 CPUs, 64 GB de memória). Um teste de fumaça com os dados de exemplo fornecidos pode ser executado na máquina local. As instruções e as saídas esperadas da execução do protocolo nos dados de exemplo são fornecidas no Arquivo Suplementar 1. Consulte o Arquivo de Vídeo Suplementar 1 para obter as etapas importantes do protocolo que são executadas no conjunto de dados descrito acima.
- Abra o console e escolha ou crie uma pasta onde todo o código de análise e saídas serão armazenados. Navegue até a pasta digitando o comando: cd path_to_folder no terminal.
- Baixe ou clone o repositório de código do Github (https://github.com/heiniglab/mofa_workflow) ou digitando git clone https://github.com/heiniglab/mofa_workflow.git na janela do terminal.
- Baixe a imagem que contém todas as instalações necessárias do Zenodo digitando wget https://zenodo.org/records/11192947/files/mofa_image.sif na janela do terminal.
- Gere uma pasta na qual todos os dados do resultado serão armazenados digitando mkdir results na janela do terminal.
- Gere uma pasta na qual todos os dados de entrada a serem usados na análise serão adicionados digitando mkdir input_data na janela do terminal.
- Execute o contêiner que iniciará uma sessão do JupyterLab digitando o seguinte comando no terminal: apptainer run mofa_image.sif. Copie a URL retornada pelo comando para o navegador, que abrirá uma sessão do Jupyter-lab (mais informações sobre o Jupyter-lab podem ser encontradas na documentação do software16).
NOTA: Quando o fluxo de trabalho é executado localmente em um laptop, é recomendável usar o comando apptainer exec mofa_image.sif jupyter-lab , que retornará diretamente um endereço de host local. Caso o contêiner seja executado em um ambiente de computação em cluster, pode ser necessário configurar o encaminhamento de porta, o que pode ser feito via ssh.
2. Inicialização e preparação de dados
- Na sessão do Jupyter-Lab, use o menu de navegação no lado esquerdo. Navegue até a pasta input_data clicando duas vezes em input_data.
- Copie todos os conjuntos de dados que serão usados como entrada para a análise para o diretório input_data usando Arrastar e soltar. Arraste o arquivo da pasta em que ele está localizado no momento e solte-o na sessão do Jupyter-lab na área abaixo da pasta input_data.
NOTA: Todos os conjuntos de dados precisam estar no formato .csv ou .h5ad (no caso de dados de célula única). Todos os arquivos .csv devem conter uma coluna sample_id correspondente (IDs idênticas devem ser usadas nos conjuntos de dados). Todas as outras colunas serão usadas como recursos. Dentro do arquivo h5ad-, a anotação da célula deve conter dois identificadores especificando o sample_id e o cluster_id. Eles serão usados para agregação e correspondência. Os conjuntos de dados ômicos em outros formatos precisam ser convertidos para o formato .csv especificado antes do uso (Figura 3). Os conjuntos de dados scRNA-seq fornecidos no formato .h5seurat podem ser convertidos em .h5ad executando o Jupyter-notebook: 00_Data_Conversion.ipynb. - Navegue até a pasta de configurações clicando no símbolo da pasta e, em seguida, clicando duas vezes nas pastas mofa_workflow, scripts e configurações. Dentro da pasta, abra o arquivo Data_configs.csv clicando duas vezes nele.
- Na coluna de valor , adicione os caminhos às pastas das pastas input_data (data_path) e resultados (result_path). Adicione um nome que será adicionado como uma extensão de arquivo a todos os arquivos salvos na coluna de valor do configuration_name (este protocolo usado MI_v1 [Infarto do Miocárdio versão 1]) (Figura 4).
- Salve as alterações clicando em Arquivo > Salvar arquivo CSV no menu na parte superior.
- Use o menu de navegação no lado esquerdo para navegar até a pasta de scripts clicando em scripts. Abra o bloco de anotações de inicialização clicando duas vezes em 00_Configuration_Update.ipynb. Execute o script clicando no botão Reiniciar kernel e executar todas as células na parte superior e clicando em Reiniciar no pop-up (Figura 5).
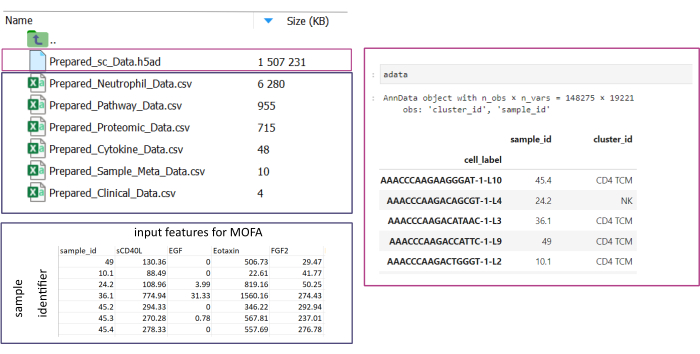
Figura 3: Entrada e configuração de dados. Para a execução do fluxo de trabalho, todos os dados precisam ser armazenados em uma pasta input_data especificada. Para cada conjunto de dados de entrada, um arquivo separado deve ser fornecido. Os dados de célula única devem ser fornecidos como .h5ad contendo anotação de célula no cluster_id (resultante, por exemplo, de etapas anteriores de anotação de tipo de célula) e uma coluna sample_id (identificando exclusivamente cada amostra separada que deve ser analisada). Todos os outros conjuntos de dados de entrada devem ser apresentados no formato «.csv», incluindo uma coluna que especifica o sample_id (correspondente à coluna correspondente dos dados unicelulares) e as características a utilizar na análise MOFA em todas as outras colunas. Clique aqui para ver uma versão maior desta figura.
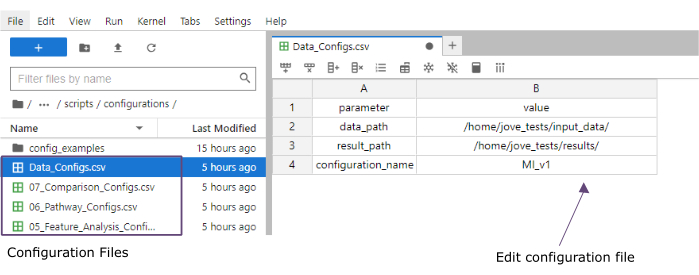
Figura 4: Arquivos de configuração do Jupyter-lab. Durante a execução do fluxo de trabalho, as alterações nos parâmetros (por exemplo, ajuste das opções de filtragem, etc.) são especificadas por meio de arquivos de configuração '.csv'. No repositório clonado, os arquivos de configuração padrão para cada etapa são incluídos. Eles podem ser editados diretamente no console do jupyter-lab, da mesma forma que em uma planilha. Clique aqui para ver uma versão maior desta figura.
Figura 5: scripts Jupyter-notebooks. O fluxo de trabalho completo consiste em uma série de blocos de anotações Jupyter que serão executados sequencialmente após a modificação dos arquivos de configuração correspondentes. Ao clicar duas vezes no bloco de anotações Jupyter no lado esquerdo, o arquivo correspondente será aberto no lado direito. A execução completa do arquivo pode ser iniciada com o botão destacado na parte superior. Clique aqui para ver uma versão maior desta figura.
3. Pré-processamento e harmonização de dados
- Pré-processamento - Converta dados sc em pseudobulk.
NOTA: Esta etapa só precisa ser executada se dados de célula única forem usados na análise.- Use o menu de navegação no lado esquerdo para navegar até a pasta de configurações clicando duas vezes em configuração. Abra o 01_Preprocessing_SC_Data.csv de arquivos clicando duas vezes. Verifique os valores preenchidos automaticamente no arquivo e, se necessário, ajuste os valores na coluna data_name para corresponder aos nomes dos conjuntos de dados de célula única na pasta input_data que será usada para a análise.
NOTA: Por padrão, todos os nomes dos arquivos .h5ad na pasta de dados de entrada serão adicionados ao arquivo de configuração no script de inicialização. Se alguns dos conjuntos de dados não devem ser usados para a análise, eles podem ser removidos aqui. - Salve as alterações feitas clicando em Arquivo > Salvar arquivo CSV no menu na parte superior.
- Use o menu de navegação no lado esquerdo para navegar até a pasta de scripts clicando em scripts. Abra o notebook 01_Prepare_Pseudobulk.ipynb clicando duas vezes nele. Execute o script clicando no botão Reiniciar kernel e executar todas as células na parte superior e clicando em Reiniciar no pop-up.
- Use o menu de navegação do lado esquerdo para navegar até a pasta de figuras clicando duas vezes primeiro nas figuras e depois em 01_figures. Abra o FIG01_Amount_of_Cells_overview de plotagem recém-gerado clicando duas vezes nele.
NOTA: A execução do notebook pode levar vários minutos. Quando o notebook for executado com sucesso, um pop-up aparecerá e o FIG01_Amount_of_Cells_Overview de arquivo será atualizado pelo notebook ou gerado novamente. A coluna Última modificação pode indicar quando o arquivo foi gerado para avaliar se é um arquivo novo ou antigo. - Investigue o gráfico para identificar clusters de tipo de célula com um número muito baixo de células por amostra. Anote os nomes desses cluster_ids para excluí-los nas etapas subsequentes (Figura 6).
- Use o menu de navegação no lado esquerdo para navegar de volta para a pasta de configurações clicando em ... e, em seguida, clique duas vezes em configurações. Abra o 02_Preprocessing_Configs_SC.csv de arquivos clicando duas vezes nele.
- Verifique os valores nas colunas configuration_name e data_name e ajuste-os, se necessário.
NOTA: No script de inicialização, esses valores são pré-preenchidos com todos os nomes dos arquivos .h5ad na pasta de dados de entrada e o valor configuration_name definido no arquivo Data_Configs.csv anteriormente. Caso os arquivos devam ser excluídos da análise ou outra extensão para nomes de arquivos deva ser usada, isso pode ser ajustado aqui. - Ajuste o valor na coluna cell_type_exclusion e adicione todos os cluster_id que foram identificados para excluir na etapa anterior separados por ','.
- Salve as alterações clicando em Arquivo > Salvar arquivo CSV na barra de navegação na parte superior.
- Use o menu de navegação no lado esquerdo para navegar até a pasta de configurações clicando duas vezes em configuração. Abra o 01_Preprocessing_SC_Data.csv de arquivos clicando duas vezes. Verifique os valores preenchidos automaticamente no arquivo e, se necessário, ajuste os valores na coluna data_name para corresponder aos nomes dos conjuntos de dados de célula única na pasta input_data que será usada para a análise.
- Pré-processamento - Harmonize e integre outras fontes de dados ômicas.
- Abra o 02_Preprocessing_Configs.csv de arquivos clicando duas vezes nele e ajuste a configuração de pré-processamento para cada um dos conjuntos de dados que serão incluídos e armazenados na pasta data_input (uma linha por conjunto de dados).
- Verifique os valores nas colunas configuration_name e data_name e ajuste-os, se necessário.
- Ajuste os outros parâmetros nas colunas de acordo, dependendo de quais etapas de pré-processamento devem ser aplicadas.
NOTA: Os valores padrão são adicionados para cada conjunto de dados encontrado na pasta input_dataset, mas não são específicos para os tipos de dados únicos dos dados. Portanto, ajustes serão necessários. Uma documentação detalhada dos parâmetros é fornecida no arquivo Documentation_Config_Parameter.doc . - Salve as alterações clicando em Arquivo > Salvar arquivo CSV.
- Use o menu de navegação no lado esquerdo para navegar até a pasta de scripts clicando em scripts. Abra o notebook 02_Integrate_and_Normalized_Data_Sources.ipynb clicando duas vezes nele. Execute o script clicando no botão Reiniciar kernel e executar todas as células na parte superior e clicando em Reiniciar no pop-up.
- Use o menu de navegação no lado esquerdo para navegar até a pasta 02_results gerada clicando no símbolo da pasta e, em seguida, clicando duas vezes em resultados e 02_results. Verifique se ele inclui o arquivo 02_Combined_data_'configuration_name'_Integrated.csv contendo o arquivo de entrada de dados pré-processados combinados.
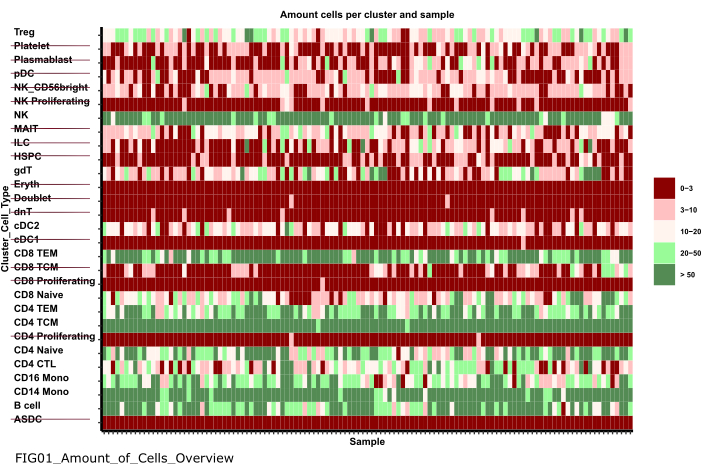
Figura 6: Pré-processamento e harmonização de dados. Uma saída da etapa '01_Prepare_Pseudobulk' é o gráfico 'Fig01_Amount_of_Cells_Overview'. Aqui, para cada cluster_id (eixo y indicando o tipo de célula das etapas anteriores de anotação do tipo de célula), o número de células por amostra ('sample_id') é fornecido. Dentro dos resultados apresentados, os tipos de células com baixa quantidade de células por amostra são excluídos da análise subsequente (indicada pelo tachado). Clique aqui para ver uma versão maior desta figura.
4. Executando o MOFA
- No Jupyter-Lab, use o menu de navegação no lado esquerdo para navegar até a pasta de configurações clicando no símbolo da pasta e, em seguida, clicando duas vezes em mofa_workflow, seguido de um clique duplo em scripts e configurações. Abra o arquivo 03_MOFA_Configs.csv clicando duas vezes nele.
- Verifique as entradas das colunas configuration_name e mofa_result_name e ajuste as entradas se nomes alternativos devem ser usados.
NOTA: O mofa_result_name será anexado como uma extensão de arquivo a todos os arquivos de resultados gerados com base no MOFA. Isso pode ser diferente do valor configuration_name , pois diferentes configurações de MOFA podem ser executadas com os mesmos dados de entrada (esse protocolo usa MI_v1_MOFA). - Insira a quantidade de fatores que devem ser estimados no modelo MOFA (coluna amount_of_factors ) e defina se a ponderação e o dimensionamento devem ser aplicados (weighting_of_views e scale_views colunas) ajustando os valores no arquivo.
- Salve as alterações clicando em Arquivo > Salvar arquivo CSV.
- Use o menu de navegação no lado esquerdo para navegar até a pasta de scripts clicando em 'scripts'. Abra o notebook 03_Run_MOFA.ipynb clicando duas vezes no arquivo. Execute o script clicando no botão Reiniciar kernel e executar todas as células na parte superior e clicando em Reiniciar no pop-up.
- Navegue até a pasta 03_figures clicando duas vezes nas figuras e depois em 03_figures. Abra o gráfico gerado FIG03_Overview_Variance_Decomposition_'mofa_result_name e investigue o resultado do modelo (Figura 7A).
- Use o menu de navegação no lado esquerdo para navegar até a pasta 03_results gerada clicando no símbolo da pasta e, em seguida, clicando duas vezes nos resultados e 03_results. Verifique se ele inclui o arquivo de valor do fator de amostra 03_Factor_Data_'mofa_result_name'.csv e o arquivo de peso do fator de recurso 03_Weight_Data_'mofa_result_name'.csv.
5. Análise a jusante
- Interpretação fatorial.
- Use o menu de navegação no lado esquerdo para navegar até a pasta input_data clicando no símbolo da pasta e, em seguida, clicando duas vezes em input_data.
- Prepare um arquivo .csv (Prepared_Sample_Meta_Data.csv) que contenha todos os metadados (covariáveis) das amostras que serão analisadas em associação com os fatores gerados. Copie o arquivo para a pasta input_data usando Arrastar e Soltar , soltando o arquivo na visão geral da pasta input_data .
NOTA: Ele precisa conter a coluna sample_id para combiná-lo com os dados usados anteriormente e outras colunas para cada feição que deve ser analisada. - No Jupyter-Lab, use o menu de navegação à esquerda para navegar de volta para a pasta de configurações clicando no símbolo da pasta e, em seguida, clicando duas vezes em mofa_workflow, seguido por scripts e configuração. Abra o arquivo 04_Factor_Analysis.csv clicando duas vezes nele.
- Verifique se as entradas para o configuration_name e mofa_result_name contêm os nomes da configuração e os resultados do MOFA que serão analisados no script e ajuste-os se necessário.
- Na coluna numeric_covariates , adicione o nome de todas as colunas numéricas no arquivo Prepared_Sample_Meta_Data.csv que serão investigadas em relação aos fatores MOFA separados por vírgula (Este protocolo usa CRP,CK).
- Na coluna categorical_covariates, adicione o nome de todas as colunas categóricas no arquivo Prepared_Sample_Meta_Data.csv que serão investigadas em relação aos fatores MOFA separados por vírgula (Este protocolo usa medição).
- Salve as alterações clicando em Arquivo > Salvar arquivo CSV.
- Use o menu de navegação à esquerda para navegar até a pasta 'scripts' clicando em scripts. Abra o notebook 04_Downstream_Factor_Analysis.ipynb clicando duas vezes nele. Execute o script clicando no botão Reiniciar kernel e executar todas as células na parte superior e clicando em reiniciar no pop-up.
- Use o menu de navegação à esquerda para navegar até a pasta 04_figures clicando duas vezes nas figuras e depois em 04_figures. Abra os gráficos gerados clicando duas vezes neles e investigue os fatores para padrões e associações interessantes: FIG04_Factor_Association_with_numeric_features_
'mofa_result_name.pdf (Figura 7B). FIG04_Factor_Association_
with_categorical_features_ mofa_result_name.pdf (Figura 7C). FIG04_Top_Feature_Overview_per_Factor _'mofa_result_name.pdf (Figura 8A).
- Análise de recursos
- Use o menu de navegação à esquerda para navegar de volta para a pasta de configurações clicando em ... e, em seguida, clique duas vezes em configurações. Abra o arquivo 05_Feature_Analysis_Configs.csv clicando duas vezes nele.
- Verifique se as entradas das colunas configuration_name e mofa_result_name correspondem aos nomes da configuração e do resultado MOFA gerado que será usado para a análise downstream e ajuste-os, se necessário.
- Na coluna de fatores , adicione o fator para o qual os principais recursos serão plotados no próximo script.
- Na coluna faceting_variable, adicione um nome de coluna de uma coluna categórica no Prepared_Sample_Meta_Data.csv que será usado para agrupar as amostras no gráfico (Este protocolo usa medição)
- Salve as alterações clicando em Arquivo > Salvar arquivo CSV.
- Use o menu de navegação à esquerda para navegar até a pasta de scripts clicando em scripts. Abra o notebook 05_Downstream_Investigate_Features_Heatmap.ipynb clicando duas vezes nele. Execute o script clicando no botão Reiniciar kernel e executar todas as células na parte superior e clicando em Reiniciar no pop-up.
- Use o menu de navegação à esquerda para navegar até a pasta 05_figures clicando duas vezes primeiro nas figuras e depois em 05_figures. Abra e investigue o gráfico gerado FIG05_Heatmap_Feature_Overview__ 'mofa_result_name'.pdf clicando duas vezes no arquivo (Figura 8B).
NOTA: Dependendo da quantidade de recursos que serão mostrados no gráfico, pode ser necessário ajustar os parâmetros plot_width e plot_height dentro do 05_Feature_Analysis_Configs.csv e executar novamente o script para garantir que tudo se encaixe no gráfico.
- Análise de vias
- Use o menu de navegação no lado esquerdo para navegar até a pasta input_data clicando no símbolo da pasta e, em seguida, clicando duas vezes em input_data.
- Prepare um arquivo .csv (Prepared_Pathway_Data.csv) que contenha uma lista de caminhos que serão testados quanto ao enriquecimento. Copie o arquivo para a pasta input_data usando Arrastar e Soltar, soltando o arquivo na visão geral da pasta input_data.
NOTA: Ele precisa conter três colunas: ID (um identificador exclusivo da via), gene (os genes dados pelo nome do gene (SÍMBOLO) pertencente à via, uma linha por gene) pathway_name (um nome/descrição textual das vias). - Na sessão do Jupyter-Lab, use o menu de navegação à esquerda para navegar até a pasta de configurações clicando em ... e, em seguida, clique duas vezes em configurações. Abra o 06_Pathway_Configs.csv de arquivo clicando duas vezes nele.
- Verifique a entrada da coluna mofa_result_name e certifique-se de que ela corresponda ao nome do resultado MOFA gerado que será usado para o cálculo do enriquecimento do caminho.
- Verifique a entrada na coluna de tipos e remova as entradas na coluna de tipos que não contêm recursos que correspondem à coluna de genes no arquivo Prepared_Pathway_Data.csv .
NOTA: Por padrão, todas as diferentes visualizações que foram usadas no modelo MOFA são adicionadas a esse arquivo durante a execução do fluxo de trabalho. Caso haja visualizações que não contenham feições que correspondam a pelo menos um caminho, elas precisam ser removidas; caso contrário, a execução falhará. Um exemplo seria o arquivo de caminho conter apenas anotações de caminho para genes, mas há uma exibição contendo nomes de proteínas. - Salve as alterações clicando em Arquivo > Salvar arquivo CSV.
- Use o menu de navegação para navegar até a pasta de scripts clicando em scripts. Abra o notebook 06_Downstream_Pathways.ipynb clicando duas vezes nele. Execute o script clicando no botão Reiniciar kernel e executar todas as células na parte superior e clicando em Reiniciar no pop-up.
- Use o menu de navegação à esquerda para navegar até a pasta 06_figures clicando primeiro duas vezes nas figuras e depois em 06_figures. Abra o gráfico gerado FIG06_Pathways_and_Genes_ 'mofa_result_name clicando duas vezes nele e investigue os caminhos visualizados (Figura 8C).
NOTA: A forma como os caminhos visualizados são selecionados pode ser configurada por meio do arquivo de configuração. Para obter mais detalhes, consulte a documentação dos parâmetros. - Use o menu de navegação no lado esquerdo para navegar até a pasta 06_results gerada clicando no símbolo da pasta e, em seguida, clicando duas vezes nos resultados e 06_results. Verifique se ele inclui o arquivo que inclui as vias enriquecidas 06_Pathway_enrichment__'mofa_result_name'.
6. Comparação de diferentes configurações e versões (Figura Suplementar 1, Figura Suplementar 2, Figura Suplementar 3, Figura Suplementar 4)
- Para comparar o efeito do uso de diferentes parâmetros/configurações em todo o fluxo de trabalho, execute novamente as seções 3 a 5, modificando os parâmetros nos arquivos de configuração e usando diferentes identificadores de configuration_name e mofa_result_name .
NOTA: Novos resultados serão armazenados com esses nomes para serem usados para comparar diferentes execuções. - No Jupyter-Lab, use o menu de navegação à esquerda para navegar até a pasta de configurações . Abra o arquivo 07_Comparison_Configs.csv clicando duas vezes nele.
- Na coluna mofa_result_name , adicione os nomes de todas as execuções anteriores do MOFA que serão comparadas (uma linha por nome/configuração, por exemplo, MI_v1_MOFA, MI_v2_MOFA).
- Na coluna compare_factors, adicione os fatores que serão comparados entre os modelos. Por padrão, é Factor1,Factor2,Factor3. (Figura 2A suplementar).
NOTA: Neste script, os valores de características e fatores dos diferentes modelos serão comparados correlacionando-os. Isso só funciona para modelos baseados nas mesmas amostras (indicadas por sample_id) e no mesmo conjunto de recursos. Caso as amostras ou recursos não correspondam entre as versões comparadas, eles serão excluídos da comparação. - Salve as alterações clicando em Arquivo > Salvar arquivo CSV.
- Use o menu à esquerda para navegar até a pasta de scripts clicando em scripts. Abra o notebook 07_Compare_Models.ipynb clicando duas vezes nele. Execute o script clicando no botão Reiniciar kernel e executar todas as células e clicando em Reiniciar no pop-up.
- Use o menu à esquerda para navegar até a pasta 06_figures clicando duas vezes primeiro nas figuras e depois em 06_figures. Abra os gráficos gerados clicando duas vezes nos arquivos para analisar a semelhança das diferentes versões:
FIG07_Variance_Model_Comparison.pdf (Figura Suplementar 2B)
FIG07_Factor_Correlations.pdf
(Figura Suplementar 2C)
FIG07_Feature_Correlations.pdf
(Figura 3C suplementar)
7. Estendendo o fluxo de trabalho: Adicionando outros parâmetros e configurações
NOTA: Além dos parâmetros que atualmente podem ser configurados nos arquivos de configuração, outros ajustes no código ou outros parâmetros podem ser incluídos. Por exemplo, o próprio modelo MOFA oferece vários outros parâmetros de treinamento17 que podem ser modificados diretamente no código ou ajustáveis por meio dos arquivos de configuração. A próxima seção do protocolo descreverá um exemplo de como fazer isso para parâmetros adicionais de treinamento do modelo MOFA. Para esta parte, é necessário conhecimento de programação em R.
- No Jupyter-Lab, use o menu de navegação à esquerda para navegar até a pasta de scripts . Abra o notebook 03_Run_MOFA.ipynb clicando duas vezes nele.
- Clique na guia Sumário no lado esquerdo e, em seguida, navegue até a subseção 4.3 Definir opções de treinamento MOFA e execute o treinamento do modelo clicando nele. Role para baixo para ver a saída impressa do modelo MOFA de parâmetros configuráveis no notebook.
- Dentro do loop for do R no código abaixo do título, todos os dados, modelo e opções de treinamento do MOFA são definidos. Abaixo da linha model_opts$num_factors = mofa_configs$amount_of_factors[i], adicione outra linha com o código abaixo
model_opts$probabilidades['data_type'] = 'poisson'.
NOTA: Isso alterará a distribuição que o modelo usa como entrada para a vista especificada pelo nome data_type para todas as execuções do MOFA. Ao especificar poisson para um tipo de dados, o modelo só será executado quando os recursos para esse tipo de dados forem inteiros (por exemplo, contagens de leitura de RNA-seq). Para obter mais informações sobre os dados, treinamento e opções de modelo do MOFA, também é possível consultar os tutoriais e a documentação do MOFA17. - Salve as alterações no bloco de anotações clicando no botão Salvar na parte superior.
- Para entregar novos parâmetros por meio dos arquivos de configuração .csv , use a navegação no lado esquerdo para navegar até a pasta de configurações clicando duas vezes nas configurações e abra o 03_MOFA_Configs.csv de arquivos clicando duas vezes.
- Adicione uma nova coluna especificando o nome do parâmetro, por exemplo, number_iterations e insira um valor, por exemplo, 1000. Salve as alterações clicando em Arquivo > Salvar arquivo CSV.
- Use o menu de navegação para navegar na pasta de scripts clicando em scripts. Abra o notebook 03_Run_Mofa.ipynb clicando duas vezes nele. Clique na guia Sumário no lado esquerdo e, em seguida, navegue até a subseção 4.3 Definir opções de treinamento MOFA e execute o treinamento do modelo clicando nele.
- Substitua a linha train_opts$maxiter = 50000 por train_opts$maxiter = mofa_configs$column_name[i] (quando o nome da coluna adicionada é number_of_iterations é train_opts$maxiter = mofa_configs$number_of_iterations[i])).
NOTA: O arquivo de configuração 03_MOFA_Configs.csv neste notebook é lido no início do notebook (subseção: Configurações e parâmetros de pré-requisitos) como mofa_config data.frame na sessão e, portanto, nesta linha de código, esse objeto e a coluna recém-gerada correspondente são referenciados. Como várias configurações podem ser executadas ao mesmo tempo, o i identifica a linha do data.frame à medida que a estimativa do modelo é executada em um loop for em todas as diferentes linhas no arquivo .csv . O princípio de leitura no arquivo de configuração no início do notebook na seção 'Pré-requisitos, Configurações e Parâmetros' é o mesmo para todos os notebooks, e outras modificações podem ser feitas como esta. - Salve as alterações no bloco de anotações clicando no botão Salvar .
Access restricted. Please log in or start a trial to view this content.
Resultados
Após a execução bem-sucedida do fluxo de trabalho, várias tabelas e figuras são geradas, conforme indicado na Figura 2. As figuras são colocadas na pasta /figures (Figura 6, Figura 7, Figura 8, Figura Suplementar 1, Figura Suplementar 2, Figura Suplementar 3, Figura Suplementar 4)...
Access restricted. Please log in or start a trial to view this content.
Discussão
Com o protocolo descrito, é apresentado um fluxo de trabalho modular e extensível baseado em Jupyter-notebook que pode ser usado para explorar rapidamente um conjunto de dados multi-ômicos complexo. As principais partes do fluxo de trabalho consistem na parte de pré-processamento e harmonização de dados (oferecendo diferentes etapas padrão para filtragem e normalização dos dados), estimativa do modelo MOFA9 e algumas análises exemplares a jusante. Uma da...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores declaram não haver conflito de interesses.
Agradecimentos
C.L. é apoiado pela Associação Helmholtz sob a escola de pesquisa conjunta "Munich School for Data Science - MUDS".
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Apptainer | NA | NA | https://apptainer.org/docs/admin/main/installation.html |
| Compute server or workstation or cloud (Linux, Mac or Windows environment). Depending on the size of the different input datasets we recommend running the workflow on a suitable machine (in our setting we use: 16 CPU, 64GB Memory) | Any manufacturer | 16 CPU, 64GB Memory | Large Memory is only required for the processing of the raw single cell data. After preprocessing the later analysis steps can also be performed on regular desktop or laptop computers |
| git | NA | NA | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git |
| GitHub | GitHub | NA | https://github.com/heiniglab/mofa_workflow |
Referências
- Lähnemann, D., et al. Eleven grand challenges in single-cell data science. Genome Biol. 21 (1), 31(2020).
- Colomé-Tatché, M., Theis, F. J. Statistical single cell multi-omics integration. Curr Opin Syst Biol. 7, 54-59 (2018).
- Hawe, J., Theis, F., Heinig, M. Inferring interaction networks from multi-omics data. Front Genet. 10, 535(2019).
- Hawe, J. S., et al. Network reconstruction for trans acting genetic loci using multi-omics data and prior information. Genome Med. 14 (1), 125(2022).
- Koh, H. W. L., Fermin, D., Vogel, C., Choi, K. P., Ewing, R. M., Choi, H. iOmicsPASS: network-based integration of multiomics data for predictive subnetwork discovery. NPJ Syst Biol Appl. 5, 22(2019).
- Ogris, C., Hu, Y., Arloth, J., Müller, N. S. Versatile knowledge guided network inference method for prioritizing key regulatory factors in multi-omics data. Sci Rep. 11, 6806(2021).
- Lee, C., vander Schaar, M. A variational information bottleneck approach to multi-omics data integration. Proceedings of The 24th International Conference on Artificial Intelligence and Statistics. 130, 1513-1521 (2021).
- Singh, A., et al. DIABLO: an integrative approach for identifying key molecular drivers from multi-omics assays. Bioinformatics. 35 (17), 3055-3062 (2019).
- Argelaguet, R., et al. Multi-omics factor analysis-a framework for unsupervised integration of multi-omics data sets. Mol Syst Biol. 14 (6), e8124(2018).
- Cantini, L., et al. Benchmarking joint multi-omics dimensionality reduction approaches for the study of cancer. Nature Commun. 12 (1), 124(2021).
- Pekayvaz, K., et al. Multiomic analyses uncover immunological signatures in acute and chronic coronary syndromes. Nature Medicine. 30 (6), 1696-1710 (2024).
- Swirski, F. K., Nahrendorf, M. Cardioimmunology: the immune system in cardiac homeostasis and disease. Nat Rev Immunol. 18 (12), 733-744 (2018).
- Janjic, A., et al. Prime-seq, efficient and powerful bulk RNA sequencing. Genome Biol. 23 (1), 88(2022).
- Wolf, F. A., Angerer, P., Theis, F. J. SCANPY: large-scale single-cell gene expression data analysis. Genome Biol. 19 (1), 15(2018).
- Cao, Y., et al. Integrated analysis of multimodal single-cell data with structural similarity. Nucleic Acids Res. 50 (21), e121(2022).
- Get Started - JupyterLab 4.1.0a4 documentation. , Available from: https://jupyterlab.readthedocs.io/en/latest/getting_started/overview.html (2024).
- MOFA2: training a model in R. , Available from: https://raw.githack.com/bioFAM/MOFA2_tutorials/master/R_tutorials/getting_started_R.html (2020).
- De Silva, D., et al. Robust T cell activation requires an eIF3-driven burst in T cell receptor translation. eLife. 10, e74272(2021).
- Li, G., Liang, X., Lotze, M. HMGB1: The central cytokine for all lymphoid cells. Front Immunol. 4, 68(2013).
- Jassal, B., et al. The reactome pathway knowledgebase. Nucleic Acids Res. 48 (D1), D498-D503 (2020).
- Argelaguet, R., et al. MOFA+: a statistical framework for comprehensive integration of multimodal single-cell data. Genome Biol. 21 (1), 111(2020).
- Velten, B., et al. Identifying temporal and spatial patterns of variation from multimodal data using MEFISTO. Nat Methods. 19 (2), 179-186 (2022).
- Qoku, A., Buettner, F. Encoding domain knowledge in multi-view latent variable models: A Bayesian approach with structured sparsity. Proceedings of The 26th International Conference on Artificial Intelligence and Statistics. 206, 11545-11562 (2022).
- Multi-Omics Factor Analysis. MOFA. , Available from: https://biofam.github.io/MOFA2/ (2024).
- Mitchel, J., et al. Tensor decomposition reveals coordinated multicellular patterns of transcriptional variation that distinguish and stratify disease individuals. bioRxiv. , (2023).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados