È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Applicazione dell'analisi fattoriale multi-omica non supervisionata per scoprire modelli di variazione e processi molecolari legati alle malattie cardiovascolari
In questo articolo
Riepilogo
Presentiamo un flusso di lavoro flessibile ed estendibile basato su Jupyter-lab per l'analisi non supervisionata di set di dati multi-omici complessi che combina diverse fasi di pre-elaborazione, stima del modello di analisi fattoriale multi-omica e diverse analisi a valle.
Abstract
I meccanismi della malattia sono solitamente complessi e governati dall'interazione di diversi processi molecolari distinti. I set di dati complessi e multidimensionali sono una risorsa preziosa per generare maggiori informazioni su tali processi, ma l'analisi di tali set di dati può essere impegnativa a causa dell'elevata dimensionalità risultante, ad esempio, da diverse condizioni di malattia, punti temporali e aspetti omici che catturano il processo a diverse risoluzioni.
Qui, mostriamo un approccio per analizzare ed esplorare un set di dati multiomici così complesso in modo non supervisionato applicando l'analisi fattoriale multi-omica (MOFA) a un set di dati generato da campioni di sangue che catturano la risposta immunitaria nelle sindromi coronariche acute e croniche. Il set di dati è costituito da diversi saggi a diverse risoluzioni, tra cui dati di citochine a livello di campione, proteomica plasmatica e prime-seq dei neutrofili e dati di RNA-seq a singola cellula (scRNA-seq). Un'ulteriore complessità è aggiunta dalla misurazione di diversi punti temporali per paziente e di diversi sottogruppi di pazienti.
Il flusso di lavoro dell'analisi delinea come integrare e analizzare i dati in diverse fasi: (1) Pre-elaborazione e armonizzazione dei dati, (2) Stima del modello MOFA, (3) Analisi a valle. Il passaggio 1 illustra come elaborare le caratteristiche dei diversi tipi di dati, filtrare le funzionalità di bassa qualità e normalizzarle per armonizzare le loro distribuzioni per ulteriori analisi. Il passaggio 2 mostra come applicare il modello MOFA ed esplorare le principali fonti di varianza all'interno del set di dati in tutte le omiche e le funzionalità. La fase 3 presenta diverse strategie per l'analisi a valle dei pattern catturati, collegandoli alle condizioni patologiche e ai potenziali processi molecolari che governano tali condizioni.
Nel complesso, presentiamo un flusso di lavoro per l'esplorazione non supervisionata dei dati di set di dati multi-omici complessi per consentire l'identificazione dei principali assi di variazione composti da diverse caratteristiche molecolari che possono essere applicate anche ad altri contesti e set di dati multi-omici (inclusi altri saggi presentati nel caso d'uso esemplare).
Introduzione
I meccanismi della malattia sono solitamente complessi e governati dall'interazione di diversi processi molecolari distinti. Decifrare i complessi meccanismi molecolari che portano a malattie specifiche o governano l'evoluzione di una malattia è un compito di grande rilevanza medica in quanto potrebbe rivelare nuove intuizioni per la comprensione e il trattamento delle malattie.
I recenti progressi tecnologici consentono di misurare questi processi a una risoluzione più elevata (ad esempio, a livello di singola cellula) e a vari strati biologici (ad esempio, DNA, mRNA, accessibilità della cromatina, metilazione del DNA, proteomica) allo stesso tempo. Ciò porta alla crescente generazione di grandi set di dati biologici multidimensionali, che possono essere analizzati congiuntamente per generare maggiori informazioni sui processi sottostanti. Allo stesso tempo, combinare e analizzare le diverse fonti di dati in modo biologicamente significativo rimane un compito impegnativo1.
I diversi limiti tecnologici, i rumori e le gamme di variabilità tra le diverse omiche rappresentano una sfida. Ad esempio, i dati di sequenziamento dell'RNA a singola cellula (scRNA-seq) sono molto scarsi e spesso influenzati da grandi effetti tecnici o batch. Inoltre, lo spazio delle caratteristiche è spesso molto ampio, e varia da diverse migliaia di geni o proteine misurati, mentre le dimensioni del campione sono limitate. Ciò è ulteriormente complicato da disegni complessi, che potrebbero includere diverse condizioni patologiche, fattori confondenti, punti temporali e risoluzioni. Ad esempio, nel caso d'uso presentato, erano disponibili diversi tipi di dati a livello di singola cella o di campione (bulk). Oltre a ciò, i dati potrebbero essere incompleti e non tutte le misurazioni potrebbero essere disponibili per tutti i soggetti analizzati.
A causa di queste sfide, le diverse omiche e le caratteristiche incluse vengono spesso analizzate solo separatamente2 anche se l'esecuzione di un'analisi integrata non solo non può fornire un quadro completo del processo, ma i rumori biologici e tecnici di un'omica potrebbero anche essere compensati da altre omiche 3,4. Sono stati proposti diversi metodi per eseguire un'analisi integrata di dati multi-omici, inclusi metodi bayesiani, metodi basati su reti 5,6, deep learning multimodale7 e metodi di riduzione della dimensionalità tramite fattorizzazione matriciale 8,9. Per quest'ultimo, i risultati di un ampio studio di benchmarking10 hanno dimostrato che il metodo MOFA9 (analisi fattoriale multi-omica) è uno degli strumenti più adatti quando i dati devono essere collegati alle annotazioni cliniche.
Soprattutto in contesti complessi, i metodi di fattorizzazione di matrici non supervisionati sono un approccio utile per ridurre la complessità ed estrarre segnali condivisi e complementari da diverse fonti di dati e caratteristiche. Scomponendo lo spazio complesso in rappresentazioni latenti di rango inferiore, le principali fonti di varianza all'interno dei dati possono essere rapidamente esplorate e collegate a covariate note. Nel caso in cui lo stesso modello di variazione sia condiviso tra più caratteristiche (ad esempio, geni o proteine), questo può essere aggregato a pochi fattori mentre il rumore è ridotto. La regolarizzazione può essere utilizzata per aumentare la sparsità dei coefficienti del modello, il che rende l'approccio adatto in ambienti in cui lo spazio delle caratteristiche è ampio mentre il numero di campioni è limitatoa 9.
Questo protocollo presenta un flusso di lavoro di analisi flessibile che utilizza il modello MOFA per mostrare come esplorare rapidamente un complesso set di dati multi-omici e distillare i principali modelli di variazione che caratterizzano questo set di dati. Il flusso di lavoro è costituito da tre passaggi principali. Nella prima fase, la pre-elaborazione e l'armonizzazione dei dati, vengono presentate diverse strategie per la pre-elaborazione dei dati basate su diversi tipi di dati di input (scRNA-seq, proteomica, citochine, dati clinici). Il protocollo elabora il modo in cui elaborare le caratteristiche dei diversi set di dati di input, filtrare le funzionalità di bassa qualità e normalizzarle per armonizzare le loro distribuzioni. Mostriamo anche come queste decisioni di pre-elaborazione potrebbero influenzare i risultati a valle. Nella seconda fase, il modello MOFA viene applicato ai dati e la scomposizione della varianza risultante può essere utilizzata per valutare l'integrazione dei diversi set di dati. Il terzo passo mostra come collegare i fattori catturati alle covariate e scoprire i programmi molecolari che definiscono tali fattori. Con il flusso di lavoro presentato, siamo stati in grado di estrarre diversi fattori latenti legati alle covariate cliniche in un set di dati di pazienti affetti da sindromi coronariche e di identificare potenziali programmi immunitari multicellulari sottostanti da un precedente progetto11. Utilizzeremo questo set di dati qui, ma il protocollo può essere facilmente applicato ad altri contesti, comprese altre omici.
Il set di dati è costituito da campioni di pazienti con sindromi coronariche croniche stabili (CCS), sindromi coronariche acute (ACS) e un gruppo di controllo con coronarie sane (non CCS) (Figura 1). La SCA è causata dalla rottura della placca in CCS preesistente, che porta a un'interruzione acuta del flusso sanguigno al miocardio e a una conseguente lesione ischemica del cuore. Questa lesione provoca una risposta infiammatoria da parte del sistema immunitario seguita da una fase riparativa, che dura fino a diversi giorni dopo l'evento acuto12. Per essere in grado di caratterizzare questa risposta immunitaria per i pazienti con SCA, sono stati prelevati campioni di sangue in quattro diversi punti temporali: acuta (TP1); dopo ricanalizzazione (14 [± 8] h) (TP2); 60 [± 12] h dopo (TP3); prima della dimissione (6,5 [±1,5] giorni) (TP4) (Figura 1A). Per la CCS e i pazienti con coronarie sane, era disponibile un solo punto temporale- (TP0). Per tutti i pazienti e i punti temporali sono stati misurati diversi saggi basati sui campioni di sangue: marcatori clinici di infiammazione (Creatina-chinasi (CK), CK-MB, Tropolina, proteina C-reattiva (CRP)), scRNA-seq di cellule mononucleate del sangue periferico (PBMC), analisi delle citochine, proteomica plasmatica e dati prime-seq13 di neutrofili.
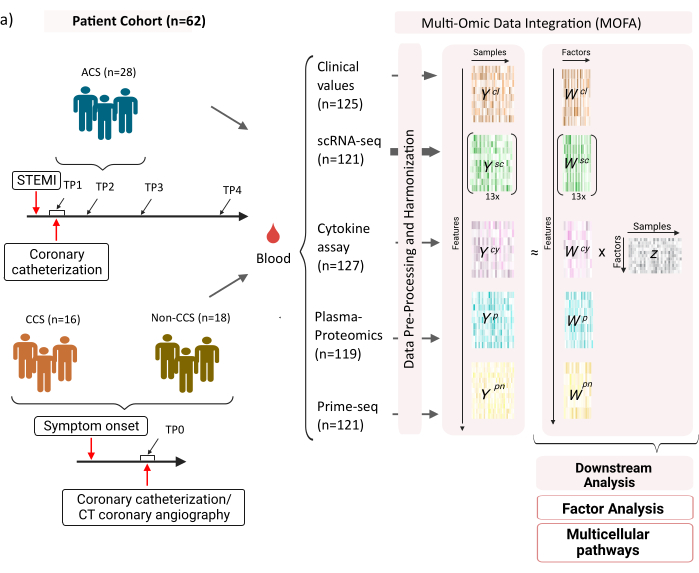
Figura 1: Set di dati di input multi-omici sull'infarto del miocardio. Set di dati di input: i dati analizzati includono campioni di sangue di pazienti (n = 62) con sindrome coronarica acuta (SCA), sindromi coronariche croniche (CCS) e pazienti con coronarie sane (non CCS). Per i pazienti con SCA sono stati inclusi campioni di sangue in quattro diversi punti temporali (TP1-4), per i pazienti con CCS e non CCS in un unico punto temporale (TP0). Ogni combinazione di pazienti e punti temporali viene trattata come un campione separato nell'analisi. Sui campioni sono stati misurati diversi test omici: esami del sangue clinici (n = 125), scRNA-seq (n = 121), proteomica del plasma (n = 119), test delle citochine (n = 127) e prime-seq dei neutrofili (n = 121). Successivamente, il protocollo descritto è stato applicato per integrare i dati in tutte le omiche ed esplorarli utilizzando il modello MOFA e ulteriori analisi a valle (analisi fattoriale, arricchimento del percorso). Clicca qui per visualizzare una versione più grande di questa figura.
Come input per il flusso di lavoro come presentato qui, prendiamo i conteggi grezzi dai dati scRNA-seq dopo l'elaborazione con cellranger e il controllo di qualità (QC) come, ad esempio, delineato nel tutorial di pre-elaborazione scanpy14 . Per l'annotazione del tipo di cella, abbiamo utilizzato la pipeline automatizzata Azimuth15 . I conteggi vengono quindi aggregati a livello di campione per ciascun tipo di cellula prendendo la media di tutte le cellule per ciascun campione e tipo di cellula (aggregazione pseudobulk). La proteomica del plasma è inclusa come intensità normalizzate e mediane, e per i neutrofili, prendiamo la conta degli esoni dell'identificatore molecolare unico umi (UMI) dal prime-seq. Sulle citochine e sui valori clinici, non è stato applicato alcun preprocessing precedente. Ulteriori dettagli sulla generazione di dati (sperimentali) sono descritti nel corrispondente manoscritto11. Poiché i risultati qui presentati si basano sull'utilizzo dell'annotazione automatica dell'azimut per i tipi di cellule nei dati scRNA-seq rispetto alla strategia basata su marcatori utilizzata nella pubblicazione di riferimento, i risultati qui presentati sono simili ma non esattamente gli stessi presentati nella pubblicazione. Nel manoscritto si può dimostrare che la strategia di annotazione del tipo di cellula non cambia i principali modelli e interpretazioni biologiche dell'analisi, ma piccoli cambiamenti nei valori esatti risultanti dal modello possono variare. Nel complesso, i dati di input erano un complesso set di dati multidimensionali che comprendeva diversi punti temporali e livelli di misurazione (singole cellule vs. bulk) di oltre 10.000 diverse caratteristiche (geni, proteine, valori clinici). Una rigorosa strategia di pre-elaborazione e armonizzazione dei dati seguita dall'analisi MOFA si è dimostrata uno strumento utile e rapido per esplorare i dati ed estrarre il programma immunitario pertinente. Ogni punto temporale e combinazione di pazienti viene trattato come un campione indipendente nell'analisi MOFA. Ogni tipo di dati e tipo di cella è considerato una vista separata nell'analisi MOFA.
Questo protocollo fornisce istruzioni per la preparazione dei dati di input per il flusso di lavoro, l'esecuzione dei diversi passaggi del flusso di lavoro, la personalizzazione delle configurazioni, l'interpretazione delle cifre risultanti e la regolazione iterativa delle configurazioni in base alle interpretazioni. Una panoramica delle diverse fasi del protocollo, dei set di dati di input richiesti in ogni fase e delle cifre e dei set di dati risultanti è fornita dalla panoramica del flusso di lavoro tecnico (Figura 2).
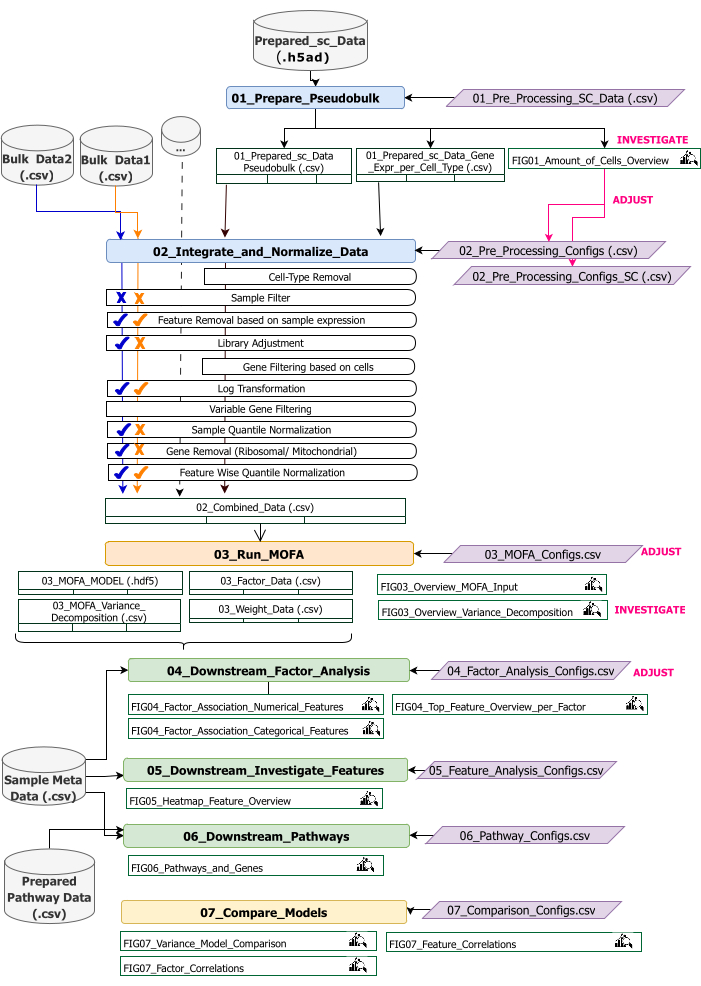
Figura 2: Panoramica del flusso di lavoro tecnico. Cenni sul flusso di lavoro per l'analisi del dataset multi-omico. I diversi elementi sono evidenziati da colori e simboli diversi. I Jupyter Notebook appartenenti alla fase di pre-elaborazione e armonizzazione dei dati (1) sono colorati in blu. I Jupyter Notebook appartenenti al passaggio 'MOFA Model' (2) sono colorati in arancione. I Jupyter Notebook appartenenti alla fase "Analisi a valle" (3) sono colorati in verde. Un Jupyter Notebook da utilizzare per il confronto dei risultati è colorato in giallo. I file di configurazione in cui è possibile modificare i parametri per l'esecuzione del flusso di lavoro sono evidenziati in viola. I set di dati di input necessari per eseguire il flusso di lavoro sono indicati dal simbolo del set di dati ed evidenziati in grigio. Tutti gli output delle figure generati durante l'esecuzione del flusso di lavoro sono indicati dal simbolo della lente di ingrandimento. I set di dati generati durante l'esecuzione del flusso di lavoro sono indicati come tabelle. In generale, il flusso di lavoro viene eseguito in sequenza: (1) La pre-elaborazione e l'armonizzazione dei dati consiste in due fasi: prima generazione di una tabella pseudobulk basata sui dati di input scRNA-seq (01_Prepare_Pseudobulk) e successiva integrazione e normalizzazione di questi dati insieme a tutti gli altri input a livello di campione (bulk) (02_Integrate_and_Normalize_Data). All'interno di questo passaggio, tramite i file di configurazione, è possibile configurare separatamente per ciascun set di dati quali dei passaggi di pre-elaborazione e normalizzazione indicati (ad esempio, il filtro di esempio) devono essere applicati. (2) «Modello MOFA»: esegue il modello MOFA sull'input generato nella prima fase con le configurazioni specificate nel file di configurazione (03_MOFA_configs.csv) (3) «Analisi a valle»: consiste in tre diversi quaderni che possono essere eseguiti indipendentemente l'uno dall'altro per generare informazioni sui risultati MOFA generati e associarli ai metadati campione (covariate) forniti come input tramite il file «Meta Data.csv campione». (4) «Confronto tra modelli»: è una piccola fase separata che può essere utilizzata per confrontare diversi modelli generati nella fase 2. Clicca qui per visualizzare una versione più grande di questa figura.
Il flusso di lavoro è costituito da diversi Jupyter Notebook scritti in R e Python (la conoscenza del linguaggio R e Python non è necessaria per eseguire il flusso di lavoro, ma potrebbe essere utile nel caso in cui vengano visualizzati errori). In vari passaggi del protocollo, i parametri vengono modificati tramite file di configurazione (file '.csv' contenenti il suffisso '_Configs' nel nome). All'interno del protocollo, delineiamo solo i parametri che devono essere modificati a partire dalla configurazione predefinita.
È possibile modificare anche diversi altri parametri, ad esempio per personalizzare la pre-elaborazione. Una documentazione di questi parametri e spiegazioni è fornita nel file 'Documentation_Config_Parameter', che è incluso nel repository scaricato.
Protocollo
1. Preparativi: configurazione tecnica e installazione
NOTA: Per eseguire questo programma, avere wget, git e Apptainer preinstallati sul dispositivo. Una guida per l'installazione di Apptainer su diversi sistemi (Linux, Windows, Mac) è disponibile qui: https://apptainer.org/docs/admin/main/installation.html. Le informazioni sull'installazione su git possono essere trovate qui: https://git-scm.com/book/en/v2/Getting-Started-Installing-Git. A seconda delle dimensioni dei diversi set di dati di input, si consiglia di eseguire il flusso di lavoro su un computer adatto (16 CPU, 64 GB di memoria). Un smoke test con i dati di esempio forniti può essere eseguito sul computer locale. Le istruzioni e gli output attesi dall'esecuzione del protocollo sui dati di esempio sono forniti nel file supplementare 1. Fare riferimento al file video supplementare 1 per i passaggi importanti del protocollo che vengono eseguiti sul set di dati descritto sopra.
- Apri la console e scegli o crea una cartella in cui verranno memorizzati tutto il codice di analisi e gli output. Navigare nella cartella digitando il comando: cd path_to_folder nel terminale.
- Scarica o clona il repository di codice da Github (https://github.com/heiniglab/mofa_workflow) o digitando git clone https://github.com/heiniglab/mofa_workflow.git nella finestra del terminale.
- Scarica l'immagine che contiene tutte le installazioni richieste da Zenodo digitando wget https://zenodo.org/records/11192947/files/mofa_image.sif nella finestra del terminale.
- Genera una cartella in cui verranno memorizzati tutti i dati dei risultati digitando mkdir results nella finestra del terminale.
- Genera una cartella in cui verranno aggiunti tutti i dati di input da utilizzare nell'analisi digitando mkdir input_data nella finestra del terminale.
- Esegui il container che avvierà una sessione JupyterLab digitando il seguente comando nel terminale: apptainer run mofa_image.sif. Copiare l'URL restituito dal comando nel browser, che aprirà una sessione di Jupyter-lab (ulteriori informazioni su Jupyter-lab sono disponibili nella documentazione del software16).
NOTA: quando il flusso di lavoro viene eseguito localmente su un laptop, si consiglia di utilizzare invece il comando apptainer exec mofa_image.sif jupyter-lab , che restituirà direttamente un indirizzo host locale. Nel caso in cui il container venga eseguito all'interno di un ambiente di calcolo cluster, potrebbe essere necessario impostare il port forwarding, che può essere fatto tramite ssh.
2. Inizializzazione e preparazione dei dati
- Nella sessione Jupyter-Lab, utilizzare il menu di navigazione sul lato sinistro. Passare alla cartella input_data facendo doppio clic su input_data.
- Copia tutti i dataset che verranno utilizzati come input per l'analisi nella directory input_data utilizzando il Drag&Drop. Trascinare il file dalla cartella in cui si trova attualmente e rilasciarlo nella sessione Jupyter-lab nell'area sotto la cartella input_data.
NOTA: tutti i set di dati devono essere in formato .csv o .h5ad (nel caso di dati a cella singola). Tutti i file .csv devono contenere una colonna sample_id corrispondente (gli ID identici devono essere utilizzati in tutti i set di dati). Tutte le altre colonne verranno utilizzate come funzionalità. All'interno del file h5ad-, l'annotazione della cella deve contenere due identificatori che specificano il sample_id e il cluster_id. Questi verranno utilizzati per l'aggregazione e la corrispondenza. I set di dati omici in altri formati devono essere convertiti nel formato .csv specificato prima dell'uso (Figura 3). I set di dati scRNA-seq forniti in formato .h5seurat potrebbero essere convertiti in .h5ad eseguendo il Jupyter-notebook: 00_Data_Conversion.ipynb. - Passare alla cartella delle configurazioni facendo clic sul simbolo della cartella e quindi facendo doppio clic sulle cartelle mofa_workflow, script e configurazioni. All'interno della cartella, apri il file Data_configs.csv facendo doppio clic su di esso.
- Nella colonna del valore , aggiungere i percorsi delle cartelle delle cartelle input_data (data_path) e dei risultati (result_path). Aggiungere un nome che verrà aggiunto come estensione di file a tutti i file salvati nella colonna dei valori per il configuration_name (questo protocollo utilizzato MI_v1 [Infarto del miocardio versione1]) (Figura 4).
- Salva le modifiche cliccando su File > Salva file CSV nel menu in alto.
- Utilizzare il menu di navigazione sul lato sinistro per accedere alla cartella degli script facendo clic su script. Aprire il taccuino di inizializzazione facendo doppio clic su 00_Configuration_Update.ipynb. Eseguire lo script facendo clic sul pulsante Riavvia il kernel ed esegui tutte le celle in alto e facendo clic su Riavvia nel pop-up (Figura 5).
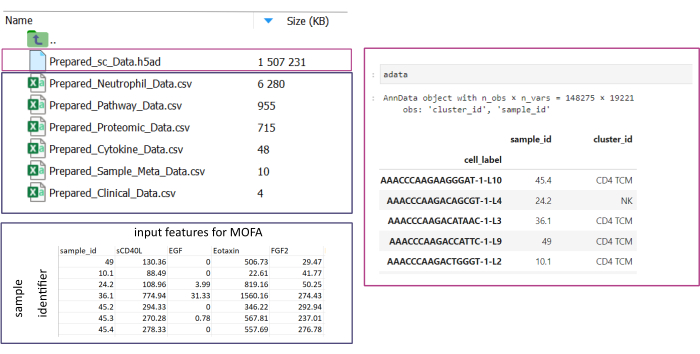
Figura 3: Inserimento e configurazione dei dati. Per l'esecuzione del flusso di lavoro, tutti i dati devono essere archiviati in una cartella input_data specificata. Per ogni set di dati di input deve essere fornito un file separato. I dati di una singola cella devono essere forniti come .h5ad contenente l'annotazione della cella sulla cluster_id (risultante, ad esempio, dai precedenti passaggi di annotazione del tipo di cella) e una colonna sample_id (che identifica in modo univoco ogni campione separato che deve essere analizzato). Tutti gli altri set di dati di input devono essere forniti in formato ".csv", compresa una colonna che specifichi il sample_id (corrispondente alla colonna corrispondente dei dati a cella singola) e le caratteristiche da utilizzare nell'analisi MOFA in tutte le altre colonne. Clicca qui per visualizzare una versione più grande di questa figura.
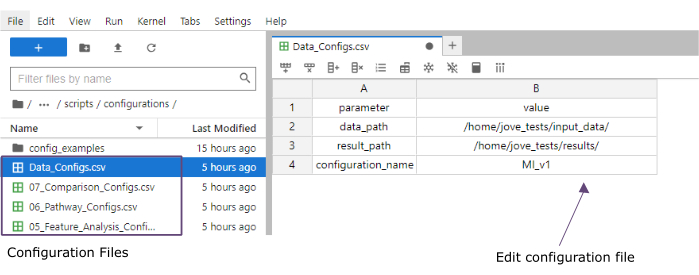
Figura 4: File di configurazione di Jupyter-lab. Durante l'esecuzione del flusso di lavoro, le modifiche ai parametri (ad esempio, la regolazione delle opzioni di filtraggio, ecc.) vengono specificate tramite i file di configurazione ".csv". All'interno del repository clonato, sono inclusi i file di configurazione predefiniti per ogni passaggio. Possono essere modificati direttamente nella console jupyter-lab, in modo simile a un foglio di calcolo. Clicca qui per visualizzare una versione più grande di questa figura.
Figura 5: script Jupyter-notebooks. Il flusso di lavoro completo è costituito da una serie di notebook Jupyter che verranno eseguiti in sequenza dopo la modifica dei file di configurazione corrispondenti. Facendo doppio clic sul taccuino Jupyter sul lato sinistro, il file corrispondente verrà aperto sul lato destro. L'esecuzione completa del file può essere avviata con il pulsante evidenziato in alto. Clicca qui per visualizzare una versione più grande di questa figura.
3. Pre-elaborazione e armonizzazione dei dati
- Pre-elaborazione: converti i dati sc in pseudobulk.
NOTA: questo passaggio deve essere eseguito solo se nell'analisi vengono utilizzati dati a cella singola.- Utilizzare il menu di navigazione sul lato sinistro per accedere alla cartella delle configurazioni facendo doppio clic su configurazione. Aprire il file 01_Preprocessing_SC_Data.csv facendo doppio clic. Controllare i valori compilati automaticamente nel file e, se necessario, regolare i valori nella colonna data_name in modo che corrispondano ai nomi dei set di dati a cella singola nella cartella input_data che verrà utilizzata per l'analisi.
NOTA: Per impostazione predefinita, tutti i nomi dei file .h5ad nella cartella dei dati di input verranno aggiunti al file di configurazione nello script di inizializzazione. Se alcuni dei set di dati non devono essere utilizzati per l'analisi, possono essere rimossi qui. - Salva le modifiche apportate cliccando su File > Salva file CSV nel menu in alto.
- Utilizzare il menu di navigazione sul lato sinistro per accedere alla cartella degli script facendo clic su script. Apri il notebook 01_Prepare_Pseudobulk.ipynb facendo doppio clic su di esso. Eseguire lo script facendo clic sul pulsante Riavvia il kernel ed esegui tutte le celle in alto e facendo clic su Riavvia nel pop-up.
- Utilizzare il menu di navigazione sul lato sinistro per accedere alla cartella delle figure facendo doppio clic prima sulle figure e poi su 01_figures. Apri il FIG01_Amount_of_Cells_overview del grafico appena generato facendo doppio clic su di esso.
NOTA: l'esecuzione del notebook potrebbe richiedere alcuni minuti. Quando il blocco appunti è stato eseguito correttamente, viene visualizzato un popup e il file FIG01_Amount_of_Cells_Overview viene aggiornato dal blocco note o generato nuovamente. La colonna Ultima modifica può indicare quando il file è stato generato per valutare se si tratta di un file nuovo o vecchio. - Esaminare il grafico per identificare i cluster di tipo cellulare con un numero molto basso di cellule per campione. Annotare i nomi di tali cluster_ids per escluderli nei passaggi successivi (Figura 6).
- Utilizzare il menu di navigazione sul lato sinistro per tornare alla cartella delle configurazioni facendo clic su ... e quindi fare doppio clic su Configurazioni. Apri il file 02_Preprocessing_Configs_SC.csv facendo doppio clic su di esso.
- Controllare i valori nelle colonne configuration_name e data_name e modificarli se necessario.
NOTA: all'interno dello script di inizializzazione, tali valori sono precompilati con tutti i nomi dei file .h5ad nella cartella dei dati di input e il valore configuration_name impostato in precedenza all'interno del file Data_Configs.csv . Nel caso in cui i file debbano essere esclusi dall'analisi o debba essere utilizzata un'altra estensione per i nomi dei file, questo può essere regolato qui. - Regola il valore nella colonna cell_type_exclusion e aggiungi tutti i cluster_id che sono stati identificati per escludere nel passaggio precedente separati da ','.
- Salva le modifiche cliccando su File > Salva file CSV nella barra di navigazione in alto.
- Utilizzare il menu di navigazione sul lato sinistro per accedere alla cartella delle configurazioni facendo doppio clic su configurazione. Aprire il file 01_Preprocessing_SC_Data.csv facendo doppio clic. Controllare i valori compilati automaticamente nel file e, se necessario, regolare i valori nella colonna data_name in modo che corrispondano ai nomi dei set di dati a cella singola nella cartella input_data che verrà utilizzata per l'analisi.
- Pre-elaborazione - Armonizza e integra altre fonti di dati omici.
- Aprire il file 02_Preprocessing_Configs.csv facendo doppio clic su di esso e regolare la configurazione di pre-elaborazione per ciascuno dei set di dati che verranno inclusi e vengono memorizzati nella cartella data_input (una riga per set di dati).
- Controllare i valori nelle colonne configuration_name e data_name e modificarli se necessario.
- Regolare di conseguenza gli altri parametri nelle colonne, a seconda delle fasi di pre-elaborazione da applicare.
NOTA: i valori predefiniti vengono aggiunti per ogni set di dati trovato all'interno della cartella input_dataset, ma non sono specifici per i singoli tipi di dati dei dati. Pertanto, saranno necessari degli aggiustamenti. Una documentazione dettagliata dei parametri è fornita nel file Documentation_Config_Parameter.doc . - Salva le modifiche facendo clic su File > Salva file CSV.
- Utilizzare il menu di navigazione sul lato sinistro per accedere alla cartella degli script facendo clic su script. Aprire il notebook 02_Integrate_and_Normalized_Data_Sources.ipynb facendo doppio clic su di esso. Eseguire lo script facendo clic sul pulsante Riavvia il kernel ed esegui tutte le celle in alto e facendo clic su Riavvia nel pop-up.
- Utilizzare il menu di navigazione sul lato sinistro per accedere alla cartella 02_results generata facendo clic sul simbolo della cartella e quindi facendo doppio clic su risultati e 02_results. Verificare se include il file 02_Combined_data_'configuration_name'_Integrated.csv contenente il file di input dei dati pre-elaborato combinato.
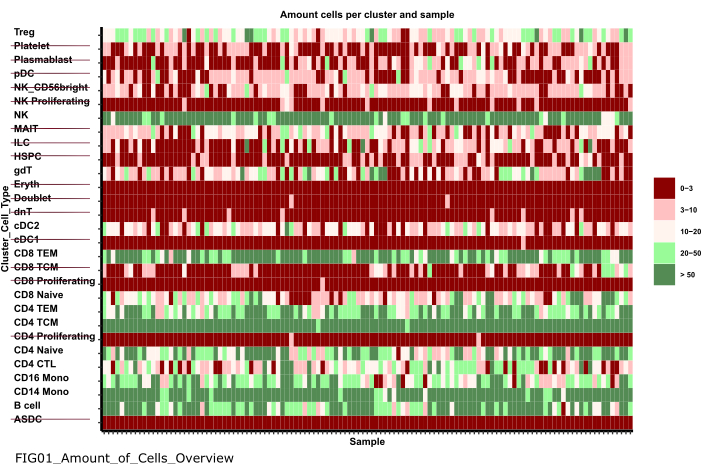
Figura 6: Pre-elaborazione e armonizzazione dei dati. Un output della fase '01_Prepare_Pseudobulk' è il grafico 'Fig01_Amount_of_Cells_Overview'. Qui, per ogni cluster_id (asse y che indica il tipo di cella dai precedenti passaggi di annotazione del tipo di cella), viene fornito il numero di celle per campione ("sample_id"). All'interno dei risultati presentati, i tipi di cellule con una bassa quantità di cellule per campione sono esclusi dall'analisi successiva (indicata dal barrato). Clicca qui per visualizzare una versione più grande di questa figura.
4. Esecuzione di MOFA
- In Jupyter-Lab, utilizzare il menu di navigazione sul lato sinistro per navigare nella cartella delle configurazioni facendo clic sul simbolo della cartella e quindi facendo doppio clic su mofa_workflow, quindi facendo doppio clic su script e configurazioni. Apri il file 03_MOFA_Configs.csv facendo doppio clic su di esso.
- Controllare le voci per le colonne configuration_name e mofa_result_name e regolare le voci se è necessario utilizzare nomi alternativi.
NOTA: Il mofa_result_name verrà aggiunto come estensione di file a tutti i file dei risultati generati in base al MOFA. Questo può essere diverso dal valore configuration_name poiché diverse configurazioni MOFA potrebbero essere eseguite con gli stessi dati di input (questo protocollo utilizza MI_v1_MOFA). - Immettere la quantità di fattori che devono essere stimati nel modello MOFA (colonna amount_of_factors ) e definire se applicare la ponderazione e la scala (colonne weighting_of_views e scale_views ) regolando i valori nel file.
- Salva le modifiche facendo clic su File > Salva file CSV.
- Utilizzare il menu di navigazione sul lato sinistro per accedere alla cartella degli script facendo clic su "script". Aprire il notebook 03_Run_MOFA.ipynb facendo doppio clic sul file. Esegui lo script facendo clic sul pulsante Riavvia il kernel ed esegui tutte le celle in alto e facendo clic su Riavvia nel pop-up.
- Passare alla cartella 03_figures facendo doppio clic sulle figure e quindi 03_figures. Aprire il mofa_result_name FIG03_Overview_Variance_Decomposition_ del grafico generato e analizzare il risultato del modello (Figura 7A).
- Utilizzare il menu di navigazione sul lato sinistro per accedere alla cartella 03_results generata facendo clic sul simbolo della cartella e quindi facendo doppio clic su risultati e 03_results. Verificare se include il file del valore del fattore di esempio 03_Factor_Data_'mofa_result_name'.csv e il file del peso del fattore di funzione 03_Weight_Data_'mofa_result_name'.csv.
5. Analisi a valle
- Interpretazione dei fattori.
- Utilizzare il menu di navigazione sul lato sinistro per accedere alla cartella input_data facendo clic sul simbolo della cartella , quindi facendo doppio clic su input_data.
- Preparare un file .csv (Prepared_Sample_Meta_Data.csv) che contenga tutti i metadati (covariate) dei campioni che verranno analizzati in associazione con i fattori generati. Copiare il file nella cartella input_data utilizzando il Drag&Drop rilasciando il file nella panoramica della cartella input_data .
NOTA: Deve contenere la colonna sample_id per abbinarla ai dati utilizzati in precedenza e ulteriori colonne per ogni funzionalità che deve essere analizzata. - In Jupyter-Lab, utilizzare il menu di navigazione a sinistra per tornare alla cartella delle configurazioni facendo clic sul simbolo della cartella e quindi facendo doppio clic su mofa_workflow, seguito da script e configurazione. Apri il file 04_Factor_Analysis.csv facendo doppio clic su di esso.
- Verificare che le voci per il configuration_name e il mofa_result_name contengano i nomi della configurazione e i risultati MOFA che verranno analizzati nello script e modificarli se necessario.
- Nella colonna numeric_covariates aggiungere il nome di tutte le colonne numeriche nel file Prepared_Sample_Meta_Data.csv che verranno esaminate in relazione ai fattori MOFA separati da virgola (questo protocollo utilizza CRP,CK).
- Nella colonna categorical_covariates', aggiungi il nome di tutte le colonne categoriche nel file Prepared_Sample_Meta_Data.csv che verranno esaminate in relazione ai fattori MOFA separati da virgola (questo protocollo utilizza la misurazione).
- Salva le modifiche facendo clic su File > Salva file CSV.
- Utilizzare il menu di navigazione a sinistra per accedere alla cartella "script" facendo clic su script. Apri il notebook 04_Downstream_Factor_Analysis.ipynb facendo doppio clic su di esso. Esegui lo script facendo clic sul pulsante Riavvia kernel ed esegui tutte le celle in alto e facendo clic su Riavvia nel pop-up.
- Utilizzare il menu di navigazione a sinistra per navigare nella cartella 04_figures facendo doppio clic sulle figure e quindi 04_figures. Apri i grafici generati facendo doppio clic su di essi e indaga i fattori per modelli e associazioni interessanti: FIG04_Factor_Association_with_numeric_features_
'mofa_result_name.pdf (Figura 7B). FIG04_Factor_Association_
with_categorical_features_'mofa_result_name.pdf (Figura 7C). FIG04_Top_Feature_Overview_per_Factor _'mofa_result_name.pdf (Figura 8A).
- Analisi delle caratteristiche
- Utilizzare il menu di navigazione a sinistra per tornare alla cartella delle configurazioni facendo clic su ... e quindi fare doppio clic su Configurazioni. Apri il file 05_Feature_Analysis_Configs.csv facendo doppio clic su di esso.
- Verificare che le voci per le colonne configuration_name e mofa_result_name corrispondano ai nomi della configurazione e al risultato MOFA generato che verrà utilizzato per l'analisi a valle e regolarle se necessario.
- Nella colonna del fattore , aggiungere il fattore per il quale le funzioni principali verranno tracciate nello script successivo.
- Nella colonna faceting_variable, aggiungere il nome di una colonna categorica nel Prepared_Sample_Meta_Data.csv che verrà utilizzato per raggruppare i campioni nel grafico (questo protocollo utilizza la misurazione)
- Salva le modifiche facendo clic su File > Salva file CSV.
- Utilizzare il menu di navigazione a sinistra per accedere alla cartella degli script facendo clic su script. Apri il notebook 05_Downstream_Investigate_Features_Heatmap.ipynb facendo doppio clic su di esso. Eseguire lo script facendo clic sul pulsante Riavvia il kernel ed esegui tutte le celle in alto e facendo clic su Riavvia nel pop-up.
- Utilizzare il menu di navigazione a sinistra per navigare nella cartella 05_figures facendo doppio clic prima sulle figure e poi su 05_figures. Aprire ed esaminare il grafico generato FIG05_Heatmap_Feature_Overview__ 'mofa_result_name'.pdf facendo doppio clic sul file (Figura 8B).
NOTA: A seconda della quantità di funzioni che verranno mostrate nel grafico, potrebbe essere necessario regolare i parametri plot_width e plot_height all'interno del 05_Feature_Analysis_Configs.csv ed eseguire nuovamente lo script per assicurarsi che tutto si adatti al grafico.
- Analisi dei percorsi
- Utilizzare il menu di navigazione sul lato sinistro per accedere alla cartella input_data facendo clic sul simbolo della cartella , quindi facendo doppio clic su input_data.
- Preparare un file .csv (Prepared_Pathway_Data.csv) contenente un elenco di percorsi che verranno testati per l'arricchimento. Copiare il file nella cartella input_data utilizzando il Drag&Drop rilasciando il file nella panoramica della cartella input_data .
NOTA: Deve contenere tre colonne: ID (un identificatore univoco della via), gene (i geni dati dal loro nome genetico (SYMBOL) appartenenti alla via, una riga per gene), pathway_name (un nome/descrizione testuale delle vie). - Nella sessione Jupyter-Lab, utilizzare il menu di navigazione a sinistra per navigare nella cartella delle configurazioni facendo clic su ... e quindi fare doppio clic su Configurazioni. Apri il file 06_Pathway_Configs.csv facendo doppio clic su di esso.
- Controllare la voce per la colonna mofa_result_name e assicurarsi che corrisponda al nome del risultato MOFA generato che verrà utilizzato per il calcolo dell'arricchimento del percorso.
- Controllare la voce nella colonna dei tipi e rimuovere le voci all'interno della colonna dei tipi che non contengono funzionalità che corrispondono alla colonna del gene nel file Prepared_Pathway_Data.csv .
NOTA: per impostazione predefinita, tutte le diverse viste che sono state utilizzate all'interno del modello MOFA vengono aggiunte a questo file durante l'esecuzione del flusso di lavoro. Nel caso in cui ci siano viste che non contengono elementi che corrispondono ad almeno un percorso, devono essere rimosse; In caso contrario, l'esecuzione avrà esito negativo. Un esempio potrebbe essere il file del percorso che contiene solo annotazioni del percorso per i geni, ma c'è una vista che contiene i nomi delle proteine. - Salva le modifiche facendo clic su File > Salva file CSV.
- Utilizzare il menu di navigazione per accedere alla cartella degli script facendo clic su script. Apri il notebook 06_Downstream_Pathways.ipynb facendo doppio clic su di esso. Eseguire lo script facendo clic sul pulsante Riavvia il kernel ed esegui tutte le celle in alto e facendo clic su Riavvia nel pop-up.
- Utilizzare il menu di navigazione a sinistra per accedere alla cartella 06_figures facendo doppio clic sulle figure e poi 06_figures. Aprire il grafico generato FIG06_Pathways_and_Genes_ 'mofa_result_name facendo doppio clic su di esso e analizzare i percorsi visualizzati (Figura 8C).
NOTA: La modalità di selezione dei percorsi visualizzati può essere configurata tramite il file di configurazione. Per maggiori dettagli, fare riferimento alla documentazione dei parametri. - Utilizzare il menu di navigazione sul lato sinistro per accedere alla cartella 06_results generata facendo clic sul simbolo della cartella e quindi facendo doppio clic su risultati e 06_results. Controlla se include il file che include i percorsi arricchiti 06_Pathway_enrichment__'mofa_result_name".
6. Confronto tra diverse configurazioni e versioni (Figura supplementare 1, Figura supplementare 2, Figura supplementare 3, Figura supplementare 4)
- Per confrontare l'effetto dell'utilizzo di parametri/configurazioni diversi in tutto il flusso di lavoro, eseguire nuovamente le sezioni 3-5, modificando i parametri nei file di configurazione e utilizzando identificatori di configuration_name e mofa_result_name diversi.
NOTA: I nuovi risultati verranno memorizzati con questi nomi da utilizzare per confrontare diverse esecuzioni. - In Jupyter-Lab, utilizzare il menu di navigazione a sinistra per accedere alla cartella delle configurazioni . Apri il file 07_Comparison_Configs.csv facendo doppio clic su di esso.
- Nella colonna mofa_result_name , aggiungi i nomi di tutte le esecuzioni MOFA precedenti che verranno confrontate (una riga per nome/configurazione, ad es. MI_v1_MOFA, MI_v2_MOFA).
- Nella colonna compare_factors aggiungere i fattori che verranno confrontati tra i modelli. Per impostazione predefinita, è Fattore1,Fattore2,Fattore3. (Figura supplementare 2A).
NOTA: In questo script verranno confrontati i valori delle caratteristiche e dei fattori dei diversi modelli correlandoli. Questo funziona solo per i modelli basati sugli stessi campioni (indicati da sample_id) e sullo stesso set di funzionalità. Nel caso in cui i campioni o le funzionalità non corrispondano tra le versioni confrontate, verranno esclusi dal confronto. - Salva le modifiche facendo clic su File > Salva file CSV.
- Utilizzare il menu a sinistra per accedere alla cartella degli script facendo clic su script. Apri il notebook 07_Compare_Models.ipynb facendo doppio clic su di esso. Eseguire lo script facendo clic sul pulsante Riavvia il kernel ed esegui tutte le celle e facendo clic su Riavvia nel pop-up.
- Utilizzare il menu a sinistra per navigare nella cartella 06_figures facendo doppio clic prima sulle figure e poi su 06_figures. Apri i grafici generati facendo doppio clic sui file per analizzare la somiglianza delle diverse versioni:
FIG07_Variance_Model_Comparison.pdf (Figura supplementare 2B)
FIG07_Factor_Correlations.pdf
(Figura 2C supplementare)
FIG07_Feature_Correlations.pdf
(Figura supplementare 3C)
7. Estensione del flusso di lavoro: aggiunta di altri parametri e configurazioni
NOTA: Oltre ai parametri attualmente configurabili nei file di configurazione, potrebbero essere incluse altre regolazioni nel codice o altri parametri. Ad esempio, il modello MOFA stesso offre diversi altri parametri di addestramento17 che possono essere modificati direttamente nel codice o resi regolabili tramite i file di configurazione. La sezione successiva del protocollo delineerà un esempio di come eseguire questa operazione per ulteriori parametri di addestramento del modello MOFA. Per questa parte è richiesta la conoscenza della programmazione R.
- In Jupyter-Lab, utilizzare il menu di navigazione a sinistra per passare alla cartella degli script . Apri il notebook 03_Run_MOFA.ipynb facendo doppio clic su di esso.
- Fare clic sulla scheda Sommario sul lato sinistro, quindi accedere alla sottosezione 4.3 Impostare le opzioni di formazione MOFA ed eseguire l'addestramento del modello facendo clic su di essa. Scorri verso il basso per vedere l'output stampato del modello MOFA dei parametri configurabili nel notebook.
- All'interno del ciclo for R nel codice sotto l'intestazione, vengono impostati tutti i dati, il modello e le opzioni di training MOFA. Sotto la riga model_opts$num_factors = mofa_configs$amount_of_factors[i], aggiungi un'altra riga con il codice seguente
model_opts$probabilità['data_type'] = 'Poisson'.
NOTA: Questo cambierà la distribuzione che il modello prende come input per la vista specificata dal nome data_type per tutte le esecuzioni MOFA. Quando si specifica poisson per un tipo di dati, il modello verrà eseguito solo quando le funzionalità per questo tipo di dati sono interi (ad esempio, conteggi di lettura da RNA-seq). Per ottenere maggiori informazioni sui dati MOFA, sull'addestramento e sulle opzioni del modello, si può anche fare riferimento ai tutorial e alla documentazione MOFA17. - Salva le modifiche nel taccuino facendo clic sul pulsante Salva in alto.
- Per trasferire nuovi parametri tramite i file di configurazione .csv , utilizzare la navigazione sul lato sinistro per accedere alla cartella delle configurazioni facendo doppio clic sulle configurazioni e aprire il 03_MOFA_Configs.csv dei file facendo doppio clic.
- Aggiungi una nuova colonna specificando il nome del parametro, ad esempio number_iterations e inserisci un valore, ad esempio 1000. Salva le modifiche facendo clic su File > Salva file CSV.
- Utilizzare il menu di navigazione per navigare nella cartella degli script facendo clic su script. Apri il notebook 03_Run_Mofa.ipynb facendo doppio clic su di esso. Fare clic sulla scheda Sommario sul lato sinistro, quindi accedere alla sottosezione 4.3 Impostare le opzioni di formazione MOFA ed eseguire l'addestramento del modello facendo clic su di essa.
- Sostituisci la riga train_opts$maxiter = 50000 con train_opts$maxiter = mofa_configs$column_name[i] (quando il nome della colonna aggiunta è number_of_iterations è train_opts$maxiter = mofa_configs$number_of_iterations[i])).
NOTA: Il file di configurazione 03_MOFA_Configs.csv in questo notebook viene letto all'inizio del notebook (sottosezione: Prerequisiti Configurazioni e parametri) come mofa_config data.frame nella sessione e quindi, in questa riga di codice, si fa riferimento a questo oggetto e alla corrispondente colonna appena generata. Poiché è possibile eseguire più configurazioni contemporaneamente, i identifica la riga del data.frame poiché la stima del modello viene eseguita in un ciclo for su tutte le diverse righe del file .csv . Il principio di lettura nel file di configurazione all'inizio del notebook nella sezione "Prerequisiti, configurazioni e parametri" è lo stesso per tutti i notebook, e ulteriori modifiche possono essere apportate in questo modo. - Salvare le modifiche nel blocco appunti facendo clic sul pulsante Salva .
Risultati
Dopo la corretta esecuzione del flusso di lavoro, vengono generate diverse tabelle e figure, come indicato nella Figura 2. Le figure vengono inserite nella cartella /figures (Figura 6, Figura 7, Figura 8, Figura supplementare 1, Figura supplementare 2, Figura supplementare 3, Figura supplementar...
Discussione
Con il protocollo delineato, viene presentato un flusso di lavoro modulare ed estendibile basato su Jupyter-notebook che può essere utilizzato per esplorare rapidamente un complesso set di dati multi-omici. Le parti principali del flusso di lavoro consistono nella parte di pre-elaborazione e armonizzazione dei dati (che offre diversi passaggi standard per il filtraggio e la normalizzazione dei dati), la stima del modello MOFA9 e alcune analisi esemplari a valle. ...
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
C.L. è supportato dall'Associazione Helmholtz nell'ambito della scuola di ricerca congiunta "Munich School for Data Science - MUDS".
Materiali
| Name | Company | Catalog Number | Comments |
| Apptainer | NA | NA | https://apptainer.org/docs/admin/main/installation.html |
| Compute server or workstation or cloud (Linux, Mac or Windows environment). Depending on the size of the different input datasets we recommend running the workflow on a suitable machine (in our setting we use: 16 CPU, 64GB Memory) | Any manufacturer | 16 CPU, 64GB Memory | Large Memory is only required for the processing of the raw single cell data. After preprocessing the later analysis steps can also be performed on regular desktop or laptop computers |
| git | NA | NA | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git |
| GitHub | GitHub | NA | https://github.com/heiniglab/mofa_workflow |
Riferimenti
- Lähnemann, D., et al. Eleven grand challenges in single-cell data science. Genome Biol. 21 (1), 31 (2020).
- Colomé-Tatché, M., Theis, F. J. Statistical single cell multi-omics integration. Curr Opin Syst Biol. 7, 54-59 (2018).
- Hawe, J., Theis, F., Heinig, M. Inferring interaction networks from multi-omics data. Front Genet. 10, 535 (2019).
- Hawe, J. S., et al. Network reconstruction for trans acting genetic loci using multi-omics data and prior information. Genome Med. 14 (1), 125 (2022).
- Koh, H. W. L., Fermin, D., Vogel, C., Choi, K. P., Ewing, R. M., Choi, H. iOmicsPASS: network-based integration of multiomics data for predictive subnetwork discovery. NPJ Syst Biol Appl. 5, 22 (2019).
- Ogris, C., Hu, Y., Arloth, J., Müller, N. S. Versatile knowledge guided network inference method for prioritizing key regulatory factors in multi-omics data. Sci Rep. 11, 6806 (2021).
- Lee, C., vander Schaar, M. A variational information bottleneck approach to multi-omics data integration. Proceedings of The 24th International Conference on Artificial Intelligence and Statistics. 130, 1513-1521 (2021).
- Singh, A., et al. DIABLO: an integrative approach for identifying key molecular drivers from multi-omics assays. Bioinformatics. 35 (17), 3055-3062 (2019).
- Argelaguet, R., et al. Multi-omics factor analysis-a framework for unsupervised integration of multi-omics data sets. Mol Syst Biol. 14 (6), e8124 (2018).
- Cantini, L., et al. Benchmarking joint multi-omics dimensionality reduction approaches for the study of cancer. Nature Commun. 12 (1), 124 (2021).
- Pekayvaz, K., et al. Multiomic analyses uncover immunological signatures in acute and chronic coronary syndromes. Nature Medicine. 30 (6), 1696-1710 (2024).
- Swirski, F. K., Nahrendorf, M. Cardioimmunology: the immune system in cardiac homeostasis and disease. Nat Rev Immunol. 18 (12), 733-744 (2018).
- Janjic, A., et al. Prime-seq, efficient and powerful bulk RNA sequencing. Genome Biol. 23 (1), 88 (2022).
- Wolf, F. A., Angerer, P., Theis, F. J. SCANPY: large-scale single-cell gene expression data analysis. Genome Biol. 19 (1), 15 (2018).
- Cao, Y., et al. Integrated analysis of multimodal single-cell data with structural similarity. Nucleic Acids Res. 50 (21), e121 (2022).
- . Get Started - JupyterLab 4.1.0a4 documentation Available from: https://jupyterlab.readthedocs.io/en/latest/getting_started/overview.html (2024)
- . MOFA2: training a model in R Available from: https://raw.githack.com/bioFAM/MOFA2_tutorials/master/R_tutorials/getting_started_R.html (2020)
- De Silva, D., et al. Robust T cell activation requires an eIF3-driven burst in T cell receptor translation. eLife. 10, e74272 (2021).
- Li, G., Liang, X., Lotze, M. HMGB1: The central cytokine for all lymphoid cells. Front Immunol. 4, 68 (2013).
- Jassal, B., et al. The reactome pathway knowledgebase. Nucleic Acids Res. 48 (D1), D498-D503 (2020).
- Argelaguet, R., et al. MOFA+: a statistical framework for comprehensive integration of multimodal single-cell data. Genome Biol. 21 (1), 111 (2020).
- Velten, B., et al. Identifying temporal and spatial patterns of variation from multimodal data using MEFISTO. Nat Methods. 19 (2), 179-186 (2022).
- Qoku, A., Buettner, F. Encoding domain knowledge in multi-view latent variable models: A Bayesian approach with structured sparsity. Proceedings of The 26th International Conference on Artificial Intelligence and Statistics. 206, 11545-11562 (2022).
- Multi-Omics Factor Analysis. MOFA Available from: https://biofam.github.io/MOFA2/ (2024)
- Mitchel, J., et al. Tensor decomposition reveals coordinated multicellular patterns of transcriptional variation that distinguish and stratify disease individuals. bioRxiv. , (2023).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon