Method Article
铜绿假单胞菌和金黄色葡萄球菌异质表型的延时落射荧光显微镜成像
* 这些作者具有相同的贡献
摘要
在本手稿中,我们提供了一个全面的方案,用于评估 铜绿假单胞菌 和 金黄色葡萄球菌的抗生素存活率,将质粒转化为 铜绿假单 胞菌和 金黄色葡萄球菌 ,以产生报告菌株并通过延时落射荧光显微镜可视化表型变体,例如持久性。
摘要
抗生素持久性是一种现象,其中遗传易感人群中的少数细菌细胞在杀死其他基因相同的细胞的抗生素治疗后存活下来。一旦抗生素治疗结束,细菌持续存在即可恢复复制,通常被认为是临床治疗失败的原因。最近的工作利用延时荧光显微镜的功效,其中细菌用荧光转录报告基因、翻译报告基因和/或各种细胞特征的染料标记,这推进了我们对 大肠杆菌 持久性的理解,超出了从种群水平抗生素存活测定中可以学到的。这种单细胞方法,而不是大量群体测定,对于描述持久性形成、损伤反应和存活的机制至关重要。然而,在这种详细程度上研究其他重要致病物种的持久性的方法仍然有限。
本研究为抗生素治疗和恢复期间铜 绿假单胞 菌 (一种革兰氏阴性杆菌) 和 金黄色葡萄球菌 (一种革兰氏阳性球菌) 的延时成像提供了一种适应性强的方法。我们讨论了将荧光报告基因引入这些细菌的分子遗传学方法。使用这些报告基因以及染料,我们可以追踪响应抗生素处理的单个细胞的表型变化、形态特征和命运。此外,我们能够观察个体持续者在治疗后复苏时的表型。总而言之,这项工作为那些有兴趣追踪单个抗生素处理细胞的存活和基因表达的人提供了资源,包括治疗期间和治疗后临床重要病原体的持久性细胞。
引言
细菌病原体可以通过两种主要机制逃避抗生素的影响:抗生素耐药性(涉及遗传变化)和表型耐受性(涉及非遗传变化)。抗生素耐药性是一种遗传编码现象,它赋予给定细菌细胞在抗生素存在下不仅能够生存而且能够复制的能力 1。表型耐受性可以包括抗生素耐受或抗生素持久性细菌,当细胞承受杀菌抗生素处理而没有获得在抗生素抑制浓度存在下复制的能力时,就会发生表型耐受性 1,2。耐受性与持久性的区别在于,耐受性是指整个人群在治疗中存活的能力,而持久性是指在抗生素治疗中存活下来的同基因但表型异质性人群的一个子集。当克隆培养物用杀菌抗生素处理时,将留在培养物中的幸存者与时间的关系图对对数线性标度,当存在持续存在时,通常会检测到双相曲线。在这些曲线上,第一阶段表明大多数种群被相对较快地杀死,第二阶段表明抗生素持久性部分以较慢的速度被杀死或根本没有被杀死 1,2。
抗生素持久性给全球医疗保健系统带来了沉重负担。例如,本文重点介绍的金黄色葡萄球菌和铜绿假单胞菌持久性被认为会导致抗生素顽固性感染,包括囊性纤维化和慢性伤口感染患者的复发性气道感染 3,4。因此,进一步阐明持久性细胞生物学和表型程序至关重要。虽然在了解持久性细胞如何形成和复苏方面取得了进展,但与支撑持久性的单个细胞中代谢重编程和分子事件的协调有关的关键知识差距仍然存在 5,6,7,8。
事实证明,有效地研究持久性是一项技术挑战。由于持久性只能在细菌种群的一小部分中观察到,因此对大量细菌种群进行采样的技术通常无法捕获相关的生物学信息 1,2,8,9,10。此外,由于支撑持久性的表型变化是短暂的且不可遗传的,因此追踪持久性细胞的命运可能很复杂 1,8,9,10,11。一旦细菌持久性恢复生长,它们就会分裂并产生持久性和非持久性,这使得无法通过培养来富集纯持久性种群。这些挑战突出了对能够满足以下标准的技术的需求:1) 能够捕获活的单细胞的生物学信息,以及 2) 能够与荧光染料、探针、传感器和报告基因配合使用,从而随着时间的推移询问异质群体中单个细胞的表型。
单细胞技术的最新进展为有效研究细菌异质性并克服研究持久性的这些障碍提供了途径12,13。其中一些技术包括荧光显微镜检查、流式细胞术/荧光激活细胞分选、微流体和单细胞 RNA 测序12,13。在这里,我们描述了使用转录或翻译报告菌株的落射荧光延时显微镜阐明单细胞持久性生理学的方案。荧光显微镜是一种强大的技术,可以满足研究持久性表型的标准,即能够识别大量细胞群中的哪些单个细胞在抗生素去除后繁殖,因此可以定义为持久性细胞。随着自动相机技术和培养室的引入,捕获活细菌细胞在整个微生物学领域得到了广泛应用。至关重要的是,延时显微镜能够在数小时甚至数天内实时观察单个细胞,从而可以在抗生素治疗之前、期间和之后跟踪细菌 14,15,16。利用延时显微镜的这些研究的见解具有巨大的潜力,可以深入了解持久性生物学的复杂机制。
研究方案
1.通过转化和转导产生金黄色葡萄球菌的荧光报告菌株
注:报告菌株含有荧光蛋白,以指示目标基因或蛋白质的表达。转录报告基因具有荧光蛋白上游目标基因的天然启动子序列的重复拷贝,因此荧光随着目标基因表达的增加而增加。翻译报告基因将荧光蛋白和目标蛋白的开放阅读框与灵活的肽连接器连接起来。使用活细胞显微镜观察报告基因菌株可以揭示给定的目标基因/蛋白质是否与特定细胞形态或细胞命运相关(图 1、补充视频 1 和补充视频 2)。选择针对金黄色葡萄球菌进行密码子优化的荧光蛋白。虽然本研究使用了 Alexander Horswill 博士赠送的 pCM29 的 sGFP,但内布拉斯加州转座子突变文库遗传工具包包含带有密码子优化的 sGFP、eYFP、eCFP、DsRed.T3 和 eqFP650 的质粒17,18。
- 将报告质粒转化到 金黄色葡萄球菌 RN4220 中。
注意:以下协议部分改编自内布拉斯加大学医学中心的葡萄球菌遗传学和代谢研讨会要点19,20。- 首先,准备电感受态 金黄色葡萄球菌 RN4220。通过将 2 g 酪蛋白氨基酸、5 g 酵母提取物、2 mL 10% K2HPO4(pH 值为 7.5)、2 mL 50% 葡萄糖溶液和 5 g NaCl 混合,制备 200 mL B2 肉汤。用无菌超纯水将体积调至 100 mL。在 37 °C 下短暂混合以溶解组分并使用瓶顶过滤器(0.22 μm 孔径)进行消毒。
- 将 金黄色 葡萄球菌 RN4220 从储存在 -80 °C 的 25% 甘油中的冷冻原液中接种到 250 mL 挡板烧瓶中的 30 mL B 2 肉汤中,并在 30 °C 下孵育 18 小时,以 125 rpm 摇动。
- 将两个 250 mL 带挡板的烧瓶与 50 mL B2 肉汤在 37 °C 下孵育过夜,以预热第二天的培养基。
- 第二天,测量过夜培养物的 OD600 。向每个含有 50 mL B2 肉汤的培养瓶中加入足够的过夜培养物,使最终 OD600 为 0.25。
- 在 37 °C 下孵育,以 250 rpm 振荡,直到 OD600 达到 0.35-0.4。
注意:传代培养后约 30 分钟检查 OD600 ,以确保培养物不会生长超过 0.35-0.4。如果 OD600 在传代培养后 1 小时内未达到 0.35-0.4,则放弃该程序并在第二天重试。细菌很可能在一夜之间生长过多,并且进入固定相太深。 - 一旦培养物达到对数中期,将每个培养瓶中的培养物倒入无菌 50 mL 锥形管中,并以 4,000 x g 离心 10 分钟使细胞沉淀。
- 倒出上清液,并将沉淀重悬于 1 mL 无菌室温 (RT) 水中。将细胞转移到微量离心管中。
- 再次以 21,000 x g 离心细胞 30 秒,去除上清液,然后重悬于 1 mL 无菌 RT 水中。重复离心,去除上清液,并在 1 mL 水步骤中再重悬 2 次,总共洗涤 3 次。
- 离心细胞,除去水,然后将细胞重悬于 1 mL 无菌 RT 10% 甘油(水中,无菌过滤)中,并在 RT 下放置 15 分钟。
- 以 21,000 x g 离心 30 秒,使细胞沉淀,去除上清液,然后将细胞重悬于另外 1 mL 无菌 RT 10% 甘油中。这些电池现在是电感受态的。
- 将 70 μL 电感受态细胞分装到卡口盖微量离心管中,并储存在 -80 °C 以备将来使用。
- 为了转化电感受态 金黄色葡萄球菌 RN4220,在冰上解冻等分试样的细胞。向细胞中加入 1 μg 质粒 DNA,轻弹样品混合,并在冰上孵育 5 分钟。然后,对细胞进行电穿孔,让它们在 390 μL 的 B2 培养基中在 37 °C 下回收 1-2 小时,以 225 rpm 的速度振荡,并在选择性胰蛋白酶大豆琼脂上铺板转化体。
注:本研究中使用的大多数穿梭载体均具有氨苄青霉素抗性盒,用于大 肠 杆菌 (100 μg/mL) 的选择,以及氯霉素 (10 μg/mL) 或红霉素 (Erm,10 μg/mL) 抗性盒,用于金 黄色葡萄球菌的选择。 实验室中常规使用的许多 金黄色 葡萄球菌菌株都对 Erm 产生抗性。在使用给定的穿梭载体之前,请确保目标菌株与选择性抗生素兼容。重要的是,将添加到细胞中的 DNA 体积降至最低(即 <5 μL),以防止电弧,当样品在电穿孔过程中导电性过强时,就会发生电弧。从 大肠 杆菌中纯化高浓度 (≥300 ng/μL) 的质粒 DNA 将有助于保持低体积。
- 在转化的噬菌体上繁殖 S. aureus RN4220 的。
注意:以下协议部分改编自 Krausz 和 Bose 的方法21和奥尔森22.两种最常用的噬菌体用于 S. aureus 转导是 φ11 和 80α。收获这两个的过程是相同的,只是我们从 S. aureus RN451 和 80α 来自 S. aureus RN10359。φ11 已成功用于常见菌株 JE2 和 HG003 等,并用于该方案23,24.然而,这些噬菌体可能无法有效地转导具有不同噬菌体耐药性的临床分离株。请参阅 Krausz 和 Bose 的出版物21 和奥尔森22 用于制备 φ11 和测定噬菌体滴度的方法。- 为了转导报告质粒,用 5 mM CaCl2 和适当浓度的选择性抗生素制备六个胰蛋白酶大豆琼脂 (TSA) 板。用报告质粒将金 黄色葡萄球菌 RN4220 连贯到含有选择性抗生素且不含 CaCl2 的 TSA 板上并孵育过夜。
- 第二天,将培养箱或水浴打开至 56 °C。 标记 6 个 5 mL 卡口盖管 1 x 104 至 1 x 109,并将 40 μL 500 mM CaCl2 分装到每个卡口管中。
- 在微波炉中融化软 TSA(0.5% 琼脂),松开盖子,以防止压力积聚,同时保持无菌。然后,将 4 mL 分装到每个卡口盖管中。倒置试管进行混合,然后将它们放入 56 °C 培养箱中,以防止软琼脂凝固。
- 在胰蛋白酶大豆汤 (TSB) + 5 mM CaCl 中将噬菌体原液稀释至 1 x10 10 PFU/mL,2。将 100 μL 1 x10 10 PFU/mL 噬菌体添加到含有 900 μL TSB + 5 mM CaCl2 的微量离心管中,最终浓度为 1 x 109 PFU/mL。然后,连续稀释到含有 900 μL TSB + 5 mM CaCl2 的其他微量离心管中,直至浓度为 1 x 104 PFU/mL。用连续稀释度标记六个 TSA + 5 mM CaCl2 + 选择性抗生素琼脂板中的每一个:1 x104 到 1 x 109 PFU/mL。
- 在 1 mL TSB + 5 mM CaCl2 中重悬含有质粒的过夜金黄色葡萄球菌 RN4220。从培养箱中取出 109 卡口盖管,快速加入 10 μL 含有质粒的重悬 RN4220 和 100 μL 1 x 109 PFU/mL 噬菌体稀释液,倒置混合,然后将内容物倒入相应的 TSA + 5 mM CaCl2 + 选择性抗生素琼脂平板上。
- 对剩余的噬菌体稀释液及其各自的板执行此程序。让软琼脂在平板上凝固,然后在 37 °C 下将软琼脂面朝上孵育过夜。
- 第二天,确保在板上可以看到斑块(细菌草坪上的清除区域)(图 2A)。噬菌斑代表噬菌体成功感染 RN4220 的裂解区域,一些噬菌体会占据感兴趣的质粒。选择裂解裂解度接近汇合的噬菌体储液稀释度最低的板(例如:1 x 109 PFU/mL 噬菌体),以及接受低于此两种稀释度的板(在本例中为 1 x 108 PFU/mL 和 1 x 107 PFU/mL,它们都应该具有接近汇合的裂解)。
- 将 3 mL TSB + 5 mM CaCl2 添加到这些板中稀释度最低的板中(在本例中为 1 x 109 PFU/mL)。使用无菌 L 形细胞扩散器轻轻地从普通琼脂上刮下软琼脂层(图 2B)。使用细胞涂布器尽可能多地破坏琼脂,以促进噬菌体从软琼脂中释放。
- 一旦第一个板上的软琼脂被彻底破坏,轻轻地将软琼脂和肉汤浆液倒到下一个板上(在本例中为 1 x 108 PFU/mL)。刮擦并破坏该板上的软琼脂,然后将所得浆液倒入最终板上并重复。
- 从所有三个板中制备软琼脂浆液后,将浆液轻轻倒入 50 mL 锥形管中。轻轻移液以进一步破坏软琼脂,但要避免形成气泡。不要涡旋,因为它可能会损坏噬菌体并降低滴度。
- 在 RT 下以 4,000 x g 离心软琼脂浆液 10 分钟。将上清液轻轻倒入连接到 0.45 μm 过滤器的注射器中。
注意:必须轻轻倾倒,避免琼脂碎片进入过滤器。这些碎片会堵塞过滤器并阻止过滤剩余的上清液,从而降低滴度。 - 使用野生型 RN422021 测定所得噬菌体裂解物的滴度。
- 通过转导将报告质粒引入感兴趣的 金黄色 葡萄球菌菌株中。
- 准备 8 个 TSA 板,其中含有 500 μg/mL 柠檬酸钠和适当浓度的选择性抗生素。将所需的受体 金黄色葡萄球菌 菌株密集划线到普通 TSA 上,并在 37 °C 下孵育过夜。
- 第二天,擦拭板并将受体菌株重悬于 1 mL TSB + 5 mM CaCl2 中。
- 将携带所需报告质粒的噬菌体储液稀释至 1 x1010 PFU/mL。
- 在 50 mL 锥形管中混合 1.5 mL TSB + 5 mM CaCl2、0.5 mL 重悬的受体菌株和 0.5 mL 稀释的噬菌体原液。在 37 °C 下孵育,以 225 rpm 振荡,持续 20 分钟。
注:不建议使用较长的孵育时间,因为当噬菌体开始裂解受体细胞时,它们可能会降低转导效率。 - 然后,立即向锥形管中加入 1 mL 无菌过滤的冰冷 0.02 M 柠檬酸钠(水溶液),并在 4 °C 下以 4,000 x g 离心 10 分钟。 然后,将沉淀重悬于 1 mL 冰冷的 0.02 M 柠檬酸钠中。
- 将 100 μL 等分试样涂布到六个 TSA 板中,每个板含有 500 μg/mL 柠檬酸钠和适当浓度的选择性抗生素,以选择转导剂。在 37 °C 下孵育过夜。
- 第二天,将转导板中的单个菌落划线到其余两个含有 500 μg/mL 柠檬酸钠和适当浓度的选择性抗生素的 TSA 板上。在 37 °C 下孵育过夜。
注:其他柠檬酸钠平板上的传代步骤有助于降低转导细胞周围噬菌体的滴度,防止裂解22。 - 从条纹板中挑选单个菌落,在含有选择性抗生素的 TSB 中将细胞生长至指数期,并在 -80 °C 下储存在 25% 甘油中以备将来使用。
2.通过偶联产生铜绿假单胞菌的荧光报告菌株
注:可以通过三亲交配25 将报告质粒从大肠杆菌克隆菌株转移到铜绿假单胞菌中。携带目标质粒的大肠杆菌供体菌株(应包含用于共轭转移的 oriT)、大肠杆菌 HB101 + pRK2013 - 促进共轭的辅助菌株(使用 50 μg/mL 卡那霉素维持 pRK2013 质粒)和将接收质粒的铜绿假单胞菌受体菌株是必需的。对于本例,目标质粒具有四环素 (Tet) 抗性标记,因此使用质粒的任何大肠杆菌培养物都需要 10 μg/mL Tet,而使用质粒的铜绿假单胞菌需要 75 μg/mL Tet 才能选择26。
- 将细菌从储存在-80°C的25%甘油中的冷冻原液中接种到溶原肉汤(LB)中,用适当的抗生素进行过夜生长,在37°C下以225rpm摇动。
- 第二天,将 200 μL 每种液体过夜培养物涂在 LB 琼脂平板上,并加入适当的选择性抗生素。根据要进行的转化交配次数,为辅助菌株和供体菌株铺布更多的板。建议每次转化一个帮助者/供体板。将板在 37 °C 下孵育过夜,以获得每种菌株的茂密草坪。
- 第二天,用无菌棉签擦拭每个板表面并沿着微量离心管的内部擦拭,收集生长的草坪。短暂离心每个试管(脉冲 5 秒)以将细胞收集到试管底部。将每个细胞沉淀重悬于 200 μL LB 中。
注意:重悬液看起来应该非常厚、不透明且没有结块。 - 在每次交配的新微量离心管中,混合 5 μL 受体 铜绿假单 胞菌菌株、160 μL 携带目标质粒的供体 大肠 杆菌菌株和 160 μL 辅助 性大肠 杆菌 HB101 + pRK2013。轻轻混匀,上下移液,然后将 50 μL 混合物点到预干燥的 LB 琼脂平板上。此外,将 20 μL 每种菌株点样到 LB 琼脂中用于对照。让斑点完全干燥到板中,然后在 37 °C 下孵育 3-6 小时。
注:预干燥 LB 琼脂可防止 50 μL 斑点在整个表面扩散,从而迫使细胞在较小的区域内相互作用。 - 孵育后,用移液器吸头轻轻扫过交配点,将一些细胞转移到新的微量离心管中。短暂离心以收集管底部的细胞。重悬于 200 μL LB 中,并将整个重悬液接种到 LB + Irgasan (Igr;25 μg/mL) + Tet (75 μg/mL) 琼脂平板上。
注意: 铜绿假单 胞菌对 Igrasan 具有内在抗性,并且感兴趣的质粒含有 Tet 抗性标记,因此只有成功转化的铜 绿假单 胞菌才能在 LB + Irgasan + Tet 平板上生长。根据转化效率,重悬和铺板整个交配点可能会导致第二天出现转化体草坪。为了获得单个菌落,(i) 将整个交配点重悬于更大体积的 LB 中,然后接种 200 μL 的 LB,或 (ii) 将一小部分交配点转移到微量离心管中,然后重悬于 200 μL 中并铺板。 - 对于每个菌株的对照点,将一些致密的细胞点轻轻地扫到移液器吸头上,转移到新的微量离心管中,重悬于 200 μL 的 LB 中,然后将 50 μL 点到 LB + Irgasan + Tet 琼脂平板上(多个控制点可以放置在同一个琼脂平板上)。让斑点完全干燥。将所有板在 37 °C 下孵育 16-20 小时过夜。
注意:对于对照点,目标是验证抗生素板对 铜绿假单 胞菌转化体具有选择性。如果在 LB + Irgasan + Tet 琼脂平板上点样时对照点过于集中,则很难确定第二天的选择是否有效,因为会出现可能被误认为生长的死细胞光晕。 - 第二天,通过注意单个菌株对照点缺乏细胞生长来确认 LB + Irgasan + Tet 板的抗生素选择性。
注:出现在三亲交配 LB + Igrasan + Tet 平板上的菌落是携带目标质粒的铜 绿假单 胞菌转化体。 - 可选择将菌落采摘并划线到 LB + Irgasan + Tet 琼脂上并生长过夜,以去除交配混合物中多余的死 大肠杆菌 。
- 挑选单个菌落,在液体 LB + Tet (75 μg/mL) 中生长 3-6 小时,直至明显浑浊,然后将冷冻原液储存在 25% 甘油(500 μL 细胞培养物 + 500 μL 50% 甘油水溶液)中。验证克隆是否通过菌落 PCR 和 Sanger 或全质粒测序成功转化。
3. 确定持久性测定的抗生素剂量
注:要选择给定抗生素的剂量来处理持久性实验的细菌种群,请首先测量抗生素对目标细菌菌株的最低抑制浓度 (MIC)。这可以使用肉汤微量稀释法(临床和实验室标准协会 (CLSI) 认可的一种方法)或 Epsilometer 测试 (E-test) 来实现,该方法使用具有一系列抗生素剂量的试纸完成27。一旦确定了 MIC,就选择至少五种浓度的抗生素,范围为 1 至 100 倍 MIC 用于细胞处理。
- 将细菌从储存在 -80 °C 的 25% 甘油中的冷冻原液中接种到 2 mL 阳离子调整的 MHB (CA-MHB) 或其他营养丰富的培养基中。将细胞在 37 °C 下以 250 rpm 振荡培养约 4 小时,然后将 125 μL 培养物转移到 250 mL 带挡板的锥形瓶中的 25 mL 新鲜化学定义的培养基中。将细菌在 37 °C 下培养 16 小时(至固定相),以 250 rpm 摇动。
注:以琥珀酸盐为唯一碳源的基础盐培养基 (BSM) 通常用于铜绿假单胞菌实验,而富含化学成分的培养基通常用于金黄色葡萄球菌实验 28,29,30,31。 - 第二天早上,为所需的超 MIC 浓度范围准备 100x 抗生素原液(在各自的溶剂中)。将 10 μL 的每种稀释液添加到单个试管中。
- 测量过夜培养物的 OD600 以确认静止相浊度。连续稀释 10 μL 培养物,并将稀释液接种到营养琼脂平板(如 LB 琼脂、CA-MHB 琼脂或 TSA)上,以确定抗生素处理前的菌落形成单位 (CFU)。
- 对于抗生素处理,将 1 mL 等分试样的培养物分配到含有 10 μL 100 倍抗生素浓度的试管中。将样品在 37 °C 下孵育,以 250 rpm 振荡,其持续时间足以杀死群体中的非持久性细胞,留下持久性细胞作为唯一的集落形成细胞。
注意:治疗持续时间可能因实验和被测试的细菌菌株而异。通常, 金黄色葡萄球菌 处理 5 小时或 7 小时, 铜绿假单 胞菌处理 7 小时或 24 小时。通常,可以使用时间依赖性存活测定第二阶段中的任何时间点,因为预计留在第二阶段的细胞将是持久性细胞。 - 抗生素处理后,将 100 μL 细胞从试管转移到含有 900 μL 无菌磷酸盐缓冲盐水 (PBS) 的微量离心管中。通过在 RT 下以 21,000 x g 离心 3 分钟来沉淀细胞。去除 900 μL 上清液,将沉淀重悬于 900 μL 无菌 PBS 中。至少再重复一次洗涤步骤,以将残留抗生素减少到亚 MIC 水平。
- 连续稀释培养物 10 倍(6 次),并将每种稀释液的 10 μL 接种到营养琼脂平板上。将板在 37 °C 下孵育过夜。
注:使用未经处理的圆底 96 孔板进行连续稀释,并使用多通道移液器将细胞铺在方形琼脂平板上。 - 第二天,计算每种抗生素浓度的菌落。在对数线性刻度上绘制生存分数(治疗结束时的 CFU/治疗前 CFU)与浓度的关系。要为未来的持久性测定选择药物浓度,请在双相曲线的第二阶段选择浓度(图 3)。
4. 抗生素治疗或恢复期间的细胞成像
- 准备琼脂糖垫和细胞样品以进行延时成像
注:以下样品制备方案是作为传统琼脂糖"夹心"方法的用户友好、经济高效的替代方案而开发的14,16.使用可互换的盖玻片培养皿无需使用凌乱的油脂或指甲油来密封样品,这可能会进一步限制样品通气。将琼脂糖放入可互换的盖玻片培养皿中,盖玻片已就位,与徒手琼脂糖垫制备相比,可形成可靠的平面琼脂糖表面。总体而言,这种方法允许对样品进行成像,在整个视场中具有更好的聚焦效果,并且由于稳定的加湿效果而延长了持续时间。此处描述的方法用于在抗生素治疗期间可视化细菌 (图 4, 补充视频 3和 补充视频 4).为了可视化持续存在者在抗生素治疗后复苏和重新觉醒 (图 5, 补充视频 5和 补充视频 6),用新鲜培养基而不是用过的培养基制备琼脂糖垫,并且不要在垫中添加抗生素(为质粒维持/抗生素选择而添加的抗生素除外)。- 将无菌的 30 mm 盖玻片(#1.5 厚度)放入可互换盖玻片培养皿("腔室")的不锈钢底座底部。轻轻地将聚碳酸酯插件拧入底座,使 30 mm 盖玻片形成腔室的底座,然后通过压缩连接的硅胶 O 形圈将其密封到位。重复此步骤以准备一个重复的腔室。
注意:建议为每个实验准备副本,以防其中一个腔室的制备不理想。在 30 mm 盖玻片上放置一个定制的 3D 打印隔板,以防止铜 绿假单 胞菌等运动细胞交叉污染,并为样品位置提供标志。用于 3D 打印的 STL 文件位于 补充文件 1 中。 - 使用所选培养基在 50 mL 锥形管中制备 1.5% 琼脂糖。轻轻旋转混合。
注:对于处理期间的静止期细胞成像,使用来自静止期过夜培养物的无细胞用培养基作为基础培养基。通常,琼脂糖培养基在 7 mL 培养基中制备为 0.105 g 琼脂糖,用于制备两个腔室;每个腔室需要 2 mL 琼脂糖培养基,过量有助于避免移液时出现气泡。 - 将 50 mL 锥形瓶放入玻璃烧杯或微波炉安全支架中(确保瓶盖松动)。微波炉高速加热,每 3-4 秒停止旋转和混合。经常暂停和混合,以防止琼脂糖混合物冒泡。
- 总加热时间 ~1 分钟后,检查混合物中是否留下任何未熔化的可见琼脂糖。如果混合物均匀透明,则让琼脂糖培养基短暂冷却至 ~60 °C,然后再添加任何其他药物或染料。轻轻旋转以混合并避免形成气泡。
注:可以添加碘化丙啶(铜 绿假单 胞菌为 16 μM,金黄色葡萄球 菌为 1.6 μM)以观察细胞何时失去活力。
- 总加热时间 ~1 分钟后,检查混合物中是否留下任何未熔化的可见琼脂糖。如果混合物均匀透明,则让琼脂糖培养基短暂冷却至 ~60 °C,然后再添加任何其他药物或染料。轻轻旋转以混合并避免形成气泡。
- 为防止琼脂糖培养基凝固,将 2 mL 琼脂糖培养基移液到装有 30 mm 盖玻片的腔室中。将无菌的 25 mm 盖玻片(#1.5 厚度)轻轻放在顶部腔室开口的琼脂糖培养基上;这有助于防止琼脂糖垫脱水,并确保琼脂糖的顶面平坦,以实现最佳相差成像。让垫子凝固 1-2 小时。
- 垫凝固后,通过在 PBS 中将细胞稀释至适当稀疏的密度来准备要成像的细胞,以可视化单个细胞。在这里,将细胞稀释至 OD600 0.01-0.05。
- 在 25 mm 盖玻片上,使用细尖永久性记号笔标记接种在垫上的每个样品的位置/身份。倒置腔室,使不锈钢底座环朝上,并在松开底座时小心地将聚碳酸酯插件固定在下方。将不锈钢底座放在一边。小心地将 30 mm 盖玻片从垫上滑下并丢弃。
注意:水平滑动以去除 30 mm 盖玻片,并注意不要压痕琼脂糖表面。此外,避免在裸露的琼脂糖表面工作:落在上面的任何灰尘都会影响图像质量,并可能污染垫。 - 使用 25 mm 盖玻片上的标记作为指导,将 5 μL 稀释的细胞点到琼脂糖垫上的相应位置。添加 3 个 5 μL 点(根据需要进行调整,例如 4 x 4 μL 点等)。斑点完全干燥后,轻轻地将一个新的、无菌的 30 毫米盖玻片放在垫的中心。
- 用一只手握住聚碳酸酯插件,然后用另一只手慢慢地将不锈钢底座重新拧回新的盖玻片上。穿线至刚好用手拧紧就足以压缩硅胶 O 形圈并密封腔室。
注意:小心不要过度拧紧 - 如果靠在样品表面的 30 mm 盖玻片被不锈钢底座压缩并反复扭曲,则样品池会以径向模式散开,可能导致样品交叉污染。 - 密封腔室后,观察琼脂糖是否接触 30 mm 盖玻片的表面;此时,通常会有一些但不完全的表面接触。使用镊子或类似工具的钝端轻轻按压 25 mm 盖玻片,直到琼脂糖在整个表面上均匀地接触 30 mm 盖玻片。
注意:如果使用过大的力,此步骤可能会导致 25 mm 盖玻片破裂。 - 一旦琼脂糖充分压在 30 mm 盖玻片上,使没有大气泡残留,垫就可以进行成像了。
- 将无菌的 30 mm 盖玻片(#1.5 厚度)放入可互换盖玻片培养皿("腔室")的不锈钢底座底部。轻轻地将聚碳酸酯插件拧入底座,使 30 mm 盖玻片形成腔室的底座,然后通过压缩连接的硅胶 O 形圈将其密封到位。重复此步骤以准备一个重复的腔室。
- 设置显微镜和图像。
- 准备显微镜和成像室环境控制。将舞台顶部培养箱和大室培养箱温度设置为 37 °C,然后打开腔室加湿器。将一种琼脂糖垫制剂放入载物台顶部培养箱中,然后关闭孵育室以使样品平衡。此时,载物台顶部培养箱加湿盖保持打开状态,以避免冷凝水落在腔室的 25 mm 盖玻片上。
注意:由于温度变化导致的焊盘收缩/膨胀会导致时间点之间的漂移超出自动对焦算法的扫描范围。在成像前将琼脂糖垫平衡至稳定温度至关重要,这通常至少需要 15 分钟。充分加热还可以防止在加湿盖就位时冷凝水到顶部盖玻片上。成像系统的硬件组件为(材料表):活细胞培养室、带有 Plan-Apochromat 63x/1.40 Oil Ph3 M27 物镜的倒置显微镜、Spectra 7 LED 光引擎和 sCMOS 相机(6.5 μm 像素大小)。显微镜的电动组件的控制和图像采集是使用显微镜分析软件应用程序完成的。 - 准备用于图像采集的软件。将 MetaMorph(显微镜分析软件)的内置自动对焦算法设置为在多维采集期间使用相位通道。
- 在 Stage 选项卡中,为每个样品设置多个载物台位置,瞄准细胞密度均匀分布的视野。在 Timelapse 选项卡中,设置要拍摄的图像的所需持续时间和频率(时间间隔)。在 Wavelengths 选项卡中,设置所需的采集通道,并调整样品信号强度的曝光时间。
注意:每个位置的图像每 10 分钟拍摄一次,最长 24 小时。对于本文中提到的染料/荧光基团的荧光成像,使用了以下光引擎激发设置:碘化丙啶(Cy3;555/15 nm)和 GFP(GFP/FITC;470/24 nm)。使用的典型荧光激发曝光时间为 100 毫秒。使用用于碘化丙啶的滤光片组 15(分光镜 FT580,发射 LP590)和用于 GFP 的滤光片组 44(分光镜 FT500,发射 BP 530/50)。
- 在 Stage 选项卡中,为每个样品设置多个载物台位置,瞄准细胞密度均匀分布的视野。在 Timelapse 选项卡中,设置要拍摄的图像的所需持续时间和频率(时间间隔)。在 Wavelengths 选项卡中,设置所需的采集通道,并调整样品信号强度的曝光时间。
- 琼脂糖垫在腔室中预热后,将加湿盖添加到载物台顶部培养箱中。这将有助于防止垫子在成像过程中脱水和移动。
- 打开加湿盖,从相位更改为微分干涉对比 (DIC),并调整显微镜的聚光镜和孔径光阑以获得适当的科勒照明。
- 调整后,切换回同相。导航到每个载物台位置,调整焦点,然后将载物台位置重置为新的焦平面。现在,设置已准备就绪,可以开始映像了。
注意:在开始采集之前重新访问每个载物台位置以确保焊盘没有移动非常重要。如果在给定的舞台位置重新访问单元格时失焦,请重新关注它们并覆盖上一个舞台位置。自动对焦算法设置使用最后一个时间点的焦点 z 位置± 3 μm 作为搜索范围,因此如果焊盘偏移超过 3 μm,则算法将无法正确聚焦。 - 单击 Acquire 按钮开始多维采集。
- 实验完成后,将每个通道中的各个图像编译到堆栈中,以便作为视频查看或进行其他分析。使用 MetaMorph 或 ImageJ 执行此作。
- 要在 MetaMorph 中编译图像(首选),请在 Review Multi Dimensional Data 应用程序中,选择感兴趣的通道/波长,并为给定的载物台位置选择所有时间点。单击 Load Images (加载图像)。将出现每个通道/波长的窗口。将每个编译区及其各自的通道名称存储为 .tiff 文件。
- 要在 ImageJ 中编译,请打开一个阶段位置和一个通道的所有时间点的文件。使用 Images to Stack 进行编译。
- 准备显微镜和成像室环境控制。将舞台顶部培养箱和大室培养箱温度设置为 37 °C,然后打开腔室加湿器。将一种琼脂糖垫制剂放入载物台顶部培养箱中,然后关闭孵育室以使样品平衡。此时,载物台顶部培养箱加湿盖保持打开状态,以避免冷凝水落在腔室的 25 mm 盖玻片上。
5. 使用 Fiji/ImageJ 创建延时视频
注意:斐济(斐济只是 ImageJ)是一款免费提供的图像处理和分析软件,可在此处下载:"https://imagej.net/software/fiji/downloads32"32。Fiji/ImageJ2 1.54f 用于下述图像处理方法。
- 使用 BaSiC33 对相位通道图像堆栈进行阴影校正。在斐济打开所需的相位图像堆栈,然后从 Plugins 选项卡中选择 BaSiC。阴影校正后的堆栈将显示在标题为 Corrected:ImageName 的单独窗口中。
注意:BaSiC.jar可在此处下载:"https://github.com/marrlab/BaSiC33"33。按照开发人员的说明将 BaSiC 正确安装到斐济。 - 使用 Image > Color > Merge Channels(合并通道)将阴影校正的相位通道堆栈与任何其他通道/波长合并。使用 Image > Adjust > Brightness/Contrast 为每个通道调整背景和信号强度。将合并的文件另存为 .tiff。
- 然后,使用 Correct 3D Drift 稳定图像。选择阴影校正堆栈后,转到 Plugins > Registrations > Correct 3D Drift。在打开的对话框窗口中,将 channel for registration 设置为与相位堆栈对应的 channel number。校正后的结果堆栈将命名为 registered time points。裁剪到所需的视野,然后将校正后的文件存储为 .tiff。
注意:如果使用 MetaMorph 创建已编译的图像堆栈,则还有一个额外的处理步骤:转到 Image > Properties 并交换 Slices (z) 和 Frames (t) 字段中的数字。漂移校正现在可以正确地将每个帧解释为一个时间点,而不是 z 堆栈的切片。阴影校正和漂移校正的 .tiff 文件可用于后续图像分析。有许多软件包可用于测量荧光信号强度、量化形态学特征、追踪单个细胞的命运等。两个常用的程序是 Fiji 的 MicrobeJ 插件和 Oufti34,35。 - 使用 Image > Stacks > Label 将时间戳和文本标签添加到堆栈中。
- 要添加比例尺,首先要知道相机的像素大小和显微镜物镜放大倍率。计算像素:微米比为像素大小除以放大倍率。在 Analyze > Set Scale 对话框中,输入 已知距离 字段中的像素:微米比率。然后,使用 Analyze > Tools (分析工具) > Scale Bar (缩放条) 向堆栈中添加比例尺。
注意:显微镜的精确刻度应使用载物台千分尺校准。然而,所使用的显微镜物镜和相机可以提供对比例的粗略估计。例如,这里使用的 PCO sCMOS 相机的像素尺寸为 6.5 x 6.5 μm 2,63x 物镜用于成像,因此像素大小除以放大倍率为 6.5/63 = 0.1032 μm/像素。在 Set Scale 对话框的已知距离字段中输入 0.1032。 - 要将完成的图像堆栈导出为可在 QuickTime Player、Microsoft PowerPoint 等中播放的视频,请另存为 .avi 文件。
结果
将报告质粒成功引入 铜绿假单 胞菌和 金黄色葡萄 球菌中,这可以通过正确的选择性抗生素生长来表明,并且可以通过菌落 PCR 和/或测序来确认。应通过将修饰菌株置于已知诱导目标基因的条件下来验证其表型报告基因,并且所得荧光可以通过流式细胞术、分光光度法或落射荧光显微镜测量(图 1)。
为了便于选择将用于后续实验的抗生素剂量,对感兴趣的铜 绿假单 胞菌或 金黄色葡萄球菌 菌株进行浓度依赖性抗生素持久性测定。浓度依赖性测定通常会导致双相曲线,在较低抗生素浓度下具有陡峭的初始斜率,在较高浓度下具有平台期或不太陡峭的斜率。然而,对于某些抗生素-物种对,可能不会产生明显的双相曲线。例如, 金黄色葡萄球菌 delafloxacin 曲线的曲线显然是双相的(图 3A),但 铜绿假单 胞菌左氧氟沙星曲线不是(图 3B)15。在这种情况下,我们将选择至少是 MIC 10 倍的浓度(例如,5 μg/mL,大约是 铜绿假单胞菌 MIC 的 15 倍)15。然而,由于 15x 左氧氟沙星 MIC 仅导致 ~0.001% 铜绿假单 胞菌存活者,因此如果我们想在对细胞进行成像时看到持久性,则使用 1 μg/mL 左氧氟沙星处理,因为它们在不含抗生素的琼脂糖垫上恢复(补充视频 6);否则,对多个持久化程序进行成像所需的视野数量将变得令人望而却步。
在成像开始时,理想的样品和琼脂糖垫制备应在整个视野中呈平面状,没有大碎片、皱纹或气泡,并且具有均匀分布的单个细胞。获得分布均匀的单细胞可能需要优化样品稀释或重悬。对于金黄色葡萄球菌,细胞往往会形成小簇,需要在接种到琼脂糖垫之前彻底涡旋(图 4 和图 5)。对于铜绿假单胞菌,细胞可能会形成包裹在悬浮液中的粘性细胞外基质中的聚集体;有必要彻底移液这些样品并破坏聚集体以对单细胞进行成像。
成像实验结束后,图像的成功延时图像将出现在焦点上,稳定照明,并且在整个实验过程中 x-y 平面上的漂移最小。 补充视频 7 代表了最佳图像采集:t 是 补充视频 4 在阴影或漂移校正之前的相位通道。如果冷凝(由于过度加湿或样品加热不足)导致水滴在顶部 25 mm 盖玻片上形成,从而扭曲光线并将焦平面推到自动对焦算法的最大搜索范围之外,则可能会发生失焦(补充视频 8)。照明的变化通常表明成像时浸油不足。如果载物台移动得太快,物镜上的油可能会拖到后面,并且在采集图像时仍然会赶上。这可以通过调整采集控件以减慢移动速度或在移动到下一个位置和图像采集之间添加暂停来缓解。主要样品漂移看起来像许多细胞在视野中划过,而一些细胞则保持在原位(补充视频 9)。这通常发生在实验后期,因为琼脂糖垫由于湿度控制不足而脱水。本文中介绍的琼脂糖垫制备旨在促进样品稳定性,但适当加热/加湿样品及其周围环境对于最佳图像采集是必要的。
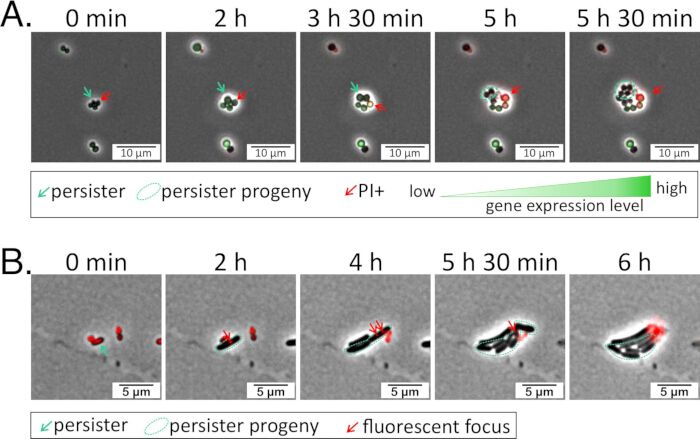
图 1: 荧光报告基因菌株阐明了目标基因的表达。 (A) 根据方案 1 用 GFP 转录报告基因转导目标基因的金黄色葡萄球 菌 。用抗生素处理报告菌株 24 小时,用 PBS 洗涤,然后接种到由 CA-MHB 加碘化丙啶 (1.6 μM) 和氯霉素 (10 μg/mL,用于报告质粒维持) 制成的琼脂糖垫上,用于恢复过程中的成像(补充视频 1)。(B) 用带有 mScarlet 连锁翻译报告基因的质粒转化铜 绿假单 胞菌,用于目标蛋白质26。用抗生素处理报告菌株 5 小时,用 PBS 洗涤,然后接种到由 BSM 加 Tet 制成的琼脂糖垫上(75 μg/mL;用于报告质粒维持),以便在恢复过程中成像(补充视频 2)。 请单击此处查看此图的较大版本。
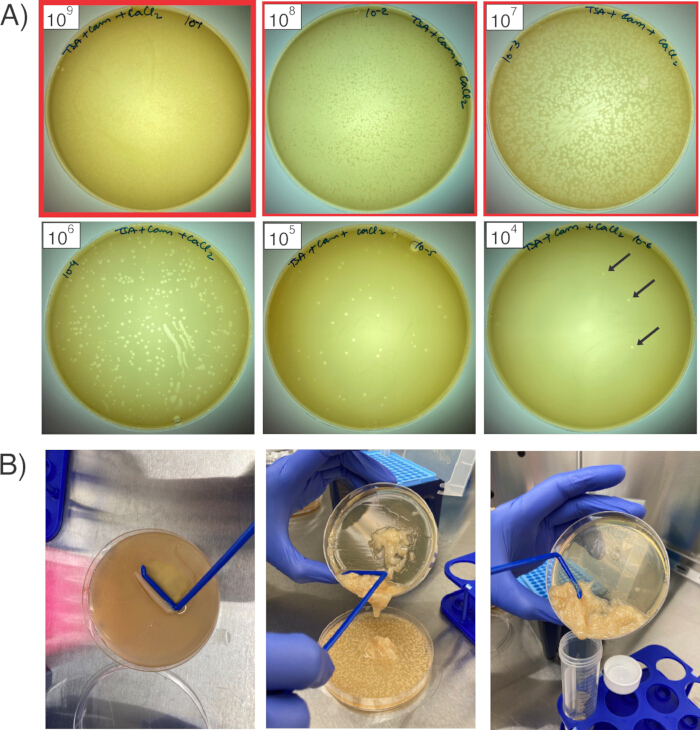
图 2:繁殖和收获噬菌体。 (A) 这六个板显示了金黄色葡萄球 菌 RN4220 草坪上六种不同量的稀释噬菌体原液。红色轮廓表示将收获的三个板,从透明度最高的板(粗体红色轮廓;1 x109 PFU/mL)到接下来的两个稀释液(1 x108 和 1 x 107 PFU/mL)。黑色箭头指向单个斑块。(B) 要从平板中收获噬菌体,刮掉软琼脂层(左),将浆液转移到下一个稀释板(中),然后将所有三个平板的软琼脂汇集在一起后,合并成锥形管进行离心(右)。 请单击此处查看此图的较大版本。
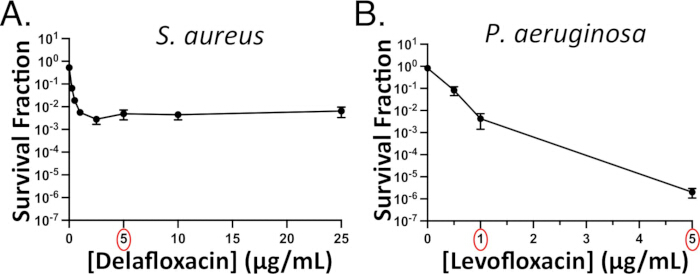
图 3:代表性的浓度依赖性持久性测定。在静止期 (A) 金黄色葡萄球菌 (针对德拉沙星) 和 (B) 铜绿假单胞菌 (针对左氧氟沙星) 中评估浓度依赖性氟喹诺酮类药物持久性。随后的实验使用 5 μg/mL 德拉沙星(红色圆圈),因为金黄色葡萄球菌杀伤已在此浓度下趋于稳定。至少 1 μg/mL 左氧氟沙星(红色圆圈)的剂量将用于铜绿假单胞菌的后续实验。请注意,铜绿假单胞菌的细菌杀灭并未趋于稳定,但双相曲线的"第二阶段"仍然不那么陡峭,表明存在持续的亚群。面板 3B 已获得 Hare 等人的许可15。请单击此处查看此图的较大版本。
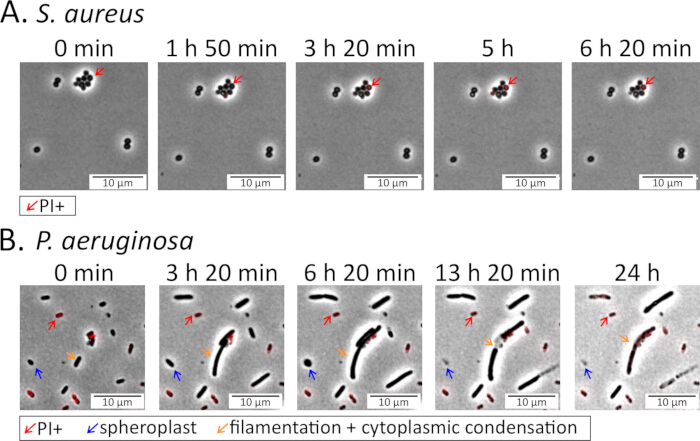
图 4:抗生素治疗期间的细菌表型成像。 将固定期 (A) 金黄色葡萄球菌 和 (B) 铜绿假单 胞菌细胞接种到含有氟喹诺酮类抗生素的琼脂糖垫上,并在处理过程中进行监测:金 黄色 葡萄球菌 5 μg/mL 德拉氟沙星(补充视频 3)和铜 绿假单 胞菌 5 μg/mL 左氧氟沙星(补充视频 4)15。将碘化丙啶(PI; 铜绿假单胞菌为 16 μM, 金黄色葡萄球菌为 1.6 μM)添加到垫中以标记死亡或垂死的细胞。 金黄色葡萄球菌 细胞在 FQ 存在下基本保持完整和活力,而大多数 铜绿假单 胞菌细胞在裂解和死亡之前会发生剧烈的形态变化,包括形成圆形原生质球。 请单击此处查看此图的较大版本。
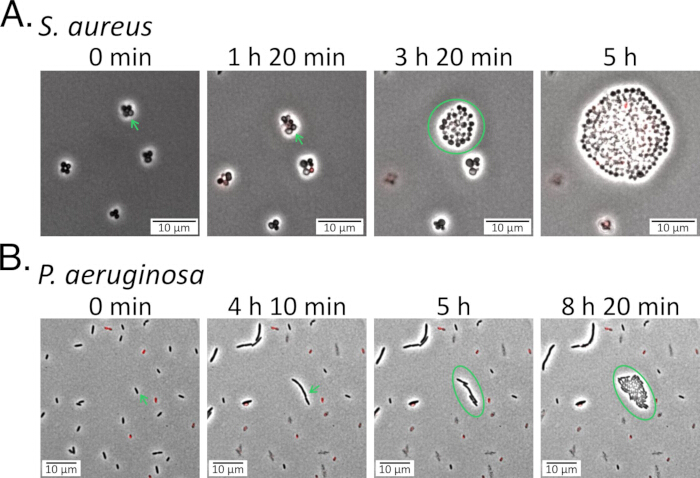
图 5:在恢复过程中跟踪持久性。 (A) 金黄色 葡萄球菌和 (B) 铜绿假 单胞菌种群在用氟喹诺酮类药物(金 黄色葡萄球菌 5 μg/mL 德拉沙星和铜 绿假单胞菌 1 μg/mL 左氧氟沙星)处理后,将它们接种到含有新鲜培养基的琼脂糖垫上,并在治疗后恢复期间进行监测(补充视频 5 和 补充视频 6).在每个面板的前两个框架中用绿色箭头表示所看到的 perisisters,并且在抗生素治疗期间它们保持完整和活力。在最初的滞后期之后,持久性开始分裂并产生新的后代(用绿色圆圈表示)。 请单击此处查看此图的较大版本。
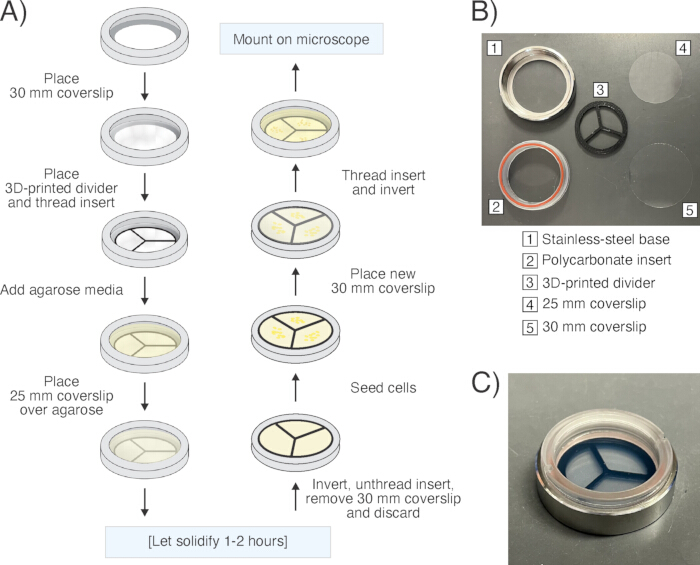
图 6:显微镜样品制备。 (A) 使用可互换盖玻片培养皿("腔室")的样品制备工作流程示意图。 (B) 拆卸后的腔室及其各个组件的图片。(C) 完全组装的腔室的照片。 请单击此处查看此图的较大版本。
补充视频 1: 金黄色葡萄球菌 持久性。 包含 图 1A 中图像的视频文件。简而言之,用抗生素处理带有目标基因 GFP 转录报告基因的 金黄色 葡萄球菌 24 小时,用 PBS 洗涤,然后接种到用 CA-MHB 加碘化丙啶 (1.6 μM) 和氯霉素 (10 μg/mL) 制成的琼脂糖垫上,用于在恢复过程中成像。 请点击此处下载此视频。
补充视频 2:铜绿假单胞菌持久性。 包含图 1B 中图像的视频文件。简而言之,用抗生素处理目标蛋白质的带有 mScarlet 连锁翻译报告基因的铜绿假单胞菌 5 小时,用 PBS 洗涤,然后接种到用 BSM 加 Tet (75 μg/mL) 制成的琼脂糖垫上,以便在恢复期间成像26。请点击此处下载此视频。
补充视频 3:抗生素治疗期间的金黄色葡萄球菌。将生长在丰富的化学成分明确的培养基中的金黄色葡萄球菌固定期培养物接种到由含有碘化丙啶 (1.6 μM) 和德拉沙星 (5 μg/mL) 的培养物的无细胞条件培养基制成的琼脂糖垫中。请点击此处下载此视频。
补充视频 4:抗生素治疗期间的铜绿假单胞菌。将在 BSM 中生长的铜绿假单胞菌的固定期培养物接种到由无细胞条件培养基制成的琼脂糖垫上,这些培养基来自在 BSM 中平行生长的铜绿假单胞菌培养物;琼脂糖垫还含有碘化丙啶 (16 μM) 和左氧氟沙星 (5 μg/mL)。本视频经 Hare et al.15 许可改编。请点击此处下载此视频。
补充视频 5:抗生素后恢复过程中的金黄色葡萄球菌。 金黄色葡萄球菌在丰富的化学成分明确的培养基中生长至固定相。将固定期培养物在试管中用 5 μg/mL 德拉沙星处理 24 小时,用 PBS 洗涤,然后接种到含有碘化丙啶 (1.6 μM) 的无抗生素 CA-MHB 琼脂糖垫中进行成像。请点击此处下载此视频。
补充视频 6:抗生素后恢复期间的铜绿假单胞菌。将 BSM 中生长的铜绿假单胞菌固定期培养物在试管中用 1 μg/mL 左氧氟沙星处理 7 小时,用 PBS 洗涤,然后接种到含有碘化丙啶 (16 μM) 的无抗生素 BSM 琼脂糖垫中用于成像。请点击此处下载此视频。
补充视频 7:最佳图像采集示例。 该视频是图像处理前补充 视频 4 的相位通道,作为最佳延时采集的示例。请注意,在整个实验过程中,漂移最小,照明稳定,焦点保持。 请点击此处下载此视频。
补充视频 8:由于冷凝而导致的次优图像采集示例。 该视频展示了图像采集受到聚焦不良影响的部分实验,这可能是由于样品加热不当和/或成像环境过度加湿导致腔室冷凝。成像的样品是左氧氟沙星处理 的铜绿假单 胞菌,在 BSM 琼脂糖垫上进行抗生素后回收。 请点击此处下载此视频。
补充视频 9:由于漂移导致的图像采集不理想示例。 该视频显示了图像采集受样品漂移影响的实验的一部分,这可能是由于脱水和琼脂糖垫从盖玻片上收缩/抬起。被成像的样品是铜 绿假单 胞菌,位于含有左氧氟沙星和碘化丙啶的琼脂糖垫上。 请点击此处下载此视频。
补充文件 1:25mm-3D-divider-for-35mmBioptechs.stl 请单击此处下载此文件。
讨论
我们发现,延时显微镜实验的成功取决于琼脂糖垫的质量及其在整个成像过程中的稳定性。不锈钢腔室封闭的琼脂糖垫相对容易制备,从而获得一致的平面样品,可在数十小时内稳定成像。这使得可以在单个实验中对数万个细胞进行成像,并增加在群体中检测到罕见表型变异(如持久性细胞)的可能性。
这种琼脂糖垫制备方法为以前发布的方法提供了一种易于实施的替代方案。我们的方案不需要微流体装置制造的技术精度或琼脂糖"夹心"方法的灵巧作,从而更容易实现从运行到运行的一致制备 14,16,36。此外,该系统具有成本效益。不锈钢腔室可消毒和重复使用(与一次性塑料腔室不同),并且设置不需要专用设备16,37。使用市售的载物台插件,该腔室可轻松安装到不同的显微镜系统。此外,由于细菌被固定在琼脂糖-盖玻片界面处,我们已经成功地追踪了高度运动的细菌,如铜绿假单胞菌,同时仍然允许形态变化(图 4,补充视频 4)。其他单细胞成像技术,例如"母机",将细胞限制在无法观察除细丝以外的形态变化的通道中36。
为了成功使用该协议,需要牢记一些关键步骤和参数。对于安片制备,彻底加热和熔化琼脂糖很重要,因为任何残留的琼脂糖晶体都会导致光衍射并影响图像质量。同样,必须注意将琼脂糖移液到腔室中,而不会引入气泡。为了确保琼脂糖垫的厚度保持一致并限制样品漂移,在成像开始之前,重要的是让琼脂糖垫在加湿、温控的外壳中平衡(通常为 15 分钟)。另一个可能导致图像质量不佳的因素是湿度控制:低湿度会导致琼脂糖垫脱水和收缩,而高湿度(或腔室中的样品加热不当)可能导致暖空气在样品上凝结并扭曲成像。由于冷凝而导致的延时成像效果不佳的示例可以在补充视频 8 中找到。
当前设置的一个限制是培养基不能更换,这妨碍了在抗生素处理之前、期间和之后连续跟踪单个细菌。我们预计,将琼脂糖垫与允许培养基交换的流动池或微流控装置耦合,可以在营养或环境变化期间跟踪种群。当前设计中另一个可以改进的参数是样品曝气。与需要使用蜡或油脂基密封剂密封垫16 的设置相比,可互换盖玻片培养皿的 O 形圈密封、螺帽设计可实现更好的样品通气。然而,密封室中的曝气可能仍然有限,并且可能不支持专性需氧菌的生长,尽管这仍有待测试。
我们在本文中介绍的延时成像样品制备方案可以跟踪数千种细菌对抗生素治疗的反应或从抗生素治疗中恢复。这种方法还具有高度的可推广性,并且在持久性生物学之外具有多种潜在应用。例如,琼脂糖垫和分隔器设置允许接种空间分离的细胞样品,但允许通过琼脂糖垫的扩散进行细胞间通讯。我们目前正在探索这种设置的潜力,以测试分泌产物的交换如何影响多物种群落中的细胞生长。我们预计该协议将为新研究人员提供延时显微镜的低准入门槛,并为经验丰富的微生物学家提供无限的变化来探索。
披露声明
作者没有需要声明的利益冲突。
致谢
我们感谢康涅狄格大学健康中心细胞分析和建模显微镜设施的 Susan Staurovsky 女士在显微镜实验方面的帮助。我们感谢 Mona Wu Orr 博士和葡萄球菌遗传学和代谢要点研讨会分别提供关于 铜绿假单胞 菌和 金黄色葡萄球菌克隆的方案和建议。这项工作得到了美国国立卫生研究院 (NIH;DP2GM146456-01 和 1R01AI167886-01A1 到 W.W.K.M.,1F30DE032598-01A1 到 PJH,以及 1F31DK136259-01A1 到 T.J.L.)。资助者在我们的实验设计或这份手稿的准备中没有作用。
材料
| Name | Company | Catalog Number | Comments |
| BaSiC | GitHub | https://github.com/marrlab/BaSiC | |
| Certified Molecular Biology Agarose | Biorad | 1613101 | |
| Fiji-ImageJ | NIH | https://imagej.net/software/fiji/downloads | |
| Interchangeable Coverglass Dish | Bioptechs | 190310-35 | 35 mm ICD for preparing agarose pads; comes with 30 mm (#1.5) coverslips |
| Lumencor Spectra 7 LED light engine | Lumencor | https://lumencor.com/products/spectra-light-engine | Spectra 7 LED light engine |
| MetaMorph | Molecular Devices | Premier version 7.10.5 | |
| pco.edge 4.2 bi sCMOS camera | Excelitas | https://www.excelitas.com/product/pcoedge-42-bi-usb-scmos-camera | sCMOS camera |
| PeCon live cell incubation chamber | peCon | https://www.pecon.biz/ | |
| Thomas Scientific Round cover glass, #1.5 thickness, 25 mm, 100 pack | Fisher Scientific | NC1272770 | 25 mm (#1.5) coverslips |
| Zeiss Axiovert 200M microscope | Zeiss | https://www.zeiss.com/microscopy/en/products/light-microscopes/widefield-microscopes/axiovert-for-materials.html | Inverted microscope with Plan-Apochromat 63x/1.40 Oil Ph3 M27 objective, Lumencor Spectra 7 LED light engine, and pco.edge 4.2 bi sCMOS camera (6.5 mm pixel size) |
参考文献
- Brauner, A., Fridman, O., Gefen, O., Balaban, N. Q. Distinguishing between resistance, tolerance and persistence to antibiotic treatment. Nat Rev Microbiol. 14, 320-330 (2016).
- Balaban, N. Q., et al. Definitions and guidelines for research on antibiotic persistence. Nat Rev Microbiol. 17 (7), 441-448 (2019).
- Conlon, B. P. Staphylococcus aureus chronic and relapsing infections: evidence of a role for persister cells. Bioessays. 36 (10), 991-996 (2014).
- Mulcahy, L. R., Burns, J. L., Lory, S., Lewis, K. Emergence of Pseudomonas aeruginosa strains producing high levels of persister cells in patients with cystic fibrosis. J Bacteriol. 192 (23), 6191-6199 (2010).
- Verstraete, L., Van den Bergh, B., Verstraeten, N., Michiels, J. Ecology and evolution of antibiotic persistence. Trends Microbiol. 30 (5), 466-479 (2021).
- Sett, A., Dubey, V., Bhowmik, S., Pathania, R. Decoding bacterial persistence: Mechanisms and strategies for effective eradication. ACS Infect Dis. 10 (8), 2525-2539 (2024).
- Mempin, R., et al. Release of extracellular ATP by bacteria during growth. BMC Microbiol. 13, 301 (2013).
- Lewis, K. Persister cells, dormancy and infectious disease. Nat Rev Microbiol. 5 (1), 48-56 (2006).
- Fauvart, M., De Groote, V. N., Michiels, J. Role of persister cells in chronic infections: clinical relevance and perspectives on anti-persister therapies. J Med Microbiol. 60 (pt 6), 699-709 (2011).
- Van den Bergh, B., Fauvart, M., Michiels, J. Formation, physiology, ecology, evolution and clinical importance of bacterial persisters. FEMS Microbiol Rev. 41, 219-251 (2017).
- Levin, B. R., Rozen, D. E. Non-inherited antibiotic resistance. Nat Rev Microbiol. 4 (7), 556-562 (2006).
- Davis, K. M., Isberg, R. R. Defining heterogeneity within bacterial populations via single cell approaches. Bioessays. 38 (8), 782-790 (2016).
- Hare, P. J., LaGree, T. J., Byrd, B. A., DeMarco, A. M., Mok, W. W. K. Single-cell technologies to study phenotypic heterogeneity and bacterial persisters. Microorganisms. 9 (11), 2277 (2021).
- de Jong, I. G., Beilharz, K., Kuipers, O. P., Veening, J. W. Live cell imaging of Bacillus subtilis and Streptococcus pneumoniae using automated time-lapse microscopy. J Vis Exp. 53, e3145 (2011).
- Hare, P. J., Gonzalez, J. R., Quelle, R. M., Wu, Y. I., Mok, W. W. K. Metabolic and transcriptional activities underlie stationary-phase Pseudomonas aeruginosa sensitivity to levofloxacin. Microbiol Spectr. 12 (1), e0356723 (2024).
- Young, J. W., et al. Measuring single-cell gene expression dynamics in bacteria using fluorescence time-lapse microscopy. Nat Protoc. 7 (1), 80-88 (2011).
- Pang, Y. Y., et al. agr-Dependent interactions of Staphylococcus aureus USA300 with human polymorphonuclear neutrophils. J Innate Immun. 2 (6), 546-559 (2010).
- Bose, J. L., Fey, P. D., Bayles, K. W. Genetic tools to enhance the study of gene function and regulation in Staphylococcus aureus. Appl Environ Microbiol. 79 (7), 2218 (2013).
- Schenk, S., Laddaga, R. A. Improved method for electroporation of Staphylococcus aureus. FEMS Microbiol Lett. 73, 133-138 (1992).
- Grosser, M. R., Richardson, A. R. Method for Preparation and Electroporation of S. aureus and S. epidermidis. The Genetic Manipulation of Staphylococci. Methods Molecular Biology. , (2016).
- Krausz, K. L., Bose, J. L. Bacteriophage Transduction in Staphylococcus aureus: Broth-Based Method. The Genetic Manipulation of Staphylococci. Methods Molecular Biology. , (2016).
- Olson, M. E. Bacteriophage Transduction in Staphylococcus aureus. The Genetic Manipulation of Staphylococci. Methods Molecular Biology. , (2016).
- Fey, P. D., et al. A genetic resource for rapid and comprehensive phenotype screening of nonessential Staphylococcus aureus genes. mBio. 4 (1), e00537-e00612 (2013).
- Coe, K. A., et al. Multi-strain Tn-Seq reveals common daptomycin resistance determinants in Staphylococcus aureus. PLoS Pathog. 15 (11), e1007862 (2019).
- Figurski, D. H., Helinski, D. R. Replication of an origin-containing derivative of plasmid RK2 dependent on a plasmid function provided in trans. Proc Natl Acad Sci U S A. 76 (4), 1648 (1979).
- Shee, C., et al. Engineered proteins detect spontaneous DNA breakage in human and bacterial cells. eLife. 2, e01222 (2013).
- Kowalska-Krochmal, B., Dudek-Wicher, R. The minimum inhibitory concentration of antibiotics: methods, interpretation, clinical relevance. Pathogens. 10 (2), 165 (2021).
- Robinson, J. L., Jaslove, J. M., Murawski, A. M., Fazen, C. H., Brynildsen, M. P. An integrated network analysis reveals that nitric oxide reductase prevents metabolic cycling of nitric oxide by Pseudomonas aeruginosa. Metab Eng. 41, 67-81 (2017).
- Wolff, J. A., MacGregor, C. H., Eisenberg, R. C., Phibbs, P. V. Isolation and characterization of catabolite repression control mutants of Pseudomonas aeruginosa PAO. J Bacteriol. 173 (15), 4700-4706 (1991).
- Batchelder, J. I., Taylor, A. J., Mok, W. W. K. Metabolites augment oxidative stress to sensitize antibiotic-tolerant Staphylococcus aureus to fluoroquinolones. mBio. , e0271424 (2024).
- Power, A. D., Mok, W. W. K. Agar and agarose used for Staphylococcus aureus biofilm cultivation impact fluoroquinolone tolerance. J Appl Microbiol. 135 (8), lxae191 (2024).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Peng, T., et al. A BaSiC tool for background and shading correction of optical microscopy images. Nat Commun. 8, 14836 (2017).
- Ducret, A., Quardokus, E., Brun, Y. V. MicrobeJ, a tool for high throughput bacterial cell detection and quantitative analysis. Nat Microbiol. 1 (7), 16077 (2016).
- Paintdakhi, A., et al. Oufti: an integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Mol Microbiol. 99 (4), 767-777 (2016).
- Wang, P., et al. Robust growth of Escherichia coli. Curr Biol. 20 (12), 1099-1103 (2010).
- Moffitt, J. R., Lee, J. B., Cluzel, P. The single-cell chemostat: an agarose-based, microfluidic device for high-throughput, single-cell studies of bacteria and bacterial communities. Lab Chip. 12 (8), 1487-1494 (2012).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。