Method Article
Imaging al microscopio a epifluorescenza time-lapse di fenotipi eterogenei di Pseudomonas aeruginosa e Staphylococcus aureus
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
In questo manoscritto, forniamo un protocollo completo per valutare la sopravvivenza agli antibiotici per Pseudomonas aeruginosa e Staphylococcus aureus, trasformando i plasmidi in P. aeruginosa e S. aureus per creare ceppi reporter e visualizzare varianti fenotipiche, come i persistenti, mediante microscopia a epifluorescenza time-lapse.
Abstract
La persistenza degli antibiotici è un fenomeno in cui un piccolo numero di cellule batteriche in una popolazione geneticamente suscettibile sopravvive al trattamento antibiotico che uccide le altre cellule geneticamente identiche. I persistenti batterici possono riprendere la replicazione una volta terminato il trattamento antibiotico e si ritiene comunemente che siano alla base del fallimento del trattamento clinico. Un recente lavoro che sfrutta la potenza della microscopia a fluorescenza time-lapse, in cui i batteri sono marcati con reporter trascrizionali fluorescenti, reporter traslazionali e/o coloranti per una varietà di caratteristiche cellulari, ha fatto progredire la nostra comprensione dei persistenti di Escherichia coli oltre ciò che potrebbe essere appreso dai test di sopravvivenza agli antibiotici a livello di popolazione. Tali approcci a singola cellula, piuttosto che saggi di popolazione di massa, sono essenziali per delineare i meccanismi di formazione dei persistenti, risposta al danno e sopravvivenza. Tuttavia, i metodi per studiare i persistenti in altre importanti specie patogene a questo livello di dettaglio rimangono limitati.
Questo studio fornisce un approccio adattabile per l'imaging time-lapse di Pseudomonas aeruginosa (un bastoncello gram-negativo) e Staphylococcus aureus (un cocco gram-positivo) durante il trattamento antibiotico e il recupero. Discutiamo gli approcci di genetica molecolare per introdurre i reporter fluorescenti in questi batteri. Utilizzando questi reporter, così come i coloranti, possiamo tracciare i cambiamenti fenotipici, le caratteristiche morfologiche e i destini delle singole cellule in risposta al trattamento antibiotico. Inoltre, siamo in grado di osservare i fenotipi dei singoli persistenti mentre rianimano dopo il trattamento. Nel complesso, questo lavoro funge da risorsa per coloro che sono interessati a monitorare la sopravvivenza e l'espressione genica di singole cellule trattate con antibiotici, compresi i persistenti, sia durante che dopo il trattamento, in patogeni clinicamente importanti.
Introduzione
I patogeni batterici possono eludere gli effetti degli antibiotici attraverso due meccanismi principali: la resistenza agli antibiotici, che comporta cambiamenti genetici, e la tolleranza fenotipica, che comporta cambiamenti non genetici. La resistenza agli antibiotici è un fenomeno geneticamente codificato che conferisce la capacità di una data cellula batterica non solo di sopravvivere, ma anche di replicarsi in presenza di un antibiotico1. La tolleranza fenotipica, che può comprendere batteri tolleranti agli antibiotici o persistenti agli antibiotici, si verifica quando le cellule resistono al trattamento antibiotico battericida senza acquisire la capacità di replicarsi in presenza di una concentrazione inibitoria dell'antibiotico 1,2. Ciò che differenzia la tolleranza dalla persistenza è che la tolleranza si riferisce alla capacità dell'intera popolazione di sopravvivere al trattamento, mentre la persistenza si riferisce a un sottoinsieme di una popolazione isogenica ma fenotipicamente eterogenea che sopravvive al trattamento antibiotico. Quando una coltura clonale viene trattata con antibiotici battericidi e i sopravvissuti che rimangono nella coltura vengono tracciati nel tempo su una scala log-lineare, una curva bifasica viene solitamente rilevata quando sono presenti persistenti. Su queste curve, la prima fase mostra che la maggior parte della popolazione viene uccisa in tempi relativamente brevi, e la seconda fase indica che una frazione persistente di antibiotici viene uccisa a un ritmo più lento o non viene uccisa affatto 1,2.
La persistenza degli antibiotici rappresenta un onere importante per i sistemi sanitari globali. Ad esempio, si ritiene che Staphylococcus aureus e Pseudomonas aeruginosa persisters, che sono al centro di questo articolo, causino infezioni antibiotico-recalcitranti, comprese le infezioni ricorrenti delle vie aeree nei pazienti con fibrosi cistica e le infezioni croniche delle ferite 3,4. Pertanto, è fondamentale chiarire ulteriormente la biologia cellulare persistente e i programmi fenotipici. Sebbene siano stati compiuti progressi nella comprensione di come i persistenti si formino e si rianimano, rimangono lacune di conoscenza critiche relative al coordinamento della riprogrammazione metabolica e degli eventi molecolari nelle singole cellule che sono alla base della persistenza 5,6,7,8.
Studiare efficacemente la persistenza si è rivelata una sfida tecnica. Poiché la persistenza è osservabile solo in un piccolo sottoinsieme di una popolazione batterica, le tecniche che campionano popolazioni batteriche di massa spesso non riescono a catturare informazioni biologiche rilevanti 1,2,8,9,10. Inoltre, poiché i cambiamenti fenotipici che sono alla base della persistenza sono transitori e non ereditabili, tracciare il destino delle cellule persistenti può essere complesso 1,8,9,10,11. Una volta che i persistenti batterici riprendono a crescere, possono dividersi e dare origine sia a persistenti che a non persistenti, il che rende impossibile l'arricchimento per le popolazioni di persister puri mediante coltura. Queste sfide evidenziano la necessità di tecniche in grado di soddisfare i seguenti criteri: 1) la capacità di catturare informazioni biologiche di singole cellule vive e 2) la capacità di essere utilizzate in tandem con coloranti fluorescenti, sonde, sensori e reporter che consentono di interrogare nel tempo i fenotipi di singole cellule in popolazioni eterogenee.
I recenti progressi nelle tecnologie a singola cellula hanno fornito una strada per studiare efficacemente l'eterogeneità batterica e superare questi ostacoli nello studio della persistenza12,13. Alcune di queste tecniche includono la microscopia a fluorescenza, la citometria a flusso/smistamento cellulare attivato da fluorescenza, la microfluidica e il sequenziamento dell'RNA a singola cellula12,13. Qui, descriviamo i protocolli per chiarire la fisiologia del persister a singola cellula utilizzando la microscopia time-lapse a epifluorescenza di ceppi reporter trascrizionali o traduzionali. La microscopia a fluorescenza è una tecnica potente che soddisfa i criteri per lo studio dei fenotipi persistenti, vale a dire la capacità di identificare quali singole cellule in una grande popolazione si propagano dopo la rimozione dell'antibiotico e possono quindi essere definite persistenti. Con l'introduzione delle tecnologie delle telecamere automatizzate e delle camere incubate, la cattura di cellule batteriche vive è ampiamente accessibile in tutto il campo della microbiologia. Fondamentalmente, la microscopia time-lapse offre la possibilità di visualizzare singole cellule in tempo reale, nel corso di ore e persino giorni, il che rende possibile tracciare i batteri prima, durante e dopo il trattamento antibiotico 14,15,16. Le intuizioni di queste indagini che sfruttano la microscopia time-lapse hanno un immenso potenziale per generare informazioni sui complessi meccanismi della biologia dei persistenti.
Protocollo
1. Generazione di ceppi reporter fluorescenti di S. aureus mediante trasformazione e trasduzione
NOTA: I ceppi reporter ospitano una proteina fluorescente per indicare l'espressione di un gene o di una proteina di interesse. I reporter trascrizionali presentano una copia duplicata della sequenza del promotore nativo per un gene di interesse a monte di una proteina fluorescente in modo che la fluorescenza aumenti all'aumentare dell'espressione di un gene di interesse. I reporter traslazionali collegano i frame di lettura aperti di una proteina fluorescente e di una proteina di interesse con un connettore peptidico flessibile. La visualizzazione dei ceppi reporter con la microscopia su cellule vive può rivelare se un determinato gene/proteina di interesse è associato a specifiche morfologie cellulari o destini cellulari (Figura 1, Video supplementare 1 e Video supplementare 2). Scegli una proteina fluorescente ottimizzata per il codone per S. aureus. Mentre questo studio utilizza sGFP da pCM29 donato dal Dr. Alexander Horswill, il Nebraska Transposon Mutant Library Genetic Toolkit contiene plasmidi contenenti sGFP, eYFP, eCFP, DsRed.T3 e eqFP65017,18 ottimizzati per il codone.
- Trasformare i plasmidi reporter in S. aureus RN4220.
NOTA: La seguente sezione del protocollo è stata adattata dal workshop Essentials of Staphylococcal Genetics and Metabolism presso l'University of Nebraska Medical Center19,20.- Innanzitutto, preparare l'elettrocompetente S. aureus RN4220. Preparare 200 ml di brodo B2 combinando 2 g di casamino acidi, 5 g di estratto di lievito, 2 ml di 10% K2HPO4, pH 7,5, 2 ml di soluzione di glucosio al 50% e 5 g di NaCl. Portare il volume a 100 ml con acqua ultrapura sterile. Mescolare brevemente a 37 °C per sciogliere i componenti e sterilizzare utilizzando un filtro per flaconi (dimensione dei pori di 0,22 μm).
- Inoculare S. aureus RN4220 da un brodo congelato conservato a -80 °C in glicerolo al 25% in 30 mL di brodo B2 in un pallone da 250 mL e incubare per 18 ore a 30 °C, agitando a 125 giri/min.
- Incubare due fiasche da 250 mL con 50 mL di brodo B2 ciascuna a 37 °C per una notte per preriscaldare il terreno per il giorno successivo.
- Il giorno successivo, misurare l'OD600 della coltura notturna. Aggiungere una quantità sufficiente di coltura notturna a ciascun pallone contenente 50 ml di brodo B2 per ottenere un OD600 finale di 0,25.
- Incubare a 37 °C, agitando a 250 giri/min, fino a quando l'OD600 raggiunge 0,35-0,4.
NOTA: Controllare l'OD600 circa 30 minuti dopo la subcoltura per assicurarsi che la coltura non cresca oltre 0,35-0,4. Se l'OD600 non raggiunge 0,35-0,4 entro 1 ora dalla subcoltura, abbandonare la procedura e riprovare il giorno successivo. Molto probabilmente i batteri sono cresciuti troppo durante la notte ed erano troppo in profondità nella fase stazionaria. - Una volta che la coltura raggiunge la fase di metà log, versare la coltura da ciascun pallone in una provetta conica sterile da 50 ml e pellettare le celle mediante centrifugazione a 4.000 x g per 10 minuti.
- Decantare il surnatante e risospendere il pellet in 1 mL di acqua sterile a temperatura ambiente (RT). Trasferire le cellule in una provetta da microcentrifuga.
- Centrifugare nuovamente le cellule a 21.000 x g per 30 s, rimuovere il surnatante e risospendere in 1 mL di acqua RT sterile. Ripetere la centrifugazione, la rimozione del surnatante e la risospensione in 1 mL di passaggi d'acqua altre due volte per un totale di tre lavaggi.
- Centrifugare le cellule, rimuovere l'acqua, quindi risospendere le cellule in 1 mL di RT 10% glicerolo sterile (in acqua, filtrato sterile) e lasciare in RT per 15 minuti.
- Pellettare le cellule mediante centrifugazione a 21.000 x g per 30 s, rimuovere il surnatante e risospendere le cellule in un altro 1 mL di glicerolo sterile, RT 10%. Queste celle sono ora elettrocompetenti.
- Aliquotare 70 μL di celle elettrocompetenti in provette per microcentrifuga con tappo a scatto e conservarle a -80 °C per un uso futuro.
- Per trasformare l'elettrocompetente S. aureus RN4220, scongelare un'aliquota di cellule sul ghiaccio. Aggiungere 1 μg di DNA plasmidico alle cellule, muovere il campione per mescolarlo e incubare su ghiaccio per 5 minuti. Quindi, elettroporare le cellule e lasciarle recuperare in 390 μL di terreno B2 per 1-2 ore a 37 °C, agitando a 225 giri/min e trasformanti su piastre su agar di soia triptico selettivo.
NOTA: La maggior parte dei vettori shuttle utilizzati in questo studio presenta una cassetta di resistenza all'ampicillina per la selezione in E. coli (100 μg/mL) e una cassetta di resistenza al cloramfenicolo (10 μg/mL) o all'eritromicina (Erm, 10 μg/mL) per la selezione in S. aureus. Molti ceppi di S. aureus che vengono abitualmente utilizzati in laboratorio sono resistenti all'Erm. Assicurarsi che un ceppo di interesse sia compatibile con l'antibiotico selettivo prima di utilizzare un determinato vettore shuttle. È importante che il volume di DNA aggiunto alle cellule sia ridotto al minimo (ad esempio, <5 μL) per prevenire la formazione di archi elettrici, che si verificano quando il campione è troppo conduttivo durante l'elettroporazione. La purificazione di alte concentrazioni (≥300 ng/μL) del DNA plasmidico da E. coli aiuterà a mantenere bassi i volumi.
- Propagare il batteriofago su trasformato S. aureus RN4220.
NOTA: La seguente sezione del protocollo è stata adattata dai metodi di Krausz e Bose21e Olson22. Due dei batteriofagi più comunemente usati per S. aureus trasduzione sono φ11 e 80α. La procedura per la raccolta di entrambi è la stessa, tranne per il fatto che otteniamo φ11 da S. aureus RN451 e 80α da S. aureus RN10359. φ11 è stato utilizzato con successo con ceppi comuni JE2 e HG003, tra gli altri, ed è utilizzato in questo protocollo23,24. Tuttavia, questi fagi potrebbero non trasdurre in modo efficiente isolati clinici che hanno diverse resistenze ai fagi. Fare riferimento alle pubblicazioni di Krausz e Bose21 e Olson22 per un metodo per preparare φ11 e determinare il titolo fagico.- Per trasdurre il plasmide reporter, preparare sei piastre di agar di soia triptica (TSA) con 5 mM di CaCl2 e la corretta concentrazione dell'antibiotico selettivo. La striscia S. aureus RN4220 si è trasformata con il plasmide reporter su una piastra TSA contenente l'antibiotico selettivo e l'assenza di CaCl2 e ha incubato durante la notte.
- Il giorno successivo, accendere un'incubatrice o un bagnomaria a 56 °C. Etichettare sei provette da 5 mL con tappo a scatto da 1 x 104 a 1 x 109 e aliquotare 40 μL di 500 mM di CaCl2 in ciascuna provetta con tappo a scatto.
- Sciogliere il TSA morbido (0,5% di agar) in un forno a microonde con il tappo allentato per evitare l'accumulo di pressione mantenendo la sterilità. Quindi, aliquotare 4 mL in ciascuna delle provette con tappo a scatto. Capovolgere le provette per mescolare, quindi metterle nell'incubatore a 56 °C per evitare che l'agar morbido si solidifichi.
- Diluire il brodo di fagi a 1 x 1010 PFU/mL in brodo di soia triptico (TSB) + 5 mM di CaCl2. Aggiungere 100 μl di 1 x 1010 PFU/mL di fago a una provetta da microcentrifuga contenente 900 μl di TSB + 5 mM di CaCl2 per una concentrazione finale di 1 x 109 PFU/mL. Quindi, diluire in serie in ulteriori provette da microcentrifuga contenenti 900 μL di TSB + 5 mM di CaCl2 fino a una concentrazione di 1 x 104 PFU/mL. Etichettare ciascuna delle sei piastre di agar antibiotico selettivo TSA + 5 mM CaCl+ con una diluizione seriale: da 1 x104 a 1 x 109 PFU/mL.
- Risospendere per una notte S. aureus RN4220 contenente il plasmide in 1 mL di TSB + 5 mM di CaCl2. Rimuovere la provetta con tappo a scatto 109 dall'incubatore e aggiungere rapidamente 10 μL di RN4220 risospeso contenente il plasmide e 100 μL di diluizione di fagi 1 x 109 PFU/mL, capovolgere per miscelare e versare il contenuto sulla corrispondente piastra di agar antibiotico selettivo TSA + 5 mM CaCl2 .
- Eseguire questa procedura per le restanti diluizioni dei fagi e le rispettive piastre. Lasciare solidificare l'agar morbido sulle piastre, quindi incubare con l'agar morbido rivolto verso l'alto per una notte a 37 °C.
- Il giorno successivo, assicurarsi che le placche (aree di spazio libero sul prato batterico) siano visibili sulle piastre (Figura 2A). Le placche rappresentano aree di lisi in cui i fagi hanno infettato con successo RN4220 e alcuni fagi avranno assorbito il plasmide di interesse. Selezionare la piastra con il fago meno diluito che ha una lisi quasi confluente (esempio: 1 x 109 PFU/mL fago) e le piastre che hanno ricevuto le due diluizioni inferiori (in questo caso, 1 x 108 PFU/mL e 1 x 107 PFU/mL, che dovrebbero avere entrambe una lisi quasi confluente).
- Aggiungere 3 mL di TSB + 5 mM di CaCl2 alla meno diluita di queste piastre (1 x 109 PFU/mL, per questo esempio). Utilizzare un divaricatore di cellule sterile a forma di L per raschiare delicatamente lo strato di agar morbido dall'agar normale (Figura 2B). Usa il divaricatore cellulare per distruggere il più possibile l'agar per facilitare il rilascio del fago dall'agar morbido.
- Una volta che l'agar morbido sulla prima piastra è stato completamente distrutto, versare delicatamente l'agar morbido e l'impasto di brodo sulla piastra successiva (in questo caso, 1 x 108 PFU/mL). Raschiare e rompere l'agar morbido su questa piastra, quindi versare l'impasto risultante sulla piastra finale e ripetere.
- Dopo aver preparato l'impasto di agar morbido da tutte e tre le piastre, versare delicatamente l'impasto in una provetta conica da 50 ml. Pipettare delicatamente per interrompere ulteriormente l'agar morbido, ma evitare la formazione di bolle. Non vortice, poiché potrebbe danneggiare il fago e diminuire il titolo.
- Centrifugare l'impasto di agar morbido a 4.000 x g per 10 minuti a RT. Versare delicatamente il surnatante in una siringa collegata a un filtro da 0,45 μm.
NOTA: È essenziale versare delicatamente ed evitare di far entrare pezzi di agar nel filtro. Questi pezzi ostruiscono il filtro e impediscono di filtrare il surnatante rimanente, diminuendo il titolo. - Determinare il titolo del lisato fagico risultante utilizzando RN422021 wild-type.
- Introdurre plasmidi reporter in ceppi di S. aureus di interesse per trasduzione.
- Preparare otto piastre TSA contenenti 500 μg/mL di citrato di sodio e la concentrazione appropriata di antibiotico selettivo. Strisciare densamente il ceppo di S. aureus ricevente desiderato sul TSA semplice e incubare per una notte a 37 °C.
- Il giorno successivo, tamponare la piastra e risospendere il ceppo ricevente in 1 mL di TSB + 5 mM di CaCl2.
- Diluire il phage stock contenente il plasmide reporter desiderato a 1 x1010 PFU/mL.
- Miscelare 1,5 mL di TSB + 5 mM di CaCl2, 0,5 mL di ceppo ricevente risospeso e 0,5 mL di stock fagico diluito in una provetta conica da 50 mL. Incubare a 37 °C, agitando a 225 giri/min, per 20 min.
NOTA: Tempi di incubazione più lunghi non sono raccomandati perché probabilmente diminuiranno l'efficienza di trasduzione quando il fago inizia a lisare le cellule riceventi. - Subito dopo, aggiungere 1 mL di citrato di sodio 0,02 M (in acqua) freddo e filtrato sterile nella provetta conica e centrifugare a 4.000 x g per 10 minuti a 4 °C. Quindi, risospendere il pellet in 1 mL di citrato di sodio 0,02 M ghiacciato.
- Distribuire 100 μL di aliquote su ciascuna delle sei piastre TSA contenenti 500 μg/mL di citrato di sodio e la concentrazione appropriata di antibiotico selettivo da selezionare per i trasduttori. Incubare per una notte a 37 °C.
- Il giorno successivo, strisciare le singole colonie dalle piastre di trasduzione sulle restanti due piastre TSA contenenti 500 μg/mL di citrato di sodio e la concentrazione appropriata di antibiotico selettivo. Incubare per una notte a 37 °C.
NOTA: Questo passaggio su piastre aggiuntive di citrato di sodio aiuta a ridurre il titolo del fago attorno alle cellule trasdotte, prevenendo la lisi22. - Prelevare le singole colonie dalla piastra di striatura, far crescere le cellule fino alla fase esponenziale in TSB contenente l'antibiotico selettivo e conservare il glicerolo al 25% a -80 °C per un uso futuro.
2. Generazione di ceppi reporter fluorescenti di P. aeruginosa mediante coniugazione
NOTA: Lo spostamento di un plasmide reporter in P. aeruginosa da un ceppo di clonazione di E. coli può essere effettuato mediante accoppiamento triparentale25. Sono necessari un ceppo donatore di E. coli che trasporta il plasmide di interesse (che dovrebbe contenere un oriT per il trasferimento coniugale), E. coli HB101 + pRK2013 - un ceppo helper per facilitare la coniugazione (utilizzare 50 μg/mL di kanamicina per il mantenimento del plasmide pRK2013) e il ceppo ricevente di P. aeruginosa che riceverà il plasmide. Per questo esempio, il plasmide di interesse ha un marcatore di resistenza alla tetraciclina (Tet), quindi qualsiasi coltura di E. coli con il plasmide avrà bisogno di 10 μg/mL di Tet, e P. aeruginosa con il plasmide avrà bisogno di 75 μg/mL di Tet per la selezione26.
- Inoculare i batteri provenienti da ceppi congelati conservati a -80 °C in glicerolo al 25% nel brodo di lisogenia (LB) con l'antibiotico appropriato per la crescita notturna, agitando a 225 giri/min a 37 °C.
- Il giorno successivo, distribuire 200 μl di ciascuna coltura liquida durante la notte sulla piastra di agar LB con l'antibiotico selettivo appropriato. A seconda del numero di accoppiamenti di trasformazione da effettuare, stendere più piastre per i ceppi helper e donatore. Si consiglia una piastra di aiutante/donatore per trasformazione. Incubare le piastre per una notte a 37 °C per ottenere prati densi di ogni ceppo.
- Il giorno successivo, raccogli i prati di crescita tamponando ogni superficie della piastra con un batuffolo di cotone sterile e strofinandolo lungo l'interno di una provetta da microcentrifuga. Centrifugare brevemente ciascuna provetta (pulsare per 5 s) per raccogliere le cellule sul fondo della provetta. Risospendere ogni pellet di cella in 200 μL di LB.
NOTA: Le risospensioni dovrebbero apparire molto spesse, opache e prive di grumi. - In nuove provette da microcentrifuga per ogni accoppiamento, combinare 5 μL di ceppo di P. aeruginosa ricevente, 160 μL di ceppo di E. coli donatore con il plasmide di interesse e 160 μL di E. coli helper HB101 + pRK2013. Miscelare delicatamente, pipettando su e giù, quindi posizionare 50 μl di miscela su una piastra di agar LB pre-essiccata. Inoltre, posizionare 20 μl di ogni singolo ceppo nell'agar LB per i controlli. Lasciare asciugare completamente le macchie nella piastra, quindi incubare a 37 °C per 3-6 ore.
NOTA: La pre-essiccazione dell'agar LB impedisce che la macchia da 50 μL si diffonda sulla superficie e quindi costringe le cellule a interagire all'interno di un'area più piccola. - Dopo l'incubazione, far scorrere delicatamente la punta di una pipetta sul punto di accoppiamento per trasferire alcune cellule in una nuova provetta per microcentrifuga. Centrifugare brevemente per raccogliere le cellule sul fondo della provetta. Risospendere in 200 μL di LB e placcare l'intera risospensione in una piastra di agar LB + Irgasan (Igr; 25 μg/mL) + Tet (75 μg/mL).
NOTA: P. aeruginosa è intrinsecamente resistente all'Igrasan, e il plasmide di interesse contiene il marcatore di resistenza Tet, quindi solo P. aeruginosa trasformata con successo dovrebbe essere in grado di crescere sulle piastre LB + Irgasan + Tet. A seconda dell'efficienza della trasformazione, la risospensione e la placcatura dell'intero punto di accoppiamento possono provocare un prato di trasformanti il giorno successivo. Per ottenere colonie individuali, (i) risospendere l'intero punto di accoppiamento in un volume maggiore di LB e quindi piastrarne 200 μL o (ii) trasferire una piccola porzione del punto di accoppiamento in una provetta da microcentrifuga e quindi risospendere in 200 μL e piastrare. - Con i punti di controllo di ogni singolo ceppo, far scorrere delicatamente una parte del punto di cellule dense su un puntale di pipetta, trasferire in una nuova provetta da microcentrifuga, risospendere in 200 μL di LB, quindi posizionare 50 μL su una piastra di agar LB + Irgasan + Tet (è possibile posizionare più punti di controllo sulla stessa piastra di agar). Lascia asciugare completamente le macchie. Incubare tutte le piastre per 16-20 ore durante la notte a 37 °C.
NOTA: Per gli spot di controllo, l'obiettivo è verificare che la piastra antibiotica sia selettiva per i trasformanti di P. aeruginosa . Se i punti di controllo sono troppo concentrati quando vengono individuati sulle piastre di agar LB + Irgasan + Tet, sarà difficile determinare se la selezione ha funzionato il giorno successivo perché ci sarà un alone di cellule morte che potrebbero essere scambiate per crescita. - Il giorno successivo, confermare la selettività antibiotica delle piastre LB + Irgasan + Tet notando la mancanza di crescita cellulare per i singoli punti di controllo del ceppo.
NOTA: Le colonie che appaiono sulla piastra di accoppiamento triparentale LB + Igrasan + Tet sono trasformanti di P. aeruginosa che trasportano il plasmide di interesse. - Facoltativo raccogliere e striare le colonie su LB + Irgasan + Tet agar e crescere durante la notte per eliminare l'eccesso di E. coli morto dalla miscela di accoppiamento.
- Raccogliere le singole colonie, coltivare per 3-6 ore in LB + Tet liquido (75 μg/mL) fino a quando non diventano visibilmente torbide, quindi conservare le scorte congelate in glicerolo al 25% (500 μL di coltura cellulare + 500 μL di glicerolo al 50% in acqua). Verificare che i cloni vengano trasformati con successo mediante PCR a base di colonia seguita da Sanger o sequenziamento dell'intero plasmide.
3. Determinazione delle dosi di antibiotici per i saggi persistenti
NOTA: Per selezionare una dose di un determinato antibiotico con cui trattare la popolazione batterica per gli esperimenti persistenti, misurare prima la concentrazione inibitoria minima (MIC) dell'antibiotico rispetto al ceppo batterico di interesse. Ciò può essere ottenuto utilizzando il metodo di microdiluizione in brodo, un approccio approvato dal Clinical and Laboratory Standards Institute (CLSI), o il test dell'epsilometro (E-test), che viene eseguito utilizzando strisce reattive con una gamma di dosi di antibiotici27. Una volta determinata la MIC, scegliere almeno cinque concentrazioni dell'antibiotico che vanno da 1 a 100 volte la MIC per il trattamento cellulare.
- Inoculare i batteri provenienti da ceppi congelati conservati a -80 °C in glicerolo al 25% in 2 mL di MHB CATION-ADJUSTED (CA-MHB) o in un altro terreno ricco di sostanze nutritive. Far crescere le cellule per circa 4 ore a 37 °C con agitazione a 250 giri/min prima di trasferire 125 μL di coltura in 25 mL di terreno fresco e chimicamente definito in un pallone di Erlenmeyer da 250 mL sfalsato. Far crescere i batteri per 16 ore (fino alla fase stazionaria) a 37 °C, agitando a 250 giri/min.
NOTA: Il terreno salino basale (BSM) con succinato come unica fonte di carbonio viene tipicamente utilizzato per gli esperimenti di P. aeruginosa e il terreno ricco chimicamente definito viene tipicamente utilizzato per gli esperimenti di S. aureus 28,29,30,31. - La mattina successiva, preparare 100 scorte di antibiotici (nei rispettivi solventi) per l'intervallo desiderato di concentrazioni di sovra-MIC. Aggiungere 10 μl di ciascuna diluizione alle singole provette.
- Misurare l'OD600 della coltura notturna per confermare la torbidità in fase stazionaria. Diluire in serie 10 μL di coltura e disporre delle diluizioni su piastre di agar nutritive, come l'agar LB, l'agar CA-MHB o la TSA, per determinare le unità formanti colonie (CFU) prima del trattamento antibiotico.
- Per il trattamento antibiotico, erogare 1 mL di aliquote della coltura nelle provette contenenti 10 μL di concentrazioni di antibiotico 100x. Incubare i campioni a 37 °C, agitando a 250 giri/min, per una durata sufficiente a uccidere i non persistenti nella popolazione, lasciando i persistenti come uniche cellule formanti colonie.
NOTA: La durata del trattamento può variare a seconda dell'esperimento e del ceppo batterico da testare. Tipicamente S. aureus viene trattato per 5 ore o 7 ore e P. aeruginosa per 7 ore o 24 ore. In generale, è possibile utilizzare qualsiasi punto temporale nella seconda fase del test di sopravvivenza tempo-dipendente perché ci si aspetta che le cellule rimanenti nella seconda fase siano persistenti. - Dopo il trattamento antibiotico, trasferire 100 μL di cellule dalle provette alle provette per microcentrifuga contenenti 900 μL di soluzione salina tamponata con fosfato sterile (PBS). Pellettare le celle mediante centrifugazione a RT a 21.000 x g per 3 min. Rimuovere 900 μl di surnatante e risospendere il pellet in 900 μl di PBS sterile. Ripetere la fase di lavaggio almeno una volta per ridurre gli antibiotici residui a livelli inferiori alla MIC.
- Diluire in serie la coltura 10 volte (sei volte) e impiattare 10 μl di ciascuna diluizione su piastre di agar nutritive. Incubare le piastre a 37 °C per una notte.
NOTA: Utilizzare piastre a 96 pozzetti a fondo tondo non trattate per la diluizione seriale e piastre su piastre di agar quadrate utilizzando una pipetta multicanale. - Il giorno successivo, contare le colonie ad ogni concentrazione di antibiotico. Tracciare la frazione di sopravvivenza (CFU alla fine del trattamento/CFU prima del trattamento) rispetto alla concentrazione su una scala log-lineare. Per scegliere una concentrazione di farmaco per i futuri saggi persistenti, selezionare una concentrazione nella seconda fase della curva bifasica (Figura 3).
4. Imaging delle cellule durante il trattamento antibiotico o il recupero
- Preparazione di tamponi di agarosio e campioni cellulari per l'imaging time-lapse
NOTA: Il seguente protocollo di preparazione dei campioni è stato sviluppato come alternativa facile da usare ed economica ai tradizionali metodi "sandwich" di agarosio14,16. L'uso di un coprioggetto intercambiabile evita la necessità di grasso o smalto per sigillare il campione, il che potrebbe limitare ulteriormente l'aerazione del campione. L'agarosio viene inserito nella capsula intercambiabile del vetrino coprioggetti con il vetrino coprioggetti già in posizione, creando superfici di agarosio planari affidabili rispetto alle preparazioni di tamponi di agarosio a mano libera. Nel complesso, questo metodo ha permesso di ottenere campioni di imaging con una migliore messa a fuoco in tutto il campo visivo e per durate più lunghe grazie all'umidificazione stabile. Il metodo qui descritto viene utilizzato per visualizzare i batteri durante il trattamento antibiotico (Figura 4, Video supplementare 3e Video supplementare 4). Per visualizzare i persistenti mentre rianimano e si risvegliano dopo il trattamento antibiotico (Figura 5, Video supplementare 5e Video supplementare 6), preparare i tamponi di agarosio con terreni di coltura freschi anziché con terreni esauriti e non aggiungere antibiotici ai tamponi (ad eccezione di quelli aggiunti per il mantenimento dei plasmidi/la selezione degli antibiotici).- Posizionare un vetrino coprioggetti sterile da 30 mm (spessore #1,5) sul fondo della base in acciaio inossidabile di un coprioggetto intercambiabile ("camera"). Infilare delicatamente l'inserto in policarbonato nella base in modo che il vetrino coprioggetti da 30 mm formi la base della camera e sigillarlo in posizione comprimendo l'O-ring in silicone attaccato. Ripetere questo passaggio per preparare una camera duplicata.
NOTA: Si raccomanda di preparare dei duplicati per ogni esperimento nel caso in cui la preparazione di una delle camere non sia ottimale. Un divisore personalizzato stampato in 3D è posizionato contro il vetrino coprioggetti da 30 mm per prevenire la contaminazione incrociata di cellule mobili come P. aeruginosa e per fornire punti di riferimento per le posizioni dei campioni. Il file STL per la stampa 3D è disponibile nel file supplementare 1. - Preparare l'agarosio all'1,5% in una provetta conica da 50 mL utilizzando il terreno di coltura scelto. Agitare delicatamente per mescolare.
NOTA: Per l'imaging di cellule in fase stazionaria durante il trattamento, come terreno di base viene utilizzato un terreno esaurito privo di cellule proveniente da una coltura notturna in fase stazionaria. Tipicamente, il terreno di agarosio viene preparato come 0,105 g di agarosio in 7 ml di terreno per la preparazione di due camere; ogni camera richiede 2 mL di terreno di agarosio e l'eccesso è utile per evitare bolle d'aria durante il pipettaggio. - Posizionare il conico da 50 ml in un bicchiere di vetro o in un supporto adatto al microonde (assicurarsi che il tappo sia allentato). Microonde alla massima potenza, fermandosi ogni 3-4 s per far roteare e mescolare. Mettere in pausa e mescolare spesso per evitare che la miscela di agarosio trabocchi.
- Dopo ~1 min tempo di riscaldamento totale, controllare se c'è dell'agarosio visibile che non si è sciolto nella miscela. Se la miscela è uniformemente limpida, lasciare raffreddare brevemente il terreno di agarosio a ~60 °C prima di aggiungere ulteriori farmaci o coloranti. Agitare delicatamente per amalgamare ed evitare la formazione di bolle d'aria.
NOTA: Lo ioduro di propidio (16 μM per P. aeruginosa o 1,6 μM per S. aureus) può essere aggiunto per visualizzare quando le cellule perdono la vitalità.
- Dopo ~1 min tempo di riscaldamento totale, controllare se c'è dell'agarosio visibile che non si è sciolto nella miscela. Se la miscela è uniformemente limpida, lasciare raffreddare brevemente il terreno di agarosio a ~60 °C prima di aggiungere ulteriori farmaci o coloranti. Agitare delicatamente per amalgamare ed evitare la formazione di bolle d'aria.
- Muovendosi rapidamente per evitare che il terreno di agarosio si solidifichi, pipettare 2 mL di terreno di agarosio nella camera con il vetrino coprioggetti da 30 mm. Stendere delicatamente un vetrino coprioggetti sterile da 25 mm (spessore #1,5) sul terreno di agarosio nell'apertura superiore della camera; Questo aiuta a prevenire la disidratazione del tampone di agarosio e garantisce una superficie superiore piatta dell'agarosio per un imaging ottimale a contrasto di fase. Lasciare solidificare il tampone per 1-2 ore.
- Dopo che il tampone si è solidificato, preparare le cellule da sottoporre all'imaging diluendole a una densità adeguatamente sparsa in PBS per la visualizzazione di singole cellule. Qui, le celle vengono diluite a OD600 0,01-0,05.
- Sul vetrino coprioggetti da 25 mm, utilizzare un pennarello indelebile a punta fine per contrassegnare le posizioni/identità di ciascun campione seminato sul tampone. Capovolgere la camera in modo che l'anello di base in acciaio inossidabile sia rivolto verso l'alto e tenere con cura l'inserto in policarbonato sottostante mentre si svita la base. Posizionare la base in acciaio inossidabile di lato. Far scivolare con cautela il vetrino coprioggetti da 30 mm dal tampone e gettarlo.
NOTA: Rimuovere il vetrino coprioggetti da 30 mm facendolo scorrere orizzontalmente e fare attenzione a non far rientrare la superficie dell'agarosio. Inoltre, evitare di lavorare sulla superficie esposta dell'agarosio: la polvere che cade su di essa influirà sulla qualità dell'immagine e può potenzialmente contaminare il tampone. - Utilizzando i segni sul vetrino coprioggetti da 25 mm come guida, individuare 5 μl di cellule diluite nelle rispettive posizioni sul tampone di agarosio. Aggiungere tre spot da 5 μl (regolarli secondo necessità, ad es. 4 spot da 4 μl, ecc.). Una volta che le macchie si sono asciugate completamente, posizionare leggermente un nuovo vetrino coprioggetti sterile da 30 mm centrato sul tampone.
- Tenere l'inserto in policarbonato con una mano e, con l'altra, reinserire lentamente la base in acciaio inossidabile sopra il nuovo vetrino coprioggetti. L'infilatura fino a quando non è ben serrata con le dita è sufficiente per comprimere l'O-ring in silicone e sigillare la camera.
NOTA: Fare attenzione a non stringere eccessivamente: se il vetrino coprioggetti da 30 mm contro la superficie del campione viene compresso dalla base in acciaio inossidabile e ruotato ripetutamente, le celle possono diffondersi in uno schema radiale, causando potenzialmente la contaminazione incrociata del campione. - Una volta sigillata la camera, osservare se l'agarosio è a contatto con la superficie del vetrino coprioggetti da 30 mm; A questo punto, di solito c'è un po', ma non completamente, contatto superficiale. Utilizzare l'estremità smussata di un paio di pinzette o di uno strumento simile per premere delicatamente contro il vetrino coprioggetti da 25 mm fino a quando l'agarosio tocca il vetrino coprioggetti da 30 mm in modo uniforme su tutta la superficie.
ATTENZIONE: Questo passaggio potrebbe causare la rottura del vetrino coprioggetti da 25 mm se viene utilizzata una forza eccessiva. - Una volta che l'agarosio è ben premuto contro il vetrino coprioggetti da 30 mm in modo che non rimangano grandi bolle d'aria, il tampone è pronto per essere riprodotto.
- Posizionare un vetrino coprioggetti sterile da 30 mm (spessore #1,5) sul fondo della base in acciaio inossidabile di un coprioggetto intercambiabile ("camera"). Infilare delicatamente l'inserto in policarbonato nella base in modo che il vetrino coprioggetti da 30 mm formi la base della camera e sigillarlo in posizione comprimendo l'O-ring in silicone attaccato. Ripetere questo passaggio per preparare una camera duplicata.
- Imposta il microscopio e l'immagine.
- Preparare i controlli ambientali del microscopio e della camera di imaging. Impostare le temperature dell'incubatore a tavolino e dell'incubatore a camera grande a 37 °C e accendere l'umidificatore a camera. Posizionare uno dei preparati di tampone di agarosio nell'incubatore del tavolino e chiudere la camera di incubazione per consentire al campione di equilibrarsi. A questo punto, il coperchio umidificatore dell'incubatore stage-top viene lasciato aperto per evitare la formazione di condensa sul vetrino coprioggetti da 25 mm della camera.
NOTA: Il restringimento/espansione del pad dovuto alle variazioni di temperatura può causare una deriva tra i punti temporali che va oltre l'intervallo di scansione dell'algoritmo di messa a fuoco automatica. È fondamentale equilibrare il tampone di agarosio a una temperatura stabile prima dell'imaging, che in genere richiede almeno 15 minuti. Un riscaldamento sufficiente impedisce inoltre la formazione di condensa sul vetrino coprioggetto superiore quando si posiziona il coperchio dell'umidificatore. I componenti hardware del sistema di imaging erano (Tabella dei materiali): camera di incubazione di cellule vive, microscopio invertito con obiettivo Plan-Apochromat 63x/1.40 Oil Ph3 M27, motore di luce LED Spectra 7 e fotocamera sCMOS (dimensione pixel 6,5 μm). Il controllo dei componenti motorizzati del microscopio e l'acquisizione delle immagini vengono effettuati utilizzando un'applicazione software di analisi al microscopio. - Preparare il software per l'acquisizione delle immagini. Impostate l'algoritmo di messa a fuoco automatica integrato di MetaMorph (software di analisi al microscopio) per utilizzare il canale di fase durante l'acquisizione multidimensionale.
- Nella scheda Stage , impostare più posizioni del tavolino per ciascun campione, puntando a campi visivi in cui la densità delle celle è distribuita uniformemente. Nella scheda Timelapse , impostare la durata e la frequenza desiderate (intervallo di tempo) delle immagini da scattare. Nelle schede Lunghezze d'onda , impostare i canali desiderati per l'acquisizione e regolare i tempi di esposizione in base all'intensità del segnale dei campioni.
NOTA: Le immagini di ciascuna posizione vengono scattate ogni 10 minuti per un massimo di 24 ore. Per l'imaging a fluorescenza dei coloranti/fluorofori menzionati in questo articolo, sono state utilizzate le seguenti impostazioni di eccitazione del motore luminoso: ioduro di propidio (Cy3; 555/15 nm) e GFP (GFP/FITC; 470/24 nm). Il tempo di esposizione tipico all'eccitazione della fluorescenza utilizzato è di 100 ms. Vengono utilizzati il set di filtri 15 (Beamsplitter FT580, Emission LP590) per lo ioduro di propidio e il set di filtri 44 (Beamsplitter FT500, Emission BP 530/50) per GFP.
- Nella scheda Stage , impostare più posizioni del tavolino per ciascun campione, puntando a campi visivi in cui la densità delle celle è distribuita uniformemente. Nella scheda Timelapse , impostare la durata e la frequenza desiderate (intervallo di tempo) delle immagini da scattare. Nelle schede Lunghezze d'onda , impostare i canali desiderati per l'acquisizione e regolare i tempi di esposizione in base all'intensità del segnale dei campioni.
- Dopo che il tampone di agarosio si è riscaldato nella camera, aggiungere il coperchio umidificatore all'incubatore da palco. Ciò contribuirà a prevenire la disidratazione e lo spostamento del pad durante l'imaging.
- Con il coperchio dell'umidificatore inserito, passare dal contrasto di fase a quello di interferenza differenziale (DIC) e regolare il condensatore e il diaframma di apertura del microscopio per una corretta illuminazione Köhler.
- Dopo la regolazione, tornare alla fase. Passare a ciascuna posizione del tavolino, regolare la messa a fuoco e reimpostare la posizione del tavolino sul nuovo piano focale. Le impostazioni sono ora a posto per iniziare l'imaging.
NOTA: È importante rivedere ogni posizione dello stadio prima di iniziare l'acquisizione per assicurarsi che il pad non si sia spostato. Se le celle non sono a fuoco quando vengono rivisitate in una determinata posizione dello stage, rifocalizzare su di esse e ignorare la posizione dello stage precedente. L'impostazione dell'algoritmo di messa a fuoco automatica utilizza la posizione focale z dall'ultimo punto temporale ± 3 μm come intervallo di ricerca, quindi se il pad si sposta di più di 3 μm, l'algoritmo non riuscirà a mettere a fuoco correttamente. - Avviare l'acquisizione multidimensionale facendo clic sul pulsante Acquisisci .
- Al termine dell'esperimento, compila le singole immagini di ciascun canale in una pila per visualizzarle come video o per altre analisi. A tale scopo, utilizzare MetaMorph o ImageJ.
- Per compilare le immagini in MetaMorph (preferibile), nell'app Review Multi Dimensional Data , selezionare i canali/lunghezze d'onda di interesse e selezionare tutti i punti temporali per una determinata posizione del palco. Fai clic su Carica immagini. Appariranno finestre per ogni canale/lunghezza d'onda. Salva ogni compilation con il rispettivo nome del canale come file .tiff.
- Per eseguire la compilazione in ImageJ, aprire i file per tutti i punti temporali per una posizione dello stage e un canale. Compila utilizzando le immagini per impilare.
- Preparare i controlli ambientali del microscopio e della camera di imaging. Impostare le temperature dell'incubatore a tavolino e dell'incubatore a camera grande a 37 °C e accendere l'umidificatore a camera. Posizionare uno dei preparati di tampone di agarosio nell'incubatore del tavolino e chiudere la camera di incubazione per consentire al campione di equilibrarsi. A questo punto, il coperchio umidificatore dell'incubatore stage-top viene lasciato aperto per evitare la formazione di condensa sul vetrino coprioggetti da 25 mm della camera.
5. Creazione di video time-lapse utilizzando Fiji/ImageJ
NOTA: Fiji (Fiji è semplicemente ImageJ) è un software disponibile gratuitamente per l'elaborazione e l'analisi delle immagini che può essere scaricato qui: "https://imagej.net/software/fiji/downloads32"32. Fiji/ImageJ2 1.54f è stato utilizzato per i metodi di elaborazione delle immagini descritti di seguito.
- Correzione dell'ombreggiatura dello stack di immagini del canale di fase utilizzando BaSiC33. Apri lo stack di immagini di fase desiderato nelle Fiji, quindi seleziona BaSiC dalla scheda Plugin . Lo stack con correzione dell'ombreggiatura verrà visualizzato in una finestra separata denominata Corrected:ImageName.
NOTA: BaSiC.jar può essere scaricato qui: "https://github.com/marrlab/BaSiC33"33. Segui le istruzioni dello sviluppatore per installare correttamente BaSiC nelle Fiji. - Unisci lo stack di canali di fase con correzione dell'ombra con qualsiasi altro canale/lunghezza d'onda utilizzando Image > Color > Unisci canali. Regola lo sfondo e l'intensità del segnale con Image > Regola > luminosità/contrasto per ciascun canale. Salvare il file unito come .tiff.
- Quindi, stabilizza le immagini utilizzando la deviazione 3D corretta. Con lo stack con correzione dell'ombreggiatura selezionato, vai su Plug-in > Registrazioni > Correggi deviazione 3D. Nella finestra di dialogo che si apre, impostare il canale per la registrazione sul numero di canale corrispondente allo stack di fasi. Lo stack risultante dopo la correzione sarà intitolato punti temporali registrati. Ritaglia il campo visivo desiderato, quindi salva il file corretto come .tiff.
NOTA: Se MetaMorph è stato utilizzato per creare la pila di immagini compilata, allora c'è un ulteriore passaggio di elaborazione: vai su Proprietà > immagine e scambia i numeri nei campi Sezioni (z) e Fotogrammi (t). La correzione della deriva ora può interpretare correttamente ogni fotogramma come un punto temporale e non come una fetta di uno z-stack. Il file di .tiff con correzione dell'ombra e della deriva può essere utilizzato nelle successive analisi delle immagini. Sono disponibili molti pacchetti software per misurare l'intensità del segnale fluorescente, quantificare le caratteristiche morfologiche, tracciare il destino delle singole cellule e altro ancora. Due programmi comunemente usati sono il plug-in MicrobeJ per Fiji e Oufti34,35. - Aggiungi timestamp ed etichette di testo alla pila utilizzando l'opzione Pile di immagini > > etichetta.
- Per aggiungere una barra della scala, per prima cosa, conosci la dimensione dei pixel della fotocamera e l'ingrandimento dell'obiettivo del microscopio. Calcola il rapporto pixel:micron come dimensione dei pixel divisa per l'ingrandimento. Nella finestra di dialogo Analizza > imposta scala , immettere il rapporto pixel:micron nel campo della distanza nota . Quindi, aggiungi una barra di scala alla pila con Strumenti di analisi > > barra di scala.
NOTA: La scala precisa di un microscopio deve essere calibrata utilizzando un micrometro da tavolino. Tuttavia, l'obiettivo del microscopio e la fotocamera utilizzati possono fornire una stima approssimativa della scala. Ad esempio, la fotocamera PCO sCMOS utilizzata qui ha una dimensione dei pixel di 6,5 x 6,5 μm2 e l'obiettivo 63x è stato utilizzato per l'imaging, quindi la dimensione dei pixel divisa per l'ingrandimento è 6,5/63 = 0,1032 μm per pixel. Immettere 0,1032 nel campo della distanza nota per la finestra di dialogo Imposta scala . - Per esportare la pila di immagini finita come video che può essere riprodotto in QuickTime Player, Microsoft PowerPoint, ecc., salva come file .avi.
Risultati
Il successo dell'introduzione di plasmidi reporter in P. aeruginosa e S. aureus è indicato dalla crescita con gli antibiotici selettivi corretti e può essere confermato dalla PCR delle colonie e/o dal sequenziamento. I ceppi modificati devono essere verificati come reporter fenotipici sottoponendoli a condizioni in cui è noto che il gene di interesse è indotto e la fluorescenza risultante può essere misurata mediante citometria a flusso, spettrofotometria o microscopia a epifluorescenza (Figura 1).
Per facilitare la selezione di una o più dosi di antibiotico che verranno utilizzate per esperimenti successivi, eseguire saggi di persistenza antibiotica concentrazione-dipendenti per i ceppi di P. aeruginosa o S. aureus di interesse. I saggi concentrazione-dipendenti in genere determinano una curva bifasica con una pendenza iniziale ripida a concentrazioni di antibiotici più basse e una pendenza plateau o meno ripida a concentrazioni più elevate. Tuttavia, per alcune coppie antibiotico-specie, potrebbe non risultare una curva bifasica distinta. Ad esempio, la curva per S. aureus delafloxacina è chiaramente bifasica (Figura 3A), ma la curva della levofloxacina di P. aeruginosa non lo è (Figura 3B)15. In questo scenario, sceglieremmo una concentrazione che sia almeno 10 volte la MIC (ad esempio, 5 μg/mL, che è circa 15 volte la MIC per P. aeruginosa)15. Tuttavia, poiché la MIC 15x levofloxacina produce solo ~0,001% di sopravvissuti a P. aeruginosa , utilizziamo il trattamento con levofloxacina da 1 μg/mL se vogliamo vedere i persistenti durante l'imaging delle cellule mentre si riprendono su cuscinetti di agarosio privi di antibiotici (Video supplementare 6); In caso contrario, il numero di campi visivi necessari per visualizzare più persister diventa proibitivo.
All'inizio dell'imaging, il campione ideale e la preparazione del tampone di agarosio devono apparire planari in tutto il campo visivo, privi di detriti di grandi dimensioni, rughe o bolle d'aria e con singole cellule distribuite uniformemente. L'ottenimento di singole cellule ben distribuite può richiedere l'ottimizzazione della diluizione o della risospensione del campione. Per S. aureus, le cellule tendono a formare piccoli grappoli e devono essere vorticate accuratamente prima di essere seminate nel cuscinetto di agarosio (Figura 4 e Figura 5). Per P. aeruginosa, le cellule possono formare aggregati racchiusi in una matrice extracellulare appiccicosa in sospensione; È necessario pipettare accuratamente questi campioni e interrompere gli aggregati per l'imaging di singole cellule.
Dopo la conclusione di un esperimento di imaging, un time-lapse di successo delle immagini apparirà a fuoco, illuminato in modo stabile e con una deriva minima nel piano x-y durante l'esperimento. Il Video supplementare 7 rappresenta un'acquisizione ottimale dell'immagine: t è il canale di fase del Video supplementare 4 prima della correzione dell'ombra o della deriva. La perdita di messa a fuoco può verificarsi se la condensa (dovuta a un'eccessiva umidificazione o a un riscaldamento insufficiente del campione) provoca la formazione di goccioline d'acqua sul vetrino coprioggetto superiore da 25 mm, distorcendo la luce e spingendo il piano focale al di fuori del campo di ricerca massimo dell'algoritmo di messa a fuoco automatica (Video supplementare 8). La variazione dell'illuminazione di solito indica una quantità insufficiente di olio da immersione al momento dell'imaging. Se il tavolino si muove troppo velocemente, l'olio sull'obiettivo potrebbe trascinarsi dietro e continuare a recuperare terreno quando le immagini vengono acquisite. Questo può essere mitigato regolando i controlli di acquisizione per rallentare la velocità di movimento o aggiungendo una pausa tra il movimento alla posizione successiva e l'acquisizione dell'immagine. La deriva del campione principale sarà simile a molte cellule che attraversano il campo visivo, mentre alcune rimangono al loro posto (Video supplementare 9). Questo si verifica in genere più tardi negli esperimenti perché il tampone di agarosio si è disidratato a causa di un controllo dell'umidità insufficiente. La preparazione del tampone di agarosio presentata in questo articolo è stata progettata per facilitare la stabilità del campione, ma per un'acquisizione ottimale dell'immagine è necessario riscaldare/umidificare adeguatamente il campione e l'ambiente circostante.
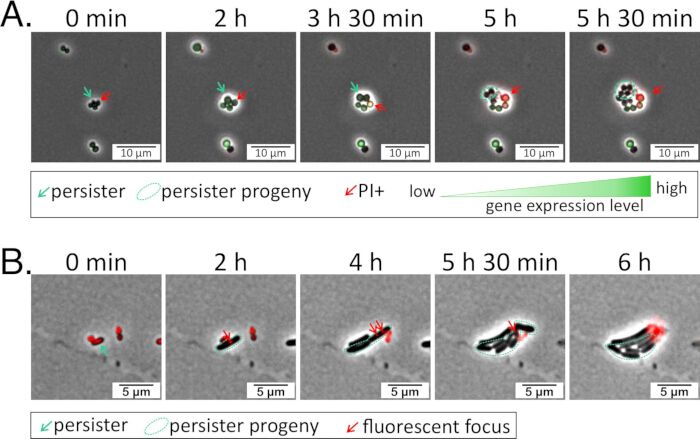
Figura 1: I ceppi reporter fluorescenti illuminano l'espressione di un gene di interesse. (A) S. aureus è stato trasdotto con un reporter trascrizionale GFP per un gene di interesse per il Protocollo 1. Il ceppo reporter è stato trattato per 24 ore con antibiotico, lavato con PBS e quindi seminato su un tampone di agarosio a base di CA-MHB più ioduro di propidio (1,6 μM) e cloramfenicolo (10 μg/mL per il mantenimento del plasmide reporter) per l'imaging durante il recupero (Video supplementare 1). (B) P. aeruginosa è stato trasformato con un plasmide contenente un reporter traslazionale legato a mScarlet per una proteina di interesse26. Il ceppo reporter è stato trattato per 5 ore con antibiotico, lavato con PBS e quindi seminato su un tampone di agarosio a base di BSM più Tet (75 μg/mL; per il mantenimento del plasmide reporter) per l'imaging durante il recupero (Video supplementare 2). Clicca qui per visualizzare una versione più grande di questa figura.
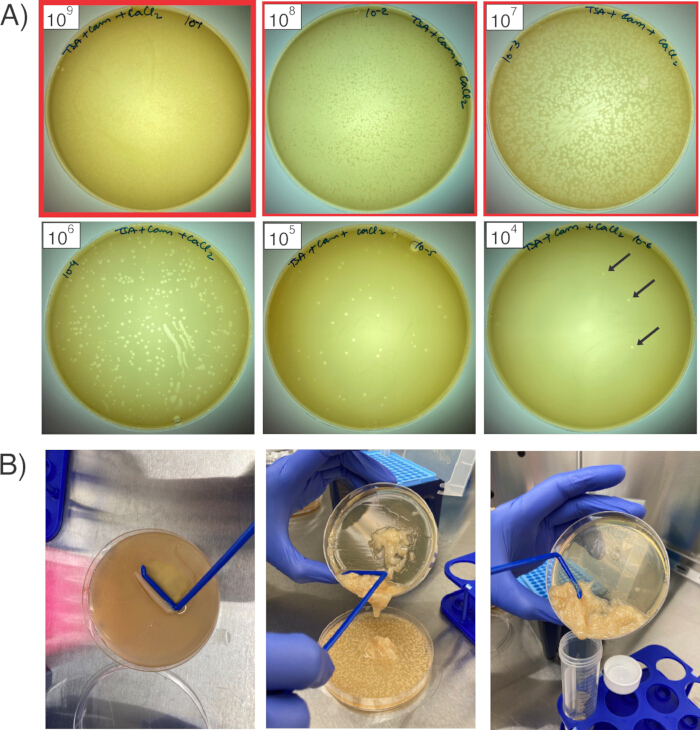
Figura 2: Propagazione e raccolta dei batteriofagi. (A) Le sei tavole mostrano sei diverse quantità di stock di fagi diluiti sui prati di S. aureus RN4220. I contorni rossi indicano le tre piastre che verrebbero prelevate, dalla piastra con la maggiore chiarifica (contorno rosso in grassetto; 1 x109 PFU/mL) alle due diluizioni successive (1 x108 e 1 x 107 PFU/mL). Le frecce nere indicano le singole placche. (B) Per raccogliere il fago dalle piastre, raschiare lo strato di agar morbido (a sinistra), trasferire l'impasto alla successiva piastra di diluizione (al centro) e, dopo aver raccolto l'agar morbido da tutte e tre le piastre, unire in una provetta conica per la centrifugazione (a destra). Clicca qui per visualizzare una versione più grande di questa figura.
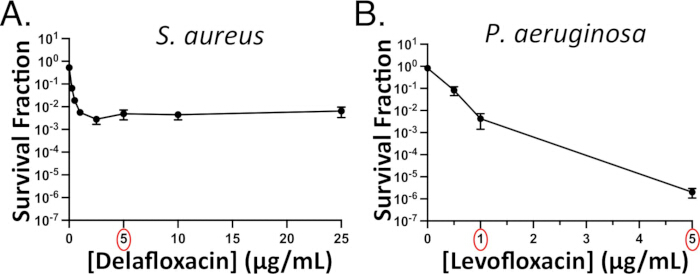
Figura 3: Saggi rappresentativi di persistenza concentrazione-dipendente. La persistenza dei fluorochinoloni concentrazione-dipendente è stata valutata in fase stazionaria (A) S. aureus (contro delafloxacina) e (B) P. aeruginosa (contro levofloxacina). Gli esperimenti successivi utilizzano 5 μg/mL di delafloxacina (cerchio rosso) perché l'uccisione di S. aureus si era stabilizzata a questa concentrazione. Un dosaggio di almeno 1 μg/mL di levofloxacina (cerchio rosso) sarebbe stato utilizzato per i successivi esperimenti con P. aeruginosa. Si noti che l'abbattimento batterico non si stabilizza per P. aeruginosa, ma c'è ancora una "seconda fase" meno ripida della curva bifasica che indica una sottopopolazione persistente. Il pannello 3B è stato adattato con il permesso di Hare et al.15. Clicca qui per visualizzare una versione più grande di questa figura.
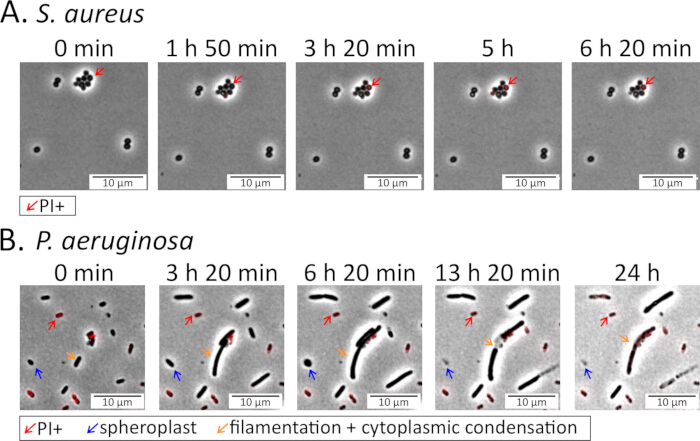
Figura 4: Imaging dei fenotipi batterici durante il trattamento antibiotico. Le cellule in fase stazionaria (A) di S. aureus e (B) di P. aeruginosa sono state seminate su tamponi di agarosio contenenti antibiotici fluorochinolonici e monitorate durante il trattamento: 5 μg/mL di delafloxacina per S. aureus (Video supplementare 3) e 5 μg/mL di levofloxacina per P. aeruginosa (Video supplementare 4)15. Lo ioduro di propidio (PI; 16 μM per P. aeruginosa, 1,6 μM per S. aureus) è stato aggiunto ai cuscinetti per contrassegnare le cellule morte o morenti. Le cellule di S. aureus rimangono in gran parte intatte e vive in presenza di FQ, mentre la maggior parte delle cellule di P. aeruginosa subisce drastici cambiamenti morfologici, inclusa la formazione di sferoplasti rotondi, prima di lisi e morire. Clicca qui per visualizzare una versione più grande di questa figura.
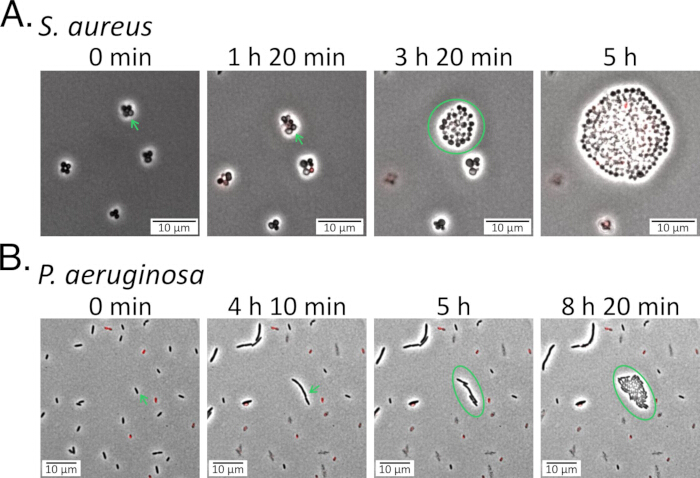
Figura 5: Monitoraggio dei persistenti durante il recupero. (A) Le popolazioni di S. aureus e (B) P. aeruginosa sono state seminate su tamponi di agarosio contenenti terreni freschi dopo essere state trattate con fluorochinoloni (5 μg/mL di delafloxacina per S. aureus e 1 μg/mL di levofloxacina per P. aeruginosa) e monitorate durante il loro recupero post-trattamento (Video supplementare 5 e Video supplementare 6). I perisori osservati sono indicati con frecce verdi nei primi due fotogrammi di ciascun pannello e sono rimasti intatti e vitali durante il trattamento antibiotico. Dopo un iniziale periodo di ritardo, i persister hanno iniziato a dividersi e hanno dato origine a una nuova progenie (indicata con cerchi verdi). Clicca qui per visualizzare una versione più grande di questa figura.
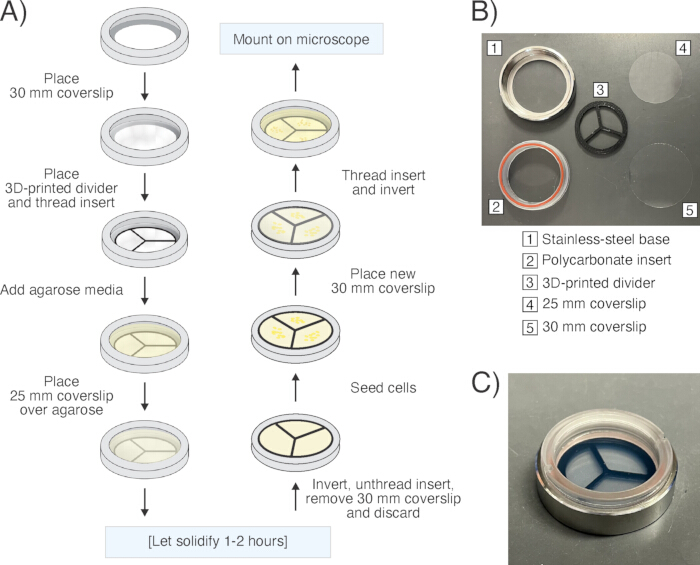
Figura 6: Preparazione del campione al microscopio. (A) Schema del flusso di lavoro di preparazione del campione utilizzando un coprioggetto intercambiabile ("camera"). (B) Immagine della camera smontata e dei suoi singoli componenti. (C) Immagine della camera completamente assemblata. Clicca qui per visualizzare una versione più grande di questa figura.
Video supplementare 1: S. aureus persister. File video contenente le immagini nella Figura 1A. In breve, S. aureus che portava un reporter trascrizionale GFP per un gene di interesse è stato trattato per 24 ore con antibiotico, lavato con PBS, quindi seminato su un tampone di agarosio a base di CA-MHB più ioduro di propidio (1,6 μM) e cloramfenicolo (10 μg/mL) per l'imaging durante il recupero. Clicca qui per scaricare questo video.
Video supplementare 2: P. aeruginosa persister. File video contenente immagini nella Figura 1B. In breve, P. aeruginosa contenente un reporter traslazionale legato a mScarlet per una proteina di interesse è stato trattato per 5 ore con antibiotico, lavato con PBS, quindi seminato su un tampone di agarosio realizzato con BSM più Tet (75 μg/mL) per l'imaging durante il recupero26. Clicca qui per scaricare questo video.
Video supplementare 3: S. aureus durante il trattamento antibiotico. Una coltura in fase stazionaria di S. aureus coltivata in terreni ricchi chimicamente definiti è stata seminata in tamponi di agarosio realizzati con terreni condizionati privi di cellule della coltura con ioduro di propidio (1,6 μM) e delafloxacina (5 μg/mL). Clicca qui per scaricare questo video.
Video supplementare 4: P. aeruginosa durante il trattamento antibiotico. Una coltura in fase stazionaria di P. aeruginosa coltivata in BSM è stata seminata in cuscinetti di agarosio realizzati con terreni condizionati privi di cellule da una coltura di P. aeruginosa coltivata in BSM in parallelo; il tampone di agarosio conteneva anche ioduro di propidio (16 μM) e levofloxacina (5 μg/mL). Questo video è stato adattato con il permesso di Hare et al.15. Clicca qui per scaricare questo video.
Video supplementare 5: S. aureus durante il recupero post-antibiotico. S. aureus è stato coltivato fino alla fase stazionaria in terreni ricchi chimicamente definiti. Le colture in fase stazionaria sono state trattate con 5 μg/mL di delafloxacina in provette per 24 ore, lavate con PBS e quindi seminate in tamponi di agarosio CA-MHB privi di antibiotici contenenti ioduro di propidio (1,6 μM) per l'imaging. Clicca qui per scaricare questo video.
Video supplementare 6: P. aeruginosa durante il recupero post-antibiotico. Una coltura in fase stazionaria di P. aeruginosa coltivata in BSM è stata trattata con 1 μg/mL di levofloxacina in provetta per 7 ore, lavata con PBS, quindi seminata in tamponi di agarosio BSM privi di antibiotici contenenti ioduro di propidio (16 μM) per l'imaging. Clicca qui per scaricare questo video.
Video supplementare 7: Esempio di acquisizione ottimale delle immagini. Questo video è il canale di fase del Video supplementare 4 prima dell'elaborazione delle immagini come esempio di un'acquisizione time-lapse ottimale. Si noti la deriva minima, l'illuminazione stabile e il mantenimento della messa a fuoco durante l'esperimento. Clicca qui per scaricare questo video.
Video supplementare 8: Esempio di acquisizione di immagini non ottimale a causa della condensa. Questo video mostra parte di un esperimento in cui l'acquisizione dell'immagine è stata influenzata da una scarsa messa a fuoco, probabilmente a causa della condensa sulla camera dovuta a un riscaldamento improprio del campione e/o all'eccessiva umidificazione dell'ambiente di imaging. Il campione oggetto di imaging era P. aeruginosa trattato con levofloxacina durante il recupero post-antibiotico su un tampone di agarosio BSM. Clicca qui per scaricare questo video.
Video supplementare 9: Esempio di acquisizione di immagini non ottimale a causa di una deriva. Questo video mostra parte di un esperimento in cui l'acquisizione dell'immagine è stata influenzata dalla deriva del campione, probabilmente a causa della disidratazione e del restringimento/sollevamento del tampone di agarosio dal vetrino coprioggetti. Il campione oggetto dell'imaging era P. aeruginosa su un tampone di agarosio contenente levofloxacina e ioduro di propidio. Clicca qui per scaricare questo video.
File supplementare 1: 25mm-3D-divider-for-35mmBioptechs.stl Fare clic qui per scaricare questo file.
Discussione
Abbiamo scoperto che il successo di un esperimento di microscopia time-lapse dipende dalla qualità dei cuscinetti di agarosio e dalla loro stabilità nel corso dell'imaging. I tamponi di agarosio racchiusi in una camera di acciaio inossidabile sono relativamente facili da preparare, il che si traduce in campioni costantemente planari che possono essere visualizzati in modo stabile per decine di ore. Ciò consente l'imaging di decine di migliaia di cellule in un singolo esperimento e aumenta la probabilità di rilevare varianti fenotipiche rare, come le cellule persistenti, in una popolazione.
Questo metodo di preparazione del tampone di agarosio rappresenta un'alternativa facilmente implementabile ai metodi pubblicati in precedenza. Il nostro protocollo non richiede la precisione tecnica della fabbricazione di dispositivi microfluidici o le abili manipolazioni dei metodi "sandwich" di agarosio, rendendo più facile ottenere preparazioni coerenti da una corsa all'altra 14,16,36. Inoltre, il sistema è conveniente. La camera in acciaio inossidabile è sterilizzabile e riutilizzabile (a differenza delle camere in plastica monouso) e l'installazione non richiede attrezzature specializzate 16,37. La camera si adatta facilmente a diversi sistemi di microscopia utilizzando inserti per tavolini disponibili in commercio. Inoltre, poiché i batteri sono immobilizzati all'interfaccia agarosio-vetro di copertura, abbiamo avuto successo nel tracciare batteri altamente mobili come P. aeruginosa, pur consentendo cambiamenti morfologici (Figura 4, Video supplementare 4). Altre tecniche di imaging a singola cellula, come la "macchina madre", confinano le cellule a canali che precludono l'osservazione di cambiamenti morfologici diversi dalla filamentazione36.
Per il successo con questo protocollo, ci sono alcuni passaggi e parametri critici da tenere a mente. Per la preparazione del tampone, è importante riscaldare e fondere accuratamente l'agarosio, poiché eventuali cristalli di agarosio rimanenti causeranno diffrazione della luce e influenzeranno la qualità dell'immagine. Allo stesso modo, bisogna fare attenzione a pipettare l'agarosio nella camera senza introdurre bolle d'aria. Per garantire che lo spessore dei tamponi di agarosio rimanga costante e per limitare la deriva del campione, è importante lasciare che il tampone si equilibri, in genere per 15 minuti, nell'involucro umidificato e a temperatura controllata prima dell'inizio dell'imaging. Un altro fattore che può causare una scarsa qualità dell'immagine è il controllo dell'umidità: una bassa umidità provocherà la disidratazione e il restringimento del tampone di agarosio, mentre un'elevata umidità (o un riscaldamento improprio del campione nella camera) potrebbe causare la condensazione dell'aria calda sul campione e distorcere l'imaging. Un esempio di imaging time-lapse non ottimale a causa della condensa è disponibile nel video supplementare 8.
Una limitazione dell'attuale configurazione è che i terreni di coltura non possono essere scambiati, il che impedisce il monitoraggio continuo dei singoli batteri prima, durante e dopo il trattamento antibiotico. Prevediamo che l'accoppiamento del tampone di agarosio con celle a flusso o dispositivi microfluidici che consentono lo scambio di terreni di coltura potrebbe consentire di tracciare le popolazioni durante i cambiamenti nutrizionali o ambientali. Un altro parametro dell'attuale progetto che potrebbe essere migliorato è l'aerazione del campione. La guarnizione O-ring, il design con tappo a vite del piatto montante intercambiabile consente una migliore aerazione del campione rispetto alle configurazioni che richiedono l'uso di cera o sigillante a base di grasso per sigillare i tamponi16. Tuttavia, l'aerazione nella camera stagna può essere ancora limitata e potrebbe non supportare la crescita degli aerobi obbligati, anche se questo aspetto deve ancora essere testato.
Il protocollo di preparazione dei campioni con imaging time-lapse che presentiamo in questo articolo consente di monitorare migliaia di batteri mentre rispondono o si riprendono dal trattamento antibiotico. Questo metodo è anche altamente generalizzabile e ha una varietà di potenziali applicazioni oltre alla biologia dei persistenti. Ad esempio, il tampone di agarosio e il divisore consentono la semina di campioni cellulari spazialmente separati, ma consentono la comunicazione cellula-cellula attraverso la diffusione attraverso il tampone di agarosio. Attualmente stiamo esplorando il potenziale di questa configurazione per testare come lo scambio di prodotti secreti influenzi la crescita cellulare nelle comunità multispecie. Prevediamo che questo protocollo fornirà una bassa barriera all'ingresso nella microscopia time-lapse per il nuovo ricercatore e variazioni illimitate da esplorare per il microbiologo esperto.
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
Ringraziamo la signora Susan Staurovsky dell'UConn Health Center for Cell Analysis and Modeling Microscopy Facility per la sua assistenza negli esperimenti di microscopia. Ringraziamo la Dott.ssa Mona Wu Orr e l'Essentials of Staphylococcal Genetics and Metabolism Workshop per i loro protocolli e consigli sulla clonazione rispettivamente in P. aeruginosa e S. aureus. Questo lavoro è stato sostenuto dai finanziamenti del National Institutes of Health (NIH; DP2GM146456-01 e 1R01AI167886-01A1 a W.W.K.M., 1F30DE032598-01A1 a P.J.H. e 1F31DK136259-01A1 a T.J.L.). I finanziatori non hanno avuto alcun ruolo nella progettazione dei nostri esperimenti o nella preparazione di questo manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| BaSiC | GitHub | https://github.com/marrlab/BaSiC | |
| Certified Molecular Biology Agarose | Biorad | 1613101 | |
| Fiji-ImageJ | NIH | https://imagej.net/software/fiji/downloads | |
| Interchangeable Coverglass Dish | Bioptechs | 190310-35 | 35 mm ICD for preparing agarose pads; comes with 30 mm (#1.5) coverslips |
| Lumencor Spectra 7 LED light engine | Lumencor | https://lumencor.com/products/spectra-light-engine | Spectra 7 LED light engine |
| MetaMorph | Molecular Devices | Premier version 7.10.5 | |
| pco.edge 4.2 bi sCMOS camera | Excelitas | https://www.excelitas.com/product/pcoedge-42-bi-usb-scmos-camera | sCMOS camera |
| PeCon live cell incubation chamber | peCon | https://www.pecon.biz/ | |
| Thomas Scientific Round cover glass, #1.5 thickness, 25 mm, 100 pack | Fisher Scientific | NC1272770 | 25 mm (#1.5) coverslips |
| Zeiss Axiovert 200M microscope | Zeiss | https://www.zeiss.com/microscopy/en/products/light-microscopes/widefield-microscopes/axiovert-for-materials.html | Inverted microscope with Plan-Apochromat 63x/1.40 Oil Ph3 M27 objective, Lumencor Spectra 7 LED light engine, and pco.edge 4.2 bi sCMOS camera (6.5 mm pixel size) |
Riferimenti
- Brauner, A., Fridman, O., Gefen, O., Balaban, N. Q. Distinguishing between resistance, tolerance and persistence to antibiotic treatment. Nat Rev Microbiol. 14, 320-330 (2016).
- Balaban, N. Q., et al. Definitions and guidelines for research on antibiotic persistence. Nat Rev Microbiol. 17 (7), 441-448 (2019).
- Conlon, B. P. Staphylococcus aureus chronic and relapsing infections: evidence of a role for persister cells. Bioessays. 36 (10), 991-996 (2014).
- Mulcahy, L. R., Burns, J. L., Lory, S., Lewis, K. Emergence of Pseudomonas aeruginosa strains producing high levels of persister cells in patients with cystic fibrosis. J Bacteriol. 192 (23), 6191-6199 (2010).
- Verstraete, L., Van den Bergh, B., Verstraeten, N., Michiels, J. Ecology and evolution of antibiotic persistence. Trends Microbiol. 30 (5), 466-479 (2021).
- Sett, A., Dubey, V., Bhowmik, S., Pathania, R. Decoding bacterial persistence: Mechanisms and strategies for effective eradication. ACS Infect Dis. 10 (8), 2525-2539 (2024).
- Mempin, R., et al. Release of extracellular ATP by bacteria during growth. BMC Microbiol. 13, 301 (2013).
- Lewis, K. Persister cells, dormancy and infectious disease. Nat Rev Microbiol. 5 (1), 48-56 (2006).
- Fauvart, M., De Groote, V. N., Michiels, J. Role of persister cells in chronic infections: clinical relevance and perspectives on anti-persister therapies. J Med Microbiol. 60 (pt 6), 699-709 (2011).
- Van den Bergh, B., Fauvart, M., Michiels, J. Formation, physiology, ecology, evolution and clinical importance of bacterial persisters. FEMS Microbiol Rev. 41, 219-251 (2017).
- Levin, B. R., Rozen, D. E. Non-inherited antibiotic resistance. Nat Rev Microbiol. 4 (7), 556-562 (2006).
- Davis, K. M., Isberg, R. R. Defining heterogeneity within bacterial populations via single cell approaches. Bioessays. 38 (8), 782-790 (2016).
- Hare, P. J., LaGree, T. J., Byrd, B. A., DeMarco, A. M., Mok, W. W. K. Single-cell technologies to study phenotypic heterogeneity and bacterial persisters. Microorganisms. 9 (11), 2277 (2021).
- de Jong, I. G., Beilharz, K., Kuipers, O. P., Veening, J. W. Live cell imaging of Bacillus subtilis and Streptococcus pneumoniae using automated time-lapse microscopy. J Vis Exp. 53, e3145 (2011).
- Hare, P. J., Gonzalez, J. R., Quelle, R. M., Wu, Y. I., Mok, W. W. K. Metabolic and transcriptional activities underlie stationary-phase Pseudomonas aeruginosa sensitivity to levofloxacin. Microbiol Spectr. 12 (1), e0356723 (2024).
- Young, J. W., et al. Measuring single-cell gene expression dynamics in bacteria using fluorescence time-lapse microscopy. Nat Protoc. 7 (1), 80-88 (2011).
- Pang, Y. Y., et al. agr-Dependent interactions of Staphylococcus aureus USA300 with human polymorphonuclear neutrophils. J Innate Immun. 2 (6), 546-559 (2010).
- Bose, J. L., Fey, P. D., Bayles, K. W. Genetic tools to enhance the study of gene function and regulation in Staphylococcus aureus. Appl Environ Microbiol. 79 (7), 2218 (2013).
- Schenk, S., Laddaga, R. A. Improved method for electroporation of Staphylococcus aureus. FEMS Microbiol Lett. 73, 133-138 (1992).
- Grosser, M. R., Richardson, A. R. Method for Preparation and Electroporation of S. aureus and S. epidermidis. The Genetic Manipulation of Staphylococci. Methods Molecular Biology. , (2016).
- Krausz, K. L., Bose, J. L. Bacteriophage Transduction in Staphylococcus aureus: Broth-Based Method. The Genetic Manipulation of Staphylococci. Methods Molecular Biology. , (2016).
- Olson, M. E. Bacteriophage Transduction in Staphylococcus aureus. The Genetic Manipulation of Staphylococci. Methods Molecular Biology. , (2016).
- Fey, P. D., et al. A genetic resource for rapid and comprehensive phenotype screening of nonessential Staphylococcus aureus genes. mBio. 4 (1), e00537-e00612 (2013).
- Coe, K. A., et al. Multi-strain Tn-Seq reveals common daptomycin resistance determinants in Staphylococcus aureus. PLoS Pathog. 15 (11), e1007862 (2019).
- Figurski, D. H., Helinski, D. R. Replication of an origin-containing derivative of plasmid RK2 dependent on a plasmid function provided in trans. Proc Natl Acad Sci U S A. 76 (4), 1648 (1979).
- Shee, C., et al. Engineered proteins detect spontaneous DNA breakage in human and bacterial cells. eLife. 2, e01222 (2013).
- Kowalska-Krochmal, B., Dudek-Wicher, R. The minimum inhibitory concentration of antibiotics: methods, interpretation, clinical relevance. Pathogens. 10 (2), 165 (2021).
- Robinson, J. L., Jaslove, J. M., Murawski, A. M., Fazen, C. H., Brynildsen, M. P. An integrated network analysis reveals that nitric oxide reductase prevents metabolic cycling of nitric oxide by Pseudomonas aeruginosa. Metab Eng. 41, 67-81 (2017).
- Wolff, J. A., MacGregor, C. H., Eisenberg, R. C., Phibbs, P. V. Isolation and characterization of catabolite repression control mutants of Pseudomonas aeruginosa PAO. J Bacteriol. 173 (15), 4700-4706 (1991).
- Batchelder, J. I., Taylor, A. J., Mok, W. W. K. Metabolites augment oxidative stress to sensitize antibiotic-tolerant Staphylococcus aureus to fluoroquinolones. mBio. , e0271424 (2024).
- Power, A. D., Mok, W. W. K. Agar and agarose used for Staphylococcus aureus biofilm cultivation impact fluoroquinolone tolerance. J Appl Microbiol. 135 (8), lxae191 (2024).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Peng, T., et al. A BaSiC tool for background and shading correction of optical microscopy images. Nat Commun. 8, 14836 (2017).
- Ducret, A., Quardokus, E., Brun, Y. V. MicrobeJ, a tool for high throughput bacterial cell detection and quantitative analysis. Nat Microbiol. 1 (7), 16077 (2016).
- Paintdakhi, A., et al. Oufti: an integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Mol Microbiol. 99 (4), 767-777 (2016).
- Wang, P., et al. Robust growth of Escherichia coli. Curr Biol. 20 (12), 1099-1103 (2010).
- Moffitt, J. R., Lee, J. B., Cluzel, P. The single-cell chemostat: an agarose-based, microfluidic device for high-throughput, single-cell studies of bacteria and bacterial communities. Lab Chip. 12 (8), 1487-1494 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon