Method Article
Time-Lapse Epifluorescence Microscopy Imaging of Pseudomonas aeruginosa 및 Staphylococcus aureus 이종 표현형
* 이 저자들은 동등하게 기여했습니다
요약
이 원고에서는 녹농균(Pseudomonas aeruginosa ) 및 황색포도상구균(Staphylococcus aureus)의 항생제 생존을 평가하고, 플라스미드를 녹농균(P. aeruginosa) 및 S. 아우레우스(S. aureus )로 변환하여 리포터 균주를 생성하고, 타임랩스 형광 현미경 검사로 지속형(persister)와 같은 표현형 변이를 시각화하기 위한 포괄적인 프로토콜을 제공합니다.
초록
항생제 지속성은 유전적으로 취약한 집단에서 소수의 박테리아 세포가 유전적으로 동일한 다른 세포를 죽이는 항생제 치료에서 살아남는 현상입니다. 세균 지속인자는 항생제 치료가 끝나면 복제를 재개할 수 있으며, 일반적으로 임상 치료 실패의 기저에 있는 것으로 생각됩니다. 다양한 세포 특징에 대한 형광 전사 리포터, 번역 리포터 및/또는 염료로 박테리아를 라벨링하는 타임 랩스 형광 현미경 검사의 힘을 활용하는 최근 연구는 대장 균 생존 분석에서 알 수 있는 것 이상으로 대장균 생존 바이러스에 대한 이해를 발전시켰습니다. 벌크 집단 분석이 아닌 이러한 단세포 접근법은 지속성 형성, 손상 반응 및 생존 메커니즘을 설명하는 데 필수적입니다. 그러나 이 세부 수준에서 다른 중요한 병원성 종의 지속재를 연구하는 방법은 여전히 제한적입니다.
이 연구는 항생제 치료 및 회복 중 녹농균( Pseudomonas aeruginosa , 그람 음성 간상균) 및 황색포도상구균(Staphylococcus acorus , 그람 양성 구균)의 타임 랩스 이미징을 위한 적응 가능한 접근 방식을 제공합니다. 우리는 이러한 박테리아에 형광 리포터를 도입하기 위한 분자 유전학적 접근 방식에 대해 논의합니다. 이러한 리포터와 염료를 사용하여 항생제 치료에 대한 반응으로 개별 세포의 표현형 변화, 형태학적 특징 및 운명을 추적할 수 있습니다. 또한, 우리는 치료 후 소생하는 개별 지속인자의 표현형을 관찰할 수 있습니다. 전체적으로 이 연구는 임상적으로 중요한 병원체에서 치료 중 및 치료 후에 지속생존자를 포함하여 개별 항생제 투여된 세포의 생존 및 유전자 발현을 추적하는 데 관심이 있는 사람들을 위한 리소스 역할을 합니다.
서문
세균 병원체는 두 가지 주요 메커니즘, 즉 유전적 변화를 수반하는 항생제 내성과 비유전적 변화를 수반하는 표현형 내성을 통해 항생제의 영향을 피할 수 있습니다. 항생제 내성은 유전적으로 암호화된 현상으로, 항생제가 있는 상태에서 박테리아 세포가 생존할 뿐만 아니라 복제할 수 있는 능력을 부여합니다1. 항생제 내성 또는 항생제 잔류 박테리아를 포함할 수 있는 표현형 내성은 세포가 항생제의 억제 농도가 있는 상태에서 복제 능력을 얻지 않고 살균 항생제 처리에 견딜 때 발생합니다 1,2. 내성과 지속성을 구별하는 것은 관용은 전체 개체군이 치료를 견뎌낼 수 있는 능력을 의미하는 반면, 지속성은 항생제 치료에서 살아남는 동질적이지만 표현형적으로 이질적인 집단의 하위 집합을 의미한다는 것입니다. 클론 배양액을 살균 항생제로 처리하고 배양액에 남아 있는 생존자를 로그 선형 척도로 시간에 대해 그래프화하는 경우, 지속생존자가 존재할 때 일반적으로 이위상 곡선이 감지됩니다. 이러한 곡선에서 첫 번째 단계는 인구의 대다수가 상대적으로 빨리 사멸한다는 것을 보여주고, 두 번째 단계는 항생제 지속성 분획이 더 느린 속도로 사멸되거나 전혀 사멸되지 않음을 나타냅니다 1,2.
항생제 지속성은 전 세계 의료 시스템에 큰 부담이 되고 있습니다. 예를 들어, 이 글에서 중점적으로 다루고 있는 황색포도상구균(Staphylococcus aureus)과 녹농균(Pseudomonas aeruginosa) 지속인자(Pseudomonas aeruginosa persisters)는 낭포성 섬유증(cystic fibrosis) 및 만성 상처 감염 환자에서 재발성 기도 감염을 포함한 항생제 난치성 감염을 유발하는 것으로 생각된다 3,4. 따라서 지속세포 생물학 및 표현형 프로그램을 추가로 규명하는 것이 중요합니다. 지속생존자가 어떻게 형성되고 소생하는지에 대한 이해가 진전되었지만, 지속성의 기초가 되는 개별 세포의 대사 재프로그래밍과 분자 사건의 조정과 관련된 중요한 지식 격차는 여전히남아 있습니다 5,6,7,8.
끈기를 효과적으로 연구하는 것은 기술적인 도전임이 입증되었습니다. 지속성은 박테리아 집단의 작은 하위 집합에서만 관찰할 수 있기 때문에 벌크 박테리아 집단을 샘플링하는 기술은 종종 관련 생물학적 정보를 캡처하지 못합니다 1,2,8,9,10. 더욱이, 지속성의 기저에 있는 표현형 변화는 일시적이며 유전될 수 없기 때문에 지속세포 의 운명을 추적하는 것은 복잡할 수 있습니다 1,8,9,10,11. 박테리아 지속생존 개체군이 성장을 재개하면 지속개체와 비지속개체군이 분열되어 발생할 수 있으며, 이로 인해 배양을 통해 순수한 지속생존 개체군을 풍부하게 만드는 것이 불가능합니다. 이러한 과제는 1) 살아있는 단일 세포의 생물학적 정보를 캡처할 수 있는 능력과 2) 이질적인 집단에 있는 개별 세포의 표현형을 시간이 지남에 따라 조사할 수 있는 형광 염료, 프로브, 센서 및 리포터와 함께 사용할 수 있는 능력과 같은 기준을 충족할 수 있는 기술의 필요성을 강조합니다.
최근 단세포 기술의 발전은 박테리아의 이질성을 효과적으로 조사하고 지속성 연구에서 이러한 장애물을 극복할 수 있는 방법을 제공했습니다12,13. 이러한 기술 중 일부에는 형광 현미경 검사, 유세포 분석/형광 활성화 세포 분류, 미세유체역학 및 단일 세포 RNA 시퀀싱12,13이 포함됩니다. 여기에서는 전사 또는 번역 리포터 균주의 epifluorescence time-lapse microscopy를 사용하여 단일 세포 지속 생리학을 설명하기 위한 프로토콜에 대해 설명합니다. 형광 현미경 검사는 지속형 표현형을 연구하기 위한 기준, 즉 항생제 제거 후 대규모 집단에서 어떤 개별 세포가 증식하는지 식별하여 지속형으로 정의할 수 있는 능력을 충족하는 강력한 기술입니다. 자동 카메라 기술과 배양 챔버의 도입으로 살아있는 박테리아 세포를 캡처하는 것은 미생물학 분야 전반에 걸쳐 널리 접근할 수 있습니다. 결정적으로, 타임 랩스 현미경 검사는 몇 시간, 심지어 며칠에 걸쳐 단일 세포를 실시간으로 시각화할 수 있는 기능을 제공하므로 항생제 치료 전, 치료 중, 치료 후에 박테리아를 추적할 수 있습니다 14,15,16. 타임 랩스 현미경을 활용한 이러한 조사에서 얻은 통찰력은 지속 생물학의 복잡한 메커니즘에 대한 통찰력을 생성할 수 있는 엄청난 잠재력을 가지고 있습니다.
프로토콜
1.형질전환 및 형질전환에 의한S. aureus 의 형광리포터 균주 생성
참고: 리포터 균주는 관심 유전자 또는 단백질의 발현을 나타내는 형광 단백질을 보유하고 있습니다. 전사 리포터는 형광 단백질의 업스트림에 있는 관심 유전자에 대한 네이티브 프로모터 염기서열의 중복 사본을 특징으로 하여 관심 유전자의 발현이 증가함에 따라 형광이 증가합니다. Translational Reporter는 형광 단백질과 관심 단백질의 개방형 판독 프레임을 유연한 펩타이드 커넥터와 연결합니다. 생세포 현미경 검사로 리포터 균주를 시각화하면 특정 관심 유전자/단백질이 특정 세포 형태 또는 세포 운명과 관련이 있는지 여부를 밝힐 수 있습니다(그림 1, 보충 동영상 1 및 보충 동영상 2). S. aureus 에 대해 코돈에 최적화된 형광 단백질을 선택하십시오.이 연구는 Alexander Horswill 박사가 기증한 pCM29의 sGFP를 사용하지만, Nebraska Transposon Mutant Library Genetic Toolkit에는 코돈에 최적화된 sGFP, eYFP, eCFP, DsRed.T3 및 eqFP650을 함유한 플라스미드가 포함되어 있습니다17,18.
- 리포터 플라스미드를 S. aureus RN4220으로 변환합니다.
참고: 다음 프로토콜 섹션은 University of Nebraska Medical Center19,20의 Essentials of Staphylococcal Genetics and Metabolism Workshop에서 발췌한 것입니다.- 먼저 전기적격성 S. aureus RN4220을 준비합니다. 카사미노산 2g, 효모 추출물 5g, 10% K2HPO4 2mL, pH 7.5, 50% 포도당 용액 2mL, NaCl 5g을 혼합하여 B2 육수 200mL를 준비합니다. 멸균 초순수로 부피를 100mL로 가져옵니다. 37°C에서 잠시 혼합하여 구성 요소를 용해하고 보틀탑 필터(0.22μm 공극 크기)를 사용하여 멸균합니다.
- -80°C에서 25% 글리세롤을 섞어 보관한 냉동 스톡에서 250mL 배플 플라스크에 B2 육수 30mL를 넣고 125rpm으로 진탕하면서 30°C에서 18시간 동안 배양합니다.
- 250mL 배플 플라스크 2개를 각각 50mL의 B2 육수와 함께 37°C에서 하룻밤 동안 배양하여 다음 날을 위해 배지를 예열합니다.
- 다음날, 하룻밤 배양의 OD600 을 측정합니다. B2 육수 50mL가 들어 있는 각 플라스크에 충분한 하룻밤 배양을 첨가하여 0.25의 최종 OD600 을 얻습니다.
- OD600 이 0.35-0.4에 도달 할 때까지 37 ° C에서 250rpm으로 흔들어 배양합니다.
참고: 서브컬처링 후 약 30분 후에 OD600 을 확인하여 배양이 0.35-0.4 이상으로 성장하지 않는지 확인하십시오. OD600 이 subculturing 후 1 시간 이내에 0.35-0.4에 도달하지 않으면 절차를 중단하고 다음날 다시 시도하십시오. 박테리아는 하룻밤 사이에 너무 많이 자라서 고정상으로 너무 깊숙이 들어갔을 가능성이 큽니다. - 배양액이 mid-log 단계에 도달하면 각 플라스크의 배양액을 멸균 50mL 코니컬 튜브와 펠릿 세포에 붓고 4,000 x g 에서 10분 동안 원심분리합니다.
- 상층액을 디캔팅하고 펠릿을 1mL의 멸균 실온(RT) 물에 재현탁합니다. 세포를 마이크로 원심분리기 튜브로 옮깁니다.
- 21,000 x g 에서 30초 동안 세포를 다시 원심분리하고, 상층액을 제거한 후 1mL의 멸균 RT 물에 재현탁합니다. 원심분리, 상층액 제거 및 1mL의 물에 재현탁액 재현탁을 2회 더 반복하여 총 3회 세척합니다.
- 세포를 원심분리하고 물을 제거한 다음 멸균 RT 10% 글리세롤 1mL(물 중, 멸균 여과)에 세포를 재현탁하고 15분 동안 상온에서 그대로 둡니다.
- 21,000 x g 에서 30초 동안 원심분리하여 세포를 펠렛화하고, 상층액을 제거하고, 또 다른 1mL의 멸균 RT 10% 글리세롤에 세포를 재현탁합니다. 이 셀은 이제 전기 기능이 있습니다.
- 70μL의 전기 용해 셀을 스냅 캡 마이크로 원심분리기 튜브에 부분 표본으로 삽입하고 향후 사용을 위해 -80°C에서 보관합니다.
- 전기 기능이 있는 S. aureus RN4220을 형질전환하려면 얼음 위에서 세포 분취액을 해동합니다. 세포에 1μg의 플라스미드 DNA를 첨가하고 샘플을 튕겨 혼합한 다음 얼음 위에서 5분 동안 배양합니다. 그런 다음, 세포를 전기 천공하고 37 °C에서 1-2 시간 동안 390 μL의 B2 매체에서 225 rpm으로 진탕하고 선택적 트립 대두 한천에서 플레이트 형질전환체를 회수하도록 합니다.
참고: 이 연구에 사용된 대부분의 셔틀 벡터는 대장 균(100μg/mL)에서 선택하기 위한 암피실린 내성 카세트와 S. aureus 에서 선택하기 위한 클로람페니콜-(10μg/mL) 또는 에리트로마이신-(Erm, 10μg/mL) 저항 카세트를 특징으로 합니다.실험실에서 일상적으로 사용되는 많은 S. aureus 균주는 Erm에 내성이 있습니다. 주어진 셔틀 벡터를 사용하기 전에 관심 균주가 선택적 항생제와 호환되는지 확인합니다. 세포에 첨가되는 DNA의 부피를 최소화(즉, <5 μL)하여 전기천공법 중 샘플의 전도성이 너무 높을 때 발생하는 아크를 방지하는 것이 중요합니다. E. coli 에서 고농도(≥300ng/μL)의 플라스미드 DNA를 정제하면 부피를 낮게 유지하는 데 도움이 됩니다.
- 변형된 박테리오파지 전파 S. aureus RN4220입니다.
참고: 다음 프로토콜 섹션은 Krausz와 Bose의 방법에서 수정되었습니다.21그리고 올슨22. 가장 일반적으로 사용되는 두 가지 박테리오파지 S. aureus 형질도입은 다음과 같습니다. φ11 및 80α. 이 두 가지를 모두 수확하는 절차는 φ11을 얻는 것을 제외하고는 동일합니다. S. aureus RN451 및 80α S. aureus RN10359. φ11은 특히 일반적인 균주 JE2 및 HG003과 함께 성공적으로 사용되었으며 이 프로토콜에 사용됩니다23,24. 그러나 이러한 파지는 다양한 파지 내성을 가진 임상 분리물을 효율적으로 형질도입하지 못할 수 있습니다. Krausz와 Bose의 출판물을 참조하십시오.21 그리고 올슨22 Φ11을 준비하고 파지 역가를 측정하는 방법.- 리포터 플라스미드를 transduction하기 위해 5mM CaCl2 와 선택적 항생제의 적절한 농도가 있는 6개의 트립틱 대두 한천(TSA) 플레이트를 준비합니다. Streak S. aureus RN4220을 리포터 플라스미드와 함께 CaCl2 가 없는 선택적 항생제를 함유한 TSA 플레이트에 형질전환하고 밤새 배양했습니다.
- 다음 날, 인큐베이터나 수조를 56°C까지 켭니다. 6개의 5mL 스냅캡 튜브 1 x 104 에서 1 x 109에 라벨을 부착하고 각 스냅캡 튜브에 40μL의 500mM CaCl2 를 분취합니다.
- 부드러운 TSA(0.5% 한천)를 캡을 풀고 전자레인지에 녹여 압력 상승을 방지하고 무균 상태를 유지합니다. 그런 다음 각 스냅 캡 튜브에 4mL를 분취합니다. 튜브를 뒤집어 섞은 다음 부드러운 한천이 응고되는 것을 방지하기 위해 56 ° C 인큐베이터에 넣습니다.
- 파지 스톡을 트립틱 소이 육수(TSB) + 5mM CaCl2에 1 x 1010 PFU/mL로 희석합니다. 1 x 1010 PFU/mL 파지 100 μL를 TSB + 5 mM CaCl2 가 들어 있는 미세 원심분리 튜브에 추가하여 최종 농도 1 x109 PFU/mL를 만듭니다. 그런 다음 1 x 104 PFU/mL의 농도가 될 때까지 900μL의 TSB + 5mM CaCl2를 포함하는 추가 마이크로 원심분리 튜브에 연속적으로 희석합니다. 6개의 TSA + 5 mM CaCl2 + 선택적 항생제 한천 플레이트를 각각 1 x104 - 1 x 109 PFU/mL의 연속 희석으로 라벨링합니다.
- 1mL의 TSB + 5mM CaCl2에 플라스미드를 함유한 S. aureus RN4220을 하룻밤 동안 재현탁합니다. 인큐베이터에서 109 snap-cap tube를 제거하고 플라스미드를 포함하는 10 μL의 재현탁 RN4220과 1 x 109 PFU/mL 파지 희석 100 μL를 빠르게 첨가하고 뒤집어 혼합한 다음 내용물을 해당 TSA + 5 mM CaCl2 + 선택적 항생제 한천 플레이트에 붓습니다.
- 나머지 파지 희석액과 해당 플레이트에 대해 이 절차를 수행합니다. 부드러운 한천이 플레이트에서 굳어지도록 한 다음 37 ° C에서 밤새 부드러운 한천을 위로 향하게하여 배양합니다.
- 다음 날, 플라크(박테리아 잔디의 클리어런스 영역)가 플레이트에서 보이는지 확인합니다(그림 2A). 플라크는 파지가 RN4220을 성공적으로 감염시킨 용해 영역을 나타내며, 일부 파지는 관심 플라스미드를 차지할 것입니다. 거의 융합 용해가 있는 최소 희석 파지 스톡이 있는 플레이트(예: 1 x 109 PFU/mL 파지)와 이 미만의 두 희석을 받은 플레이트(이 경우 1 x 108 PFU/mL 및 1 x 107 PFU/mL, 둘 다 거의 융합 용해를 가져야 함)를 선택합니다.
- 3mL의 TSB + 5mM CaCl2 를 최소 희석된 플레이트에 추가합니다(이 예에서는 1 x 109 PFU/mL). 멸균 L자형 세포 스프레더를 사용하여 일반 한천에서 부드러운 한천 층을 부드럽게 긁어냅니다(그림 2B). 세포 스프레더를 사용하여 한천을 최대한 파괴하여 부드러운 한천에서 파지가 방출되는 것을 용이하게 합니다.
- 첫 번째 플레이트의 부드러운 한천이 완전히 파괴되면 부드러운 한천과 육수 슬러리를 다음 플레이트(이 경우 1 x 108 PFU/mL)에 부드럽게 붓습니다. 이 플레이트에서 부드러운 한천을 긁어 방해 한 다음 결과 슬러리를 최종 플레이트에 붓고 반복합니다.
- 세 개의 플레이트에서 부드러운 한천 슬러리를 준비한 후 슬러리를 50mL 원뿔형 튜브에 부드럽게 붓습니다. 부드러운 한천을 더 방해하기 위해 부드럽게 피펫을 사용하되 거품이 생기지 않도록 합니다. 와류는 파지를 손상시키고 역가를 감소시킬 수 있으므로 소용돌이치지 마십시오.
- 연질 한천 슬러리를 4,000 x g 의 속도로 RT에서 10분 동안 원심분리합니다.0.45μm 필터에 연결된 주사기에 상층액을 부드럽게 붓습니다.
알림: 부드럽게 붓고 한천 조각이 필터에 들어가지 않도록 하는 것이 중요합니다. 이러한 조각은 필터를 막고 남아 있는 상층액을 여과하는 것을 방해하여 역가를 감소시킵니다. - 야생형 RN422021을 사용하여 생성된 파지 용해물의 역가를 결정합니다.
- 형질도입을 통해 리포터 플라스미드를 S. aureus 관심 균주에 도입합니다.
- 500μg/mL의 구연산나트륨과 적절한 농도의 선택적 항생제를 함유한 TSA 플레이트 8개를 준비합니다. 원하는 수용체 S. aureus 균주를 일반 TSA에 조밀하게 줄무늬를 만들고 37°C에서 밤새 배양합니다.
- 다음날, 플레이트를 면봉으로 닦고 수용체 균주를 1mL의 TSB + 5mM CaCl2에 재현탁합니다.
- 원하는 리포터 플라스미드를 운반하는 파지 스톡을 1 x1010 PFU /mL로 희석합니다.
- 50mL 원뿔형 튜브에 TSB 1.5mL + 5mM CaCl2, 재현탁 수용체 균주 0.5mL 및 희석된 파지 스톡 0.5mL를 혼합합니다. 37 ° C에서 225 rpm으로 20 분 동안 진탕합니다.
참고: 배양 시간이 길면 파지가 수용 세포를 용해하기 시작할 때 형질도입 효율이 감소할 수 있으므로 권장되지 않습니다. - 그 직후, 멸균 여과된 얼음처럼 차가운 0.02M 구연산나트륨(물 중) 1mL를 원뿔형 튜브에 넣고 4,000 x g 에서 4°C에서 10분 동안 원심분리합니다. 그런 다음 펠릿을 얼음처럼 차가운 0.02M 구연산나트륨 1mL에 재현탁합니다.
- 500μg/mL의 구연산나트륨과 적절한 농도의 선택적 항생제를 함유한 6개의 TSA 플레이트에 각각 100μL 분취액을 펴 주입하여 트랜스덕턴트를 선택합니다. 37 °C에서 밤새 배양합니다.
- 다음 날, 500μg/mL 구연산나트륨과 적절한 농도의 선택적 항생제가 함유된 나머지 두 개의 TSA 플레이트에 형질도입 플레이트의 개별 콜로니를 줄무늬로 만듭니다. 37 °C에서 밤새 배양합니다.
참고: 추가적인 sodium citrate plate에 대한 이 passaging step은 transduction된 세포 주위의 파지 역가를 줄여 lysis22를 방지하는 데 도움이 됩니다. - 줄무늬 플레이트에서 개별 콜로니를 선택하고, 선택적 항생제를 포함하는 TSB에서 세포를 기하급수적으로 성장시키고, 향후 사용을 위해 -80°C에서 25% 글리세롤에 보관합니다.
2.접합에 의한 녹농균(P. aeruginosa)의 형광 리포터 균주 생성
참고: 리포터 플라스미드를 E. coli 클로닝 균주로부터 P. aeruginosa로 옮기는 것은 삼부모 짝짓기에 의해 이루어질 수 있다25. 관심 플라스미드를 운반하는 E. coli의 기증자 균주(부부 이식을 위한 oriT를 포함해야 함), E. coli HB101 + pRK2013 - 접합을 용이하게 하는 보조 균주(pRK2013 플라스미드 유지를 위해 50μg/mL kanamycin 사용) 및 플라스미드를 투여할 P. aeruginosa 수용 균주가 필요합니다. 이 예시의 경우, 관심 플라스미드에는 테트라사이클린(Tet) 내성 마커가 있으므로 플라스미드를 사용한 모든 E. coli 배양에는 10μg/mL Tet가 필요하고, 플라스미드를 사용한 녹농균에는 75μg/mL Tet가 필요합니다26.
- -80°C에서 25% 글리세롤을 함유한 냉동 재고의 박테리아를 37°C에서 225rpm으로 진탕하여 하룻밤 동안 성장할 수 있도록 적절한 항생제를 사용하여 용원 육수(LB)에 접종합니다.
- 다음 날, 적절한 선택적 항생제를 사용하여 각 액체 200μL를 LB 한천 플레이트에 하룻밤 동안 배양합니다. 얼마나 많은 형질전환 짝짓기를 수행해야 하는지에 따라 도우미 균주와 기증자 균주를 위해 더 많은 플레이트를 펼칩니다. 변환당 한 접시의 도우미/기부자가 권장됩니다. 37 ° C에서 밤새 플레이트를 배양하여 각 균주의 빽빽한 잔디를 얻습니다.
- 다음날, 멸균 면봉으로 각 판 표면을 닦고 마이크로 원심분리기 튜브 내부를 따라 닦아 성장의 잔디를 수집합니다. 각 튜브를 짧게(5초 동안 펄스) 원심분리기하여 세포를 튜브 바닥으로 모읍니다. 각 세포 펠릿을 200μL LB에 재현탁합니다.
참고: 재현탁액은 매우 두껍고 불투명하며 덩어리가 없어야 합니다. - 각 짝짓기를 위한 새로운 마이크로 원심분리 튜브에서 수용체 P. aeruginosa 균주 5μL, 관심 플라스미드를 함유한 donor E. coli 균주 160μL, helper E. coli HB101 + pRK2013 160μL를 결합합니다. 위아래로 피펫팅하여 부드럽게 혼합한 다음 혼합물 50μL를 사전 건조된 LB 한천 플레이트에 넣습니다. 또한 대조군을 위해 각 개별 균주의 20μL를 LB 한천에 찾아냅니다. 반점을 플레이트에 완전히 건조시킨 다음 37 °C에서 3-6 시간 동안 배양합니다.
참고: LB 한천을 사전 건조하면 50μL 스폿이 표면 전체로 퍼지는 것을 방지하여 세포가 더 작은 영역 내에서 상호 작용하도록 합니다. - 배양 후 피펫 팁을 짝짓기 지점을 가로질러 부드럽게 문질러 일부 세포를 새 마이크로 원심분리 튜브로 옮깁니다. 튜브 바닥에 있는 세포를 모으기 위해 잠시 원심분리기를 사용합니다. 200 μL의 LB에 재현탁하고 전체 재현탁액을 LB + Irgasan (Igr; 25 μg/mL) + Tet (75 μg/mL) 한천 플레이트에 플레이트합니다.
참고: 녹농균(P. aeruginosa) 은 이그라산(Igrasan)에 본질적으로 내성이 있으며, 관심 플라스미드(plasmid)에는 텟(Tet) 저항성 마커가 포함되어 있으므로 성공적으로 형질전환된 녹농균(P. aeruginosa )만이 LB + 이르가산 + 텟(Tet) 플레이트에서 성장할 수 있어야 합니다. 변형 효율에 따라 전체 짝짓기 지점을 다시 현탁시키고 도금하면 다음날 변형체의 잔디가 생길 수 있습니다. 개별 콜로니를 달성하려면 (i) 더 큰 부피의 LB에 전체 결합 지점을 재현탁한 다음 200μL를 플레이트에 재현탁하거나 (ii) 결합 지점의 작은 부분을 마이크로 원심분리 튜브로 옮긴 다음 200μL에 재현탁하고 플레이트합니다. - 각 개별 균주의 대조군을 사용하여 조밀한 세포 반점 중 일부를 피펫 팁에 부드럽게 문지르고, 새 마이크로 원심분리 튜브로 옮기고, 200μL의 LB에 재현탁한 다음 LB + Irgasan + Tet 한천 플레이트에 50μL를 스팟합니다(동일한 한천 플레이트에 여러 대조군을 배치할 수 있음). 반점이 완전히 마르도록 하십시오. 모든 플레이트를 37 ° C에서 16-20 시간 동안 밤새 배양하십시오.
참고: 대조군의 경우, 항생제 플레이트가 녹농 균 형질전환체에 대해 선택적인지 확인하는 것이 목표입니다. LB + Irgasan + Tet 한천 플레이트에서 대조군이 너무 집중되어 있으면 성장으로 오인될 수 있는 죽은 세포의 후광이 있기 때문에 다음 날 선택이 효과가 있었는지 여부를 결정하기 어려울 것입니다. - 다음날, 개별 균주 대조 부위에 대한 세포 성장 부족에 유의하여 LB + Irgasan + Tet 플레이트의 항생제 선택성을 확인합니다.
참고: triparental mating LB + Igrasan + Tet plate에 나타나는 콜로니는 관심 플라스미드를 운반하는 P. aeruginosa 형질전환체입니다. - LB + Irgasan + Tet 한천에 군체를 골라 줄무늬를 만들고 짝짓기 혼합물에서 과도한 죽은 대장균 을 제거하기 위해 밤새 성장시키는 것은 선택 사항입니다.
- 개별 콜로니를 선택하고 액체 LB + Tet(75μg/mL)에서 눈에 띄게 탁해질 때까지 3-6시간 동안 성장한 다음 25% 글리세롤(500μL 세포 배양 + 500μL 물 내 50% 글리세롤)에 냉동 재고를 보관합니다. 클론이 colony PCR 후 Sanger 또는 whole-plasmid 염기서열분석으로 성공적으로 형질전환되었는지 확인합니다.
3. 지속생존구 분석을 위한 항생제 용량 결정
참고: 지속인자 실험을 위해 박테리아 집단을 치료할 주어진 항생제의 용량을 선택하려면 먼저 관심 박테리아 균주에 대한 항생제의 최소 억제 농도(MIC)를 측정합니다. 이는 CLSI(Clinical and Laboratory Standards Institute)에서 승인하는 방법인 육즙 미량 희석법(Broth Microdilution Method) 또는 다양한 항생제 용량의 테스트 스트립을 사용하여 수행되는 Epsilometer 테스트(E-test)를 사용하여 달성할 수 있습니다27. MIC가 결정되면 세포 치료를 위해 1배에서 100배까지의 MIC 범위의 항생제 농도를 최소 5개 이상 선택합니다.
- -80°C에서 25% 글리세롤을 타고 보관된 냉동 재고의 박테리아를 2mL의 양이온 조정 MHB(CA-MHB) 또는 다른 영양이 풍부한 배지에 접종합니다. 250rpm에서 진탕하면서 37°C에서 약 4시간 동안 세포를 성장시킨 후 125μL의 배양액을 250mL의 당황한 삼각 플라스크에서 화학적으로 정의된 신선한 배지 25mL로 옮깁니다. 37°C에서 16시간(고정상까지) 동안 박테리아를 성장시키고 250rpm으로 흔듭니다.
참고: 숙시네이트를 유일한 탄소원으로 하는 기저염 배지(BSM)는 일반적으로 녹농균 실험에 사용되며 화학적으로 정의된 풍부한 배지는 일반적으로 S. aureus 실험에 사용됩니다 28,29,30,31. - 다음 날 아침, 원하는 범위의 초-MIC 농도에 대해 100x 항생제 스톡(각 용매)을 준비합니다. 각 희석액 10μL를 개별 시험관에 추가합니다.
- 익일 배양의 OD600 을 측정하여 고정상 탁도를 확인합니다. 배양액 10μL를 연속으로 희석하고 LB 한천, CA-MHB 한천 또는 TSA와 같은 영양 한천 플레이트에 희석액을 플레이트하여 항생제 처리 전에 집락 형성 단위(CFU)를 측정합니다.
- 항생제 치료의 경우, 100x 항생제 농도 10 μL가 함유된 시험관에 배양액 분취액 1mL를 분주합니다. 37°C에서 250rpm으로 흔들면서 개체군에서 비잔류물을 죽이기에 충분한 기간 동안 샘플을 배양하여 지속생존체만 콜로니를 형성하는 세포로 남깁니다.
참고: 치료 기간은 실험 및 테스트 중인 박테리아 균주에 따라 달라질 수 있습니다. 일반적으로 S. aureus 는 5시간 또는 7시간 동안, P. aeruginosa는 7시간 또는 24시간 동안 치료합니다. 일반적으로, 시간-의존적 생존 검정의 두 번째 단계의 임의의 시점이 사용될 수 있는데, 이는 두 번째 단계에서 남아 있는 세포가 지속자일 것으로 예상되기 때문이다. - 항생제 처리 후 시험관에서 100μL의 세포를 900μL의 멸균 인산염 완충 식염수(PBS)가 들어 있는 마이크로 원심분리 튜브로 옮깁니다. RT에서 21,000 x g 에서 3분 동안 원심분리하여 세포를 펠렛화합니다. 900μL의 상등액을 제거하고 900μL의 멸균 PBS에 펠릿을 재현탁합니다. 세척 단계를 한 번 이상 반복하여 잔류 항생제를 MIC 미만 수준으로 줄입니다.
- 배양액을 10배(6회)로 연속적으로 희석하고 각 희석액을 10μL씩 영양가 있는 한천 플레이트에 플레이트합니다. 플레이트를 37°C에서 밤새 배양합니다.
참고: 연속 희석을 위해 처리되지 않은 둥근 바닥 96웰 플레이트를 사용하고 다중 채널 피펫을 사용하여 사각 한천 플레이트에 플레이트 셀을 사용합니다. - 다음 날, 각 항생제 농도에서 군체를 세십시오. 농도에 대한 생존율(치료 종료 시 CFU/치료 전 CFU)을 로그 선형 척도로 표시합니다. 향후 지속생존 분석에 대한 약물 농도를 선택하려면 이위상 곡선의 두 번째 단계에서 농도를 선택하십시오(그림 3).
4. 항생제 치료 또는 회복 중 세포 이미징
- 타임 랩스 이미징을 위한 아가로스 패드 및 세포 샘플 준비
참고: 다음 시료 전처리 프로토콜은 기존 아가로스 "샌드위치" 방법에 대한 사용자 친화적이고 비용 효율적인 대안으로 개발되었습니다14,16. 교체 가능한 커버슬립 접시를 사용하면 샘플을 밀봉하기 위해 지저분한 그리스나 매니큐어를 칠할 필요가 없어 샘플 통기를 더욱 제한할 수 있습니다. 아가로스는 커버슬립이 이미 제자리에 있는 교체 가능한 커버슬립 접시에 설정되어 자유형 아가로스 패드 준비물에 비해 안정적인 평면 아가로스 표면을 만듭니다. 전반적으로 이 방법은 안정적인 가습으로 인해 시야 전반에 걸쳐 초점이 향상되고 더 긴 시간 동안 샘플을 이미징할 수 있습니다. 여기에 설명된 방법은 항생제 치료 중 박테리아를 시각화하는 데 사용됩니다(그림 4, 보충 비디오 3그리고 보충 비디오 4). 항생제 치료 후 지속생존자가 소생하고 다시 깨어나는 모습을 시각화하기 위해(그림 5, 보충 비디오 5그리고 보충 비디오 6), 사용한 배지 대신 신선한 배양 배지로 아가로스 패드를 준비하고 패드에 항생제를 추가하지 마십시오(플라스미드 유지/항생제 선택을 위해 추가된 것 제외).- 멸균 30mm 커버슬립(#1.5 두께)을 교체 가능한 커버슬립 접시("챔버")의 스테인리스 스틸 베이스 바닥에 놓습니다. 30mm 커버슬립이 챔버 바닥을 형성하도록 폴리카보네이트 인서트를 베이스에 부드럽게 끼우고 부착된 실리콘 O-링을 압축하여 제자리에 밀봉합니다. 이 단계를 반복하여 복제 챔버를 준비합니다.
참고: 챔버 중 하나의 준비가 최적이 아닌 경우 각 실험에 대해 복제본을 준비하는 것이 좋습니다. 30mm 커버슬립에 맞춤형 3D 프린팅 칸막이를 배치하여 녹농균(P. aeruginosa)과 같은 운동성 세포가 교차 오염되는 것을 방지하고 시료 위치에 대한 랜드마크를 제공합니다. 3D 프린팅을 위한 STL 파일은 보충 파일 1에서 사용할 수 있습니다. - 선택한 배지를 사용하여 50mL 원뿔형 튜브에 1.5% 아가로스를 준비합니다. 부드럽게 소용돌이쳐 섞습니다.
참고: 치료 중 고정상 세포를 이미징하기 위해 고정상 야간 배양에서 추출한 cell-free spent media가 base media로 사용됩니다. 일반적으로 아가로스 배지는 2개의 챔버를 준비하기 위해 7mL의 배지에 0.105g의 아가로스로 제조됩니다. 각 챔버에는 2mL의 아가로스 배지가 필요하며, 초과분은 파이펫팅 시 기포를 방지하는 데 도움이 됩니다. - 50mL 원뿔형을 유리 비커 또는 전자레인지용 홀더에 넣습니다(캡이 느슨한지 확인). 전자레인지를 센 불로 켜고 3-4초마다 멈추고 소용돌이치며 섞습니다. 아가로스 혼합물이 거품이 나는 것을 방지하기 위해 자주 일시 중지하고 혼합하십시오.
- 총 가열 시간 ~1분 후, 혼합물에 녹지 않은 아가로스가 눈에 띄는지 확인하십시오. 혼합물이 균일하게 투명하면 추가 약물이나 염료를 추가하기 전에 아가로스 배지를 ~60°C로 잠시 냉각시킵니다. 부드럽게 소용돌이쳐 섞이고 기포가 형성되지 않도록 합니다.
참고: Propidium iodide( P. aeruginosa의 경우 16μM 또는 S. aureus의 경우 1.6μM)를 추가하여 세포가 생존력을 잃을 때 시각화할 수 있습니다.
- 총 가열 시간 ~1분 후, 혼합물에 녹지 않은 아가로스가 눈에 띄는지 확인하십시오. 혼합물이 균일하게 투명하면 추가 약물이나 염료를 추가하기 전에 아가로스 배지를 ~60°C로 잠시 냉각시킵니다. 부드럽게 소용돌이쳐 섞이고 기포가 형성되지 않도록 합니다.
- 아가로스 배지가 응고되는 것을 방지하기 위해 빠르게 이동하여, 30mm 커버슬립을 사용하여 아가로스 배지 2mL를 챔버에 피펫팅합니다. 상부 챔버 개구부의 아가로스 배지 위에 멸균 25mm 커버슬립(#1.5 두께)을 부드럽게 놓습니다. 이는 아가로스 패드의 탈수를 방지하는 데 도움이 되며 최적의 위상차 이미징을 위해 아가로스의 평평한 상단 표면을 보장합니다. 패드가 1-2시간 동안 굳어지도록 합니다.
- 패드가 응고된 후, 단일 세포를 시각화하기 위해 PBS에서 적절하게 희석된 밀도로 희석하여 이미징할 세포를 준비합니다. 여기서, 세포는 OD600 0.01-0.05로 희석된다.
- 25mm 커버슬립에서 미세한 끝 영구 마커를 사용하여 패드에 파종된 각 샘플의 위치/ID를 표시합니다. 스테인리스 스틸 베이스 링이 위쪽을 향하도록 챔버를 뒤집고 베이스의 나사산을 분리하는 동안 아래에 있는 폴리카보네이트 인서트를 조심스럽게 잡습니다. 스테인리스 스틸 베이스를 옆으로 놓습니다. 패드에서 30mm 커버슬립을 조심스럽게 밀어서 버립니다.
알림: 30mm 커버슬립을 가로로 밀어 제거하고 아가로스 표면이 움푹 들어가지 않도록 주의하세요. 또한 노출된 아가로스 표면 위에서 작업하지 마십시오: 표면에 떨어지는 먼지는 이미지 품질에 영향을 미치고 패드를 잠재적으로 오염시킬 수 있습니다. - 25mm 커버슬립의 표시를 지침으로 사용하여 희석된 세포 5μL를 아가로스 패드의 해당 위치로 찾아냅니다. 3개의 5 μL 스폿을 추가합니다(예: 4 x 4 μL 스폿 등과 같이 필요에 따라 조정). 얼룩이 완전히 건조되면 새 멸균 30mm 커버슬립을 패드 중앙에 가볍게 놓습니다.
- 한 손으로 폴리카보네이트 인서트를 잡고 다른 손으로 새 커버슬립 위에 스테인리스 스틸 베이스를 천천히 다시 끼웁니다. 손가락으로 꽉 조일 때까지 나사산을 끼우는 것만으로도 실리콘 O-링을 압축하고 챔버를 밀봉할 수 있습니다.
참고: 과도하게 조이지 않도록 주의하십시오 - 샘플 표면에 대한 30mm 커버슬립이 스테인리스 스틸 베이스에 의해 압축되고 반복적으로 비틀리면 셀이 방사형 패턴으로 퍼져 잠재적으로 샘플 교차 오염을 유발할 수 있습니다. - 챔버가 밀봉되면 아가로스가 30mm 커버슬립의 표면과 접촉하는지 관찰합니다. 이 시점에는 일반적으로 완전하지는 않은 일부 표면 접촉이 있습니다. 핀셋 또는 유사한 도구의 뭉툭한 끝을 사용하여 아가로스가 전체 표면에 걸쳐 25mm 커버슬립에 고르게 닿을 때까지 30mm 커버슬립을 부드럽게 누릅니다.
주의 : 이 단계에서는 과도한 힘을 가하면 25mm 커버슬립이 파손될 수 있습니다. - 아가로스가 30mm 커버슬립에 잘 눌려 큰 기포가 남지 않도록 하면 패드를 이미지화할 준비가 된 것입니다.
- 멸균 30mm 커버슬립(#1.5 두께)을 교체 가능한 커버슬립 접시("챔버")의 스테인리스 스틸 베이스 바닥에 놓습니다. 30mm 커버슬립이 챔버 바닥을 형성하도록 폴리카보네이트 인서트를 베이스에 부드럽게 끼우고 부착된 실리콘 O-링을 압축하여 제자리에 밀봉합니다. 이 단계를 반복하여 복제 챔버를 준비합니다.
- 현미경과 이미지를 설정합니다.
- 현미경 및 이미징 챔버 환경 제어를 준비합니다. 스테이지 상단 인큐베이터와 대형 챔버 인큐베이터 온도를 37°C로 설정하고 챔버 가습기를 켭니다. 아가로스 패드 제제 중 하나를 스테이지 상단 인큐베이터에 넣고 인큐베이션 챔버를 닫아 샘플이 평형을 이룰 수 있도록 합니다. 스테이지 탑 인큐베이터 가습 뚜껑은 챔버의 25mm 커버슬립에 응결이 생기는 것을 방지하기 위해 이 시점에서 열어 둡니다.
참고: 온도 변화로 인한 패드 수축/팽창으로 인해 자동 초점 알고리즘의 스캔 범위를 벗어나는 시점 간 드리프트가 발생할 수 있습니다. 이미징 전에 아가로스 패드를 안정적인 온도로 평형을 이루는 것이 중요하며, 이는 일반적으로 최소 15분이 소요됩니다. 또한 충분한 예열은 가습 뚜껑을 제자리에 놓았을 때 상단 커버슬립에 결로 현상이 발생하는 것을 방지합니다. 이미징 시스템의 하드웨어 구성 요소는 다음과 같습니다(재료 표): 라이브 셀 배양 챔버, Plan-Apochromat 63x/1.40 Oil Ph3 M27 대물렌즈가 있는 도립 현미경, Spectra 7 LED 조명 엔진 및 sCMOS 카메라(6.5μm 픽셀 크기). 현미경의 전동 구성 요소 제어 및 이미지 획득은 현미경 분석 소프트웨어 애플리케이션을 사용하여 수행됩니다. - 이미지 획득을 위한 소프트웨어를 준비합니다. MetaMorph(현미경 분석 소프트웨어)의 내장 자동 초점 알고리즘을 설정하여 다차원 획득 중에 위상 채널을 사용합니다.
- Stage 탭에서 각 샘플에 대해 여러 스테이지 위치를 설정하여 셀 밀도가 균일하게 분포된 시야를 목표로 합니다. 타임랩스 탭에서 촬영할 이미지의 원하는 지속 시간과 빈도(시간 간격)를 설정합니다. Wavelengths(파장) 탭에서 원하는 수집 채널을 설정하고 샘플의 신호 강도에 대한 노출 시간을 조정합니다.
참고: 각 위치의 이미지는 최대 24시간 동안 10분마다 촬영됩니다. 이 기사에서 언급한 염료/형광단의 형광 이미징을 위해 프로피듐 요오드화물(Cy3, 555/15nm) 및 GFP(GFP/FITC, 470/24nm)와 같은 광 엔진 여기 설정이 사용되었습니다. 사용되는 일반적인 형광 여기 노출 시간은 100ms입니다. 프로피듐 요오드화물용 필터 세트 15(Beamsplitter FT580, Emission LP590)와 GFP용 필터 세트 44(Beamsplitter FT500, Emission BP 530/50)가 사용됩니다.
- Stage 탭에서 각 샘플에 대해 여러 스테이지 위치를 설정하여 셀 밀도가 균일하게 분포된 시야를 목표로 합니다. 타임랩스 탭에서 촬영할 이미지의 원하는 지속 시간과 빈도(시간 간격)를 설정합니다. Wavelengths(파장) 탭에서 원하는 수집 채널을 설정하고 샘플의 신호 강도에 대한 노출 시간을 조정합니다.
- 챔버에서 아가로스 패드가 예열된 후 가습 뚜껑을 스테이지 상단 인큐베이터에 추가합니다. 이렇게 하면 이미징 중에 패드가 탈수되거나 이동하는 것을 방지하는 데 도움이 됩니다.
- 가습 덮개를 켠 상태에서 위상에서 미분 간섭 대비(DIC)로 변경하고 적절한 Köhler 조명을 위해 현미경의 콘덴서와 조리개 조리개를 조정합니다.
- 조정 후 다시 위상으로 전환하십시오. 각 스테이지 위치로 이동하고, 초점을 조정하고, 스테이지 위치를 새 초점면으로 재설정합니다. 이제 이미징을 시작할 수 있는 설정이 준비되었습니다.
알림: 패드가 이동하지 않았는지 확인하기 위해 획득을 시작하기 전에 각 단계 위치를 다시 방문하는 것이 중요합니다. 주어진 스테이지 위치에서 다시 볼 때 셀의 초점이 맞지 않으면 셀에 다시 초점을 맞추고 이전 스테이지 위치를 재정의합니다. 자동 초점 알고리즘 설정은 3μm± 마지막 시점의 초점 z 위치를 검색 범위로 사용하므로 패드가 3μm 이상 이동하면 알고리즘이 제대로 초점을 맞추지 못합니다. - Acquire 버튼을 클릭하여 다차원 수집을 시작합니다.
- 실험이 완료되면 각 채널의 개별 이미지를 비디오로 보거나 다른 분석을 위해 스택으로 컴파일합니다. MetaMorph 또는 ImageJ를 사용하여 이 작업을 수행합니다.
- MetaMorph(선호)에서 이미지를 컴파일하려면 Review Multi Dimensional Data 앱에서 관심 채널/파장을 선택하고 지정된 스테이지 위치에 대한 모든 시점을 선택합니다. 이미지 로드를 클릭합니다. 각 채널/파장에 대해 창이 나타납니다. 각 컴파일을 해당 채널 이름과 함께 .tiff 파일로 저장합니다.
- ImageJ에서 컴파일하려면 한 스테이지 위치와 한 채널에 대한 모든 시점의 파일을 엽니다. 이미지를 사용하여 스택으로 컴파일합니다.
- 현미경 및 이미징 챔버 환경 제어를 준비합니다. 스테이지 상단 인큐베이터와 대형 챔버 인큐베이터 온도를 37°C로 설정하고 챔버 가습기를 켭니다. 아가로스 패드 제제 중 하나를 스테이지 상단 인큐베이터에 넣고 인큐베이션 챔버를 닫아 샘플이 평형을 이룰 수 있도록 합니다. 스테이지 탑 인큐베이터 가습 뚜껑은 챔버의 25mm 커버슬립에 응결이 생기는 것을 방지하기 위해 이 시점에서 열어 둡니다.
5. Fiji/ImageJ를 사용하여 타임랩스 비디오 만들기
참고: 피지(피지는 ImageJ)는 이미지 처리 및 분석을 위해 무료로 사용할 수 있는 소프트웨어이며 "https://imagej.net/software/fiji/downloads32"32에서 다운로드할 수 있습니다. Fiji/ImageJ2 1.54f는 아래와 같은 이미지 처리 방법에 사용되었습니다.
- BaSiC33을 사용하여 위상 채널 이미지 스택을 음영 보정합니다. 피지에서 원하는 위상 이미지 스택을 연 다음 플러그인 탭에서 BaSiC를 선택합니다. 음영 보정된 스택은 Corrected:ImageName이라는 별도의 창에 나타납니다.
참고: BaSiC.jar 여기에서 다운로드할 수 있습니다: "https://github.com/marrlab/BaSiC33"33. 피지에 BaSiC를 올바르게 설치하려면 개발자의 지침을 따르십시오. - Image > Color > Merge Channels를 사용하여 음영 보정된 위상 채널 스택을 다른 채널/파장과 병합합니다. 각 채널의 밝기/대비를 조정> Image > 를 사용하여 배경 및 신호 강도를 조정합니다. 병합된 파일을 .tiff로 저장합니다.
- 그런 다음 올바른 3D 드리프트를 사용하여 이미지를 안정화합니다. 음영 보정된 스택을 선택한 상태에서 Plugins > Registrations > Correct 3D Drift로 이동합니다. 열리는 대화 상자 창에서 위상 스택에 해당하는 채널 번호로 등록할 채널을 설정합니다. 수정 후 결과 스택의 제목은 등록된 시점으로 지정됩니다. 원하는 시야로 자른 다음 수정된 파일을 .tiff로 저장합니다.
참고: 컴파일된 이미지 스택을 생성하기 위해 MetaMorph를 사용한 경우, 추가 처리 단계가 있습니다: 이미지 > 속성으로 이동하여 슬라이스 (z) 및 프레임 (t) 필드의 숫자를 바꿉니다. 이제 드리프트 보정은 각 프레임을 z 스택의 조각이 아닌 시점으로 올바르게 해석할 수 있습니다. 음영 보정 및 드리프트 보정 .tiff 파일은 후속 이미지 분석에 사용할 수 있습니다. 형광 신호 강도를 측정하고, 형태학적 특성을 정량화하고, 개별 세포의 운명을 추적하는 등의 작업을 수행하는 데 사용할 수 있는 많은 소프트웨어 패키지가 있습니다. 일반적으로 사용되는 두 가지 프로그램은 Fiji용 MicrobeJ 플러그인과 Oufti34,35입니다. - Image > Stacks > Label을 사용하여 스택에 타임스탬프와 텍스트 레이블을 추가합니다.
- 스케일 바를 추가하려면 먼저 카메라의 픽셀 크기와 현미경 대물렌즈 배율을 알아야 합니다. 픽셀:미크론 비율을 픽셀 크기를 배율로 나눈 값으로 계산합니다. Analyze > Set Scale 대화 상자에서 known distance 필드에 픽셀:미크론 비율을 입력합니다. 그런 다음 Analyze > Tools > Scale Bar를 사용하여 스택에 배율 막대를 추가합니다.
알림: 현미경의 정확한 스케일은 스테이지 마이크로미터를 사용하여 보정해야 합니다. 그러나 사용된 현미경 대물 렌즈와 카메라는 스케일의 대략적인 추정치를 제공할 수 있습니다. 예를 들어, 여기에 사용된 PCO sCMOS 카메라의 픽셀 크기는 6.5 x 6.5 μm2이고 63x 대물렌즈는 이미징에 사용되었으므로 픽셀 크기를 배율로 나눈 값은 픽셀당 6.5/63 = 0.1032 μm입니다. 배율 설정 대화 상자의 알려진 거리 필드에 0.1032를 입력합니다. - 완성된 이미지 스택을 QuickTime Player, Microsoft PowerPoint 등에서 재생할 수 있는 비디오로 내보내려면 .avi 파일로 저장합니다.
결과
녹농균(P. aeruginosa) 및 황색포도상구균(S. aureus)에 리포터 플라스미드(reporter plasmids)를 성공적으로 도입하는 것은 올바른 선택적 항생제의 성장에 의해 나타나며 콜로니 PCR 및/또는 염기서열분석(colony PCR) 및/또는 염기서열분석(sequencing)으로 확인할 수 있습니다. 변형된 균주는 관심 유전자가 유도되는 것으로 알려진 조건에 적용하여 표현형 리포터로 검증되어야 하며, 그 결과로 발생하는 형광은 유세포 분석, 분광 광도법 또는 상피 형광 현미경으로 측정할 수 있습니다(그림 1).
후속 실험에 사용할 항생제 용량을 쉽게 선택할 수 있도록 관심 있는 녹농 균 또는 S. 황색포도상 균 균주에 대한 농도 의존성 항생제 지속요법 분석을 수행합니다. 농도 의존적 분석은 일반적으로 낮은 항생제 농도에서 가파른 초기 경사와 높은 농도에서 고원 또는 덜 가파른 경사를 가진 이위상 곡선을 초래합니다. 그러나 일부 항생제 종 쌍의 경우 뚜렷한 이위상 곡선이 발생하지 않을 수 있습니다. 예를 들어, S. aureus delafloxacin 곡선에 대한 곡선은 분명히 이위상이지만(그림 3A), P. aeruginosa levofloxacin 곡선은 그렇지 않습니다(그림 3B)15. 이 시나리오에서는 MIC의 10배 이상인 농도를 선택합니다(예: 녹 농균의 경우 MIC의 약 15배인 5μg/mL)15. 그러나 15x 레보플록사신 MIC는 녹농균 생존율이 ~0.001%에 불과하기 때문에 항생제가 없는 아가로스 패드에서 회복되는 세포를 이미징할 때 지속생존자를 확인하고자 하는 경우 1μg/mL 레보플록사신 치료를 사용합니다(보충 비디오 6). 그렇지 않으면 여러 지속자를 이미지화하는 데 필요한 시야의 수가 엄청나게 많아집니다.
이미징을 시작할 때 이상적인 샘플과 아가로스 패드 준비는 시야 전체에 걸쳐 평면으로 나타나야 하며, 큰 파편, 주름 또는 기포가 없고, 단일 세포가 고르게 분포되어 있어야 합니다. 잘 분포된 단일 세포를 얻으려면 시료 희석 또는 재현탁의 최적화가 필요할 수 있습니다. S. aureus의 경우, 세포는 작은 클러스터를 형성하는 경향이 있으며 아가로스 패드에 파종하기 전에 철저히 와류화해야 합니다(그림 4 및 그림 5). 녹농균(P. aeruginosa)의 경우, 세포는 현탁액의 끈적한 세포외 기질로 둘러싸인 응집체를 형성할 수 있습니다. 이러한 샘플을 철저히 피펫팅하고 단일 세포 이미징을 위해 응집체를 파괴해야 합니다.
이미징 실험이 종료된 후 이미지의 성공적인 타임랩스는 실험 전반에 걸쳐 x-y 평면의 드리프트를 최소화하면서 초점이 맞춰지고 안정적으로 조명되며 나타납니다. Supplementary Video 7 은 최적의 이미지 획득을 나타내며, t는 음영 또는 드리프트 보정 전 Supplementary Video 4 의 위상 채널입니다. 응결(과도한 가습 또는 불충분한 샘플 온난화로 인한)으로 인해 상단 25mm 커버슬립에 물방울이 형성되어 빛이 왜곡되고 초점면이 자동 초점 알고리즘의 최대 검색 범위 밖으로 밀려나는 경우 초점 손실이 발생할 수 있습니다(보충 비디오 8). 조명의 변화는 일반적으로 이미징 시 이멀젼 오일이 충분하지 않음을 나타냅니다. 스테이지가 너무 빨리 움직이면 대물렌즈의 오일이 뒤로 끌려갈 수 있으며 이미지를 획득할 때 여전히 따라잡을 수 있습니다. 이는 이동 속도를 늦추도록 획득 컨트롤을 조정하거나 다음 위치로의 이동과 이미지 획득 사이에 일시 중지를 추가하여 완화할 수 있습니다. 주요 샘플 드리프트는 일부는 제자리에 머무르는 동안 시야를 가로질러 줄무늬를 이루는 많은 세포처럼 보일 것입니다(보충 비디오 9). 이것은 일반적으로 실험 후반부에 발생하는데, 이는 아가로스 패드가 불충분한 습도 조절로 인해 탈수되었기 때문입니다. 이 백서에 제시된 아가로스 패드 준비는 샘플 안정성을 용이하게 하도록 설계되었지만, 최적의 이미지 획득을 위해서는 샘플과 주변 환경을 적절하게 가온/가습하는 것이 필요합니다.
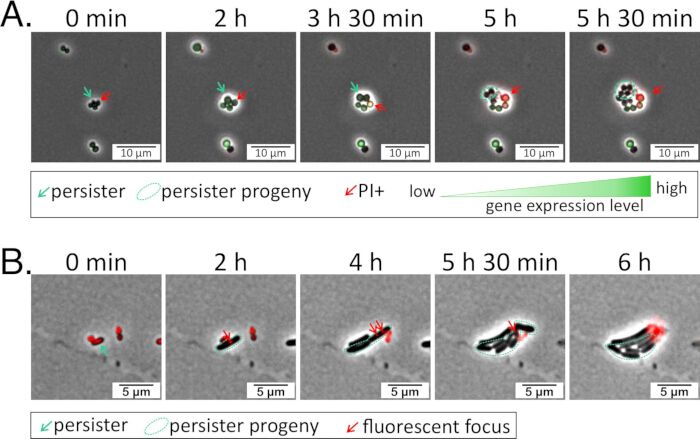
그림 1: 형광 리포터 균주는 관심 유전자의 발현을 조명합니다. (A) S. aureus 는 프로토콜 1에 따라 관심 유전자에 대한 GFP 전사 리포터로 형질주입되었습니다. 리포터 균주를 항생제로 24시간 동안 처리하고, PBS로 세척한 다음, CA-MHB와 요오드화 프로피듐(1.6μM) 및 클로람페니콜(리포터 플라스미드 유지를 위한 10μg/mL)로 만든 아가로스 패드에 파종하여 회복 중 이미징을 수행했습니다(보충 비디오 1). (B) 녹농균(P. aeruginosa )은 관심 단백질에 대한 mScarlet-linked translational reporter를 함유한 플라스미드로 형질전환시켰다26. 리포터 균주를 항생제로 5시간 동안 처리하고, PBS로 세척한 다음, 회복 중 이미징을 위해 BSM과 Tet(75μg/mL, 리포터 플라스미드 유지용)으로 만든 아가로스 패드에 파종했습니다(보충 비디오 2). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
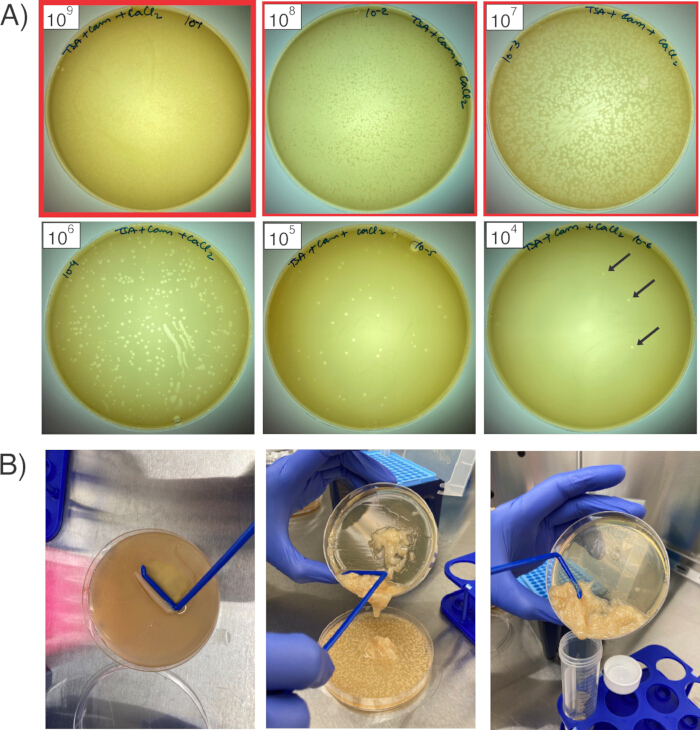
그림 2: 박테리오파지의 번식 및 수확. (A) 6개의 플레이트는 S. aureus RN4220의 잔디밭에 있는 6개의 다른 양의 희석된 파지 스톡을 보여줍니다. 빨간색 윤곽선은 투명도가 가장 높은 플레이트(굵은 빨간색 윤곽선, 1 x109 PFU/mL)부터 다음 두 희석액(1 x108 및 1 x 107 PFU/mL)까지 수확할 3개의 플레이트를 나타냅니다. 검은색 화살표는 개별 명판을 가리킵니다. (B) 플레이트에서 파지를 회수하려면 부드러운 한천 층을 긁어내고(왼쪽), 슬러리를 다음 희석 플레이트(가운데)로 옮기고, 세 플레이트의 부드러운 한천을 모두 함께 모은 후 원심분리를 위한 원뿔형 튜브로 결합합니다(오른쪽). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
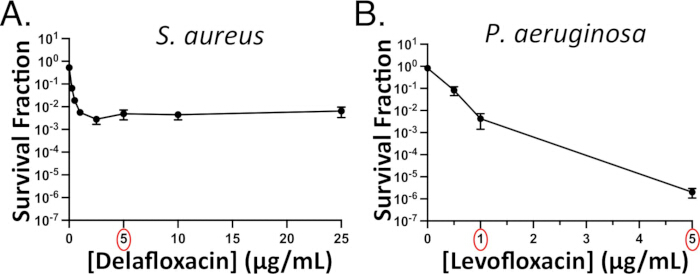
그림 3: 대표적인 농도 의존적 지속형 분석법. 농도 의존성 플루오로퀴놀론 지속성은 고정상(A) S. aureus(델라플록사신에 대하여) 및 (B) P. aeruginosa(레보플록사신에 대하여)에서 평가되었습니다. 후속 실험에서는 5μg/mL 델라플록사신(빨간색 원)을 사용했는데, 이는 S. aureus 사멸이 이 농도에서 정체되었기 때문입니다. 최소 1μg/mL의 레보플록사신(빨간색 원)의 투여량은 녹농균에 대한 후속 실험에 사용됩니다. 박테리아 사멸은 녹농균에 대해 정체되지 않지만, 지속적인 하위 집단을 나타내는 이위상 곡선의 덜 가파른 "두 번째 단계"가 여전히 있습니다. 패널 3B는 Hare et al.15의 허가를 받아 조정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
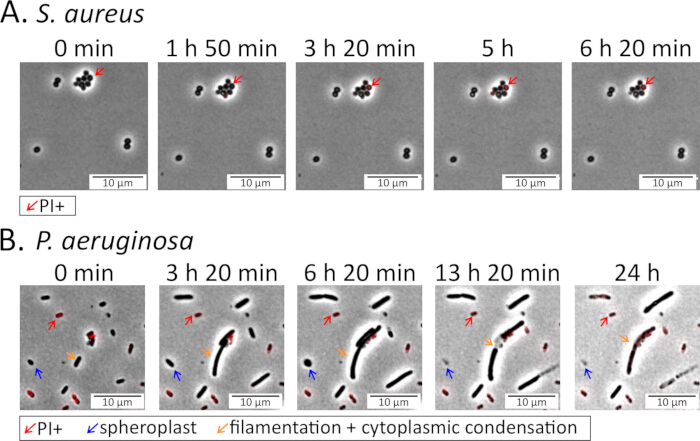
그림 4: 항생제 치료 중 박테리아 표현형 이미징. 고정상(A) S. aureus 및 (B) P. aeruginosa 세포를 플루오로퀴놀론 항생제가 함유된 아가로스 패드에 파종하고 치료 중에 모니터링했습니다: S. aureus 에 대한 5μg/mL delafloxacin(보충 비디오 3) 및 P. aeruginosa 에 대한 5μg/mL levofloxacin(보충 비디오 4)15. Propidium iodide(PI, 녹농균의 경우 16μM, S. aureus의 경우 1.6μM)를 패드에 첨가하여 죽거나 죽어가는 세포를 표시했습니다. S. aureus 세포는 FQ가 있는 상태에서 대체로 손상되지 않고 살아 있는 반면, 대부분의 녹농 균 세포는 용해되어 죽기 전에 둥근 구형체를 형성하는 것을 포함하여 급격한 형태학적 변화를 겪습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
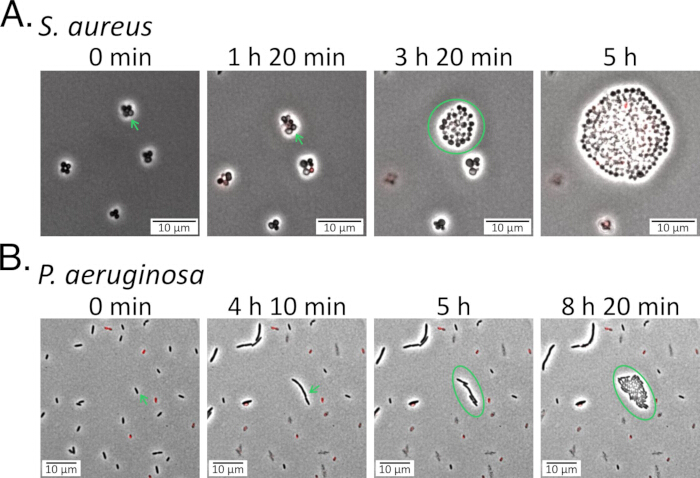
그림 5: 회복 중 지속인자 추적. (A) S. aureus 및 (B) P. aeruginosa 집단은 플루오로퀴놀론( S. aureus 의 경우 5μg/mL delafloxacin, 녹농균의 경우 1μg/mL levofloxacin)으로 처리한 후 신선한 배지가 들어 있는 아가로스 패드에 파종하고 치료 후 회복 중에 모니터링했습니다(보충 동영상 5 및 보충 동영상 6). 보이는 perisisters는 각 패널의 처음 두 프레임에 녹색 화살표로 표시되며 항생제 치료 중에도 손상되지 않고 생존 할 수 있습니다. 초기 지연 기간이 지난 후, 지속자는 분열하기 시작했고 새로운 자손(녹색 원으로 표시)을 낳았습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
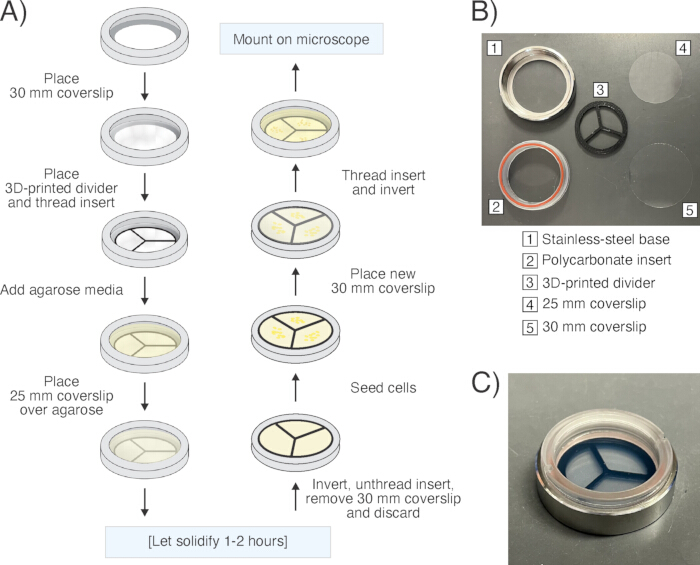
그림 6: 현미경 시료 준비. (A) 교체 가능한 커버슬립 접시("챔버")를 사용한 샘플 준비 작업 흐름의 개략도. (B) 분해된 챔버 및 개별 구성 요소의 그림. (C) 완전히 조립된 챔버의 사진. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 비디오 1: S. aureus persister. 그림 1A의 이미지를 포함하는 비디오 파일. 요컨대, 관심 유전자에 대한 GFP 전사 리포터를 지닌 S. aureus 를 항생제로 24시간 동안 처리하고, PBS로 세척한 다음, 회복 중 이미징을 위해 CA-MHB와 프로피듐 요오드화물(1.6μM) 및 클로람페니콜(10μg/mL)로 만든 아가로스 패드에 파종했습니다. 이 비디오를 다운로드하려면 여기를 클릭하십시오.
보충 비디오 2: 녹농 균 지속성. 그림 1B의 이미지를 포함하는 비디오 파일. 요컨대, 관심 단백질에 대한 mScarlet-linked translational reporter를 지닌 녹농균을 항생제로 5시간 동안 처리하고, PBS로 세척한 다음, 회복 중 이미징을 위해 BSM과 Tet(75μg/mL)로 만든 아가로스 패드에 파종했습니다26. 이 비디오를 다운로드하려면 여기를 클릭하십시오.
보충 영상 3: 항생제 치료 중 황색포도상구균. 화학적으로 규명된 풍부한 배지에서 성장한 S. aureus의 고정상 배양물을 프로피듐 요오드화물(1.6μM) 및 델라플록사신(5μg/mL)이 함유된 배양의 무세포 컨디셔닝 배지로 만든 아가로스 패드에 파종했습니다. 이 비디오를 다운로드하려면 여기를 클릭하십시오.
보충 비디오 4: 항생제 치료 중 녹농 균. BSM에서 성장한 녹농균의 고정상(stationary-phase) 배양액을 BSM에서 병렬로 성장한 녹농균(P. aeruginosa) 배양물로부터 무세포 조건화 배지로 만든 아가로스 패드에 파종하였다. 아가로스 패드에는 프로피듐 요오드화물(16μM)과 레보플록사신(5μg/mL)도 함유되어 있습니다. 이 비디오는 Hare et al.15의 허가를 받아 각색되었습니다. 이 비디오를 다운로드하려면 여기를 클릭하십시오.
보충 비디오 5: 항생제 투여 후 회복 중 S. aureus. S. aureus는 화학적으로 규명된 풍부한 배지에서 고정상으로 성장했습니다. 고정상 배양물을 시험관에서 5μg/mL 델라플록사신으로 24시간 동안 처리하고, PBS로 세척한 다음, 이미징을 위해 프로피듐 요오드화물(1.6μM)을 함유하는 항생제가 없는 CA-MHB 아가로스 패드에 파종했습니다. 이 비디오를 다운로드하려면 여기를 클릭하십시오.
보충 비디오 6: 항생제 후 회복 중 녹농 균. BSM에서 성장한 녹농균의 고정상배양을 시험관에서 1μg/mL 레보플록사신으로 7시간 동안 처리하고, PBS로 세척한 다음, 이미징을 위해 프로피듐 요오드화물(16μM)을 함유하는 무항생제 BSM 아가로스 패드에 파종했습니다. 이 비디오를 다운로드하려면 여기를 클릭하십시오.
보충 동영상 7: 최적 이미지 획득의 예. 이 비디오는 최적의 타임랩스 획득의 예로서 이미지 처리 전 보충 비디오 4 의 위상 채널입니다. 실험 전반에 걸쳐 최소한의 드리프트, 안정적인 조명 및 초점 유지에 주목하십시오. 이 비디오를 다운로드하려면 여기를 클릭하십시오.
보충 비디오 8: 응결로 인한 최적이 아닌 이미지 획득의 예. 이 비디오는 샘플의 부적절한 가열 및/또는 이미징 환경의 과도한 가습으로 인한 챔버의 응결로 인해 이미지 획득이 잘못된 초점의 영향을 받은 실험의 일부를 보여줍니다. 이미징되는 샘플은 BSM 아가로스 패드에서 항생제 후 회수 중 레보플록사신으로 처리된 녹농 균이었습니다. 이 비디오를 다운로드하려면 여기를 클릭하십시오.
보충 비디오 9: 드리프트로 인한 최적이 아닌 이미지 획득의 예. 이 비디오는 이미지 획득이 샘플 드리프트의 영향을 받았을 때 실험의 일부를 보여주는데, 이는 탈수 및 커버슬립에서 아가로스 패드의 수축/들림으로 인한 것일 수 있습니다. 이미징된 샘플은 레보플록사신(levofloxacin)과 프로피듐 요오드화물(propidium iodide)을 함유한 아가로스 패드의 녹농균(P. aeruginosa )이었습니다. 이 비디오를 다운로드하려면 여기를 클릭하십시오.
보충 파일 1: 25mm-3D-divider-for-35mmBioptechs.stl 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
우리는 타임 랩스 현미경 실험의 성공 여부가 아가로스 패드의 품질과 이미징 과정 전반에 걸친 안정성에 달려 있다는 것을 발견했습니다. 스테인리스 스틸 챔버로 둘러싸인 아가로스 패드는 비교적 쉽게 준비할 수 있어 수십 시간에 걸쳐 안정적으로 이미징할 수 있는 일관된 평면 샘플을 얻을 수 있습니다. 이를 통해 단일 실험에서 수만 개의 세포를 이미징할 수 있으며 집단에서 지속세포 세포와 같은 희귀한 표현형 변이체를 검출할 가능성을 높일 수 있습니다.
이 아가로스 패드 준비 방법은 이전에 발표된 방법에 대한 쉽게 구현할 수 있는 대안을 제시합니다. 당사의 프로토콜은 미세유체 장치 제조의 기술적 정밀도나 아가로스 "샌드위치" 방법의 민첩한 조작을 필요로 하지 않으므로 실행에서 실행까지 일관된 준비를 더 쉽게 달성할 수 있습니다 14,16,36. 또한 이 시스템은 비용 효율적입니다. 스테인리스 스틸 챔버는 멸균 및 재사용이 가능하며(일회용 플라스틱 챔버와 달리) 특수 장비가 필요하지 않습니다16,37. 이 챔버는 시중에서 판매되는 스테이지 인서트를 사용하는 다양한 현미경 시스템에 쉽게 장착할 수 있습니다. 또한 박테리아는 아가로스 커버 유리 계면에서 고정화되어 있기 때문에 녹농균과 같은 운동성이 높은 박테리아를 추적하면서 형태학적 변화를 허용하는 데 성공했습니다(그림 4, 보충 비디오 4). "마더 머신(mother machine)"과 같은 다른 단세포 이미징 기술은 세포를 필라멘트36 이외의 형태학적 변화의 관찰을 배제하는 채널로 제한합니다.
이 프로토콜의 성공을 위해 염두에 두어야 할 몇 가지 중요한 단계와 매개 변수가 있습니다. 패드 준비의 경우 남아 있는 아가로스 결정이 광 회절을 일으키고 이미지 품질에 영향을 미치므로 아가로스를 완전히 가열하고 녹이는 것이 중요합니다. 마찬가지로, 기포가 유입되지 않도록 아가로스를 챔버 안으로 피펫팅하도록 주의해야 합니다. 아가로스 패드의 두께가 일정하게 유지되고 샘플 드리프트를 제한하려면 이미징을 시작하기 전에 패드가 가습되고 온도가 제어되는 인클로저에서 평형을 이루도록(일반적으로 15분 동안) 하는 것이 중요합니다. 이미지 품질 저하를 유발할 수 있는 또 다른 요인은 습도 제어입니다: 습도가 낮으면 아가로스 패드가 탈수 및 수축되는 반면, 습도가 높으면 (또는 챔버 내의 부적절한 샘플 가열) 따뜻한 공기가 샘플에 응결되어 이미징을 왜곡할 수 있습니다. 응결로 인한 최적화되지 않은 타임 랩스 이미징의 예는 보충 비디오 8에서 찾을 수 있습니다.
현재 설정의 한계는 배양 배지를 교환할 수 없어 항생제 처리 전, 치료 중, 후에 개별 박테리아를 지속적으로 추적할 수 없다는 것입니다. 우리는 아가로스 패드를 배양 배지 교환을 허용하는 플로우 셀 또는 미세유체 장치와 결합하면 영양 또는 환경 변화 중에 개체군을 추적할 수 있을 것으로 예상합니다. 현재 설계에서 개선할 수 있는 또 다른 매개변수는 샘플 폭기입니다. 교체 가능한 커버슬립 접시의 O-링 씰, 나사 상단 디자인은 패드16을 밀봉하기 위해 왁스 또는 그리스 기반 실런트를 사용해야 하는 설정에 비해 더 나은 샘플 통기를 가능하게 합니다. 그러나 밀폐된 챔버에서의 폭기는 여전히 제한될 수 있으며 의무 호기성의 성장을 지원하지 않을 수 있지만 이는 아직 테스트되지 않았습니다.
이 기사에서 제시하는 타임 랩스 이미징 샘플 준비 프로토콜을 사용하면 항생제 치료에 반응하거나 회복하는 수천 개의 박테리아를 추적할 수 있습니다. 이 방법은 또한 일반화가 매우 용이하며 지속형 생물학을 넘어 다양한 잠재적 응용 분야를 가지고 있습니다. 예를 들어, 아가로스 패드 및 디바이더 설정은 공간적으로 분리된 세포 샘플의 시딩을 허용하면서도 아가로스 패드를 통한 확산을 통한 세포 간 통신을 허용합니다. 우리는 현재 분비된 산물의 교환이 다종 군집의 세포 성장에 어떤 영향을 미치는지 테스트하기 위해 이 설정의 잠재력을 탐구하고 있습니다. 우리는 이 프로토콜이 새로운 연구자에게는 타임 랩스 현미경에 대한 낮은 진입 장벽을 제공하고 노련한 미생물학자에게는 무한한 변형을 제공할 것으로 기대합니다.
공개
저자는 선언할 이해 상충이 없습니다.
감사의 말
현미경 실험에 도움을 주신 UConn Health Center for Cell Analysis and Modeling Microscopy Facility의 Susan Staurovsky에게 감사드립니다. 녹농균( P. aeruginosa )과 황색포도상구균(S . aureus)의 복제에 대한 프로토콜과 조언을 제공해 주신 Mona Wu Orr 박사와 Essentials of Staphylococcal Genetics and Metabolism Workshop에 감사드립니다. 이 연구는 미국 국립보건원(NIH; DP2GM146456-01 및 1R01AI167886-01A1은 W.W.K.M.으로, 1F30DE032598-01A1은 P.J.H.로, 1F31DK136259-01A1은 T.J.L.로). 자금 제공자는 우리의 실험을 설계하거나 이 원고를 준비하는 데 아무런 역할도 하지 않았습니다.
자료
| Name | Company | Catalog Number | Comments |
| BaSiC | GitHub | https://github.com/marrlab/BaSiC | |
| Certified Molecular Biology Agarose | Biorad | 1613101 | |
| Fiji-ImageJ | NIH | https://imagej.net/software/fiji/downloads | |
| Interchangeable Coverglass Dish | Bioptechs | 190310-35 | 35 mm ICD for preparing agarose pads; comes with 30 mm (#1.5) coverslips |
| Lumencor Spectra 7 LED light engine | Lumencor | https://lumencor.com/products/spectra-light-engine | Spectra 7 LED light engine |
| MetaMorph | Molecular Devices | Premier version 7.10.5 | |
| pco.edge 4.2 bi sCMOS camera | Excelitas | https://www.excelitas.com/product/pcoedge-42-bi-usb-scmos-camera | sCMOS camera |
| PeCon live cell incubation chamber | peCon | https://www.pecon.biz/ | |
| Thomas Scientific Round cover glass, #1.5 thickness, 25 mm, 100 pack | Fisher Scientific | NC1272770 | 25 mm (#1.5) coverslips |
| Zeiss Axiovert 200M microscope | Zeiss | https://www.zeiss.com/microscopy/en/products/light-microscopes/widefield-microscopes/axiovert-for-materials.html | Inverted microscope with Plan-Apochromat 63x/1.40 Oil Ph3 M27 objective, Lumencor Spectra 7 LED light engine, and pco.edge 4.2 bi sCMOS camera (6.5 mm pixel size) |
참고문헌
- Brauner, A., Fridman, O., Gefen, O., Balaban, N. Q. Distinguishing between resistance, tolerance and persistence to antibiotic treatment. Nat Rev Microbiol. 14, 320-330 (2016).
- Balaban, N. Q., et al. Definitions and guidelines for research on antibiotic persistence. Nat Rev Microbiol. 17 (7), 441-448 (2019).
- Conlon, B. P. Staphylococcus aureus chronic and relapsing infections: evidence of a role for persister cells. Bioessays. 36 (10), 991-996 (2014).
- Mulcahy, L. R., Burns, J. L., Lory, S., Lewis, K. Emergence of Pseudomonas aeruginosa strains producing high levels of persister cells in patients with cystic fibrosis. J Bacteriol. 192 (23), 6191-6199 (2010).
- Verstraete, L., Van den Bergh, B., Verstraeten, N., Michiels, J. Ecology and evolution of antibiotic persistence. Trends Microbiol. 30 (5), 466-479 (2021).
- Sett, A., Dubey, V., Bhowmik, S., Pathania, R. Decoding bacterial persistence: Mechanisms and strategies for effective eradication. ACS Infect Dis. 10 (8), 2525-2539 (2024).
- Mempin, R., et al. Release of extracellular ATP by bacteria during growth. BMC Microbiol. 13, 301 (2013).
- Lewis, K. Persister cells, dormancy and infectious disease. Nat Rev Microbiol. 5 (1), 48-56 (2006).
- Fauvart, M., De Groote, V. N., Michiels, J. Role of persister cells in chronic infections: clinical relevance and perspectives on anti-persister therapies. J Med Microbiol. 60 (pt 6), 699-709 (2011).
- Van den Bergh, B., Fauvart, M., Michiels, J. Formation, physiology, ecology, evolution and clinical importance of bacterial persisters. FEMS Microbiol Rev. 41, 219-251 (2017).
- Levin, B. R., Rozen, D. E. Non-inherited antibiotic resistance. Nat Rev Microbiol. 4 (7), 556-562 (2006).
- Davis, K. M., Isberg, R. R. Defining heterogeneity within bacterial populations via single cell approaches. Bioessays. 38 (8), 782-790 (2016).
- Hare, P. J., LaGree, T. J., Byrd, B. A., DeMarco, A. M., Mok, W. W. K. Single-cell technologies to study phenotypic heterogeneity and bacterial persisters. Microorganisms. 9 (11), 2277 (2021).
- de Jong, I. G., Beilharz, K., Kuipers, O. P., Veening, J. W. Live cell imaging of Bacillus subtilis and Streptococcus pneumoniae using automated time-lapse microscopy. J Vis Exp. 53, e3145 (2011).
- Hare, P. J., Gonzalez, J. R., Quelle, R. M., Wu, Y. I., Mok, W. W. K. Metabolic and transcriptional activities underlie stationary-phase Pseudomonas aeruginosa sensitivity to levofloxacin. Microbiol Spectr. 12 (1), e0356723 (2024).
- Young, J. W., et al. Measuring single-cell gene expression dynamics in bacteria using fluorescence time-lapse microscopy. Nat Protoc. 7 (1), 80-88 (2011).
- Pang, Y. Y., et al. agr-Dependent interactions of Staphylococcus aureus USA300 with human polymorphonuclear neutrophils. J Innate Immun. 2 (6), 546-559 (2010).
- Bose, J. L., Fey, P. D., Bayles, K. W. Genetic tools to enhance the study of gene function and regulation in Staphylococcus aureus. Appl Environ Microbiol. 79 (7), 2218 (2013).
- Schenk, S., Laddaga, R. A. Improved method for electroporation of Staphylococcus aureus. FEMS Microbiol Lett. 73, 133-138 (1992).
- Grosser, M. R., Richardson, A. R. Method for Preparation and Electroporation of S. aureus and S. epidermidis. The Genetic Manipulation of Staphylococci. Methods Molecular Biology. , (2016).
- Krausz, K. L., Bose, J. L. Bacteriophage Transduction in Staphylococcus aureus: Broth-Based Method. The Genetic Manipulation of Staphylococci. Methods Molecular Biology. , (2016).
- Olson, M. E. Bacteriophage Transduction in Staphylococcus aureus. The Genetic Manipulation of Staphylococci. Methods Molecular Biology. , (2016).
- Fey, P. D., et al. A genetic resource for rapid and comprehensive phenotype screening of nonessential Staphylococcus aureus genes. mBio. 4 (1), e00537-e00612 (2013).
- Coe, K. A., et al. Multi-strain Tn-Seq reveals common daptomycin resistance determinants in Staphylococcus aureus. PLoS Pathog. 15 (11), e1007862 (2019).
- Figurski, D. H., Helinski, D. R. Replication of an origin-containing derivative of plasmid RK2 dependent on a plasmid function provided in trans. Proc Natl Acad Sci U S A. 76 (4), 1648 (1979).
- Shee, C., et al. Engineered proteins detect spontaneous DNA breakage in human and bacterial cells. eLife. 2, e01222 (2013).
- Kowalska-Krochmal, B., Dudek-Wicher, R. The minimum inhibitory concentration of antibiotics: methods, interpretation, clinical relevance. Pathogens. 10 (2), 165 (2021).
- Robinson, J. L., Jaslove, J. M., Murawski, A. M., Fazen, C. H., Brynildsen, M. P. An integrated network analysis reveals that nitric oxide reductase prevents metabolic cycling of nitric oxide by Pseudomonas aeruginosa. Metab Eng. 41, 67-81 (2017).
- Wolff, J. A., MacGregor, C. H., Eisenberg, R. C., Phibbs, P. V. Isolation and characterization of catabolite repression control mutants of Pseudomonas aeruginosa PAO. J Bacteriol. 173 (15), 4700-4706 (1991).
- Batchelder, J. I., Taylor, A. J., Mok, W. W. K. Metabolites augment oxidative stress to sensitize antibiotic-tolerant Staphylococcus aureus to fluoroquinolones. mBio. , e0271424 (2024).
- Power, A. D., Mok, W. W. K. Agar and agarose used for Staphylococcus aureus biofilm cultivation impact fluoroquinolone tolerance. J Appl Microbiol. 135 (8), lxae191 (2024).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Peng, T., et al. A BaSiC tool for background and shading correction of optical microscopy images. Nat Commun. 8, 14836 (2017).
- Ducret, A., Quardokus, E., Brun, Y. V. MicrobeJ, a tool for high throughput bacterial cell detection and quantitative analysis. Nat Microbiol. 1 (7), 16077 (2016).
- Paintdakhi, A., et al. Oufti: an integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Mol Microbiol. 99 (4), 767-777 (2016).
- Wang, P., et al. Robust growth of Escherichia coli. Curr Biol. 20 (12), 1099-1103 (2010).
- Moffitt, J. R., Lee, J. B., Cluzel, P. The single-cell chemostat: an agarose-based, microfluidic device for high-throughput, single-cell studies of bacteria and bacterial communities. Lab Chip. 12 (8), 1487-1494 (2012).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유