Method Article
Zeitraffer-Epifluoreszenzmikroskopie: Bildgebung von Pseudomonas aeruginosa und Staphylococcus aureus : Heterogene Phänotypen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
In diesem Manuskript stellen wir ein umfassendes Protokoll zur Beurteilung des Überlebens von Antibiotika für Pseudomonas aeruginosa und Staphylococcus aureus zur Verfügung, indem wir Plasmide in P. aeruginosa und S. aureus transformieren, um Reporterstämme zu erzeugen und phänotypische Varianten, wie z.B. Persistere, durch Zeitraffer-Epifluoreszenzmikroskopie sichtbar zu machen.
Zusammenfassung
Die Persistenz von Antibiotika ist ein Phänomen, bei dem eine kleine Anzahl von Bakterienzellen in einer genetisch anfälligen Population eine Antibiotikabehandlung überlebt, bei der die anderen genetisch identischen Zellen abgetötet werden. Bakterielle Persistere können sich nach Beendigung der Antibiotikabehandlung wieder vermehren und es wird allgemein angenommen, dass sie dem klinischen Behandlungsversagen zugrunde liegen. Jüngste Arbeiten, die sich die Leistungsfähigkeit der Zeitraffer-Fluoreszenzmikroskopie zunutze machen, bei der Bakterien mit fluoreszierenden Transkriptionsreportern, Translationsreportern und/oder Farbstoffen für eine Vielzahl von zellulären Merkmalen markiert werden, haben unser Verständnis von Escherichia coli-Persistern über das hinaus erweitert, was aus Antibiotika-Überlebenstests auf Bevölkerungsebene gelernt werden konnte. Solche Einzelzellansätze sind anstelle von Massenpopulationsassays unerlässlich, um die Mechanismen der Persisterbildung, der Schadensreaktion und des Überlebens abzugrenzen. Die Methoden zur Untersuchung von Persistern in anderen wichtigen pathogenen Spezies auf diesem Detaillierungsgrad sind jedoch nach wie vor begrenzt.
Diese Studie bietet einen anpassungsfähigen Ansatz für die Zeitrafferbildgebung von Pseudomonas aeruginosa (einem gramnegativen Stäbchen) und Staphylococcus aureus (einem grampositiven Kokkus) während der Antibiotikabehandlung und -genesung. Wir diskutieren molekulargenetische Ansätze, um fluoreszierende Reporter in diese Bakterien einzuführen. Mit diesen Reportern sowie Farbstoffen können wir die phänotypischen Veränderungen, morphologischen Merkmale und das Schicksal einzelner Zellen als Reaktion auf eine Antibiotikabehandlung verfolgen. Darüber hinaus sind wir in der Lage, die Phänotypen einzelner Persister zu beobachten, während sie nach der Behandlung wiederbelebt werden. Alles in allem dient diese Arbeit als Ressource für diejenigen, die daran interessiert sind, das Überleben und die Genexpression einzelner mit Antibiotika behandelter Zellen, einschließlich Persister, sowohl während als auch nach der Behandlung in klinisch wichtigen Krankheitserregern zu verfolgen.
Einleitung
Bakterielle Krankheitserreger können sich der Wirkung von Antibiotika durch zwei Hauptmechanismen entziehen: Antibiotikaresistenz, die genetische Veränderungen beinhaltet, und phänotypische Toleranz, die nicht-genetische Veränderungen beinhaltet. Antibiotikaresistenz ist ein genetisch kodiertes Phänomen, das einer bestimmten Bakterienzelle die Fähigkeit verleiht, in Gegenwart eines Antibiotikums nicht nur zu überleben, sondern sich auch zu vermehren. Phänotypische Toleranz, die antibiotikatolerante oder antibiotikapersistente Bakterien umfassen kann, tritt auf, wenn Zellen einer bakteriziden Antibiotikabehandlung standhalten, ohne die Fähigkeit zu erlangen, sich in Gegenwart einer hemmenden Konzentration des Antibiotikums zu replizieren 1,2. Was Toleranz von Persistenz unterscheidet, ist, dass sich Toleranz auf die Fähigkeit der gesamten Population bezieht, eine Behandlung zu überleben, während sich Persistenz auf eine Untergruppe einer isogenen, aber phänotypisch heterogenen Population bezieht, die eine Antibiotikabehandlung überlebt. Wenn eine klonale Kultur mit bakteriziden Antibiotika behandelt wird und die in der Kultur verbleibenden Überlebenden auf einer logarithmisch-linearen Skala gegen die Zeit aufgetragen werden, wird in der Regel eine biphasische Kurve nachgewiesen, wenn Persister vorhanden sind. Auf diesen Kurven zeigt die erste Phase, dass die Mehrheit der Bevölkerung relativ schnell getötet wird, und die zweite Phase, dass eine antibiotische persistente Fraktion langsamer oder gar nicht abgetötet wird 1,2.
Die Persistenz von Antibiotika stellt eine große Belastung für die globalen Gesundheitssysteme dar. Zum Beispiel wird angenommen, dass Staphylococcus aureus und Pseudomonas aeruginosa Persister, die im Mittelpunkt dieses Artikels stehen, Antibiotika-widerspenstige Infektionen verursachen, einschließlich rezidivierender Atemwegsinfektionen bei Patienten mit Mukoviszidose und chronischen Wundinfektionen 3,4. Daher ist die weitere Aufklärung der Persisterzellbiologie und der phänotypischen Programme von entscheidender Bedeutung. Während Fortschritte beim Verständnis der Bildung und Wiederbelebung von Persistern erzielt wurden, bestehen nach wie vor kritische Wissenslücken in Bezug auf die Koordination der metabolischen Reprogrammierung und der molekularen Ereignisse in einzelnen Zellen, die der Persistenz zugrunde liegen, 5,6,7,8.
Die effektive Untersuchung der Persistenz hat sich als technische Herausforderung erwiesen. Da Persistenz nur in einer kleinen Untergruppe einer Bakterienpopulation beobachtbar ist, können Techniken, die Bakterienpopulationen in großen Mengen beproben, oft keine relevanten biologischen Informationen erfassen 1,2,8,9,10. Da phänotypische Veränderungen, die der Persistenz zugrunde liegen, vorübergehend und nicht vererbbar sind, kann die Verfolgung des Schicksals von Persisterzellen komplex sein 1,8,9,10,11. Sobald bakterielle Persister ihr Wachstum wieder aufnehmen, können sie sich teilen und sowohl Persister als auch Nicht-Persister hervorbringen, was es unmöglich macht, reine Persisterpopulationen durch Kultivierung anzureichern. Diese Herausforderungen unterstreichen den Bedarf an Techniken, die die folgenden Kriterien erfüllen können: 1) die Fähigkeit, biologische Informationen lebender, einzelner Zellen zu erfassen, und 2) die Fähigkeit, zusammen mit Fluoreszenzfarbstoffen, Sonden, Sensoren und Reportern verwendet zu werden, die es ermöglichen, Phänotypen einzelner Zellen in heterogenen Populationen im Laufe der Zeit abzufragen.
Jüngste Fortschritte in der Einzelzelltechnologie haben einen Weg eröffnet, die bakterielle Heterogenität effektiv zu untersuchen und diese Hürden bei der Untersuchung der Persistenz zu überwinden12,13. Zu diesen Techniken gehören Fluoreszenzmikroskopie, Durchflusszytometrie/fluoreszenzaktivierte Zellsortierung, Mikrofluidik und Einzelzell-RNA-Sequenzierung12,13. In dieser Arbeit beschreiben wir Protokolle zur Aufklärung der Physiologie von Einzelzellpersistern mittels Epifluoreszenz-Zeitraffermikroskopie von transkriptionellen oder translationalen Reporterstämmen. Die Fluoreszenzmikroskopie ist eine leistungsfähige Technik, die die Kriterien für die Untersuchung von Persister-Phänotypen erfüllt, nämlich die Fähigkeit zu identifizieren, welche einzelnen Zellen in einer großen Population sich nach der Entfernung von Antibiotika vermehren und somit als Persister definiert werden können. Mit der Einführung automatisierter Kameratechnologien und Inkubationskammern ist die Erfassung lebender Bakterienzellen im gesamten Bereich der Mikrobiologie allgemein zugänglich. Entscheidend ist, dass die Zeitraffermikroskopie die Möglichkeit bietet, einzelne Zellen in Echtzeit über Stunden und sogar Tage hinweg zu visualisieren, was es ermöglicht, Bakterien vor, während und nach der Antibiotikabehandlung zu verfolgen 14,15,16. Die Erkenntnisse aus diesen Untersuchungen, die sich die Zeitraffermikroskopie zunutze machen, haben ein immenses Potenzial, Einblicke in die komplexen Mechanismen der Persisterbiologie zu gewinnen.
Protokoll
1. Erzeugung fluoreszierender Reporterstämme von S. aureus durch Transformation und Transduktion
HINWEIS: Reporterstämme beherbergen ein fluoreszierendes Protein, um die Expression eines Gens oder Proteins von Interesse anzuzeigen. Transkriptionsreporter weisen eine doppelte Kopie der nativen Promotorsequenz für ein Gen von Interesse stromaufwärts eines fluoreszierenden Proteins auf, so dass die Fluoreszenz zunimmt, wenn die Expression eines Gens von Interesse zunimmt. Translationale Reporter verknüpfen die offenen Leserahmen eines fluoreszierenden Proteins und eines Proteins von Interesse mit einem flexiblen Peptidkonnektor. Die Visualisierung von Reporterstämmen mit der Lebendzellmikroskopie kann Aufschluss darüber geben, ob ein bestimmtes Gen/Protein von Interesse mit bestimmten Zellmorphologien oder Zellschicksalen assoziiert ist (Abbildung 1, Ergänzendes Video 1 und Ergänzendes Video 2). Wählen Sie ein fluoreszierendes Protein, das für S. aureus codon-optimiert ist.Während in dieser Studie sGFP aus pCM29 verwendet wird, das von Dr. Alexander Horswill zur Verfügung gestellt wurde, enthält das Nebraska Transposon Mutant Library Genetic Toolkit Plasmide mit Codon-optimiertem sGFP, eYFP, eCFP, DsRed.T3 und eqFP650 17,18.
- Transformieren Sie Reporterplasmide in S. aureus RN4220.
HINWEIS: Der folgende Protokollabschnitt wurde aus dem Workshop "Grundlagen der Staphylokokkengenetik und des Stoffwechsels" am University of Nebraska Medical Center19,20 übernommen.- Bereiten Sie zunächst elektrokompetentes S. aureus RN4220 vor. Bereiten Sie 200 ml B2-Brühe zu, indem Sie 2 g Casaminosäuren, 5 g Hefeextrakt, 2 ml 10% K2HPO4, pH 7,5, 2 ml 50%ige Glukoselösung und 5 g NaCl kombinieren. Bringen Sie das Volumen mit sterilem Reinstwasser auf 100 ml. Kurz bei 37 °C mischen, um die Bestandteile aufzulösen, und mit einem Flaschenfilter (0,22 μm Porengröße) sterilisieren.
- S. aureus RN4220 aus einer gefrorenen Brühe, die bei -80 °C in 25 % Glycerin gelagert wurde, in 30 ml B2-Bouillon in einem 250-ml-Kolben impfen und 18 h bei 30 °C inkubieren und bei 125 U/min schütteln.
- Zwei 250-ml-Isolierkolben mit je 50 ml B2-Bouillon werden über Nacht bei 37 °C inkubiert, um das Medium für den nächsten Tag vorzuwärmen.
- Messen Sie am nächsten Tag den OD600 der Nachtkultur. Geben Sie genügend Übernachtkultur in jeden Kolben mit 50 ml B2-Brühe, um einen endgültigen OD600 von 0,25 zu erhalten.
- Inkubieren Sie bei 37 °C und schütteln Sie bei 250 U/min, bis der OD600 0,35-0,4 erreicht.
HINWEIS: Überprüfen Sie den OD600 etwa 30 Minuten nach der Subkultur, um sicherzustellen, dass die Kultur nicht über 0,35-0,4 wächst. Wenn der OD600 innerhalb von 1 h nach der Subkultivierung nicht 0,35-0,4 erreicht, brechen Sie das Verfahren ab und versuchen Sie es am nächsten Tag erneut. Die Bakterien wuchsen höchstwahrscheinlich über Nacht zu stark und waren zu tief in der stationären Phase. - Sobald die Kultur die mittlere logarithmische Phase erreicht hat, gießen Sie die Kultur aus jedem Kolben in ein steriles konisches 50-ml-Röhrchen und Pelletzellen durch Zentrifugation bei 4.000 x g für 10 Minuten.
- Dekantieren Sie den Überstand und resuspendieren Sie das Pellet in 1 mL sterilem Wasser mit Raumtemperatur (RT). Übertragen Sie die Zellen in ein Mikrozentrifugenröhrchen.
- Die Zellen werden erneut bei 21.000 x g für 30 s zentrifugiert, der Überstand entfernt und in 1 mL sterilem RT-Wasser resuspendiert. Wiederholen Sie das Zentrifugieren, Entfernen des Überstands und Resuspension in 1 ml Wasserschritt zwei weitere Male für insgesamt drei Wäschen.
- Zentrifugieren Sie die Zellen, entfernen Sie das Wasser, resuspendieren Sie die Zellen dann in 1 mL sterilem RT 10% Glycerin (in Wasser, steril gefiltert) und lassen Sie sie 15 Minuten lang bei RT.
- Pelletieren Sie die Zellen durch Zentrifugation bei 21.000 x g für 30 s, entfernen Sie den Überstand und resuspendieren Sie die Zellen in einem weiteren 1 ml sterilem RT 10%igem Glycerin. Diese Zellen sind nun elektrokompetent.
- Aliquotieren Sie 70 μl elektrokompetenter Zellen in Mikrozentrifugenröhrchen mit Schnappverschluss und lagern Sie sie bei -80 °C für die zukünftige Verwendung.
- Um das elektrokompetente S. aureus RN4220 umzuwandeln, taut man ein Aliquot von Zellen auf Eis auf. Geben Sie 1 μg Plasmid-DNA zu den Zellen, schnippen Sie die Probe zum Mischen und inkubieren Sie sie 5 Minuten lang auf Eis. Dann elektroporieren Sie die Zellen und lassen sie 1-2 h lang bei 37 μC in 390 μl B2-Medien zurückgewinnen, schütteln bei 225 U/min und plattieren Transformanten auf selektivem tryptischem Soja-Agar.
HINWEIS: Die meisten der in dieser Studie verwendeten Shuttle-Vektoren weisen eine Ampicillin-Resistenzkassette zur Selektion in E. coli (100 μg/ml) und eine Chloramphenicol- (10 μg/ml) oder Erythromycin- (Erm, 10 μg/ml) Widerstandskassette zur Selektion in S. aureus auf. Viele S. aureus-Stämme , die routinemäßig im Labor verwendet werden, sind Erm-resistent. Stellen Sie sicher, dass ein interessierender Stamm mit dem selektiven Antibiotikum kompatibel ist, bevor Sie einen bestimmten Shuttle-Vektor verwenden. Es ist wichtig, dass das Volumen der DNA, die den Zellen zugesetzt wird, minimiert wird (d. h. <5 μl), um Lichtbögen zu vermeiden, die auftreten, wenn die Probe während der Elektroporation zu leitfähig ist. Die Reinigung hoher Konzentrationen (≥300 ng/μl) der Plasmid-DNA aus E. coli trägt dazu bei, das Volumen gering zu halten.
- Vermehren Sie Bakteriophagen auf transformierten S. aureus RN4220.
HINWEIS: Der folgende Protokollabschnitt wurde von Methoden von Krausz und Bose übernommen21und Olson22. Zwei der am häufigsten verwendeten Bakteriophagen für S. aureus Transduktion sind φ11 und 80α. Das Verfahren zur Ernte dieser beiden ist das gleiche, nur dass wir φ11 erhalten von S. aureus RN451 und 80α ab S. aureus RN10359. φ11 wurde unter anderem erfolgreich mit den gängigen Stämmen JE2 und HG003 eingesetzt und wird in diesem Protokoll verwendet23,24. Diese Phagen transduzieren klinische Isolate mit unterschiedlichen Phagenresistenzen jedoch möglicherweise nicht effizient. Siehe die Veröffentlichungen von Krausz und Bose21 und Olson22 für ein Verfahren zur Herstellung von φ11 und zur Bestimmung des Phagentiters.- Um das Reporterplasmid zu transduzieren, bereiten Sie sechs tryptische Soja-Agar (TSA)-Platten mit 5 mM CaCl2 und der richtigen Konzentration des selektiven Antibiotikums vor. Streak S. aureus RN4220 transformierte sich mit dem Reporterplasmid auf eine TSA-Platte, die das selektive Antibiotikum und kein CaCl2 enthielt, und inkubierte über Nacht.
- Schalten Sie am nächsten Tag einen Inkubator oder ein Wasserbad auf 56 °C ein. Etikettieren Sie sechs 5-ml-Schnappverschlussröhrchen 1 x 104 bis 1 x 109 und aliquotieren Sie 40 μl 500 mM CaCl2 in jedes Schnappverschlussröhrchen.
- Schmelzen Sie weiches TSA (0,5 % Agar) in der Mikrowelle und lösen Sie die Kappe, um einen Druckaufbau zu verhindern und gleichzeitig die Sterilität zu erhalten. Aliquotieren Sie dann 4 mL in jedes der Schnappverschlussröhrchen. Drehen Sie die Röhrchen zum Mischen um und legen Sie sie dann in den 56 °C heißen Inkubator, um zu verhindern, dass sich der weiche Agar verfestigt.
- Verdünnen Sie die Phagenbrühe auf 1 x 1010 PFU/ml in tryptischer Sojabouillon (TSB) + 5 mM CaCl2. 100 μl 1 x 1010 PFU/ml Phage in ein Mikrozentrifugenröhrchen mit 900 μl TSB + 5 mM CaCl2 geben, um eine Endkonzentration von 1 x109 PFU/ml zu erreichen. Anschließend in zusätzliche Mikrozentrifugenröhrchen mit 900 μl TSB + 5 mM CaCl2 nacheinander verdünnen, bis zu einer Konzentration von 1 x 104 PFU/ml. Jede der sechs TSA + 5 mM CaCl2 + selektiven Antibiotika-Agarplatten wird mit einer seriellen Verdünnung von 1 x104 bis 1 x 109 PFU/ml gekennzeichnet.
- Resuspendieren Sie das über Nacht verabreichte S. aureus RN4220, das das Plasmid in 1 mL TSB + 5 mM CaCl2 enthält. Nehmen Sie das 109 Schnappverschlussröhrchen aus dem Inkubator und fügen Sie schnell 10 μl resuspendiertes RN4220 mit dem Plasmid und 100 μl 1 x 109 PFU/ml-Phagenverdünnung hinzu, drehen Sie es um, um es zu mischen, und gießen Sie den Inhalt auf die entsprechende TSA + 5 mM CaCl2 + selektive antibiotische Agarplatte.
- Führen Sie dieses Verfahren für die verbleibenden Phagenverdünnungen und ihre jeweiligen Platten durch. Lassen Sie den weichen Agar auf den Platten fest werden und inkubieren Sie ihn dann mit dem weichen Agar nach oben über Nacht bei 37 °C.
- Stellen Sie am nächsten Tag sicher, dass Plaques (Bereiche mit Abstand auf dem Bakterienrasen) auf den Platten sichtbar sind (Abbildung 2A). Plaques stellen Lysebereiche dar, in denen Phagen RN4220 erfolgreich infiziert haben, und einige Phagen haben das gewünschte Plasmid aufgenommen. Wählen Sie die Platte mit dem am wenigsten verdünnten Phagenbestand aus, der eine nahezu konfluente Lyse aufweist (Beispiel: 1 x 109 PFU/ml-Phagen) sowie die Platten, die die beiden Verdünnungen darunter erhalten haben (in diesem Fall 1 x 108 PFU/ml und 1 x 107 PFU/ml, die beide eine nahezu konfluente Lyse aufweisen sollten).
- Fügen Sie 3 mL TSB + 5 mM CaCl2 zur am wenigsten verdünnten dieser Platten hinzu (1 x 109 PFU/ml, für dieses Beispiel). Verwenden Sie einen sterilen L-förmigen Zellspreizer, um die Schicht des weichen Agars vorsichtig vom normalen Agar abzukratzen (Abbildung 2B). Verwenden Sie den Zellspreader, um den Agar so weit wie möglich zu zerkleinern, um die Freisetzung von Phagen aus dem weichen Agar zu erleichtern.
- Sobald der weiche Agar auf der ersten Platte gründlich zerkleinert wurde, gießen Sie den weichen Agar und die Brüheaufschlämmung vorsichtig auf die nächste Platte (in diesem Fall 1 x 108 PFU/ml). Kratzen und zerkleinern Sie das weiche Agar auf dieser Platte, gießen Sie dann die resultierende Aufschlämmung auf die endgültige Platte und wiederholen Sie den Vorgang.
- Nachdem Sie die weiche Agar-Aufschlämmung aus allen drei Platten zubereitet haben, gießen Sie die Aufschlämmung vorsichtig in ein konisches 50-ml-Röhrchen. Pipettieren Sie vorsichtig, um das weiche Agar weiter zu zerkleinern, aber vermeiden Sie die Bildung von Blasen. Nicht vortexen, da dies den Phagen schädigen und den Titer verringern könnte.
- Zentrifugieren Sie die weiche Agar-Aufschlämmung bei 4.000 x g für 10 min bei RT. Gießen Sie den Überstand vorsichtig in eine Spritze, die mit einem 0,45 μm Filter verbunden ist.
HINWEIS: Es ist wichtig, vorsichtig zu gießen und zu vermeiden, dass Agarstücke in den Filter gelangen. Diese Stücke verstopfen den Filter und verhindern, dass der verbleibende Überstand gefiltert wird, wodurch der Titer verringert wird. - Bestimmen Sie den Titer des resultierenden Phagenlysats mit Wildtyp RN422021.
- Führen Sie Reporterplasmide durch Transduktion in S. aureus-Stämme von Interesse ein.
- Bereiten Sie acht TSA-Platten mit 500 μg/ml Natriumcitrat und der entsprechenden Konzentration des selektiven Antibiotikums vor. Den gewünschten S. aureus-Stamm dicht auf einfaches TSA streichen und über Nacht bei 37 °C inkubieren.
- Am nächsten Tag tupfen Sie die Platte ab und suspendieren Sie den Empfängerstamm in 1 ml TSB + 5 mM CaCl2.
- Verdünnen Sie den Phagenstamm mit dem gewünschten Reporterplasmid auf 1 x1010 PFU/ml.
- Mischen Sie 1,5 ml TSB + 5 mM CaCl2, 0,5 ml resuspendierten Empfängerstamm und 0,5 ml verdünnte Phagenbrühe in einem konischen 50-ml-Röhrchen. Inkubieren bei 37 °C, Schütteln bei 225 U/min, für 20 min.
HINWEIS: Längere Inkubationszeiten werden nicht empfohlen, da sie wahrscheinlich die Transduktionseffizienz verringern, wenn der Phagen beginnt, Empfängerzellen zu lysieren. - Unmittelbar danach 1 mL steril filtriertes eiskaltes 0,02 M Natriumcitrat (in Wasser) in das konische Röhrchen geben und bei 4.000 x g für 10 min bei 4 °C zentrifugieren. Dann resuspendieren Sie das Pellet in 1 ml eiskaltem 0,02 M Natriumcitrat.
- Verteilen Sie 100 μl Aliquots auf jede der sechs TSA-Platten mit 500 μg/ml Natriumcitrat und der entsprechenden Konzentration des selektiven Antibiotikums zur Auswahl der Transduktivitäten. Über Nacht bei 37 °C inkubieren.
- Am nächsten Tag streichen Sie einzelne Kolonien von den Transduktionsplatten auf die verbleibenden zwei TSA-Platten, die 500 μg/ml Natriumcitrat und die entsprechende Konzentration des selektiven Antibiotikums enthalten. Über Nacht bei 37 °C inkubieren.
HINWEIS: Dieser Passage-Schritt auf zusätzlichen Natriumcitratplatten hilft, den Titer von Phagen um die transduzierten Zellen zu reduzieren und so eine Lysezu verhindern 22. - Entnehmen Sie einzelne Kolonien aus der Streifenplatte, züchten Sie die Zellen in TSB, das das selektive Antibiotikum enthält, in exponentielle Phase und lagern Sie sie in 25 % Glycerin bei -80 °C für die zukünftige Verwendung.
2. Erzeugung fluoreszierender Reporterstämme von P. aeruginosa durch Konjugation
HINWEIS: Das Verschieben eines Reporterplasmids in P. aeruginosa von einem E. coli-Klonierungsstamm kann durch triparentale Paarungerfolgen 25. Ein Spenderstamm von E. coli , der das gewünschte Plasmid trägt (das ein oriT für den ehelichen Transfer enthalten sollte), E. coli HB101 + pRK2013 - ein Helferstamm zur Erleichterung der Konjugation (verwenden Sie 50 μg/ml Kanamycin zur Aufrechterhaltung des pRK2013-Plasmids) und der P. aeruginosa-Empfängerstamm , der das Plasmid erhalten wird, sind erforderlich. In diesem Beispiel hat das Plasmid von Interesse einen Tetracyclin (Tet)-Resistenzmarker, so dass jede E. coli-Kultur mit dem Plasmid 10 μg/ml Tet und P. aeruginosa mit dem Plasmid 75 μg/ml Tet für die Selektion26 benötigt.
- Impfen Sie Bakterien aus gefrorenen Stoffen, die bei -80 °C in 25 % Glycerin gelagert wurden, in Lysogenbrühe (LB) mit dem geeigneten Antibiotikum für das Wachstum über Nacht und schütteln Sie sie bei 225 U/min bei 37 °C.
- Verteilen Sie am nächsten Tag 200 μl jeder flüssigen Übernachtkultur mit dem entsprechenden selektiven Antibiotikum auf die LB-Agarplatte. Je nachdem, wie viele Transformationspaarungen durchgeführt werden sollen, verteilen Sie weitere Platten für die Helfer- und Donorstämme. Empfohlen wird eine Platte Helfer/Spender pro Transformation. Inkubieren Sie die Platten über Nacht bei 37 °C, um dichte Rasenflächen jeder Sorte zu erhalten.
- Sammeln Sie am nächsten Tag den Rasen des Wachstums, indem Sie jede Plattenoberfläche mit einem sterilen Wattestäbchen abtupfen und an der Innenseite eines Mikrozentrifugenröhrchens entlangwischen. Jedes Röhrchen kurz zentrifugieren (5 s pulsieren), um die Zellen am Boden des Röhrchens zu sammeln. Resuspendieren Sie jedes Zellpellet in 200 μl LB.
HINWEIS: Die Resuspensionen sollten sehr dick, undurchsichtig und frei von Klumpen aussehen. - Kombinieren Sie in neuen Mikrozentrifugenröhrchen für jede Paarung 5 μl des Empfängerstamms P. aeruginosa , 160 μl des Spenderstamms E. coli mit dem gewünschten Plasmid und 160 μl des Helferstamms E . coli HB101 + pRK2013. Mischen Sie vorsichtig, pipettieren Sie auf und ab und geben Sie dann 50 μl der Mischung auf eine vorgetrocknete LB-Agarplatte. Geben Sie außerdem 20 μl jedes einzelnen Stammes zur Kontrolle auf den LB-Agar. Lassen Sie die Flecken vollständig in der Platte trocknen, dann bei 37 °C 3-6 h inkubieren.
HINWEIS: Das Vortrocknen des LB-Agars verhindert, dass sich der 50-μl-Spot über die Oberfläche ausbreitet und zwingt die Zellen somit, in einem kleineren Bereich zu interagieren. - Streichen Sie nach der Inkubation vorsichtig mit einer Pipettenspitze über die Gegenstelle, um einige Zellen in ein neues Mikrozentrifugenröhrchen zu übertragen. Kurz zentrifugieren, um die Zellen am Boden des Röhrchens zu sammeln. Resuspendieren Sie in 200 μl LB und plattieren Sie die gesamte Resuspension auf eine LB + Irgasan (Igr; 25 μg/ml) + Tet (75 μg/ml) Agarplatte.
HINWEIS: P. aeruginosa ist intrinsisch resistent gegen Igrasan, und das Plasmid von Interesse enthält den Tet-Resistenzmarker, so dass nur erfolgreich transformiertes P. aeruginosa in der Lage sein sollte, auf den LB + Irgasan + Tet-Platten zu wachsen. Abhängig von der Umwandlungseffizienz kann das Resuspendieren und Plattieren der gesamten Paarungsstelle am nächsten Tag zu einem Rasen mit Transformatmitteln führen. Um einzelne Kolonien zu erhalten, resuspendieren Sie entweder (i) den gesamten Gegenwert in einem größeren Volumen LB und plattieren Sie dann 200 μl davon oder (ii) übertragen Sie einen kleinen Teil des Gegenpunkts in ein Mikrozentrifugenröhrchen und resuspendieren Sie ihn dann in 200 μl und Platte. - Wischen Sie mit den Kontrollspots jedes einzelnen Stammes vorsichtig über einen Teil des dichten Zellspots auf eine Pipettenspitze, geben Sie ihn in ein neues Mikrozentrifugenröhrchen, resuspendieren Sie 200 μl LB und spotten Sie dann 50 μl auf eine LB + Irgasan + Tet-Agarplatte (mehrere Kontrollpunkte können auf derselben Agarplatte platziert werden). Lassen Sie die Flecken vollständig eintrocknen. Alle Platten über Nacht bei 37 °C 16-20 h inkubieren.
HINWEIS: Bei den Kontrollspots besteht das Ziel darin, zu überprüfen, ob die Antibiotikaplatte selektiv für P. aeruginosa-Transformanten ist. Wenn die Kontrollflecken zu konzentriert sind, wenn sie auf den LB + Irgasan + Tet-Agarplatten entdeckt werden, dann wird es schwierig sein, am nächsten Tag festzustellen, ob die Selektion funktioniert hat, da es einen Halo aus abgestorbenen Zellen gibt, der mit Wachstum verwechselt werden kann. - Am nächsten Tag bestätigen Sie die Antibiotikaselektivität der LB + Irgasan + Tet-Platten, indem Sie das fehlende Zellwachstum für die einzelnen Stammkontrollpunkte feststellen.
HINWEIS: Kolonien, die auf der triparentalen Paarungsplatte LB + Igrasan + Tet erscheinen, sind P. aeruginosa-Transformanten , die das interessierende Plasmid tragen. - Optional, um Kolonien auf LB + Irgasan + Tet-Agar zu pflücken und über Nacht zu züchten, um überschüssige tote E. coli aus der Paarungsmischung zu entfernen.
- Einzelne Kolonien auswählen, 3-6 h in flüssigem LB + Tet (75 μg/mL) züchten, bis sie sichtbar trüb sind, dann gefrorene Bestände in 25% Glycerin lagern (500 μL Zellkultur + 500 μL 50% Glycerin in Wasser). Stellen Sie sicher, dass die Klone erfolgreich transformiert wurden, indem Sie eine Kolonie-PCR durchführen, gefolgt von einer Sanger- oder Ganzplasmid-Sequenzierung.
3. Bestimmung der Antibiotikadosis für Persister-Assays
HINWEIS: Um eine Dosis eines bestimmten Antibiotikums auszuwählen, mit der die Bakterienpopulation für Persister-Experimente behandelt werden soll, messen Sie zunächst die minimale Hemmkonzentration (MIC) des Antibiotikums gegen den interessierenden Bakterienstamm. Dies kann entweder mit der Bouillon-Mikrodilutionsmethode erreicht werden – ein Ansatz, den das Clinical and Laboratory Standards Institute (CLSI) befürwortet – oder mit dem Epsilometer-Test (E-Test), der unter Verwendung von Teststreifen mit einer Reihe von Antibiotikadosen durchgeführt wird27. Sobald die MHK bestimmt ist, wählen Sie mindestens fünf Konzentrationen des Antibiotikums, die zwischen dem 1- und 100-fachen MHK für die Zellbehandlung liegen.
- Impfen Sie Bakterien aus gefrorenen Stoffen, die bei -80 °C in 25 % Glycerin gelagert wurden, in 2 ml kationenadjustiertes MHB (CA-MHB) oder ein anderes nährstoffreiches Medium. Die Zellen werden ca. 4 h lang bei 37 °C gezüchtet und bei 250 U/min geschüttelt, bevor 125 μl der Kultur in einem 250 ml-Erlenmeyerkolben auf 25 ml frisches, chemisch definiertes Medium überführt werden. Züchten Sie die Bakterien 16 h lang (in die stationäre Phase) bei 37 °C und schütteln Sie sie bei 250 U/min.
HINWEIS: Basalsalzmedien (BSM) mit Succinat als einzige Kohlenstoffquelle werden typischerweise für P. aeruginosa-Experimente verwendet, und reichhaltige chemisch definierte Medien werden typischerweise für S. aureus-Experimente verwendet 28,29,30,31. - Bereiten Sie am nächsten Morgen 100x Antibiotika-Brühen (in den entsprechenden Lösungsmitteln) für den gewünschten Bereich der supra-MHK-Konzentrationen vor. Geben Sie 10 μl jeder Verdünnung in einzelne Reagenzgläser.
- Messen Sie den OD600 der Übernachtkultur, um die Trübung in der stationären Phase zu bestätigen. Verdünnen Sie seriell 10 μl Kultur und plattieren Sie die Verdünnungen auf Nähragarplatten – wie LB-Agar, CA-MHB-Agar oder TSA –, um die koloniebildenden Einheiten (KBE) vor der Antibiotikabehandlung zu bestimmen.
- Für die Antibiotikabehandlung geben Sie 1 ml Aliquote der Kultur in die Reagenzgläser mit 10 μl 100-facher Antibiotikakonzentration. Inkubieren Sie die Proben bei 37 °C und schütteln Sie sie bei 250 U/min für eine Dauer, die ausreicht, um Nicht-Persister in der Population abzutöten, wobei die Persister die einzigen koloniebildenden Zellen sind.
HINWEIS: Die Dauer der Behandlung kann je nach Experiment und getestetem Bakterienstamm variieren. Typischerweise wird S. aureus für 5 h oder 7 h und P. aeruginosa für 7 h oder 24 h behandelt. Im Allgemeinen kann jeder Zeitpunkt in der zweiten Phase des zeitabhängigen Überlebensassays verwendet werden, da davon ausgegangen wird, dass die in der zweiten Phase verbleibenden Zellen Persister sind. - Nach der Antibiotikabehandlung werden 100 μl Zellen aus den Reagenzgläsern in Mikrozentrifugenröhrchen mit 900 μl steriler phosphatgepufferter Kochsalzlösung (PBS) überführt. Pelletieren Sie die Zellen durch Zentrifugation bei RT bei 21.000 x g für 3 min. Entfernen Sie 900 μl Überstand und resuspendieren Sie das Pellet in 900 μl sterilem PBS. Wiederholen Sie den Waschschritt mindestens noch einmal, um die Antibiotikareste auf ein Niveau unterhalb der MHK zu reduzieren.
- Die Kultur wird nacheinander 10-fach (sechsmal) verdünnt und 10 μl jeder Verdünnung auf Nähragarplatten aufgebracht. Inkubieren Sie die Platten über Nacht bei 37 °C.
HINWEIS: Verwenden Sie unbehandelte 96-Well-Platten mit rundem Boden für die serielle Verdünnung und Plattenzellen auf quadratischen Agarplatten mit einer Mehrkanalpipette. - Zählen Sie am nächsten Tag die Kolonien bei jeder Antibiotikakonzentration. Plotten Sie die Überlebensfraktion (KBE am Ende der Behandlung/KBE vor der Behandlung) gegen die Konzentration auf einer logarithmisch-linearen Skala. Um eine Wirkstoffkonzentration für zukünftige Persister-Assays zu wählen, wählen Sie eine Konzentration in der zweiten Phase der biphasischen Kurve (Abbildung 3).
4. Bildgebung von Zellen während der Antibiotikabehandlung oder -genesung
- Bereiten Sie Agarose-Pads und Zellproben für die Zeitraffer-Bildgebung vor
HINWEIS: Das folgende Probenvorbereitungsprotokoll wurde als benutzerfreundliche und kostengünstige Alternative zu herkömmlichen Agarose-"Sandwich"-Methoden entwickelt14,16. Durch die Verwendung einer austauschbaren Deckglasschale wird vermieden, dass unordentliches Fett oder Nagellack zur Versiegelung der Probe erforderlich ist, was die Belüftung der Probe weiter einschränken könnte. Die Agarose wird in die austauschbare Deckglasschale eingelegt, während das Deckglas bereits an Ort und Stelle ist, wodurch im Vergleich zu freihändigen Agarose-Pad-Präparaten zuverlässig planare Agarose-Oberflächen entstehen. Insgesamt hat diese Methode die Bildgebung von Proben mit verbesserter Fokussierung über das gesamte Sichtfeld und für längere Zeiträume aufgrund der stabilen Befeuchtung ermöglicht. Die hier beschriebene Methode dient der Visualisierung von Bakterien während einer Antibiotikabehandlung (Abbildung 4, Ergänzendes Video 3und Ergänzendes Video 4). Um Persister zu visualisieren, wie sie nach einer Antibiotikabehandlung wiederbelebt und wieder erwachen (Abbildung 5, Ergänzendes Video 5und Ergänzendes Video 6), bereiten Sie die Agarose-Pads mit frischen Nährmedien anstelle von verbrauchten Medien vor und fügen Sie den Pads keine Antibiotika hinzu (außer denen, die zur Plasmiderhaltung/Antibiotikaauswahl hinzugefügt werden).- Legen Sie ein steriles 30 mm Deckglas (#1,5 Dicke) in den Boden des Edelstahlbodens einer austauschbaren Deckglasschale ("Kammer"). Schrauben Sie den Polycarbonat-Einsatz vorsichtig in den Boden ein, so dass das 30-mm-Deckglas den Boden der Kammer bildet, und dichten Sie ihn durch Zusammendrücken des angebrachten Silikon-O-Rings ab. Wiederholen Sie diesen Schritt, um eine doppelte Kammer vorzubereiten.
HINWEIS: Es wird empfohlen, für jeden Versuch Duplikate anzufertigen, falls die Vorbereitung einer der Kammern suboptimal ist. Eine speziell angefertigte 3D-gedruckte Trennwand wird auf dem 30 mm Deckglas platziert, um eine Kreuzkontamination von beweglichen Zellen wie P. aeruginosa zu verhindern und Orientierungspunkte für Probenstandorte zu setzen. Die STL-Datei für den 3D-Druck ist in der Zusatzdatei 1 verfügbar. - Bereiten Sie 1,5 % Agarose in einem konischen 50-ml-Röhrchen mit dem Medium Ihrer Wahl vor. Zum Vermischen vorsichtig schwenken.
HINWEIS: Für die Bildgebung von Zellen in stationärer Phase während der Behandlung wird zellfreies verbrauchtes Medium aus einer stationären Übernachtkultur als Basismedium verwendet. Typischerweise wird Agarosemedium als 0,105 g Agarose in 7 ml Medium zur Vorbereitung von zwei Kammern hergestellt; Jede Kammer benötigt 2 ml Agarosemedium, und der Überschuss ist hilfreich, um Luftblasen beim Pipettieren zu vermeiden. - Legen Sie den konischen 50-ml-Behälter in ein Becherglas oder einen mikrowellengeeigneten Halter (stellen Sie sicher, dass die Kappe locker ist). Mikrowelle auf hoher Stufe stellen und alle 3-4 s anhalten, um zu schwenken und zu mischen. Pause machen und oft mischen, damit die Agarosemischung nicht übersprudelt.
- Überprüfen Sie nach ~1 min Gesamtaufheizzeit, ob sich in der Mischung sichtbare Agarose befindet, die nicht geschmolzen ist. Wenn die Mischung gleichmäßig klar ist, lassen Sie das Agarosemedium kurz auf ~60 °C abkühlen, bevor Sie zusätzliche Medikamente oder Farbstoffe hinzufügen. Vorsichtig schwenken, um sich zu vermischen und die Bildung von Luftblasen zu vermeiden.
HINWEIS: Propidiumiodid (16 μM für P. aeruginosa oder 1,6 μM für S. aureus) kann hinzugefügt werden, um zu visualisieren, wann Zellen ihre Lebensfähigkeit verlieren.
- Überprüfen Sie nach ~1 min Gesamtaufheizzeit, ob sich in der Mischung sichtbare Agarose befindet, die nicht geschmolzen ist. Wenn die Mischung gleichmäßig klar ist, lassen Sie das Agarosemedium kurz auf ~60 °C abkühlen, bevor Sie zusätzliche Medikamente oder Farbstoffe hinzufügen. Vorsichtig schwenken, um sich zu vermischen und die Bildung von Luftblasen zu vermeiden.
- Um ein Verfestigen des Agarosemediums zu verhindern, pipettieren Sie 2 mL des Agarosemediums in die Kammer mit dem 30-mm-Deckglas. Legen Sie vorsichtig ein steriles 25 mm Deckglas (#1,5 Dicke) über das Agarosemedium in der oberen Kammeröffnung; Dies hilft, eine Austrocknung des Agarose-Pads zu verhindern und sorgt für eine flache Oberseite der Agarose für eine optimale Phasenkontrastbildgebung. Lassen Sie das Pad 1-2 h lang festigen.
- Nachdem das Pad erstarrt ist, bereiten Sie die Zellen für die Abbildung vor, indem Sie sie auf eine entsprechend spärliche Dichte in PBS verdünnen, um einzelne Zellen sichtbar zu machen. Hier werden die Zellen auf OD600 0,01-0,05 verdünnt.
- Verwenden Sie auf dem 25-mm-Deckglas einen Permanentmarker mit feiner Spitze, um die Positionen/Identitäten jeder Probe zu markieren, die auf dem Pad ausgesät wurde. Drehen Sie die Kammer so um, dass der Basisring aus Edelstahl nach oben zeigt, und halten Sie den Polycarbonat-Einsatz vorsichtig darunter, während Sie den Boden ausfädeln. Legen Sie den Edelstahlsockel zur Seite. Schieben Sie das 30 mm Deckglas vorsichtig vom Pad ab und entsorgen Sie es.
HINWEIS: Entfernen Sie das 30 mm Deckglas, indem Sie es horizontal schieben, und achten Sie darauf, die Agarose-Oberfläche nicht einzudrücken. Vermeiden Sie auch Arbeiten über die freiliegende Agarose-Oberfläche: Staub, der darauf fällt, beeinträchtigt die Bildqualität und kann das Pad möglicherweise verunreinigen. - Anhand der Markierungen auf dem 25-mm-Deckglas 5 μl der verdünnten Zellen an den entsprechenden Stellen auf dem Agarose-Pad platzieren. Fügen Sie drei 5-μL-Spots hinzu (passen Sie dies nach Bedarf an, z. B. 4 x 4 μL-Spots usw.). Sobald die Flecken vollständig eingetrocknet sind, legen Sie leicht ein neues, steriles 30 mm Deckglas mittig über das Pad.
- Halten Sie den Polycarbonat-Einsatz mit einer Hand fest und fädeln Sie mit der anderen den Edelstahlsockel langsam wieder über das neue Deckglas. Das Einfädeln bis zum Fingerfest reicht aus, um den Silikon-O-Ring zusammenzudrücken und die Kammer abzudichten.
HINWEIS: Achten Sie darauf, dass Sie es nicht zu fest anziehen - wenn das 30-mm-Deckglas gegen die Probenoberfläche durch den Edelstahlboden zusammengedrückt und wiederholt verdreht wird, können sich die Küvetten in einem radialen Muster ausbreiten, was zu einer Kreuzkontamination der Probe führen kann. - Sobald die Kammer verschlossen ist, beobachten Sie, ob die Agarose die Oberfläche des 30-mm-Deckglases berührt; An dieser Stelle besteht in der Regel ein gewisser, aber nicht vollständiger Oberflächenkontakt. Drücken Sie mit dem stumpfen Ende einer Pinzette oder eines ähnlichen Instruments vorsichtig gegen das 25 mm Deckglas, bis die Agarose das 30 mm Deckglas gleichmäßig über die gesamte Oberfläche berührt.
ACHTUNG: Dieser Schritt kann dazu führen, dass das 25-mm-Deckglas bricht, wenn übermäßige Kraft angewendet wird. - Sobald die Agarose gut gegen das 30 mm Deckglas gedrückt ist, so dass keine großen Luftblasen zurückbleiben, ist das Pad bereit für die Abbildung.
- Legen Sie ein steriles 30 mm Deckglas (#1,5 Dicke) in den Boden des Edelstahlbodens einer austauschbaren Deckglasschale ("Kammer"). Schrauben Sie den Polycarbonat-Einsatz vorsichtig in den Boden ein, so dass das 30-mm-Deckglas den Boden der Kammer bildet, und dichten Sie ihn durch Zusammendrücken des angebrachten Silikon-O-Rings ab. Wiederholen Sie diesen Schritt, um eine doppelte Kammer vorzubereiten.
- Richten Sie das Mikroskop und das Bild ein.
- Bereiten Sie die Umgebungskontrollen des Mikroskops und der Bildgebungskammer vor. Stellen Sie die Temperaturen des Stage-Top-Inkubators und des Großkammer-Inkubators auf 37 °C ein und schalten Sie den Kammerbefeuchter ein. Legen Sie eines der Agarose-Pad-Präparate in den Inkubator auf der Bühne und schließen Sie die Inkubationskammer, damit sich die Probe äquilibrieren kann. Der Befeuchtungsdeckel des Inkubators auf der Bühne wird an dieser Stelle offen gelassen, um Kondensation auf dem 25-mm-Deckglas der Kammer zu vermeiden.
HINWEIS: Das Schrumpfen/Ausdehnen des Pads aufgrund von Temperaturänderungen kann zu einer Drift zwischen den Zeitpunkten führen, die über den Scanbereich des Autofokus-Algorithmus hinausgehen. Es ist wichtig, das Agarose-Pad vor der Bildgebung auf eine stabile Temperatur auszugleichen, was in der Regel mindestens 15 Minuten dauert. Eine ausreichende Erwärmung verhindert auch die Bildung von Kondenswasser auf dem oberen Deckglas, wenn der Befeuchtungsdeckel aufgesetzt wird. Die Hardwarekomponenten des Bildgebungssystems waren (Table of Materials): Inkubationskammer für lebende Zellen, inverses Mikroskop mit einem Plan-Apochromat 63x/1.40 Oil Ph3 M27 Objektiv, Spectra 7 LED-Light-Engine und sCMOS-Kamera (6,5 μm Pixelgröße). Die Steuerung der motorisierten Komponenten des Mikroskops und die Bildaufnahme erfolgt über eine Mikroskopie-Analysesoftware. - Bereiten Sie die Software für die Bildaufnahme vor. Stellen Sie den integrierten Autofokus-Algorithmus von MetaMorph (Mikroskopie-Analysesoftware) so ein, dass der Phasenkanal während der mehrdimensionalen Erfassung verwendet wird.
- Legen Sie auf der Registerkarte "Tisch" mehrere Tischpositionen für jede Probe fest, wobei Sie auf Sichtfelder abzielen, in denen die Zelldichte gleichmäßig verteilt ist. Legen Sie auf der Registerkarte Zeitraffer die gewünschte Dauer und Häufigkeit (Zeitintervall) der aufzunehmenden Bilder fest. Legen Sie auf den Registerkarten Wellenlängen die gewünschten Kanäle für die Erfassung fest und passen Sie die Belichtungszeiten für die Signalintensitäten der Proben an.
HINWEIS: Bilder von jeder Position werden alle 10 Minuten für bis zu 24 Stunden aufgenommen. Für die Fluoreszenzbildgebung der in diesem Artikel erwähnten Farbstoffe/Fluorophore wurden die folgenden Light-Engine-Anregungseinstellungen verwendet: Propidiumiodid (Cy3; 555/15 nm) und GFP (GFP/FITC; 470/24 nm). Die typische Expositionszeit bei Fluoreszenzanregung beträgt 100 ms. Zum Einsatz kommen das Filterset 15 (Beamsplitter FT580, Emission LP590) für Propidiumiodid und das Filterset 44 (Beamsplitter FT500, Emission BP 530/50) für GFP.
- Legen Sie auf der Registerkarte "Tisch" mehrere Tischpositionen für jede Probe fest, wobei Sie auf Sichtfelder abzielen, in denen die Zelldichte gleichmäßig verteilt ist. Legen Sie auf der Registerkarte Zeitraffer die gewünschte Dauer und Häufigkeit (Zeitintervall) der aufzunehmenden Bilder fest. Legen Sie auf den Registerkarten Wellenlängen die gewünschten Kanäle für die Erfassung fest und passen Sie die Belichtungszeiten für die Signalintensitäten der Proben an.
- Nachdem sich das Agarose-Pad in der Kammer erwärmt hat, setzen Sie den Befeuchtungsdeckel in den Inkubator auf der Bühne ein. Dadurch wird verhindert, dass das Pad während der Bildgebung austrocknet und sich verschiebt.
- Wechseln Sie bei aufgesetztem Befeuchtungsdeckel von Phasen- auf differentiellen Interferenzkontrast (DIC) und stellen Sie den Kondensor und die Aperturblende des Mikroskops für eine korrekte Köhler-Beleuchtung ein.
- Schalten Sie nach dem Einstellen wieder auf Phase um. Navigieren Sie zu den einzelnen Tischpositionen, passen Sie den Fokus an und setzen Sie die Tischposition auf die neue Fokusebene zurück. Die Einstellungen sind jetzt vorhanden, um mit dem Imaging zu beginnen.
HINWEIS: Es ist wichtig, jede Position der Stufe vor Beginn der Aufnahme erneut zu überprüfen, um sicherzustellen, dass sich das Pad nicht verschoben hat. Wenn die Zellen unscharf sind, wenn sie an einer bestimmten Bühnenposition erneut aufgerufen werden, fokussieren Sie erneut auf sie und überschreiben Sie die vorherige Phasenposition. Die Einstellung des Autofokus-Algorithmus verwendet die fokale Z-Position ab dem letzten Zeitpunkt ± 3 μm als Suchbereich, wenn sich das Pad also mehr als 3 μm entfernt, kann der Algorithmus nicht richtig fokussieren. - Starten Sie die mehrdimensionale Erfassung, indem Sie auf die Schaltfläche Erfassen klicken.
- Nachdem das Experiment abgeschlossen ist, kompilieren Sie die einzelnen Bilder aus jedem Kanal in einem Stapel, um sie als Video oder für andere Analysen anzuzeigen. Verwenden Sie dazu entweder MetaMorph oder ImageJ.
- Um Bilder in MetaMorph (bevorzugt) zu kompilieren, wählen Sie in der App "Multidimensionale Daten überprüfen " die gewünschten Kanäle/Wellenlängen aus und wählen Sie alle Zeitpunkte für eine bestimmte Tischposition aus. Klicken Sie auf Bilder laden. Für jeden Kanal/jede Wellenlänge werden Fenster angezeigt. Speichern Sie jede Kompilierung mit dem jeweiligen Kanalnamen als .tiff Datei.
- Um in ImageJ zu kompilieren, öffnen Sie die Dateien für alle Zeitpunkte für eine Tischposition und einen Kanal. Kompilieren Sie mit Images to Stack.
- Bereiten Sie die Umgebungskontrollen des Mikroskops und der Bildgebungskammer vor. Stellen Sie die Temperaturen des Stage-Top-Inkubators und des Großkammer-Inkubators auf 37 °C ein und schalten Sie den Kammerbefeuchter ein. Legen Sie eines der Agarose-Pad-Präparate in den Inkubator auf der Bühne und schließen Sie die Inkubationskammer, damit sich die Probe äquilibrieren kann. Der Befeuchtungsdeckel des Inkubators auf der Bühne wird an dieser Stelle offen gelassen, um Kondensation auf dem 25-mm-Deckglas der Kammer zu vermeiden.
5. Erstellen von Zeitraffervideos mit Fiji/ImageJ
HINWEIS: Fiji (Fiji ist nur ImageJ) ist eine frei verfügbare Software zur Bildverarbeitung und -analyse, die hier heruntergeladen werden kann: "https://imagej.net/software/fiji/downloads32"32. Für die unten beschriebenen Bildverarbeitungsverfahren wurde Fiji/ImageJ2 1.54f verwendet.
- Korrigieren Sie den Phasenkanal-Bildstapel mit BaSiC33. Öffnen Sie den gewünschten Phasen-Image-Stack in Fiji und wählen Sie dann BaSiC auf der Registerkarte Plugins aus. Der schattierungskorrigierte Stapel wird in einem separaten Fenster mit dem Titel Corrected:ImageName angezeigt.
HINWEIS: BaSiC.jar können hier heruntergeladen werden: "https://github.com/marrlab/BaSiC33"33. Befolgen Sie die Anweisungen des Entwicklers, um BaSiC korrekt auf Fidschi zu installieren. - Führen Sie den farbtonkorrigierten Phasenkanalstapel mit allen anderen Kanälen/Wellenlängen zusammen, indem Sie die > "Farbe" > "Kanäle zusammenführen" verwenden. Passen Sie den Hintergrund und die Signalintensität mit Image > Passen Sie > Helligkeit/Kontrast für jeden Kanal an. Speichern Sie die zusammengeführte Datei als .tiff.
- Stabilisieren Sie dann die Bilder mit "3D-Drift korrigieren". Wenn der schattierungskorrigierte Stack ausgewählt ist, gehen Sie zu Plugins > Registrierungen > 3D-Drift korrigieren. Legen Sie im sich öffnenden Dialogfenster den Kanal für die Registrierung auf die Kanalnummer fest, die dem Phasenstack entspricht. Der resultierende Stapel nach der Korrektur wird mit registrierten Zeitpunkten betitelt. Schneiden Sie die Datei auf das gewünschte Sichtfeld zu und speichern Sie die korrigierte Datei als .tiff.
HINWEIS: Wenn MetaMorph verwendet wurde, um den kompilierten Bildstapel zu erstellen, gibt es einen zusätzlichen Verarbeitungsschritt: Gehen Sie zu Image > Properties und tauschen Sie die Zahlen in den Feldern Slices (z) und Frames (t) aus. Die Drift-Korrektur kann jetzt jeden Frame korrekt als Zeitpunkt und nicht als Slice eines Z-Stapels interpretieren. Die schattierungskorrigierte und driftkorrigierte .tiff Datei kann für nachfolgende Bildanalysen verwendet werden. Es gibt viele Softwarepakete, um die Intensität von Fluoreszenzsignalen zu messen, morphologische Eigenschaften zu quantifizieren, das Schicksal einzelner Zellen zu verfolgen und vieles mehr. Zwei häufig verwendete Programme sind das MicrobeJ Plug-in für Fidschi und Oufti34,35. - Fügen Sie dem Stapel Zeitstempel und Textbeschriftungen hinzu, indem Sie > > Beschriftung Stapel verwenden.
- Um eine Maßstabsleiste hinzuzufügen, müssen Sie zunächst die Pixelgröße der Kamera und die Vergrößerung des Mikroskopobjektivs kennen. Berechnen Sie das Pixel:Mikron-Verhältnis als Pixelgröße geteilt durch die Vergrößerung. Geben Sie im Dialogfeld Analysieren > Maßstab festlegen das Pixel-Mikron-Verhältnis in das Feld für den bekannten Abstand ein. Fügen Sie dann dem Stapel mit den Tools "> analysieren" > "Maßstabsleiste" eine Maßstabsleiste hinzu.
HINWEIS: Die genaue Skala eines Mikroskops sollte mit einem Tischmikrometer kalibriert werden. Das verwendete Mikroskopobjektiv und die Kamera können jedoch eine grobe Abschätzung des Maßstabs liefern. So hat die hier verwendete PCO sCMOS-Kamera eine Pixelgröße von 6,5 x 6,5 μm2, und für die Bildgebung wurde das 63x Objektiv verwendet, so dass die Pixelgröße geteilt durch die Vergrößerung 6,5/63 = 0,1032 μm pro Pixel beträgt. Geben Sie 0,1032 in das Feld "Bekannte Entfernung" für das Dialogfeld "Maßstab festlegen " ein. - Um den fertigen Bildstapel als Video zu exportieren, das in QuickTime Player, Microsoft PowerPoint usw. abgespielt werden kann, speichern Sie es als .avi Datei.
Ergebnisse
Die erfolgreiche Einführung von Reporterplasmiden in P. aeruginosa und S. aureus wird durch das Wachstum mit den richtigen selektiven Antibiotika angezeigt und kann durch Kolonie-PCR und/oder Sequenzierung bestätigt werden. Die modifizierten Stämme sollten als phänotypische Reporter verifiziert werden, indem sie Bedingungen ausgesetzt werden, von denen bekannt ist, dass das interessierende Gen induziert wird, und die resultierende Fluoreszenz kann durch Durchflusszytometrie, Spektrophotometrie oder Epifluoreszenzmikroskopie gemessen werden (Abbildung 1).
Um die Auswahl einer oder mehrerer Antibiotikadosen zu erleichtern, die für nachfolgende Experimente verwendet werden, führen Sie konzentrationsabhängige Antibiotika-Persister-Assays für die interessierenden P . aeruginosa - oder S. aureus-Stämme durch. Konzentrationsabhängige Assays führen in der Regel zu einer biphasischen Kurve mit einer steilen anfänglichen Steigung bei niedrigeren Antibiotikakonzentrationen und einer plateauartigen oder weniger steilen Steigung bei höheren Konzentrationen. Bei einigen Antibiotika-Spezies-Paaren kann es jedoch nicht zu einer eindeutigen biphasischen Kurve kommen. Zum Beispiel ist die Kurve für S. aureus dElafloxacin-Kurve eindeutig zweiphasig (Abbildung 3A), aber die P . aeruginosa Levofloxacin-Kurve ist es nicht (Abbildung 3B)15. In diesem Szenario würden wir eine Konzentration wählen, die mindestens das 10-fache der MHK beträgt (z. B. 5 μg/ml, was etwa dem 15-fachen der MHK für P. aeruginosa entspricht)15. Da jedoch eine 15-fache Levofloxacin-MHK nur zu ~0,001 % P . aeruginosa-Überlebenden führt, verwenden wir eine Behandlung mit 1 μg/ml Levofloxacin, wenn wir bei der Bildgebung von Zellen Persister sehen möchten, wie sie sich auf antibiotikafreien Agarose-Pads erholen (Ergänzendes Video 6); Andernfalls wird die Anzahl der Sichtfelder, die zum Abbilden mehrerer Persister erforderlich sind, unerschwinglich.
Zu Beginn der Bildgebung sollte die ideale Proben- und Agarose-Pad-Vorbereitung über das gesamte Sichtfeld planar erscheinen, frei von großen Ablagerungen, Falten oder Luftblasen und mit gleichmäßig verteilten Einzelzellen. Um gut verteilte Einzelzellen zu erhalten, kann eine Optimierung der Probenverdünnung oder -resuspension erforderlich sein. Bei S. aureus neigen die Zellen dazu, kleine Cluster zu bilden und müssen gründlich vortexiert werden, bevor sie auf das Agarosepad ausgesät werden (Abbildung 4 und Abbildung 5). Bei P. aeruginosa können Zellen Aggregate bilden, die in einer klebrigen extrazellulären Matrix in Suspension eingeschlossen sind; Es ist notwendig, diese Proben gründlich zu pipettieren und die Aggregate für die Bildgebung einzelner Zellen aufzubrechen.
Nach Abschluss eines bildgebenden Experiments erscheint ein erfolgreicher Zeitraffer von Bildern im Fokus, stabil beleuchtet und mit minimaler Drift in der x-y-Ebene während des gesamten Experiments. Ergänzendes Video 7 stellt eine optimale Bildaufnahme dar: t ist der Phasenkanal von Ergänzendes Video 4 vor der Schatten- oder Driftkorrektur. Ein Fokusverlust kann auftreten, wenn sich durch Kondensation (durch Überfeuchtung oder unzureichende Probenerwärmung) Wassertropfen auf dem oberen 25-mm-Deckglas bilden, die das Licht verzerren und die Fokusebene außerhalb des maximalen Suchbereichs des Autofokus-Algorithmus verschieben (Ergänzendes Video 8). Schwankungen in der Beleuchtung deuten in der Regel auf ein unzureichendes Immersionsöl zum Zeitpunkt der Bildgebung hin. Wenn sich der Tisch zu schnell bewegt, kann es sein, dass das Öl auf dem Objektiv hinterherschleift und bei der Aufnahme der Bilder immer noch aufholt. Dies kann abgemildert werden, indem Sie die Aufnahmesteuerung so anpassen, dass die Bewegungsgeschwindigkeit verlangsamt wird, oder indem Sie eine Pause zwischen der Bewegung zur nächsten Position und der Bildaufnahme hinzufügen. Eine größere Probendrift sieht so aus, als würden viele Zellen über das Sichtfeld streifen, während andere an Ort und Stelle bleiben (Ergänzendes Video 9). Dies tritt in der Regel später in Experimenten auf, da das Agarose-Pad aufgrund unzureichender Feuchtigkeitskontrolle dehydriert ist. Die in dieser Arbeit vorgestellte Agarose-Pad-Vorbereitung wurde entwickelt, um die Probenstabilität zu verbessern, aber eine ordnungsgemäße Erwärmung/Befeuchtung der Probe und ihrer Umgebung ist für eine optimale Bildaufnahme erforderlich.
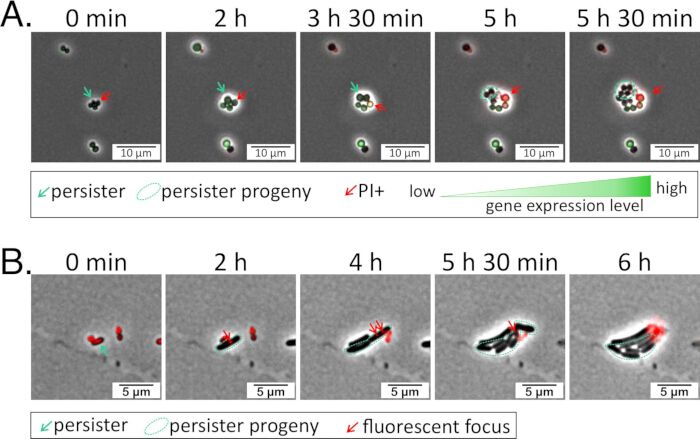
Abbildung 1: Fluoreszierende Reporterstämme beleuchten die Expression eines Gens von Interesse. (A) S. aureus wurde mit einem GFP-Transkriptionsreporter für ein Gen von Interesse gemäß Protokoll 1 transduziert. Der Reporterstamm wurde 24 Stunden lang mit einem Antibiotikum behandelt, mit PBS gewaschen und dann auf ein Agarosepad aus CA-MHB plus Propidiumiodid (1,6 μM) und Chloramphenicol (10 μg/ml zur Aufrechterhaltung des Reporterplasmids) zur Bildgebung während der Genesung ausgesät (Ergänzendes Video 1). (B) P. aeruginosa wurde mit einem Plasmid transformiert, das einen mScarlet-verknüpften translationalen Reporter für ein Protein von Interessetrug 26. Der Reporterstamm wurde 5 Stunden lang mit einem Antibiotikum behandelt, mit PBS gewaschen und dann auf ein Agarose-Pad aus BSM plus Tet (75 μg/ml; zur Erhaltung des Reporterplasmids) zur Bildgebung während der Genesung ausgesät (Ergänzendes Video 2). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
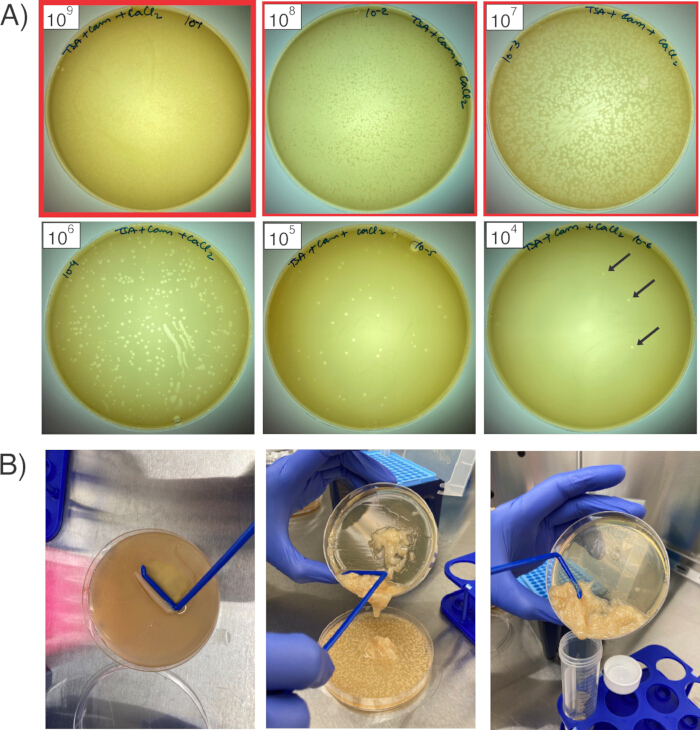
Abbildung 2: Vermehrung und Ernte von Bakteriophagen. (A) Die sechs Tafeln zeigen sechs verschiedene Mengen an verdünntem Phagenbestand auf den Rasenflächen von S. aureus RN4220. Die roten Umrisse zeigen die drei Platten, die geerntet werden würden, von der Platte mit der größten Klarheit (fette rote Umrandung; 1 x109 PFU/ml) bis zu den nächsten beiden Verdünnungen (1 x108 und 1 x 107 PFU/ml). Die schwarzen Pfeile zeigen auf einzelne Tafeln. (B) Um Phagen aus den Platten zu ernten, kratzen Sie die weiche Agarschicht (links) ab, übertragen Sie die Aufschlämmung auf die nächste Verdünnungsplatte (Mitte) und kombinieren Sie sie, nachdem Sie den weichen Agar von allen drei Platten zusammengepoolt haben, zu einem konischen Röhrchen für die Zentrifugation (rechts). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
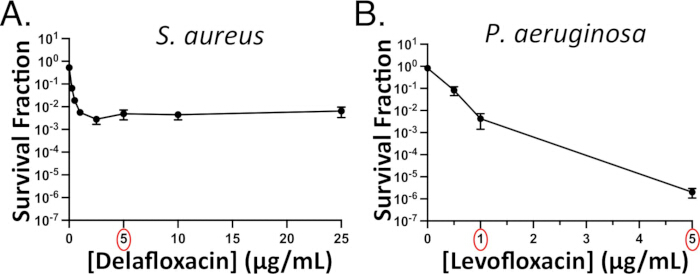
Abbildung 3: Repräsentative konzentrationsabhängige Persister-Assays. Die konzentrationsabhängige Fluorchinolon-Persistenz wurde in stationärer Phase (A) S. aureus (gegen Delafloxacin) und (B) P. aeruginosa (gegen Levofloxacin) untersucht. Nachfolgende Experimente verwenden 5 μg/ml Delafloxacin (roter Kreis), da die Tötung von S. aureus bei dieser Konzentration ein Plateau erreicht hatte. Eine Dosierung von mindestens 1 μg/ml Levofloxacin (roter Kreis) würde für nachfolgende Experimente mit P. aeruginosa verwendet werden. Beachten Sie, dass die bakterielle Abtötung für P. aeruginosa kein Plateau erreicht, aber es gibt immer noch eine weniger steile "zweite Phase" der biphasischen Kurve, die auf eine persistente Subpopulation hinweist. Panel 3B wurde mit Genehmigung von Hare et al.15 adaptiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
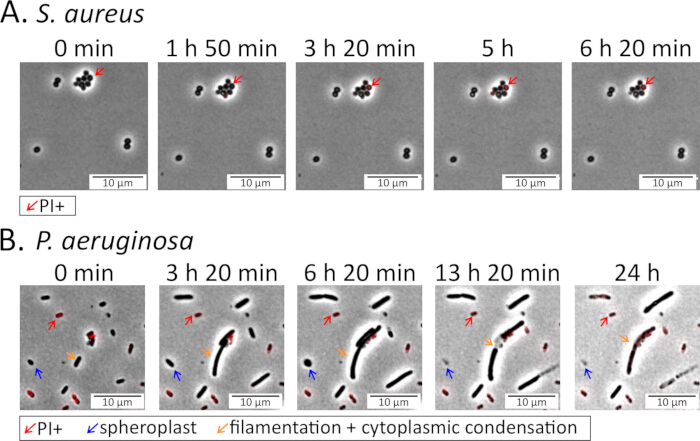
Abbildung 4: Bildgebung bakterieller Phänotypen während der Antibiotikabehandlung. Stationäre (A) S. aureus - und (B) P. aeruginosa-Zellen wurden auf Agarose-Pads, die Fluorchinolon-Antibiotika enthielten, ausgesät und während der Behandlung überwacht: 5 μg/ml Delafloxacin für S. aureus (Ergänzendes Video 3) und 5 μg/ml Levofloxacin für P. aeruginosa (Ergänzendes Video 4)15. Propidiumiodid (PI; 16 μM für P. aeruginosa, 1,6 μM für S. aureus) wurde den Pads zugesetzt, um tote oder sterbende Zellen zu markieren. S. aureus-Zellen bleiben in Gegenwart des FQ weitgehend intakt und am Leben, während die meisten P. aeruginosa-Zellen drastische morphologische Veränderungen durchlaufen, einschließlich der Bildung runder Sphäroplasten, bevor sie lysieren und absterben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
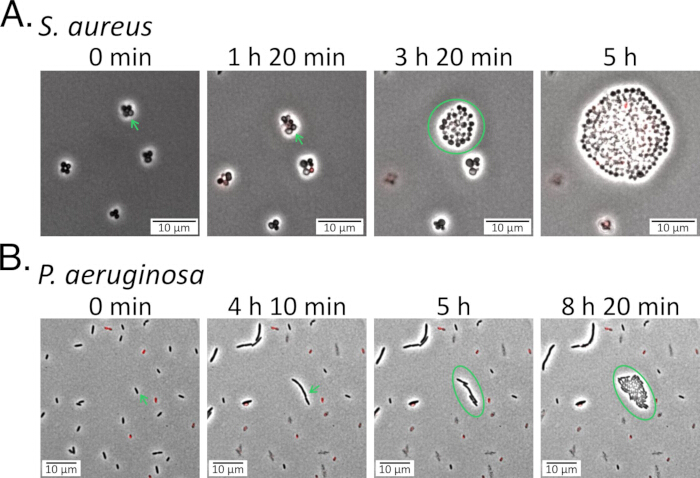
Abbildung 5: Verfolgung von Persistern während der Erholung. (A) S. aureus - und (B) P. aeruginosa-Populationen wurden auf Agarose-Pads mit frischem Medium ausgesät, nachdem sie mit Fluorchinolonen (5 μg/ml Delafloxacin für S. aureus und 1 μg/ml Levofloxacin für P. aeruginosa) behandelt und während ihrer Genesung nach der Behandlung überwacht worden waren (Ergänzendes Video 5 und Ergänzendes Video 6). Die Perisisters sind in den ersten beiden Bildern in jedem Panel mit grünen Pfeilen gekennzeichnet und blieben während der Antibiotikabehandlung intakt und lebensfähig. Nach einer anfänglichen Verzögerungsphase begannen sich die Persister zu teilen und brachten neue Nachkommen hervor (gekennzeichnet durch grüne Kreise). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
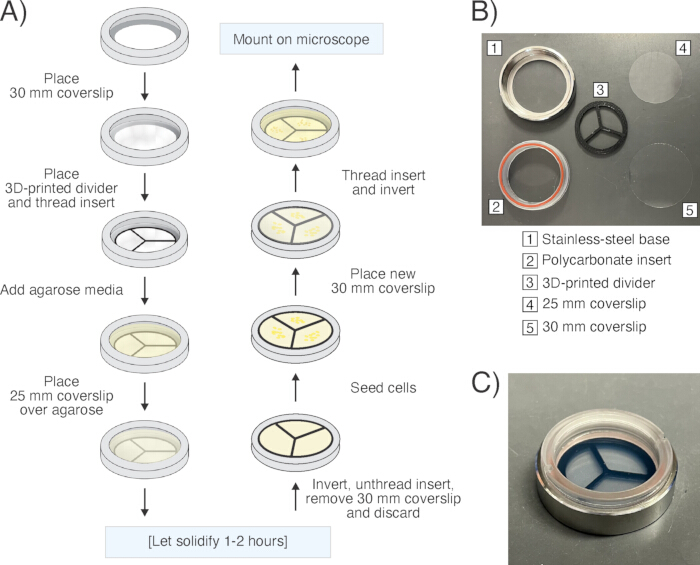
Abbildung 6: Probenvorbereitung für das Mikroskop. (A) Schematische Darstellung des Arbeitsablaufs bei der Probenvorbereitung unter Verwendung einer austauschbaren Deckglasschale ("Kammer"). (B) Bild der zerlegten Kammer und ihrer einzelnen Komponenten. (C) Bild der vollständig zusammengebauten Kammer. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzendes Video 1: S. aureus Persister. Videodatei mit den Bildern in Abbildung 1A. Kurz gesagt, S. aureus , der einen GFP-Transkriptionsreporter für ein Gen von Interesse trug, wurde 24 Stunden lang mit einem Antibiotikum behandelt, mit PBS gewaschen und dann auf ein Agarose-Pad aus CA-MHB plus Propidiumiodid (1,6 μM) und Chloramphenicol (10 μg/ml) zur Bildgebung während der Genesung ausgesät. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Ergänzendes Video 2: P. aeruginosa Persister. Videodatei mit Bildern in Abbildung 1B. Kurz gesagt, P. aeruginosa mit einem mScarlet-linked translationalen Reporter für ein Protein von Interesse wurde 5 Stunden lang mit einem Antibiotikum behandelt, mit PBS gewaschen und dann auf ein Agarose-Pad mit BSM plus Tet (75 μg/ml) ausgesät, um während der Genesung bildgebendzu machen 26. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Ergänzendes Video 3: S. aureus während der Antibiotikabehandlung. Eine stationäre Phasenkultur von S. aureus, die in reichhaltigen, chemisch definierten Medien gezüchtet wurde, wurde auf Agarose-Pads ausgesät, die aus den zellfreien konditionierten Medien der Kultur mit Propidiumiodid (1,6 μM) und Delafloxacin (5 μg/ml) hergestellt wurden. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Ergänzendes Video 4: P. aeruginosa während der Antibiotikabehandlung. Eine stationäre Phasenkultur von P. aeruginosa , die in BSM gezüchtet wurde, wurde auf Agarose-Pads ausgesät, die aus zellfrei konditionierten Medien aus einer parallel in BSM gezüchteten Kultur von P. aeruginosa hergestellt wurden; Das Agarose-Pad enthielt auch Propidiumiodid (16 μM) und Levofloxacin (5 μg/ml). Dieses Video wurde mit Genehmigung von Hare et al.15 adaptiert. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Ergänzendes Video 5: S. aureus während der Genesung nach Antibiotika. S. aureus wurde in reichhaltigen, chemisch definierten Medien in die stationäre Phase gezüchtet. Die stationären Phasenkulturen wurden 24 Stunden lang mit 5 μg/ml Delafloxacin in Reagenzgläsern behandelt, mit PBS gewaschen und dann für die Bildgebung auf antibiotikafreie CA-MHB-Agarose-Pads mit Propidiumiodid (1,6 μM) ausgesät. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Ergänzendes Video 6: P. aeruginosa während der postantibiotischen Erholung. Eine stationäre Phasenkultur von P. aeruginosa , die in BSM gezüchtet wurde, wurde 7 Stunden lang mit 1 μg/ml Levofloxacin in Reagenzgläsern behandelt, mit PBS gewaschen und dann zur Bildgebung auf antibiotikafreie BSM-Agarose-Pads mit Propidiumiodid (16 μM) ausgesät. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Ergänzendes Video 7: Beispiel für eine optimale Bildaufnahme. Dieses Video ist der Phasenkanal von Supplementary Video 4 vor der Bildverarbeitung als Beispiel für eine optimale Zeitrafferaufnahme. Beachten Sie die minimale Drift, die stabile Beleuchtung und die Beibehaltung des Fokus während des gesamten Experiments. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Ergänzendes Video 8: Beispiel für eine suboptimale Bildaufnahme durch Kondensation. Dieses Video zeigt einen Teil eines Experiments, bei dem die Bildaufnahme durch eine schlechte Schärfe beeinträchtigt wurde, wahrscheinlich aufgrund von Kondensation in der Kammer aufgrund unsachgemäßer Erwärmung der Probe und/oder Überbefeuchtung der Bildgebungsumgebung. Bei der abgebildeten Probe handelte es sich um mit Levofloxacin behandelte P. aeruginosa während der postantibiotischen Erholung auf einem BSM-Agarose-Pad. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Ergänzendes Video 9: Beispiel für eine suboptimale Bildaufnahme aufgrund von Drift. Dieses Video zeigt einen Teil eines Experiments, bei dem die Bildaufnahme durch Probendrift beeinträchtigt wurde, wahrscheinlich aufgrund von Dehydrierung und dem Schrumpfen/Abheben des Agarose-Pads vom Deckglas. Bei der abgebildeten Probe handelte es sich um P. aeruginosa auf einem Agarose-Pad, das Levofloxacin und Propidiumiodid enthielt. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Ergänzende Datei 1: 25mm-3D-Teiler-für-35mmBioptechs.stl Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Wir haben festgestellt, dass der Erfolg eines Zeitraffermikroskopie-Experiments von der Qualität der Agarose-Pads und ihrer Stabilität im Verlauf der Bildgebung abhängt. Die kammergekapselten Agarose-Pads aus Edelstahl sind relativ einfach herzustellen, was zu gleichbleibend planaren Proben führt, die über Dutzende von Stunden stabil abgebildet werden können. Dies ermöglicht die Bildgebung von Zehntausenden von Zellen in einem einzigen Experiment und erhöht die Wahrscheinlichkeit, seltene phänotypische Varianten, wie z. B. Persisterzellen, in einer Population zu erkennen.
Diese Methode zur Herstellung von Agarose-Pads stellt eine leicht umsetzbare Alternative zu bisher veröffentlichten Methoden dar. Unser Protokoll erfordert nicht die technische Präzision der Herstellung von mikrofluidischen Geräten oder die geschickten Manipulationen von Agarose-"Sandwich"-Methoden, was es einfacher macht, konsistente Präparationen von Lauf zu Lauf zu erreichen 14,16,36. Darüber hinaus ist das System kostengünstig. Die Edelstahlkammer ist sterilisierbar und wiederverwendbar (im Gegensatz zu Einweg-Plastikkammern), und für den Aufbau ist keine spezielle Ausrüstung erforderlich16,37. Die Kammer lässt sich mit handelsüblichen Tischeinsätzen problemlos an verschiedene Mikroskopiesysteme anpassen. Da die Bakterien an der Grenzfläche zwischen Agarose und Deckglas immobilisiert sind, ist es uns außerdem gelungen, hochbewegliche Bakterien wie P. aeruginosa zu verfolgen und gleichzeitig morphologische Veränderungen zu ermöglichen (Abbildung 4, Ergänzendes Video 4). Andere Einzelzell-Bildgebungsverfahren, wie z. B. die "Muttermaschine", beschränken Zellen auf Kanäle, die die Beobachtung anderer morphologischer Veränderungen als der Filamentierung ausschließen36.
Für den Erfolg dieses Protokolls gibt es einige kritische Schritte und Parameter, die Sie beachten sollten. Für die Padvorbereitung ist es wichtig, die Agarose gründlich zu erhitzen und zu schmelzen, da verbleibende Agarosekristalle Lichtbeugung verursachen und die Bildqualität beeinträchtigen. Ebenso muss man darauf achten, dass die Agarose in die Kammer pipettiert wird, ohne dass Luftblasen entstehen. Um sicherzustellen, dass die Dicke der Agarose-Pads konstant bleibt, und um die Probendrift zu begrenzen, ist es wichtig, das Pad vor Beginn der Bildgebung in dem befeuchteten, temperaturkontrollierten Gehäuse äquilibrieren zu lassen – in der Regel 15 Minuten lang. Ein weiterer Faktor, der zu einer schlechten Bildqualität führen kann, ist die Feuchtigkeitskontrolle: Eine niedrige Luftfeuchtigkeit führt dazu, dass das Agarose-Pad austrocknet und schrumpft, während eine hohe Luftfeuchtigkeit (oder eine unsachgemäße Erwärmung der Probe in der Kammer) dazu führen kann, dass die warme Luft auf der Probe kondensiert und die Bildgebung verzerrt. Ein Beispiel für suboptimale Zeitrafferaufnahmen aufgrund von Kondensation finden Sie im Ergänzenden Video 8.
Eine Einschränkung des derzeitigen Aufbaus besteht darin, dass die Nährmedien nicht ausgetauscht werden können, was eine kontinuierliche Verfolgung einzelner Bakterien vor, während und nach der Antibiotikabehandlung verhindert. Wir gehen davon aus, dass die Kopplung des Agarose-Pads mit Durchflusszellen oder mikrofluidischen Geräten, die den Austausch von Kulturmedien ermöglichen, die Verfolgung von Populationen während des Ernährungs- oder Umweltwandels ermöglichen könnte. Ein weiterer Parameter des aktuellen Designs, der verbessert werden könnte, ist die Belüftung der Probe. Die O-Ring-Dichtung und das Schraubverschluss-Design der austauschbaren Deckglasschale ermöglichen eine bessere Belüftung der Probe im Vergleich zu Setups, die die Verwendung von Dichtmittel auf Wachs- oder Fettbasis zur Abdichtung der Padserfordern 16. Die Belüftung in der geschlossenen Kammer kann jedoch immer noch begrenzt sein und das Wachstum obligater Aerobier möglicherweise nicht unterstützen, obwohl dies noch getestet werden muss.
Das Zeitraffer-Imaging-Probenvorbereitungsprotokoll, das wir in diesem Artikel vorstellen, ermöglicht die Verfolgung von Tausenden von Bakterien, während sie auf eine Antibiotikabehandlung ansprechen oder sich davon erholen. Diese Methode ist zudem hochgradig verallgemeinerbar und hat eine Vielzahl von Anwendungsmöglichkeiten über die Persisterbiologie hinaus. Zum Beispiel ermöglicht der Aufbau des Agarose-Pads und des Teilers die Aussaat von räumlich getrennten Zellproben, ermöglicht aber auch die Zell-Zell-Kommunikation durch Diffusion durch das Agarose-Pad. Wir untersuchen derzeit das Potenzial dieses Aufbaus, um zu testen, wie sich der Austausch von sekretierten Produkten auf das Zellwachstum in Multispezies-Gemeinschaften auswirkt. Wir gehen davon aus, dass dieses Protokoll dem neuen Forscher eine niedrige Einstiegshürde in die Zeitraffermikroskopie und dem erfahrenen Mikrobiologen unbegrenzte Variationen zur Erkundung bietet.
Offenlegungen
Die Autoren haben keine Interessenkonflikte anzugeben.
Danksagungen
Wir danken Frau Susan Staurovsky von der UConn Health Center for Cell Analysis and Modeling Microscopy Facility für ihre Unterstützung bei den Mikroskopie-Experimenten. Wir danken Dr. Mona Wu Orr und dem Essentials of Staphylococcal Genetics and Metabolism Workshop für ihre Protokolle und Ratschläge zum Klonen bei P. aeruginosa bzw . S. aureus. Diese Arbeit wurde mit Mitteln der National Institutes of Health (NIH; DP2GM146456-01 und 1R01AI167886-01A1 bis W.W.K.M., 1F30DE032598-01A1 bis P.J.H. und 1F31DK136259-01A1 bis T.J.L.). Die Geldgeber spielten keine Rolle bei der Gestaltung unserer Experimente oder der Vorbereitung dieses Manuskripts.
Materialien
| Name | Company | Catalog Number | Comments |
| BaSiC | GitHub | https://github.com/marrlab/BaSiC | |
| Certified Molecular Biology Agarose | Biorad | 1613101 | |
| Fiji-ImageJ | NIH | https://imagej.net/software/fiji/downloads | |
| Interchangeable Coverglass Dish | Bioptechs | 190310-35 | 35 mm ICD for preparing agarose pads; comes with 30 mm (#1.5) coverslips |
| Lumencor Spectra 7 LED light engine | Lumencor | https://lumencor.com/products/spectra-light-engine | Spectra 7 LED light engine |
| MetaMorph | Molecular Devices | Premier version 7.10.5 | |
| pco.edge 4.2 bi sCMOS camera | Excelitas | https://www.excelitas.com/product/pcoedge-42-bi-usb-scmos-camera | sCMOS camera |
| PeCon live cell incubation chamber | peCon | https://www.pecon.biz/ | |
| Thomas Scientific Round cover glass, #1.5 thickness, 25 mm, 100 pack | Fisher Scientific | NC1272770 | 25 mm (#1.5) coverslips |
| Zeiss Axiovert 200M microscope | Zeiss | https://www.zeiss.com/microscopy/en/products/light-microscopes/widefield-microscopes/axiovert-for-materials.html | Inverted microscope with Plan-Apochromat 63x/1.40 Oil Ph3 M27 objective, Lumencor Spectra 7 LED light engine, and pco.edge 4.2 bi sCMOS camera (6.5 mm pixel size) |
Referenzen
- Brauner, A., Fridman, O., Gefen, O., Balaban, N. Q. Distinguishing between resistance, tolerance and persistence to antibiotic treatment. Nat Rev Microbiol. 14, 320-330 (2016).
- Balaban, N. Q., et al. Definitions and guidelines for research on antibiotic persistence. Nat Rev Microbiol. 17 (7), 441-448 (2019).
- Conlon, B. P. Staphylococcus aureus chronic and relapsing infections: evidence of a role for persister cells. Bioessays. 36 (10), 991-996 (2014).
- Mulcahy, L. R., Burns, J. L., Lory, S., Lewis, K. Emergence of Pseudomonas aeruginosa strains producing high levels of persister cells in patients with cystic fibrosis. J Bacteriol. 192 (23), 6191-6199 (2010).
- Verstraete, L., Van den Bergh, B., Verstraeten, N., Michiels, J. Ecology and evolution of antibiotic persistence. Trends Microbiol. 30 (5), 466-479 (2021).
- Sett, A., Dubey, V., Bhowmik, S., Pathania, R. Decoding bacterial persistence: Mechanisms and strategies for effective eradication. ACS Infect Dis. 10 (8), 2525-2539 (2024).
- Mempin, R., et al. Release of extracellular ATP by bacteria during growth. BMC Microbiol. 13, 301 (2013).
- Lewis, K. Persister cells, dormancy and infectious disease. Nat Rev Microbiol. 5 (1), 48-56 (2006).
- Fauvart, M., De Groote, V. N., Michiels, J. Role of persister cells in chronic infections: clinical relevance and perspectives on anti-persister therapies. J Med Microbiol. 60 (pt 6), 699-709 (2011).
- Van den Bergh, B., Fauvart, M., Michiels, J. Formation, physiology, ecology, evolution and clinical importance of bacterial persisters. FEMS Microbiol Rev. 41, 219-251 (2017).
- Levin, B. R., Rozen, D. E. Non-inherited antibiotic resistance. Nat Rev Microbiol. 4 (7), 556-562 (2006).
- Davis, K. M., Isberg, R. R. Defining heterogeneity within bacterial populations via single cell approaches. Bioessays. 38 (8), 782-790 (2016).
- Hare, P. J., LaGree, T. J., Byrd, B. A., DeMarco, A. M., Mok, W. W. K. Single-cell technologies to study phenotypic heterogeneity and bacterial persisters. Microorganisms. 9 (11), 2277 (2021).
- de Jong, I. G., Beilharz, K., Kuipers, O. P., Veening, J. W. Live cell imaging of Bacillus subtilis and Streptococcus pneumoniae using automated time-lapse microscopy. J Vis Exp. 53, e3145 (2011).
- Hare, P. J., Gonzalez, J. R., Quelle, R. M., Wu, Y. I., Mok, W. W. K. Metabolic and transcriptional activities underlie stationary-phase Pseudomonas aeruginosa sensitivity to levofloxacin. Microbiol Spectr. 12 (1), e0356723 (2024).
- Young, J. W., et al. Measuring single-cell gene expression dynamics in bacteria using fluorescence time-lapse microscopy. Nat Protoc. 7 (1), 80-88 (2011).
- Pang, Y. Y., et al. agr-Dependent interactions of Staphylococcus aureus USA300 with human polymorphonuclear neutrophils. J Innate Immun. 2 (6), 546-559 (2010).
- Bose, J. L., Fey, P. D., Bayles, K. W. Genetic tools to enhance the study of gene function and regulation in Staphylococcus aureus. Appl Environ Microbiol. 79 (7), 2218 (2013).
- Schenk, S., Laddaga, R. A. Improved method for electroporation of Staphylococcus aureus. FEMS Microbiol Lett. 73, 133-138 (1992).
- Grosser, M. R., Richardson, A. R. Method for Preparation and Electroporation of S. aureus and S. epidermidis. The Genetic Manipulation of Staphylococci. Methods Molecular Biology. , (2016).
- Krausz, K. L., Bose, J. L. Bacteriophage Transduction in Staphylococcus aureus: Broth-Based Method. The Genetic Manipulation of Staphylococci. Methods Molecular Biology. , (2016).
- Olson, M. E. Bacteriophage Transduction in Staphylococcus aureus. The Genetic Manipulation of Staphylococci. Methods Molecular Biology. , (2016).
- Fey, P. D., et al. A genetic resource for rapid and comprehensive phenotype screening of nonessential Staphylococcus aureus genes. mBio. 4 (1), e00537-e00612 (2013).
- Coe, K. A., et al. Multi-strain Tn-Seq reveals common daptomycin resistance determinants in Staphylococcus aureus. PLoS Pathog. 15 (11), e1007862 (2019).
- Figurski, D. H., Helinski, D. R. Replication of an origin-containing derivative of plasmid RK2 dependent on a plasmid function provided in trans. Proc Natl Acad Sci U S A. 76 (4), 1648 (1979).
- Shee, C., et al. Engineered proteins detect spontaneous DNA breakage in human and bacterial cells. eLife. 2, e01222 (2013).
- Kowalska-Krochmal, B., Dudek-Wicher, R. The minimum inhibitory concentration of antibiotics: methods, interpretation, clinical relevance. Pathogens. 10 (2), 165 (2021).
- Robinson, J. L., Jaslove, J. M., Murawski, A. M., Fazen, C. H., Brynildsen, M. P. An integrated network analysis reveals that nitric oxide reductase prevents metabolic cycling of nitric oxide by Pseudomonas aeruginosa. Metab Eng. 41, 67-81 (2017).
- Wolff, J. A., MacGregor, C. H., Eisenberg, R. C., Phibbs, P. V. Isolation and characterization of catabolite repression control mutants of Pseudomonas aeruginosa PAO. J Bacteriol. 173 (15), 4700-4706 (1991).
- Batchelder, J. I., Taylor, A. J., Mok, W. W. K. Metabolites augment oxidative stress to sensitize antibiotic-tolerant Staphylococcus aureus to fluoroquinolones. mBio. , e0271424 (2024).
- Power, A. D., Mok, W. W. K. Agar and agarose used for Staphylococcus aureus biofilm cultivation impact fluoroquinolone tolerance. J Appl Microbiol. 135 (8), lxae191 (2024).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Peng, T., et al. A BaSiC tool for background and shading correction of optical microscopy images. Nat Commun. 8, 14836 (2017).
- Ducret, A., Quardokus, E., Brun, Y. V. MicrobeJ, a tool for high throughput bacterial cell detection and quantitative analysis. Nat Microbiol. 1 (7), 16077 (2016).
- Paintdakhi, A., et al. Oufti: an integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Mol Microbiol. 99 (4), 767-777 (2016).
- Wang, P., et al. Robust growth of Escherichia coli. Curr Biol. 20 (12), 1099-1103 (2010).
- Moffitt, J. R., Lee, J. B., Cluzel, P. The single-cell chemostat: an agarose-based, microfluidic device for high-throughput, single-cell studies of bacteria and bacterial communities. Lab Chip. 12 (8), 1487-1494 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten