Method Article
Imagerie par microscopie à épifluorescence time-lapse de Pseudomonas aeruginosa et Staphylococcus aureus Phénotypes hétérogènes
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Dans ce manuscrit, nous fournissons un protocole complet pour évaluer la survie aux antibiotiques de Pseudomonas aeruginosa et Staphylococcus aureus, en transformant les plasmides en P. aeruginosa et S. aureus pour créer des souches rapporteures et visualiser des variantes phénotypiques, telles que les persistants, par microscopie à épifluorescence time-lapse.
Résumé
La persistance des antibiotiques est un phénomène dans lequel un petit nombre de cellules bactériennes d’une population génétiquement sensible survivent à un traitement antibiotique qui tue les autres cellules génétiquement identiques. Les bactéries persistantes peuvent reprendre la réplication une fois le traitement antibiotique terminé et sont généralement considérées comme à l’origine de l’échec du traitement clinique. Des travaux récents exploitant la puissance de la microscopie à fluorescence time-lapse, dans laquelle les bactéries sont marquées avec des rapporteurs transcriptionnels fluorescents, des rapporteurs translationnels et/ou des colorants pour une variété de caractéristiques cellulaires, ont fait progresser notre compréhension des persisteurs d’Escherichia coli au-delà de ce qui pourrait être appris à partir d’essais de survie aux antibiotiques au niveau de la population. De telles approches unicellulaires, plutôt que des tests de population en vrac, sont essentielles pour délimiter les mécanismes de formation de plantes persistantes, de réponse aux dommages et de survie. Cependant, les méthodes d’étude des persistants chez d’autres espèces pathogènes importantes à ce niveau de détail restent limitées.
Cette étude fournit une approche adaptable pour l’imagerie en accéléré de Pseudomonas aeruginosa (un bâtonnet à Gram négatif) et de Staphylococcus aureus (un coccus à Gram positif) pendant le traitement antibiotique et la récupération. Nous discutons des approches de génétique moléculaire pour introduire des rapporteurs fluorescents dans ces bactéries. À l’aide de ces rapporteurs, ainsi que des colorants, nous pouvons suivre les changements phénotypiques, les caractéristiques morphologiques et le destin des cellules individuelles en réponse à un traitement antibiotique. De plus, nous sommes en mesure d’observer les phénotypes des individus persistants lorsqu’ils se réaniment après le traitement. Dans l’ensemble, ce travail sert de ressource pour ceux qui s’intéressent au suivi de la survie et de l’expression génique des cellules individuelles traitées aux antibiotiques, y compris les cellules persistantes, pendant et après le traitement, chez les agents pathogènes cliniquement importants.
Introduction
Les agents pathogènes bactériens peuvent échapper aux effets des antibiotiques par deux mécanismes principaux : la résistance aux antibiotiques, qui implique des changements génétiques, et la tolérance phénotypique, qui implique des changements non génétiques. La résistance aux antibiotiques est un phénomène génétiquement codé qui confère à une cellule bactérienne donnée la capacité non seulement de survivre, mais aussi de se répliquer en présence d’un antibiotique1. La tolérance phénotypique, qui peut englober des bactéries tolérantes aux antibiotiques ou persistantes aux antibiotiques, se produit lorsque les cellules résistent à un traitement antibiotique bactéricide sans acquérir la capacité de se répliquer en présence d’une concentration inhibitrice de l’antibiotique 1,2. Ce qui différencie la tolérance de la persistance, c’est que la tolérance fait référence à la capacité de l’ensemble de la population à survivre au traitement, tandis que la persistance fait référence à un sous-ensemble d’une population isogénique mais phénotypiquement hétérogène qui survit au traitement antibiotique. Lorsqu’une culture clonale est traitée avec des antibiotiques bactéricides et que les survivants qui restent dans la culture sont tracés en fonction du temps sur une échelle log-linéaire, une courbe biphasique est généralement détectée en présence de persistants. Sur ces courbes, la première phase montre que la majorité de la population est tuée relativement rapidement, et la deuxième phase indique qu’une fraction persistante d’antibiotiques est tuée à un rythme plus lent ou pas du tout 1,2.
La persistance des antibiotiques représente un fardeau majeur pour les systèmes de santé mondiaux. Par exemple, on pense que Staphylococcus aureus et Pseudomonas aeruginosa persistants, qui sont au centre de cet article, provoquent des infections antibiotiques-récalcitrantes, y compris des infections récurrentes des voies respiratoires chez les patients atteints de mucoviscidose et des infections chroniques des plaies 3,4. Par conséquent, il est essentiel d’élucider davantage la biologie des cellules persistantes et les programmes phénotypiques. Bien que des progrès aient été réalisés dans la compréhension de la formation et de la réanimation des plantes persistantes, des lacunes critiques dans les connaissances relatives à la coordination de la reprogrammation métabolique et des événements moléculaires dans les cellules individuelles qui sous-tendent la persistance subsistent 5,6,7,8.
L’étude efficace de la persistance s’est avérée être un défi technique. Étant donné que la persistance n’est observable que dans un petit sous-ensemble d’une population bactérienne, les techniques qui échantillonnent des populations bactériennes en vrac ne parviennent souvent pas à recueillir des informations biologiques pertinentes 1,2,8,9,10. De plus, étant donné que les changements phénotypiques qui sous-tendent la persistance sont transitoires et non héréditaires, le suivi du destin des cellules persistantes peut être complexe 1,8,9,10,11. Une fois que les bactéries persistantes reprennent leur croissance, elles peuvent se diviser et donner naissance à des persistants et des non-persistants, ce qui rend impossible l’enrichissement des populations de persistants purs par la culture. Ces défis mettent en évidence la nécessité de techniques capables de répondre aux critères suivants : 1) la capacité de capturer des informations biologiques de cellules uniques vivantes et 2) la capacité d’être utilisées en tandem avec des colorants fluorescents, des sondes, des capteurs et des rapporteurs qui permettent d’interroger les phénotypes de cellules individuelles dans des populations hétérogènes au fil du temps.
Les progrès récents des technologies unicellulaires ont permis d’étudier efficacement l’hétérogénéité bactérienne et de surmonter ces obstacles dans l’étude de la persistance12,13. Certaines de ces techniques comprennent la microscopie à fluorescence, la cytométrie en flux/le tri cellulaire activé par fluorescence, la microfluidique et le séquençage de l’ARN unicellulaire12,13. Ici, nous décrivons des protocoles pour élucider la physiologie persistante unicellulaire à l’aide de la microscopie à intervalle de temps d’épifluorescence de souches rapporteures transcriptionnelles ou translationnelles. La microscopie à fluorescence est une technique puissante qui répond aux critères d’étude des phénotypes persistants, à savoir la capacité d’identifier les cellules individuelles d’une grande population qui se propagent après l’élimination de l’antibiotique et peuvent donc être définies comme persistantes. Avec l’introduction de technologies de caméras automatisées et de chambres incubées, la capture de cellules bactériennes vivantes est largement accessible dans le domaine de la microbiologie. De manière cruciale, la microscopie time-lapse offre la possibilité de visualiser des cellules individuelles en temps réel, sur des heures et même des jours, ce qui permet de suivre les bactéries avant, pendant et après le traitement antibiotique 14,15,16. Les résultats de ces recherches, qui exploitent la microscopie time-lapse, ont un immense potentiel pour mieux comprendre les mécanismes complexes de la biologie persistante.
Protocole
1. Génération de souches rapporteures fluorescentes de S. aureus par transformation et transduction
REMARQUE : Les souches rapporteures hébergent une protéine fluorescente pour indiquer l’expression d’un gène ou d’une protéine d’intérêt. Les rapporteurs transcriptionnels présentent une copie dupliquée de la séquence promotrice native d’un gène d’intérêt en amont d’une protéine fluorescente, de sorte que la fluorescence augmente à mesure que l’expression d’un gène d’intérêt augmente. Les rapporteurs translationnels relient les cadres de lecture ouverts d’une protéine fluorescente et d’une protéine d’intérêt avec un connecteur peptidique flexible. La visualisation des souches rapporteures à l’aide de la microscopie à cellules vivantes peut révéler si un gène/protéine d’intérêt donné est associé à des morphologies cellulaires ou à des destins cellulaires spécifiques (Figure 1, Vidéo supplémentaire 1 et Vidéo supplémentaire 2). Choisissez une protéine fluorescente optimisée pour S. aureus en termes de codons. Alors que cette étude utilise la sGFP de pCM29 offerte par le Dr Alexander Horswill, la Nebraska Transposon Mutant Library Genetic Toolkit contient des plasmides portant des codons optimisés pour les codons sGFP, eYFP, eCFP, DsRed.T3 et eqFP65017,18.
- Transformer les plasmides rapporteurs en S. aureus RN4220.
REMARQUE : La section suivante du protocole a été adaptée de l’atelier Essentials of Staphylococcal Genetics and Metabolism au Centre médical de l’Université du Nebraska19,20.- Tout d’abord, préparez électrocompétent S. aureus RN4220. Préparez 200 ml de bouillon B2 en combinant 2 g d’acides casaminés, 5 g d’extrait de levure, 2 ml de 10 % de K2HPO4, pH 7,5, 2 ml de solution de glucose à 50 % et 5 g de NaCl. Porter le volume à 100 mL avec de l’eau stérile ultrapure. Mélanger brièvement à 37 °C pour dissoudre les composants et stériliser à l’aide d’un filtre sur le dessus du flacon (pores de 0,22 μm).
- Inocuculer S. aureus RN4220 à partir d’un produit congelé conservé à -80 °C dans du glycérol à 25 % dans 30 mL de bouillon B2 dans une fiole à chicanes de 250 mL et incuber pendant 18 h à 30 °C, en agitant à 125 tr/min.
- Incuber pendant la nuit deux flacons à chicanes de 250 ml avec 50 ml de bouillon B2 chacun à 37 °C afin de préchauffer le milieu pour le lendemain.
- Le lendemain, mesurez la DO600 de la culture de nuit. Ajouter suffisamment de culture pendant la nuit dans chaque flacon contenant 50 mL de bouillon B2 pour obtenir une DOfinale de 600 de 0,25.
- Incuber à 37 °C, en agitant à 250 tr/min, jusqu’à ce que la DO600 atteigne 0,35-0,4.
REMARQUE : Vérifiez l’OD600 environ 30 minutes après la sous-culture pour vous assurer que la culture ne dépasse pas 0,35-0,4. Si l’OD600 n’atteint pas 0,35-0,4 dans l’heure qui suit la sous-culture, abandonnez la procédure et réessayez le lendemain. Les bactéries se sont probablement trop développées pendant la nuit et étaient trop profondes dans la phase stationnaire. - Une fois que la culture a atteint la phase mi-logarithmique, verser la culture de chaque flacon dans un tube conique stérile de 50 mL et des cellules de granules par centrifugation à 4 000 x g pendant 10 min.
- Décantez le surnageant et remettez la pastille en suspension dans 1 mL d’eau stérile à température ambiante. Transférez les cellules dans un tube de microcentrifugation.
- Centrifugez à nouveau les cellules à 21 000 x g pendant 30 s, retirez le surnageant et mettez-les en suspension dans 1 mL d’eau RT stérile. Répéter la centrifugation, l’élimination du surnageant et la remise en suspension dans 1 mL d’eau deux fois de plus pour un total de trois lavages.
- Centrifugez les cellules, retirez l’eau, puis remettez les cellules en suspension dans 1 mL de glycérol stérile à 10 % (dans de l’eau, filtré stérilement) et laissez à RT pendant 15 min.
- Granulez les cellules par centrifugation à 21 000 x g pendant 30 s, retirez le surnageant et remettez les cellules en suspension dans 1 autre mL de glycérol stérile à 10 %. Ces cellules sont aujourd’hui électrocompétentes.
- Aliquote 70 μL de cellules électrocompétentes dans des tubes de microcentrifugation à bouchon encliquetable et stockez-les à -80 °C pour une utilisation future.
- Pour transformer le S. aureus RN4220 électrocompétent, décongelez une aliquote de cellules sur de la glace. Ajoutez 1 μg d’ADN plasmidique dans les cellules, mélangez l’échantillon et incubez sur de la glace pendant 5 min. Ensuite, électroporatez les cellules et laissez-les récupérer dans 390 μL de milieu B2 pendant 1 à 2 h à 37 °C, en agitant à 225 tr/min, et des transformants sur plaque sur gélose de soja tryptique sélective.
REMARQUE : La plupart des vecteurs navettes utilisés dans cette étude sont dotés d’une cassette de résistance à l’ampicilline pour la sélection chez E. coli (100 μg/mL) et d’une cassette de résistance au chloramphénicol (10 μg/mL) ou à l’érythromycine (Erm, 10 μg/mL) pour la sélection chez S. aureus. De nombreuses souches de S. aureus qui sont couramment utilisées en laboratoire sont résistantes à l’Erm. S’assurer qu’une souche d’intérêt est compatible avec l’antibiotique sélectif avant d’utiliser un vecteur navette donné. Il est important de minimiser le volume d’ADN ajouté aux cellules (c’est-à-dire <5 μL) pour éviter la formation d’arcs électriques, qui se produit lorsque l’échantillon est trop conducteur lors de l’électroporation. La purification de concentrations élevées (≥300 ng/μL) de l’ADN plasmidique d’E. coli aidera à maintenir les volumes bas.
- Propager le bactériophage sur transformé S. aureus RN4220.
REMARQUE : La section suivante du protocole a été adaptée des méthodes de Krausz et Bose21et Olson22. Deux des bactériophages les plus couramment utilisés pour S. aureus transduction sont φ11 et 80α. La procédure de récolte des deux est la même, sauf que nous obtenons φ11 à partir de S. aureus RN451 et 80α à partir de S. aureus RN10359. φ11 a été utilisé avec succès avec les souches courantes JE2 et HG003, entre autres, et est utilisé dans ce protocole23,24. Cependant, ces phages peuvent ne pas transduire efficacement les isolats cliniques qui ont des résistances aux phages variables. Reportez-vous aux publications de Krausz et Bose21 et Olson22 pour une méthode de préparation de φ11 et de détermination du titre de phage.- Pour transduire le plasmide rapporteur, préparez six plaques de gélose tryptique de soja (TSA) avec 5 mM de CaCl2 et la concentration appropriée de l’antibiotique sélectif. Streak S. aureus RN4220 s’est transformé avec le plasmide rapporteur sur une plaque TSA contenant l’antibiotique sélectif et pas de CaCl2 et a incubé pendant la nuit.
- Le lendemain, allumez un incubateur ou un bain-marie à 56 °C. Étiquetez six tubes à encliquetage de 5 mL 1 x 104 à 1 x 109, et aliquote 40 μL de 500 mM de CaCl2 dans chaque tube à encliquetage.
- Faites fondre du TSA mou (0,5 % d’agar-agar) dans un micro-ondes avec le capuchon desserré pour éviter l’accumulation de pression tout en maintenant la stérilité. Ensuite, aliquote 4 mL dans chacun des tubes à encliquetage. Retournez les tubes pour mélanger, puis placez-les dans l’incubateur à 56 °C pour éviter que la gélose molle ne se solidifie.
- Diluer le stock de phages à 1 x10 10 UFP/mL dans un bouillon de soja tryptique (BST) + 5 mM de CaCl2. Ajouter 100 μL de 1 x 1010 PFU/mL dans un tube de microcentrifugation contenant 900 μL de TSB + 5 mM de CaCl2 pour une concentration finale de 1 x 109 PFU/mL. Ensuite, diluer en série dans des tubes de microcentrifugation supplémentaires contenant 900 μL de TSB + 5 mM de CaCl2 jusqu’à une concentration de 1 x 104 PFU/mL. Étiquetez chacune des six plaques de gélose antibiotique sélective TSA + 5 mM CaCl2 + avec une dilution en série : 1 x 104 à 1 x 109 PFU/mL.
- Remettre en suspension le S. aureus RN4220 contenant le plasmide pendant la nuit dans 1 mL de TSB + 5 mM de CaCl2. Retirez le tube à bouchon pression 109 de l’incubateur et ajoutez rapidement 10 μL de RN4220 remis en suspension contenant le plasmide et 100 μL de dilution de phage 1 x 109 PFU/mL, retournez pour mélanger et versez le contenu sur la plaque de gélose antibiotique sélective TSA + 5 mM CaCl2 correspondante.
- Effectuez cette procédure pour les dilutions de phages restantes et leurs plaques respectives. Laissez la gélose molle se solidifier sur les plaques, puis incubez avec la gélose molle vers le haut pendant une nuit à 37 °C.
- Le lendemain, assurez-vous que les plaques (zones d’élimination sur la pelouse bactérienne) sont visibles sur les plaques (figure 2A). Les plaques représentent des zones de lyse où les phages ont réussi à infecter RN4220, et certains phages auront absorbé le plasmide d’intérêt. Sélectionnez la plaque avec le stock de phages le moins dilué qui a une lyse presque confluente (exemple : 1 x 109 PFU/mL de phage) ainsi que les plaques qui ont reçu les deux dilutions en dessous (dans ce cas, 1 x 108 PFU/mL et 1 x 107 PFU/mL, qui doivent toutes deux avoir une lyse presque confluente).
- Ajouter 3 mL de TSB + 5 mM de CaCl2 à la moins diluée de ces plaques (1 x 109 PFU/mL, dans cet exemple). À l’aide d’un épandeur de cellules stérile en forme de L, grattez doucement la couche de gélose molle de la gélose ordinaire (figure 2B). Utilisez l’épandeur de cellules pour perturber la gélose autant que possible afin de faciliter la libération du phage de la gélose molle.
- Une fois que la gélose molle de la première assiette a été complètement perturbée, versez doucement la bouillie de gélose molle et de bouillon dans la plaque suivante (dans ce cas, 1 x 108 PFU/mL). Grattez et perturbez la gélose molle sur cette plaque, puis versez la bouillie obtenue sur la dernière plaque et répétez.
- Après avoir préparé la bouillie de gélose molle à partir des trois plaques, versez doucement la bouillie dans un tube conique de 50 ml. Pipetez doucement pour perturber davantage l’agar-agar, mais évitez de former des bulles. Ne faites pas de vortex, car cela pourrait endommager le phage et diminuer le titre.
- Centrifuger la bouillie de gélose molle à 4 000 x g pendant 10 min à RT. Verser doucement le surnageant dans une seringue reliée à un filtre de 0,45 μm.
REMARQUE : Il est essentiel de verser doucement et d’éviter de mettre des morceaux de gélose dans le filtre. Ces pièces obstrueront le filtre et empêcheront de filtrer le surnageant restant, diminuant ainsi le titre. - Déterminer le titre du lysat de phage résultant à l’aide du type sauvage RN422021.
- Introduire des plasmides rapporteurs dans les souches de S. aureus d’intérêt par transduction.
- Préparez huit plaques TSA contenant 500 μg/mL de citrate de sodium et la concentration appropriée d’antibiotique sélectif. Striez densément la souche réceptrice souhaitée de S. aureus sur du TSA nature et incubez toute la nuit à 37 °C.
- Le lendemain, frottez la plaque et remettez en suspension la souche réceptrice dans 1 mL de TSB + 5 mM de CaCl2.
- Diluer le stock de phages portant le plasmide rapporteur souhaité à 1 x1010 PFU/mL.
- Mélanger 1,5 mL de TSB + 5 mM de CaCl2, 0,5 mL de souche réceptrice en suspension et 0,5 mL de stock de phages dilué dans un tube conique de 50 mL. Incuber à 37 °C, en secouant à 225 tr/min, pendant 20 min.
REMARQUE : Des temps d’incubation plus longs ne sont pas recommandés car ils diminueront probablement l’efficacité de la transduction lorsque le phage commence à lyser les cellules réceptrices. - Immédiatement après, ajouter 1 ml de citrate de sodium 0,02 M froid et filtré stérilement (dans de l’eau) dans le tube conique et centrifuger à 4 000 x g pendant 10 min à 4 °C. Ensuite, remettre la pastille en suspension dans 1 mL de citrate de sodium 0,02 M glacé.
- Étaler 100 μL d’aliquotes sur chacune des six plaques TSA contenant 500 μg/mL de citrate de sodium et la concentration appropriée d’antibiotique sélectif à sélectionner pour les transducteurs. Incuber toute la nuit à 37 °C.
- Le lendemain, striez les colonies individuelles des plaques de transduction sur les deux autres plaques TSA contenant 500 μg/mL de citrate de sodium et la concentration appropriée d’antibiotique sélectif. Incuber toute la nuit à 37 °C.
REMARQUE : Cette étape de passage sur des plaques de citrate de sodium supplémentaires aide à réduire le titre de phage autour des cellules transduites, empêchant ainsi la lyse22. - Prélevez des colonies individuelles dans la plaque striée, faites croître les cellules jusqu’à la phase exponentielle dans du TSB contenant l’antibiotique sélectif et stockez-les dans du glycérol à 25 % à -80 °C pour une utilisation future.
2. Génération de souches rapporteures fluorescentes de P. aeruginosa par conjugaison
REMARQUE : Le déplacement d’un plasmide rapporteur dans P. aeruginosa à partir d’une souche clonée d’E. coli peut se faire par accouplement triparental25. Une souche donneuse d’E. coli qui porte le plasmide d’intérêt (qui doit contenir un oriT pour le transfert conjugal), E . coli HB101 + pRK2013 - une souche auxiliaire pour faciliter la conjugaison (utiliser 50 μg/mL de kanamycine pour l’entretien du plasmide pRK2013) et la souche receveuse de P. aeruginosa qui recevra le plasmide sont nécessaires. Pour cet exemple, le plasmide d’intérêt a un marqueur de résistance à la tétracycline (Tet), de sorte que toute culture d’E. coli avec le plasmide aura besoin de 10 μg/mL de Tet, et P. aeruginosa avec le plasmide aura besoin de 75 μg/mL de Tet pour la sélection26.
- Inoculer des bactéries à partir de stocks congelés conservés à -80 °C dans du glycérol à 25 % dans un bouillon de lysogénie (LB) avec l’antibiotique approprié pour la croissance pendant la nuit, en agitant à 225 tr/min à 37 °C.
- Le lendemain, étalez 200 μL de chaque culture liquide pendant la nuit sur la plaque de gélose LB avec l’antibiotique sélectif approprié. En fonction du nombre d’accouplements de transformation à effectuer, étalez plus de plaques pour les souches auxiliaires et donneuses. Une plaque d’aide/donateur par transformation est recommandée. Incuber les plaques pendant la nuit à 37 °C pour obtenir des pelouses denses de chaque variété.
- Le lendemain, récupérez les pelouses en tamponnant chaque surface de plaque avec un coton-tige stérile et en l’essuyant à l’intérieur d’un tube de microcentrifugation. Centrifugez brièvement chaque tube (impulsion pendant 5 s) pour recueillir les cellules au fond du tube. Remettre en suspension chaque pastille de cellule dans 200 μL de LB.
REMARQUE : Les resuspensions doivent avoir l’air très épaisses, opaques et débarrassées des grumeaux. - Dans de nouveaux tubes de microcentrifugation pour chaque accouplement, combiner 5 μL de souche réceptrice de P. aeruginosa , 160 μL de souche d’E. coli donneuse portant le plasmide d’intérêt et 160 μL d’E. coli HB101 + pRK2013. Mélangez doucement, en pipetant de haut en bas, puis placez 50 μL du mélange sur une plaque de gélose LB pré-séchée. De plus, placez 20 μL de chaque souche dans la gélose LB pour les témoins. Laissez les taches sécher complètement dans la plaque, puis incubez à 37 °C pendant 3 à 6 h.
REMARQUE : Le pré-séchage de la gélose LB empêche la tache de 50 μL de s’étendre sur la surface et force ainsi les cellules à interagir dans une zone plus petite. - Après l’incubation, passez doucement la pointe d’une pipette sur le point d’accouplement pour transférer certaines cellules dans un nouveau tube de microcentrifugation. Centrifugez brièvement pour recueillir les cellules au fond du tube. Remettre en suspension dans 200 μL de LB et mettre en plaque la totalité de la remise en suspension dans une plaque de gélose LB + Irgasan (Igr ; 25 μg/mL) + Tet (75 μg/mL).
REMARQUE : P. aeruginosa est intrinsèquement résistant à l’Igrasan, et le plasmide d’intérêt contient le marqueur de résistance Tet, de sorte que seul P. aeruginosa transformé avec succès devrait pouvoir se développer sur les plaques LB + Irgasan + Tet. Selon l’efficacité de la transformation, la remise en suspension et le placage de l’ensemble du lieu d’accouplement peuvent entraîner une pelouse de transformants le lendemain. Pour obtenir des colonies individuelles, soit (i) remettre en suspension l’ensemble du point d’accouplement dans un plus grand volume de LB, puis une plaque de 200 μL de celui-ci, soit (ii) transférer une petite partie du point d’accouplement dans un tube de microcentrifugation, puis remettre en suspension dans 200 μL et une plaque. - Avec les points de contrôle de chaque souche individuelle, balayez doucement une partie du point cellulaire dense sur une pointe de pipette, transférez dans un nouveau tube de microcentrifugation, remettez en suspension dans 200 μL de LB, puis placez 50 μL sur une plaque de gélose LB + Irgasan + Tet (plusieurs points de contrôle peuvent être placés sur la même plaque de gélose). Laissez les taches sécher complètement. Incuber toutes les plaques pendant 16 à 20 h pendant la nuit à 37 °C.
REMARQUE : Pour les points témoins, l’objectif est de vérifier que la plaque d’antibiotique est sélective pour les transformants de P. aeruginosa . Si les taches de contrôle sont trop concentrées lorsqu’elles sont repérées sur les plaques de gélose LB + Irgasan + Tet, il sera difficile de déterminer si la sélection a fonctionné le lendemain car il y aura un halo de cellules mortes qui peuvent être confondues avec la croissance. - Le lendemain, confirmez la sélectivité antibiotique des plaques LB + Irgasan + Tet en notant l’absence de croissance cellulaire pour les points de contrôle de la souche individuelle.
REMARQUE : Les colonies qui apparaissent sur la plaque d’accouplement triparental LB + Igrasan + Tet sont des transformants de P. aeruginosa portant le plasmide d’intérêt. - Facultatif de cueillir et de strier les colonies sur LB + Irgasan + gélose Tet et de les cultiver pendant la nuit pour se débarrasser de l’excès d’E. coli mort du mélange d’accouplement.
- Cueillir des colonies individuelles, les faire croître pendant 3 à 6 h dans du LB + Tet liquide (75 μg/mL) jusqu’à ce qu’elles soient visiblement troubles, puis stocker des stocks congelés dans 25 % de glycérol (500 μL de culture cellulaire + 500 μL de glycérol à 50 % dans de l’eau). Vérifiez que les clones sont transformés avec succès par PCR de colonie suivie d’un séquençage de Sanger ou d’un séquençage du plasmide entier.
3. Détermination des doses d’antibiotiques pour les tests persistants
REMARQUE : Pour sélectionner une dose d’un antibiotique donnée avec laquelle traiter la population bactérienne pour les expériences persistantes, mesurez d’abord la concentration minimale inhibitrice (CMI) de l’antibiotique par rapport à la souche bactérienne d’intérêt. Cela peut être réalisé en utilisant soit la méthode de microdilution en bouillon – une approche approuvée par le Clinical and Laboratory Standards Institute (CLSI) – soit le test Epsilometer (E-test), qui est effectué à l’aide de bandelettes réactives avec une gamme de doses d’antibiotiques27. Une fois la CMI déterminée, choisissez au moins cinq concentrations de l’antibiotique allant de 1 à 100 fois la CMI pour le traitement cellulaire.
- Inoculez des bactéries à partir de stocks congelés conservés à -80 °C dans du glycérol à 25 % dans 2 mL de MHB AJUSTÉ AUX CATIONS (CA-MHB) ou un autre milieu riche en nutriments. Faire croître les cellules pendant environ 4 h à 37 °C en les agitant à 250 tr/min avant de transférer 125 μL de la culture dans 25 mL de milieu frais chimiquement défini dans un erlenmeyer à chicanes de 250 mL. Cultivez les bactéries pendant 16 h (jusqu’à la phase stationnaire) à 37 °C, en secouant à 250 tr/min.
REMARQUE : Les milieux salins de base (BSM) avec le succinate comme seule source de carbone sont généralement utilisés pour les expériences sur P. aeruginosa et les milieux riches chimiquement définis sont généralement utilisés pour les expériences sur S. aureus 28,29,30,31. - Le lendemain matin, préparez 100 stocks d’antibiotiques (dans les solvants respectifs) pour la plage souhaitée de concentrations supra-CMI. Ajouter 10 μL de chaque dilution dans des tubes à essai individuels.
- Mesurez la DO600 de la culture de nuit pour confirmer la turbidité en phase stationnaire. Diluez en série 10 μL de culture et placez les dilutions sur des plaques de gélose nutritive, telles que la gélose LB, la gélose CA-MHB ou la TSA, afin de déterminer les unités formant colonies (UFC) avant le traitement antibiotique.
- Pour le traitement antibiotique, verser 1 mL d’aliquotes de la culture dans les tubes à essai contenant 10 μL de concentrations d’antibiotiques 100x. Incuber les échantillons à 37 °C, en les agitant à 250 tr/min, pendant une durée suffisante pour tuer les non-persistants dans la population, laissant les persistants comme seules cellules formant des colonies.
REMARQUE : La durée du traitement peut varier en fonction de l’expérience et de la souche bactérienne testée. Généralement, S. aureus est traité pendant 5 h ou 7 h et P. aeruginosa pendant 7 h ou 24 h. En général, n’importe quel point temporel de la deuxième phase de l’essai de survie dépendant du temps peut être utilisé, car on s’attend à ce que les cellules restantes dans la deuxième phase soient persistantes. - Après le traitement antibiotique, transférez 100 μL de cellules des tubes à essai dans des tubes de microcentrifugation contenant 900 μL de solution saline tamponnée au phosphate (PBS) stérile. Granuler les cellules par centrifugation à RT à 21 000 x g pendant 3 min. Retirer 900 μL de surnageant et remettre la pastille en suspension dans 900 μL de PBS stérile. Répétez l’étape de lavage au moins une fois de plus pour réduire les antibiotiques résiduels à des niveaux inférieurs à la CMI.
- Diluer la culture 10 fois (six fois) en série et déposer 10 μL de chaque dilution sur des plaques de gélose nutritive. Incuber les plaques à 37 °C pendant la nuit.
REMARQUE : Utilisez des plaques non traitées, à fond rond, à 96 puits pour la dilution en série et des cellules sur plaques sur des plaques de gélose carrées à l’aide d’une pipette multicanaux. - Le lendemain, comptez les colonies à chaque concentration d’antibiotique. Tracer la fraction de survie (UFC à la fin du traitement/UFC avant le traitement) en fonction de la concentration sur une échelle log-linéaire. Pour choisir une concentration de médicament pour les futurs essais persistants, sélectionnez une concentration dans la deuxième phase de la courbe biphasique (Figure 3).
4. Imagerie des cellules pendant le traitement antibiotique ou la récupération
- Préparer des tampons d’agarose et des échantillons de cellules pour l’imagerie en accéléré
REMARQUE : Le protocole de préparation d’échantillons suivant a été mis au point comme une solution de rechange conviviale et rentable aux méthodes traditionnelles d’agarose « sandwich »14,16. L’utilisation d’un plat interchangeable évite d’avoir à utiliser de la graisse ou du vernis à ongles pour sceller l’échantillon, ce qui pourrait limiter davantage l’aération de l’échantillon. L’agarose est placée dans le plat interchangeable avec la lamelle déjà en place, créant ainsi des surfaces d’agarose planes fiables par rapport aux préparations de tampons d’agarose à main levée. Dans l’ensemble, cette méthode a permis d’imager des échantillons avec une meilleure mise au point dans le champ de vision et pendant de plus longues durées en raison d’une humidification stable. La méthode décrite ici est utilisée pour visualiser les bactéries pendant le traitement antibiotique (Graphique 4, Vidéo supplémentaire 3et Vidéo supplémentaire 4). Pour visualiser les personnes persistantes lorsqu’elles se réaniment et se réveillent après un traitement antibiotique (Graphique 5, Vidéo supplémentaire 5et Vidéo supplémentaire 6), préparez les tampons d’agarose avec des milieux de culture frais au lieu de milieux épuisés et n’ajoutez pas d’antibiotiques aux tampons (sauf ceux ajoutés pour l’entretien des plasmides ou la sélection des antibiotiques).- Placez une lamelle stérile de 30 mm (épaisseur #1,5) dans le fond de la base en acier inoxydable d’une coupelle à lamelles interchangeables (« chambre »). Enfilez doucement l’insert en polycarbonate dans la base de sorte que la lamelle de 30 mm forme la base de la chambre, et scellez-le en place en comprimant le joint torique en silicone attaché. Répétez cette étape pour préparer une chambre dupliquée.
REMARQUE : Il est recommandé de préparer des duplicatas pour chaque expérience au cas où la préparation de l’une des chambres ne serait pas optimale. Un séparateur personnalisé imprimé en 3D est placé contre la lamelle de 30 mm pour empêcher les cellules mobiles comme P. aeruginosa de se contaminer et pour fournir des points de repère pour les emplacements des échantillons. Le fichier STL pour l’impression 3D est disponible dans le fichier supplémentaire 1. - Préparez de l’agarose à 1,5 % dans un tube conique de 50 mL en utilisant le milieu de votre choix. Agitez doucement pour mélanger.
REMARQUE : Pour l’imagerie de cellules en phase stationnaire pendant le traitement, on utilise comme milieu de base un milieu épuisé non acellulaire provenant d’une culture de nuit en phase stationnaire. En règle générale, l’agarose est préparée sous forme de 0,105 g d’agarose dans 7 mL de milieu pour préparer deux chambres ; chaque chambre nécessite 2 ml de milieu d’agarose, et l’excès est utile pour éviter les bulles d’air lors du pipetage. - Placez le cône de 50 ml dans un bécher en verre ou un support allant au micro-ondes (assurez-vous que le capuchon est desserré). Cuire au micro-ondes à puissance élevée, en s’arrêtant toutes les 3-4 s pour tourbillonner et mélanger. Faites une pause et mélangez souvent pour éviter que le mélange d’agarose ne bouillonne.
- Après ~1 min de chauffage total, vérifiez s’il n’y a pas d’agarose visible qui n’a pas fondu dans le mélange. Si le mélange est uniformément clair, laissez le milieu d’agarose refroidir brièvement à ~60 °C avant d’ajouter des médicaments ou des colorants supplémentaires. Agitez doucement pour mélanger et éviter la formation de bulles d’air.
REMARQUE : De l’iodure de propidium (16 μM pour P. aeruginosa ou 1,6 μM pour S. aureus) peut être ajouté pour visualiser quand les cellules perdent leur viabilité.
- Après ~1 min de chauffage total, vérifiez s’il n’y a pas d’agarose visible qui n’a pas fondu dans le mélange. Si le mélange est uniformément clair, laissez le milieu d’agarose refroidir brièvement à ~60 °C avant d’ajouter des médicaments ou des colorants supplémentaires. Agitez doucement pour mélanger et éviter la formation de bulles d’air.
- En se déplaçant rapidement pour éviter que le milieu d’agarose ne se solidifie, pipetez 2 ml de milieu d’agarose dans la chambre avec la lamelle de 30 mm. Posez délicatement une lamelle stérile de 25 mm (épaisseur #1,5) sur le milieu d’agarose dans l’ouverture de la chambre supérieure ; Cela permet d’éviter la déshydratation du tampon d’agarose et garantit une surface supérieure plane de l’agarose pour une imagerie optimale à contraste de phase. Laissez le tampon se solidifier pendant 1 à 2 h.
- Une fois que le tampon s’est solidifié, préparez les cellules à imager en les diluant à une densité clairsemée appropriée dans le PBS pour visualiser les cellules individuelles. Ici, les cellules sont diluées àOD 600 0,01-0,05.
- Sur la lamelle de 25 mm, utiliser un marqueur permanent à pointe fine pour marquer l’emplacement et l’identité de chaque échantillon ensemencé sur le tampon. Retournez la chambre de manière à ce que l’anneau de base en acier inoxydable soit orienté vers le haut et tenez soigneusement l’insert en polycarbonate en dessous tout en dévissant la base. Placez la base en acier inoxydable sur le côté. Faites glisser avec précaution la lamelle de 30 mm du tampon et jetez-la.
REMARQUE : Retirez la lamelle de 30 mm en la faisant glisser horizontalement et veillez à ne pas indenter la surface de l’agarose. Évitez également de travailler sur la surface exposée de l’agarose : toute poussière qui tombe dessus affectera la qualité de l’image et peut potentiellement contaminer le tampon. - À l’aide des marques sur la lamelle de 25 mm à titre indicatif, repérez 5 μL des cellules diluées aux endroits respectifs sur le tampon d’agarose. Ajoutez trois spots de 5 μL (ajustez cela si nécessaire, par exemple, 4 spots de 4 μL, etc.). Une fois que les taches sont complètement sèches, placez légèrement une nouvelle lamelle stérile de 30 mm centrée sur le tampon.
- Tenez l’insert en polycarbonate d’une main et, de l’autre, réenfilez lentement la base en acier inoxydable sur la nouvelle lamelle. L’enfilage jusqu’à ce qu’il soit juste serré à la main suffit à comprimer le joint torique en silicone et à sceller la chambre.
REMARQUE : Veillez à ne pas trop serrer - si la lamelle de 30 mm contre la surface de l’échantillon est comprimée par la base en acier inoxydable et tordue à plusieurs reprises, les cellules peuvent s’étaler selon un motif radial, provoquant potentiellement une contamination croisée de l’échantillon. - Une fois la chambre scellée, observez si l’agarose entre en contact avec la surface de la lamelle de 30 mm ; À ce stade, il y a généralement un certain contact de surface, mais pas complet. À l’aide de l’extrémité émoussée d’une pince à épiler ou d’un instrument similaire, appuyez doucement contre la lamelle de 25 mm jusqu’à ce que l’agarose touche uniformément la lamelle de 30 mm sur toute la surface.
ATTENTION : Cette étape peut entraîner la rupture de la lamelle de 25 mm si une force excessive est utilisée. - Une fois que l’agarose est bien pressée contre la lamelle de 30 mm de sorte qu’il ne reste plus de grosses bulles d’air, le tampon est prêt à être imagé.
- Placez une lamelle stérile de 30 mm (épaisseur #1,5) dans le fond de la base en acier inoxydable d’une coupelle à lamelles interchangeables (« chambre »). Enfilez doucement l’insert en polycarbonate dans la base de sorte que la lamelle de 30 mm forme la base de la chambre, et scellez-le en place en comprimant le joint torique en silicone attaché. Répétez cette étape pour préparer une chambre dupliquée.
- Installez le microscope et l’image.
- Préparez les contrôles environnementaux du microscope et de la chambre d’imagerie. Réglez la température de l’incubateur sur scène et de l’incubateur à grande chambre à 37 °C et allumez l’humidificateur à chambre. Placez l’une des préparations de tampons d’agarose dans l’incubateur sur le dessus de la scène et fermez la chambre d’incubation pour permettre à l’échantillon de s’équilibrer. Le couvercle d’humidification de l’incubateur est laissé ouvert à ce stade pour éviter la condensation sur la lamelle de 25 mm de la chambre.
REMARQUE : Le rétrécissement/l’expansion du tampon dû aux changements de température peut provoquer une dérive entre les points temporels qui dépassent la plage de balayage de l’algorithme de mise au point automatique. Il est crucial d’équilibrer le tampon d’agarose à une température stable avant l’imagerie, ce qui prend généralement au moins 15 minutes. Un réchauffement suffisant empêche également la condensation sur la lamelle supérieure lorsque le couvercle d’humidification est mis en place. Les composants matériels du système d’imagerie étaient (Table des matériaux) : chambre d’incubation de cellules vivantes, microscope inversé avec un objectif Plan-Apochromat 63x/1.40 Oil Ph3 M27, moteur de lumière LED Spectra 7 et caméra sCMOS (taille de pixel de 6,5 μm). Le contrôle des composants motorisés du microscope et l’acquisition d’images se font à l’aide d’un logiciel d’analyse de microscopie. - Préparez le logiciel pour l’acquisition d’images. Réglez l’algorithme de mise au point automatique intégré de MetaMorph (logiciel d’analyse de microscopie) pour utiliser le canal de phase lors de l’acquisition multidimensionnelle.
- Dans l’onglet Étape , définissez plusieurs positions de scène pour chaque échantillon, en visant les champs de vision où la densité cellulaire est uniformément répartie. Dans l’onglet Timelapse , définissez la durée et la fréquence souhaitées (intervalle de temps) des images à prendre. Dans les onglets Longueurs d’onde , définissez les voies souhaitées pour l’acquisition et ajustez les temps d’exposition en fonction de l’intensité du signal des échantillons.
REMARQUE : Les images de chaque position sont prises toutes les 10 minutes pendant un maximum de 24 heures. Pour l’imagerie par fluorescence des colorants/fluorophores mentionnés dans cet article, les paramètres d’excitation du moteur de lumière suivants ont été utilisés : iodure de propidium (Cy3 ; 555/15 nm) et GFP (GFP/FITC ; 470/24 nm). Le temps d’exposition typique utilisé pour l’excitation de fluorescence est de 100 ms. Le jeu de filtres 15 (Beamsplitter FT580, Emission LP590) pour l’iodure de propidium et le jeu de filtres 44 (Beamsplitter FT500, Emission BP 530/50) pour GFP sont utilisés.
- Dans l’onglet Étape , définissez plusieurs positions de scène pour chaque échantillon, en visant les champs de vision où la densité cellulaire est uniformément répartie. Dans l’onglet Timelapse , définissez la durée et la fréquence souhaitées (intervalle de temps) des images à prendre. Dans les onglets Longueurs d’onde , définissez les voies souhaitées pour l’acquisition et ajustez les temps d’exposition en fonction de l’intensité du signal des échantillons.
- Une fois que le tampon d’agarose s’est réchauffé dans la chambre, ajoutez le couvercle d’humidification dans l’incubateur de la scène. Cela aidera à empêcher le tampon de se déshydrater et de se déplacer pendant l’imagerie.
- Avec le couvercle humidificateur ouvert, passez du contraste interférentiel différentiel (DIC) à la phase et ajustez le condenseur et le diaphragme d’ouverture du microscope pour un éclairage Köhler approprié.
- Après l’ajustement, revenez en phase. Naviguez jusqu’à chaque position de la scène, ajustez la mise au point et réinitialisez la position de la scène sur le nouveau plan focal. Les paramètres sont maintenant en place pour commencer l’imagerie.
REMARQUE : Il est important de revoir chaque position de l’étape avant de commencer l’acquisition pour s’assurer que le tampon n’a pas bougé. Si les cellules ne sont pas nettes lorsqu’elles sont revisitées à une position d’étape donnée, concentrez-vous à nouveau sur elles et remplacez la position d’étape précédente. Le réglage de l’algorithme de mise au point automatique utilise la position z focale du dernier point temporel ± 3 μm comme plage de recherche, donc si le pad se décale de plus de 3 μm, l’algorithme ne parviendra pas à faire la mise au point correctement. - Lancez l’acquisition multidimensionnelle en cliquant sur le bouton Acquérir .
- Une fois l’expérience terminée, compilez les images individuelles de chaque canal dans une pile pour les visionner sous forme de vidéo ou pour d’autres analyses. Pour ce faire, utilisez MetaMorph ou ImageJ.
- Pour compiler des images dans MetaMorph (de préférence), dans l’application Review Multi Dimensional Data , sélectionnez les canaux/longueurs d’onde qui vous intéressent et sélectionnez tous les points temporels pour une position de scène donnée. Cliquez sur Charger les images. Des fenêtres apparaîtront pour chaque canal/longueur d’onde. Enregistrez chaque compilation avec son nom de canal respectif dans un fichier .tiff.
- Pour compiler dans ImageJ, ouvrez les fichiers pour tous les points temporels d’une position de scène et d’un canal. Compilez à l’aide d’images à empiler.
- Préparez les contrôles environnementaux du microscope et de la chambre d’imagerie. Réglez la température de l’incubateur sur scène et de l’incubateur à grande chambre à 37 °C et allumez l’humidificateur à chambre. Placez l’une des préparations de tampons d’agarose dans l’incubateur sur le dessus de la scène et fermez la chambre d’incubation pour permettre à l’échantillon de s’équilibrer. Le couvercle d’humidification de l’incubateur est laissé ouvert à ce stade pour éviter la condensation sur la lamelle de 25 mm de la chambre.
5. Création de vidéos time-lapse à l’aide de Fiji/ImageJ
REMARQUE : Fiji (Fiji est simplement ImageJ) est un logiciel de traitement et d’analyse d’images disponible gratuitement qui peut être téléchargé ici : « https://imagej.net/software/fiji/downloads32"32. Fiji/ImageJ2 1.54f a été utilisé pour les méthodes de traitement d’image décrites ci-dessous.
- Correction de l’ombrage de la pile d’images de canal de phase à l’aide de BaSiC33. Ouvrez la pile d’images de phase souhaitée dans Fidji, puis sélectionnez BaSiC dans l’onglet Plugins. La pile corrigée apparaîtra dans une fenêtre séparée intitulée Corrigé : NomImage.
REMARQUE : BaSiC.jar peut être téléchargé ici : « https://github.com/marrlab/BaSiC33"33. Suivez les instructions du développeur pour installer correctement BaSiC dans Fidji. - Fusionnez l’empilement de couches de phase corrigé de l’ombre avec d’autres couches/longueurs d’onde à l’aide des options Image > Couleur > Fusion des couches. Ajustez l’arrière-plan et l’intensité du signal avec Image > Ajustez > luminosité/contraste pour chaque canal. Enregistrez le fichier fusionné en tant que .tiff.
- Ensuite, stabilisez les images à l’aide de Correct 3D Drift. Une fois la pile corrigée sélectionnée, allez dans Plugins > Registrations > Corriger la dérive 3D. Dans la fenêtre de dialogue qui s’ouvre, définissez le canal d’enregistrement sur le numéro de canal correspondant à la pile de phases. La pile résultante après correction sera intitulée points de temps enregistrés. Recadrez jusqu’au champ de vision souhaité, puis enregistrez le fichier corrigé en tant que .tiff.
REMARQUE : Si MetaMorph a été utilisé pour créer la pile d’images compilée, il y a une étape de traitement supplémentaire : allez dans les propriétés de l’image > et échangez les numéros dans les champs Tranches (z) et Cadres (t). La correction de la dérive peut désormais interpréter correctement chaque image comme un point temporel et non comme une tranche d’une pile z. Le fichier .tiff corrigé des nuances et de la dérive peut être utilisé dans les analyses d’images ultérieures. Il existe de nombreux logiciels pour mesurer l’intensité des signaux fluorescents, quantifier les caractéristiques morphologiques, suivre le destin des cellules individuelles, etc. Deux programmes couramment utilisés sont le plug-in MicrobeJ pour Fidji et Oufti34,35. - Ajoutez des horodatages et des étiquettes de texte sur la pile à l’aide des piles d’images > > des étiquettes.
- Pour ajouter une barre d’échelle, vous devez d’abord connaître la taille en pixels de l’appareil photo et le grossissement de l’objectif du microscope. Calculez le rapport pixel/micron en divisant la taille du pixel par le grossissement. Dans la boîte de dialogue Analyser > Définir l’échelle , entrez le rapport pixel/micron dans le champ de distance connue . Ensuite, ajoutez une barre d’échelle à la pile à l’aide des outils d’analyse > > de la barre d’échelle.
REMARQUE : L’échelle précise d’un microscope doit être calibrée à l’aide d’un micromètre à platine. Cependant, l’objectif du microscope et la caméra utilisés peuvent fournir une estimation approximative de l’échelle. Par exemple, la caméra PCO sCMOS utilisée ici a une taille de pixel de 6,5 x 6,5 μm2, et l’objectif 63x a été utilisé pour l’imagerie, de sorte que la taille de pixel divisée par le grossissement est de 6,5/63 = 0,1032 μm par pixel. Entrez 0,1032 dans le champ de distance connue de la boîte de dialogue Définir l’échelle . - Pour exporter la pile d’images terminée sous forme de vidéo pouvant être lue dans QuickTime Player, Microsoft PowerPoint, etc., enregistrez-la en tant que fichier .avi.
Résultats
L’introduction réussie de plasmides rapporteurs dans P. aeruginosa et S. aureus est indiquée par la croissance des antibiotiques sélectifs appropriés et peut être confirmée par PCR de colonie et/ou séquençage. Les souches modifiées doivent être vérifiées en tant que rapporteurs phénotypiques en les soumettant à des conditions dans lesquelles le gène d’intérêt est connu pour être induit, et la fluorescence résultante peut être mesurée par cytométrie en flux, spectrophotométrie ou microscopie à épifluorescence (figure 1).
Pour faciliter la sélection d’une ou plusieurs doses d’antibiotiques qui seront utilisées pour des expériences ultérieures, effectuer des dosages d’antibiotiques persistants dépendants de la concentration pour les souches d’intérêt de P. aeruginosa ou de S. aureus . Les essais dépendant de la concentration entraînent généralement une courbe biphasique avec une pente initiale abrupte à des concentrations d’antibiotiques plus faibles et un plateau ou une pente moins abrupte à des concentrations plus élevées. Cependant, pour certaines paires d’espèces d’antibiotiques, il peut ne pas en résulter une courbe biphasique distincte. Par exemple, la courbe de S. aureus delafloxacine est clairement biphasique (Figure 3A), mais la courbe de P . aeruginosa levofloxacine ne l’est pas (Figure 3B)15. Dans ce scénario, nous choisirions une concentration qui est au moins 10 fois la CMI (p. ex. 5 μg/mL, soit environ 15 fois la CMI de P. aeruginosa)15. Cependant, étant donné que la CMI de lévofloxacine 15x ne donne que ~0,001 % de survivants à P. aeruginosa , nous utilisons un traitement à la lévofloxacine de 1 μg/mL si nous voulons voir des cellules persistantes lors de l’imagerie des cellules lorsqu’elles se rétablissent sur des coussinets d’agarose sans antibiotiques (vidéo supplémentaire 6) ; Sinon, le nombre de champs de vision nécessaires pour imager plusieurs persisteurs devient prohibitif.
Au début de l’imagerie, la préparation idéale de l’échantillon et du tampon d’agarose doit apparaître plane dans tout le champ de vision, exempte de gros débris, de rides ou de bulles d’air, et avec des cellules uniques uniformément réparties. L’obtention de cellules uniques bien distribuées peut nécessiter une optimisation de la dilution ou de la remise en suspension de l’échantillon. Dans le cas de S. aureus, les cellules ont tendance à former de petits amas et doivent être soigneusement tourbillonnées avant d’être ensemencées dans le tampon d’agarose (figures 4 et 5). Pour P. aeruginosa, les cellules peuvent former des agrégats enfermés dans une matrice extracellulaire collante en suspension ; Il est nécessaire de pipeter soigneusement ces échantillons et de perturber les agrégats pour l’imagerie des cellules individuelles.
Après la conclusion d’une expérience d’imagerie, un time-lapse réussi d’images apparaîtra nette, éclairé de manière stable et avec une dérive minimale dans le plan x-y tout au long de l’expérience. La vidéo supplémentaire 7 représente une acquisition d’image optimale : t est le canal de phase de la vidéo supplémentaire 4 avant la correction de l’ombre ou de la dérive. Une perte de mise au point peut se produire si la condensation (due à une surhumidification ou à un réchauffement insuffisant de l’échantillon) provoque la formation de gouttelettes d’eau sur la lamelle supérieure de 25 mm, déformant la lumière et poussant le plan focal en dehors de la plage de recherche maximale de l’algorithme de mise au point automatique (Vidéo supplémentaire 8). La variation de l’éclairage indique généralement une huile d’immersion insuffisante au moment de l’imagerie. Si la platine se déplace trop rapidement, l’huile sur l’objectif peut traîner derrière et rattraper son retard lorsque les images sont acquises. Cela peut être atténué en ajustant les commandes d’acquisition pour ralentir la vitesse du mouvement ou en ajoutant une pause entre le mouvement vers la position suivante et l’acquisition de l’image. La dérive majeure de l’échantillon se présentera comme si de nombreuses cellules traversaient le champ de vision tandis que certaines restaient en place (vidéo supplémentaire 9). Cela se produit généralement plus tard dans les expériences, car le tampon d’agarose s’est déshydraté en raison d’un contrôle insuffisant de l’humidité. La préparation du tampon d’agarose présentée dans cet article a été conçue pour faciliter la stabilité de l’échantillon, mais il est nécessaire de réchauffer/humidifier correctement l’échantillon et son environnement pour une acquisition d’image optimale.
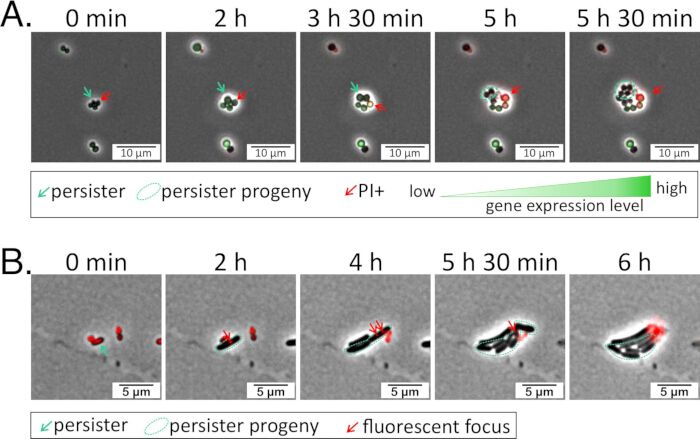
Figure 1 : Les souches rapporteures fluorescentes éclairent l’expression d’un gène d’intérêt. (A) S. aureus a été transduit avec un rapporteur transcriptionnel GFP pour un gène d’intérêt selon le protocole 1. La souche rapporteure a été traitée pendant 24 heures avec un antibiotique, lavée avec du PBS, puis ensemencée sur un tampon d’agarose fabriqué à partir de CA-MHB plus de l’iodure de propidium (1,6 μM) et du chloramphénicol (10 μg/mL pour l’entretien du plasmide rapporteur) pour l’imagerie pendant la récupération (vidéo supplémentaire 1). (B) P. aeruginosa a été transformé avec un plasmide portant un rapporteur translationnel lié à mScarlet pour une protéine d’intérêt26. La souche rapporteure a été traitée pendant 5 h avec un antibiotique, lavée avec du PBS, puis ensemencée sur un tampon d’agarose fabriqué à partir de BSM plus Tet (75 μg/mL ; pour l’entretien du plasmide rapporteur) à des fins d’imagerie pendant la récupération (vidéo supplémentaire 2). Veuillez cliquer ici pour voir une version agrandie de cette figure.
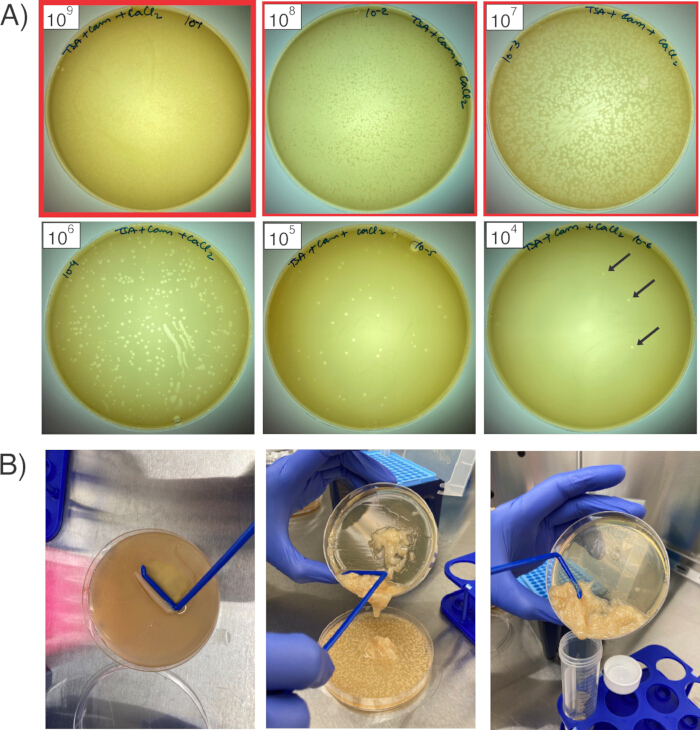
Figure 2 : Multiplication et récolte du bactériophage. (A) Les six planches montrent six quantités différentes de stock de phages dilués sur les pelouses de S. aureus RN4220. Les contours rouges indiquent les trois plaques qui seraient récoltées, de la plaque la plus claire (contour rouge gras ; 1 x 109 UFP/mL) aux deux dilutions suivantes (1 x 108 et 1 x 107 UFP/mL). Les flèches noires pointent vers des plaques individuelles. (B) Pour récolter le phage des plaques, grattez la couche de gélose molle (à gauche), transférez la boue sur la plaque de dilution suivante (au centre) et, après avoir regroupé la gélose molle des trois plaques ensemble, combinez-la en un tube conique pour la centrifugation (à droite). Veuillez cliquer ici pour voir une version agrandie de cette figure.
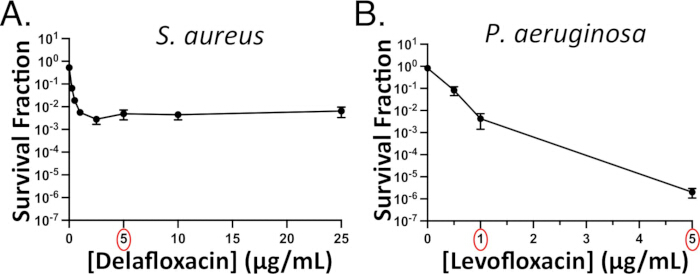
Figure 3 : Dosages représentatifs de persistance dépendant de la concentration. La persistance des fluoroquinolones dépendante de la concentration a été évaluée chez les bactéries stationnaires (A) S. aureus (contre la délafloxacine) et (B) P. aeruginosa (contre la lévofloxacine). Des expériences subséquentes utilisent 5 μg/mL de délafloxacine (cercle rouge) parce que la destruction de S. aureus avait atteint un plateau à cette concentration. Une dose d’au moins 1 μg/mL de lévofloxacine (cercle rouge) serait utilisée pour les expériences subséquentes avec P. aeruginosa. Il convient de noter que la destruction bactérienne ne plafonne pas chez P. aeruginosa, mais qu’il existe tout de même une « deuxième phase » moins abrupte de la courbe biphasique qui indique une sous-population persistante. Le panneau 3B a été adapté avec la permission de Hare et al.15. Veuillez cliquer ici pour voir une version agrandie de cette figure.
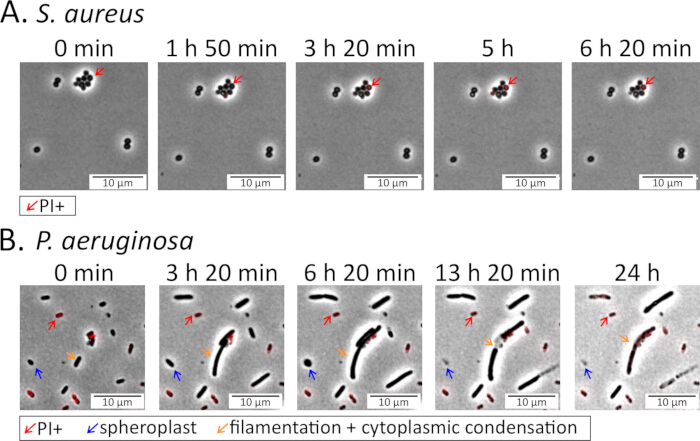
Figure 4 : Imagerie des phénotypes bactériens pendant le traitement antibiotique. Des cellules de S. aureus et de P. aeruginosa en phase stationnaire (A) et (B) ont été ensemencées sur des tampons d’agarose contenant des antibiotiques fluoroquinolones et surveillées pendant le traitement : 5 μg/mL de délafloxacine pour S. aureus (Vidéo supplémentaire 3) et 5 μg/mL de lévofloxacine pour P. aeruginosa (Vidéo supplémentaire 4)15. De l’iodure de propidium (PI ; 16 μM pour P. aeruginosa, 1,6 μM pour S. aureus) a été ajouté aux coussinets pour marquer les cellules mortes ou mourantes. Les cellules de S. aureus restent en grande partie intactes et vivantes en présence du FQ, tandis que la plupart des cellules de P. aeruginosa subissent des changements morphologiques drastiques, y compris la formation de sphéroplastes ronds, avant de se lyser et de mourir. Veuillez cliquer ici pour voir une version agrandie de cette figure.
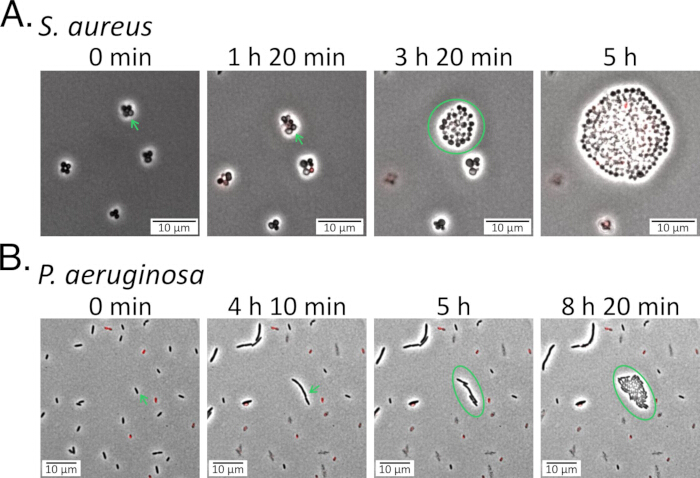
Figure 5 : Suivi des populations persistantes pendant le rétablissement. (A) Les populations de S. aureus et (B) de P. aeruginosa ont été ensemencées sur des tampons d’agarose contenant des milieux frais après avoir été traitées avec des fluoroquinolones (5 μg/mL de délafloxacine pour S. aureus et 1 μg/mL de lévofloxacine pour P. aeruginosa) et surveillées pendant leur rétablissement après le traitement (Vidéo supplémentaire 5 et Vidéo supplémentaire 6). Les périssœurs observées sont indiquées par des flèches vertes dans les deux premiers cadres de chaque panneau, et elles sont restées intactes et viables pendant le traitement antibiotique. Après une période de latence initiale, les persistants ont commencé à se diviser et ont donné naissance à une nouvelle progéniture (indiquée par des cercles verts). Veuillez cliquer ici pour voir une version agrandie de cette figure.
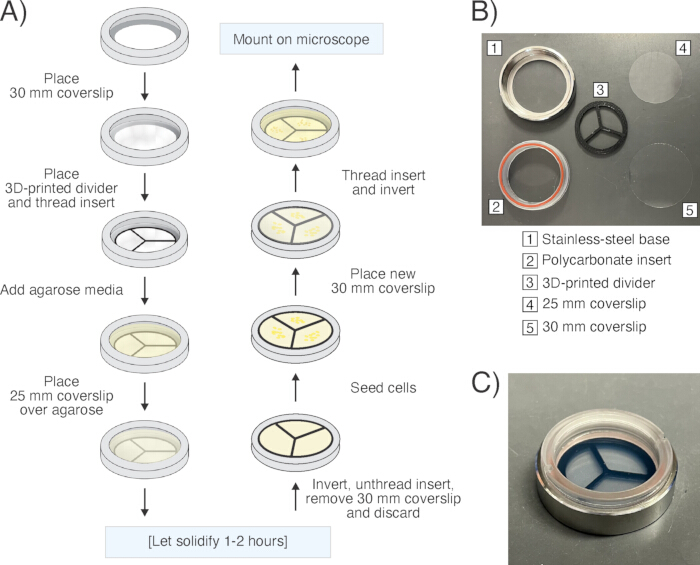
Figure 6 : Préparation d’un échantillon au microscope. (A) Schéma du flux de préparation des échantillons à l’aide d’une boîte de lamelle interchangeable (« chambre »). (B) Photo de la chambre démontée et de ses composants individuels. (C) Photo de la chambre entièrement assemblée. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Vidéo supplémentaire 1 : S. aureus persister. Fichier vidéo contenant les images de la figure 1A. En bref, S. aureus portant un rapporteur transcriptionnel GFP pour un gène d’intérêt a été traité pendant 24 heures avec un antibiotique, lavé avec du PBS, puis ensemencé sur un tampon d’agarose fabriqué avec du CA-MHB plus de l’iodure de propidium (1,6 μM) et du chloramphénicol (10 μg/mL) pour l’imagerie pendant la récupération. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire 2 : P. aeruginosa persistant. Fichier vidéo contenant les images de la figure 1B. En bref, P. aeruginosa portant un rapporteur translationnel lié à mScarlet pour une protéine d’intérêt a été traité pendant 5 h avec un antibiotique, lavé avec du PBS, puis ensemencé sur un tampon d’agarose fabriqué avec BSM plus Tet (75 μg/mL) pour l’imagerie pendant la récupération26. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire 3 : S. aureus pendant le traitement antibiotique. Une culture stationnaire de S. aureus cultivée dans un milieu riche chimiquement défini a été ensemencée dans des coussinets d’agarose fabriqués à partir du milieu conditionné sans cellules de la culture avec de l’iodure de propidium (1,6 μM) et de la délafloxacine (5 μg/mL). Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire 4 : P. aeruginosa pendant le traitement antibiotique. Une culture en phase stationnaire de P. aeruginosa cultivée dans des MSB a été ensemencée sur des coussinets d’agarose fabriqués à partir de milieux conditionnés sans cellules à partir d’une culture de P. aeruginosa cultivée dans des MSB en parallèle ; le tampon d’agarose contenait également de l’iodure de propidium (16 μM) et de la lévofloxacine (5 μg/mL). Cette vidéo a été adaptée avec la permission de Hare et al.15. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire 5 : S. aureus pendant la récupération post-antibiotique. S. aureus a été cultivé jusqu’à la phase stationnaire dans des milieux riches chimiquement définis. Les cultures en phase stationnaire ont été traitées avec 5 μg/mL de délafloxacine dans des tubes à essai pendant 24 h, lavées avec du PBS, puis ensemencées dans des tampons d’agarose CA-MHB sans antibiotiques contenant de l’iodure de propidium (1,6 μM) pour l’imagerie. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire 6 : P. aeruginosa pendant la récupération post-antibiotique. Une culture stationnaire de P. aeruginosa cultivée dans des MSB a été traitée avec 1 μg/mL de lévofloxacine dans des tubes à essai pendant 7 h, lavée avec du PBS, puis ensemencée dans des tampons d’agarose BSM sans antibiotiques contenant de l’iodure de propidium (16 μM) pour l’imagerie. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire 7 : Exemple d’acquisition d’image optimale. Cette vidéo est le canal de phase de la vidéo supplémentaire 4 avant le traitement de l’image, comme exemple d’une acquisition time-lapse optimale. Notez la dérive minimale, l’éclairage stable et le maintien de la mise au point tout au long de l’expérience. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire 8 : Exemple d’acquisition d’image sous-optimale en raison de la condensation. Cette vidéo montre une partie d’une expérience où l’acquisition de l’image a été affectée par une mauvaise mise au point, probablement due à la condensation sur la chambre due à un chauffage inadéquat de l’échantillon et/ou à une surhumidification de l’environnement d’imagerie. L’échantillon imagé était un P. aeruginosa traité à la lévofloxacine pendant la récupération post-antibiotique sur un tampon d’agarose BSM. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire 9 : Exemple d’acquisition d’image sous-optimale en raison de la dérive. Cette vidéo montre une partie d’une expérience où l’acquisition de l’image a été affectée par la dérive de l’échantillon, probablement en raison de la déshydratation et du rétrécissement/soulèvement du tampon d’agarose de la lamelle. L’échantillon imagé était celui de P. aeruginosa sur un tampon d’agarose contenant de la lévofloxacine et de l’iodure de propidium. Veuillez cliquer ici pour télécharger cette vidéo.
Fichier supplémentaire 1 : 25mm-3D-divider-for-35mmBioptechs.stl Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Nous avons constaté que le succès d’une expérience de microscopie time-lapse dépend de la qualité des coussinets d’agarose et de leur stabilité tout au long de l’imagerie. Les tampons d’agarose enfermés dans une chambre en acier inoxydable sont relativement faciles à préparer, ce qui permet d’obtenir des échantillons toujours planaires qui peuvent être imagés de manière stable sur des dizaines d’heures. Cela permet d’imager des dizaines de milliers de cellules en une seule expérience et augmente la probabilité de détecter des variants phénotypiques rares, comme les cellules persistantes, dans une population.
Cette méthode de préparation des tampons d’agarose présente une alternative facilement réalisable aux méthodes publiées précédemment. Notre protocole ne nécessite pas la précision technique de la fabrication de dispositifs microfluidiques ou les manipulations habiles des méthodes « sandwich » à l’agarose, ce qui facilite la réalisation de préparations cohérentes d’une série à l’autre 14,16,36. De plus, le système est rentable. La chambre en acier inoxydable est stérilisable et réutilisable (contrairement aux chambres en plastique à usage unique) et l’installation ne nécessite pas d’équipement spécialisé16,37. La chambre s’adapte facilement à différents systèmes de microscopie à l’aide d’inserts de platine disponibles dans le commerce. De plus, comme les bactéries sont immobilisées à l’interface entre l’agarose et le verre, nous avons réussi à suivre les bactéries très mobiles telles que P. aeruginosa tout en tenant compte des changements morphologiques (Figure 4, Vidéo supplémentaire 4). D’autres techniques d’imagerie unicellulaire, telles que la « machine mère », confinent les cellules à des canaux qui empêchent l’observation de changements morphologiques autres que la filamentation36.
Pour réussir avec ce protocole, il y a quelques étapes et paramètres critiques à garder à l’esprit. Pour la préparation des tampons, il est important de bien chauffer et fondre l’agarose, car les cristaux d’agarose restants provoqueront une diffraction de la lumière et affecteront la qualité de l’image. De même, il faut prendre soin de pipeter l’agarose dans la chambre sans introduire de bulles d’air. Pour s’assurer que l’épaisseur des tampons d’agarose reste constante et pour limiter la dérive de l’échantillon, il est important de laisser le tampon s’équilibrer, généralement pendant 15 minutes, dans l’enceinte humidifiée à température contrôlée avant le début de l’imagerie. Un autre facteur qui peut entraîner une mauvaise qualité d’image est le contrôle de l’humidité : une faible humidité entraînera la déshydratation et le rétrécissement du tampon d’agarose, tandis qu’une humidité élevée (ou un réchauffement inadéquat de l’échantillon dans la chambre) pourrait provoquer la condensation de l’air chaud sur l’échantillon et déformer l’imagerie. Un exemple d’imagerie time-lapse sous-optimale due à la condensation peut être trouvé dans la vidéo supplémentaire 8.
Une limite de la configuration actuelle est que les milieux de culture ne peuvent pas être échangés, ce qui empêche le suivi continu des bactéries individuelles avant, pendant et après le traitement antibiotique. Nous prévoyons que le couplage de la plaque d’agarose avec des cellules d’écoulement ou des dispositifs microfluidiques qui permettent l’échange de milieux de culture pourrait permettre de suivre les populations lors de changements nutritionnels ou environnementaux. Un autre paramètre de la conception actuelle qui pourrait être amélioré est l’aération des échantillons. Le joint torique et la conception vissée de la boîte interchangeable permettent une meilleure aération de l’échantillon par rapport aux configurations qui nécessitent l’utilisation d’un mastic à base de cire ou de graisse pour sceller les tampons16. Cependant, l’aération dans la chambre scellée peut encore être limitée et ne pas favoriser la croissance des aérobies obligatoires, bien que cela reste à tester.
Le protocole de préparation d’échantillons d’imagerie en accéléré que nous présentons dans cet article permet de suivre des milliers de bactéries pendant qu’elles réagissent ou se rétablissent d’un traitement antibiotique. Cette méthode est également hautement généralisable et a une variété d’applications potentielles au-delà de la biologie persistante. Par exemple, la configuration du tampon d’agarose et du diviseur permet l’ensemencement d’échantillons de cellules séparés spatialement tout en permettant la communication entre les cellules par diffusion à travers le tampon d’agarose. Nous explorons actuellement le potentiel de cette configuration pour tester comment l’échange de produits sécrétés affecte la croissance cellulaire dans les communautés multi-espèces. Nous prévoyons que ce protocole fournira une faible barrière d’entrée à la microscopie time-lapse pour le nouvel investigateur et des variations illimitées à explorer pour le microbiologiste chevronné.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
Nous remercions Mme Susan Staurovsky du UConn Health Center for Cell Analysis and Modeling Microscopy Facility pour son aide dans les expériences de microscopie. Nous remercions le Dr Mona Wu Orr et l’atelier sur l’essentiel de la génétique et du métabolisme des staphylocoques pour leurs protocoles et leurs conseils sur le clonage chez P. aeruginosa et S. aureus, respectivement. Ce travail a été soutenu par le financement des National Institutes of Health (NIH ; DP2GM146456-01 et 1R01AI167886-01A1 à W.W.K.M., 1F30DE032598-01A1 à P.J.H. et 1F31DK136259-01A1 à T.J.L.). Les bailleurs de fonds n’ont joué aucun rôle dans la conception de nos expériences ou la préparation de ce manuscrit.
matériels
| Name | Company | Catalog Number | Comments |
| BaSiC | GitHub | https://github.com/marrlab/BaSiC | |
| Certified Molecular Biology Agarose | Biorad | 1613101 | |
| Fiji-ImageJ | NIH | https://imagej.net/software/fiji/downloads | |
| Interchangeable Coverglass Dish | Bioptechs | 190310-35 | 35 mm ICD for preparing agarose pads; comes with 30 mm (#1.5) coverslips |
| Lumencor Spectra 7 LED light engine | Lumencor | https://lumencor.com/products/spectra-light-engine | Spectra 7 LED light engine |
| MetaMorph | Molecular Devices | Premier version 7.10.5 | |
| pco.edge 4.2 bi sCMOS camera | Excelitas | https://www.excelitas.com/product/pcoedge-42-bi-usb-scmos-camera | sCMOS camera |
| PeCon live cell incubation chamber | peCon | https://www.pecon.biz/ | |
| Thomas Scientific Round cover glass, #1.5 thickness, 25 mm, 100 pack | Fisher Scientific | NC1272770 | 25 mm (#1.5) coverslips |
| Zeiss Axiovert 200M microscope | Zeiss | https://www.zeiss.com/microscopy/en/products/light-microscopes/widefield-microscopes/axiovert-for-materials.html | Inverted microscope with Plan-Apochromat 63x/1.40 Oil Ph3 M27 objective, Lumencor Spectra 7 LED light engine, and pco.edge 4.2 bi sCMOS camera (6.5 mm pixel size) |
Références
- Brauner, A., Fridman, O., Gefen, O., Balaban, N. Q. Distinguishing between resistance, tolerance and persistence to antibiotic treatment. Nat Rev Microbiol. 14, 320-330 (2016).
- Balaban, N. Q., et al. Definitions and guidelines for research on antibiotic persistence. Nat Rev Microbiol. 17 (7), 441-448 (2019).
- Conlon, B. P. Staphylococcus aureus chronic and relapsing infections: evidence of a role for persister cells. Bioessays. 36 (10), 991-996 (2014).
- Mulcahy, L. R., Burns, J. L., Lory, S., Lewis, K. Emergence of Pseudomonas aeruginosa strains producing high levels of persister cells in patients with cystic fibrosis. J Bacteriol. 192 (23), 6191-6199 (2010).
- Verstraete, L., Van den Bergh, B., Verstraeten, N., Michiels, J. Ecology and evolution of antibiotic persistence. Trends Microbiol. 30 (5), 466-479 (2021).
- Sett, A., Dubey, V., Bhowmik, S., Pathania, R. Decoding bacterial persistence: Mechanisms and strategies for effective eradication. ACS Infect Dis. 10 (8), 2525-2539 (2024).
- Mempin, R., et al. Release of extracellular ATP by bacteria during growth. BMC Microbiol. 13, 301 (2013).
- Lewis, K. Persister cells, dormancy and infectious disease. Nat Rev Microbiol. 5 (1), 48-56 (2006).
- Fauvart, M., De Groote, V. N., Michiels, J. Role of persister cells in chronic infections: clinical relevance and perspectives on anti-persister therapies. J Med Microbiol. 60 (pt 6), 699-709 (2011).
- Van den Bergh, B., Fauvart, M., Michiels, J. Formation, physiology, ecology, evolution and clinical importance of bacterial persisters. FEMS Microbiol Rev. 41, 219-251 (2017).
- Levin, B. R., Rozen, D. E. Non-inherited antibiotic resistance. Nat Rev Microbiol. 4 (7), 556-562 (2006).
- Davis, K. M., Isberg, R. R. Defining heterogeneity within bacterial populations via single cell approaches. Bioessays. 38 (8), 782-790 (2016).
- Hare, P. J., LaGree, T. J., Byrd, B. A., DeMarco, A. M., Mok, W. W. K. Single-cell technologies to study phenotypic heterogeneity and bacterial persisters. Microorganisms. 9 (11), 2277 (2021).
- de Jong, I. G., Beilharz, K., Kuipers, O. P., Veening, J. W. Live cell imaging of Bacillus subtilis and Streptococcus pneumoniae using automated time-lapse microscopy. J Vis Exp. 53, e3145 (2011).
- Hare, P. J., Gonzalez, J. R., Quelle, R. M., Wu, Y. I., Mok, W. W. K. Metabolic and transcriptional activities underlie stationary-phase Pseudomonas aeruginosa sensitivity to levofloxacin. Microbiol Spectr. 12 (1), e0356723 (2024).
- Young, J. W., et al. Measuring single-cell gene expression dynamics in bacteria using fluorescence time-lapse microscopy. Nat Protoc. 7 (1), 80-88 (2011).
- Pang, Y. Y., et al. agr-Dependent interactions of Staphylococcus aureus USA300 with human polymorphonuclear neutrophils. J Innate Immun. 2 (6), 546-559 (2010).
- Bose, J. L., Fey, P. D., Bayles, K. W. Genetic tools to enhance the study of gene function and regulation in Staphylococcus aureus. Appl Environ Microbiol. 79 (7), 2218 (2013).
- Schenk, S., Laddaga, R. A. Improved method for electroporation of Staphylococcus aureus. FEMS Microbiol Lett. 73, 133-138 (1992).
- Grosser, M. R., Richardson, A. R. Method for Preparation and Electroporation of S. aureus and S. epidermidis. The Genetic Manipulation of Staphylococci. Methods Molecular Biology. , (2016).
- Krausz, K. L., Bose, J. L. Bacteriophage Transduction in Staphylococcus aureus: Broth-Based Method. The Genetic Manipulation of Staphylococci. Methods Molecular Biology. , (2016).
- Olson, M. E. Bacteriophage Transduction in Staphylococcus aureus. The Genetic Manipulation of Staphylococci. Methods Molecular Biology. , (2016).
- Fey, P. D., et al. A genetic resource for rapid and comprehensive phenotype screening of nonessential Staphylococcus aureus genes. mBio. 4 (1), e00537-e00612 (2013).
- Coe, K. A., et al. Multi-strain Tn-Seq reveals common daptomycin resistance determinants in Staphylococcus aureus. PLoS Pathog. 15 (11), e1007862 (2019).
- Figurski, D. H., Helinski, D. R. Replication of an origin-containing derivative of plasmid RK2 dependent on a plasmid function provided in trans. Proc Natl Acad Sci U S A. 76 (4), 1648 (1979).
- Shee, C., et al. Engineered proteins detect spontaneous DNA breakage in human and bacterial cells. eLife. 2, e01222 (2013).
- Kowalska-Krochmal, B., Dudek-Wicher, R. The minimum inhibitory concentration of antibiotics: methods, interpretation, clinical relevance. Pathogens. 10 (2), 165 (2021).
- Robinson, J. L., Jaslove, J. M., Murawski, A. M., Fazen, C. H., Brynildsen, M. P. An integrated network analysis reveals that nitric oxide reductase prevents metabolic cycling of nitric oxide by Pseudomonas aeruginosa. Metab Eng. 41, 67-81 (2017).
- Wolff, J. A., MacGregor, C. H., Eisenberg, R. C., Phibbs, P. V. Isolation and characterization of catabolite repression control mutants of Pseudomonas aeruginosa PAO. J Bacteriol. 173 (15), 4700-4706 (1991).
- Batchelder, J. I., Taylor, A. J., Mok, W. W. K. Metabolites augment oxidative stress to sensitize antibiotic-tolerant Staphylococcus aureus to fluoroquinolones. mBio. , e0271424 (2024).
- Power, A. D., Mok, W. W. K. Agar and agarose used for Staphylococcus aureus biofilm cultivation impact fluoroquinolone tolerance. J Appl Microbiol. 135 (8), lxae191 (2024).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Peng, T., et al. A BaSiC tool for background and shading correction of optical microscopy images. Nat Commun. 8, 14836 (2017).
- Ducret, A., Quardokus, E., Brun, Y. V. MicrobeJ, a tool for high throughput bacterial cell detection and quantitative analysis. Nat Microbiol. 1 (7), 16077 (2016).
- Paintdakhi, A., et al. Oufti: an integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Mol Microbiol. 99 (4), 767-777 (2016).
- Wang, P., et al. Robust growth of Escherichia coli. Curr Biol. 20 (12), 1099-1103 (2010).
- Moffitt, J. R., Lee, J. B., Cluzel, P. The single-cell chemostat: an agarose-based, microfluidic device for high-throughput, single-cell studies of bacteria and bacterial communities. Lab Chip. 12 (8), 1487-1494 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon