Method Article
定量玉米 (Zea mays) 非生物胁迫表型的成像和分析
摘要
提出了一种低成本植物成像以测量表型的方法,以及图像捕获的最佳实践和用于量化植物性状的图像分析管道。这些方法用于测量玉米 (Zea mays) 在高温、干旱和非生物综合胁迫条件下的表型。
摘要
测量定量植物表型或性状对于了解植物对环境因素(包括生物和非生物胁迫)的反应至关重要。然而,传统的植物测量方法可能耗时、不准确且具有破坏性,尤其是在评估个体生物量和高度等性状时。基于图像的分析提供了减少劳动力、提高准确性和精密度的机会,并支持在多个时间点对同一工厂进行重复测量。虽然高通量表型分析工具提供了一种解决方案,但它们通常非常昂贵,并且在全球范围内的可用性有限。描述了一种在照相馆设置中使用低成本单板计算机和数码相机捕获植物图像的方法,并附有基于免费开源软件包 PlantCV 的图像分析管道。该方法为图像采集、标注和传输提供了一种快速准确的方案,以及确保高质量图像采集的最佳实践。通过图像分析,同时对实验中所有图像的 10 多种表型进行量化,包括植物大小、高度和颜色等性状。这些方法用于表征 玉米 (玉米) 自交品种 B73 对高温、干旱以及高温和干旱非生物胁迫条件的响应。
引言
测量植物性状或表型对于回答植物科学中的基本和应用问题至关重要。例如,寻求了解基因突变对植物大小影响的研究人员需要定量测量植物高度和叶面积1。应用生物胁迫(如细菌、病毒或真菌感染)或非生物胁迫(如干旱、高温、寒冷或营养枯竭)的研究人员需要研究压力对植物颜色或“绿色度”的影响,以测量萎黄症 2,3。
虽然测量这些性状对于基础植物生物学和应用植物育种至关重要,但手动测量这些性状可能非常耗时、不精确且不准确 4,5,6,7。例如,测量植物的高度包括手动使用尺子测量每株植物,通常是在炎热或潮湿的环境条件下,从测量者的角度来看,哪个点被认为是植物的“顶部”可能会出错。此外,生物量和色素含量等测量具有破坏性,同一株植物不能测量随时间的变化或用于多次采样,因为它必须在进行这些测量的过程中被销毁 4,5,6,7。这些测量耗时、容易出错的性质阻碍了评估植物表型和生理胁迫反应的进展。
捕获植物图像并从图像中测量数量性状为这些问题提供了解决方案 4,5,6,7。图像分析提高了准确性和精密度,同时减少了研究人员进行测量的时间,并且由于无损性状量化 4,5,6,7,同一株植物可以多次成像或用于其他采样。此外,在实验完成并且植物被丢弃后很长一段时间内,图像可以存储和重新分析 4,5,6,7。可以使用低通量图像编辑工具(如 ImageJ (斐济)8)单独完成图像分析,也可以使用计算机视觉和机器学习(如 PlantCV9)等平台以更高的通量完成。PlantCV 是一个免费的开源图像分析平台,使研究人员能够首先从图像背景中遮罩(或分离)植物或其他物体,然后测量该植物或物体的大小、形态、颜色等9。使用模块化工作流程,研究人员首先使用样本图像构建不同模块的灵活工作流程(或管道),然后将剩余图像置于高通量工作流程中,而无需与每个单独的图像交互9。输出数量性状并分析组间的统计学显著差异。在捕获了足够的图像并使用适当的特征测量或元数据进行标记后,可以开发深度学习管道以进行高效的自动化图像分析10。
“高通量”表型分析可以提高数据收集和/或数据分析过程中的速度并减少人力 4,5,6,7。高通量表型分析工具实现了受控环境中图像采集的自动化,其中植物通过传送带或通过机器人移动到成像站,并自动使用与植物相关的名称(即带有元数据的标签)进行标记 6,11,12,13,14.虽然这些传送带或机器人支持的设施能够在没有人工干预的情况下捕获和标记大量图像,但世界上很少,而且它们的建造成本可能高达数百万美元,这使得许多研究人员无法获得它们。为了以更低的成本应用这些自动成像的概念,Raspberry Pi(此处称为单板计算机,SBC)是低成本计算机(每台不到 35 美元),用于捕获、命名、存储和传输在受控环境中生长的植物的图像。这些设置使用多达 72 个 Raspberry Pi 12,15,16 的阵列以自动、定时的间隔捕获图像。虽然比可能花费数百万美元的传送带系统更具成本效益,但前面描述的 SBC 生长室最适用于自上而下的小型植物生长视图,并且仍然需要大量的 SBC15。在野外环境中,龙门架、无人机等 UAS(无人航空系统)和卫星都会捕获图像以分析植物特性17,18。现场系统和高通量受控环境设施的前期成本、维护和构建和维护系统以及分析图像所需的专业知识都可能令人望而却步17,18。单独用于植物摄影的手机和数码相机在图像传输和元数据命名方面存在耗时的挑战,这对于下游图像分析至关重要。
这里描述的方法为植物成像提供了一种低成本的解决方案,可以解决其他系统无法解决的挑战。将植物单独运送到工作室的摄影装置可以有效地测量花盆中各种大小的植物。使用连接到数码相机的 SBC,该方法首先提供图像的标准化捕获、标记和传输。使用键盘或 QR/条形码扫描仪在捕获图像时命名图像,可以避免在捕获和存储图像后使用元数据重命名图像的耗时且容易出错的活动。图像的合成和推荐的相机设置对于实现下游图像分析非常重要,此处详细介绍了相关准则。最后,在 PlantCV 中提供了一个图像分析管道,用于从捕获的图像中提取数量性状,例如叶面积、植物高度、宽度和颜色。
相对于传送带系统,这种方法成本低,并且需要单个 SBC。此处描述的表型分析设置旨在消除传送带或机器人系统的限制和成本 - 可以测量任何规模的植物,因为该设置可以与生长空间分离,只需要一个 SBC,并且它适用于多种相机类型,以实现最大的灵活性和经济性。与此处介绍的方法类似的方法包括 PhenoBox 或 PhenoRig12,19。与手动测量相比,这种方法节省时间,减少错误,并且不会破坏植物 4,12,15,19。
研究方案
有关本协议中使用的试剂和设备的详细信息,请参见 材料表。
1. 照相馆的准备
- 将黑色摄影级织物挂在墙壁或天花板上,用于侧视成像。将织物悬垂到地板上至少 0.5 m,以便将植物放置在织物顶部(图 1)。
注意:织物应比要成像的植物至少高 100 厘米和宽 100 厘米。 - 将色卡放在背景上,靠在盒子等物体上,使其在框架中可见(图 1)。对于高度或宽度大于 30 厘米的植物,请使用至少 20 厘米宽的色卡。对于高度或宽度小于 30 厘米的植物,请使用 4-8 厘米宽的色卡。
- 将 SD 卡插入笔记本电脑或台式计算机。从网站 (https://www.raspberrypi.com/software/) 上的软件部分下载 Raspberry Pi Imager,然后将提供的 raspberrypi-image-capture.img.gz 文件 (https://github.com/danforthcenter/photo-maize-paper) 下载到笔记本电脑或台式计算机。
- 通过打开下载的成像器 (.exe) 文件来启动 Imager。点击 选择作系统 按钮,然后选择 使用自定义 底部的按钮。选择 image-capture.img.gz 文件。
- 接下来,点击 选择存储 按钮并选择插入计算机的 SD 卡。点击 Write 按钮。
注意:确保选择了正确的 SD 卡,因为此过程将删除卡上存储的所有数据。
- 接下来,点击 选择存储 按钮并选择插入计算机的 SD 卡。点击 Write 按钮。
- 使用 USB 线将最新版本的 SBC 连接到键盘,使用 HDMI 线连接到外部显示器,并使用带开关的电源线连接到电源板。使用显示器随附的电源线将显示器连接到电源板。将 SD 卡放入 SBC。有关设置的标记示意图,请参见 图 1 。
- 按下电源板上的开关打开电源板,使用显示器上的按钮打开显示器,然后将电源线上的开关拨到“打开”位置来打开 SBC。
- 使用 WIFI 或以太网电缆将 SBC 连接到本地互联网。
- 如果要将照片直接传输到服务器或云存储:
- 在 SBC 上编辑文件 “CAPTURE-PHOTO.py” 以包含完成传输的文件路径。
- 在终端中使用以下命令使用服务器或云存储的路径和密码编辑 win-credentials 文件: sudo nano /etc/win-credentials。
注意:请与 Internet 系统管理员联系,以确保该文件夹具有传输文件的权限。
- 按照条形码扫描仪随附的制造商说明,使用 USB 或蓝牙将无线条形码扫描仪连接到 SBC。
- 通过单击屏幕左上角的 Raspberry Pi 按钮,单击“注销”,然后单击 “关闭” 以安全地关闭 SBC,以关闭 SBC。关机后,将 SBC 电源开关“打开”,将电源线切换到“关闭”,将电源板切换到“关闭”。
2. 植物生长和胁迫处理
注意:任何植物都适合这种表型分析方法;这些植物生长方法适用于此处介绍的代表性结果。
- 从库存中心获取 B73 或其他玉米品种的种子。
- 准备土壤介质和生长条件:
- 用 Turface MVP 和 Berger BM7-35 的 1:1 体积混合物和 3 磅/立方码的缓释肥料 14-14-14 (NPK) 填充 3.5 英寸花盆 (0.7 L)。将花盆放入有孔的托盘中,并用反渗透 (RO) 水彻底注水。
- 将生长室设置为以下环境条件:白天 30 °C/夜间 20 °C,白天长度 16 小时,湿度 60%,白天花盆水平的光照为 500 mmol.m -2s -1 。设置具有相同条件的单独生长室,除了 38 °C 白天/28 °C 夜间用于热应激。
- 种植 20 颗玉米种子,每颗种子放在一个 3.5 英寸的花盆中,将土壤深 ~2 厘米,并用移位的土壤覆盖。用 RO 水彻底浇水,然后放入对照生长室中。每天用液体肥料浇水(根据制造商的说明混合;见 材料表)。
注意:在开始施用压力之前,种植实验所需的额外植物将导致发芽不良和缺乏均匀性。发芽率会因品种而异。 - 生长 21 天后,将 16 株最均匀的植物(每次处理 n = 4 株植物)移植到 3.79 升花盆中,花盆中装满相同的盆栽土壤混合物,用 RO 水预先湿润。将植物放回对照生长室。
- 在第 25 天(移植后 4 天),在室内灯打开时将一半的植物 (n = 8) 移至热应激室。停止给两个腔室中受干旱胁迫的植物浇水(每个腔室 n = 4)。
注意:干旱胁迫植物可能需要浇水,以便植物存活到实验结束;在这里,干旱植物在第 31 天和第 37 天用肥料浇水。 - 在第 38 天(应激开始后 13 天),从生长室中取出植物进行成像。
3. 植物成像
- 启动 SBC(参见 图 2):
- 使用电源板上的开关打开电源板。
- 使用显示器上的 按钮打开显示器。
注意: 如果在显示器打开之前打开 SBC,它将无法正常启动。 - 将 SBC 电源线上的开关转到 “on”(打开)。
注意:这将打开 SBC,并将在显示器上启动;SBC 本身没有 “on” 开关,只有电源线上。
- 将相机放在三脚架或固定的桌子上。
注意: 整个植物和色卡应在相机屏幕上可见。相机应垂直于地面,并能直接看到植物的中心(无角度)。相机必须与 gphoto2 软件包兼容,可在此处找到列表:http://www.gphoto.org/proj/libgphoto2/support.php。 - 通过相机或电池的电源线为相机供电。使用相机上的按钮开启相机。
- 使用相机随附的数据传输电缆将相机连接到 SBC 上的 USB 端口。
- 在摄影设置中排列植物:
- 在放置花盆的摄影级织物上放一条深色胶带,以确保每株植物的位置相同。确保色卡与胶带对齐。
注意:将色卡放置在距离植物边缘至少 10 厘米的地方。在 PlantCV 工作流程(如下)中进行质量控制期间,应能够裁剪图像以仅包含植物和花盆,而不包括色卡,以确保色卡不包含在最终特征提取中。有关高质量和低质量图像的示例,请参见 图 2 。 - 将花盆放在胶带上。对于在平面上生长的植物,例如玉米 (Zea mays) 和高粱 (Sorghum bicolor),请将植物的最宽角度朝向相机。对于不在平面中生长的植物,捕获植物的 0 度和 90 度角,并在图像分析完成后对得到的定量测量值进行平均。
- 在放置花盆的摄影级织物上放一条深色胶带,以确保每株植物的位置相同。确保色卡与胶带对齐。
- 在显示器上显示的 SBC 桌面屏幕上,单击左上角菜单栏上的 终端 按钮打开终端。
- 如果直接保存到服务器或云存储,请使用以下命令挂载存储,方法是编辑必要的云存储路径(以下示例中的 cloud-storage-path)和密码,在打开的终端窗口中键入代码行,然后按 键盘上的 Enter 。如果出厂设置保持不变,则用户 ID (UID) 将为“pi”。
注意:
sudo mount -t cifs -o credentials=/etc/win-credentials,uid=pi //cloud-storage-path /mnt/cloud-storage - 在终端中键入以下代码,然后按键盘上的 Enter 。将出现一个新窗口。
./RUN-PHOTOSTUDIO-SHELL.sh
注意:RUN-PHOTOSTUDIO-SHELL.sh 文件调用 CAPTURE-PHOTO.py 文件,如果使用上述方法加载,则会将其预加载到 pi 上。或者,这些脚本已在 GitHub 上提供,用于放入现有的 Raspberry Pi SD 卡。 - 将打开一个图形用户界面窗口。输入图像名称,或使用条形码扫描仪扫描植物的条形码或 QR 码。
- 选择一个选项以将映像保存到本地或保存到挂载的服务器/云存储。
- 点击 点击这里拍照! 按钮。
- 如果选择了本地存储,请打开桌面上的 PHOTOS 文件夹以检查图像。如果选择了服务器或云存储,请在该位置打开图像。
- 如有必要,请调整摄像机的设置,方法是将摄像机从 SBC 上拔下,按照制造商的说明进行调整,然后将其重新插入 SBC。
注意: 可能需要根据房间中的照明和被成像的物体调整曝光、亮度对比度等设置。一个常见的错误是过度曝光图像。要检查测试图像的曝光,请通过 PlantCV 图像分析工作流程继续处理测试图像,其中包括曝光的质量控制检查。根据需要调整相机设置以减少或增加曝光,直到直方图得到适当分布。 - 捕获完所有图像后,使用首选方法(如 USB 存储、Internet 浏览器或 SSH 传输)将图像从 SBC 传输到本地计算机或云存储。
4. 从图像中提取特征
- 将以下文件下载到本地计算机或服务器:“photo-studio-SV-notebook.ipynb”、“workflow.py”和“photo-studio-SV-config.json”。所有文件都可以在 GitHub (https://github.com/danforthcenter/photo-maize-paper) 上找到。
- 使用所述步骤 (https://plantcv.readthedocs.io/en/latest/installation/) 在本地计算机或服务器上安装 PlantCV 。
- 使用首选代码编辑器(如 JupyterLab 或 Visual Studio Code)打开文件 photo-studio-SV-notebook.ipynb 。
- 运行每个代码块,根据代码块中概述的参数根据需要进行编辑,以获得仅图像中植物的干净掩码。
- 如果对示例图像的分析感到满意,请在首选代码编辑器中打开文件 workflow.py 。使用在 “photo-studio-SV-notebook.ipynb” 中更改的任何参数编辑此文件并保存。
- 打开文件 photo-studio-SV-config.json 并编辑文件路径,以定向到输入图像文件夹、所需的输出文件夹等,如文件中所示。
- 打开终端并运行以下代码以激活 PlantCV。为此,请在终端中键入代码 conda activate plantcv,然后按键盘上的 Enter 。
- 将以下代码行更改为 “photo-studio-SV-config.json” 文件的文件路径(这是以下代码中唯一要进行的更改)。
- 在终端中,键入编辑后的代码并按键盘上的 Enter 。这将对实验中捕获的所有图像运行工作流。
注意:
plantcv-run-workflow --config /用户/用户名/photo-studio-SV-config.json
- 在终端中,键入编辑后的代码并按键盘上的 Enter 。这将对实验中捕获的所有图像运行工作流。
- 检查输出图像以确定代码是否适合所有图像。
注意:植物的蓝色和粉红色轮廓应位于植物的边缘周围,并且图像中不应有其他对象。如有必要,请返回“photo-studio-SV-analysis.ipynb”并测试有问题的图像并编辑代码,特别是蒙版步骤和降噪,以获得所需的结果。有关高质量、可接受和低质量图像分割的示例,请参见 图 3 。 - 输出文件将采用 .json 文件的形式。返回终端(确保 conda activate plantcv 已运行),键入以下代码,然后按键盘上的 Enter 键将文件转换为 .csv 以进行下游统计分析,将文件路径更改为“results-photo-studio.json”文件的路径。
plantcv-utils json2csv -j /用户/用户名/results-photo-studio.json -c /用户/用户名/results-photo-studio.csv
注意:要将测量值从像素转换为标准测量单位(如厘米),请使用 PlantCV 工作流程中 find_color_card 函数提供的转换。 - 对输出数量性状进行统计分析,以确定由于不同植物物种、品种、处理和时间点而导致的植物表型差异。
注意:R Studio 中的 pcvr 包已制定用于分析 PlantCV 的输出的统计显着性和结果绘图20。请参阅 图 4 中收集的特征的摘要和可视化表示。
结果
为了开发这种方法,对 B73 玉米植株在受控温度、高温、浇水充足和干旱条件下及其组合进行了定量表型评估,共进行了四个处理。B73 是一种著名的自交玉米品种,拥有丰富的遗传数据21。
PlantCV 中的图像分析成功地对图像进行了色彩校正,用植物元数据标记了定量结果,并将植物与背景分割(换句话说,分离),如图 3 所示。不成功的分析将提供输出图像,这些图像不会显示植物外部周围的蓝色轮廓或粉红色形状,而是包含额外的噪声或植物的缺失部分(图 3,低质量示例)。双通道阈值是必要的,以便在掩模中包含尽可能多的植物(参见“photo-studio-SV-notebook.ipynb”中的替代单通道策略)。然而,一些植物,特别是那些具有紫色茎的植物,没有完美的分割,但足以进行下游分析(图 3)。
PlantCV 分析提供了 16 个定量的单值性状,如图 4 所示。单值性状为每株植物提供一个值 - 例如,叶面积、高度、宽度和色调圆形平均值。使用 “analyze.shape” 函数(图 4)考虑叶面积,并考虑整个植物。然而,使用“analyze.bound_horizontal函数”考虑高度和宽度,在植物土壤线上绘制边界。这是因为将植物作为一个整体考虑,下垂在花盆侧面的植物将具有更大的高度,而应该只从它与土壤相遇的植物基部开始测量。除了单值特征之外,PlantCV 还输出多值特征,这些特征是直方图,特别是颜色值,表示植物的每个像素。色相圆平均值是一个单值性状,它是整个植物的平均色相值。
为了确定哪些性状最有趣,计算了每个性状的处理解释的方差,以及使用 pcvr R Studio 包20 中的 “frem” 函数不同性状之间的相关性(图 5A)。选择叶面积、高度、宽度和色调圆平均值进行下游分析,因为它们解释了超过 50% 的处理方差,并且是植物生长(面积、高度和宽度)和健康状况(色调)的相关指标。具有最大和最小叶面积的植物的代表性图像展示了本实验中收集的表型的变化(分别为图 5B、C)。叶面积最大的植物也具有最高的平均色调,并且是 B73、浇水充足的热胁迫植物(图 5B)。最小的叶面积具有最小的平均色调(图 5C),并且是 B73、干旱胁迫、热胁迫的植物。
水处理在确定叶面积 (F(1,13) = 226.5,p = 1.32 x 10-9)、高度 (F(1,13) = 21.1,p = 0.0005)、宽度 (F(1,13) = 75.5,p = 8.92 x 10-7) 和色调圆平均值 (F(1,13) = 27.8,p = 0.0002)方面具有显着影响,使用处理效果的线性回归模型和 R Studio 中的双向方差分析确定(完整结果见表 1)。 温度处理对叶高有显著影响 (F(1,13) = 5.94,p = 0.03),但对叶面积、宽度或色调圆平均值没有影响 (p > 0.05,表 1)。 温度和水状况之间的交互作用在任何测量的性状中都不是一个重要因素(p > 0.05,表 1)。
接下来,使用线性回归和事后检验 (估计的边际平均值) 将单个处理与对照组进行比较,并对多个比较进行 Sidak 校正 (图 6)。在两种温度条件下,与浇水良好的植物相比,干旱胁迫植物的叶面积、株高、植株宽度和色调圆形平均值均显著降低(p < 0.05, 图 6)。浇水充足的热胁迫仅降低株高(p < 0.05, 图 6),但没有显着减少叶面积、植株宽度或色调圆形平均值。可以通过评估多值颜色特征色调来进一步研究这种色调的差异,如图 7 中绘制为直方图,而不仅仅是平均值。干旱导致的平均色调降低是由于像素从绿色变为黄色,称为萎黄(图 7)。虽然热量没有显示出平均色调的显着差异,但由于绿色变黄(萎黄)和变暗,它确实表现出减少和增加(图 7)。
成像后,在根-芽交界处切割植物,并记录地上组织的重量。用毛巾轻轻清洗和擦干根部,然后称重,以计算植物总生物量(也称为重量),以便与所描述的基于图像的表型方法进行比较。 通过 图像分析测量的叶面积与植物生物量密切相关(R2 = 0.84, 图 8)。

图 1:带有标记组件的照相馆设置图像,用于收集植物图像。 (A) 摄影级织物,(B) 色卡,(C) 用于均匀放置花盆的胶带线,(D) 数码相机,(E) 单板计算机,(F) 显示图像捕获过程的显示器。 请单击此处查看此图的较大版本。

图 2:成像工作流程概述。 提供了具有代表性的“高质量”和“低质量”图像。 请单击此处查看此图的较大版本。
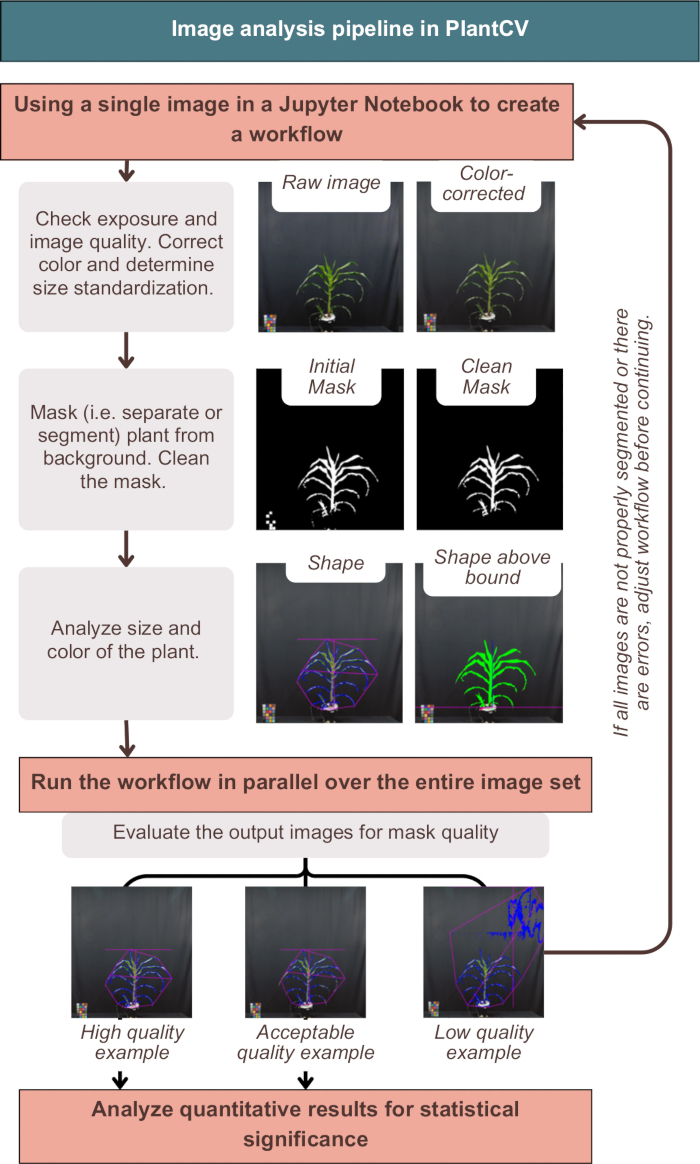
图 3:PlantCV 中的图像分析工作流程概述。 呈现了“高”、“可接受”和“低”质量分割的代表性图像;PlantCV 性状使用整株植物形状的粉红色轮廓和植物区域的蓝色轮廓表示。 请单击此处查看此图的较大版本。
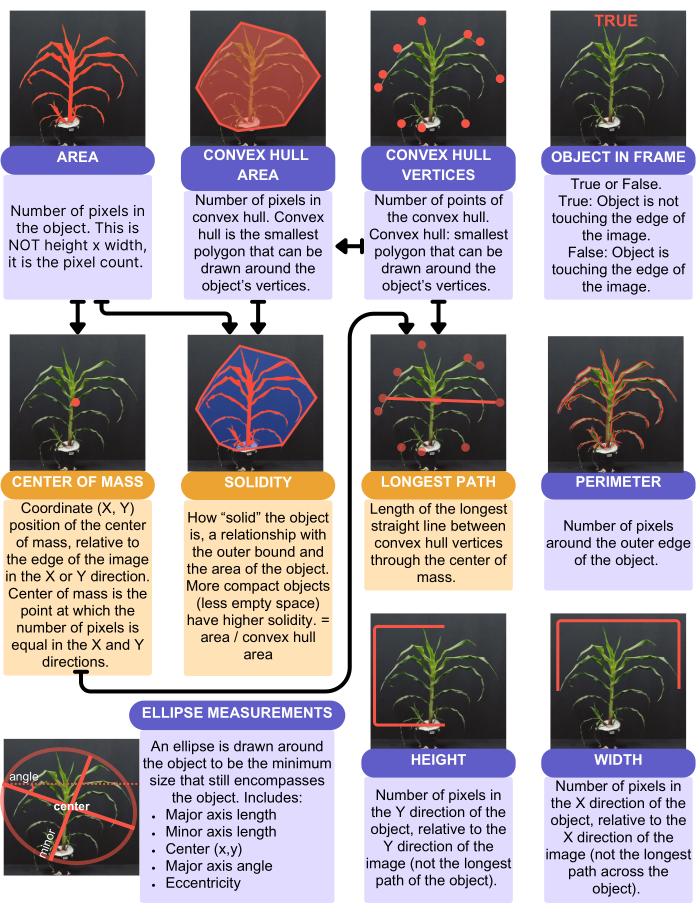
图 4:从分析中捕获的形状特征的摘要和可视化表示。 Shape 函数。紫色框表示在计算中不使用其他特征的特征。橙色框表示在计算中使用其他特征的特征;箭头指示这些计算中使用了哪些特征。 请单击此处查看此图的较大版本。
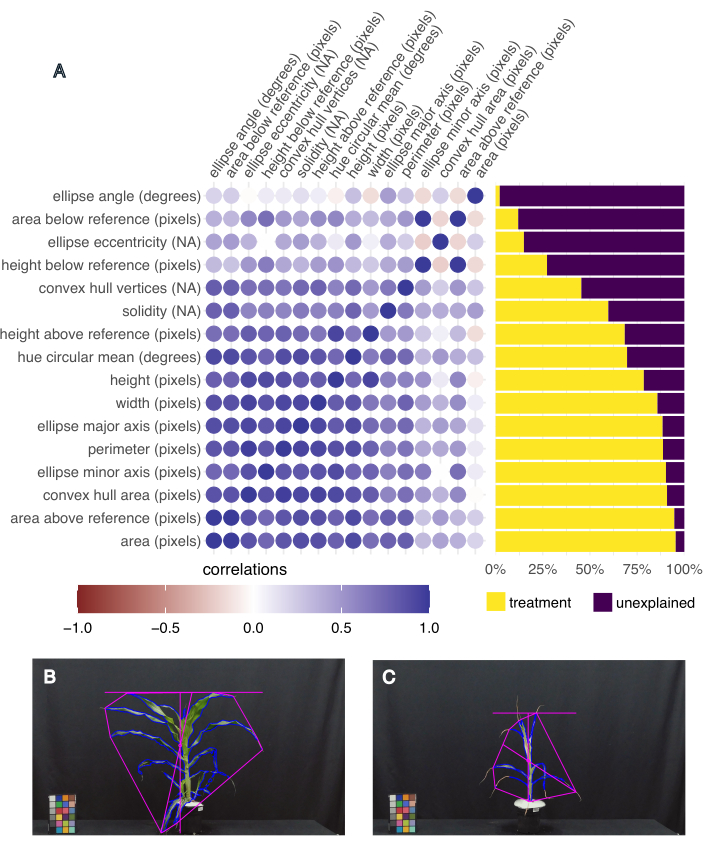
图 5:基于图像的表型分析捕获了表型方差和相关性状。 (A) 由处理解释的性状方差(右)和性状之间的相关性(左)。每个特征都标记了,后跟其度量单位;“NA” 表示它是无单位的测量。(B) 具有最高叶面积和最高色调圆平均值(度)的植物的图像。(C) 具有最小叶面积和最低色调圆平均值(度)的植物图像。 请单击此处查看此图的较大版本。
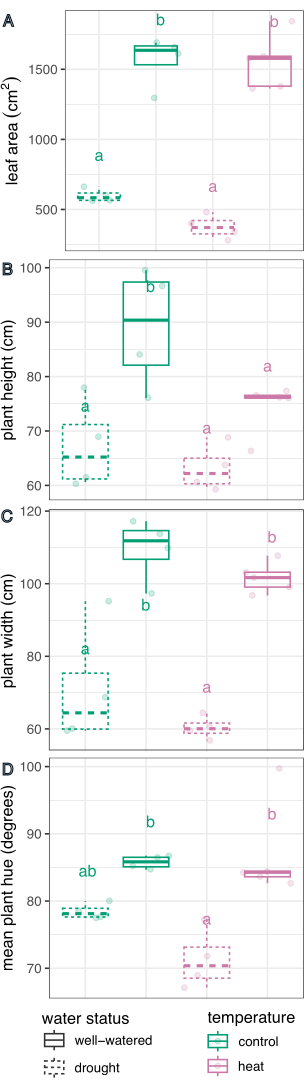
图 6:处理对植物表型影响的箱线图比较。 比较了叶面积 (A)、高度 (B)、宽度 (C) 和色调圆平均值 (D) 受干旱、高温和综合胁迫的影响。n = 4。字母表示处理之间的统计学显着差异 (p < 0.05);使用线性回归和事后检验 (估计的边际平均值) 与 Sidak 校正进行多重比较进行比较。 请单击此处查看此图的较大版本。
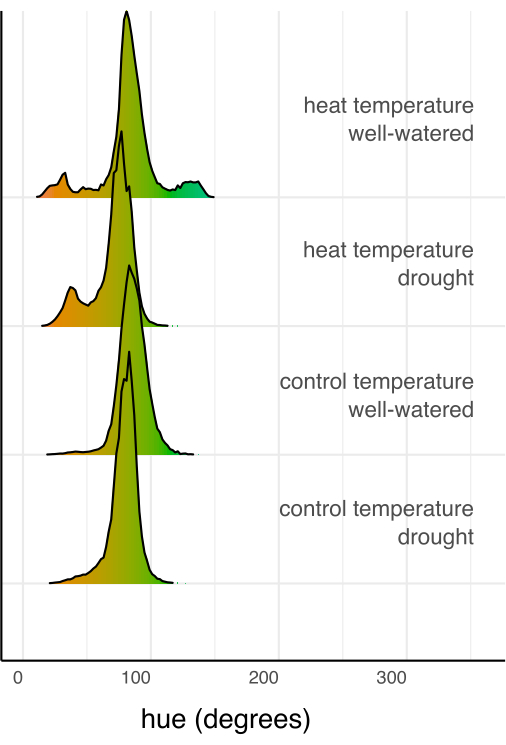
图 7:每个处理组合的所有像素相加的颜色(色调)直方图,按处理组合中的像素总数进行归一化。 直方图按色相程度的颜色值着色。 请单击此处查看此图的较大版本。
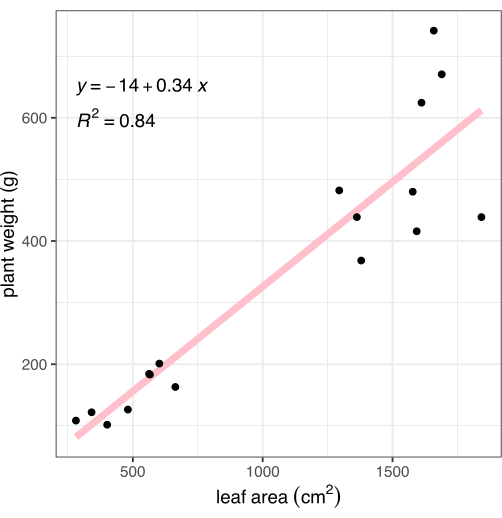
图 8:从图像测得的叶面积与全植物生物量的相关性。 每个黑点代表一种植物。粉色线表示叶面积 (X) 和生物量 (Y) 之间的线性相关性。 请单击此处查看此图的较大版本。
| 特性 | 因素 | 自由度 | F 值 | p 值 |
| 叶面积 (cm^2) | 温度 | 1 | 0.5372 | 0.4766 |
| 叶面积 (cm^2) | 水 | 1 | 226.5318 | 1.32E-09 |
| 叶面积 (cm^2) | 温度:水 | 1 | 2.1526 | 0.1661 |
| 叶面积 (cm^2) | 残 差 | 13 | ||
| 高度 (cm) | 温度 | 1 | 5.9426 | 0.0298935 |
| 高度 (cm) | 水 | 1 | 21.107 | 0.0005029 |
| 高度 (cm) | 温度:水 | 1 | 2.1752 | 0.1640545 |
| 高度 (cm) | 残 差 | 13 | ||
| 宽度(cm) | 温度 | 1 | 2.2495 | 0.1575 |
| 宽度(cm) | 水 | 1 | 75.5474 | 8.92E-07 |
| 宽度(cm) | 温度:水 | 1 | 0.0839 | 0.7766 |
| 宽度(cm) | 残 差 | 13 | ||
| 色调圆平均值(度) | 温度 | 1 | 0.9154 | 0.3561432 |
| 色调圆平均值(度) | 水 | 1 | 27.7988 | 0.0001509 |
| 色调圆平均值(度) | 温度:水 | 1 | 3.4792 | 0.0848724 |
| 色调圆平均值(度) | 残 差 | 13 |
表 1:线性回归模型和双向方差分析的结果,检查了水处理、温度处理及其相互作用对植物性状的影响。
讨论
基于图像的表型分析可准确、快速、定量地测量植物表型 4,5,6,7。与手动使用尺子记录植物高度或定性测量可能与健康相关的植物颜色特征相比,这种方法明显更快、更精确、更准确,并且提供了更多的测量 4,5,6,7。这里可以看到叶面积,在以前的研究10 中,叶面积与植物生物量密切相关(图 8),这表明该指标可以可靠地取代手动称量全植物生物量的耗时、破坏性和容易出错的过程。虽然可以单独使用相机来捕获图像,但添加低成本 SBC 通过自动化图像收集和具有必要元数据的图像标记提高了吞吐量15。这种方法适用于每天成像一次(或频率较低)的 1 到 ~300 株植物。对于实验中大量的植物,基于传送带的、高架龙门架或机器人支持的成像系统更合适,因为单独移动植物进行成像的时间可能比工作日更长11,13。对于更频繁的时间点(例如,每小时一次图像,而不是每天一次或频率较低),生长室内的相机设置更合适,因为它们可以自动捕获图像,但受植物大小的限制,并且需要额外的 SBC15。
虽然 PlantCV 在此处作为图像分析选项提供,但存在其他软件。ImageJ (Fiji) 是一个用于图像分析的免费软件包,也可以通过图形用户界面 (GUI) 用于植物图像8;然而,它并不是专门为全厂图像分析而开发的,对高通量计算的适应性较差,并且比 PlantCV 中可以轻松应用于新图像的已经工作的管道更难重复9。深度学习(例如 BioDock,参见 材料表)和 Segment Anything22)方法很有吸引力,因为它们能够轻松地从后台分割植物,并且可以用于此处描述的分割任务。然而,这些深度学习模型不提供此处描述的植物表型测量10,并且要进行这些更复杂的定量表型测量,可能需要大量的训练图像(通常大于 10,000 张)和超出基本笔记本电脑或台式电脑的计算能力 10。这些模型还需要大量的计算机内存和处理能力,而许多研究人员无法获得这些10。
该协议最关键的步骤是安排植物成像设置,并确保感兴趣的植物附近没有物体(例如其他植物、色卡或植物碎片)14。如果图像设置不当,或者未使用色卡,则以后将无法对其进行分析,因为没有标准化机制。虽然该协议不需要校准步骤,但处理过程中的色彩校正和尺寸标准化是必不可少的。始终确保整个色卡都在框架中,但不与植物重叠(图 2)。另一个常见的陷阱是照明不均匀,这可能会导致阴影或明亮镜头;我们建议使用明亮、漫射的顶灯并覆盖房间内的窗户,以防止光线不均匀(图 2)。如果顶灯造成阴影,可以以正面对植物的角度添加额外的灯。灯泡的类型不是问题,因为将使用色卡校正图像中的颜色。模糊的图像不可用于分析 - 使用相机上的自动对焦或制造商的手动对焦说明,以确保图像清晰。
同样,确保图像不会过度曝光(一个常见的错误)可以进行适当的分析(图 2);在图像分析期间,无法在颜色校正中修复曝光过度的图像,因为像素值超出了相机在当前设置14 下的检测范围。为防止过度曝光,请使用此处描述的 PlantCV 工作流程评估测试图像的颜色直方图或 RGB 值(图 3),如果曝光测试确定图像曝光过度,请降低相机的亮度设置。RGB 值(以及直方图)的值不应为 0 或 255,这是值范围,因为这些值可能高于或低于检测范围,并且只能记录最小值或最大值,而不是真实值。应始终使用这些方法评估测试图像的过度曝光(图 2 和 图 3);我们建议使用本出版物中提供的 PlantCV 曝光质量控制作为检查,它确定质量图像的像素在 0 或 255 时小于 5%。根据经验,相机和分析软件提供直方图,直方图应具有相等且完整的直方图尾部;当被截断的值低于 0(曝光不足)或高于 255(曝光过度)时,应使用调整后的照相机设定重新拍摄。我们建议选择一个有明亮、均匀、人工照明且没有窗户照明的位置,因为窗户可能太亮、不均匀,并且经常改变空间的光线照射。图像应均匀显示,色卡的灰色方块不应显示为白色,这表明曝光过度。
可能需要对图像捕捉代码进行故障排除,具体取决于用户机构的 IP 和安全设置。要进行故障排除,请联系 IT 部门以解决 Internet 连接和文件传输问题。一个常见的错误是为 SBC 使用错误大小的 SD 卡,或者在打开连接的显示器之前打开 SBC。要进行故障排除,请严格按照上述协议作,然后重新加载 SBC 软件。在线论坛是解决和寻找使用 gphoto2 时所选特定相机的解决方案的绝佳资源。要对 PlantCV 图像分析进行故障排除并使用最新的版本控制,请使用文档页面 (https://plantcv.readthedocs.io/en/latest/),或在 GitHub 上发布问题 (https://github.com/danforthcenter/plantcv/issues)。特别是,如果本协议中的阈值方法不能将植物与背景充分分离,请使用“photo-studio-SV-notebook.ipynb”或文档页面上提供的替代阈值方法。
该方法可应用于任何植物物种和处理,这些植物可以运输到所描述的组装照相装置中。例如,它不适用于在田间生长的植物,因为如果不挖出植物,就无法将它们运输到摄影装置中。在这种情况下,可以砍伐植物,然后使用该协议进行成像,尽管测量会具有破坏性。可以在单个图像中捕获多个植物并进行分析,但用户应注意防止植物相互接触(图 2),并且每次将植物放置在背景的相同位置,以确保工作流程可以在整个图像集上自动运行。如果拟 南芥等植物需要自上而下的视图,请使用三脚架将相机移动到放置在摄影级织物上的植物的顶部;其余的图像捕获和分析方法保持不变。
正如这里用玉米所展示的那样,这种方法对于植物育种和了解植物如何耐受非生物胁迫(如高温和干旱)的基本见解都很重要。高温和干旱胁迫共同导致全球玉米每年损失 15%-20%23。预计产量损失只会随着全球气候变化而增加,因为预计到 2100 年气温将升高 1.4-4.4°C24。评估玉米抗逆性的多样性,以及确定非生物抗逆性的表型、生理和分子机制,是全球粮食安全的关键研究目标25。为了解决这些问题,所有相邻领域的研究人员——生理学家、遗传学家、育种家、分子生物学家等——必须能够快速、有效且通常无损地评估非生物胁迫下玉米植物的表型25。此处显示的方法和示例结果满足了这一需求,是研究人员应对实际农业挑战的重要工具。
根据先前的大量研究,这里观察到的表型是假设的——干旱胁迫会降低植物的大小和绿色度(用色调圆平均值衡量)26 (图 6、 图 7 和 表 1)。浇水良好的热胁迫表型出乎意料地没有显着减少叶面积,尽管重复次数越多,这种关系可以更好地统计评估,并且高度如预期的那样降低(图 6)。虽然色调圆形平均值没有因热量而改变,但颜色分布确实发生了变化,以包括更多的黄色和更深的绿色像素,这表明仅评估平均色调是不够的,应考虑颜色的多值特征直方图(图 7)。这些表型变化描述了植物对环境的反应,可以采用其他技术,如气体交换测量、显微镜成像等,来评估响应压力而发生的生理变化。这一结果证明了精确、定量测量的力量,否则使用手动或定性植物健康测量会错过这些测量。这些见解对于培育和改造抗气候变化的作物至关重要,气候变化会显著降低全球作物产量,并且预计未来几年会恶化。
披露声明
没有。
致谢
这项工作是在唐纳德丹佛斯植物科学中心 (RRID: SCR_019049) 的表型核心设施进行的,该中心为 KMM 和 J.G.D. 提供了资金支持。这项工作得到了 Donald Danforth 植物科学中心企业租车研究所资助的支持。D.S. 由泰勒地理空间研究所整笔赠款和美国经济发展管理中心农业科技和应用位置科学与技术中心 (CATALST) 赠款 (ED21HDQ0240072) 资助。我们感谢 Kevin Reilly 和 Kris Haines(综合植物生长设施,唐纳德丹佛斯植物科学中心,RRID:SCR_024902)在植物生长方面的专业知识和帮助。我们感谢 Noah Fahlgren、Malia Gehan 和 Haley Schuhl 对 PlantCV 的建议。
材料
| Name | Company | Catalog Number | Comments |
| 15W USB-C Power Supply | CanaKit | DCAR-RSP-3A5-C | |
| 25604 UC-E6 USB Cable | Nikon | 18208256044 | |
| 3.5 inch pots | Hummert International | 13006000 | |
| Berger BM7 35% Bark HP | Hummert International | 10121500 | |
| Biodock 2024 | AI Software Platform | www.biodock.ai. | |
| Code editing software | Jupyter | https://jupyter.org/install | |
| Code editing software | Visual Studio Code | https://code.visualstudio.com/download | |
| Color Card ColorChecker Classic | Calibrite | CCC | |
| Color Card ColorChecker Passport | Calibrite | CCPP2 | |
| Conviron Growth Chamber | Conviron | BDW80 model | |
| Dell 22 Monitor | Dell | SE2222H | |
| EH-67 AC Adapter | Nikon | AZ185 | |
| Image analysis software | PlantCV | https://plantcv.readthedocs.io/en/latest/installation/ | |
| Jack's Water Soluble Fertilizer 15-5-15 | Hummert International | 7590200 | liquid fertilizer |
| Keyboard and Mouse | Logitech | 1440638 | MK270 wireless keyboard and mouse |
| Maize seed | USDA-ARS Germplasm Resources Information Network (GRIN) | B73 | https://www.ars-grin.gov/ |
| Micro HDMI to HDMI cable | Amazon | B07KSDB25X | |
| Nikon Coolpix B500 camera | Nikon | ||
| Nikon D7200 camera | Nikon | ||
| Osmocote 14-14-14 | Hummert International | 7630000 | |
| Photography-grade fabric | Yayoya | Polyester Fabric Black Photo Backdrop for Photo Video Studio Televison | |
| Raspberry Pi 4 Case with heatsink | iUniker | B07ZVKN262 | |
| Raspberry Pi 4 Model B Rev 1.1 | Raspberry Pi Foundation | RP4B4GB_BP | |
| Raspberry Pi 4 PiSwitch (USB-C) | CanaKit | RSP-PISWITCH-USBC | |
| Raspberry Pi software | Raspberry Pi Imager | https://www.raspberrypi.com/software/ | |
| Statistical analysis software | Rstudio | https://posit.co/downloads/ | |
| Tripod | K&F Concept | T255A3+BH-28L | |
| Turface MVP | Hummert International | 10240000 | |
| Wireless Barcode Scanner | Netumscan | B08X4NMX2M | 1D and 2D barcode scanner |
参考文献
- Xiao, Q., Bai, X., Zhang, C., He, Y. Advanced high-throughput plant phenotyping techniques for genome-wide association studies: A review. J Adv Res. 35, 215-230 (2022).
- Mutka, A. M., et al. image-based phenotyping methods provide insight into spatial and temporal dimensions of plant disease. Plant Physiol. 172 (2), 650-660 (2016).
- Mutka, A. M., Bart, R. S. Image-based phenotyping of plant disease symptoms. Front Plant Sci. 5, 734 (2014).
- Fahlgren, N., Gehan, M. A., Baxter, I. Lights, camera, action: High-throughput plant phenotyping is ready for a close-up. Curr Opin Plant Biol. 24 (2), 93-99 (2015).
- Das Choudhury, S., Samal, A., Awada, T. Leveraging image analysis for high-throughput plant phenotyping. Front Plant Sci. 10, 508 (2019).
- Yang, W., et al. Crop phenomics and high-throughput phenotyping: Past decades, current challenges, and future perspectives. Mol Plant. 13 (2), 187-214 (2020).
- Omari, M. K., et al. Digital image-based plant phenotyping: A review. Korean J Agric Sci. 47 (1), 119-130 (2020).
- Schindelin, J., et al. Fiji: An open-source platform for biolFiogical-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Gehan, M. A., et al. PlantCV v2: Image analysis software for high-throughput plant phenotyping. PeerJ. 5, e4088 (2017).
- Murphy, K. M., Ludwig, E., Gutierrez, J., Gehan, M. A. Deep learning in image-based plant phenotyping. Annu Rev Plant Biol. 75 (1), 771-795 (2024).
- Fahlgren, N., et al. A versatile phenotyping system and analytics platform reveals diverse temporal responses to water availability in Setaria. Mol Plant. 8 (3), 1520-1535 (2015).
- Yu, L., et al. Development of a mobile, high-throughput, and low-cost image-based plant growth phenotyping system. bioRxiv. , (2023).
- Atefi, A., Ge, Y., Pitla, S., Schnable, J. Robotic technologies for high-throughput plant phenotyping: Contemporary reviews and future perspectives. Front Plant Sci. 12, 611940 (2021).
- Berry, J. C., Fahlgren, N., Pokorny, A. A., Bart, R. S., Veley, K. M. An automated, high-throughput method for standardizing image color profiles to improve image-based plant phenotyping. PeerJ. 6, e5727 (2018).
- Tovar, J. C., et al. Raspberry Pi-powered imaging for plant phenotyping. Appl Plant Sci. 6, e1031 (2018).
- Jolles, J. W. Broad-scale applications of the Raspberry Pi: A review and guide for biologists. Methods Ecol Evol. 12 (5), 1562-1579 (2021).
- Watt, M., et al. Phenotyping: New windows into the plant for breeders. Annu Rev Plant Biol. 71 (1), 689-712 (2020).
- Xie, C., Yang, C. A review on plant high-throughput phenotyping traits using UAV-based sensors. Comput Electron Agric. 178, 105731 (2020).
- Czedik-Eysenberg, A., et al. The "PhenoBox", a flexible, automated, open-source plant phenotyping solution. New Phytol. 219 (4), 808-823 (2018).
- Sumner, J., Fahlgren, N., Murphy, K. M. PCVR: An R package and tutorials for guided statistical analysis of plant phenotyping data. Authorea Preprints. , (2023).
- Schnable, P. S., et al. The B73 maize genome: complexity, diversity, and dynamics. Science. 326 (5956), 1112-1115 (2009).
- Kirillov, A., et al. Segment Anything. , 3992-4003 (2023).
- Gabaldón-Leal, C., et al. Modelling the impact of heat stress on maize yield formation. Field Crops Res. 198 (3), 226-237 (2016).
- Intergovernmental Panel on Climate Change. . SYNTHESIS REPORT OF THE IPCC SIXTH ASSESSMENT REPORT (AR6). , (2023).
- Quiñones, A., et al. Optimized methods for applying and assessing heat, drought, and nutrient stress of maize seedlings in controlled environment experiments. Cold Spring Harb Protoc. , (2024).
- Murphy, K. M., et al. Maize abiotic stress treatments in controlled environments. Cold Spring Harb Protoc. , (2024).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。