Method Article
Imagem e análise para quantificar fenótipos de estresse abiótico de milho (Zea mays)
Neste Artigo
Resumo
Um método é apresentado para imagens de baixo custo de plantas para medir fenótipos, juntamente com as melhores práticas para captura de imagens e um pipeline de análise de imagens para quantificar as características das plantas. Esses métodos foram aplicados para medir os fenótipos do milho (Zea mays) sob condições de calor, seca e estresse abiótico combinado.
Resumo
A medição quantitativa dos fenótipos das plantas, ou características, é essencial para entender as respostas das plantas aos fatores ambientais, incluindo estresses bióticos e abióticos. No entanto, os métodos tradicionais de medição de plantas podem ser demorados, imprecisos e destrutivos, principalmente ao avaliar características como biomassa e altura individualmente. A análise baseada em imagem oferece uma oportunidade de reduzir a mão de obra, melhorar a exatidão e a precisão e permitir medições repetidas da mesma planta em vários pontos de tempo. Embora as instalações de fenotipagem de alto rendimento forneçam uma solução, elas geralmente são proibitivamente caras e limitadas em disponibilidade em todo o mundo. Um método é descrito para capturar imagens de plantas usando computadores de placa única de baixo custo e câmeras digitais dentro de uma configuração de estúdio fotográfico, acompanhado por um pipeline de análise de imagem baseado no pacote gratuito e de código aberto PlantCV. Esse método oferece um protocolo rápido e preciso para captura, rotulagem e transferência de imagens, juntamente com as melhores práticas para garantir a aquisição de imagens de alta qualidade. Por meio da análise de imagens, mais de 10 fenótipos foram quantificados para todas as imagens do experimento simultaneamente, incluindo características como tamanho, altura e cor da planta. Esses métodos foram usados para caracterizar a resposta da variedade B73 de Zea mays (milho) ao calor, à seca e às condições combinadas de calor e estresse hibótico da seca.
Introdução
Medir as características das plantas, ou fenótipos, é fundamental para responder a questões fundamentais e aplicadas na ciência das plantas. Por exemplo, um pesquisador que busca entender o impacto de uma mutação genética no tamanho da planta precisará medir quantitativamente a altura da planta e a área foliar1. Um pesquisador que aplica um estresse biótico (como infecção bacteriana, viral ou fúngica) ou um estresse abiótico (como seca, calor, frio ou esgotamento de nutrientes) precisará investigar o efeito do estresse na cor da planta, ou "verdura", para medir a clorose 2,3.
Embora a medição dessas características seja essencial para a biologia vegetal básica, bem como para o melhoramento de plantas aplicadas, as medições dessas características manualmente podem ser demoradas, imprecisas e imprecisas 4,5,6,7. Por exemplo, medir a altura das plantas inclui o uso manual de uma régua para cada planta, muitas vezes em condições ambientais quentes ou úmidas, e está sujeito a erros do ponto de vista da pessoa que está medindo, bem como qual ponto é considerado o "topo" da planta. Além disso, medições como biomassa e teor de pigmento são destrutivas, e a mesma planta não pode ser medida quanto a mudanças ao longo do tempo ou usada para várias amostragens porque deve ser destruída no processo de fazer essas medições 4,5,6,7. A natureza demorada e propensa a erros dessas medições dificulta o progresso na avaliação das plantas quanto às suas respostas fenotípicas e fisiológicas ao estresse.
A captura de imagens de plantas e a medição de características quantitativas da imagem fornecem soluções para esses problemas 4,5,6,7. A análise de imagens melhora a exatidão e a precisão, reduzindo o tempo que um pesquisador tem para fazer as medições, e a mesma planta pode ser fotografada várias vezes ou usada para outras amostragens devido à quantificação de características não destrutivas 4,5,6,7. Além disso, as imagens podem ser armazenadas e reanalisadas muito depois de um experimento ser concluído e as plantas serem descartadas 4,5,6,7. A análise de imagens pode ser concluída individualmente com ferramentas de edição de imagens de baixo rendimento, como ImageJ (Fiji)8, ou em maior rendimento usando visão computacional e aprendizado de máquina com plataformas como PlantCV9. O PlantCV é uma plataforma de análise de imagens gratuita e de código aberto que permite aos pesquisadores primeiro mascarar (ou separar) uma planta ou outro objeto do fundo de uma imagem e fazer medições de tamanho, morfologia, cor e muito mais dessa planta ou objeto9. Usando um fluxo de trabalho modular, um pesquisador primeiro cria um fluxo de trabalho flexível (ou pipeline) de diferentes módulos com uma imagem de amostra e, em seguida, submete as imagens restantes ao fluxo de trabalho de alto rendimento sem interagir com cada imagem individual9. Características quantitativas são produzidas e analisadas quanto a diferenças estatisticamente significativas entre os grupos. Os pipelines de Deep Learning podem ser desenvolvidos para análise de imagem eficiente e automatizada depois que imagens suficientes forem capturadas e rotuladas com medições de características ou metadados apropriados10.
A fenotipagem de "alto rendimento" pode aumentar a velocidade e reduzir o trabalho humano durante a coleta de dados e/ou durante a análise de dados 4,5,6,7. As instalações de fenotipagem de alto rendimento permitiram a automação da coleta de imagens em ambientes controlados, onde as plantas se movem em correias transportadoras ou por meio de robôs para uma estação de imagem e são automaticamente rotuladas com o nome (ou seja, rótulo com metadados) associado à planta 6,11,12,13,14 . Embora essas correias transportadoras ou instalações habilitadas por robôs permitam a captura e rotulagem de grandes quantidades de imagens sem intervenção humana, existem poucas no mundo e podem custar milhões de dólares para serem construídas, tornando-as inacessíveis para muitos pesquisadores. Para aplicar esses conceitos de imagem automatizada a um custo menor, o Raspberry Pi (aqui chamado de computadores de placa única, SBC) são computadores de baixo custo (menos de US$ 35 cada) que foram usados para capturar, nomear, armazenar e transferir imagens de plantas cultivadas em ambientes controlados. Essas configurações capturam imagens em intervalos automatizados e cronometrados usando uma matriz de até 72 Raspberry Pi 12,15,16. Embora mais econômicos do que os sistemas de correia transportadora que podem custar milhões de dólares, as câmaras de crescimento SBC descritas anteriormente são mais aplicáveis a uma visão de cima para baixo do cultivo de pequenas plantas e ainda requerem vários SBC15. Em ambientes de campo, pórticos, UAS (sistemas aéreos não tripulados), como drones, e satélites capturam imagens para análise de características da planta17,18. Tanto os sistemas de campo quanto as instalações de ambiente controlado de alto rendimento podem ser proibitivos em seu custo inicial, manutenção e conhecimento especializado necessários para construir e manter os sistemas, bem como analisar imagens17,18. Telefones celulares e câmeras digitais usados sozinhos para fotografia de plantas apresentam desafios demorados de transferência e nomeação de imagens com metadados, o que é essencial para a análise de imagens a jusante.
Os métodos descritos aqui apresentam uma solução de baixo custo para a geração de imagens de plantas para os desafios não enfrentados por outros sistemas. Uma configuração de fotografia, onde as plantas são transportadas individualmente para o estúdio, permite a medição eficiente de plantas de todos os tamanhos em vasos. Usando um SBC conectado a uma câmera digital, esse método primeiro fornece captura, rotulagem e transferência padronizadas de imagens. Usar um teclado ou um leitor de QR/código de barras para nomear imagens à medida que elas estão sendo capturadas contorna a atividade demorada e propensa a erros de renomear imagens com metadados depois de terem sido capturadas e armazenadas. A composição da imagem e as configurações recomendadas da câmera são importantes para possibilitar a análise de imagem downstream, e as diretrizes são detalhadas aqui. Por fim, um pipeline de análise de imagem no PlantCV é fornecido para extrair características quantitativas das imagens capturadas, como área foliar, altura, largura e cor da planta.
Em relação aos sistemas de correias transportadoras, esse método é de baixo custo e requer um único SBC. A configuração de fenotipagem descrita aqui foi construída para remover as restrições e custos de uma correia transportadora ou sistemas robóticos - qualquer tamanho de planta pode ser medido porque a configuração pode ser separada do espaço de crescimento, apenas um único SBC é necessário e funciona com vários tipos de câmeras para máxima flexibilidade e acessibilidade. Abordagens semelhantes à apresentada aqui incluem o PhenoBox ou o PhenoRig12,19. Comparado às medições manuais, esse método economiza tempo, reduz erros e não destrói as plantas 4,12,15,19.
Protocolo
Os detalhes sobre os reagentes e os equipamentos usados neste protocolo estão listados na Tabela de Materiais.
1. Preparação do estúdio fotográfico
- Pendure tecido preto de qualidade fotográfica em uma parede ou teto para imagens de vista lateral. Coloque o tecido pelo menos 0,5 m no chão para que a planta seja colocada em cima do tecido (Figura 1).
NOTA: O tecido deve ser pelo menos 100 cm mais alto e mais largo do que a planta a ser fotografada. - Coloque um cartão colorido no pano de fundo, apoiado em um objeto, como uma caixa, para que fique visível no quadro (Figura 1). Para plantas maiores que 30 cm de altura ou largura, use um cartão colorido com pelo menos 20 cm de largura. Para plantas menores que 30 cm de altura ou largura, use uma cartela colorida com 4-8 cm de largura.
- Conecte um cartão SD a um laptop ou computador desktop. Baixe o Raspberry Pi Imager na seção de software do site (https://www.raspberrypi.com/software/) e baixe o arquivo raspberrypi-image-capture.img.gz fornecido (https://github.com/danforthcenter/photo-maize-paper) para o laptop ou computador desktop.
- Inicie o Imager abrindo o arquivo de imagem (.exe) baixado. Clique no botão Escolher sistema operacional e selecione o botão Usar personalizado na parte inferior. Selecione o arquivo image-capture.img.gz.
- Em seguida, clique no botão Escolher armazenamento e selecione o cartão SD conectado ao computador. Clique no botão Escrever .
NOTA: Certifique-se de que o cartão SD correto foi selecionado, pois esse processo excluirá todos os dados armazenados no cartão.
- Em seguida, clique no botão Escolher armazenamento e selecione o cartão SD conectado ao computador. Clique no botão Escrever .
- Conecte a versão mais recente do SBC a um teclado usando um cabo USB, a um monitor externo usando um cabo HDMI e a um filtro de linha usando um cabo de alimentação com um interruptor. Conecte o monitor ao filtro de linha usando o cabo de alimentação fornecido com o monitor. Coloque o cartão SD no SBC. Consulte a Figura 1 para obter um esquema rotulado da configuração.
- Ligue o filtro de linha pressionando o interruptor no filtro, ligue o monitor usando o botão no monitor e, em seguida, ligue o SBC girando o interruptor no cabo de alimentação para "ligado".
- Conecte o SBC à Internet local usando WIFI ou um cabo Ethernet.
- Se as fotos forem transferidas diretamente para um servidor ou armazenamento em nuvem:
- Edite o arquivo "CAPTURE-PHOTO.py" no SBC para conter o caminho do arquivo para concluir a transferência.
- Edite o arquivo win-credentials com o caminho e a senha do servidor ou armazenamento em nuvem usando o seguinte comando no terminal: sudo nano /etc/win-credentials.
NOTA: Entre em contato com o administrador do sistema de Internet para garantir que a pasta tenha permissão para transferir arquivos.
- Conecte um leitor de código de barras sem fio ao SBC usando as instruções do fabricante fornecidas com o leitor de código de barras, usando um USB ou Bluetooth.
- Desligue o SBC clicando no botão Raspberry Pi no canto superior esquerdo da tela, clicando em Logout e, em seguida, clicando em Desligar para desligar o SBC com segurança. Após o desligamento, ligue o interruptor de alimentação SBC "on" o cabo de alimentação para "off" e o filtro de linha para "off".
2. Crescimento das plantas e tratamento do estresse
NOTA: Qualquer planta é adequada para este método de fenotipagem; Esses métodos de crescimento de plantas são aplicáveis aos resultados representativos apresentados aqui.
- Obtenha sementes de B73 ou outra variedade de milho em um centro de estoque.
- Prepare o meio do solo e as condições de crescimento:
- Encha vasos de 3,5 polegadas (0,7 L) com uma mistura de 1: 1 por volume de Turface MVP e Berger BM7-35 com 3 lb / jarda cúbica de fertilizante de liberação lenta 14-14-14 (NPK). Coloque os potes em uma bandeja com furos e regue bem com água de osmose reversa (RO).
- Defina uma câmara de crescimento para as seguintes condições ambientais: 30 °C dia/20 °C noite, 16 h de duração do dia, 60% de umidade e 500 mmol.m-2 s-1 de luz no nível do vaso durante o dia. Defina uma câmara de crescimento separada com as mesmas condições, exceto 38 °C dia / 28 °C noite para estresse térmico.
- Plante 20 sementes de milho, cada uma em um vaso de 3,5 polegadas, colocando ~ 2 cm de profundidade no solo e cobrindo-o com o solo deslocado. Regue bem com água RO e coloque na câmara de controle de crescimento. Regue diariamente com fertilizante líquido (misturado de acordo com as instruções do fabricante; consulte a Tabela de Materiais).
NOTA: O plantio de plantas extras necessárias para o experimento será responsável pela germinação deficiente e falta de uniformidade antes do início de uma aplicação de estresse. A taxa de germinação varia de acordo com a variedade. - Após 21 dias de crescimento, transplantar 16 das plantas mais uniformes (n = 4 plantas por tratamento) para vasos de 3,79 L preenchidos com a mesma mistura de solo para vasos, pré-umedecidos com água RO. Retorne as plantas para a câmara de crescimento de controle.
- No dia 25 (4 dias após o transplante), mova metade das plantas (n = 8) para a câmara de estresse térmico no momento em que as luzes acenderem na câmara. Pare de regar as plantas estressadas pela seca em ambas as câmaras (n = 4 por câmara).
NOTA: As plantas de estresse hídrico podem precisar ser regadas para que sobrevivam até o final do experimento; Aqui, as plantas de seca foram regadas no dia 31 e no dia 37 com água fertilizante. - No dia 38 (13 dias após o início do estresse), remova as plantas das câmaras de crescimento para obter imagens.
3. Imagem da planta
- Inicie o SBC (consulte a Figura 2):
- Ligue o filtro de linha usando o interruptor no filtro de linha.
- Ligue o monitor usando o botão no monitor.
NOTA: Se o SBC for ligado antes de o monitor ser ligado, ele não iniciará corretamente. - Ligue o interruptor do cabo de alimentação SBC.
NOTA: Isso ativará o SBC e ele inicializará no monitor; não há interruptor "on" no próprio SBC, apenas no cabo de alimentação.
- Coloque a câmera em um tripé ou mesa estável.
NOTA: Toda a planta e o cartão de cores devem estar em view na tela da câmera. A câmera deve ser perpendicular ao solo e ter uma visão direta (sem ângulo) do centro da planta. A câmera deve ser compatível com o pacote gphoto2 e uma lista pode ser encontrada aqui: http://www.gphoto.org/proj/libgphoto2/support.php. - Forneça energia à câmera por meio de um cabo de alimentação com a câmera ou baterias. Ligue a câmera usando o botão na câmera.
- Conecte a câmera à porta USB do SBC usando o cabo de transferência de dados fornecido com a câmera.
- Organize a planta na configuração de fotografia:
- Coloque um pedaço de fita adesiva de cor escura no tecido de fotografia onde o vaso será colocado para garantir que a colocação seja a mesma para cada planta. Certifique-se de que o cartão colorido esteja alinhado com o pedaço de fita.
NOTA: Coloque o cartão colorido a pelo menos 10 cm da borda da planta. Durante o controle de qualidade no fluxo de trabalho do PlantCV (abaixo), a imagem deve poder ser cortada para incluir apenas a planta e o vaso, mas não o cartão de cores, para garantir que os chips de cores não sejam incluídos na extração final de características. Consulte a Figura 2 para obter exemplos de imagens de alta e baixa qualidade. - Coloque o pote no pedaço de fita adesiva. Para plantas que crescem em um plano, como milho (Zea mays) e sorgo (Sorghum bicolor), fique de frente para o ângulo mais amplo da planta em direção à câmera. Para plantas que não crescem em um plano, capture ângulos de 0 e 90 graus da planta e calcule a média das medições quantitativas resultantes após a conclusão da análise da imagem.
- Coloque um pedaço de fita adesiva de cor escura no tecido de fotografia onde o vaso será colocado para garantir que a colocação seja a mesma para cada planta. Certifique-se de que o cartão colorido esteja alinhado com o pedaço de fita.
- Na tela da área de trabalho do SBC mostrada no monitor, abra o terminal clicando no botão do terminal na barra de menu superior esquerda.
- Se estiver salvando diretamente em um servidor ou armazenamento em nuvem, monte o armazenamento usando o seguinte comando editando o caminho de armazenamento em nuvem necessário (cloud-storage-path no exemplo abaixo) e a senha, digitando a linha de código na janela aberta do terminal e pressionando Enter no teclado. Se não for alterado em relação às configurações de fábrica, o ID do usuário (UID) será "pi".
NOTA:
sudo mount -t cifs -o credentials=/etc/win-credentials,uid=pi //caminho-de-armazenamento-de-nuvem /mnt/armazenamento-de-nuvem - Digite o seguinte código no terminal e pressione Enter no teclado. Uma nova janela aparecerá.
./RUN-PHOTOSTUDIO-SHELL.sh
NOTA: O arquivo RUN-PHOTOSTUDIO-SHELL.sh chama o arquivo CAPTURE-PHOTO.py, que é pré-carregado no pi se ele foi carregado usando o método acima. Como alternativa, esses scripts foram fornecidos no GitHub para serem colocados em um cartão SD Raspberry Pi existente. - Uma janela de interface gráfica do usuário será aberta. Digite o nome da imagem ou escaneie o código de barras ou código QR da planta usando um leitor de código de barras.
- Selecione uma opção para salvar a imagem localmente ou em um servidor/armazenamento em nuvem montado.
- Clique aqui para tirar uma foto! botão.
- Se o armazenamento local foi escolhido, abra a pasta FOTOS na área de trabalho para examinar a imagem. Se um servidor ou armazenamento em nuvem foi escolhido, abra a imagem nesse local.
- Se necessário, ajuste as configurações da câmera desconectando-a do SBC, ajustando usando as instruções do fabricante e conectando-a novamente ao SBC.
NOTA: Configurações como exposição, brilho, contraste, etc., podem precisar ser ajustadas dependendo da iluminação da sala e do objeto que está sendo fotografado. Um erro comum é expor demais as imagens. Para verificar a exposição de uma imagem de teste, prossiga com a imagem de teste por meio do fluxo de trabalho de análise de imagem do PlantCV, que inclui uma verificação de controle de qualidade para exposição. Ajuste as configurações da câmera conforme necessário para reduzir ou aumentar a exposição até que os histogramas sejam distribuídos adequadamente. - Depois que todas as imagens forem capturadas, transfira-as do SBC para um computador local ou armazenamento em nuvem usando o método preferido, como armazenamento USB, navegador da Internet ou transferência SSH.
4. Extração de características de imagens
- Baixe os seguintes arquivos para um computador ou servidor local: "photo-studio-SV-notebook.ipynb", "workflow.py" e "photo-studio-SV-config.json". Todos os arquivos estão disponíveis no GitHub (https://github.com/danforthcenter/photo-maize-paper).
- Instale o PlantCV no computador ou servidor local usando as etapas descritas (https://plantcv.readthedocs.io/en/latest/installation/).
- Abra o arquivo photo-studio-SV-notebook.ipynb usando o editor de código preferencial, como JupyterLab ou Visual Studio Code.
- Execute cada bloco de código, editando conforme necessário, dados os parâmetros descritos no bloco de código para obter uma máscara limpa apenas da planta na imagem.
- Quando estiver satisfeito com a análise da imagem de exemplo, abra o workflow.py de arquivo no editor de código preferido. Edite este arquivo com quaisquer parâmetros alterados em "photo-studio-SV-notebook.ipynb" e salve-o.
- Abra o photo-studio-SV-config.json de arquivos e edite os caminhos do arquivo para direcionar para uma pasta de imagens de entrada, uma pasta de saída desejada, etc., conforme indicado no arquivo.
- Abra um terminal e execute o código a seguir para ativar o PlantCV. Para fazer isso, digite o código conda activate plantcv no terminal e pressione Enter no teclado.
- Altere a seguinte linha de código para o caminho do arquivo "photo-studio-SV-config.json" (esta é a única alteração a ser feita no código abaixo).
- No terminal, digite o código editado e pressione Enter no teclado. Isso executará o fluxo de trabalho em todas as imagens capturadas no experimento.
NOTA:
plantcv-run-workflow --config /Usuários/NOME DE USUÁRIO/photo-studio-SV-config.json
- No terminal, digite o código editado e pressione Enter no teclado. Isso executará o fluxo de trabalho em todas as imagens capturadas no experimento.
- Inspecione as imagens de saída para determinar se o código era apropriado para todas as imagens.
NOTA: O contorno azul e rosa da planta deve estar ao redor das bordas da planta e não deve haver outros objetos na imagem. Se necessário, volte para o "photo-studio-SV-analysis.ipynb" e teste imagens problemáticas e edite o código, especificamente as etapas de mascaramento e redução de ruído, para obter os resultados desejados. Consulte a Figura 3 para obter exemplos de segmentação de imagem de alta, aceitável e baixa qualidade. - O arquivo de saída estará na forma de um arquivo .json. Volte para o terminal (certifique-se de que conda activate plantcv já foi executado), digite o seguinte código e pressione Enter no teclado para converter o arquivo em .csv para análise estatística downstream, alterando o caminho do arquivo para o do arquivo "results-photo-studio.json".
plantcv-utils json2csv -j /Usuários/NOME DE USUÁRIO/results-photo-studio.json -c /Usuários/NOME DE USUÁRIO/results-photo-studio.csv
NOTA: Para converter as medidas de pixels em uma unidade de medida padrão, como cm, utilize a conversão fornecida pela função find_color_card no fluxo de trabalho do PlantCV. - Realize análises estatísticas das características quantitativas de saída para determinar diferenças nos fenótipos de plantas devido a diferentes espécies de plantas, variedades, tratamentos e pontos de tempo.
NOTA: O pacote pcvr no R Studio foi formulado para analisar as saídas do PlantCV quanto à significância estatística e plotagem dos resultados20. Veja um resumo e uma representação visual das características coletadas na Figura 4.
Resultados
Para desenvolver esse método, plantas de milho B73 foram avaliadas quanto a fenótipos quantitativos sob condições de controle de temperaturas, calor, bem irrigadas e secas, bem como suas combinações, para um total de quatro tratamentos. B73 é uma variedade de milho endogâmica bem conhecida com uma riqueza de dados genéticos21.
A análise de imagens no PlantCV corrigiu com sucesso as cores das imagens, rotulou os resultados quantitativos com os metadados da planta e segmentou (em outras palavras, separou) a planta do fundo, como demonstrado na Figura 3. Uma análise malsucedida forneceria imagens de saída que não mostram o contorno azul ou a forma rosa ao redor da parte externa da planta, mas incluem ruído adicional ou peças ausentes da planta (Figura 3, exemplo de baixa qualidade). Um limite de canal duplo foi necessário para incluir o máximo possível da planta na máscara (veja estratégias alternativas de canal único no "photo-studio-SV-notebook.ipynb"). No entanto, algumas plantas, particularmente aquelas com caules de cor roxa, não tiveram uma segmentação perfeita, mas foram suficientes para análise a jusante (Figura 3).
A análise PlantCV forneceu 16 características quantitativas de valor único, resumidas na Figura 4. Uma característica de valor único fornece um valor por planta - por exemplo, a área da folha, a altura, a largura e a média circular de matiz. A área foliar foi considerada por meio da função "analyze.shape" (Figura 4) e considerada a planta inteira. A altura e a largura, no entanto, foram consideradas usando a "função analyze.bound_horizontal", com um limite desenhado na linha do solo da planta. Isso ocorre porque as plantas que caem na lateral do vaso terão uma altura maior considerando a planta como um objeto inteiro e, em vez disso, devem medir apenas a partir da base da planta onde encontra o solo. Além das características de valor único, o PlantCV gera características de vários valores, que são histogramas, particularmente dos valores de cor, que representam cada pixel da planta. A média circular de matiz é uma característica de valor único que é o valor médio de matiz para toda a planta.
Para determinar quais características são as mais interessantes, foi calculada a variância explicada pelo tratamento para cada característica, bem como a correlação entre as diferentes características usando a função "frem" no pacote pcvr R Studio20 (Figura 5A). A área foliar, altura, largura e média circular de matiz foram selecionadas para análise a jusante porque explicaram mais de 50% da variância devido ao tratamento e são medidas relevantes de crescimento da planta (área, altura e largura) e saúde (matiz). Imagens representativas da planta com a maior e a menor área foliar demonstram a variação nos fenótipos coletados neste experimento (Figura 5B, C, respectivamente). A planta com a maior área foliar também teve a maior tonalidade média e foi uma planta B73, bem regada e estressada pelo calor (Figura 5B). A menor área foliar teve a menor tonalidade média (Figura 5C) e foi uma planta B73, estressada pela seca e pelo calor.
O tratamento da água teve um efeito significativo na determinação da área foliar (F(1,13) = 226,5, p = 1,32 x 10-9), altura (F(1,13) = 21,1, p = 0,0005), largura (F(1,13) = 75,5, p = 8,92 x 10-7) e média circular de matiz (F(1,13) = 27,8, p = 0,0002), determinada usando um modelo de regressão linear para o efeito do tratamento e uma ANOVA de duas vias no R Studio (resultados completos na Tabela 1). O tratamento com temperatura teve um efeito significativo na altura (F (1,13) = 5,94, p = 0,03), mas não na área foliar, largura ou média circular da tonalidade (p > 0,05, Tabela 1). A interação entre temperatura e estado hídrico não foi fator significativo em nenhuma das características medidas (p > 0,05, Tabela 1).
Em seguida, os tratamentos individuais foram comparados com o controle e entre si por meio de regressão linear e teste post-hoc (médias marginais estimadas) com correção de Sidak para comparações múltiplas (Figura 6). A área foliar, a altura da planta, a largura da planta e a média circular da tonalidade foram significativamente reduzidas em plantas estressadas pela seca em comparação com bem regadas em ambas as condições de temperatura (p < 0,05, Figura 6). O estresse térmico quando bem regado apenas reduziu a altura da planta (p < 0,05, Figura 6), mas não reduziu significativamente a área foliar, a largura da planta ou a média circular da tonalidade. Essa diferença de matiz pode ser investigada avaliando a tonalidade do traço de cor de vários valores, plotada como um histograma na Figura 7, em vez de apenas uma média. A redução na tonalidade média devido à seca foi devido a uma mudança de pixels verdes para amarelos, conhecida como clorose (Figura 7). Embora o calor não tenha mostrado uma diferença significativa na tonalidade média, ele exibiu uma redução e aumento devido ao amarelecimento (clorose) e ao escurecimento da cor verde (Figura 7).
Após a imagem, as plantas foram cortadas na junção raiz-parte aérea e o peso do tecido acima do solo foi registrado. As raízes foram lavadas e secas suavemente com uma toalha, depois pesadas, para calcular a biomassa total da planta (também conhecida como peso) para comparação com o método de fenotipagem baseado em imagem descrito. A área foliar medida por meio de análise de imagem correlacionou-se fortemente com a biomassa vegetal (R2 = 0,84, Figura 8).

Figura 1: Imagem da configuração do estúdio fotográfico com componentes rotulados para coletar imagens de plantas. (A) Tecido de grau fotográfico, (B) cartão colorido, (C) linha de fita para colocar vasos uniformemente, (D) câmera digital, (E) computador de placa única, (F) monitor exibindo o processo de captura de imagem. Clique aqui para ver uma versão maior desta figura.

Figura 2: Visão geral do fluxo de trabalho de imagem. Uma imagem representativa de "alta qualidade" e "baixa qualidade" é fornecida. Clique aqui para ver uma versão maior desta figura.
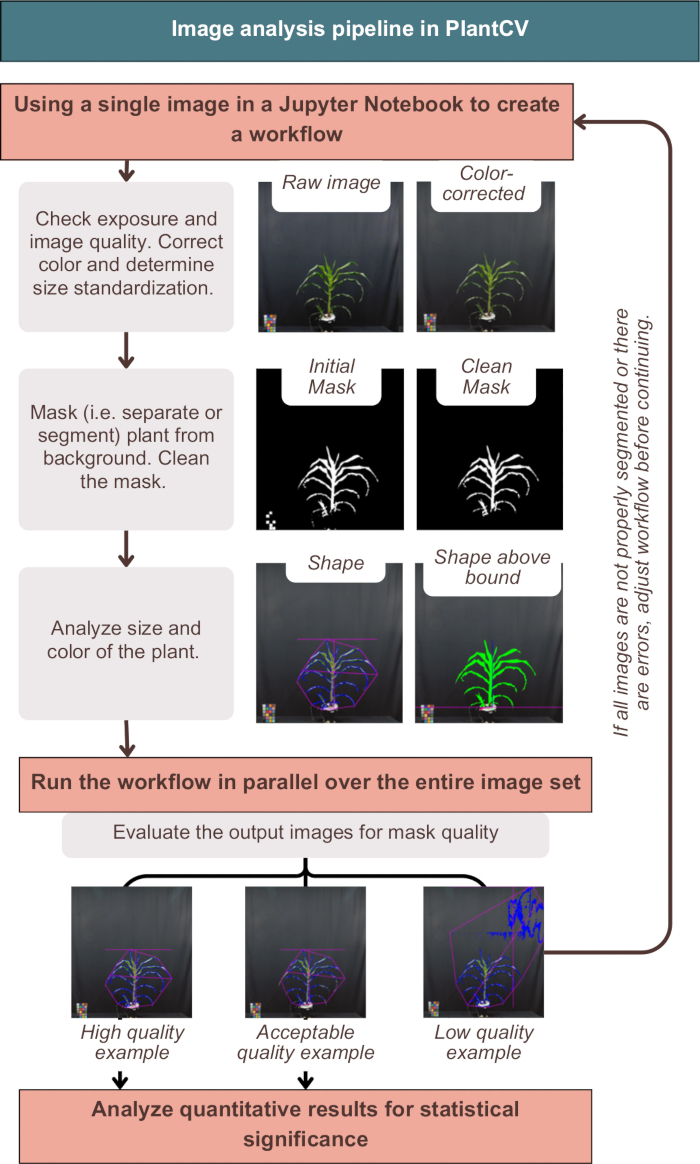
Figura 3: Visão geral do fluxo de trabalho de análise de imagens no PlantCV. Imagens representativas de segmentação de qualidade "alta", "aceitável" e "baixa" são apresentadas; As características do PlantCV são representadas usando um contorno rosa da forma da planta inteira e um contorno azul da área da planta. Clique aqui para ver uma versão maior desta figura.
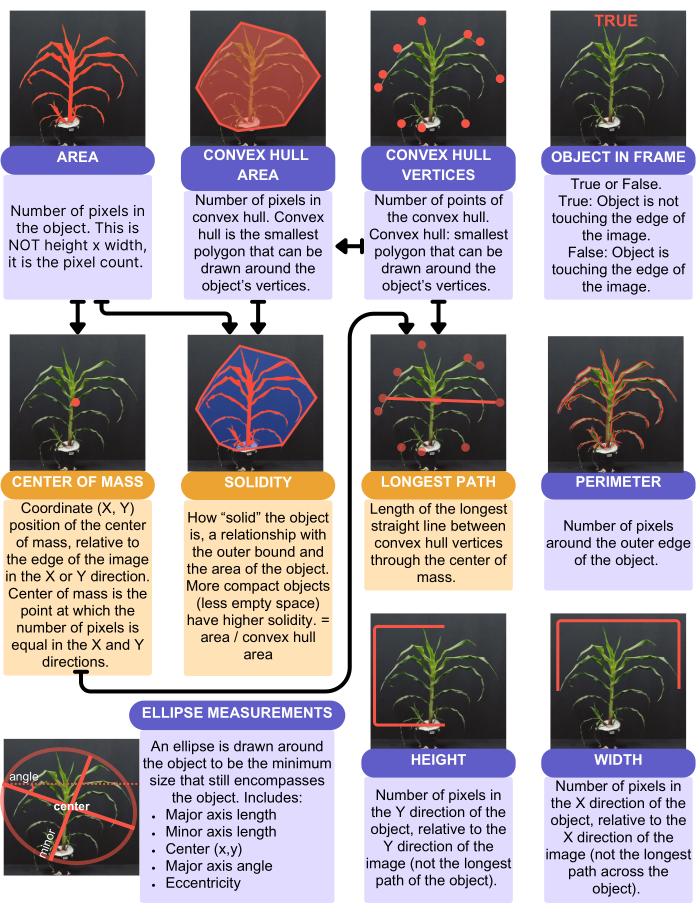
Figura 4: Resumo e representação visual dos traços de forma capturados da análise. Função de forma em PlantCV. As caixas roxas representam características que não utilizam outra característica em seus cálculos. As caixas laranja representam características que utilizam outras características em seus cálculos; As setas indicam quais características são usadas nesses cálculos. Clique aqui para ver uma versão maior desta figura.
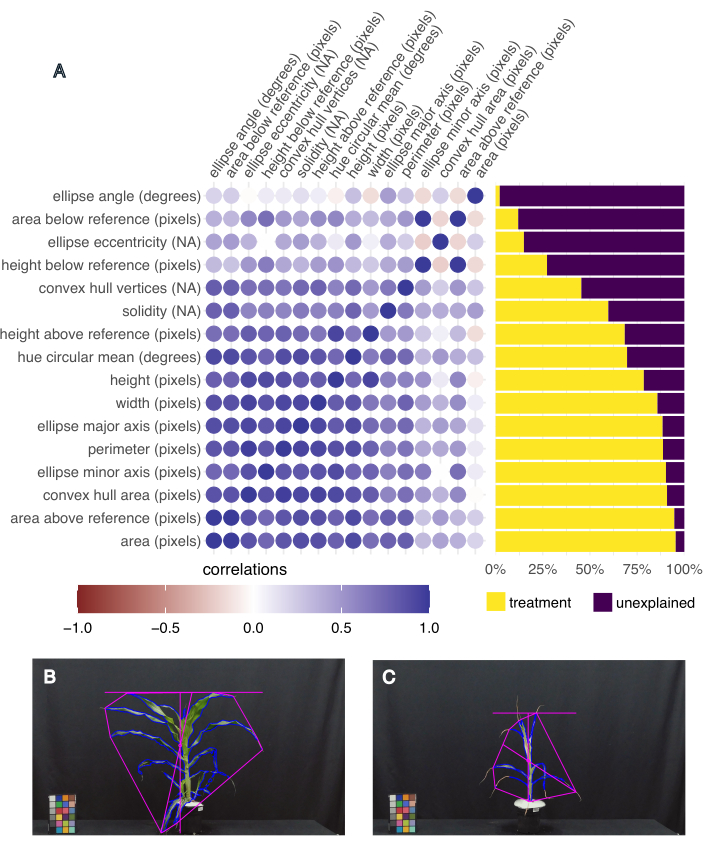
Figura 5: A fenotipagem baseada em imagem captura a variância fenotípica e as características correlacionadas. (A) Variância de características explicadas pelo tratamento (direita) e correlação de características entre si (esquerda). Cada característica é rotulada, seguida por sua unidade de medida; "NA" significa que é uma medida sem unidade. (B) Imagem da planta que apresentou a maior área foliar e maior tonalidade média circular (graus). (C) Imagem da planta que apresentou a menor área foliar e menor média circular de matiz (graus). Clique aqui para ver uma versão maior desta figura.
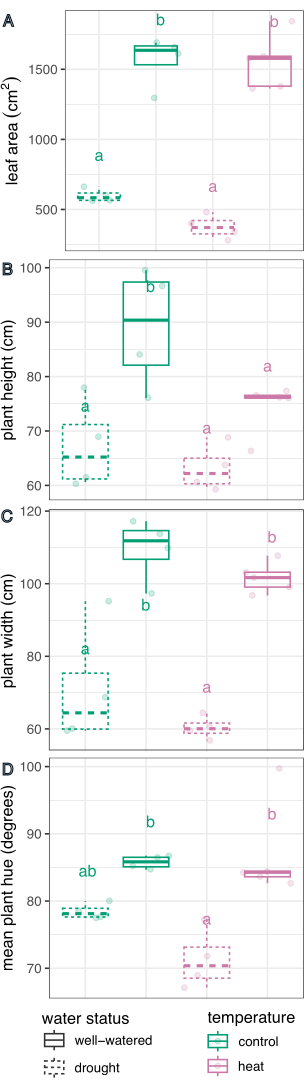
Figura 6: Comparação boxplot dos efeitos do tratamento nos fenótipos das plantas. A área foliar (A), a altura (B), a largura (C) e a média circular de matiz (D) são comparadas quanto ao efeito da seca, do calor e das tensões combinadas. n = 4. As letras representam diferenças estatisticamente significativas entre os tratamentos (p < 0,05); Os tratamentos foram comparados por meio de regressão linear e teste post-hoc (médias marginais estimadas) com correção de Sidak para comparações múltiplas. Clique aqui para ver uma versão maior desta figura.
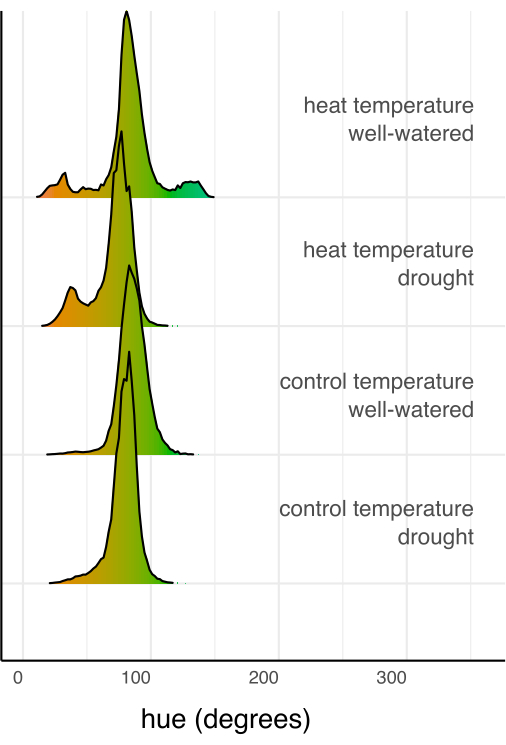
Figura 7: Histogramas de cor (matiz) somados de todos os pixels de cada combinação de tratamento, normalizados pelo número total de pixels na combinação de tratamento. Os histogramas são coloridos pelo valor de cor para o grau de matiz. Clique aqui para ver uma versão maior desta figura.
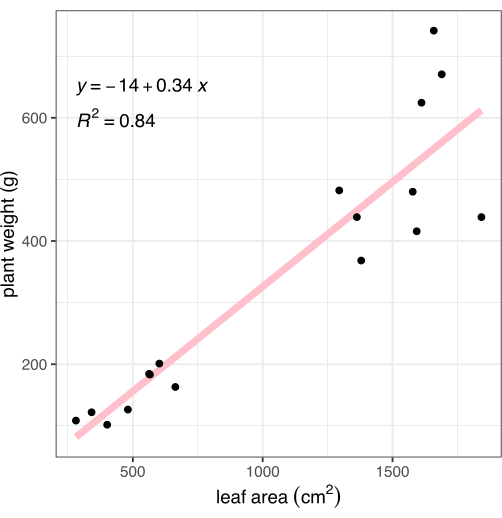
Figura 8: Correlação da área foliar medida a partir de imagens com a biomassa de toda a planta. Cada ponto preto representa uma planta. A linha rosa representa a correlação linear entre a área foliar (X) e a biomassa (Y). Clique aqui para ver uma versão maior desta figura.
| Traço | Fator | graus de liberdade | Valor F | Valor de p |
| área foliar (cm^2) | Temperatura | 1 | 0.5372 | 0.4766 |
| área foliar (cm^2) | Água | 1 | 226.5318 | 1.32E-09 |
| área foliar (cm^2) | Temperatura: Água | 1 | 2.1526 | 0.1661 |
| área foliar (cm^2) | Resíduos | 13 | ||
| Altura (cm) | Temperatura | 1 | 5.9426 | 0.0298935 |
| Altura (cm) | Água | 1 | 21.107 | 0.0005029 |
| Altura (cm) | Temperatura: Água | 1 | 2.1752 | 0.1640545 |
| Altura (cm) | Resíduos | 13 | ||
| largura (cm) | Temperatura | 1 | 2.2495 | 0.1575 |
| largura (cm) | Água | 1 | 75.5474 | 8.92E-07 |
| largura (cm) | Temperatura: Água | 1 | 0.0839 | 0.7766 |
| largura (cm) | Resíduos | 13 | ||
| Matiz Circular Médio (graus) | Temperatura | 1 | 0.9154 | 0.3561432 |
| Matiz Circular Médio (graus) | Água | 1 | 27.7988 | 0.0001509 |
| Matiz Circular Médio (graus) | Temperatura: Água | 1 | 3.4792 | 0.0848724 |
| Matiz Circular Médio (graus) | Resíduos | 13 |
Tabela 1: Resultados do modelo de regressão linear e análises ANOVA bidirecionais examinando os efeitos do tratamento da água, tratamento da temperatura e sua interação nas características das plantas.
Discussão
A fenotipagem baseada em imagem fornece medições precisas, rápidas e quantitativas dos fenótipos de plantas 4,5,6,7. Este método é significativamente mais rápido, mais preciso, mais preciso e fornece um número maior de medições do que manualmente usando uma régua para registrar a altura da planta ou medidas qualitativas das características da cor da planta que podem estar relacionadas à saúde 4,5,6,7. A área foliar foi vista aqui, e em pesquisas anteriores10, fortemente correlacionada à biomassa vegetal (Figura 8), sugerindo que essa medida poderia substituir de forma confiável o processo demorado, destrutivo e propenso a erros de pesagem manual da biomassa de toda a planta. Embora uma câmera sozinha pudesse ser usada para capturar as imagens, a adição do SBC de baixo custo aumentou o rendimento por meio da automação da coleta de imagens e rotulagem de imagens com os metadados necessários15. Este método é adequado para uma a ~ 300 plantas que são fotografadas uma vez por dia (ou com menos frequência). Para um número maior de plantas em um experimento, um sistema de imagem baseado em transportador, pórtico suspenso ou habilitado para robô é mais adequado, pois o tempo para mover as plantas individualmente para geração de imagens pode ser maior do que um dia útil11,13. Para pontos de tempo mais frequentes (por exemplo, uma imagem a cada hora em vez de uma vez por dia ou com menos frequência), as configurações de câmera dentro de uma câmara de crescimento são mais adequadas porque capturam imagens automaticamente, mas são limitadas pelo tamanho das plantas e precisam de SBC15 adicional.
Embora o PlantCV seja apresentado como a opção de análise de imagem aqui, existem outros softwares. O ImageJ (Fiji) é um pacote de software gratuito para análise de imagens que também pode ser usado para imagens de plantas por meio de uma interface gráfica do usuário (GUI)8; no entanto, não é particularmente desenvolvido para análise de imagens de plantas inteiras, é menos adaptável à computação de alto rendimento e é menos facilmente repetível do que pipelines já em funcionamento, como os do PlantCV, que podem ser facilmente aplicados a novas imagens9. Os métodos de Deep Learning (como BioDock, consulte Tabela de Materiais) e Segment Anything22) são atraentes por sua capacidade de segmentar facilmente plantas a partir do plano de fundo e podem ser usados para as tarefas de segmentação descritas aqui. No entanto, esses modelos de Deep Learning não fornecem as medições de fenótipo de planta descritas aqui10, e para fazer essas medições mais complexas de fenótipos quantitativos pode exigir uma grande quantidade de imagens de treinamento (muitas vezes maiores que 10.000) e poder de computação além de um laptop ou computador de mesa básico10. Esses modelos também requerem memória e poder de processamento significativos do computador, que não estão disponíveis para muitos pesquisadores10.
A etapa mais crítica do protocolo é organizar a configuração da imagem da planta e garantir que não haja objetos (como outras plantas, um cartão colorido ou restos de plantas) perto da planta de interesse14. Se as imagens não forem definidas adequadamente ou um cartão colorido não for usado, elas serão impossíveis de analisar posteriormente porque não há mecanismo de padronização. Embora nenhuma etapa de calibração seja necessária neste protocolo, a correção de cores e a padronização de tamanho durante o processamento são essenciais. Certifique-se sempre de que todo o cartão colorido esteja no quadro, mas não sobreposto à planta (Figura 2). Outra armadilha comum é a iluminação irregular, que pode causar sombras ou fotos brilhantes; recomendamos o uso de luz brilhante e difusa no teto e a cobertura das janelas da sala para evitar iluminação irregular (Figura 2). Lâmpadas adicionais podem ser adicionadas em um ângulo frontal à planta se as luzes do teto estiverem causando sombras. O tipo de lâmpada não é uma preocupação, uma vez que a cor será corrigida na imagem usando um cartão de cores. Imagens desfocadas não são aceitáveis para análise - use o foco automático na câmera ou as instruções do fabricante para foco manual para garantir que as imagens sejam nítidas.
Da mesma forma, garantir que as imagens não sejam superexpostas (um erro comum) permite uma análise apropriada (Figura 2); As imagens superexpostas não podem ser corrigidas em uma correção de cores durante a análise da imagem porque os valores de pixel estão além da faixa de detecção da câmera com as configurações atuais14. Para evitar a superexposição, avalie os histogramas de cores ou valores RGB para uma imagem de teste usando o fluxo de trabalho PlantCV descrito aqui (Figura 3) e reduza a configuração de brilho na câmera se o teste de exposição determinar que a imagem está superexposta. Os valores RGB (e, portanto, os histogramas) não devem ter valores em 0 ou 255, que é o intervalo de valores porque é possível que esses valores estejam acima ou abaixo do intervalo de detecção e só possam registrar o mínimo ou o máximo em vez do valor verdadeiro. Uma imagem de teste deve sempre ser avaliada usando esses métodos para superexposição (Figura 2 e Figura 3); sugerimos utilizar o controle de qualidade de exposição PlantCV fornecido nesta publicação como uma verificação, que determina que uma imagem de qualidade tenha menos de 5% de pixels em 0 ou 255 para cada cor. Como regra geral, câmeras e software de análise fornecem histogramas, que devem ter caudas de histograma iguais e completas; Os valores cortados abaixo de 0 (subexposto) ou acima de 255 (superexposto) devem ser retomados com as configurações da câmera ajustadas. Sugerimos escolher um local com iluminação artificial brilhante e uniforme, sem janelas para iluminação, pois as janelas podem ser muito claras, irregulares e alterar frequentemente a exposição à luz do espaço. As imagens devem aparecer uniformemente iluminadas e os quadrados cinza de um cartão colorido não devem aparecer brancos, o que sugere superexposição.
A solução de problemas pode ser necessária para o código de captura de imagem, dependendo do IP e das configurações de segurança da instituição do usuário. Para solucionar problemas, entre em contato com o departamento de TI para problemas de conectividade com a Internet e transferência de arquivos. Um erro comum é usar o cartão SD de tamanho errado para o SBC ou ligar o SBC antes que o monitor conectado seja ligado. Para solucionar o problema, siga o protocolo acima com precisão e recarregue o software SBC. Os fóruns online são um excelente recurso para solucionar problemas e encontrar soluções para a câmera específica de sua escolha ao usar o gphoto2. Para solucionar problemas de análise de imagem do PlantCV e usar o controle de versão mais atualizado, utilize a página de documentação (https://plantcv.readthedocs.io/en/latest/) ou publique um problema no GitHub (https://github.com/danforthcenter/plantcv/issues). Em particular, se o método de limiar neste protocolo não separar suficientemente a planta do plano de fundo, utilize métodos alternativos de limiar fornecidos em "photo-studio-SV-notebook.ipynb" ou na página de documentação.
Este método pode ser aplicado a qualquer espécie de planta e tratamento que possa ser transportado para a configuração de fototografia montada descrita. Por exemplo, não é aplicável a plantas que crescem em um campo, pois não podem ser transportadas para a configuração de fotografia sem desenterrar a planta. Nessa situação, as plantas poderiam ser cortadas e posteriormente fotografadas usando o protocolo, embora a medição fosse destrutiva. Várias plantas podem ser capturadas em uma única imagem e analisadas, mas o usuário deve tomar cuidado para evitar que as plantas se toquem (Figura 2), e as plantas são colocadas no mesmo local no pano de fundo todas as vezes para garantir que o fluxo de trabalho possa ser executado automaticamente em todo o conjunto de imagens. Se uma visão de cima para baixo for necessária para plantas como Arabidopsis thaliana, use um tripé para mover a câmera para o topo das plantas colocadas no tecido de grau fotográfico; Os demais métodos de captura e análise de imagens permanecem os mesmos.
Como demonstrado aqui com o milho, esse método é importante tanto para o melhoramento de plantas quanto para insights fundamentais sobre como as plantas toleram estresses abióticos, como calor e seca. O estresse térmico e hídrico combinados contribuem para a perda de rendimento de 15% a 20% no milho anualmente em todo o mundo23. As perdas de rendimento só devem aumentar com as mudanças climáticas globais, já que as temperaturas devem aumentar de 1,4 a 4,4 ° C até o ano 210024. Avaliar a diversidade do milho quanto à tolerância ao estresse, bem como determinar os mecanismos fenotípicos, fisiológicos e moleculares da tolerância ao estresse abiótico, são objetivos críticos da pesquisa para a segurança alimentar global25. Para abordar essas questões, pesquisadores de todos os campos adjacentes - fisiologistas, geneticistas, melhoristas, biólogos moleculares, etc. - devem ser capazes de avaliar de forma rápida, eficaz e muitas vezes não destrutiva os fenótipos de plantas de milho sob estresse abiótico25. O método e os resultados de exemplo mostrados aqui atendem a essa necessidade e são uma ferramenta importante para os pesquisadores enfrentarem os desafios agrícolas do mundo real.
Os fenótipos observados aqui foram hipotetizados, dada uma riqueza de pesquisas anteriores - o estresse hídrico reduz o tamanho e o verde da planta (medido pela média circular da tonalidade)26 (Figura 6, Figura 7 e Tabela 1). O fenótipo do estresse térmico com rega inesperada não reduziu significativamente a área foliar, embora com mais repetições, essa relação possa ser melhor avaliada estatisticamente, e a altura foi reduzida conforme o esperado (Figura 6). Embora a média circular de matiz não tenha sido alterada devido ao calor, o perfil de cores foi de fato alterado para incluir pixels mais amarelos e verdes mais escuros, mostrando que a avaliação apenas da tonalidade média é insuficiente, e o histograma de cor de traço de múltiplos valores deve ser considerado (Figura 7). Essas mudanças fenotípicas descrevem a resposta de uma planta ao ambiente, e técnicas adicionais, como medições de trocas gasosas, imagens microscópicas e muito mais, podem ser empregadas para avaliar as mudanças fisiológicas que ocorrem em resposta ao estresse. Este resultado demonstra o poder de medições precisas e quantitativas que, de outra forma, seriam perdidas usando medidas manuais ou qualitativas de fitossanidade. Esses insights são essenciais para o melhoramento e engenharia de culturas resistentes às mudanças climáticas, o que reduz significativamente o rendimento global das safras e deve piorar nos próximos anos.
Divulgações
Nenhum.
Agradecimentos
Este trabalho foi conduzido no Phenotyping Core Facility, Donald Danforth Plant Science Center (RRID: SCR_019049), que forneceu apoio financeiro para K.M.M. e J.G.D. Este trabalho foi apoiado pelo Donald Danforth Plant Science Center Enterprise Rent-A-Car Research Institute Grant. DS foi financiado pelo Taylor Geospatial Institute Block Grant e um Centro de Administração de Desenvolvimento Econômico dos EUA para AgTech e Applied Location Science and Technology (CATALST) grant (ED21HDQ0240072). Agradecemos a Kevin Reilly e Kris Haines (Integrated Plant Growth Facility, Donald Danforth Plant Science Center, RRID: SCR_024902) por sua experiência e assistência no crescimento de plantas. Agradecemos a Noah Fahlgren, Malia Gehan e Haley Schuhl por seus conselhos sobre o PlantCV.
Materiais
| Name | Company | Catalog Number | Comments |
| 15W USB-C Power Supply | CanaKit | DCAR-RSP-3A5-C | |
| 25604 UC-E6 USB Cable | Nikon | 18208256044 | |
| 3.5 inch pots | Hummert International | 13006000 | |
| Berger BM7 35% Bark HP | Hummert International | 10121500 | |
| Biodock 2024 | AI Software Platform | www.biodock.ai. | |
| Code editing software | Jupyter | https://jupyter.org/install | |
| Code editing software | Visual Studio Code | https://code.visualstudio.com/download | |
| Color Card ColorChecker Classic | Calibrite | CCC | |
| Color Card ColorChecker Passport | Calibrite | CCPP2 | |
| Conviron Growth Chamber | Conviron | BDW80 model | |
| Dell 22 Monitor | Dell | SE2222H | |
| EH-67 AC Adapter | Nikon | AZ185 | |
| Image analysis software | PlantCV | https://plantcv.readthedocs.io/en/latest/installation/ | |
| Jack's Water Soluble Fertilizer 15-5-15 | Hummert International | 7590200 | liquid fertilizer |
| Keyboard and Mouse | Logitech | 1440638 | MK270 wireless keyboard and mouse |
| Maize seed | USDA-ARS Germplasm Resources Information Network (GRIN) | B73 | https://www.ars-grin.gov/ |
| Micro HDMI to HDMI cable | Amazon | B07KSDB25X | |
| Nikon Coolpix B500 camera | Nikon | ||
| Nikon D7200 camera | Nikon | ||
| Osmocote 14-14-14 | Hummert International | 7630000 | |
| Photography-grade fabric | Yayoya | Polyester Fabric Black Photo Backdrop for Photo Video Studio Televison | |
| Raspberry Pi 4 Case with heatsink | iUniker | B07ZVKN262 | |
| Raspberry Pi 4 Model B Rev 1.1 | Raspberry Pi Foundation | RP4B4GB_BP | |
| Raspberry Pi 4 PiSwitch (USB-C) | CanaKit | RSP-PISWITCH-USBC | |
| Raspberry Pi software | Raspberry Pi Imager | https://www.raspberrypi.com/software/ | |
| Statistical analysis software | Rstudio | https://posit.co/downloads/ | |
| Tripod | K&F Concept | T255A3+BH-28L | |
| Turface MVP | Hummert International | 10240000 | |
| Wireless Barcode Scanner | Netumscan | B08X4NMX2M | 1D and 2D barcode scanner |
Referências
- Xiao, Q., Bai, X., Zhang, C., He, Y. Advanced high-throughput plant phenotyping techniques for genome-wide association studies: A review. J Adv Res. 35, 215-230 (2022).
- Mutka, A. M., et al. image-based phenotyping methods provide insight into spatial and temporal dimensions of plant disease. Plant Physiol. 172 (2), 650-660 (2016).
- Mutka, A. M., Bart, R. S. Image-based phenotyping of plant disease symptoms. Front Plant Sci. 5, 734 (2014).
- Fahlgren, N., Gehan, M. A., Baxter, I. Lights, camera, action: High-throughput plant phenotyping is ready for a close-up. Curr Opin Plant Biol. 24 (2), 93-99 (2015).
- Das Choudhury, S., Samal, A., Awada, T. Leveraging image analysis for high-throughput plant phenotyping. Front Plant Sci. 10, 508 (2019).
- Yang, W., et al. Crop phenomics and high-throughput phenotyping: Past decades, current challenges, and future perspectives. Mol Plant. 13 (2), 187-214 (2020).
- Omari, M. K., et al. Digital image-based plant phenotyping: A review. Korean J Agric Sci. 47 (1), 119-130 (2020).
- Schindelin, J., et al. Fiji: An open-source platform for biolFiogical-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Gehan, M. A., et al. PlantCV v2: Image analysis software for high-throughput plant phenotyping. PeerJ. 5, e4088 (2017).
- Murphy, K. M., Ludwig, E., Gutierrez, J., Gehan, M. A. Deep learning in image-based plant phenotyping. Annu Rev Plant Biol. 75 (1), 771-795 (2024).
- Fahlgren, N., et al. A versatile phenotyping system and analytics platform reveals diverse temporal responses to water availability in Setaria. Mol Plant. 8 (3), 1520-1535 (2015).
- Yu, L., et al. Development of a mobile, high-throughput, and low-cost image-based plant growth phenotyping system. bioRxiv. , (2023).
- Atefi, A., Ge, Y., Pitla, S., Schnable, J. Robotic technologies for high-throughput plant phenotyping: Contemporary reviews and future perspectives. Front Plant Sci. 12, 611940 (2021).
- Berry, J. C., Fahlgren, N., Pokorny, A. A., Bart, R. S., Veley, K. M. An automated, high-throughput method for standardizing image color profiles to improve image-based plant phenotyping. PeerJ. 6, e5727 (2018).
- Tovar, J. C., et al. Raspberry Pi-powered imaging for plant phenotyping. Appl Plant Sci. 6, e1031 (2018).
- Jolles, J. W. Broad-scale applications of the Raspberry Pi: A review and guide for biologists. Methods Ecol Evol. 12 (5), 1562-1579 (2021).
- Watt, M., et al. Phenotyping: New windows into the plant for breeders. Annu Rev Plant Biol. 71 (1), 689-712 (2020).
- Xie, C., Yang, C. A review on plant high-throughput phenotyping traits using UAV-based sensors. Comput Electron Agric. 178, 105731 (2020).
- Czedik-Eysenberg, A., et al. The "PhenoBox", a flexible, automated, open-source plant phenotyping solution. New Phytol. 219 (4), 808-823 (2018).
- Sumner, J., Fahlgren, N., Murphy, K. M. PCVR: An R package and tutorials for guided statistical analysis of plant phenotyping data. Authorea Preprints. , (2023).
- Schnable, P. S., et al. The B73 maize genome: complexity, diversity, and dynamics. Science. 326 (5956), 1112-1115 (2009).
- Kirillov, A., et al. Segment Anything. , 3992-4003 (2023).
- Gabaldón-Leal, C., et al. Modelling the impact of heat stress on maize yield formation. Field Crops Res. 198 (3), 226-237 (2016).
- Intergovernmental Panel on Climate Change. . SYNTHESIS REPORT OF THE IPCC SIXTH ASSESSMENT REPORT (AR6). , (2023).
- Quiñones, A., et al. Optimized methods for applying and assessing heat, drought, and nutrient stress of maize seedlings in controlled environment experiments. Cold Spring Harb Protoc. , (2024).
- Murphy, K. M., et al. Maize abiotic stress treatments in controlled environments. Cold Spring Harb Protoc. , (2024).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados